È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Monitoraggio temporale del ciclo cellulare progressione citometria a flusso, senza la necessità di sincronizzazione
In questo articolo
Riepilogo
Questo protocollo descrive l'uso di bromodeossiuridina (BrdU) uptake per consentire il monitoraggio temporale di cellule che erano in fase S in un determinato momento. L'aggiunta di coloranti DNA e l'etichettatura degli anticorpi facilita l'analisi dettagliata del destino delle cellule in fase S in tempi successivi.
Abstract
This protocol describes a method to permit the tracking of cells through the cell cycle without requiring the cells to be synchronized. Achieving cell synchronization can be difficult for many cell systems. Standard practice is to block cell cycle progression at a specific stage and then release the accumulated cells producing a wave of cells progressing through the cycle in unison. However, some cell types find this block toxic resulting in abnormal cell cycling, or even mass death. Bromodeoxyuridine (BrdU) uptake can be used to track the cell cycle stage of individual cells. Cells incorporate this synthetic thymidine analog, while synthesizing new DNA during S phase. By providing BrdU for a brief period it is possible to mark a pool of cells that were in S phase while the BrdU was present. These cells can then be tracked through the remainder of the cell cycle and into the next round of replication, permitting the duration of the cell cycle phases to be determined without the need to induce a potentially toxic cell cycle block. It is also possible to determine and correlate the expression of both internal and external proteins during subsequent stages of the cell cycle. These can be used to further refine the assignment of cell cycle stage or assess effects on other cellular functions such as checkpoint activation or cell death.
Introduzione
La valutazione delle caratteristiche del ciclo cellulare e cambiamenti che si verificano nelle cellule durante la progressione del ciclo cellulare è fondamentale per capire molti aspetti della biologia, in particolare biologia del cancro. Molti agenti di sviluppo per il trattamento di tumori maligni hanno effetti profondi sulla progressione del ciclo cellulare o indurre la morte cellulare per ciclo cellulare dipendente-meccanismi. Per studiare la dinamica del ciclo cellulare o cellule in una particolare fase del ciclo cellulare, si è soliti sincronizzare cellule. Tuttavia metodi di sincronizzazione possono avere effetti negativi sulle cellule in fase di studio, potenzialmente confondere i risultati ottenuti. 1 analisi Recentemente l'uso di proteine con tag fluorescenza che sono presenti solo in determinati fasi del ciclo di cellule hanno permesso di progressione del ciclo cellulare in cellule singole nel tempo 2, tuttavia le cellule da studiare bisogno di essere manipolato geneticamente per esprimere queste proteine taggati, limitando il loro utilizzo ai sistemi in cui questo può essere lettoily raggiunto.
Il ciclo cellulare è costituito da due fasi attive: la fase di sintesi (S), in cui il DNA si replica e la mitosi (M), dove la divisione cellulare si svolge. Queste fasi sono separate da tre fasi gap, G 0, G 1 e G 2. G 0 o quiescenza, è una fase di riposo in cui la cellula ha lasciato il ciclo, G 1 è dove le cellule aumentano di dimensione prima replicazione del DNA e G 2 dove la crescita cellulare continua tra il completamento della replicazione del DNA ma prima divisione cellulare. La progressione attraverso il ciclo cellulare è controllato da una serie di punti di controllo. Il G 1 checkpoint si attiva quando le condizioni ambientali non sono favorevoli della sintesi del DNA e impedisce l'entrata in fase S. La fase di checkpoint o ritardo intra-S possono essere attivati da danni al DNA che può provocare forche replica stallo. Durante G 2 la fedeltà del DNA replicato è confermata e se il danno è rilevato poi il G 2 checkpoint è attivata permettendo la riparazione del DNA prima di divisione cellulare. Un punto di controllo finale durante la mitosi assicura che cromatidi sono state allineate correttamente la piastra mitotico in modo che la divisione cellulare può essere completata con successo. 3 L'attivazione di questi punti di controllo è comunemente usato per sincronizzare popolazioni cellulari. Checkpoint del ciclo cellulare possono essere attivati da una serie di fattori, ma in biologia del cancro il più comune è il rilevamento dei danni al DNA. La risposta al danno al DNA è iniziata dalla PI3-chinasi-like chinasi telangiectasia atassia e RAD3 connessi (ATR) e atassia telangiectasia mutato (ATM) che attivano chinasi effettori a valle Chk1 e CHK2 rispettivamente. 3 una serie di eventi attiva Chk1 compresi stallo forche replica, legami crociati DNA, e danni radiazione ultravioletta mentre Chk2 è attivata principalmente da rotture del doppio filamento.
Il metodo usuale per studiare l'effetto di mutate condizioni della lunghezza del ciclo cellulare is per sincronizzare le cellule in una particolare fase del ciclo cellulare. 1 Ciò può essere ottenuto mediante diversi metodi. Le celle possono essere fisicamente separate in base alle dimensioni, densità, dispersione laterale (granularità), e marcatori di espressione di superficie cellulare. Più in concreto, le cellule possono essere sincronizzati con mezzi chimici. Diversi agenti come timidina, citosina arabinoside e idrossiurea possono essere utilizzati per inibire la sintesi del DNA nella fase S del ciclo cellulare risultante in un accumulo di cellule nella fase S che continuano ciclismo dopo che gli agenti sono rimossi. Le cellule trattate con nocodazole, che impedisce la formazione del fuso mitotico, l'arresto a G 2 - o M-fase contenuto di DNA. Eliminazione di siero dai risultati medi cultura in accumulo di cellule in G 0 fase. Tale aggiunta dei nutrienti nei siero cultura ri-avvia il normale bicicletta delle cellule. Tuttavia, tutti questi metodi di sincronizzazione interferisce con il ciclismo normale e la crescita delle cellule e possono Result significativa morte cellulare.
La sincronizzazione delle cellule di leucemia linfoblastica acuta è particolarmente impegnativo e queste cellule non sono suscettibili di manipolazione genetica. Il metodo qui descritto consente di stimare le dinamiche del ciclo cellulare e lo studio delle cellule in particolari fasi del ciclo cellulare senza sincronizzazione tradizionale o modificazione genetica. Questo metodo può anche essere utile per altri tipi di cellule in cui la modificazione genetica e procedure di sincronizzazione tradizionali non sono facilmente ottenibili. Il metodo si basa sull'uso consolidata di bromodeossiuridina (BrdU), che ha un impatto sulla crescita a breve termine e la proliferazione delle cellule. 4 protocolli BrdU fondazione sfruttano l'incorporazione di BrdU nel DNA di nuova sintesi in fase S . Questo segna definitivamente le cellule come dopo essere stato in fase S durante l'esposizione BrdU. Questa popolazione può essere identificato in momenti successivi con la colorazione per BrdU Incorporzione e quindi agiscono come una popolazione sincronizzato che può essere seguito e valutati nel corso del tempo permettendo lo studio degli effetti dei farmaci sul transito del ciclo cellulare. BrdU deve essere esposto prima della colorazione degli anticorpi, di solito raggiunti per DNasi o trattamento con l'acido. 6,7 citometria a flusso per rilevare BrdU incorporata consente l'inserimento di altri marker. La più importante è l'uso di coloranti per misurare il contenuto di DNA, consentendo la valutazione del ciclo cellulare distribuzione di fase delle cellule che erano in fase S all'inizio dello studio. 8 Inoltre aggiuntivi antigeni di superficie o intracellulari possono essere studiate. 9 Questi possono riguardare eventi del ciclo cellulare, come Ki67 o funzioni cellulari per apparentemente non correlate, come marcatori di apoptosi, come spaccati caspasi-3. Le potenziali applicazioni sono limitate dalla fantasia del ricercatore.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Il protocollo qui descritto utilizza la linea cellulare linfoblastica acuta leucemia NALM6 ma può essere applicato ad altri tipi di cellule.
1. Soluzioni e reagenti
- Completa RPMI
- Aggiungere 56 ml di siero fetale bovino (FCS) e 5,5 ml di 200 mM di L-glutammina in un flacone da 500 ml di mezzo RPMI-1640.
- BrdU Stock Solution
- Preparare 32.5 BrdU mm (10 mg / ml) in Dulbecco tampone fosfato (DPBS).
- BrdU completo RPMI
- Aggiungere 6,2 ml di soluzione madre BrdU per 10 ml di RPMI completo.
- DNase Soluzione
- Preparare 1 mg DNase / ml in DPBS.
- Colorazione Buffer
- Preparare 3% FCS inattivato al calore e 0,09% di sodio azide in DPBS.
- Vedi Materiali Lista per le definizioni di Fixation Buffer, Permeabilization tampone e tampone di lavaggio.
2. Cellule
NoTE: Le cellule non sono state coltivate per oltre 6 mesi. Questo metodo è direttamente adattabile a qualsiasi linea cellulare non aderente con adattamenti densità cellulare e coltura. Utilizzare le cellule che stanno crescendo in modo esponenziale a inizio dell'esperimento.
- Mantenere le cellule NALM6 in T-75 fiasche di coltura in RPMI completo. Eseguire tutte le operazioni in condizioni sterili utilizzando una classe II biosicurezza Cabinet.
- Mantenere le cellule NALM6 tra 1-2 x 10 6 cellule per ml dividendo cultura tre volte alla settimana.
- Incubare a 37 ° C in 5% CO 2 in aria.
3. Pulse Etichettatura di Celle con BrdU
ATTENZIONE: Maneggiare con cura BrdU in quanto è un potenziale mutageno e teratogeno.
- Cellule centrifugare a 150 xg per 5 min. Nota: Trasferimento di cellule in mezzi freschi migliora la riproducibilità dei risultati.
- Eseguire un numero di celle e risospendere le cellule in RPMI completo a 2 x 10 6 cells / ml.
- Diluire le cellule in 1 2 con BrdU completo RPMI producendo una concentrazione cellulare finale di 1 x 10 6 cellule / ml.
- Incubare a 37 ° C con 5% di CO 2 per 45 minuti, poi diluito 1 a 10 celle con RPMI completo. Cellule centrifugare a 150 xg per 5 minuti e con attenzione scartano tutto il surnatante.
- Risospendere le cellule in un piccolo volume (~ 100 ml) di RPMI completo, eseguire un conteggio delle cellule e regolare per 1 x 10 6 cellule / ml.
- Pipettare 1 ml di cellule nei pozzetti di una piastra ben 48. Pipettare 1 ml di DPBS in qualsiasi pozzetti non occupate per ottenere risultati più riproducibili.
- Incubare a 37 ° C in 5% CO 2 in aria per timepoints desiderati, qui 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17 , 18, 19, 20, 21, 22, 23, e 24 ore. Nota: La lunghezza del tempo dipenderà da ciò che il disegno sperimentale mira a misurare.
- Trasferire tutte le cellule in tubi FACS con una pipetta. Sciacquare la sequenza bene con 1 ml volumes di PBS per un volume totale finale di 5 ml.
- Centrifugare a 150 xg per 5 minuti e rimuovere con attenzione tutto il surnatante. Le cellule sono pronte per la colorazione, eseguire questa (sezione 4) immediatamente.
Colorazione 4. cellulare
Nota: Se è richiesta superficie colorazione di cellule eseguirla prima del fissaggio, in modo che le cellule sono conservati a 4 ° C per tutta.
- Risospendere le cellule in 100 ml di tampone colorazione (per facoltativo colorazione della superficie, aggiungere il volume consigliato di anticorpi di superficie antigeni e incubare per 30 minuti a 4 ° C).
- Aggiungere 1 ml di tampone colorazione, centrifugare per 5 min a 150 xg ed eliminare il surnatante.
Nota: l'anticorpo specifico, la concentrazione, il tempo di incubazione ecc varierà a seconda degli obiettivi sperimentali specifici. - Fissazione e permeabilizzazione
- Risospendere le cellule in 100 microlitri di tampone di fissaggio e incubare per 15 min a temperatura ambiente.
- Aggiungere 1 ml of tampone di lavaggio, centrifugare per 5 min a 150 xg ed eliminare il surnatante.
- Risospendere le cellule in 100 microlitri di tampone permeabilizzazione e incubare le cellule per 10 min in ghiaccio.
- Aggiungere 1 ml di tampone di lavaggio, centrifuga per 5 minuti a 150 xg, e scartare il surnatante.
- Risospendere le cellule in 100 microlitri di tampone di fissaggio per tubi e incubare per 5 min a temperatura ambiente.
- Aggiungere 1 ml di tampone di lavaggio, centrifuga per 5 minuti a 150 xg, e scartare il surnatante.
Nota: Il protocollo può essere messo in pausa qui, se necessario. Le cellule fissate sono stabili per diversi giorni a 4 ° C se risospeso in tampone colorazione. Rimuovere il buffer di colorazione seguente centrifugazione prima di procedere.
- DNase Trattamento
- Risospendere le cellule in 100 microlitri di soluzione di DNasi (30 microgrammi di DNasi / 10 6 cellule) e incubare le cellule per 1 ora a 37 ° C.
- Aggiungere 1 ml di tampone di lavaggio, centrifugare a 150 xg per 5 minuti e scartare il surnatante.
- Anticorpi colorazione
Nota: colorazione di marcatori intracellulari diversi BrdU può essere eseguita contemporaneamente alla colorazione BrdU.- IMPORTANTE: Preparare controlli di compensazione costituiti da cellule non colorati e cellule marcate con ogni singolo fluorocromo. Idealmente, utilizzare gli stessi anticorpi per i controlli di compensazione come quelle utilizzate nei tubi sperimentali. Tuttavia, se ciò non è fattibile, anticorpi sostituto antigeni altamente espressi coniugati allo stesso fluorocromo.
- Risospendere le cellule in 50 ml di tampone di lavaggio e aggiungere 1 ml / 10 6 cellule di anticorpi BrdU. Nota: anticorpi coniugati direttamente ad altri antigeni intracellulari specifici possono anche essere aggiunti.
NOTA:. Gli anticorpi anti istone H3 fosforilata in Ser10 possono essere utilizzati per discriminare tra cellule in G2 e M, istone H3 è fosforilata in Ser10 durante la mitosi 10 Anticorpi anti cdc2 fosforilati su Tyr15 può essere utilizzato per rilevare le cellule che HAVe impegnata per mitosi. 11 - Incubare le cellule per 20 minuti a temperatura ambiente.
- Aggiungere 1 ml di tampone di lavaggio, le cellule centrifugare a 150 xg per 5 minuti e scartare il surnatante.
- DNA Stain per l'analisi del ciclo cellulare
- Allentare pellet e aggiungere 20 ml di soluzione di 7-AAD (0,25 mg). Nota: è fondamentale utilizzare una quantità costante di 7-AAD / cell.
- Risospendere le cellule in 1 ml di tampone di colorazione.
5. Raccolta di Citometria a Flusso di dati
Nota: La macchina richiesta dipenderà dal numero e natura dei fluorocromi usati.
- Raccogliere i seguenti parametri: FSC-A, SSC-A, FSC-H (FSC-W possono essere utilizzati al posto di FSC-H) e 7-AAD fluorescenza su una scala lineare. Raccogliere il canale APC su una scala logaritmica. Raccogliere eventuali canali aggiuntivi necessari per la valutazione della superficie o interne etichette utilizzando una scala logaritmica.
- Eseguire comcompensazione dei segnali in spettri di emissione osservata tra diversi fluorocromi prima di analizzare i campioni si sovrappongono. Nota: La maggior parte dei citofluorimetri eseguirà automaticamente.
- Raccogliere almeno 10.000 eventi per ciascun campione.
6. Analisi di citometria a flusso di dati
Nota: FlowJo stato utilizzato in questo studio per citometria di flusso possono essere utilizzati anche l'analisi dei dati, ma altri pacchetti software. La strategia di gating è illustrato nella figura 1.
- Identificare la popolazione di cellule vitali con i parametri FSC-A e SSC-A.
- All'interno di questa popolazione escludere doppietti e aggregati utilizzando FSC-A e FSC-H (FSC-W possono anche essere usato qui).
- All'interno di questa popolazione impostare un diagramma a punti con 7-AAD sulla xe BrdU-APC sul y.
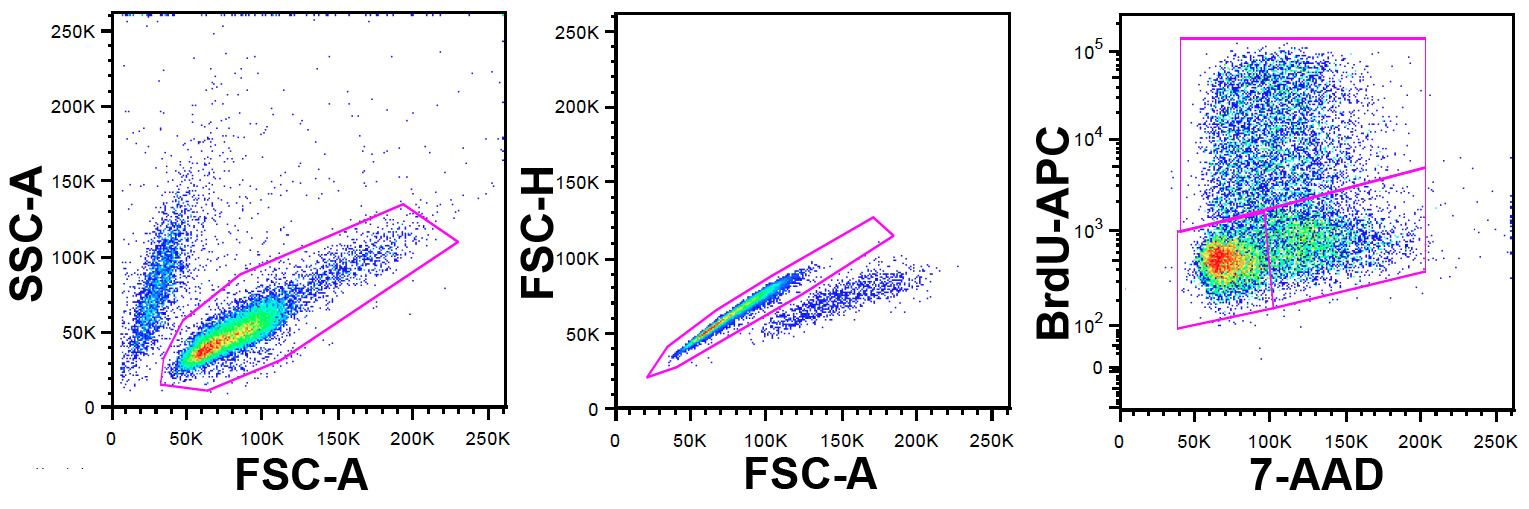
Figura 1: strategia di gating pa sinistra.nel: cellule ungated vengono visualizzati su un FCS-A e SSC-A dot plot. La popolazione di cellule vitali è identificata dalla porta mostrato. Pannello centrale: le cellule gated dal pannello a sinistra vengono visualizzati su un FSC A vs. FSC-H dot plot (FSC-W può essere usato al posto di altezza). Farsetti e aggregati sono identificati, ed esclusi dalla porta mostrato. A destra: le cellule gated dalla data di esclusione doppietto nel pannello centrale vengono visualizzati su un 7-AAD vs. APC-A diagramma a punti. L'anticorpo è marcato con BrdU APC consentono di identificare le cellule che hanno incorporato BrdU durante l'etichettatura impulso. 7-AAD fornisce informazioni sul contenuto di DNA. La porta superiore definisce cellule positive per BrdU e quindi in fase S al tempo dell'impulso BrdU, porta inferiore sinistra, cellule in G 0/1 e la porta inferiore destro quelli in G 2 / M. Fare click qui per visualizzare un versione più grande di questa figura.
- Cell Cycle A nalisi
- Aprire il primo file di dati porta e sulle cellule del cancello esclusione doppietto.
- Analizzare questa popolazione per la distribuzione del ciclo cellulare (che si trova sotto le piattaforme di software Flojo) e utilizzare il modello Dean-Jett-Fox.
- Ottenere le posizioni del G 0/1 e G 2 / M vette utilizzando creare cancelli.
- Porta sul BrdU cellule positive e soggetti a questa popolazione stessa analisi del ciclo cellulare.
- Fornire le posizioni per la G 0/1 e G 2 / M picchi applicando le stesse porte da creare porte e impostare vincoli (utilizzando le porte create) per le posizioni del G 0/1 e G 2 / M picchi. Questo è illustrato nei primi 2 pannelli di figura 2.
Nota: altri software può essere utilizzato anche per analizzare i dati e le istruzioni varierebbe di conseguenza.
840 / 52840fig2highres.jpg "width =" 700 "/>
Figura 2:. Cell Cycle Progression Il primo pannello (tutte le celle) è recintato sulla popolazione di cellule definito dalla porta esclusione farsetto. Questa popolazione è stata visualizzata in un istogramma con 7-AAD sulla X-asse. Il picco della G 0/1 picco è indicata dalla freccia sotto dell'asse. In pannelli successivi cellule positive BrdU sono stati gated come mostrato in Figura 1. Il valore per la posizione G 0/1 ottenuto quando gating del cancello esclusione doppietto è applicato alle cellule BrdU positive gated nel software ciclo cellulare FlowJo. Ogni pannello posteriore sia stato gating sulla popolazione positiva BrdU come mostrato nella figura 1 e la posizione del G 0/1 picco in base al valore ottenuto quando si analizza l'intera popolazione come mostrato nelle prime due pannelli. Utilizzando la frazione negativo BrdU per identificare la posizione della popolazione G 0/1 per le cellule positive BrdU nella stessa SAMPle controlla per eventuali lievi differenze nell'intensità della macchia DNA tra i campioni. Il numero indicato su ogni pannello rappresenta il tempo da quando l'impulso BrdU conclusa. Le fasi del ciclo cellulare calcolati sono mostrati in verde ombreggiato. Cliccate qui per vedere una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Risultati
Questo metodo può essere utilizzato per ottenere una serie di informazioni. Alcune applicazioni sono elencate qui.
Valutazione della durata del ciclo cellulare
Per determinare il tempo necessario per le cellule di transito attraverso il ciclo cellulare, le cellule vengono raccolte in vari momenti seguenti dell'impulso BrdU. Gli intervalli tra le valutazioni possono essere adattati alle particolari cellule essere analizzati. Linee cellulari ematopoietiche son...
Access restricted. Please log in or start a trial to view this content.
Discussione
La capacità di analizzare il ciclo cellulare è importante per la comprensione della biologia del cancro e il meccanismo di azione di entrambi i farmaci e geni che influenzano la proliferazione e la crescita cellulare. Mentre ci sono una moltitudine di saggi che misurano riferito proliferazione cellulare, la maggioranza solo fornire una misura che indica le cellule numerici presenti. Questi includono test che misurano il numero di cellulare per la visualizzazione diretta e il conteggio, l'attività metabolica o la ...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
The work was funded by the Leukemia and Lymphoma Society of the USA (6105-08), a Cancer Council NSW grant (13-02), an NHMRC Senior Research Fellowship (LJB) (1042305) and project grant (1041614).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| APC BrdU Flow Kit | BD Biosciences | 552598 | Contains BrdU antibody, 7-AAD and BD Cytofix/Cytoperm Buffer (referred to as Fixation Buffer) |
| BD Cytoperm Permeabilization Buffer Plus | BD Biosciences | 561651 | Referred to as Permeabilization buffer |
| BD Perm/Wash Buffer | BD Biosciences | 554723 | Referred to as Wash buffer |
| DNase | Sigma | D-4513 | |
| BD Falcon 12 x 75 mm FACS tubes | BD Biosciences | 352008 | |
| BD Pharmingen Stain Buffer | BD Biosciences | 554656 | |
| BD LSR FORTESSA flow cytometer | BD Biosciences | FORTESSA | |
| Pipetman | Gilson | P2, P20, P100, P1000 | |
| RPMI 1,640 w/o L-Gln 500 ml | Lonza | 12-167F | |
| DPBS | Lonza | 17-512F | |
| Fetal Bovine Serum | FisherBiotec | FBS-7100113 | |
| L-Glutamine | Sigma | G7513-100ML | |
| 5-Bromo-2′-deoxyuridine | Sigma | B5002-1G | |
| Falcon TC 150 cm2 vented Flasks | BD Biosciences | 355001 | |
| Pipettes 25 ml | Greiner | 760180 | |
| Aersol Pipettes 200 µl | Interpath | 24700 | |
| Aersol Pipettes 1 ml | Interpath | 24800 | |
| Centrifuge | Spintron | GT-175R | |
| CO2 incubator | Binder | C 150 | |
| AF488 anti-Histone H3 Phospho (Ser10) Antibody | Cell Signalling | 9708S | |
| Phospho-Chk2 (Thr68) (C13C1) Rabbit mAb | Cell Signalling | 2197S | |
| Phospho-Chk1 (Ser345) (133D3) Rabbit mAb | Cell Signalling | 2348S | |
| NALM6 | DSMZ | ACC-128 |
Riferimenti
- Banfalvi, G. Methods Mol Biol. Banfalvi, G. 761, Humana Press. New York, Dordrecht, Heidelberg, London. 1-23 (2011).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132, 487-498 (2008).
- Harper, J. W., Elledge, S. J. The DNA damage response: ten years after. Molecular Cell. 28, 739-745 (2007).
- Latt, S. A., George, Y. S., Gray, J. W. Flow cytometric analysis of bromodeoxyuridine-substituted cells stained with 33258 Hoechst. The Journal of Histochemistry and Cytochemistry. 25, 927-934 (1977).
- Gratzner, H. G. Monoclonal antibody to 5-bromo- and 5-iododeoxyuridine: A new reagent for detection of DNA replication. Science. 218, 474-475 (1982).
- Carayon, P., Bord, A. Identification of DNA-replicating lymphocyte subsets using a new method to label the bromo-deoxyuridine incorporated into the DNA. Journal of Immunological Methods. 147, 225-230 (1992).
- Gonchoroff, N. J., et al. S-phase detection with an antibody to bromodeoxyuridine. Role of DNase pretreatment. Journal of Immunological Methods. 93, 97-101 (1986).
- Rabinovitch, P. S., Torres, R. M., Engel, D. Simultaneous cell cycle analysis and two-color surface immunofluorescence using 7-amino-actinomycin D and single laser excitation: applications to study of cell activation and the cell cycle of murine Ly-1 B cells. Journal of Immunology. 136, 2769-2775 (1986).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. 32, 4789-4797 (2013).
- Hendzel, M. J., et al. Mitosis-specific phosphorylation of histone H3 initiates primarily within pericentromeric heterochromatin during G2 and spreads in an ordered fashion coincident with mitotic chromosome condensation. Chromosoma. 106, 348-360 (1997).
- Draetta, G., Beach, D. Activation of cdc2 protein kinase during mitosis in human cells: cell cycle-dependent phosphorylation and subunit rearrangement. Cell. 54, 17-26 (1988).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. , (2012).
- Knudsen, R. C., Ahmed, A. A., Sell, K. W. An in vitro microassay for lymphotoxin using microculture plates and the multiple automated sample harvester. Journal of Immunological Methods. 5, 55-63 (1974).
- Beck, H. P. Proliferation kinetics of perturbed cell populations determined by the bromodeoxyuridine-33258 technique: radiotoxic effects of incorporated [3H]thymidine. Cytometry. 2, 170-174 (1981).
- Miller, M. J., Wei, S. H., Parker, I., Cahalan, M. D. Two-photon imaging of lymphocyte motility and antigen response in intact lymph node. Science. 296, 1869-1873 (2002).
- Collins, J. M., Berry, D. E., Bagwell, C. B. Different rates of DNA synthesis during the S phase of log phase HeLa S3, WI-38, and 2RA cells. Journal of Biological Chemistry. 255, 3585-3590 (1980).
- Schwarting, R., Gerdes, J., Niehus, J., Jaeschke, L., Stein, H. Determination of the growth fraction in cell suspensions by flow cytometry using the monoclonal antibody Ki-67. Journal of Immunological Methods. 90, 65-70 (1986).
- Danova, M., et al. Cell cycle-related proteins: a flow cytofluorometric study in human tumors. Biology of the Cell. 64, 23-28 (1988).
- Juan, G., et al. Histone H3 phosphorylation and expression of cyclins A and B1 measured in individual cells during their progression through G2 and mitosis. Cytometry. 32, 71-77 (1998).
- Langan, T. J., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 761, 75-83 (2011).
- Kues, W. A., et al. Cell cycle synchronization of porcine fetal fibroblasts: effects of serum deprivation and reversible cell cycle inhibitors. Biology of Reproduction. 62, 412-419 (2000).
- Urbani, L., Sherwood, S. W., Schimke, R. T. Dissociation of nuclear and cytoplasmic cell cycle progression by drugs employed in cell synchronization. Experimental Cell Research. 219, 159-168 (1995).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon