JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
동기화를 필요로하지 않고 유동 세포 계측법을 사용하여 세포주기 진행의 시간적 추적
요약
이 프로토콜은 특정 시점에서 S 상에 있었던 셀의 시간 추적을 허용 브로 모데 옥시 우리 딘 (BrdU의) 흡수의 사용을 설명한다. DNA 염료 및 표지 항체의 첨가는 이후 시간에 S 상 세포의 운명의 상세한 분석을 용이하게한다.
초록
This protocol describes a method to permit the tracking of cells through the cell cycle without requiring the cells to be synchronized. Achieving cell synchronization can be difficult for many cell systems. Standard practice is to block cell cycle progression at a specific stage and then release the accumulated cells producing a wave of cells progressing through the cycle in unison. However, some cell types find this block toxic resulting in abnormal cell cycling, or even mass death. Bromodeoxyuridine (BrdU) uptake can be used to track the cell cycle stage of individual cells. Cells incorporate this synthetic thymidine analog, while synthesizing new DNA during S phase. By providing BrdU for a brief period it is possible to mark a pool of cells that were in S phase while the BrdU was present. These cells can then be tracked through the remainder of the cell cycle and into the next round of replication, permitting the duration of the cell cycle phases to be determined without the need to induce a potentially toxic cell cycle block. It is also possible to determine and correlate the expression of both internal and external proteins during subsequent stages of the cell cycle. These can be used to further refine the assignment of cell cycle stage or assess effects on other cellular functions such as checkpoint activation or cell death.
서문
세포주기 진행 동안에 세포주기 및 세포 기능에서 발생하는 변화의 평가는 생물, 특히 암 생물학의 여러 측면의 이해에 기초한다. 악성 종양의 치료를 위해 개발에 많은 제제는 세포주기 진행에 지대한 영향을 미칠 또는 세포주기 - 종속 메카니즘을 통해 세포 사멸을 유도한다. 세포주기의 특정 단계에서 세포주기 역학 또는 세포를 연구하기 위해, 세포를 동기화하는 것이 일반적이다. 그러나 동기화 방법은 잠재적 결과 얻어진 교란, 검토되고 세포에 해로운 영향을 가질 수있다. 1 시간에 걸쳐 하나의 세포에서 세포주기 진행의 최근 세포주기의 특정 단계에서만 존재하는 형광 태그 된 단백질의 사용이 허용 된 분석 (2) 그러나,이 세포는 유전자가 읽을 수있는 시스템의 사용을 제한하는, 이들 태그가 단백질을 발현하도록 조작 할 필요가 공부 될ILY 달성했다.
합성 (S) 상, DNA 복제와 유사 분열 (M) 여기서 세포 분열이 발생되어 세포주기는 두 개의 활성 단계로 구성됩니다. 이 단계는 세 갭 단계, G 0, G 1 및 G 2로 구분됩니다. 셀의 크기가 종래 세포 성장은 DNA 복제의 완료 사이하지만 세포 분열 전에 계속 DNA 복제와 G이 증가 여기서 G는 0 또는 정지, 세포주기를 떠난 쉬고 위상이고, G 1이다. 세포주기를 통해 진행이 체크 포인트의 수에 의해 제어된다. 환경 조건이 DNA 합성을지지하지 않으며 S 단계 진입을 방지 할 때 G 1 체크 포인트가 활성화됩니다. 인트라 S 상 검문소 또는 지연 실속 복제 포크 초래할 수 DNA 손상에 의해 유발 될 수있다. 손상 후 G 2 검출되면 G 2 중 DNA의 복제 충실도를 확인되고 체크 포인트는 분열을 셀에 DNA 복구 전에 허용 활성화됩니다. 유사 분열 동안 최종 체크 포인트는 세포 분열이 성공적으로 완료 될 수 있도록 염색 분체가 제대로 유사 분열 판에 정렬되어 있는지 확인합니다.이 체크 포인트 3 활성화는 일반적으로 세포 인구를 동기화하는 데 사용됩니다. 세포주기 체크 포인트는 다수의 인자에 의해되지만 가장 일반적인 DNA 손상의 검출의 암 생물학에서 활성화 될 수있다. DNA 손상 응답 (ATR) 관련 PI3 키나아제 형 키나제의 실조증 모세 혈관과 Rad3 의해 개시 및 운동 실조의 모세 혈관이 (ATM) 각각 하류 효과기 키나아제 CHK1 및 Chk2를 활성화하는 돌연변이. 3 이벤트 범위가 정지 포함 CHK1를 기동 복제 포크 DNA 가교 결합 및 자외선 손상 Chk2는 주로 이중 가닥 바꿈에 의해 활성화된다.
세포주기의 전 길이에 변경된 조건의 효과를 연구하기위한 통상의 방법S는 세포주기의 특정 단계에서 세포를 동기화한다. 한 이것은 여러 가지 방법을 통해 달성 될 수있다. 세포를 물리적 크기, 밀도, 측면 스 캐터 (입상), 및 세포 표면 마커의 발현에 기초하여 분리 될 수있다. 더 실제적으로, 세포는 화학적 수단에 의해 동기화 될 수있다. 예컨대 티미 딘, 히드 록시 우레아 및 시토신 아라 비노 여러 에이전트는 에이전트가 제거 된 후 순환을 계속 S 단계에서 세포의 축적으로 인한 세포주기의 S 단계에서 DNA 합성을 억제하는데 사용될 수있다. 또는 M 상 DNA 함량 - 세포는 G 2 방추사 형성 체포 방지 nocodazole으로 처리. G 0 단계에서 세포의 축적 배지 결과에서 혈청의 제거. 배양 혈청 내의 영양소의 재 첨가 세포의 정상적인 순환을 재 - 시작한다. 그러나 이러한 동기화 방법은 모든 세포의 정상적인 순환과 성장을 방해 할 수 resul중요한 세포 사멸에 T.
급성 림프 구성 백혈병 세포의 동기화는 특히 도전이며,이 세포는 유전자 조작 의무가 없습니다. 여기에 설명 된 방법은 세포주기 역학 평가 전통적인 동기화 또는 유전자 조작없이 세포주기의 특정 단계에서 세포의 연구를 허용한다. 이 방법은 유전자 변형 전통적인 동기화 과정이 용이하게 달성되지 않은 다른 세포 유형에 유용 할 수있다. 방법은 세포의 단기 성장과 증식에 거의 영향을 미치지 브로 모데 옥시 우리 딘 (BrdU의) 혼입의 노포 사용에 기초한다. 4 설립 BrdU의 프로토콜은 S 단계에서 새로 합성 된 DNA 내로 BrdU의 혼입을 활용 . 이것은 영구적 BrdU의 노광 중에 S 단계에서 된 것으로 세포를 표시한다. 이는 인구의 BrdU의 incorpor위한 염색하여 나중 시점에서 식별 될 수있다ATION 이에 따라야하고 세포주기 교통 약물 효과의 연구를 허용하는 시간에 걸쳐 평가 될 수 동기 인구로서 작용한다. BrdU의 보통의 DNase 또는 산 처리 다음 달성 전에 항체 염색에 노출 될 필요가있다. -6,7- BrdU의 도입은 부가적인 마커의 포함을 가능하게 검출하기 위해 유세포 사용. 가장 중요한 연구의 개시시에 S 상에 있던 세포의 세포주기 위상 분포의 평가를 가능하게 DNA 함량을 측정하기 위해 염료를 사용하는 것이다. (8) 또한 추가적인 표면 또는 세포 항원은 또한 조사 할 수있다. (9) 이러한 이러한 사료된다 등의 세포주기 이벤트 관련 또는 절단 카스파 제 -3와 같은 세포 사멸 마커 등으로 관련이 없어 보이는 세포의 기능을 할 수 있습니다. 잠재적 인 응용 프로그램은 연구자의 상상력에 의해 제한됩니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
여기에 설명 된 프로토콜은 급성 림프 모구 백혈병 세포주 NALM6를 사용하지만, 다른 유형의 세포에도 적용 할 수있다.
1. 솔루션 및 시약
- 전체 RPMI
- 56 ml의 소 태아 혈청 (FCS) 및 RPMI-1640 배지 500㎖의 병에 200 ml의 5.5 mm의 L - 글루타민 추가.
- BrdU의 원액
- 둘 베코 인산염 완충 생리 식염수 (DPBS)에서 32.5 mM의 BrdU의 (10 ㎎ / ㎖)을 준비합니다.
- BrdU의 전체 RPMI
- 전체 RPMI 10 ㎖에 BrdU의 주식 솔루션의 6.2 μl를 추가합니다.
- DNase의 솔루션
- 1 밀리그램의 DNase / DPBS에 mL로 준비합니다.
- 염색 버퍼
- DPBS에서 3 % 열 - 불 활성화 FCS 및 0.09 %의 아 지드 화 나트륨을 준비한다.
- 고정 버퍼, 투과성으로 버퍼 및 세척 버퍼의 정의에 대한 자료 목록을 참조하십시오.
2. 세포
아니테 : 세포 6 개월을 초과하는 배양하지 않았다. 이 방법은 셀 밀도 및 배양 배지에 대한 조정과 비 - 부착 세포 라인에 직접 적용 할 것이다. 실험의 개시에 기하 급수적으로 증가하고 세포를 사용합니다.
- 전체 RPMI에서 T-75 배양 플라스크에 NALM6 세포를 유지한다. 클래스 II 바이오 안전성 캐비닛을 사용하여 무균 조건 하에서 모든 단계를 수행합니다.
- 세 번 주간 문화를 분할하여 ml의 당 6 10 × 1-2 사이의 세포를 NALM6 세포를 유지한다.
- 공기에서 이산화탄소 5 %에서 37 ° C에서 품어.
BrdU의와 세포의 3 펄스 라벨링
주의 : 잠재적 돌연변이와 기형은주의하여 BrdU의를 처리합니다.
- 5 분 150 XG에 원심 분리기 세포. 참고 : 신선한 매체에 세포를 전송하는 것은 결과의 재현성을 향상시킨다.
- 2 × 106 (C)에서 완전 RPMI에서 세포 수와 세포를 재현 탁 수행ELL 학생 / ㎖.
- BrdU를 완전 RPMI 1 × 106 세포 / ml의 최종 세포 농도를 생산하는 두 셀 1 희석.
- 다음 완전한 RPMI 10 셀 1 희석, 45 분 동안 5 % CO 2, 37 ℃에서 인큐베이션. 조심스럽게 (150) 5 분 XG와 원심 분리기 세포 상층 액을 모두 폐기합니다.
- 완전한 RPMI의 작은 부피 (~ 100 μL)에 재현 탁 된 세포는 세포 수를 수행하고 1 × 106 세포 / ml로 조정한다.
- 피펫 48 웰 플레이트로 세포를 1 ㎖. 모든 빈 우물에 DPBS의 피펫 1 ml를 더 재현 가능한 결과를 얻을 수있다.
- (17) 여기에서, 원하는 시점들, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 공기 중 5 %에서 37 ° C에서 CO 2 부화 , 18, 19, 20, 21, 22, 23, 및 24 시간. 참고 : 시간의 길이는 실험 설계 측정하는 것을 목표로 무엇에 따라 달라집니다.
- 피펫을 사용하여 외과 튜브로 모든 세포를 전송합니다. 1ml의 volu로 잘 헹구고 순차적5 ㎖의 최종 총 부피의 PBS MES.
- 5 분 150 XG에 원심 분리기 조심스럽게 모든 뜨는을 제거합니다. 세포를 염색 할 준비가 즉시 (4 절)을 수행합니다.
4. 세포 얼룩
주 : 세포 표면 염색이 필요한 경우 세포에 걸쳐 4 ℃로 유지되는 것을 보장하기 전에 고정을 수행한다.
- 염색 버퍼 100 ㎕에 재현 탁 세포 (옵션 표면 염색은 항원을 표면 및 4 ℃에서 30 분 동안 배양 항체의 권장 볼륨을 추가).
- 150 XG에서 5 분 동안 염색 버퍼 1 ㎖, 원심 분리기를 추가하고 상층 액을 버린다.
참고 : 등 특정 항체 농도, 배양 시간을 특정 실험 목적에 따라 달라질 수 있습니다. - 고정과 투과성으로
- 고정 버퍼 100 ㎕에 세포를 재현 탁하고 실온에서 15 분 동안 배양한다.
- 1 ml의 O 추가F 세척 버퍼, 150 XG에서 5 분 동안 원심 분리하고 상층 액을 버린다.
- 투과성으로 완충액 100 ㎕에 세포를 재현 탁하고 얼음에 10 분 동안 세포를 배양한다.
- 150 XG에서 5 분 동안 세척 버퍼 1 ㎖, 원심 분리기를 추가하고, 상층 액을 버린다.
- 튜브 당 고정 버퍼 100 ㎕에 세포를 재현 탁하고 실온에서 5 분 동안 배양한다.
- 150 XG에서 5 분 동안 세척 버퍼 1 ㎖, 원심 분리기를 추가하고, 상층 액을 버린다.
주 : 필요한 경우, 프로토콜은 여기에서 일시 정지 될 수있다. 염색 완충액 경우 고정 된 세포는 4 ° C에서 며칠 동안 안정하다. 진행하기 전에 원심 분리 다음 염색 버퍼를 제거합니다.
- DNase의 치료
- DNase의 용액 100 ㎕ (30 DNase의 / 10 6 세포의 μg의), 37 ℃에서 1 시간 동안 세포를 배양에 재현 탁 세포.
- 5 분 150 XG에 세척 버퍼, 원심 분리기 1 ML을 추가하고 상층 액을 버린다.
- 항체 염색
주 : BrdU의 이외의 세포 마커 염색 BrdU의 염색과 동시에 행할 수있다.- 중요 : 각 하나의 형광 색소로 표지 흠없는 세포와 세포로 구성된 보상 컨트롤을 준비합니다. 이상적으로, 실험 튜브에 사용되는 것과 같은 보상 컨트롤 같은 항체를 사용합니다. 이것이 가능하지 않을 경우, 고도로 발현 항원 항체 대신 동일한 형광 색소에 접합.
- 세척 완충액 50 μL에 재현 탁하고 세포를 1 μL / BrdU의 항체 10 6 세포를 추가한다. 참고 : 다른 특정 세포 내 항원에 직접 결합하는 항체도 추가 할 수 있습니다.
주 :. cdc2에 Ser10 인산화 히스톤 H3에 항체가 G2 및 M에서 세포를 구별하는 데 사용할 수있는, 히스톤 H3 유사 분열 동안 Ser10에 인산화 10 항체는 근래 세포를 검출하는데 사용될 수 Tyr15 인산화전자 유사 분열에 최선을 다하고 있습니다. (11) - 실온에서 20 분 동안 세포를 인큐베이션.
- 5 분 동안 150 XG에서 1 세척 버퍼의 ml의 원심 분리기 세포를 추가하고 상층 액을 버린다.
- 세포주기 분석을위한 얼룩의 DNA
- 펠렛을 풀고 7-AAD 용액 (0.25 μg의) 20 μl를 추가합니다. 주 : 7-AAD / 셀의 일정한 양을 사용하는 것이 중요합니다.
- 염색 완충액 1 ml의 세포를 재현 탁.
유동 세포 계측법 데이터 5. 컬렉션
참고 : 사용되는 형광 색소의 수와 특성에 따라 달라집니다 필요한 기계.
- 다음과 같은 파라미터를 수집 FSC-A, SSC-A, FSC-H 선형 스케일 7- AAD 형광 (FSC-W는 FSC-H 대신에 사용될 수있다). 로그 스케일 APC 채널 모아서. 로그 스케일을 사용하여 표면 또는 내부 라벨의 평가를 위해 필요한 추가 채널을 수집합니다.
- COM을 수행시료를 분석하기 전에 다른 형광 색소 사이의 발광 스펙트럼 관찰에 신호를 중첩 보정 :. 참고 : 대부분의 유동 세포 계측기가이 작업을 자동으로 수행합니다.
- 각 샘플에 대해 적어도 10,000 이벤트를 수집합니다.
유동 세포 계측법 데이터의 분석 (6)
주 : 데이터 해석되지만, 다른 소프트웨어 패키지가 또한 사용될 수있다 계측법 FlowJo 흐름이 연구에 사용 하였다. 게이팅 전략은도 1에 도시되어있다.
- FSC-A와 SSC-매개 변수를 사용하여 실행 가능한 세포 인구를 식별합니다.
- 이 인구 내에서 이중선과 (도 여기에 사용할 수있는 FSC-W) FSC-A 및 FSC-H를 사용하여 집계를 제외합니다.
- 모집단 내의 Y 축의 X 축 및 BrdU의 APC-7-AAD를 사용하여 도트 플롯을 설정한다.
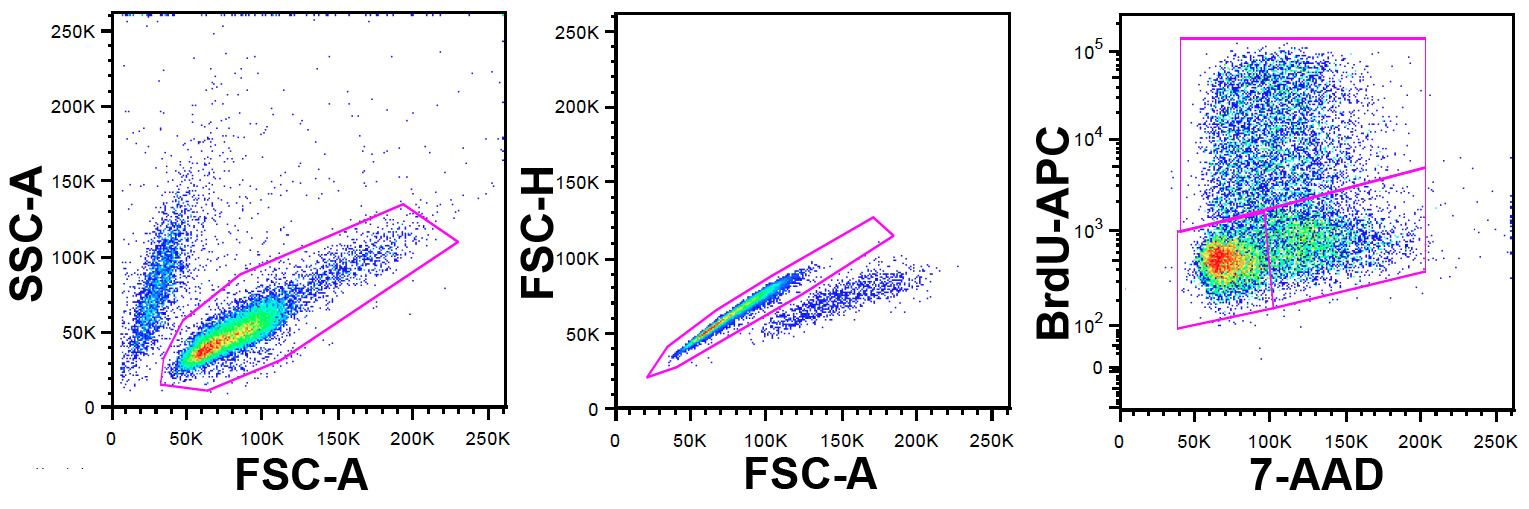
그림 1 : 게이팅 전략 왼쪽 PA.Nel 보낸 : ungated 세포는 SSC-도트 플롯 대 FCS-에 표시됩니다. 가능한 세포 인구가 도시 게이트로 식별됩니다. 센터 패널 : 왼쪽 패널에서 문이 세포는 FSC-H 도트 플롯 (대신 높이를 사용할 수 있습니다 FSC-W) 대 FSC-에 표시됩니다. 이중선과 집계가 확인하고 그림과 게이트에 의해 제외됩니다. 오른쪽 패널 : 중앙 패널에 이중 배제 날로부터 게이트 세포가 APC-도트 플롯 대 7-AAD에 표시됩니다. BrdU의 항체는 APC는 펄스 라벨 동안 BrdU의를 통합 한 세포의 식별을 가능하게 표시되어 있습니다. 7-AAD는 DNA 콘텐츠에 대한 정보를 제공합니다. 상단 게이트는 BrdU의 펄스, 왼쪽 하단 게이트, 세포 G 0/1 및 G에 그 오른쪽 게이트의 시간에 S 단계에 따라서 BrdU의 양성 세포를 정의 / M 2. 를 보려면 여기를 클릭하십시오 이 그림의 더 큰 버전.
- 세포주기 nalysis
- 이중 제외 게이트의 셀의 첫 번째 데이터 파일과 문을 엽니 다.
- (FloJo 소프트웨어 플랫폼 아래에있는) 세포주기 분포이 인구를 분석하고 딘 - 제트 - 폭스 모델을 사용합니다.
- G 0/1 및 G 게이트를 작성하여 2 / M 피크의 위치를 가져옵니다.
- BrdU의 양성 세포와 주제에 게이트 같은 세포주기 분석이 인구.
- 만들 게이트에서 같은 게이트를 적용하고 G 0/1 및 G 2 / M 피크의 위치에 대한 (생성 된 게이트를 사용하여) 제약 조건을 설정하여 G 0/1 및 G 2 / M 피크의 위치를 제공합니다. 이는도 2의 제 2 패널에 도시되어있다.
주의 : 다른 소프트웨어는 데이터를 분석하는데 사용될 수 있으며, 설명은 따라서 변할 것이다.
840 / 52840fig2highres.jpg "폭 ="700 "/>
그림 2. 세포주기 진행 첫번째 패널 (모든 셀)을 겹 배제 게이트에 의해 정의 세포군에 게이팅된다. 이 인구는 X 축에 7-AAD와 히스토그램 표시했다. G 0/1 피크의 피크 축 아래 화살표로 표시된다. 도 1에 도시 된 바와 같이 후속하는 패널에서의 BrdU 양성 세포상의 게이팅되었다. 이중선 배제 게이트의 게이팅 때 얻어지는 G 0/1 위치에 대한 값은 FlowJo 세포주기 소프트웨어 내에서의 BrdU 양성 게이팅 셀에인가된다. 도 1과 처음 두 패널에 도시 된 바와 같이 전체 모집단을 분석 할 때 얻어진 값에 기초하여 G 0/1 피크의 위치에서와 같이 이후의 각 패널은 BrdU의 양 인구에 게이팅되었다. 동일한 SAMP에서의 BrdU 양성 세포에 대해 G 0/1 인구의 위치를 식별하는 BrdU의 음 부분을 사용하여제작 샘플의 DNA를 얼룩의 강도에 어떤 약간의 차이를 제어합니다. 각각의 패널 상에 표시된 숫자는 BrdU의 펄스가 종료 된 이후의 시간을 나타낸다. 계산 된 세포주기 단계가 음영 녹색으로 표시되어있다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Access restricted. Please log in or start a trial to view this content.
결과
이 방법은 다양한 정보를 얻기 위해 사용될 수있다. 일부 응용 프로그램은 여기에 설명되어 있습니다.
세포주기의 지속 기간의 평가
세포주기를 통해 세포로 운송하는데 필요한 시간을 결정하기 위해, 세포를 BrdU의 펄스 아래의 다양한 시점에서 수확한다. 평가의 간격은 분석되는 특정 셀에 적용될 수있다. 조혈 세포주는 표준 배양 조건에서 세포주기 ?...
Access restricted. Please log in or start a trial to view this content.
토론
세포주기 분석하는 능력은 암 생물학을 이해하고 세포 증식 및 성장에 영향을 미치는 두 약물 및 유전자의 작용 메카니즘에 중요하다. 전하는 세포 증식을 측정하는 검정법 다수 존재하지만, 대부분은 단지 본 셀 번호를 나타내는 측정 값을 제공한다. 이들은 직접 시각화 및 계산, 신진 대사 활동 또는 ATP 농도로 세포 수를 측정 분석을 포함한다. 이러한 방법의 많은의 주요 장점은 조건 또는 화합...
Access restricted. Please log in or start a trial to view this content.
공개
The authors have nothing to disclose.
감사의 말
The work was funded by the Leukemia and Lymphoma Society of the USA (6105-08), a Cancer Council NSW grant (13-02), an NHMRC Senior Research Fellowship (LJB) (1042305) and project grant (1041614).
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| APC BrdU Flow Kit | BD Biosciences | 552598 | Contains BrdU antibody, 7-AAD and BD Cytofix/Cytoperm Buffer (referred to as Fixation Buffer) |
| BD Cytoperm Permeabilization Buffer Plus | BD Biosciences | 561651 | Referred to as Permeabilization buffer |
| BD Perm/Wash Buffer | BD Biosciences | 554723 | Referred to as Wash buffer |
| DNase | Sigma | D-4513 | |
| BD Falcon 12 x 75 mm FACS tubes | BD Biosciences | 352008 | |
| BD Pharmingen Stain Buffer | BD Biosciences | 554656 | |
| BD LSR FORTESSA flow cytometer | BD Biosciences | FORTESSA | |
| Pipetman | Gilson | P2, P20, P100, P1000 | |
| RPMI 1,640 w/o L-Gln 500 ml | Lonza | 12-167F | |
| DPBS | Lonza | 17-512F | |
| Fetal Bovine Serum | FisherBiotec | FBS-7100113 | |
| L-Glutamine | Sigma | G7513-100ML | |
| 5-Bromo-2′-deoxyuridine | Sigma | B5002-1G | |
| Falcon TC 150 cm2 vented Flasks | BD Biosciences | 355001 | |
| Pipettes 25 ml | Greiner | 760180 | |
| Aersol Pipettes 200 µl | Interpath | 24700 | |
| Aersol Pipettes 1 ml | Interpath | 24800 | |
| Centrifuge | Spintron | GT-175R | |
| CO2 incubator | Binder | C 150 | |
| AF488 anti-Histone H3 Phospho (Ser10) Antibody | Cell Signalling | 9708S | |
| Phospho-Chk2 (Thr68) (C13C1) Rabbit mAb | Cell Signalling | 2197S | |
| Phospho-Chk1 (Ser345) (133D3) Rabbit mAb | Cell Signalling | 2348S | |
| NALM6 | DSMZ | ACC-128 |
참고문헌
- Banfalvi, G. Methods Mol Biol. Banfalvi, G. 761, Humana Press. New York, Dordrecht, Heidelberg, London. 1-23 (2011).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132, 487-498 (2008).
- Harper, J. W., Elledge, S. J. The DNA damage response: ten years after. Molecular Cell. 28, 739-745 (2007).
- Latt, S. A., George, Y. S., Gray, J. W. Flow cytometric analysis of bromodeoxyuridine-substituted cells stained with 33258 Hoechst. The Journal of Histochemistry and Cytochemistry. 25, 927-934 (1977).
- Gratzner, H. G. Monoclonal antibody to 5-bromo- and 5-iododeoxyuridine: A new reagent for detection of DNA replication. Science. 218, 474-475 (1982).
- Carayon, P., Bord, A. Identification of DNA-replicating lymphocyte subsets using a new method to label the bromo-deoxyuridine incorporated into the DNA. Journal of Immunological Methods. 147, 225-230 (1992).
- Gonchoroff, N. J., et al. S-phase detection with an antibody to bromodeoxyuridine. Role of DNase pretreatment. Journal of Immunological Methods. 93, 97-101 (1986).
- Rabinovitch, P. S., Torres, R. M., Engel, D. Simultaneous cell cycle analysis and two-color surface immunofluorescence using 7-amino-actinomycin D and single laser excitation: applications to study of cell activation and the cell cycle of murine Ly-1 B cells. Journal of Immunology. 136, 2769-2775 (1986).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. 32, 4789-4797 (2013).
- Hendzel, M. J., et al. Mitosis-specific phosphorylation of histone H3 initiates primarily within pericentromeric heterochromatin during G2 and spreads in an ordered fashion coincident with mitotic chromosome condensation. Chromosoma. 106, 348-360 (1997).
- Draetta, G., Beach, D. Activation of cdc2 protein kinase during mitosis in human cells: cell cycle-dependent phosphorylation and subunit rearrangement. Cell. 54, 17-26 (1988).
- Saunders, P. O., et al. RAD001 (everolimus) induces dose-dependent changes to cell cycle regulation and modifies the cell cycle response to vincristine. Oncogene. , (2012).
- Knudsen, R. C., Ahmed, A. A., Sell, K. W. An in vitro microassay for lymphotoxin using microculture plates and the multiple automated sample harvester. Journal of Immunological Methods. 5, 55-63 (1974).
- Beck, H. P. Proliferation kinetics of perturbed cell populations determined by the bromodeoxyuridine-33258 technique: radiotoxic effects of incorporated [3H]thymidine. Cytometry. 2, 170-174 (1981).
- Miller, M. J., Wei, S. H., Parker, I., Cahalan, M. D. Two-photon imaging of lymphocyte motility and antigen response in intact lymph node. Science. 296, 1869-1873 (2002).
- Collins, J. M., Berry, D. E., Bagwell, C. B. Different rates of DNA synthesis during the S phase of log phase HeLa S3, WI-38, and 2RA cells. Journal of Biological Chemistry. 255, 3585-3590 (1980).
- Schwarting, R., Gerdes, J., Niehus, J., Jaeschke, L., Stein, H. Determination of the growth fraction in cell suspensions by flow cytometry using the monoclonal antibody Ki-67. Journal of Immunological Methods. 90, 65-70 (1986).
- Danova, M., et al. Cell cycle-related proteins: a flow cytofluorometric study in human tumors. Biology of the Cell. 64, 23-28 (1988).
- Juan, G., et al. Histone H3 phosphorylation and expression of cyclins A and B1 measured in individual cells during their progression through G2 and mitosis. Cytometry. 32, 71-77 (1998).
- Langan, T. J., Chou, R. C. Synchronization of mammalian cell cultures by serum deprivation. Methods in Molecular Biology. 761, 75-83 (2011).
- Kues, W. A., et al. Cell cycle synchronization of porcine fetal fibroblasts: effects of serum deprivation and reversible cell cycle inhibitors. Biology of Reproduction. 62, 412-419 (2000).
- Urbani, L., Sherwood, S. W., Schimke, R. T. Dissociation of nuclear and cytoplasmic cell cycle progression by drugs employed in cell synchronization. Experimental Cell Research. 219, 159-168 (1995).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유