Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Usando un dispositivo de microfluidos para la estimulación mecánica y alta resolución de imagen de C. elegans
* Estos autores han contribuido por igual
En este artículo
Resumen
Nuevas herramientas para la investigación de la mecanobiología se necesitan entender cómo mecánico estrés activa vías bioquímicas y produce respuestas biológicas. Aquí, nos muestra un nuevo método de estimulación mecánica selectiva de animales inmovilizados con una trampa de microfluidos que permite proyección de imagen de alta resolución de las respuestas celulares.
Resumen
Un objetivo central de la mecanobiología es entender el efecto recíproco del estrés mecánico sobre las proteínas y las células. A pesar de su importancia, la influencia del estrés mecánico sobre la función celular es todavía mal entendida. En parte, esta brecha de conocimiento existe porque algunas herramientas permiten deformación simultánea de tejidos y células, la proyección de imagen de la actividad celular en los animales vivos y eficiente de movilidad en organismos modelo lo contrario altamente móviles, tales como el nematodo Elegans de Caenorhabditis. El tamaño pequeño de C. elegans hace un excelente partido a los dispositivos de investigación basada en microfluídica, y se presentan soluciones para la inmovilización con dispositivos microfluídicos. Aunque estos dispositivos permiten obtener imágenes de alta resolución, el animal está completamente dentro de polidimetilsiloxano (PDMS) y el vidrio, limita el acceso físico para la entrega de fuerza mecánica o las grabaciones electrofisiológicas. Recientemente, hemos creado un dispositivo que integra los actuadores neumáticos con un diseño de captura compatible con microscopía de fluorescencia de alta resolución. El canal de actuación se separa del canal atrapa el gusano por una delgada membrana PDMS. Este diafragma se encuentra en el lado de un gusano aplicando presión desde una fuente externa. El dispositivo puede dirigirse a las neuronas individuales mechanosensitive. La activación de estas neuronas es fotografiada en alta resolución con indicadores calcio codificados genéticamente. Este artículo presenta el método general utilizando cepas de C. elegans expresando su indicador de actividad de calcio-sensible (GCaMP6s) en sus neuronas de receptor del tacto (TRNs). El método, sin embargo, no se limita a TRNs ni sensores de calcio como una punta de prueba, pero puede ser ampliado a otras células mecánicamente sensibles o sensores.
Introducción
El sentido del tacto ofrece animales información crucial acerca de su entorno. Dependiendo de la fuerza aplicada, touch es percibida como inocua, placentera o dolorosa. La deformación del tejido durante el tacto es detectada por células especializadas mechanoreceptor incrustadas en la piel que expresan proteínas del receptor, comúnmente canales del ion. Los pasos a fuerza de percepción a la activación de canal de iones durante el tacto y el dolor no se entienden completamente. Menos aún se sabe sobre cómo el tejido de la piel filtros de deformación mecánica y si mecanorreceptores detectan cambios en la tensión o estrés1,2,3. Esta brecha en la comprensión se presenta, en parte, de la falta de herramientas adecuadas para aplicar estímulos mecánicos precisos a la superficie de la piel de un animal vivo observando las respuestas a nivel celular. Mientras que la microscopía de fuerza atómica se ha utilizado extensivamente para aplicar y medir fuerzas en células aisladas4,5 y activan los receptores Piezo1 en la vida de las células6, experimentos similares usando animales vivos, especialmente C. elegans, han sido notoriamente difícil debido a la movilidad intrínseca del sujeto. Este desafío es eludido tradicionalmente mediante el uso de pegamento del cianocrilato de grado quirúrgico o veterinario para inmovilizar animales individuales en agar cojines1,7,8,9. Este enfoque ha sido productivo, pero tiene limitaciones relacionadas con la habilidad necesaria para la inmovilización por encolado y la superficie del agar suave sobre el cumplimiento de la mecánica. Una estrategia de la microfluídica es una alternativa gratuita que evita algunas de las complicaciones vinculadas a pegar.
El nematodo C. elegans es un organismo modelo genético con un sistema nervioso completamente asignado que, debido al tamaño del animal, es un buen ajuste para la tecnología de microfluídica. Dispositivos de microfluidos ofrece la ventaja que los animales lo contrario extremadamente móviles pueden ser refrenados mientras se realiza la proyección de imagen de alta resolución y entrega de los estímulos neuro-modulador pertinentes. Con la ayuda de microfluidos tecnologías, animales vivos pueden ser inmovilizadas sin daño10,11, que permite el monitoreo de la actividad conductual sobre la vida de12,13 y alta resolución proyección de imagen de la actividad neuronal14,15,16,17. Además, muchas neuronas de mechanoreceptor necesarias para el sentido del tacto y del dolor puede ser caracterizado en su fisiológico1,8, mecánico4,18,19y molecular nivel20,21,22.
C. elegans detecta estímulos mecánicos suaves a la pared del cuerpo usando seis TRNs, tres de los cuales inervan el animal anterior (ALML más baja/R y AVM) y tres de los cuales inervan la parte posterior del animal (PLML/R y PVM). Las moléculas de canal de iones necesarias para la transducción de una fuerza aplicada en una señal bioquímica se han estudiado ampliamente en su TRNs8. Este artículo presenta una plataforma de microfluidos23 que permite a los investigadores a aplicar fuerzas mecánicas precisas a la piel de un inmovilizado de C. elegans ascáride, mientras leía a la deformación de los tejidos internos por proyección de imagen óptica. Además de presentar estímulos mecánicos bien definidos, transitorios de calcio pueden ser grabados en las neuronas mechanoreceptor con resolución subcelular y correlacionados con características morfológicas y anatómicas. El dispositivo consta de un canal central reventado que tiene un solo animal y presenta su piel junto a seis canales de accionamiento neumático (figura 1 y figura 2). Los seis canales se colocan a lo largo del canal de captura para entregar estímulos mecánicos a cada uno de seis TRNs del gusano. Estos canales están separados de la cámara de captura por finas membranas PDMS, que pueden ser impulsados por una fuente de presión de aire externa (figura 1). Calibrado de la desviación con respecto a la presión y proporcionar las medidas en este artículo. Cada actuador puede ser abordado individualmente y utilizado para estimular la mechanoreceptor de elección. La presión se suministra con una bomba de presión piezo-conducido pero puede utilizarse cualquier dispositivo alternativo. Mostramos que el protocolo de presión puede utilizarse para activar TRNs en vivo y demostrar funcionamiento dispositivos adecuados para entregar estímulos mecánicos para adultos C. elegans, carga de animales adultos en dispositivos, realizar proyección de imagen del calcio experimentos y analizar los resultados. Fabricación de dispositivo consta de dos pasos principales: 1) Fotolitografía para hacer un molde de SU-8; y 2) PDMS para hacer un dispositivo de moldeo. Aras de la brevedad y claridad, los lectores se conocen previamente publicados artículos y protocolos24,25 para obtener instrucciones sobre cómo producir los moldes y dispositivos.
Protocolo
1. fabricación de dispositivo
- Descargar el archivo adjunto la máscara (1 archivo suplementario) y generar una máscara de cromo con un servicio comercial o servicio interno. Como la dimensión más pequeña en el dispositivo es el μm 10 (espesor de la membrana del actuador), asegúrese de que la máscara tiene una resolución lo suficientemente alta, dentro de ± 0.25 μm, producir confiablemente las características.
-
Seguir métodos de fotolitografía estándar SU-8 (p. ej., hace referencia a24,25,26) para fabricar el molde para la posterior producción de dispositivos PDMS; un resumen de los pasos se enumera a continuación.
Nota: SU-8 es un material fotosensible, que vínculos cruzados sobre la exposición a la luz ultravioleta (máxima absorción ~ 365 nm). Utilice las instrucciones del fabricante como punto de partida para determinar los parámetros de procesamiento para el soft-cuece al horno, exposición (UV) y después de hornear.- Depositar SU-8 2002 en una oblea de silicio y una capa de la vuelta a un espesor aproximado de 2 μm, para mejorar la adherencia de pequeñas estructuras. Suave-cueza al horno en una placa caliente durante 1 minuto a 95 ° C, ligeramente sobre-exponer superficie (Ultravioleta) el entero (~ 100 mJ/cm2) y después cocer al horno en una placa caliente durante 2 min a 95 ° C.
- Spin-capa una capa gruesa de 47 μm de 2050 SU-8 para la definición de las características del dispositivo. Utilizar una velocidad de centrifugado de 500 rpm por 15 s (130 rpm/s aceleración) y entonces 2.000 rpm durante 90 s (260 rpm/s aceleración). Si es necesario, repita y ajustar la velocidad de giro para conseguir el grosor adecuado de photoresist.
- Suave-cueza al horno en una placa caliente por 2 min a 65 ° C y luego de 7 min a 95 ° C. Exponer el photoresist según las instrucciones del fabricante (~ 160 mJ/cm2) con un filtro de máscara y pase de largo de chrome para las paredes laterales rectas.
- Retirar la oblea y la cueza al horno en una placa caliente durante 1 min a 65 ° C y 7 min a 95 ° C.
- Disolver una SU-8 con el desarrollador y limpiar con 2-propanol.
- Cueza al horno la oblea en la placa caliente a 180 ° C por 30 min (duro-cueza al horno) estabilizar la fotoresistencia características.
-
Hacer una réplica PDMS del modelo SU-8 usando réplica estándar de métodos27de moldeo.
- Tratar el molde de SU-8 con el vapor de trichloromethylsilane (TCMS) para reducir la adherencia PDMS (silanización).
PRECAUCIÓN: TCMS es tóxicos y reactivos con el agua.- Colocar la oblea con motivos en un estante de la oblea dentro de un desecador de vacío del frasco en una campana de humos libre de agua o de reactivos solubles en agua.
- Bajo el capó, utilice un gotero para aplicar 1 gota de TCMS para un plato de vidrio y coloque dentro del desecador.
- Cierre la tapa de Desecador y permiten TCMS vapor cubrir la oblea para por lo menos 20 minutos.
- De ventilación y a continuación, abra el desecador. Abra la tapa de la campana de cristal y retirar la oblea con unas pinzas de plástico. Coloque en un barco de papel aluminio o placa de Petri para el moldeado de réplica PDMS. Disponer de materiales recubiertos de TCMS en el adecuada residuos peligrosos.
- Mezcla de PDMS (proporción 10:1) utilizando 40 g de la base del polímero y 4 g PDMS agente endurecedor.
- Vierta la mezcla sobre el molde de SU-8 y desgasificar la mezcla durante al menos 30 minutos en una cámara de vacío.
- La cura por al menos 6 horas a 70 ° C en un horno.
- Tratar el molde de SU-8 con el vapor de trichloromethylsilane (TCMS) para reducir la adherencia PDMS (silanización).
- Levante la PDMS la oblea la oblea horizontal sobre una superficie plana y con cuidado, despegando el PDMS. No doble la oblea hacia arriba durante este paso, como puede romper el molde.
Nota: Adherencia incompleta de SU-8 a la oblea o silanización incompleta puede resultar en la destrucción de las estructuras SU-8 durante este paso. - Cortar PDMS alrededor de los dispositivos de fichas individuales que los dispositivos se ajustan en un cubreobjetos de 24 x 60 mm.
- Con un punzón de biopsia de 1 mm, hacer agujeros en la entrada, dos salidas y seis actuadores con un punzón de biopsia de 1 milímetro. Objetivo de los 2 mm de diámetro círculos alrededor de los bordes del dispositivo.
-
Virutas de PDMS de Bond de cubreobjetos de vidrio.
- Exponer ambas superficies para plasma de oxígeno de 80 W (30 s).
- Coloque suavemente la superficie PDMS expuesta en la superficie expuesta de la cubierta para un sello conformal.
- Recueza el dispositivo sobre una placa caliente (100 ° C, 10 minutos).
Nota: Vinculación insuficiente puede conducir a la falla del dispositivo debido a la fuga.
2. preparación del microscopio
Nota: Animales transgénicos: expresar un indicador de calcio como GCaMP6s28 u otra sonda actividad genéticamente codificados en el neuron(s) de interés (por ejemplo, TRN); Co expresan una proteína fluorescente de actividad independiente emite en una longitud de onda diferentes para corregir pequeñas laterales y artefactos de movimiento fuera de enfoque que surgen debido a la estimulación mecánica. El software de análisis automatizado de las pistas y compensa los cambios inducidos por el movimiento en la intensidad de la sonda de la actividad. El gusano cepas GN69223 (o AQ323629) expresa GCaMP6s (o GCaMP6m) y el tagRFP de calcio-independiente bajo el control del promotor del mec-7. Además, GN692 contiene la mutación lite-1(ce314), que impide la activación de TRNs debido al sensor de luz azul lite 130 durante la excitación de la fluorescencia de GCaMP6s23.
-
Establecer un sistema de microscopio para la excitación simultánea de GCaMP y solicitud de propuestas.
- Use ya sea una continua fuente de luz y un filtro de excitación de banda dual que transmite la luz sólo cian y amarillo o una fuente de luz que emite solamente longitudes de onda en un ancho de banda definido como cian simultánea (0,77 mW) y amarillo (1,21 mW) fuentes de excitación de LED para excitación simultánea de la fluorescencia dependiente de calcio e independiente, respectivamente.
- Utilizar una cámara digital para habilitar la grabación de imágenes de microscopía.
Nota: La mayoría de las cámaras cuentan con un software para la adquisición de la imagen. Alternativomente, hay software libre disponible que puede utilizarse para controlar la cámara y posiblemente también otras partes del sistema de microscopía. - Ajustar la intensidad de excitación basada en la intensidad de la fluorescencia para evitar la saturación de la cámara.
-
Utilice un cubo de fluorescencia que dependiendo de la elección de la fuente de excitación debe estar equipada con un filtro de la excitación.
Nota: Un GFP de 488 nm y 580 nm mCherry filtro de la excitación fue utilizado aquí.- Añadir un divisor de haz al cubo para reflejar luz cian y amarillo y transmitir luz verde y roja.
- Equipar al microscopio con 10 X objetivo y un objetivo de alta magnificación (p. ej., 63 X / 1.32 aceite NA) para enfocar la luz sobre la muestra de la excitación.
- Montar un divisor de haz frente a la cámara para la grabación simultánea de la GCaMP y la señal de calcio-independiente. Asegúrese de que el divisor de viga tiene un espejo dicroico (paso de largo, corte a 570 nm) para separar la luz verde y rojo utilizando un filtro de emisión verde (pasabanda centrado en 525 nm 50 ancho nm) y filtro de una emisión de luz roja (pasabanda centrado en 632 nm con 60 nm ancho).
- Proyecto fluorescencia verde y roja en la mitad superior (verde) y mitad inferior (rojo), respectivamente, de la cámara de la viruta (ver figura 3). Esta orientación es un prerrequisito para el software de análisis previsto.
3. animal preparación
- Preparar sincronizadas de edad joven adulto o adulto día uno C. elegans, como se describe anteriormente por Porta-de-la-Riva et al. 31
- Preparar el chip de microfluidos.
- Conecte el depósito de flujo de gravedad (~ 60 cm sobre el nivel del chip) que contiene filtrado buffer M9 (filtro de jeringa de 0,2 μm polyethersulfone) una salida de la viruta. Conectar la salida de otros a una salida de un contenedor de residuos de dos tomas de corriente, es decir, un matraz de filtración. Conecte la otra toma del contenedor de residuos a una bomba peristáltica.
Nota: Utilice tubería de polietileno (PE) para todas las conexiones y tubo de metal accesorios para conectar la tubería de PE para el chip. De esta manera todas las soluciones de residuos lavadas fuera de la viruta y recogidas en el contenedor de residuos. El flujo a través de los canales de salida crea una succión suave que mantendrá el gusano ajustada durante el proceso de captura más adelante. - Preparar interconexiones que consta de 50 mm largo PE tubería (0.9652 mm OD, 0.5842 mm ID) con tubería metálica (calibre tamaño 23TW, 0,635 mm OD) en ambos extremos. Press-fit estas interconexiones en cada uno de los seis dedos de actuación y en la entrada del gusano (ver figura 1). Deje estas interconexiones en el chip, como extracción repetida lleva a desgaste en los orificios PDMS.
- Conecte el depósito de flujo de gravedad (~ 60 cm sobre el nivel del chip) que contiene filtrado buffer M9 (filtro de jeringa de 0,2 μm polyethersulfone) una salida de la viruta. Conectar la salida de otros a una salida de un contenedor de residuos de dos tomas de corriente, es decir, un matraz de filtración. Conecte la otra toma del contenedor de residuos a una bomba peristáltica.
- Coloque el chip en el microscopio. Escoger de 2 a 5 gusanos en una gota de filtraron buffer M9 de (filtro de jeringa de 0,2 μm) y utilizan una jeringa de 1 mL para elaborar en una tubería de PE (0.9652 mm OD, 0.5842 mm ID) conectada a una jeringa de 1 mL, tirando el émbolo suavemente. Mantener los animales en el tubo de PE y no en la jeringa.
Nota: Muchos gusanos o soluciones sin filtrar en el chip pueden conducir a la obstrucción. - Conecte la tubería de PE de la jeringa a la interconexión en la entrada del gusano (figura 1 y figura 2) de la viruta. Activar el flujo por gravedad mediante la apertura de la válvula y encienda la bomba peristáltica. Ahora presione suavemente el émbolo de la jeringa para mover los animales en el canal de captura mientras observa el canal bajo el microscopio con objetivo 10 X en modo de brightfield.
Nota: de vez en cuando aparecen burbujas de aire no plantean un problema; generalmente automáticamente se mueven en el tomacorriente. Varios animales se pueden 'aparcados' en la sala de espera y utilizar secuencialmente.- Después de cargar el animal en el canal de captura (véase también figura 2), ajustar su posición suavemente empujando o tirando del émbolo de la jeringa a la posición de la cabeza del animal en la forma cónica de la parte delantera de la canal.
- Asegúrese de que el gusano (1 de día adulto) tiene el tamaño adecuado para ser atrapados en el chip.
- Si el gusano no llena la sección representativa entera del canal desde la nariz hasta cerca del final del cuerpo (no incluyendo la punta de la cola) o el gusano se mueve a lo largo del eje del canal sin mover el émbolo de la jeringa , retire el gusano presionando el émbolo de la jeringa hasta que desaparece desde el canal de captura y cargar uno nuevo (ver paso 3.8).
- Cambiar al modo de la fluorescencia del microscopio y una lente más alta (40 X o superior) y compruebe si la neurita de la neurona de interés viene a mentir a través de la membrana de uno de los actuadores. Si no es así, ajuste la posición del animal tirando o empujando el émbolo. Si eso no funciona, retire el gusano y cargar uno nuevo.
- Centrarse en el cuerpo celular de la neurona de interés y conecte la interconexión del actuador más cercana a la neurona en la parte anterior del cuerpo de la célula a una bomba de presión programable mediante tubo de PE.
- Si el experimento requiere medir la distancia entre el actuador y la neurona, mover el campo de visión, por lo tanto en el campo de visión y el channelwall es paralela al borde superior e inferior de la imagen.
- Definir un protocolo de presión mediante la bomba de presión programable.
Nota: Este protocolo se puede ajustar a la experiencia deseada.- Comience con una presión constante de 0 kPa durante un tiempo correspondiente a por lo menos 50 imágenes de la secuencia de imagen (necesaria para la normalización). Para una tasa de proyección de imagen de 10 Hz esto corresponde a 5 s. Añadir una forma de onda del estímulo deseado y la presión y definir la duración del estímulo como un segundo paso.
Nota: Para estimular el TRNs, una forma de onda sinusoidal (es decir, 75 kPa, 10 Hz) superpuesto con un paso (es decir, 275 kPa) es recomendable ya que genera una gran respuesta neuronal. - Si el experimento requiere estímulos adicionales, incluyen un periodo de al menos 10 s a una presión constante de 0 kPa entre estímulos. Antes de encender la bomba de presión, asegúrese de que la presión del instrumento está en kPa 0 constante evitar la estimulación accidental del gusano antes del experimento real.
- Comience con una presión constante de 0 kPa durante un tiempo correspondiente a por lo menos 50 imágenes de la secuencia de imagen (necesaria para la normalización). Para una tasa de proyección de imagen de 10 Hz esto corresponde a 5 s. Añadir una forma de onda del estímulo deseado y la presión y definir la duración del estímulo como un segundo paso.
- Ejecutar el protocolo de la proyección de imagen y presión.
- En el software de adquisición de imagen de la cámara, establecer una secuencia de imágenes de 10 Hz con un tiempo de exposición de 100 ms para la duración del Protocolo de presión. Ajustar la intensidad de excitación tal que el aumento de la fluorescencia máxima no saturar la cámara. Guardar las imágenes como un archivo *.tif con al menos 50 imágenes ante el primer estímulo para la normalización.
- Iniciar el protocolo de imagen en el software de adquisición de la imagen y el protocolo de la presión en el software de la bomba programable. Durante la grabación, observar si la neurona de interés es el punto más brillante en un área de 10 x 10 pixeles, no se mueve más allá de 10 píxeles en imágenes secuenciales, se queda en el campo de visión durante la grabación.
Nota: Si no se hace, el software de análisis no. Puntos brillantes (es decir, autofluorescencia) alrededor de la neurona que las grabaciones de las neuronas difíciles de limpiar. Para corregir esto, sacar el gusano de la trampa y cargar un nuevo gusano en el chip. Si el gusano se mueve demasiado, prueba una nueva grabación del gusano mismo y observar el movimiento del gusano. Si el problema persiste el gusano podría ser demasiado pequeño para ser atrapados. En este caso sacar este gusano y carga un gusano ligeramente más grande en la trampa. - Si lo desea, grabar señales de las neuronas múltiples en el campo de visión al mismo tiempo siempre y cuando las neuronas están separadas por al menos 10 píxeles durante toda la grabación.
- Para investigar la habituación, realizar el experimento repetidamente en un animal.
- Retire el gusano desde el canal de captura.
- Si se desea mantener el gusano para estudios posteriores, desconectar las salidas del chip hacia el flujo de gravedad y el depósito de residuos. Presione suavemente el émbolo de la jeringa hasta que el gusano entero a través del canal de captura en el canal de flujo.
- Continúe presionando el émbolo de la jeringa hasta que el animal aparezca en una gota fuera de la viruta. Desconecte la jeringa como el tubo de PE de la entrada del gusano, usarlo para aspirar los gusanos en la gota y transferirlo a una placa de agar.
- Si no se desea mantener el gusano, sacar el gusano presionando el émbolo de la jeringa hasta que el gusano entero a través del canal de captura en el canal de flujo; el flujo por gravedad y la succión de la bomba peristáltica llevan el animal fuera de la viruta y en el depósito de residuos.
4. Análisis
- Descargar e instalar el más reciente Fiji versión32.
- Descargue e instale la versión más reciente del SDK de java.
Nota: Si es necesario, eliminar o renombrar la carpeta de java dentro de la carpeta de Fiji de Fiji utilizar el compilador de java recién instalada. -
Abra el software de Fiji.
- Compruebe si el software ejecuta el compilador de java recién instalada abriendo ' Plugins | Utilidades | ImageJ | Propiedades.
- Descargar el archivo pokinganalyzer*.java (https://github.com/HFehlauer/Poking-Analyzer, ver archivo adicional 2).
- Arrastrar y soltar el archivo. Java en la ventana del software para abrirlo como un plugin.
-
Compilar y ejecutar el archivo pokinganalyzer*.java presionando las teclas Ctrl + R en Fiji; una interfaz gráfica de usuario (meter analizador) abre (figura 3).
- Haga clic en la pestaña "Ayuda" y Lea acerca de los requisitos del software y cómo realizar el análisis. Para empezar, seleccione la ficha "Abrir video" especificar la ubicación del archivo de vídeo para el analizador: haga clic en el botón "Abrir un vídeo", desplácese a la ubicación del video y abrirlo.
Nota: El software comienza con esta pestaña abierta. Si no está abierto al iniciar un nuevo análisis haga clic en la pestaña "Abrir video". Si el video está en formato a.tif, el programa abre el vídeo y mostrar la primera imagen en la parte inferior de la interfaz internamente. Si la imagen no es del video que debe ser analizado, haga clic en el botón "Abrir un vídeo" para abrir otro video. - Para continuar, haga clic en la pestaña de "Definir ROIs". En esta pestaña, definir la posición de la TRN. Tenga en cuenta que la primera imagen del vídeo abierto se muestra en la parte inferior de esta ficha haga clic en el botón "Definir TRN" y haga clic en la neurona en la parte superior de la imagen visualizada que debe mostrar la fluorescencia GCaMP.
- Opcional: Si el actuador está visible en la mitad superior de la imagen el programa puede automáticamente rastrear la distancia entre la neurona y el actuador si se desea. Para esto, consulte "Definir el actuador?" -caja, haga clic en el botón "Definir actuador" y haga clic en el actuador en la mitad superior de la imagen.
- Haga clic en la ficha "Análisis" definir la tasa de adquisición de imagen. Escriba un número en el campo o haga clic en el arriba o abajo botones para aumentar o disminuir el número. Si está en la ficha anterior la "definir el actuador?" -casilla, definir el tamaño de celda de la cámara, el aumento y el factor binning, por lo que el programa puede calcular la distancia.
- Haga clic en el botón "Iniciar análisis".
Nota: El programa ahora seguimiento de la neurona por su fluorescencia en la secuencia de imágenes en la mitad superior (dependiente de calcio) y en la mitad inferior (independiente de calcio) de la secuencia de imágenes. Para tener en cuenta para el movimiento de la neurona en el plano de grabación del programa estudiará un área de 10 x 10 píxeles alrededor de la localización de la neurona en la imagen anterior para encontrar la posición de la neurona en la siguiente imagen. Calculará la fluorescencia en ambas mitades de la corrección para la fluorescencia de fondo (Fbg) y dividiendo por la fluorescencia en las primeras 50 imágenes (F0):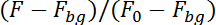
Cambios de la fluorescencia en el canal verde, dependiente de la actividad (FCa2 +) causadas por la neurona de movimiento fuera del plano de recodificación son entonces corrección mediante la fluorescencia en el canal rojo, independiente de la actividad (F corr) y la desviación estándar de pre-estímulo del calcio-dependiente (sdCa2 +) y (sdcorr) independiente de la fluorescencia por:
- El programa mostrará su progreso en la barra de estado en la parte inferior de la interfaz; Cuando la barra de estado llega al 100%, haga clic en la pestaña "Resultados". Si la barra de estado se detiene antes de 100%, el programa dará un mensaje de error indicando el motivo que el programa no podría analizar el video.
- Si la relación señal a ruido es demasiado baja, el programa no puede identificar la neurona; ajustar los parámetros de proyección de imagen en grabaciones posteriores.
- Si la neurona sale del campo de visión durante la grabación, el programa no puede seguir más y se detendrá. Para posteriores grabaciones, coloque la neurona en el medio del campo de visión al principio de la grabación.
- Si algunas regiones en el campo de visión están sobresaturados, el análisis automatizado producirá resultados no válidos. Para posteriores grabaciones, asegúrese de que la señal de fluorescencia no sature el sensor de la cámara.
- En la pestaña 'resultados', el resultado se muestra en la parte superior. Guardar un archivo *.txt ficha separada de la tabla haciendo clic en "Guardar la tabla de resultado".
Nota: Esta tabla consiste en el tiempo en segundos y la fluorescencia dependiente de calcio normalizada (corregido por la fluorescencia de calcio-independiente). Si previamente definido, también contiene la distancia total entre la TRN y el actuador en μm y la distancia entre la TRN y el actuador perpendicular a la pared del canal en μm. resultados representativos de la intensidad de la fluorescencia aumentan después de estimulación versus la distancia del cuerpo celular en el actuador más cercano se representan gráficamente en la figura 4. - Si hay múltiples neuronas en una grabación (separada por más de 10 píxeles), haga clic en la pestaña de "Definir ROIs" de nuevo y volver al paso 4.6.2 con la segunda neurona.
- Haga clic en la pestaña "Ayuda" y Lea acerca de los requisitos del software y cómo realizar el análisis. Para empezar, seleccione la ficha "Abrir video" especificar la ubicación del archivo de vídeo para el analizador: haga clic en el botón "Abrir un vídeo", desplácese a la ubicación del video y abrirlo.
Resultados
Litografía de SU-8 y la vinculación de la viruta
El protocolo de litografía y moldeado de PDMS siguen los procedimientos estándar. Detalles pueden encontrarse en otros lugares23,24,25,26. El PDMS debe despegarse de la ostia sin problemas después de curar. Si las características de SU-8 rip off durante la peladura de PDMS, la capa de adh...
Discusión
Este protocolo muestra un método para entregar precisa estimulación mecánica sobre la piel de un ascáride atrapado en un chip de microfluidos. Se pretende facilitar la integración de los estímulos físicos para responder a cuestiones biológicas y pretende agilizar investigación mecanobiología en laboratorios biológicos. Este método amplía análisis anteriores para evaluar la función de mechanosensory neuronas en C. elegans. Técnicas cuantitativas y semi cuantitativas anteriores miden fuerzas
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Sandra N. Manosalvas-Kjono, Ladpli de Purim, Farah Memon, Divya Gopisetty y Verónica Sánchez por apoyan en la generación de animales mutantes y diseño del dispositivo. Esta investigación fue apoyada por subvenciones de NIH R01EB006745 (Para BLP), R01NS092099 (a MBG), K99NS089942 (de MK), F31NS100318 (a ALN) y recibió financiación del Consejo Europeo de investigación (ERC) bajo investigación horizonte 2020 de la Unión Europea y la innovación programa () otorgar el acuerdo no. 715243 a MK).
Materiales
| Name | Company | Catalog Number | Comments |
| Chrome mask | Compugraphics (http://www.compugraphics-photomasks.com/) | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| Chrome mask | Mitani-Micronics (http://www.mitani-micro.co.jp/en/) | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| Chrome mask | Kuroda-Electric (http://www.kuroda-electric.eu/ | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| 4'' Silicon wafer (B-test) | Stanford Nanofabrication Facility | ||
| SU-8 2002 | MicroChem | ||
| SU-8 2050 | MicroChem | ||
| Spin-coater | Laurell Technologies | WS-400BZ-6NPP/LITE | |
| Exposure timer | Optical Associates, Inc | OAI 150 | |
| Illumination controller | Optical Associates, Inc | 2105C2 | |
| SU-8 developer | MicroChem | ||
| 2-Propanol | Fisher Scientific | A426F-1GAL | |
| Acetone | Fisher Scientific | A18-4 | |
| Trichloromethylsilane (TCMS) | Sigma-Aldrich | 92361-500ML | Caution: TCMS is toxic and water-reactive |
| Sylgard 184 Elastomer Kit | Dow Corning | PDMS prepolymer | |
| Biopsy punch, 1 mm | VWR | 95039-090 | |
| Oxygen Plasma Asher | Branson/IPC | ||
| Small metal tubing (0.635 mm OD, 0.4318 mm ID, 12.7 mm long); gage size 23TW | New England Small Tube Corporation | NE-1300-01 | |
| Nalgene syringe filter, 0.22 μm | Thermo Scientific | 725-2520 | to filter all solution, small particles would clog the chip |
| Polyethylene tubing; 0.9652 mm OD, 0.5842 mm ID | Solomon Scientific | BPE-T50 | |
| Syringe, 1 ml | BD Scientific | 309628 | for worm trapping and release |
| Syringe, 20 ml | BD Scientific | 309661 | for gravity-based flow |
| Gilson Minipuls 3, Peristaltic pump | Gilson | to suck solutions and worms out of the chip | |
| Microfluidic flow controller, equipped with 0–800 kPa pressure channel | Elveflow | OB1 MK3 | pressure delivery |
| Water-Resistant Clear Poly- urethane Tubing, 4 mm ID and 6 mm OD | McMaster-Carr | 5195 T52 | connection from house air to pressure pump |
| Water-Resistant Clear Polyurethane Tubing, 2.6mm ID and 4mm OD | McMaster-Carr | 5195 T51 | connect pressure pump to small tubng |
| Push-to-Connect Tube Fitting for Air | McMaster-Carr | 5111K468 | metric - imperial converter |
| Straight Connector for 6 mm × 1/4″ Tube OD | McMaster-Carr | 5779 K258 | |
| Leica DMI 4000 B microscopy system | Leica | ||
| 63×/1.32 NA HCX PL APO oil objective | Leica | 506081 | |
| Hamamatsu Orca-Flash 4.0LT digital CMOS camera | Hamamatsu | C11440-42U | |
| Lumencor Spectra X light engine | Lumencor | With cyan and green/yellow light source | |
| Excitation beam splitter | Chroma | 59022bs | in the microscope |
| Hamamatsu W-view Gemini Image splitting optics | Hamamatsu | A12801-01 | to split green and red emission and project them on different areas on the camera chip |
| Emission beam splitter | Chroma | T570lpxr | in the image splitter |
| Emission filters GCamp6s | Chroma | ET525/50m | in the image splitter |
| Emission filters mCherry | Chroma | ET632/60m | in the image splitter |
Referencias
- Eastwood, A. L., et al. Tissue mechanics govern the rapidly adapting and symmetrical response to touch. Proc. Natl. Acad. Sci. 15 (50), E6955-E6963 (2015).
- Katta, S., Krieg, M., Goodman, M. B. Feeling Force: Physical and Physiological Principles Enabling Sensory Mechanotransduction. Annu. Rev. Cell Dev. Biol. 31, 347-371 (2015).
- Krieg, M., Dunn, A. R., Goodman, M. B. Mechanical systems biology of C. elegans touch sensation. BioEssays. 37 (3), 335-344 (2015).
- Krieg, M., Dunn, A. R., Goodman, M. B. Mechanical control of the sense of touch by β-spectrin. Nat. Cell Biol. 16 (3), 224-233 (2014).
- Krieg, M., et al. Tensile forces govern germ-layer organization in zebrafish. Nat Cell Biol. 10 (4), 429-436 (2008).
- Gaub, B. M., Müller, D. J. Mechanical stimulation of Piezo1 receptors depends on extracellular matrix proteins and directionality of force. Nano Lett. 17 (3), 2064-2072 (2017).
- Geffeney, S. L., et al. DEG/ENaC but not TRP channels are the major mechanoelectrical transduction channels in a c. Elegans nociceptor. Neuron. 71 (5), 845-857 (2011).
- O'Hagan, R., Chalfie, M., Goodman, M. B. The MEC-4 DEG/ENaC channel of Caenorhabditis elegans touch receptor neurons transduces mechanical signals. Nat. Neurosci. 8 (1), 43-50 (2005).
- Suzuki, H., et al. In Vivo Imaging of C. elegans Mechanosensory Neurons Demonstrates a Specific Role for the MEC-4 Channel in the Process of Gentle Touch Sensation. Neuron. 39 (6), 1005-1017 (2003).
- Kopito, R. B., Levine, E. Durable spatiotemporal surveillance of Caenorhabditis elegans response to environmental cues. Lab Chip. 14 (4), 764-770 (2014).
- Chokshi, T. V., Ben-Yakar, A., Chronis, N. CO2 and compressive immobilization of C. elegans on-chip. Lab Chip. 9 (1), 151 (2009).
- Hulme, S. E., Shevkoplyas, S. S., McGuigan, A. P., Apfeld, J., Fontana, W., Whitesides, G. M. Lifespan-on-a-chip: microfluidic chambers for performing lifelong observation of C. elegans. Lab Chip. 10 (5), 589-597 (2010).
- Li, S., Stone, H. a., Murphy, C. T. A microfluidic device and automatic counting system for the study of C. elegans reproductive aging. Lab Chip. 15 (2), 524-531 (2015).
- Chokshi, T. V., Bazopoulou, D., Chronis, N. An automated microfluidic platform for calcium imaging of chemosensory neurons in Caenorhabditis elegans. Lab Chip. 10 (20), 2758-2763 (2010).
- Mishra, B., et al. Using microfluidics chips for live imaging and study of injury responses in Drosophila larvae. J. Vis. Exp. , e50998 (2014).
- Chronis, N., Zimmer, M., Bargmann, C. I. Microfluidics for in vivo imaging of neuronal and behavioral activity in Caenorhabditis elegans. Nat. Methods. 4 (9), 727-731 (2007).
- Krajniak, J., Lu, H. Long-term high-resolution imaging and culture of C. elegans in chip-gel hybrid microfluidic device for developmental studies. Lab Chip. 10 (14), 1862-1868 (2010).
- Vasquez, V., Krieg, M., Lockhead, D., Goodman, M. B. Phospholipids that Contain Polyunsaturated Fatty Acids Enhance Neuronal Cell Mechanics and Touch Sensation. CellReports. 6 (1), 70-80 (2013).
- Krieg, M., et al. Genetic defects in β-spectrin and tau sensitize C. elegans axons to movement-induced damage via torque-tension coupling. Elife. 6 (2010), e20172 (2017).
- Arnadóttir, J., O'Hagan, R., Chen, Y., Goodman, M. B., Chalfie, M. The DEG/ENaC protein MEC-10 regulates the transduction channel complex in Caenorhabditis elegans touch receptor neurons. J. Neurosci. 31 (35), 12695-12704 (2011).
- Lockhead, D., et al. The tubulin repertoire of Caenorhabditis elegans sensory neurons and its context-dependent role in process outgrowth. Mol. Biol. Cell. 27 (23), 3717-3728 (2016).
- Goodman, M. B., Ernstrom, G. G., Chelur, D. S., O'hagan, R., Yao, C. A., Chalfie, M. MEC-2 regulates C. elegans DEG/ENaC channels needed for mechanosensation. Nature. 415 (6875), 1039-1042 (2002).
- Nekimken, A., Fehlauer, H., Kim, A., Goodman, M., Pruitt, B. L., Krieg, M. Pneumatic stimulation of C. elegans mechanoreceptor neurons in a microfluidic trap. Lab Chip. , (2017).
- Brower, K., White, A. K., Fordyce, P. M. Multi-step Variable Height Photolithography for Valved Multilayer Microfluidic Devices. J. Vis. Exp. (119), e55276 (2017).
- Jenkins, G. Rapid prototyping of PDMS devices using SU-8 lithography. Methods Mol. Biol. 949 (1), 153-168 (2013).
- Faustino, V., Catarino, S. O., Lima, R., Minas, G. Biomedical microfluidic devices by using low-cost fabrication techniques: A review. J. Biomech. 49 (11), 2280-2292 (2016).
- Xia, Y., Whitesides, G. M. SOFT LITHOGRAPHY. Annu. Rev. Mater. Sci. 28 (1), 153-184 (1998).
- Chen, T. -. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Cho, Y., Porto, D., Hwang, H., Grundy, L., Schafer, W. R., Lu, H. Automated and controlled mechanical stimulation and functional imaging in vivo in C. elegans. Lab Chip. , (2017).
- Edwards, S. L., et al. A novel molecular solution for ultraviolet light detection in Caenorhabditis elegans. PLoS Biol. 6 (8), 1715-1729 (2008).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Cerón, J. Basic Caenorhabditis elegans methods: synchronization and observation. J. Vis. Exp. (64), e4019 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat. Methods. 9 (7), 676-682 (2012).
- Cho, Y., Porto, D. A., Hwang, H., Grundy, L. J. High-Throughput Controlled Mechanical Stimulation and Functional Imaging In Vivo. BiorXiv. , (2017).
- Petzold, B. C., Park, S. -. J., Mazzochette, E. A., Goodman, M. B., Pruitt, B. L. MEMS-based force-clamp analysis of the role of body stiffness in C. elegans touch sensation. Integr. Biol. (Camb). 5 (6), 853-864 (2013).
- Nekimken, A. L., Mazzochette, E. A., Goodman, M. B., Pruitt, B. L. Forces applied during classical touch assays for Caenorhabditis elegans. PLoS One. 12 (5), e0178080 (2017).
- Chronis, N., Zimmer, M., Bargmann, C. I. Microfluidics for in vivo imaging of neuronal and behavioral activity in Caenorhabditis elegans. Nat. Methods. 4 (9), 727-731 (2007).
- Gilleland, C. L., Rohde, C. B., Zeng, F., Yanik, M. F. Microfluidic immobilization of physiologically active Caenorhabditis elegans. Nat. Protoc. 5 (12), 1888-1902 (2010).
- Chuang, H. -. S., Raizen, D. M., Lamb, A., Dabbish, N., Bau, H. H. Dielectrophoresis of Caenorhabditis elegans. Lab Chip. 11 (4), 599 (2011).
- Christensen, A. M., Chang-Yen, D. A., Gale, B. K. Characterization of interconnects used in PDMS microfluidic systems. J. Micromechanics Microengineering. 15 (5), 928-934 (2005).
- Gilpin, W., Uppaluri, S., Brangwynne, C. P. Worms under Pressure: Bulk Mechanical Properties of C. elegans Are Independent of the Cuticle. Biophys. J. 108 (8), 1887-1898 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados