É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Utilizando um dispositivo microfluídica para estimulação mecânica e alta resolução de imagem de c. elegans
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Novas ferramentas para pesquisa de mechanobiology são necessários para compreender o estresse mecânico como ativa vias bioquímicas e elicia respostas biológicas. Aqui, vamos mostrar um novo método para a estimulação mecânica seletiva dos animais imobilizados com uma armadilha de microfluidic permitindo imagens de alta resolução de respostas celulares.
Resumo
Um objetivo central da mechanobiology é entender o efeito recíproco de estresse mecânico em proteínas e células. Apesar de sua importância, a influência do estresse mecânico na função celular é ainda mal compreendida. Em parte, essa lacuna de conhecimento existe porque alguns ferramentas permitem a deformação simultânea de tecidos e células, imagens da atividade celular em animais vivos e eficiente restrição da motilidade em organismos-modelo caso contrário altamente móveis, tais como o nematódeo Caenorhabditis elegans. O pequeno tamanho da c. elegans -los faz um excelente partido para dispositivos de microfluídica-com base em pesquisa, e foram apresentadas soluções para imobilização usando dispositivos microfluídicos. Embora estes dispositivos permitem imagens de alta resolução, o animal é totalmente envolto em polydimethylsiloxane (PDMS) e vidro, limitando o acesso físico para entrega de força mecânica ou eletrofisiológicas gravações. Recentemente, criamos um dispositivo que integra actuadores pneumáticos com um design de captura compatível com microscopia de fluorescência de alta resolução. O canal de atuação é separado pelo canal da interceptação de verme por uma fina membrana PDMS. Este diafragma é desviada para o lado de um verme aplicando pressão de uma fonte externa. O dispositivo pode direcionar os neurônios mechanosensitive individuais. A ativação desses neurônios é fotografada em alta resolução com indicadores de cálcio geneticamente codificado. Este artigo apresenta o método geral usando cepas de c. elegans , expressando o indicador de atividade de cálcio sensíveis (GCaMP6s) em seus neurônios receptores de toque (TRNs). O método, no entanto, não está limitado aos TRNs nem aos sensores de cálcio como uma sonda, mas pode ser expandido para outras células mecanicamente sensíveis ou sensores.
Introdução
O sentido do tato fornece animais com informações cruciais sobre seu ambiente. Dependendo da força aplicada, o toque é percebido como inócuo, prazeroso ou doloroso. A deformação do tecido durante o toque é detectada pelas células especializadas Mecanorreceptor incorporadas na pele que expressam proteínas do receptor, mais comumente canais iônicos. As etapas ligando a percepção da força para ativação de canal de íon durante o toque e a dor não são totalmente compreendidas. Menos ainda se sabe sobre como o tecido da pele filtra deformação mecânica e se mecanorreceptores detectam alterações na tensão ou stress1,2,3. Esta lacuna na compreensão decorre, em parte, a falta de ferramentas adequadas para aplicar estímulos mecânicos precisos para a superfície da pele de um animal vivo enquanto observa as respostas a nível celular. Considerando que a microscopia de força atômica tem sido amplamente utilizada para aplicar e medir forças em células isoladas de4,5 e também para ativar receptores Piezo1 em vida células6, experimentos semelhantes usando animais vivos, especialmente C. elegans, têm sido notoriamente desafiadora devido a mobilidade intrínseca do assunto. Tradicionalmente, este desafio é contornado usando cola de cianoacrilato de veterinária - ou -grau cirúrgico para imobilizar animais individuais em ágar almofadas1,7,8,9. Esta abordagem tem sido produtiva, mas tem limitações relacionadas com a habilidade necessária para a imobilização por colagem e superfície do agar macio na conformidade mecânica. Uma estratégia de microfluídica é uma alternativa gratuita que evita algumas das complicações ligadas à colagem.
O nemátodo c. elegans é um organismo modelo genético cujo sistema nervoso completamente mapeado que, devido ao tamanho do animal, é uma boa opção para tecnologia microfluídica. Oferta de dispositivos baseados em microfluídica a vantagem que os animais caso contrário extremamente móveis podem ser contidos durante a execução de imagens de alta resolução e entrega de estímulos neuro-moduladora relevantes. Com a ajuda de microfluidic tecnologias, vivem animais podem ser imobilizadas sem dano10,11, permitindo o monitoramento da atividade comportamental ao longo da vida inteira de12,13 e de alta resolução imagem de atividade neuronal14,15,16,17. Além disso, muitos neurônios Mecanorreceptor necessários para o sentido de toque e a dor pode ser caracterizado em sua fisiológicas1,8, mecânica4,18,19e molecular nível20,21,22.
C. elegans sentidos suaves estímulos mecânicos para sua parede do corpo usando seis TRNs, três dos quais inervam do animal anterior (ALML/R e AVM) e três dos quais inervam posterior do animal (PLML/R e PVM). As moléculas de canal de iões necessárias para transducing uma força aplicada em um sinal de bioquímico têm sido muito estudadas em seu TRNs8. Este artigo apresenta uma plataforma microfluidic23 que permite que os investigadores a aplicar forças mecânicas precisas na pele de um imobilizado c. elegans lombriga, ao ler a deformação de seus tecidos internos pela imagem latente ótica. Além de apresentar estímulos mecânicos bem definidos, transientes de cálcio podem ser gravados em neurônios Mecanorreceptor com resolução subcellular e correlacionados com características morfológicas e anatômicas. O dispositivo consiste de um canal central de interceptação que mantém um único animal e apresenta sua pele ao lado de seis canais de acionamento pneumático (Figura 1 e Figura 2). Os seis canais estão posicionados ao longo do canal de armadilhas para entregar a estímulos mecânicos para cada um dos seis TRNs do worm. Estes canais são separados da câmara de captura por diafragmas PDMS finos, que podem ser conduzidos por uma fonte de pressão do ar externo (Figura 1). Nós calibrado a deflexão em relação a pressão e fornecer as medições neste artigo. Cada atuador pode ser abordada individualmente e usado para estimular um Mecanorreceptor de escolha. A pressão é fornecida usando uma bomba de pressão piezo-driven, mas qualquer dispositivo alternativo pode ser usado. Nós mostramos que o protocolo de pressão pode ser usado para ativar TRNs na vivo e demonstrar o funcionamento dispositivos adequados para entrega de estímulos mecânicos para adultos c. elegans, carregar animais adultos em dispositivos, realizando a imagem latente de cálcio experiências e analisando os resultados. Fabricação de dispositivo consiste de duas etapas principais: 1) fotolitos para fazer um molde de SU-8; e 2) PDMS para tornar um dispositivo de moldagem. Por uma questão de brevidade e clareza, os leitores são referidos anteriormente publicados artigos e protocolos24,25 para obter instruções sobre como produzir os moldes e dispositivos.
Protocolo
1. dispositivo fabricação
- Baixe o arquivo de máscara (arquivo suplementar 1) e gerar uma máscara de cromo usando um serviço comercial ou instalações in-house. Como a menor dimensão no dispositivo é 10 µm (espessura de membrana do atuador), certifique-se de que a máscara tem resolução suficientemente alta, dentro de ± 0,25 µm, para produzir os recursos.
-
Seguir métodos de fotolitografia padrão SU-8 (por exemplo, referências24,25,26) para fabricar o molde para a posterior produção de dispositivos PDMS; um resumo das etapas está listado abaixo.
Nota: SU-8 é um material fotossensível, quais as referências cruzadas após exposição à luz ultravioleta (absorção máxima ~ 365 nm). Use as instruções do fabricante como ponto de partida para determinar os parâmetros de processamento para o macio-Asse, exposição (UV) e pós-asse.- Depósito de SU-8 2002 sobre uma bolacha de silício e rotação-casaco para uma espessura aproximada de 2 µm, para uma melhor aderência de pequenas estruturas. Macio-asse em uma chapa quente por 1 min a 95 ° C, um pouco over expor a superfície (UV) todo o (~ 100 mJ/cm2) e pós-asse em uma chapa quente por 2 min a 95 ° C.
- Rotação-casaco uma camada espessa de 47-µm de 2050 SU-8 para definir as características do dispositivo. Use uma velocidade de rotação de 500 rpm por 15 s (aceleração de 130 rpm/s) e em seguida 2.000 rpm para 90 s (aceleração de 260 rpm/s). Se necessário, repetir e ajustar a velocidade de rotação para obter a espessura adequada de fotorresiste.
- Macio-asse em uma chapa quente, por 2 min a 65 ° C e, depois, durante 7 minutos a 95 ° C. Expor o fotorresiste de acordo com as instruções do fabricante (~ 160 mJ/cm2) usando um filtro de máscara e passe longo de cromo para garantir que as paredes laterais retas.
- Retire a bolacha e pós-asse em uma chapa quente por 1 min a 65 ° C e durante 7 minutos a 95 ° C.
- Dissolver unexposed SU-8 com o desenvolvedor e limpe com 2-propanol.
- Asse a hóstia em uma placa quente a 180 ° C por 30 min (difícil-Asse) estabilizar fotorresiste características.
-
Fazer uma réplica PDMS do modelo SU-8 usando o padrão réplica métodos27de moldagem.
- Trate o molde de SU-8 com vapor de Triclorometilsilanos (TCMS) para reduzir a adesão PDMS (silanização).
Atenção: TCMS é água-reativo e tóxico.- Coloque a bolacha modelada em um rack de bolacha no exsicador campânula de vácuo em uma capa das emanações livre de água ou reagentes solúveis em água.
- Sob o capô, use um conta-gotas para aplicar 1 gota de TCMS para um prato de vidro e coloque dentro do dessecador.
- Feche a tampa do dessecador e permitir que o vapor de TCMS revestir a bolacha pelo menos 20 min.
- Desabafar e em seguida, abra o dessecador. Abra a tampa do frasco de sino e remover a bolacha usando uma pinça de plástico. Coloque em um barco de folha de Petri ou alumínio para moldagem de réplica PDMS. Descarte de materiais revestidos TCMS nos resíduos perigosos adequado.
- Mix de PDMS (proporção de 10:1) usando 40 g da base polímero e 4G PDMS agente de cura.
- Despeje a mistura sobre o molde de SU-8 e desgaseificar a mistura pelo menos 30 min em uma câmara de vácuo.
- Cura para pelo menos 6 h a 70 ° C em um forno.
- Trate o molde de SU-8 com vapor de Triclorometilsilanos (TCMS) para reduzir a adesão PDMS (silanização).
- Retire PDMS wafer definindo a bolacha horizontal sobre uma superfície plana e cuidadosamente descascando o PDMS. Não dobre a bolacha para cima durante esta etapa, como pode quebrar o molde.
Nota: Adesão incompleta de SU-8 para a bolacha ou incompleta silanização pode resultar na destruição das estruturas SU-8 durante esta etapa. - Corte o PDMS em torno dos dispositivos em fichas individuais tais que os dispositivos caberão em uma lamela de 24 x 60 mm.
- Usando um soco biópsia de 1 mm, fazer buracos na entrada, duas saídas e seis atuadores usando um soco biópsia de 1 mm. Aponta para os círculos de 2 mm de diâmetro em torno das bordas do dispositivo.
-
Circuitos de PDMS Bond para lamínulas de vidro.
- Expor as duas superfícies a plasma de oxigênio 80 W (30 s).
- Coloque delicadamente a superfície exposta do PDMS para a superfície exposta da lamínula para um selo conformal.
- Recoze o dispositivo em uma chapa quente (100 ° C, 10 min).
Nota: Ligação insuficiente pode causar a falha do dispositivo devido ao escapamento.
2. preparação do microscópio
Nota: Animais transgénicos: expressar um indicador de cálcio como GCaMP6s28 ou outra sonda de atividade geneticamente codificado no neuron(s) de interesse (por exemplo, TRN); co expresse uma proteína fluorescente independente de atividade emitem num comprimento de onda diferente para corrigir pequeno lateral e artefatos de movimento fora de foco que surgem devido à estimulação mecânica. O software de análise automatizada faixas e compensa o movimento induzido por alterações na intensidade da sonda atividade. O worm cepas GN69223 (ou AQ323629) express GCaMP6s (ou GCaMP6m) e o tagRFP de cálcio independente sob o controle do promotor do mec-7. Além disso, o GN692 contém a mutação lite-1(ce314), que impede a ativação do TRNs devido ao sensor de luz azul lite-130 durante a excitação da fluorescência GCaMP6s23.
-
Criar um sistema de microscópio para excitação simultânea de GCaMP e RFP.
- Usar ou uma fonte de luz contínua e um filtro de excitação de banda dupla que transmite a luz somente ciano e amarelo, ou usar uma fonte de luz que emite apenas comprimentos de onda em uma largura de banda definido como ciano simultânea (0,77 mW) e amarelo (1.21 mW) LED fontes de excitação para excitação simultânea da fluorescência cálcio-dependente e independente, respectivamente.
- Use uma câmera digital para habilitar a gravação de imagens de microscopia.
Nota: a maioria das câmeras vem com um software para aquisição de imagens. Alternativamente, há software livremente disponível que pode ser usado para controlar a câmera e potencialmente também outras partes do sistema de microscopia. - Ajuste a intensidade da excitação, com base na intensidade de fluorescência para evitar saturação de câmera.
-
Use um cubo de fluorescência que dependendo da escolha da fonte de excitação precisa ser equipado com um filtro de excitação.
Nota: Um 488 nm GFP e 580 nm mCherry filtro de excitação foi usado aqui.- Adicione um divisor de feixe para o cubo para refletir luz ciano e amarelo e transmitir luz verde e vermelha.
- Equipar o microscópio com um 10 X objetivo e um objetivo de alta ampliação (por exemplo, 63 X / 1.32 óleo at) para concentrar a luz sobre a amostra de excitação.
- Monte um divisor de feixe na frente da câmera para gravação simultânea do GCaMP e o sinal de cálcio-independente. Certifique-se de que o divisor de feixe tem um espelho dicroico (passe longo, corte a 570 nm) para separar a luz verde e vermelha, usando um filtro de emissão para verde (passante centrado em 525 nm, com largura de 50 nm) e de emissão de um filtro para luz vermelha (passband centrado em 632 nm com 60 nm largura).
- Projeto da fluorescência verde e vermelha para a metade superior (verde) e metade inferior (vermelho), respectivamente da câmera chip (ver Figura 3). Esta orientação é um pré-requisito para o software de análise fornecida.
3. animal preparação
- Preparar a idade-sincronizado jovem adulto ou adulto dia um c. elegans, conforme descrito anteriormente por Porta-de-la-Riva et al 31
- Prepare o chip microfluidic.
- Conecte o reservatório de fluxo de gravidade (~ 60 cm acima do nível da microplaqueta) contendo filtrada buffer de M9 (filtro de seringa de polietersulfona de 0,2 µm) para uma tomada do chip. Conecte a saída de outra para uma tomada de um contentor de resíduos de duas saídas, ou seja, um frasco de filtro. Conecte a outra saída do contêiner de resíduos uma bomba peristáltica.
Nota: Usar tubos de polietileno (PE) para todas as conexões e usar os encaixes de tubo de metal para conectar os tubos de PE para o chip. Desta forma todas as soluções de resíduos serão liberadas sem o chip e recolhidas no recipiente de resíduos. O fluxo através dos canais de saída cria uma sucção suave que manterá o worm confortável durante o processo de captura mais tarde. - Prepare-se interconecta consistindo de 50mm longo PE tubos (OD 0.9652-mm, 0.5842-mm ID) com tubos de metal (medidor de tamanho 23TW, OD 0,635 mm) em ambas as extremidades. Press-fit Estas interconexões em cada um dos seis dedos atuação e na entrada de worm (ver Figura 1). Deixe essas interligações no chip, como remoção repetida leva ao desgaste sobre os buracos PDMS.
- Conecte o reservatório de fluxo de gravidade (~ 60 cm acima do nível da microplaqueta) contendo filtrada buffer de M9 (filtro de seringa de polietersulfona de 0,2 µm) para uma tomada do chip. Conecte a saída de outra para uma tomada de um contentor de resíduos de duas saídas, ou seja, um frasco de filtro. Conecte a outra saída do contêiner de resíduos uma bomba peristáltica.
- Coloque o chip no microscópio. Escolha 2 – 5 vermes em uma gota de filtrado buffer de M9 (filtro de seringa 0,2 µm) e usam uma seringa de 1 mL para atraí-los para um tubo de PE (OD 0.9652-mm, 0.5842-mm ID) ligado a uma seringa de 1 mL, puxando o êmbolo suavemente. Manter os animais dentro do tubo de PE e não na seringa.
Nota: Muitos vermes ou soluções não filtradas no chip podem conduzir à obstrução. - Conecte o tubo de PE da seringa para a interconexão à entrada do worm (Figura 1 e Figura 2) do chip. Ativar o fluxo de gravidade, abrindo a válvula e ligue a bomba peristáltica. Em seguida, pressione suavemente o êmbolo da seringa da seringa para mover os animais para o canal de armadilhagem enquanto observa o canal sob um microscópio com uma lente X 10 no modo brightfield.
Nota: Ocasionalmente aparecem bolhas de ar não representam um problema; Eles são geralmente automaticamente movidos na tomada. Vários animais podem ser "estacionados" na câmara de espera e usados sequencialmente.- Depois de carregar o animal dentro do canal de captura (ver Figura 2), ajuste sua posição suavemente empurrando ou puxando o êmbolo da seringa para posicionar a cabeça do animal em forma cónica da frente do canal.
- Certifique-se de que o worm (adulto dia 1) tem o tamanho certo para ser preso no chip.
- Se o worm não preenche a secção inteira do canal do nariz para perto do final do corpo (não incluindo a ponta da cauda) ou o worm se move ao longo do eixo do canal sem mover o êmbolo da seringa , remover o worm, pressionando o êmbolo da seringa até desaparecer pelo canal da caça com armadilhas e carregar uma nova (consulte a etapa 3.8).
- Alternar para o modo de fluorescência do microscópio e uma lente de ampliação mais elevada (40 X ou superior) e verifique se o axônio do neurônio de interesse vem deitar o diafragma de um dos atuadores. Caso contrário, ajuste a posição do animal puxando ou empurrando o êmbolo. Se isso não ajudar, remover o worm e carregar um novo.
- Focar o corpo celular do neurônio de interesse e se conectar a interconexão do atuador mais próximo para o neurônio no lado anterior do corpo celular para uma bomba de pressão programável usando tubos de PE.
- Se o experimento requer a medição da distância entre o atuador e o neurônio, mova o campo de visão, então ambos são no campo de visão e o channelwall é paralela à borda superior e inferior da imagem.
- Define um protocolo de pressão usando a bomba de pressão programável.
Nota: Este protocolo pode ser ajustado para a experiência desejada.- Comece com uma pressão constante de 0 kPa durante um tempo correspondente a pelo menos 50 imagens da sequência de imagem (necessária para a normalização). Para uma taxa de geração de imagens de 10 Hz, isto corresponde a 5 s. adicionar uma forma de onda desejada de estímulo e pressão e definir o comprimento do estímulo como um segundo passo.
Nota: Para estimular os TRNs, uma onda senoidal de(ou seja, 75 kPa, 10 Hz) sobreposta com um passo (ou seja, 275 kPa) é recomendada como ele gera uma grande resposta neuronal. - Se o experimento requer estímulos adicionais, incluir um período de pelo menos 10 s, a uma pressão constante de 0 kPa entre estímulos. Antes de ligar a bomba de pressão, verifique se que a pressão do instrumento está em constante 0 kPa evitar estimulação acidental do worm antes a experiência.
- Comece com uma pressão constante de 0 kPa durante um tempo correspondente a pelo menos 50 imagens da sequência de imagem (necessária para a normalização). Para uma taxa de geração de imagens de 10 Hz, isto corresponde a 5 s. adicionar uma forma de onda desejada de estímulo e pressão e definir o comprimento do estímulo como um segundo passo.
- Execute o protocolo de imagem e pressão.
- O software de aquisição de imagem da câmera, defina uma sequência de imagens a 10 Hz usando um tempo de exposição de 100 ms para a duração do protocolo de pressão. Ajuste a intensidade de excitação tal que o aumento de fluorescência máxima não saturar a câmera. Salve as imagens como um arquivo *. tif, pelo menos 50 imagens antes do primeiro estímulo para normalização.
- Inicie o protocolo de imagem no software de aquisição de imagem e o protocolo de pressão no software da bomba programável. Durante a gravação, observe se o neurônio de interesse é o ponto mais brilhante em uma área de 10x10 pixels, não se move mais do que 10 pixels em imagens sequenciais e permanece no campo de visão durante a gravação.
Nota: Se isso não for feito, o software de análise irá falhar. Pontos brilhantes (i.e., autofluorescência) ao redor do neurônio fazem gravações dos neurônios difícil de limpar. Para corrigir isso, remover o worm da armadilha e carregar um novo worm no chip. Se o verme se mexer demais, tente uma nova gravação do worm mesmo e observar o movimento da minhoca. Se o problema persistir, o worm pode ser muito pequeno para ser preso. Neste caso, remover este worm e carregar um verme ligeiramente maior para a armadilha. - Se desejar, gravar os sinais de vários neurônios no campo de visão simultaneamente, enquanto os neurônios estão separados pelo menos 10 pixels durante a gravação.
- Para investigar a habituação, realizar o experimento repetidamente em um animal.
- Remova o worm do canal de armadilhagem.
- Se se deseja manter o verme para estudos posteriores, desconecte os pontos de venda do chip para o fluxo de gravidade e o recipiente de resíduos. Pressione o êmbolo da seringa cuidadosamente até que o verme inteiro é empurrado através do canal de armadilhas para o canal de fluxo.
- Continue pressionando o êmbolo da seringa até o animal aparece em uma gota fora do chip. Desconectar a seringa, incluindo os tubos de PE a partir da entrada de worm, usá-lo para aspirar os vermes em gota e transferi-lo em uma placa de ágar.
- Se não se deseja manter o verme, remover o worm, pressionando o êmbolo da seringa até que o verme inteiro é empurrado através do canal de armadilhas para o canal de fluxo; o fluxo de gravidade e a sucção da bomba peristáltica levará o animal fora do chip e para o recipiente de resíduos.
4. análise
- Baixe e instale o mais recente Fiji versão32.
- Baixe e instale a versão mais recente do SDK java.
Nota: Se necessário, excluir ou renomear a pasta java dentro da pasta de Fiji para Fiji usar o compilador de java instalado recentemente. -
Software aberto de Fiji.
- Verifique se o software está executando o compilador java instalado recentemente abrindo ' Plugins | Utilitários | ImageJ | Propriedades de.
- Baixe o arquivo pokinganalyzer*.java (https://github.com/HFehlauer/Poking-Analyzer, ver arquivo complementar 2).
- Arrastar e soltar o arquivo. Java na janela do software para abri-lo como um plugin.
-
Compilar e executar o arquivo pokinganalyzer*.java as teclas Ctrl + R em Fiji; uma interface gráfica do usuário (cutucando Analyzer) será aberta (Figura 3).
- Clique na aba "Ajuda" e leia sobre os requisitos de software e como realizar a análise. Para começar, selecione a guia "Open video" Especifique o local do arquivo de vídeo para o analisador: clique no botão "Abrir um vídeo", navegue até o local do vídeo e abri-lo.
Nota: O software começa com este guia aberto. Se não é aberta quando se inicia uma nova análise clique na guia "Open video". Se o vídeo é no formato de a.tif, o programa irá abrir o vídeo internamente e exibir a primeira imagem na parte inferior da interface. Se a imagem não é do vídeo que precisa ser analisado, clique no botão "Abrir um vídeo" para abrir um outro vídeo. - Para continuar, clique na guia "Definir ROIs". Nessa guia, defina a posição do TRN. Observe que a primeira imagem do vídeo aberto é exibida na parte inferior desta Tab. Clique no botão "Definir TRN" e clique sobre o neurônio na parte superior da imagem exibida que deve exibir a fluorescência de GCaMP.
- Opcional: Se o atuador estiver visível na metade superior da imagem o programa pode automaticamente controlar a distância entre o neurônio e o atuador se desejado. Por isso, verificar a "definir o atuador?" -caixa, clique no botão "Definir atuador" e clique sobre o atuador na metade superior da imagem.
- Clique na guia "Análise" definir a taxa de aquisição de imagem. Ou digite um número no campo ou clique em cima ou para baixo botões para aumentar ou diminuir o número. Se na aba anterior a "definir o atuador?" -caixa é verificada, definir o tamanho de célula de câmera, a ampliação e o fator binning, então o programa pode calcular a distância.
- Clique no botão "Iniciar análise".
Nota: O programa agora irá acompanhar o neurônio por sua fluorescência na sequência de imagem na metade superior (dependente de cálcio) e na metade inferior (independente de cálcio) da sequência de imagem. Para compensar o movimento do neurônio no plano de gravar o programa vai investigar uma área de 10 x 10 pixels de acordo com a localização do neurônio na imagem anterior para encontrar a posição do neurônio na imagem a seguir. Calculará a fluorescência em ambas as metades corrigindo para a fluorescência de fundo (Fbg) e dividindo por fluorescência nas primeiras 50 imagens (F0):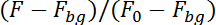
Alterações de fluorescência no canal verde, dependente da atividade (FCa2 +) que são causadas por neurônio de movimento fora do avião de gravação são então corrigido usando a fluorescência no canal vermelho, independente de atividade (F corr) e o desvio-padrão pré-estímulo de cálcio-dependente (sdCa2 +) e independente da fluorescência (sdcorr) por:
- O programa irá mostrar seu progresso na barra de status na parte inferior da interface; Quando a barra de status atinge 100%, clique na guia "Resultados". Se a barra de status parar antes 100%, o programa dará uma mensagem de erro indicando a razão pela qual que o programa não poderia analisar o vídeo.
- Se o sinal à relação de ruído é muito baixo, o programa não consegue identificar o neurônio; Ajuste os parâmetros de imagem em gravações posteriores.
- Se o neurônio deixa o campo de visão durante a gravação, o programa não pode segui-lo mais e vai parar. Para gravações posteriores, coloca o neurônio no meio do campo de visão no início da gravação.
- Se algumas regiões no campo de visão estão supersaturadas, a análise automatizada irá produzir resultados inválidos. Para gravações posteriores, certifique-se que o sinal de fluorescência não saturar o sensor da câmera.
- Na guia 'resultados', o resultado é exibido na parte superior. Salve um arquivo *. txt separado por tab da tabela resultado clicando em "Salvar a tabela de resultados".
Nota: Esta tabela consiste no tempo em segundos e a fluorescência normalizada de cálcio-dependente (corrigido por fluorescência independente de cálcio). Se anteriormente definido, ele também contém a distância total entre o TRN e o atuador em µm e a distância entre o TRN e o atuador perpendicular à parede do canal em µm. resultados representativos da intensidade da fluorescência aumentam após estimulação versus a distância do corpo celular para o atuador mais próximo são plotados na Figura 4. - Se houver vários neurônios em uma gravação (separada por mais de 10 pixels), clique na guia "Definir ROIs" novamente e volte ao passo 4.6.2 com o segundo neurônio.
- Clique na aba "Ajuda" e leia sobre os requisitos de software e como realizar a análise. Para começar, selecione a guia "Open video" Especifique o local do arquivo de vídeo para o analisador: clique no botão "Abrir um vídeo", navegue até o local do vídeo e abri-lo.
Resultados
SU-8 litografia e colagem de Chip
O protocolo de litografia e moldagem de PDMS sigam os procedimentos padrão. Detalhes podem ser encontrados em outros lugares23,24,25,26. O PDMS deve descascar fora a hóstia sem problemas após a cura. Se os recursos de SU-8 arrancar durante o descascamento de PDMS, ou a camada de aderência de SU-8 ou a sil...
Discussão
Este protocolo demonstra um método de distribuição precisa estimulação mecânica na pele de uma lombriga, presa em um chip microfluidic. Destina-se a facilitar a integração dos estímulos físicos para responder às perguntas biológicas e visa agilizar a pesquisa de mechanobiology em laboratórios biológicos. Esse método amplia anteriores ensaios para avaliar a função dos neurônios mechanosensory em c. elegans. Técnicas quantitativas e semi-quantitativa anteriores medido forças1<...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Agradecemos a Sandra N. Manosalvas-Kjono, Ladpli de Purim, Farah Memon, Divya Gopisetty e Veronica Sanchez para apoiar no projeto de dispositivo e geração de animais mutantes. Esta pesquisa foi apoiada por subsídios de NIH R01EB006745 (a BLP), R01NS092099 (para MBG), K99NS089942 (a MK), F31NS100318 (a ALN) e recebeu financiamento do Conselho Europeu de investigação (CEI), sob o Horizonte 2020 pesquisa e inovação da União Europeia (do programa Convenção de subvenção n. º 715243 para MK).
Materiais
| Name | Company | Catalog Number | Comments |
| Chrome mask | Compugraphics (http://www.compugraphics-photomasks.com/) | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| Chrome mask | Mitani-Micronics (http://www.mitani-micro.co.jp/en/) | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| Chrome mask | Kuroda-Electric (http://www.kuroda-electric.eu/ | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| 4'' Silicon wafer (B-test) | Stanford Nanofabrication Facility | ||
| SU-8 2002 | MicroChem | ||
| SU-8 2050 | MicroChem | ||
| Spin-coater | Laurell Technologies | WS-400BZ-6NPP/LITE | |
| Exposure timer | Optical Associates, Inc | OAI 150 | |
| Illumination controller | Optical Associates, Inc | 2105C2 | |
| SU-8 developer | MicroChem | ||
| 2-Propanol | Fisher Scientific | A426F-1GAL | |
| Acetone | Fisher Scientific | A18-4 | |
| Trichloromethylsilane (TCMS) | Sigma-Aldrich | 92361-500ML | Caution: TCMS is toxic and water-reactive |
| Sylgard 184 Elastomer Kit | Dow Corning | PDMS prepolymer | |
| Biopsy punch, 1 mm | VWR | 95039-090 | |
| Oxygen Plasma Asher | Branson/IPC | ||
| Small metal tubing (0.635 mm OD, 0.4318 mm ID, 12.7 mm long); gage size 23TW | New England Small Tube Corporation | NE-1300-01 | |
| Nalgene syringe filter, 0.22 μm | Thermo Scientific | 725-2520 | to filter all solution, small particles would clog the chip |
| Polyethylene tubing; 0.9652 mm OD, 0.5842 mm ID | Solomon Scientific | BPE-T50 | |
| Syringe, 1 ml | BD Scientific | 309628 | for worm trapping and release |
| Syringe, 20 ml | BD Scientific | 309661 | for gravity-based flow |
| Gilson Minipuls 3, Peristaltic pump | Gilson | to suck solutions and worms out of the chip | |
| Microfluidic flow controller, equipped with 0–800 kPa pressure channel | Elveflow | OB1 MK3 | pressure delivery |
| Water-Resistant Clear Poly- urethane Tubing, 4 mm ID and 6 mm OD | McMaster-Carr | 5195 T52 | connection from house air to pressure pump |
| Water-Resistant Clear Polyurethane Tubing, 2.6mm ID and 4mm OD | McMaster-Carr | 5195 T51 | connect pressure pump to small tubng |
| Push-to-Connect Tube Fitting for Air | McMaster-Carr | 5111K468 | metric - imperial converter |
| Straight Connector for 6 mm × 1/4″ Tube OD | McMaster-Carr | 5779 K258 | |
| Leica DMI 4000 B microscopy system | Leica | ||
| 63×/1.32 NA HCX PL APO oil objective | Leica | 506081 | |
| Hamamatsu Orca-Flash 4.0LT digital CMOS camera | Hamamatsu | C11440-42U | |
| Lumencor Spectra X light engine | Lumencor | With cyan and green/yellow light source | |
| Excitation beam splitter | Chroma | 59022bs | in the microscope |
| Hamamatsu W-view Gemini Image splitting optics | Hamamatsu | A12801-01 | to split green and red emission and project them on different areas on the camera chip |
| Emission beam splitter | Chroma | T570lpxr | in the image splitter |
| Emission filters GCamp6s | Chroma | ET525/50m | in the image splitter |
| Emission filters mCherry | Chroma | ET632/60m | in the image splitter |
Referências
- Eastwood, A. L., et al. Tissue mechanics govern the rapidly adapting and symmetrical response to touch. Proc. Natl. Acad. Sci. 15 (50), E6955-E6963 (2015).
- Katta, S., Krieg, M., Goodman, M. B. Feeling Force: Physical and Physiological Principles Enabling Sensory Mechanotransduction. Annu. Rev. Cell Dev. Biol. 31, 347-371 (2015).
- Krieg, M., Dunn, A. R., Goodman, M. B. Mechanical systems biology of C. elegans touch sensation. BioEssays. 37 (3), 335-344 (2015).
- Krieg, M., Dunn, A. R., Goodman, M. B. Mechanical control of the sense of touch by β-spectrin. Nat. Cell Biol. 16 (3), 224-233 (2014).
- Krieg, M., et al. Tensile forces govern germ-layer organization in zebrafish. Nat Cell Biol. 10 (4), 429-436 (2008).
- Gaub, B. M., Müller, D. J. Mechanical stimulation of Piezo1 receptors depends on extracellular matrix proteins and directionality of force. Nano Lett. 17 (3), 2064-2072 (2017).
- Geffeney, S. L., et al. DEG/ENaC but not TRP channels are the major mechanoelectrical transduction channels in a c. Elegans nociceptor. Neuron. 71 (5), 845-857 (2011).
- O'Hagan, R., Chalfie, M., Goodman, M. B. The MEC-4 DEG/ENaC channel of Caenorhabditis elegans touch receptor neurons transduces mechanical signals. Nat. Neurosci. 8 (1), 43-50 (2005).
- Suzuki, H., et al. In Vivo Imaging of C. elegans Mechanosensory Neurons Demonstrates a Specific Role for the MEC-4 Channel in the Process of Gentle Touch Sensation. Neuron. 39 (6), 1005-1017 (2003).
- Kopito, R. B., Levine, E. Durable spatiotemporal surveillance of Caenorhabditis elegans response to environmental cues. Lab Chip. 14 (4), 764-770 (2014).
- Chokshi, T. V., Ben-Yakar, A., Chronis, N. CO2 and compressive immobilization of C. elegans on-chip. Lab Chip. 9 (1), 151 (2009).
- Hulme, S. E., Shevkoplyas, S. S., McGuigan, A. P., Apfeld, J., Fontana, W., Whitesides, G. M. Lifespan-on-a-chip: microfluidic chambers for performing lifelong observation of C. elegans. Lab Chip. 10 (5), 589-597 (2010).
- Li, S., Stone, H. a., Murphy, C. T. A microfluidic device and automatic counting system for the study of C. elegans reproductive aging. Lab Chip. 15 (2), 524-531 (2015).
- Chokshi, T. V., Bazopoulou, D., Chronis, N. An automated microfluidic platform for calcium imaging of chemosensory neurons in Caenorhabditis elegans. Lab Chip. 10 (20), 2758-2763 (2010).
- Mishra, B., et al. Using microfluidics chips for live imaging and study of injury responses in Drosophila larvae. J. Vis. Exp. , e50998 (2014).
- Chronis, N., Zimmer, M., Bargmann, C. I. Microfluidics for in vivo imaging of neuronal and behavioral activity in Caenorhabditis elegans. Nat. Methods. 4 (9), 727-731 (2007).
- Krajniak, J., Lu, H. Long-term high-resolution imaging and culture of C. elegans in chip-gel hybrid microfluidic device for developmental studies. Lab Chip. 10 (14), 1862-1868 (2010).
- Vasquez, V., Krieg, M., Lockhead, D., Goodman, M. B. Phospholipids that Contain Polyunsaturated Fatty Acids Enhance Neuronal Cell Mechanics and Touch Sensation. CellReports. 6 (1), 70-80 (2013).
- Krieg, M., et al. Genetic defects in β-spectrin and tau sensitize C. elegans axons to movement-induced damage via torque-tension coupling. Elife. 6 (2010), e20172 (2017).
- Arnadóttir, J., O'Hagan, R., Chen, Y., Goodman, M. B., Chalfie, M. The DEG/ENaC protein MEC-10 regulates the transduction channel complex in Caenorhabditis elegans touch receptor neurons. J. Neurosci. 31 (35), 12695-12704 (2011).
- Lockhead, D., et al. The tubulin repertoire of Caenorhabditis elegans sensory neurons and its context-dependent role in process outgrowth. Mol. Biol. Cell. 27 (23), 3717-3728 (2016).
- Goodman, M. B., Ernstrom, G. G., Chelur, D. S., O'hagan, R., Yao, C. A., Chalfie, M. MEC-2 regulates C. elegans DEG/ENaC channels needed for mechanosensation. Nature. 415 (6875), 1039-1042 (2002).
- Nekimken, A., Fehlauer, H., Kim, A., Goodman, M., Pruitt, B. L., Krieg, M. Pneumatic stimulation of C. elegans mechanoreceptor neurons in a microfluidic trap. Lab Chip. , (2017).
- Brower, K., White, A. K., Fordyce, P. M. Multi-step Variable Height Photolithography for Valved Multilayer Microfluidic Devices. J. Vis. Exp. (119), e55276 (2017).
- Jenkins, G. Rapid prototyping of PDMS devices using SU-8 lithography. Methods Mol. Biol. 949 (1), 153-168 (2013).
- Faustino, V., Catarino, S. O., Lima, R., Minas, G. Biomedical microfluidic devices by using low-cost fabrication techniques: A review. J. Biomech. 49 (11), 2280-2292 (2016).
- Xia, Y., Whitesides, G. M. SOFT LITHOGRAPHY. Annu. Rev. Mater. Sci. 28 (1), 153-184 (1998).
- Chen, T. -. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Cho, Y., Porto, D., Hwang, H., Grundy, L., Schafer, W. R., Lu, H. Automated and controlled mechanical stimulation and functional imaging in vivo in C. elegans. Lab Chip. , (2017).
- Edwards, S. L., et al. A novel molecular solution for ultraviolet light detection in Caenorhabditis elegans. PLoS Biol. 6 (8), 1715-1729 (2008).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Cerón, J. Basic Caenorhabditis elegans methods: synchronization and observation. J. Vis. Exp. (64), e4019 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat. Methods. 9 (7), 676-682 (2012).
- Cho, Y., Porto, D. A., Hwang, H., Grundy, L. J. High-Throughput Controlled Mechanical Stimulation and Functional Imaging In Vivo. BiorXiv. , (2017).
- Petzold, B. C., Park, S. -. J., Mazzochette, E. A., Goodman, M. B., Pruitt, B. L. MEMS-based force-clamp analysis of the role of body stiffness in C. elegans touch sensation. Integr. Biol. (Camb). 5 (6), 853-864 (2013).
- Nekimken, A. L., Mazzochette, E. A., Goodman, M. B., Pruitt, B. L. Forces applied during classical touch assays for Caenorhabditis elegans. PLoS One. 12 (5), e0178080 (2017).
- Chronis, N., Zimmer, M., Bargmann, C. I. Microfluidics for in vivo imaging of neuronal and behavioral activity in Caenorhabditis elegans. Nat. Methods. 4 (9), 727-731 (2007).
- Gilleland, C. L., Rohde, C. B., Zeng, F., Yanik, M. F. Microfluidic immobilization of physiologically active Caenorhabditis elegans. Nat. Protoc. 5 (12), 1888-1902 (2010).
- Chuang, H. -. S., Raizen, D. M., Lamb, A., Dabbish, N., Bau, H. H. Dielectrophoresis of Caenorhabditis elegans. Lab Chip. 11 (4), 599 (2011).
- Christensen, A. M., Chang-Yen, D. A., Gale, B. K. Characterization of interconnects used in PDMS microfluidic systems. J. Micromechanics Microengineering. 15 (5), 928-934 (2005).
- Gilpin, W., Uppaluri, S., Brangwynne, C. P. Worms under Pressure: Bulk Mechanical Properties of C. elegans Are Independent of the Cuticle. Biophys. J. 108 (8), 1887-1898 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados