Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Verwendung von einem Mikrofluidik-Gerät für mechanische Stimulation und High Resolution Imaging von C. elegans
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Neue Werkzeuge für die Mechanobiology Forschung sind erforderlich, um verstehen, wie mechanischen Beanspruchung biochemische Stoffwechselwege aktiviert und biologische Reaktionen hervorruft. Hier präsentieren wir eine neue Methode zur selektiven mechanischen Stimulation der immobilisierten Tiere mit einem mikrofluidischen Trap ermöglicht hochauflösende Bildgebung der zellulären Antworten.
Zusammenfassung
Ein zentrales Ziel des Mechanobiology ist die Wechselwirkung von mechanischem Stress auf Proteine und Zellen zu verstehen. Trotz seiner Bedeutung ist der Einfluss von mechanischem Stress auf zelluläre Funktion immer noch schlecht verstanden. Unter anderem existiert diese Wissenslücke, weil einige Tools gleichzeitige Verformung von Gewebe und Zellen, Bildgebung der Zellaktivität in lebenden Tieren und effiziente Einschränkung der Beweglichkeit in ansonsten hochmobile Modellorganismen wie die Nematoden ermöglichen Caenorhabditis Elegans. Die geringe Größe von C. Elegans stellt sie eine ausgezeichnete Übereinstimmung Mikrofluidik-Forschung Geräte und Lösungen für Immobilisierung wurden mit mikrofluidischen Geräten vorgestellt. Obwohl diese Geräte hochauflösende Bildgebung zu ermöglichen, ist das Tier vollständig eingehüllt in Polydimethylsiloxan (PDMS) und Glas, physischen Zugriff für die Lieferung der mechanischen Kraft oder elektrophysiologische Aufnahmen zu begrenzen. Vor kurzem haben wir ein Gerät, das pneumatische Antriebe mit einem Trapping-Design integriert, die mit hochauflösenden Fluoreszenzmikroskopie kompatibel ist. Die Betätigung-Kanal trennt sich von den Wurm-Trapping-Kanal durch eine dünne Membran PDMS. Diese Membran ist in die Seite eines Wurms abgelenkt durch Druck aus einer externen Quelle. Das Gerät kann individuelle Mechanosensitive Neuronen ausrichten. Die Aktivierung dieser Neuronen ist abgebildet bei hochauflösenden mit genetisch codierte Kalzium Indikatoren. Dieser Artikel stellt die allgemeine Methode mit C. Elegans Stämme Calcium-Sensitive Aktivitätsanzeige (GCaMP6s) in ihrer Touch-Rezeptor-Neuronen (TRNs) zum Ausdruck zu bringen. Die Methode ist nicht beschränkt auf TRNs noch auf Kalzium-Sensoren als Sonde jedoch, auf andere mechanisch empfindlichen Zellen oder Sensoren erweitert werden kann.
Einleitung
Der Tastsinn bietet Tiere mit wichtigen Informationen über ihre Umgebung. Je nach der angewendeten Kraft wird Touch so harmlos, angenehm oder schmerzhaft empfunden. Die Gewebe-Verformung bei Berührung erkannt wird durch spezialisierte Mechanoreceptor Zellen in der Haut eingebettet, die Rezeptor-Proteine, die am häufigsten Ionenkanäle ausdrücken. Die Schritte verknüpfen Kraft Wahrnehmung mit Ionen-Kanal Aktivierung bei Berührung und Schmerz sind nicht vollständig geklärt. Noch weniger ist bekannt über das Hautgewebe filtert mechanische Verformung und ob Mechanorezeptoren erkennen von Änderungen im Stamm oder betonen,1,2,3. Diese Lücke im Verständnis ergibt sich einerseits aus einem Mangel an geeigneten Werkzeugen, präzise mechanische Reize an die Oberfläche der Haut ein lebendes Tier anzuwenden, unter Beachtung der Reaktionen auf zellulärer Ebene. Während Rasterkraftmikroskopie weitgehend verwendet worden ist, anzuwenden und messen Kräfte in isolierten Zellen4,5 und Piezo1 Rezeptoren im Leben aktivieren Zellen6, ähnliche Experimente mit lebenden Tieren, vor allem C. Elegans, wurden notorisch schwierig wegen der inhärenten Mobilität des Subjekts. Diese Herausforderung wird traditionell durch Veterinär- oder chirurgischen Klasse Cyanacrylat Klebstoff, um einzelne Tiere auf Agar-Pads1,7,8,9zu immobilisieren umgangen. Dieser Ansatz ist produktiv gewesen, aber hat Beschränkungen in Bezug auf das Geschick erforderlich für Immobilisierung durch Kleben und die weiche Agar-Oberfläche auf mechanische Compliance. Eine Mikrofluidik-Strategie ist eine kostenlose Alternative, die den Komplikationen verbunden zu kleben vermeidet.
Der Fadenwurm C. Elegans ist ein genetisches Modellorganismus mit einer völlig zugeordneten Nervensystem, das aufgrund der Größe des Tieres, eine gute Passform für Mikrofluidik-Technologie. Mikrofluidik-basierte Geräte bieten den Vorteil, dass die sonst äußerst mobilen Tiere zurückgehalten werden können, während der Durchführung hochauflösende Bildgebung und Lieferung von relevanten Neuro-modulierende reizen. Mit Hilfe von mikrofluidischen können Technologien, lebende Tiere ohne Schaden10,11, ermöglicht Überwachung des Verhaltens Aktivität über die gesamte Lebensdauer12,13 und hochauflösende immobilisiert werden Imaging von neuronaler Aktivität14,15,16,17. Weiter, viele Mechanoreceptor Neuronen benötigt für das Gefühl der Berührung und Schmerz auf ihre physiologischen1,8, mechanische4,18,19, gekennzeichnet werden kann und molekulare Level20,21,22.
C. Elegans spürt sanfte mechanische Reize an seinen Körper Wand mit sechs TRNs, von denen drei innervieren des Tieres anterior (ALML/R und AVM) und drei davon innervieren des Tieres Posterior (PLML/R und PVM). Die Ionen-Kanal Moleküle benötigt für eine ausgeübte Kraft in eine biochemische Signal transducing haben ausgiebig in seiner TRNs8untersucht. Dieser Artikel stellt eine mikrofluidischen Plattform23 , mit dem Forscher präzise mechanische Kräfte auf die Haut von einem immobilisierten C. Elegans anwenden können Fadenwurm, beim Auslesen der Verformung von seiner inneren Geweben durch optische Bildgebung. Neben der Präsentation klar definierte mechanische Reize, können Kalzium Transienten in Mechanoreceptor Neuronen mit subzellulären Auflösung aufgezeichnet und korreliert mit morphologischen und anatomischen Merkmale. Das Gerät besteht aus eine zentrale Trapping-Kanal, der hält ein einziges Tier und präsentiert seine Haut neben sechs pneumatische Betätigung-Kanälen (Abbildung 1 und Abbildung 2). Die sechs Kanäle befinden sich entlang der Trapping-Kanal, mechanische Reize zu jedem der sechs TRNs der Wurm zu liefern. Diese Kanäle sind aus der Trapping-Kammer durch dünne PDMS Membranen, getrennt durch eine externe Luftversorgung der Druck (Abbildung 1) gefahren werden kann. Wir kalibriert die Durchbiegung in Bezug auf Druck und liefern die Messungen in diesem Artikel. Jeder Antrieb kann individuell angesprochen und verwendet, um eine Mechanoreceptor Wahl zu stimulieren. Der Druck wird geliefert mit einem Piezo-gesteuerte Druckpumpe aber alternative Gerät verwendet werden kann. Wir zeigen, dass die Druck-Protokoll verwendet werden kann, TRNs in Vivo aktivieren und demonstrieren Betriebsgeräte geeignet für Erwachsene C. Elegansmechanische Stimuli bereitzustellen, ausgewachsene Tiere in Geräte laden, Kalzium Bildgebung durchführen Experimente und Analyse der Ergebnisse. Gerät Herstellung besteht aus zwei wesentlichen Schritten: 1) Photolithographie zu einer Form von SU-8; und 2) Guß PDMS um ein Gerät zu machen. Aus Gründen der Kürze und Klarheit gekennzeichnet Leser zuvor veröffentlichten Artikel und Protokolle24,25 für Anweisungen, wie man die Werkzeuge und Geräte zu produzieren.
Protokoll
1. Gerät Fertigung
- Download der angehängten Maskendatei (ergänzende Datei 1) und erzeugen eine Chrom-Maske mit einem kommerziellen Dienst oder Inhouse-Anlage. Wie die kleinste Abmessung auf dem Gerät 10 µm (Aktor Membrandicke), sichergestellt, dass die Maske ausreichend hohen Auflösung innerhalb ± 0,25 µm, für die Funktionen zuverlässig zu produzieren.
-
SU-8 Photolithographie Standardmethoden zu folgen (z. B.verweist auf24,25,26) fabrizieren die Form für die spätere Produktion von PDMS Vorrichtungen; eine Übersicht über die Schritte unten aufgeführten ist.
Hinweis: SU-8 ist ein lichtempfindliches Material, welche Querverbindungen bei Exposition gegenüber UV-Licht (maximale Absorption ~ 365 nm). Verwenden Sie die Anweisungen des Herstellers als Ausgangspunkt zu bestimmen die Verarbeitungsparameter für die Soft-backen, Exposition (UV), und nach dem Backen.- SU-8 2002 auf einem Silizium-Wafer und Spin-Mantel zu hinterlegen um eine ungefähre Dicke von 2 µm, für bessere Haftung von kleinen Strukturen. Soft-Backen auf einer heißen Platte für 1 min bei 95 ° C, leicht überbelichtet (UV) die gesamte Oberfläche (~ 100 mJ/cm2), und nach dem Backen auf einer heißen Platte für 2 min bei 95 ° C.
- Spin-Mantel eine 47-µm dicke Schicht von SU-8 2050 für die Definition der Gerätefunktionen. Verwenden Sie eine Schleuderdrehzahl von 500 u/min für 15 s (130 u/min/s Beschleunigung) und dann 2.000 u/min für 90 s (260 u/min/s Beschleunigung). Falls erforderlich, wiederholen Sie und passen Sie die Schleuderdrehzahl um die richtige Dicke von Fotolack zu erhalten.
- Backen Sie Soft-auf einer heißen Platte für 2 min bei 65 ° C, und dann für 7 min bei 95 ° C. Setzen Sie den Fotolack nach Anweisungen des Herstellers (~ 160 mJ/cm2) einen Chrom-Maske und lang-Pass-Filter mit um geraden Seitenwänden zu gewährleisten.
- Den Wafer zu entfernen und nach dem Backen auf einer heißen Platte für 1 min bei 65 ° C und 7 min bei 95 ° C.
- Unbelichtete SU-8 mit dem Entwickler zu lösen, und mit 2-Propanol reinigen.
- Backen Sie die Wafer auf eine Herdplatte bei 180 ° C für 30 min (hart-Backen), Fotolack Funktionen zu stabilisieren.
-
Machen Sie eine PDMS-Replik aus dem SU-8-Modell mit standard Replik Methoden27Guß.
- Behandeln Sie die SU-8-Form mit Trichloromethylsilane (TCMS) Dampf um PDMS Haftung (Silanisierung) zu reduzieren.
Achtung: TCMS ist giftig und Wasser reagierend.- Ort der gemusterten Wafer in einem Wafer-Rack innerhalb einer Glasglocke Vakuum Exsikkator unter einem Abzug frei von Wasser und wasserlösliche Reagenzien.
- Verwenden Sie unter der Haube eine Pipette 1 Tropfen TCMS zuweisen einer Glasschale und platzieren Sie in den Exsikkator.
- Schließen Sie den Deckel der Exsikkator und ermöglichen Sie TCMS Dampf den Wafer für mindestens 20 Minuten zu beschichten.
- Entlüften Sie und öffnen Sie dann den Exsikkator. Öffnen Sie den Deckel des Bohnenbehälters und entfernen Sie den Wafer Kunststoff Pinzette zu. Legen Sie in eine Petrischale oder Aluminium Folie Boot für PDMS-Replikat-Spritzgießen. TCMS-beschichtete Materialien in der richtigen Sondermüll entsorgen.
- Mix PDMS (Verhältnis 10:1) verwenden die Basis Polymer und 4 g PDMS Härtemittel 40 g.
- Gießen Sie die Mischung über die SU-8-Form und entgasen Sie die Mischung für mindestens 30 min in einer Vakuumkammer.
- Heilung für mindestens 6 h bei 70 ° C in einem Ofen.
- Behandeln Sie die SU-8-Form mit Trichloromethylsilane (TCMS) Dampf um PDMS Haftung (Silanisierung) zu reduzieren.
- Heben Sie PDMS durch Einstellung des Wafers horizontal auf einer ebenen Fläche und sorgfältig die PDMS Ablösen des Wafers ab. Nicht Knicken des Wafers nach oben während dieses Schrittes, da es die Form zu brechen.
Hinweis: Unvollständige Adhäsion von SU-8 auf den Wafer oder unvollständige Silanisierung führt zu Zerstörung der SU-8 Strukturen während dieses Schritts. - Schneiden Sie PDMS in einzelnen Chips um die Geräte, so dass die Geräte auf einem Deckgläschen 24 x 60 mm passt.
- Mit einem 1-mm-Biopsie-Punch, machen Sie Löcher in den Einlass, zwei Filialen und sechs Antriebe mit einem 1-mm-Biopsie-Punch. Ziel für die 2 mm Durchmesser Kreise um die Kanten des Geräts.
-
Bond PDMS-Chips Glas Deckel rutscht.
- Setzen Sie beide Oberflächen, 80 W Sauerstoffplasma (30 s).
- Legen Sie vorsichtig die freiliegende PDMS Oberfläche auf die herausgestellte Oberfläche des Deckglases für eine konforme Abdichtung.
- Tempern Sie das Gerät auf einer heißen Platte (100 ° C, 10 min).
Hinweis: Unzureichende Bindung kann zum Ausfall des Gerätes durch Leckage führen.
2. Vorbereitung des Mikroskops
Hinweis: Transgene Tiere: express einen Kalzium-Indikator wie GCaMP6s28 oder andere genetisch codierte Aktivität-Taster auf der Neuron(s) des Interesses (z.B.TRN); Co drücken Sie ein Aktivitäten-unabhängige fluoreszierenden Proteins emittierende auf einer anderen Wellenlänge, für kleine seitliche und Out-of-Focus Bewegungsartefakte durch mechanische Stimulation entstehen zu korrigieren aus. Die automatisierte Analyse-Software verfolgt und Bewegung verursachten Veränderungen in der Intensität der Aktivität Sonde kompensiert. Der Wurm Stämme GN69223 (oder AQ323629) GCaMP6s (oder GCaMP6m) ausdrücken und die Kalzium-unabhängige TagRFP unter der Kontrolle der Mec-7 Promotor. Darüber hinaus enthält GN692 die Mutation lite-1(ce314), die Aktivierung des TRNs durch das blaue Licht-Sensor lite-130 während der Erregung der GCaMP6s Fluoreszenz23verhindert.
-
Ein Mikroskopsystem für gleichzeitige Erregung GCaMP und RFP einrichten.
- Verwenden Sie entweder eine kontinuierliche Lichtquelle und ein dual-Band-Erregung-Filter, der nur Cyan und gelb Licht überträgt, oder verwenden Sie eine Lichtquelle, die nur Wellenlängen in einer definierten Bandbreite wie gleichzeitige Cyan emittiert (0,77 mW) und gelb (1.21 mW) LED Anregungsquellen für gleichzeitiger Anregung der Kalzium-abhängige und unabhängige Fluoreszenz, beziehungsweise.
- Verwenden Sie eine digitale Kamera zur Aufnahme von Mikroskopie-Bildern zu ermöglichen.
Hinweis: Die meisten Kameras verfügen über eine Software für die Bildaufnahme. Alternativ gibt es frei verfügbare Software, der verwendet werden kann, um die Kamera und möglicherweise auch andere Teile des Systems der Mikroskopie zu steuern. - Passen Sie Anregung Intensität anhand der Fluoreszenzintensität Kamera Sättigung zu vermeiden.
-
Verwenden Sie einen Fluoreszenz-Würfel, den abhängig von der Wahl der die Anregungsquelle muss mit einem Erregung-Filter ausgestattet werden.
Hinweis: Eine 488 nm GFP und 580 nm mCherry Erregung Filter wurde hier verwendet.- Fügen Sie einen Strahlteiler auf den Cube für Cyan und gelb Licht reflektiert und Übermittlung von grünem und rotem Licht.
- Statten Sie das Mikroskop mit einem 10-fach und einer hohen Vergrößerung Ziel (z.B.63 X / 1,32 NA Öl) das Anregungslicht auf die Probe zu konzentrieren.
- Montieren Sie einen Strahlteiler für gleichzeitige Aufnahme von der GCaMP und das Kalzium-unabhängige Signal vor der Kamera. Sicherzustellen, dass der Strahlteiler einen dichroitischen Spiegel hat (lange übergeben, Cut-off bei 570 nm), grünes und rotes Licht mit einer Emission Filter für Grün zu trennen (Durchlassbereich zentriert auf 525 nm mit 50 nm Breite) und eine Emission Filter für rotes Licht (Durchlassbereich zentriert auf 632 nm mit 60 nm Breite).
- Projekt-grüne und rote Fluoreszenz auf der oberen Hälfte (grün) und die untere Hälfte (rot), bzw. der Kamera chip (siehe Abbildung 3). Diese Ausrichtung ist Voraussetzung für die mitgelieferte Analysesoftware.
3. tierische Vorbereitung
- Bereiten Sie Alter-synchronisierte jungen Erwachsenen oder Erwachsenen Tag eins C. Elegans, vor, wie zuvor beschrieben von Porta-de-la-Riva Et al. 31
- Vorbereiten des Mikrofluidik-Chips.
- Schließen Sie die Schwerkraft fließen Reservoir (~ 60 cm über dem Chip) mit gefiltert (0,2 µm Polyethersulfone Spritze Filter) M9 Puffer an einer Steckdose des Chips. Schließen Sie die andere Steckdose an eine Steckdose von einem zwei-Ventil-Abfallbehälter, d. h., ein Filter-Fläschchen. Den andere Ausgang des Abfallbehälters an einer peristaltischen Pumpe anschließen.
Hinweis: Verwenden Sie Polyethylen (PE) Schläuche für alle Verbindungen und metallische Verschraubungen, um das PE-Rohr auf dem Chip zu verbinden. Auf diese Weise werden alle Lösungen aus dem Chip gespült und in den Abfallbehälter gesammelt. Der Durchfluss durch den Auslass Kanäle erzeugt eine sanfte Saugwirkung, die den Wurm bei der späteren Trapping gemütlich halten. - Bereiten Sie Verbindungen, bestehend aus 50 mm langen PE Rohre (0.9652-mm OD, 0.5842-mm ID) mit Metallmantel (Gauge Größe 23TW, 0,635 mm OD) an beiden Enden. Einpress-verbindet diese in jedem der sechs Betätigung Finger und in die Wurm-Inlet (siehe Abbildung 1). Lassen Sie diese Verbindungsleitungen in der Chip, wie wiederholte Entfernung führt auf die PDMS-Löcher zu tragen.
- Schließen Sie die Schwerkraft fließen Reservoir (~ 60 cm über dem Chip) mit gefiltert (0,2 µm Polyethersulfone Spritze Filter) M9 Puffer an einer Steckdose des Chips. Schließen Sie die andere Steckdose an eine Steckdose von einem zwei-Ventil-Abfallbehälter, d. h., ein Filter-Fläschchen. Den andere Ausgang des Abfallbehälters an einer peristaltischen Pumpe anschließen.
- Platzieren Sie den Chip am Mikroskop. Pick 2 – 5 Würmer in einem Tropfen gefiltert (0,2-µm-Spritze-Filter) M9 Puffer und eine 1-mL-Spritze verwenden, um sie in eine PE-Schlauch (0.9652-mm OD, 0.5842-mm ID) verbunden zu einer 1-mL-Spritze durch leichtes Ziehen des Kolbens aufstellen. Halten Sie die Tiere in das PE-Rohr und nicht in die Spritze.
Hinweis: Zu viele Würmer oder ungefilterte Lösungen im Chip können zu Verstopfung führen. - Verbinden Sie das PE-Rohr der Spritze die Leiterbahn an der Wurm (Abbildung 1 und Abbildung 2) des Chips. Aktivieren Sie der Schwerkraft fließen durch Öffnen des Ventils und starten Sie die peristaltische Pumpe. Drücken Sie dann sanft den Spritzenkolben der Spritze, die Tiere in der Trapping-Kanal zu bewegen, unter Beachtung des Kanals unter dem Mikroskop mit einem 10 X-Objektiv im Hellfeld-Modus.
Hinweis: Gelegentlich erscheinen Luftblasen kein Problem dar; Sie sind in der Regel automatisch in die Steckdose verschoben. Mehrere Tiere können "geparkt", in der Kammer warten und nacheinander verwendet werden.- Nach dem Laden des Tieres in der Trapping-Kanal (siehe auch Abbildung 2), stellen Sie seine Position durch sanft schieben oder ziehen den Kolben der Spritze um den Kopf des Tieres in der konischen Form der Front des Kanals zu positionieren.
- Stellen Sie sicher, dass der Wurm (Erwachsene Tag 1) hat die richtige Größe im Chip gefangen zu sein.
- Wenn der Wurm nicht den gesamten Querschnitt des Kanals aus der Nase füllt zu nahe das Ende des Körpers (nicht einschließlich die Schwanzspitze) oder der Wurm bewegt sich entlang der Achse des Kanals ohne Bewegung des Kolbens der Spritze , den Wurm durch den Kolben der Spritze drücken, bis es aus der Trapping-Kanal verschwindet zu entfernen und ein neues zu laden (siehe Punkt 3.8).
- Fluoreszenz-Mikroskop und eine höhere Vergrößerung Objektiv (40 X oder höher) Modus wechseln Sie und prüfen Sie, ob die Neuriten des Neurons von Interesse über die Membran einer der Antriebe zu liegen kommt. Wenn dies nicht der Fall ist, passen Sie die Position des Tieres durch ziehen oder schieben den Kolben. Wenn das nicht hilft, den Wurm zu entfernen und ein neues zu laden.
- Konzentrieren Sie sich auf den Zellkörper eines Neurons von Interesse und schließen Sie die Leiterbahn des Antriebs am nächsten an das Neuron auf der vorderen Seite der Zellkörper an eine programmierbare Druckpumpe mit PE-Schlauch an.
- Wenn das Experiment Messen des Abstands zwischen Antrieb und das Neuron erfordert, das Sichtfeld zu bewegen, so sind beide in das Sichtfeld und die Channelwall ist parallel zu den oberen und unteren Rand des Bildes.
- Definieren Sie eine Druck-Protokoll unter Verwendung der programmierbaren Druckpumpe.
Hinweis: Dieses Protokoll ist auf das gewünschte Experiment einstellbar.- Beginnen Sie mit einem konstanten Druck von 0 kPa eine Zeitlang mindestens 50 Bilder von der Bildsequenz (notwendig für die Normalisierung) entspricht. Für eine bildgebende Rate von 10 Hz dies entspricht 5 s. hinzufügen eine gewünschte Reiz-Wellenform und Druck und definieren die Länge der Impulse in einem zweiten Schritt.
Hinweis: Für die Stimulierung der TRNs, wird eine sinusförmige Wellenform (d.h.75 kPa, 10 Hz) überlagert mit einem Schritt (d.h. 275 kPa) empfohlen da es eine große neuronale Antwort erzeugt. - Wenn das Experiment zusätzliche Reize erfordert, umfassen einen Zeitraum von mindestens 10 s mit einem konstanten Druck von 0 kPa zwischen Reize. Vor dem Einschalten der Pumpe achten Sie Instrument Druck auf konstante 0 kPa, zufällige Stimulierung des Wurms vor dem eigentlichen Experiment zu vermeiden.
- Beginnen Sie mit einem konstanten Druck von 0 kPa eine Zeitlang mindestens 50 Bilder von der Bildsequenz (notwendig für die Normalisierung) entspricht. Für eine bildgebende Rate von 10 Hz dies entspricht 5 s. hinzufügen eine gewünschte Reiz-Wellenform und Druck und definieren die Länge der Impulse in einem zweiten Schritt.
- Die Bildgebung und Druck-Protokoll ausführen.
- Richten Sie in der Bild-Datenerfassungs-Software der Kamera eine Bildsequenz bei 10 Hz mit einer Belichtungszeit von 100 ms für die Dauer des Druck-Protokolls. Passen Sie die Anregung Intensität, so dass die Erhöhung der maximalen Fluoreszenz die Kamera nicht sättigen. Speichern Sie die Bilder als *.tif-Datei mit mindestens 50 Bilder vor den ersten Impuls für die Normalisierung.
- Starten Sie die Image-Protokoll in der Bild-Datenerfassungs-Software und dem Druck-Protokoll in der Software der programmierbaren Pumpe. Während der Aufnahme, beobachten Sie, ob das Neuron von Interesse die hellste Stelle auf einer Fläche von 10 x 10 Pixel ist, sich nicht weiter als 10 Pixel in aufeinander folgenden Bildern bewegt und während der Aufnahme in das Blickfeld bleibt.
Hinweis: Wenn dies nicht geschieht, wird die Analyse-Software nicht. Lichtblicke (d. h. Autofluoreszenz) rund um das Neuron machen Aufnahmen von den Neuronen schwierig zu reinigen. Um dieses Problem zu beheben, entfernen Sie den Wurm aus der Falle und laden Sie einen neuen Wurm in den Chip zu. Wenn der Wurm zu viel bewegt, versuchen Sie eine neue Aufnahme von den gleichen Wurm und beobachten Sie Wurm-Bewegung zu. Wenn das Problem weiterhin besteht möglicherweise der Wurm zu klein, um gefangen zu sein. In diesem Fall entfernen Sie diesen Wurm zu und laden Sie einen etwas größeren Wurm in die Falle. - Falls gewünscht, nehmen Sie Signale von mehreren Neuronen in das Sichtfeld gleichzeitig auf, solange die Neuronen während der gesamten Aufnahme von mindestens 10 Pixel getrennt sind.
- Um Gewöhnung zu untersuchen, führen Sie das Experiment wiederholt auf ein Tier.
- Entfernen Sie den Wurm aus der Trapping-Kanal.
- Wenn es gewünscht wird, um den Wurm für nachfolgende Untersuchungen zu halten, trennen Sie die Ausgänge des Chips in Richtung der Schwerkraft fließen und Abfallbehälter. Drücken Sie den Kolben der Spritze sanft, bis der ganze Wurm durch die Trapping-Kanal in den Strömungskanal geschoben wird.
- Drücken Sie den Spritzenkolben, bis das Tier in ein Tröpfchen außerhalb der Chip angezeigt wird. Trennen Sie die Spritze, einschließlich das PE-Rohr vom Wurm Einlass, aspirieren Sie die Würmer in den Tropfen und übertragen es auf einer Nährbodenplatte verwenden.
- Wenn es nicht gewünscht ist, den Wurm zu halten, entfernen Sie den Wurm durch den Kolben der Spritze drücken, bis der ganze Wurm durch die Trapping-Kanal in den Strömungskanal geschoben wird; die Schwerkraft fließen und die Absaugung der peristaltischen Pumpe tragen das Tier aus dem Chip und in den Abfallbehälter.
4. Analyse
- Downloaden Sie und installieren Sie der neuesten Fidschi Version32.
- Downloaden Sie und installieren Sie die neueste Java-SDK-Version.
Hinweis: Wenn nötig, löschen Sie oder benennen Sie den Java-Ordner im Ordner "Fidschi" Fidschi, die neu installierte Java-Compiler zu verwenden. -
Fidschi-Software zu öffnen.
- Überprüfen Sie, ob die Software durch die Eröffnung der neu installierten Java-Compiler ausgeführt wird "Plugins | Dienstprogramme | ImageJ | Eigenschaften.
- Laden Sie die Datei pokinganalyzer*.java (https://github.com/HFehlauer/Poking-Analyzer, siehe ergänzende Datei 2).
- Drag & drop die Java-Datei in das Softwarefenster zu öffnen als ein Plugin.
-
Kompilieren Sie und führen Sie die Datei pokinganalyzer*.java durch Drücken der Tasten STRG + R in Fidschi; eine grafische Benutzeroberfläche (stossen Analyzer) öffnet sich (Abbildung 3).
- Klicken Sie auf die Registerkarte "Hilfe" und lesen Sie mehr über die Anforderungen der Software und wie Sie die Analyse durchführen. Um zu beginnen, wählen Sie die Registerkarte "Open Video" und geben Sie den Speicherort der video Datei für den Analyzer: Navigieren Sie zum Speicherort des Videos klicken Sie auf "Video öffnen" und öffnen Sie es.
Hinweis: Die Software startet mit dieser Registerkarte geöffnet. Wenn es nicht geöffnet ist, beim Starten eine neue Analyse klicken Sie auf "Open Video". Wenn das Video im a.tif-Format ist, wird das Programm intern öffnen Sie das Video und das erste Bild im unteren Teil der Benutzeroberfläche angezeigt. Wenn das Bild nicht des Videos, der analysiert werden soll, klicken Sie auf die Taste "Öffnen Sie ein Video", um ein weiteres Video zu öffnen. - Um fortzufahren, klicken Sie auf der Registerkarte "Definieren ROIs". Legen Sie in dieser Registerkarte die Position von der TRN. Beachten Sie, dass das erste Bild des geöffneten Videos im unteren Teil der Registerkarte klicken Sie auf die Schaltfläche "Definieren TRN" angezeigt wird, und klicken Sie auf das Neuron im oberen Teil des angezeigten Bildes, die die GCaMP Fluoreszenz angezeigt werden sollen.
- Optional: Wenn der Antrieb in der oberen Hälfte des Bildes sichtbar ist kann das Programm automatisch den Abstand zwischen der Nervenzelle und den Antrieb verfolgen, wenn gewünscht. Überprüfen Sie hierzu "Definieren der Antrieb?" -Feld, klicken Sie auf die Schaltfläche "Definieren Antrieb" und klicken Sie auf den Antrieb in der oberen Hälfte des Bildes.
- Klicken Sie auf die Registerkarte "Analyse" und definieren die Erfassungsrate Bild. Geben Sie eine Zahl im Feld oder klicken Sie auf die oben oder unten Sie Schaltflächen zu erhöhen oder verringern Sie die Anzahl. Bei der vorherigen Registerkarte "Definieren der Antrieb?" -Kontrollkästchen ist aktiviert, die Größe der Kamera-Zellen, die Vergrößerung und den binning Faktor zu definieren, damit das Programm den Abstand berechnen kann.
- Klicken Sie auf die Schaltfläche "Analyse starten".
Hinweis: Das Programm wird nun das Neuron durch seine Fluoreszenz in der Bildsequenz in der oberen Hälfte verfolgen (Kalzium angewiesen) und in der unteren Hälfte (Kalzium unabhängige) die Bildsequenz. Eine Fläche von 10 x 10 Pixel rund um den Standort des Neurons in der vorherigen Abbildung die Position des Neurons in der folgenden Abbildung finden werden untersuchen, um Neuron Bewegung in der Ebene der Aufzeichnung des Programms zu berücksichtigen. Es wird die Fluoreszenz in beiden Hälften durch Korrektur für die Hintergrundfluoreszenz (Fbg) und dividiert durch die Fluoreszenz in den ersten 50 Bildern (F0) berechnen: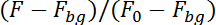
Fluoreszenz-Änderungen im grünen, aktivitätsabhängige Kanal (FCa2 +), die durch Neuronen entstehen Bewegung aus dem Flugzeug der Recodierung sind dann korrigiert mit Hilfe der Fluoreszenz in den roten, Aktivität-unabhängige Kanal (F Corr) und die Standardabweichung der Pre-Reiz der Kalzium-abhängige (sdCa2 +) und unabhängige Fluoreszenz (sdCorr) durch:
- Das Programm zeigt den Fortschritt in der Statusleiste im unteren Teil der Schnittstelle; Wenn die Statusleiste 100 % erreicht, klicken Sie auf die Registerkarte "Ergebnisse". Wenn der Fortschrittsbalken vor 100 % erteilt das Programm eine Fehlermeldung angezeigt, die den Grund, dass das Programm das Video nicht analysieren konnte.
- Wenn das Signal-Rausch-Verhältnis zu niedrig ist, kann das Programm nicht das Neuron identifizieren; passen Sie die bildgebenden Parameter im nachfolgenden Aufnahmen.
- Verlässt das Neuron das Sichtfeld während der Aufnahme, das Programm kann nicht mehr verfolgen und stoppt. Das Neuron in der Mitte das Sichtfeld Platz für spätere Aufnahmen zu Beginn der Aufnahme.
- Wenn einige Regionen in das Sichtfeld übersättigt sind, wird die automatisierte Analyse ungültige Ergebnisse produzieren. Sicherzustellen Sie für die nachfolgenden Aufnahmen, dass das Fluoreszenzsignal den Kamerasensor nicht sättigen.
- Das Ergebnis wird im Register "Ergebnisse" im oberen Teil angezeigt. Speichern Sie eine tabulatorgetrennte *.txt-Datei der Ergebnistabelle durch Klicken auf "Speichern der Tabelle".
Hinweis: Diese Tabelle besteht aus der Zeit in Sekunden und die normalisierte Kalzium-abhängige Fluoreszenz (korrigiert durch die Kalzium-unabhängige Fluoreszenz). Wenn zuvor definiert, es enthält auch der Gesamtabstand zwischen den TRN und des Antriebs in µm und der Abstand zwischen den TRN und der Betätiger senkrecht auf die Kanalwandung in µm. repräsentative Ergebnisse der Fluoreszenzintensität nach erhöhen Stimulation im Vergleich zu den Abstand der Zellkörper am nächsten Antrieb werden in Abbildung 4dargestellt. - Wenn mehrere Neuronen in einer Aufnahme (getrennt durch mehr als 10 Pixel) vorhanden sind, klicken Sie erneut auf die Registerkarte "Definieren ROIs" "und zurück zu Schritt 4.6.2 mit der zweiten Neuron.
- Klicken Sie auf die Registerkarte "Hilfe" und lesen Sie mehr über die Anforderungen der Software und wie Sie die Analyse durchführen. Um zu beginnen, wählen Sie die Registerkarte "Open Video" und geben Sie den Speicherort der video Datei für den Analyzer: Navigieren Sie zum Speicherort des Videos klicken Sie auf "Video öffnen" und öffnen Sie es.
Ergebnisse
SU-8 Lithographie und Chip Bonding
Die Lithographie Protokoll und PDMS-Spritzgießen folgen Standardverfahren. Details finden an anderer Stelle23,24,25,26. Die PDMS sollte nach dem Aushärten der Wafer ohne Probleme abziehen. Wenn die SU-8-Features während PDMS Peelings Abzocke, reichten die SU-8 Adhäsion Schicht oder der Silanisierung. Wen...
Diskussion
Dieses Protokoll veranschaulicht eine Methode für die Bereitstellung von präzisen mechanischen Stimulation der Haut ein Fadenwurm, gefangen in einem Mikrofluidik-Chip. Es soll die Integration der physikalischen Reize für biologische Fragen erleichtern und Mechanobiology Forschung im biologischen Labor optimieren soll. Diese Methode erweitert vorherigen Tests zur Beurteilung der Funktion der Mechanosensory Neuronen in C. Elegans. Vorherigen quantitative und semi-quantitativen Techniken gemessen Kräfte
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir bedanken uns bei Sandra N. Manosalvas-Kjono, Purim Ladpli, Farah Memon, Divya Gopisetty und Veronica Sanchez für Unterstützung im Gerätedesign und Generierung von mutierten Tieren. Diese Forschung wurde unterstützt durch NIH-Stipendien R01EB006745 (zur BLP), R01NS092099 (zu MBG), K99NS089942 (für MK), F31NS100318 (zu ALN) und empfangene Finanzierung vom European Research Council (ERC) unter Horizont 2020 Forschung und Innovation der Europäischen Union Programm () Finanzhilfevereinbarung Nr. 715243 MK).
Materialien
| Name | Company | Catalog Number | Comments |
| Chrome mask | Compugraphics (http://www.compugraphics-photomasks.com/) | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| Chrome mask | Mitani-Micronics (http://www.mitani-micro.co.jp/en/) | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| Chrome mask | Kuroda-Electric (http://www.kuroda-electric.eu/ | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| 4'' Silicon wafer (B-test) | Stanford Nanofabrication Facility | ||
| SU-8 2002 | MicroChem | ||
| SU-8 2050 | MicroChem | ||
| Spin-coater | Laurell Technologies | WS-400BZ-6NPP/LITE | |
| Exposure timer | Optical Associates, Inc | OAI 150 | |
| Illumination controller | Optical Associates, Inc | 2105C2 | |
| SU-8 developer | MicroChem | ||
| 2-Propanol | Fisher Scientific | A426F-1GAL | |
| Acetone | Fisher Scientific | A18-4 | |
| Trichloromethylsilane (TCMS) | Sigma-Aldrich | 92361-500ML | Caution: TCMS is toxic and water-reactive |
| Sylgard 184 Elastomer Kit | Dow Corning | PDMS prepolymer | |
| Biopsy punch, 1 mm | VWR | 95039-090 | |
| Oxygen Plasma Asher | Branson/IPC | ||
| Small metal tubing (0.635 mm OD, 0.4318 mm ID, 12.7 mm long); gage size 23TW | New England Small Tube Corporation | NE-1300-01 | |
| Nalgene syringe filter, 0.22 μm | Thermo Scientific | 725-2520 | to filter all solution, small particles would clog the chip |
| Polyethylene tubing; 0.9652 mm OD, 0.5842 mm ID | Solomon Scientific | BPE-T50 | |
| Syringe, 1 ml | BD Scientific | 309628 | for worm trapping and release |
| Syringe, 20 ml | BD Scientific | 309661 | for gravity-based flow |
| Gilson Minipuls 3, Peristaltic pump | Gilson | to suck solutions and worms out of the chip | |
| Microfluidic flow controller, equipped with 0–800 kPa pressure channel | Elveflow | OB1 MK3 | pressure delivery |
| Water-Resistant Clear Poly- urethane Tubing, 4 mm ID and 6 mm OD | McMaster-Carr | 5195 T52 | connection from house air to pressure pump |
| Water-Resistant Clear Polyurethane Tubing, 2.6mm ID and 4mm OD | McMaster-Carr | 5195 T51 | connect pressure pump to small tubng |
| Push-to-Connect Tube Fitting for Air | McMaster-Carr | 5111K468 | metric - imperial converter |
| Straight Connector for 6 mm × 1/4″ Tube OD | McMaster-Carr | 5779 K258 | |
| Leica DMI 4000 B microscopy system | Leica | ||
| 63×/1.32 NA HCX PL APO oil objective | Leica | 506081 | |
| Hamamatsu Orca-Flash 4.0LT digital CMOS camera | Hamamatsu | C11440-42U | |
| Lumencor Spectra X light engine | Lumencor | With cyan and green/yellow light source | |
| Excitation beam splitter | Chroma | 59022bs | in the microscope |
| Hamamatsu W-view Gemini Image splitting optics | Hamamatsu | A12801-01 | to split green and red emission and project them on different areas on the camera chip |
| Emission beam splitter | Chroma | T570lpxr | in the image splitter |
| Emission filters GCamp6s | Chroma | ET525/50m | in the image splitter |
| Emission filters mCherry | Chroma | ET632/60m | in the image splitter |
Referenzen
- Eastwood, A. L., et al. Tissue mechanics govern the rapidly adapting and symmetrical response to touch. Proc. Natl. Acad. Sci. 15 (50), E6955-E6963 (2015).
- Katta, S., Krieg, M., Goodman, M. B. Feeling Force: Physical and Physiological Principles Enabling Sensory Mechanotransduction. Annu. Rev. Cell Dev. Biol. 31, 347-371 (2015).
- Krieg, M., Dunn, A. R., Goodman, M. B. Mechanical systems biology of C. elegans touch sensation. BioEssays. 37 (3), 335-344 (2015).
- Krieg, M., Dunn, A. R., Goodman, M. B. Mechanical control of the sense of touch by β-spectrin. Nat. Cell Biol. 16 (3), 224-233 (2014).
- Krieg, M., et al. Tensile forces govern germ-layer organization in zebrafish. Nat Cell Biol. 10 (4), 429-436 (2008).
- Gaub, B. M., Müller, D. J. Mechanical stimulation of Piezo1 receptors depends on extracellular matrix proteins and directionality of force. Nano Lett. 17 (3), 2064-2072 (2017).
- Geffeney, S. L., et al. DEG/ENaC but not TRP channels are the major mechanoelectrical transduction channels in a c. Elegans nociceptor. Neuron. 71 (5), 845-857 (2011).
- O'Hagan, R., Chalfie, M., Goodman, M. B. The MEC-4 DEG/ENaC channel of Caenorhabditis elegans touch receptor neurons transduces mechanical signals. Nat. Neurosci. 8 (1), 43-50 (2005).
- Suzuki, H., et al. In Vivo Imaging of C. elegans Mechanosensory Neurons Demonstrates a Specific Role for the MEC-4 Channel in the Process of Gentle Touch Sensation. Neuron. 39 (6), 1005-1017 (2003).
- Kopito, R. B., Levine, E. Durable spatiotemporal surveillance of Caenorhabditis elegans response to environmental cues. Lab Chip. 14 (4), 764-770 (2014).
- Chokshi, T. V., Ben-Yakar, A., Chronis, N. CO2 and compressive immobilization of C. elegans on-chip. Lab Chip. 9 (1), 151 (2009).
- Hulme, S. E., Shevkoplyas, S. S., McGuigan, A. P., Apfeld, J., Fontana, W., Whitesides, G. M. Lifespan-on-a-chip: microfluidic chambers for performing lifelong observation of C. elegans. Lab Chip. 10 (5), 589-597 (2010).
- Li, S., Stone, H. a., Murphy, C. T. A microfluidic device and automatic counting system for the study of C. elegans reproductive aging. Lab Chip. 15 (2), 524-531 (2015).
- Chokshi, T. V., Bazopoulou, D., Chronis, N. An automated microfluidic platform for calcium imaging of chemosensory neurons in Caenorhabditis elegans. Lab Chip. 10 (20), 2758-2763 (2010).
- Mishra, B., et al. Using microfluidics chips for live imaging and study of injury responses in Drosophila larvae. J. Vis. Exp. , e50998 (2014).
- Chronis, N., Zimmer, M., Bargmann, C. I. Microfluidics for in vivo imaging of neuronal and behavioral activity in Caenorhabditis elegans. Nat. Methods. 4 (9), 727-731 (2007).
- Krajniak, J., Lu, H. Long-term high-resolution imaging and culture of C. elegans in chip-gel hybrid microfluidic device for developmental studies. Lab Chip. 10 (14), 1862-1868 (2010).
- Vasquez, V., Krieg, M., Lockhead, D., Goodman, M. B. Phospholipids that Contain Polyunsaturated Fatty Acids Enhance Neuronal Cell Mechanics and Touch Sensation. CellReports. 6 (1), 70-80 (2013).
- Krieg, M., et al. Genetic defects in β-spectrin and tau sensitize C. elegans axons to movement-induced damage via torque-tension coupling. Elife. 6 (2010), e20172 (2017).
- Arnadóttir, J., O'Hagan, R., Chen, Y., Goodman, M. B., Chalfie, M. The DEG/ENaC protein MEC-10 regulates the transduction channel complex in Caenorhabditis elegans touch receptor neurons. J. Neurosci. 31 (35), 12695-12704 (2011).
- Lockhead, D., et al. The tubulin repertoire of Caenorhabditis elegans sensory neurons and its context-dependent role in process outgrowth. Mol. Biol. Cell. 27 (23), 3717-3728 (2016).
- Goodman, M. B., Ernstrom, G. G., Chelur, D. S., O'hagan, R., Yao, C. A., Chalfie, M. MEC-2 regulates C. elegans DEG/ENaC channels needed for mechanosensation. Nature. 415 (6875), 1039-1042 (2002).
- Nekimken, A., Fehlauer, H., Kim, A., Goodman, M., Pruitt, B. L., Krieg, M. Pneumatic stimulation of C. elegans mechanoreceptor neurons in a microfluidic trap. Lab Chip. , (2017).
- Brower, K., White, A. K., Fordyce, P. M. Multi-step Variable Height Photolithography for Valved Multilayer Microfluidic Devices. J. Vis. Exp. (119), e55276 (2017).
- Jenkins, G. Rapid prototyping of PDMS devices using SU-8 lithography. Methods Mol. Biol. 949 (1), 153-168 (2013).
- Faustino, V., Catarino, S. O., Lima, R., Minas, G. Biomedical microfluidic devices by using low-cost fabrication techniques: A review. J. Biomech. 49 (11), 2280-2292 (2016).
- Xia, Y., Whitesides, G. M. SOFT LITHOGRAPHY. Annu. Rev. Mater. Sci. 28 (1), 153-184 (1998).
- Chen, T. -. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Cho, Y., Porto, D., Hwang, H., Grundy, L., Schafer, W. R., Lu, H. Automated and controlled mechanical stimulation and functional imaging in vivo in C. elegans. Lab Chip. , (2017).
- Edwards, S. L., et al. A novel molecular solution for ultraviolet light detection in Caenorhabditis elegans. PLoS Biol. 6 (8), 1715-1729 (2008).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Cerón, J. Basic Caenorhabditis elegans methods: synchronization and observation. J. Vis. Exp. (64), e4019 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat. Methods. 9 (7), 676-682 (2012).
- Cho, Y., Porto, D. A., Hwang, H., Grundy, L. J. High-Throughput Controlled Mechanical Stimulation and Functional Imaging In Vivo. BiorXiv. , (2017).
- Petzold, B. C., Park, S. -. J., Mazzochette, E. A., Goodman, M. B., Pruitt, B. L. MEMS-based force-clamp analysis of the role of body stiffness in C. elegans touch sensation. Integr. Biol. (Camb). 5 (6), 853-864 (2013).
- Nekimken, A. L., Mazzochette, E. A., Goodman, M. B., Pruitt, B. L. Forces applied during classical touch assays for Caenorhabditis elegans. PLoS One. 12 (5), e0178080 (2017).
- Chronis, N., Zimmer, M., Bargmann, C. I. Microfluidics for in vivo imaging of neuronal and behavioral activity in Caenorhabditis elegans. Nat. Methods. 4 (9), 727-731 (2007).
- Gilleland, C. L., Rohde, C. B., Zeng, F., Yanik, M. F. Microfluidic immobilization of physiologically active Caenorhabditis elegans. Nat. Protoc. 5 (12), 1888-1902 (2010).
- Chuang, H. -. S., Raizen, D. M., Lamb, A., Dabbish, N., Bau, H. H. Dielectrophoresis of Caenorhabditis elegans. Lab Chip. 11 (4), 599 (2011).
- Christensen, A. M., Chang-Yen, D. A., Gale, B. K. Characterization of interconnects used in PDMS microfluidic systems. J. Micromechanics Microengineering. 15 (5), 928-934 (2005).
- Gilpin, W., Uppaluri, S., Brangwynne, C. P. Worms under Pressure: Bulk Mechanical Properties of C. elegans Are Independent of the Cuticle. Biophys. J. 108 (8), 1887-1898 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten