Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
En utilisant un dispositif microfluidique pour une Stimulation mécanique et l’imagerie haute résolution de c. elegans
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nouveaux outils pour la recherche de mécanobiologie sont nécessaires pour comprendre les contraintes mécaniques comment active des voies biochimiques et suscite des réactions biologiques. Ici, nous présentons une nouvelle méthode de stimulation mécanique sélective des animaux immobilisée avec un piège de microfluidique permettant l’imagerie à haute résolution des réponses cellulaires.
Résumé
Un des objectifs centraux de mécanobiologie sont de comprendre l’effet réciproque de contraintes mécaniques sur les protéines et les cellules. Malgré son importance, l’influence des contraintes mécaniques sur la fonction cellulaire est encore mal compris. En partie, cette lacune existe parce que peu d’outils permettre une déformation simultanée de tissus et de cellules, imagerie de l’activité cellulaire chez les animaux vivants et restriction efficace de motilité dans les organismes modèle autrement très mobiles, tels que le nématode Caenorhabditis elegans. La petite taille de c. elegans rend un excellent match pour les dispositifs de recherche axée sur la microfluidique et des solutions pour l’immobilisation ont été présentées à l’aide de dispositifs microfluidiques. Bien que ces dispositifs permettent d’imagerie à haute résolution, l’animal est entièrement enfermée dans polydiméthylsiloxane (PDMS) et verre, limitant l’accès physique pour la livraison de force mécanique ou enregistrements électrophysiologiques. Récemment, nous avons créé un dispositif qui intègre des actionneurs pneumatiques avec un design de piégeage qui est compatible avec la microscopie de fluorescence à haute résolution. Le canal de commande est séparé du canal ver-piégeage par une fine membrane PDMS. Ce diaphragme est dévié dans le côté d’un ver en appliquant une pression d’une source externe. L’appareil peut cibler les neurones individuels mécanosensibles. L’activation de ces neurones est photographiée à haute résolution avec indicateurs de calcium codé génétiquement. Cet article présente la méthode générale en utilisant des souches de c. elegans exprimant l’indicateur d’activité sensible au calcium (GCaMP6s) dans les neurones de récepteur tactile (TRNs). La méthode, cependant, n’est pas limitée aux TRNs ni aux capteurs de calcium comme une sonde, mais peut être étendue à d’autres cellules sensibles à la mécanique ou les capteurs.
Introduction
Le sens du toucher fournit des animaux avec des informations cruciales sur leur environnement. Selon la force appliquée, touch est perçue comme anodine, agréable ou douloureux. La déformation du tissu pendant touch est détectée par des cellules spécialisées mécanorécepteur incorporés dans la peau qui expriment des protéines réceptrices, plus couramment des canaux ioniques. Les étapes reliant la perception de la force à l’activation de canal ionique lors de toucher et de la douleur ne sont pas totalement comprises. Encore moins sait comment le tissu cutané filtres déformation mécanique et si les mécanorécepteurs détecter des changements dans la souche ou souligner1,2,3. Cette lacune dans la compréhension découle, en partie, d’un manque d’outils appropriés pour appliquer les stimulations mécaniques précises à la surface de la peau d’un animal vivant tout en observant les réponses au niveau cellulaire. Considérant que la microscopie à force atomique a été largement utilisée pour appliquer et mesurer les forces dans des cellules isolées4,5 et aussi à activer les récepteurs Piezo1 la vie des cellules6, des expériences similaires à l’aide d’animaux vivants, en particulier C. elegans, ont été notoirement difficile en raison de la mobilité intrinsèque du sujet. Traditionnellement, ce défi est contourné à l’aide de colle cyanoacrylate vétérinaire - ou -qualité chirurgicale pour immobiliser les animaux individuels sur gélose tampons1,7,8,9. Cette approche a été productive, mais a des limites liées à la compétence requise pour l’immobilisation par collage et la surface de la gélose molle sur conformité mécanique. Une stratégie de microfluidique est une alternative gratuite qui permet d’éviter certaines des complications liées au collage.
Le nématode c. elegans est un organisme modèle génétique avec un système nerveux complètement mappé qui, en raison de la taille de l’animal, est un bon choix pour la technologie microfluidique. Dispositifs microfluidiques offre l’avantage que les animaux autrement extrêmement mobiles peuvent être immobilisés lors des opérations d’imagerie à haute résolution et livraison des stimuli neuro-modulateur pertinentes. Avec l’aide de microfluidique technologies, animaux vivants peuvent être immobilisés sans préjudice10,11, permettant la surveillance de l’activité comportementale sur la vie entière12,13 et haute résolution imagerie de l’activité neuronale14,15,16,17. En outre, plusieurs neurones mécanorécepteur nécessaires pour le sens du toucher et de la douleur peut être caractérisé sur leur physiologique1,8, mécanique4,18,19et moléculaire niveau20,21,22.
C. elegans détecte des stimuli mécaniques douces à son mur de corps à l’aide de six TRNs, dont trois innervent l’animal antérieur (ALML/R et AVM) et trois qui innervent postérieur de l’animal (PLML/R et PVM). Les molécules de canal d’ion nécessaires pour faire une force appliquée en un signal biochimique ont été largement étudiées dans son TRNs8. Cet article présente une plate-forme de microfluidique23 qui permet aux chercheurs d’appliquer des forces mécaniques précis sur la peau d’un immobilisé c. elegans ascaris, lors de la lecture sur la déformation de ses tissus internes par imagerie optique. En plus de présenter des stimuli mécaniques bien définis, transitoires de calcium peuvent être enregistrés dans les neurones des mécanorécepteurs avec résolution subcellulaire et en corrélation avec les caractéristiques morphologiques et anatomiques. Le dispositif se compose d’un canal central de piégeage qui détient un seul animal et présente sa peau à côté de six canaux d’actionnement pneumatique (Figure 1 et Figure 2). Les six chaînes sont positionnés le long du canal de piégeage pour livrer des stimuli mécaniques à chacun de six TRNs du ver. Ces chaînes sont séparées de la chambre de piégeage par des diaphragmes PDMS minces, qui peuvent être actionnés par une source de pression de l’air extérieur (Figure 1). Nous avons calibré la déviation par rapport à la pression et fournissent les mesures obtenues dans cet article. Chaque actionneur peut être adressée individuellement et utilisée pour stimuler un mécanorécepteur de choix. La pression est livrée à l’aide d’une pompe à pression pilotée par piezo mais n’importe quel autre dispositif peut être utilisé. Nous montrons que le protocole de pression peut être utilisé pour activer TRNs in vivo et de démontrer le fonctionnement des appareils adaptés pour fournir des stimuli mécaniques aux adultes de c. elegans, chargement des animaux adultes dans des dispositifs, exécution d’imagerie calcique expériences et analyser les résultats. Fabrication de dispositifs consistant en deux étapes principales : 1) photolithographie pour faire un moule de SU-8 ; et 2) PDMS pour faire un dispositif de moulage. Par souci de concision et de clarté, le lecteur est prié de précédemment publié articles et protocoles24,25 pour obtenir des instructions sur la façon de produire les moules et les périphériques.
Protocole
1. Fabrication de dispositifs
- Télécharger le fichier ci-joint masque (Supplemental fichier 1) et générer un masque de chrome à l’aide d’un service commercial ou une installation interne. Comme la plus petite dimension de l’appareil est de 10 µm (épaisseur de la membrane actionneur), assurez-vous que le masque a suffisamment haute résolution, au sein de ± 0,25 µm, pour produire avec fiabilité les caractéristiques.
-
Suivez les méthodes standard de photolithographie SU-8 (par exemple, fait référence à24,25,26) pour fabriquer le moule pour la production subséquente des dispositifs PDMS ; un résumé des étapes est listé ci-dessous.
NOTE : SU-8 est un matériau photosensible, quels croisements lors de l’exposition aux rayons ultraviolets (absorption maximale ~ 365 nm). Utilisez les instructions du fabricant comme point de départ pour déterminer les paramètres de traitement pour la cuisson douce, exposition (UV), les faire cuire.- Déposer les SU-8 2002 sur une plaquette de silicium et de spin-manteau d’une épaisseur approximative de 2 µm, pour une meilleure adhérence des petites structures. Soft-cuisson au four sur une plaque chauffante pendant 1 min à 95 ° C, légèrement surexposer surface (UV) l’ensemble (~ 100 mJ/cm2) et après cuisson au four sur une plaque chauffante pendant 2 min à 95 ° C.
- Spin-manteau, une couche épaisse de 47 µm de SU-8 2050 pour définir les fonctionnalités du dispositif. Utiliser une vitesse d’essorage de 500 tr/min pour 15 s (130 tr/min/s accélération) et puis 2 000 tr/min pour 90 s (260 tr/min/s accélération). Si nécessaire, répéter et régler la vitesse d’essorage pour obtenir l’épaisseur appropriée de résine photosensible.
- Soft-cuisson au four sur une plaque chauffante pendant 2 min à 65 ° C, puis 7 mn à 95 ° C. Exposer la résine photosensible selon les instructions du fabricant (~ 160 mJ/cm2) en utilisant un filtre de masque et de long-col de chrome pour assurer les parois latérales droites.
- Enlever la plaquette et les faire cuire sur une plaque chauffante pendant 1 min à 65 ° C et 7 mn à 95 ° C.
- Dissoudre les SU-8 non exposés avec le développeur et nettoyer avec 2-propanol.
- Faire cuire la galette sur une plaque de cuisson à 180 ° C pendant 30 min (dur-bake) afin de stabiliser les caractéristiques de la résine photosensible.
-
Faire une réplique PDMS du modèle SU-8 standard réplica méthodes27de moulage.
- Traiter le moule de SU-8 à vapeur trichloromethylsilane (SMTC) pour réduire l’adhérence PDMS (silanisation).
ATTENTION : SMTC est toxique et réagissant à l’eau.- Placer la plaquette à motifs dans un rack de plaquette dans un dessiccateur à vide cloche sous une hotte sans eau ou réactifs solubles dans l’eau.
- Sous le capot, utilisez un compte-gouttes pour déposer 1 goutte de SMTC dans un plat en verre et placer dans le dessiccateur.
- Fermer le couvercle de dessiccateur et permettre à vapeur de la SMTC enrober la plaquette pendant au moins 20 min.
- Ventiler et puis ouvrez le dessiccateur. Ouvrez le couvercle de la cloche de verre et enlever la plaquette à l’aide de brucelles en plastique. Placer dans un bateau de feuille boîte de Pétri ou aluminium pour le bâti de réplique PDMS. Disposer des matériaux revêtus SMTC dans les déchets dangereux appropriée.
- Mix de PDMS (ratio de 10:1) à l’aide de 40 g de la base polymère et 4 g PDMS de salaison.
- Versez le mélange sur le moule de SU-8 et dégazez le mélange pendant au moins 30 minutes dans une chambre à vide.
- Polymériser pendant au moins 6 h à 70 ° C dans un four.
- Traiter le moule de SU-8 à vapeur trichloromethylsilane (SMTC) pour réduire l’adhérence PDMS (silanisation).
- Soulevez PDMS la plaquette en définissant la gaufrette horizontale sur une surface plane et en décollant soigneusement le PDMS. Ne pas plier la plaquette vers le haut pendant cette étape, car il peut briser le moule.
NOTE : Adhésion incomplète de SU-8 à la plaquette ou la silanisation incomplète peut entraîner la destruction des structures SU-8 au cours de cette étape. - Couper PDMS autour des dispositifs en puces individuelles telles que les appareils seront adapte sur un lamelle couvre-objet 24 x 60 mm.
- À l’aide d’un poinçon de biopsie de 1 mm, percer des trous dans l’entrée, deux sorties et six actionneurs à l’aide d’un poinçon de biopsie de 1 mm. But pour les cercles de 2 mm de diamètre sur les bords de l’appareil.
-
Copeaux de PDMS Bond à lamelles de verre.
- Exposer les deux surfaces à 80 plasma d’oxygène W (30 s).
- Placez délicatement la surface PDMS exposée sur la surface exposée de la lamelle couvre-objet pour une étanchéité conforme.
- Recuire l’appareil sur une plaque chaude (100 ° C, 10 min).
NOTE : Collage insuffisante peut conduire à la défaillance de l’appareil due à une fuite.
2. préparation du Microscope
NOTE : Les animaux transgéniques : exprimer un indicateur de calcium comme GCaMP6s28 ou autre sonde activité génétiquement encodée dans le neuron(s) d’intérêt (p. ex., TRN) ; a exprimer une protéine fluorescente activité indépendante émettant à une longueur d’onde différente pour corriger les petite latérale et des artefacts de mouvement out-of-focus qui surviennent en raison d’une stimulation mécanique. Le logiciel d’analyse automatisée des pistes et compense les changements induits par le mouvement de l’intensité de la sonde de l’activité. Les souches GN692 ver23 (ou AQ323629) express GCaMP6s (ou GCaMP6m) et le tagRFP de calcium indépendantes sous le contrôle du promoteur de mec-7. En outre, GN692 contient la mutation lite-1(ce314), qui empêche l’activation de TRNs à cause du capteur de lumière bleue lite-130 au cours de l’excitation de la fluorescence de GCaMP6s23.
-
Mettre en place un système de microscope pour l’excitation simultanée de GCaMP et DP.
- Utilisez soit une source de lumière continue et un filtre d’excitation bi-bande qui transmet la lumière seulement cyan et jaune, ou une source lumineuse qui émet seulement des longueurs d’onde dans une largeur de bande définie comme cyan simultanée (0,77 mW) et jaune (1,21 mW) source d’excitation de LED pour excitation simultanée de la fluorescence de calcium-dépendante et indépendante, respectivement.
- Utiliser un appareil photo numérique pour activer l’enregistrement des images de microscopie.
NOTE : La plupart des caméras sont livrés avec un logiciel d’acquisition d’images. Sinon, il y a un logiciel librement disponible qui peut être utilisé pour contrôler la caméra et potentiellement aussi d’autres parties du système de microscopie. - Ajuster l’intensité d’excitation basée sur l’intensité de la fluorescence pour éviter la saturation de la caméra.
-
Utilisez un cube de fluorescence qui en fonction du choix de la source d’excitation doit être équipé d’un filtre d’excitation.
Remarque : Un GFP 488 nm et 580 nm mCherry filtre d’excitation ont été tenu ici.- Ajouter un séparateur de faisceau au cube réfléchissant la lumière cyan et jaune et de transmettre la lumière verte et rouge.
- Équiper le microscope avec un 10 X objectif et un fort grossissement (p. ex., 63 X / 1,32 huile NA) de se concentrer à l’excitation lumineuse sur l’échantillon.
- Monter un séparateur de faisceau devant la caméra pour l’enregistrement simultané de la GCaMP et le signal calcique indépendant. Veiller à ce que le séparateur de faisceau dispose d’un miroir dichroïque (longtemps passer, coupure à 570 nm) pour séparer la lumière verte et rouge à l’aide d’un filtre d’émission pour le vert (bande passante centrée à 525 nm à 50 nm largeur) et filtre d’une émission de lumière rouge (bande passante centrée à 632 nm avec 60 nm largeur).
- Une fluorescence verte et rouge sur la moitié supérieure du projet (vert) et la moitié inférieure (rouge), respectivement de la caméra à puce (voir Figure 3). Cette orientation est une condition sine qua non pour le logiciel d’analyse fournis.
3. animale préparation
- Préparer synchronisée à l’âge jeune adult ou adult jour un c. elegans, comme décrit plus haut par Porta-de-la-Riva et al. 31
- Préparer la puce microfluidique.
- Relier le réservoir de flux de gravité (~ 60 cm au-dessus du niveau de puce) contenant filtré tampon M9 (filtre de seringue polyéthersulfone de 0,2 µm) à une sortie de la puce. Connecter l’autre prise d’alimentation à une prise d’un conteneur à déchets deux-prise, c'est-à-dire, une fiole filtrante. Connecter l’autre sortie du récipient à déchets à une pompe péristaltique.
NOTE : Utilisez les tuyaux en polyéthylène (PE) pour toutes les connexions et raccords de tube en métal pour brancher le tuyau de PE à la puce. De cette façon toutes les solutions usées seront évacuées de la puce et recueillies dans le conteneur à déchets. Le débit à travers les canaux de sortie crée une légère succion qui gardera le ver bien ajusté au cours du processus de piégeage plus tard. - Préparer les interconnexions composé de 50 mm longs tubes PE (0.9652-mm OD, 0.5842-mm ID) avec tube métallique (jauge de taille 23TW, 0,635 mm OD) aux deux extrémités. Press-fit ces interconnexions dans chacun des six doigts actionnement et dans l’entrée de ver (voir Figure 1). Laissez ces interconnexions dans la puce, élimination répétée conduit à porter sur les trous PDMS.
- Relier le réservoir de flux de gravité (~ 60 cm au-dessus du niveau de puce) contenant filtré tampon M9 (filtre de seringue polyéthersulfone de 0,2 µm) à une sortie de la puce. Connecter l’autre prise d’alimentation à une prise d’un conteneur à déchets deux-prise, c'est-à-dire, une fiole filtrante. Connecter l’autre sortie du récipient à déchets à une pompe péristaltique.
- Placez le jeton sur le microscope. Worms 2 – 5 choix dans une goutte de filtré tampon M9 (filtre de seringue 0,2 µm) et utilisent une seringue de 1 mL pour les attirer vers le haut dans un tube PE (0.9652-mm OD, 0.5842-mm ID) relié à une seringue de 1 mL en tirant doucement sur le piston. Garder les animaux dans le tube PE et non dans la seringue.
NOTE : Trop de vers ou de solutions non filtrées dans la puce peuvent conduire à obstruer. - Raccordez le tuyau PE de la seringue à l’interconnexion à l’entrée de ver (Figure 1 et Figure 2) de la puce. Activer l’écoulement par gravité en ouvrant le robinet et démarrer la pompe péristaltique. Appuyez doucement sur le piston de la seringue de la seringue pour déplacer les animaux dans le canal de piégeage tout en observant le canal sous un microscope avec un objectif X 10 en mode fond clair.
NOTE : Parfois comparution bulles d’air ne posent un problème ; ils sont généralement automatiquement déplacés dans la prise. Plusieurs animaux peuvent être « rangé » dans la salle d’attente et utilisées dans l’ordre.- Après le chargement de l’animal dans le canal de piégeage (voir aussi la Figure 2), ajuster sa position en poussant doucement ou en tirant sur le piston de la seringue pour positionner la tête de l’animal dans la forme effilée de la façade de la chaîne.
- Assurez-vous que la vis sans fin (1 jour pour adultes) a la bonne taille pour être pris au piège dans la puce.
- Si le ver ne remplit pas la section entière de la chaîne du nez à la fin de l’organisme (sans compter l’extrémité de la queue) ou le ver se déplace le long de l’axe du chenal sans déplacer le piston de la seringue , supprimer le ver en appuyant sur le piston de la seringue jusqu'à ce qu’il disparaisse de la chaîne de piégeage et de charger une nouvelle (voir étape 3,8).
- Passer en mode de fluorescence de la loupe et une lentille de grossissement plus élevée (40 X ou plus récente) et vérifier si les neurites du neurone d’intérêt vient de mentir à travers la membrane de l’un des vérins. Si ce n’est pas le cas, ajustez la position de l’animal en tirant ou en poussant le piston. Si cela n’aide pas, supprimer le ver et charger une nouvelle.
- Mettre l’accent sur le corps cellulaire du neurone d’intérêt et connecter l’interconnexion de l’actionneur le plus proche de neurone sur la face antérieure du corps cellulaire à une pompe à pression programmable à l’aide de tubes PE.
- Si l’expérience nécessite de mesurer la distance entre l’actionneur et le neurone, déplacez le champ de vision, donc les deux sont dans le champ de vision et le Muralic est parallèle à l’arête supérieure et inférieure de l’image.
- Définir un protocole de pression à l’aide de la pompe de pression programmable.
Remarque : Ce protocole peut être ajusté à l’expérience souhaitée.- Commencez par une pression constante de 0 kPa pour un temps correspondant au moins 50 images de la séquence d’images (nécessaire pour la normalisation). Pour un taux d’imagerie de 10 Hz, cela correspond à 5 s. Ajouter une forme d’onde du stimulus désiré et pression et définir la longueur du stimulus dans un second temps.
Remarque : Pour stimuler les TRNs, une forme d’onde sinusoïdale de (c.-à-d., 75 kPa, 10Hz) superposée à une étape (c.-à-d., 275 kPa) est recommandé car il génère une grande réponse neuronale. - Si l’expérience nécessite des stimuli additionnels, incluent une période d’au moins 10 s à une pression constante de 0 kPa entre des stimuli. Avant d’allumer la pompe de pression, s’assurer que la pression de l’instrument est constante 0 kPa pour éviter la stimulation accidentelle du ver avant l’expérience réelle.
- Commencez par une pression constante de 0 kPa pour un temps correspondant au moins 50 images de la séquence d’images (nécessaire pour la normalisation). Pour un taux d’imagerie de 10 Hz, cela correspond à 5 s. Ajouter une forme d’onde du stimulus désiré et pression et définir la longueur du stimulus dans un second temps.
- Exécutez le protocole d’imagerie et de pression.
- Dans le logiciel d’acquisition image de la caméra, mettre en place une séquence d’images à 10 Hz à l’aide d’un temps d’exposition de 100 ms pour la durée du protocole de pression. Ajuster l’intensité d’excitation telle que l’augmentation de la fluorescence maximale ne pas saturer la caméra. Enregistrer les images dans un fichier *.tif au moins 50 images avant le premier stimulus pour la normalisation.
- Commencer le protocole d’imagerie dans le logiciel d’acquisition image et le protocole de pression dans le logiciel de la pompe programmable. Pendant l’enregistrement, observer si le neurone d’intérêt est l’endroit le plus lumineux dans une zone de 10 x 10 pixels, ne se déplace pas plus loin que 10 pixels en images séquentielles et reste dans le champ de vision lors de l’enregistrement.
Remarque : Si ce n’est pas fait, le logiciel d’analyse échoue. Des points lumineux (c.-à-d. l’autofluorescence) autour du neurone faire nettoyer les enregistrements des neurones difficiles. Pour corriger cela, retirez le ver du siphon et charger un nouveau ver dans la puce. Si le ver bouge trop, essayez un nouvel enregistrement du ver même et observez le mouvement de la vis sans fin. Si le problème persiste, le ver peut être trop petit pour être pris au piège. Dans ce cas, supprimer ce ver et charger un ver soit légèrement plus élevé dans le piège. - Si vous le souhaitez, enregistrer les signaux de plusieurs neurones dans le champ de vision simultanément tant que les neurones sont séparés au moins 10 pixels durant tout l’enregistrement.
- Pour étudier l’accoutumance, réaliser l’expérience de façon répétitive sur un animal.
- Supprimer le ver de la chaîne de piégeage.
- Si on désire garder le ver pour des études ultérieures, débrancher les prises de la puce vers l’écoulement par gravité et le conteneur à déchets. Appuyez doucement sur le piston de la seringue jusqu'à ce que le ver entier est poussé à travers le canal de piégeage dans le canal d’écoulement.
- Continuez à appuyer sur le piston de la seringue jusqu'à ce que l’animal apparaît dans une gouttelette à l’extérieur de la puce. Déconnecter la seringue dont le tube PE de l’entrée de ver, l’utiliser pour aspirer les vers dans la goutte et transférez-la sur une gélose.
- Si l'on ne veut pas garder le ver, supprimer le ver en appuyant sur le piston de la seringue jusqu'à ce que le ver entier est poussé à travers le canal de piégeage dans le chenal d’écoulement ; l’écoulement par gravité et l’aspiration de la pompe péristaltique porteront l’animal hors de la puce et dans le conteneur à déchets.
4. analyse
- Téléchargez et installez le plus récent Fidji version32.
- Téléchargez et installez la dernière version SDK de java.
Remarque : Si nécessaire, supprimez ou renommez le dossier java dans le dossier de Fidji pour Fidji à utiliser le compilateur java nouvellement installé. -
Ouvrez le logiciel de Fidji.
- Vérifiez si le logiciel s’exécute le compilateur java nouvellement installé en ouvrant ' Plugins | Services publics | ImageJ | Propriétés.
- Télécharger le fichier pokinganalyzer*.java (https://github.com/HFehlauer/Poking-Analyzer, voir 2 de fichier supplémentaire).
- Glissez et déposez le fichier .java dans la fenêtre du logiciel pour l’ouvrir comme un plugin.
-
Compilez et exécutez le fichier pokinganalyzer*.java en appuyant sur les touches Ctrl + R à Fidji ; une interface utilisateur graphique (analyseur de piquer) s’ouvre (Figure 3).
- Cliquez sur l’onglet « Aide » et découvrez les exigences du logiciel et comment effectuer l’analyse. Pour commencer, sélectionnez l’onglet « Open video » précisez l’emplacement du fichier vidéo de l’analyseur : cliquez sur le bouton « Ouvrir une vidéo », naviguez jusqu'à l’emplacement de la vidéo et ouvrez-le.
Remarque : Le logiciel commence avec cet onglet ouvert. S’il n’est pas ouvert lors du démarrage d’une nouvelle analyse cliquez sur l’onglet de « Open video ». Si la vidéo est au format a.tif, le programme ouvrir la vidéo en interne et afficher la première image dans la partie inférieure de l’interface. Si l’image n’est pas de la vidéo qui doit être analysée, cliquez sur le bouton « Ouvrir une vidéo » pour ouvrir une autre vidéo. - Pour continuer, cliquez sur l’onglet « Définir des ROIs ». Dans cet onglet, définir la position de la TRN. Notez que la première image de la vidéo ouverte s’affiche dans la partie inférieure de cet onglet cliquer sur le bouton « Définir la TRN » et cliquez sur le neurone dans la partie supérieure de l’image affichée, qui devrait afficher la fluorescence GCaMP.
- Facultatif : Si le vérin est visible dans la moitié supérieure de l’image le programme peut automatiquement suivre la distance entre le neurone et l’actionneur si vous le souhaitez. Pour ce faire, cochez la case « Définir l’actionneur ? » -zone, cliquez sur le bouton « Définir le déclencheur », puis cliquez sur l’actionneur dans la moitié supérieure de l’image.
- Cliquez sur l’onglet « Analyse » définir la fréquence d’acquisition des images. Entrez un nombre dans le champ ou cliquez sur le haut ou vers le bas des boutons pour augmenter ou diminuer le nombre. If dans l’onglet précédent la « définir l’actionneur ? » -case à cocher est activée, définissez la taille de cellule de caméra, le grossissement et le facteur de binning, afin que le programme peut calculer la distance.
- Cliquez sur le bouton « Démarrer l’analyse ».
NOTE : Le programme va désormais suivre le neurone par sa fluorescence dans la séquence d’images dans la moitié supérieure (calcium-dépendante) et dans la moitié inférieure (calcium indépendant) de la séquence d’images. Pour tenir compte de la proposition de neurone dans le plan de l’enregistrement du programme portera sur une superficie de 10 x 10 pixels autour de l’emplacement du neurone dans l’image précédente pour trouver la position du neurone dans l’image suivante. Il calculera la fluorescence dans les deux moitiés de correction pour la fluorescence de fond (Fbg) et en divisant par la fluorescence dans les 50 premières images (F0) :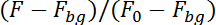
Les changements de fluorescence dans le canal vert, activité-dépendante (FCa2 +) qui sont causés par le neurone de mouvement hors de l’avion de recodage sont alors corrigées à l’aide de la fluorescence dans le canal rouge, activité indépendante (F corr) et l’écart-type de stimulation préalable de la calcium-dépendante (sdCa2 +) et la fluorescence indépendant (sdcorr) par :
- Le programme va montrer sa progression dans la barre d’État dans la partie inférieure de l’interface ; Lorsque la barre d’état atteint 100 %, cliquez sur l’onglet « Résultats ». Si la barre d’État s’arrête avant 100 %, le programme donnera un message d’erreur indiquant la raison pour laquelle que le programme ne pourrait pas analyser la vidéo.
- Si le rapport signal sur bruit est trop faible, le programme ne peut pas identifier le neurone ; Ajustez les paramètres d’imagerie dans les enregistrements ultérieurs.
- Si le neurone quitte le champ de vision lors de l’enregistrement, le programme ne peut pas le suivre plus et s’arrête. Pour les enregistrements ultérieurs, placez le neurone dans le milieu du champ de vision au début de l’enregistrement.
- Si certaines régions dans le champ de vision sont saturées, l’analyse automatique va produire des résultats non valides. Pour les enregistrements suivants, assurez-vous que le signal de fluorescence ne pas saturer le capteur de la caméra.
- Dans l’onglet « résultats », le résultat s’affiche dans la partie supérieure. Enregistrer un fichier *.txt séparées par des tabulations de la table de résultats en cliquant sur « Enregistrer la Table de résultat ».
Remarque : Ce tableau se compose du temps en secondes et la fluorescence normalisée de calcium-dépendante (corrigée par la fluorescence indépendantes du calcium). Si précédemment défini, il contient également la distance totale entre le TRN et l’actionneur en µm et la distance entre le TRN et l’actionneur perpendiculaire à la paroi du canal en µm. des résultats représentatifs de l’intensité de la fluorescence augmentent après stimulation par rapport à la distance du corps cellulaire à l’actionneur le plus proche sont tracés sur la Figure 4. - S’il y a plusieurs neurones dans un enregistrement (séparé par plus de 10 pixels), cliquez sur l’onglet de « Définir des ROIs » à nouveau et retournez à l’étape 4.6.2 avec le deuxième neurone.
- Cliquez sur l’onglet « Aide » et découvrez les exigences du logiciel et comment effectuer l’analyse. Pour commencer, sélectionnez l’onglet « Open video » précisez l’emplacement du fichier vidéo de l’analyseur : cliquez sur le bouton « Ouvrir une vidéo », naviguez jusqu'à l’emplacement de la vidéo et ouvrez-le.
Résultats
Lithographie de SU-8 et Chip Bonding
Le protocole de la lithographie et la moulure de PDMS conformément aux procédures standard. Détails peuvent être trouvés ailleurs23,24,25,26. Le PDMS devrait décoller de la plaquette sans problèmes après durcissement. Si les caractéristiques de SU-8 arnaquer au cours de l’épluchage de PDMS, soi...
Discussion
Ce protocole présente une méthode pour fournir une stimulation mécanique précise à la peau d’un ver rond, pris au piège dans une puce microfluidique. Il est destiné à faciliter l’intégration des stimuli physiques pour répondre aux questions biologiques et vise à rationaliser la mécanobiologie recherche dans les laboratoires biologiques. Cette méthode s’étend des analyses précédentes afin d’évaluer la fonction des neurones mécanosensoriels chez c. elegans. Techniques quantitatives et sem...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Sandra N. Manosalvas-Kjono, Pourim Ladpli, Farah Memon, Divya Gopisetty et Veronica Sanchez pour soutiennent dans la conception de l’appareil et la génération des animaux mutants. Cette recherche a été financée par les NIH subventions R01EB006745 (BLP), R01NS092099 (à MBG), K99NS089942 (à MK), F31NS100318 (à l’ALN) et bénéficié d’un financement du Conseil européen de recherche (CER) au titre de la recherche Horizon 2020 de l’Union européenne et de l’innovation program () Convention de subvention no 715243 à MK).
matériels
| Name | Company | Catalog Number | Comments |
| Chrome mask | Compugraphics (http://www.compugraphics-photomasks.com/) | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| Chrome mask | Mitani-Micronics (http://www.mitani-micro.co.jp/en/) | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| Chrome mask | Kuroda-Electric (http://www.kuroda-electric.eu/ | 5'', designed in AutoCAD (Autodesk, Inc.) | |
| 4'' Silicon wafer (B-test) | Stanford Nanofabrication Facility | ||
| SU-8 2002 | MicroChem | ||
| SU-8 2050 | MicroChem | ||
| Spin-coater | Laurell Technologies | WS-400BZ-6NPP/LITE | |
| Exposure timer | Optical Associates, Inc | OAI 150 | |
| Illumination controller | Optical Associates, Inc | 2105C2 | |
| SU-8 developer | MicroChem | ||
| 2-Propanol | Fisher Scientific | A426F-1GAL | |
| Acetone | Fisher Scientific | A18-4 | |
| Trichloromethylsilane (TCMS) | Sigma-Aldrich | 92361-500ML | Caution: TCMS is toxic and water-reactive |
| Sylgard 184 Elastomer Kit | Dow Corning | PDMS prepolymer | |
| Biopsy punch, 1 mm | VWR | 95039-090 | |
| Oxygen Plasma Asher | Branson/IPC | ||
| Small metal tubing (0.635 mm OD, 0.4318 mm ID, 12.7 mm long); gage size 23TW | New England Small Tube Corporation | NE-1300-01 | |
| Nalgene syringe filter, 0.22 μm | Thermo Scientific | 725-2520 | to filter all solution, small particles would clog the chip |
| Polyethylene tubing; 0.9652 mm OD, 0.5842 mm ID | Solomon Scientific | BPE-T50 | |
| Syringe, 1 ml | BD Scientific | 309628 | for worm trapping and release |
| Syringe, 20 ml | BD Scientific | 309661 | for gravity-based flow |
| Gilson Minipuls 3, Peristaltic pump | Gilson | to suck solutions and worms out of the chip | |
| Microfluidic flow controller, equipped with 0–800 kPa pressure channel | Elveflow | OB1 MK3 | pressure delivery |
| Water-Resistant Clear Poly- urethane Tubing, 4 mm ID and 6 mm OD | McMaster-Carr | 5195 T52 | connection from house air to pressure pump |
| Water-Resistant Clear Polyurethane Tubing, 2.6mm ID and 4mm OD | McMaster-Carr | 5195 T51 | connect pressure pump to small tubng |
| Push-to-Connect Tube Fitting for Air | McMaster-Carr | 5111K468 | metric - imperial converter |
| Straight Connector for 6 mm × 1/4″ Tube OD | McMaster-Carr | 5779 K258 | |
| Leica DMI 4000 B microscopy system | Leica | ||
| 63×/1.32 NA HCX PL APO oil objective | Leica | 506081 | |
| Hamamatsu Orca-Flash 4.0LT digital CMOS camera | Hamamatsu | C11440-42U | |
| Lumencor Spectra X light engine | Lumencor | With cyan and green/yellow light source | |
| Excitation beam splitter | Chroma | 59022bs | in the microscope |
| Hamamatsu W-view Gemini Image splitting optics | Hamamatsu | A12801-01 | to split green and red emission and project them on different areas on the camera chip |
| Emission beam splitter | Chroma | T570lpxr | in the image splitter |
| Emission filters GCamp6s | Chroma | ET525/50m | in the image splitter |
| Emission filters mCherry | Chroma | ET632/60m | in the image splitter |
Références
- Eastwood, A. L., et al. Tissue mechanics govern the rapidly adapting and symmetrical response to touch. Proc. Natl. Acad. Sci. 15 (50), E6955-E6963 (2015).
- Katta, S., Krieg, M., Goodman, M. B. Feeling Force: Physical and Physiological Principles Enabling Sensory Mechanotransduction. Annu. Rev. Cell Dev. Biol. 31, 347-371 (2015).
- Krieg, M., Dunn, A. R., Goodman, M. B. Mechanical systems biology of C. elegans touch sensation. BioEssays. 37 (3), 335-344 (2015).
- Krieg, M., Dunn, A. R., Goodman, M. B. Mechanical control of the sense of touch by β-spectrin. Nat. Cell Biol. 16 (3), 224-233 (2014).
- Krieg, M., et al. Tensile forces govern germ-layer organization in zebrafish. Nat Cell Biol. 10 (4), 429-436 (2008).
- Gaub, B. M., Müller, D. J. Mechanical stimulation of Piezo1 receptors depends on extracellular matrix proteins and directionality of force. Nano Lett. 17 (3), 2064-2072 (2017).
- Geffeney, S. L., et al. DEG/ENaC but not TRP channels are the major mechanoelectrical transduction channels in a c. Elegans nociceptor. Neuron. 71 (5), 845-857 (2011).
- O'Hagan, R., Chalfie, M., Goodman, M. B. The MEC-4 DEG/ENaC channel of Caenorhabditis elegans touch receptor neurons transduces mechanical signals. Nat. Neurosci. 8 (1), 43-50 (2005).
- Suzuki, H., et al. In Vivo Imaging of C. elegans Mechanosensory Neurons Demonstrates a Specific Role for the MEC-4 Channel in the Process of Gentle Touch Sensation. Neuron. 39 (6), 1005-1017 (2003).
- Kopito, R. B., Levine, E. Durable spatiotemporal surveillance of Caenorhabditis elegans response to environmental cues. Lab Chip. 14 (4), 764-770 (2014).
- Chokshi, T. V., Ben-Yakar, A., Chronis, N. CO2 and compressive immobilization of C. elegans on-chip. Lab Chip. 9 (1), 151 (2009).
- Hulme, S. E., Shevkoplyas, S. S., McGuigan, A. P., Apfeld, J., Fontana, W., Whitesides, G. M. Lifespan-on-a-chip: microfluidic chambers for performing lifelong observation of C. elegans. Lab Chip. 10 (5), 589-597 (2010).
- Li, S., Stone, H. a., Murphy, C. T. A microfluidic device and automatic counting system for the study of C. elegans reproductive aging. Lab Chip. 15 (2), 524-531 (2015).
- Chokshi, T. V., Bazopoulou, D., Chronis, N. An automated microfluidic platform for calcium imaging of chemosensory neurons in Caenorhabditis elegans. Lab Chip. 10 (20), 2758-2763 (2010).
- Mishra, B., et al. Using microfluidics chips for live imaging and study of injury responses in Drosophila larvae. J. Vis. Exp. , e50998 (2014).
- Chronis, N., Zimmer, M., Bargmann, C. I. Microfluidics for in vivo imaging of neuronal and behavioral activity in Caenorhabditis elegans. Nat. Methods. 4 (9), 727-731 (2007).
- Krajniak, J., Lu, H. Long-term high-resolution imaging and culture of C. elegans in chip-gel hybrid microfluidic device for developmental studies. Lab Chip. 10 (14), 1862-1868 (2010).
- Vasquez, V., Krieg, M., Lockhead, D., Goodman, M. B. Phospholipids that Contain Polyunsaturated Fatty Acids Enhance Neuronal Cell Mechanics and Touch Sensation. CellReports. 6 (1), 70-80 (2013).
- Krieg, M., et al. Genetic defects in β-spectrin and tau sensitize C. elegans axons to movement-induced damage via torque-tension coupling. Elife. 6 (2010), e20172 (2017).
- Arnadóttir, J., O'Hagan, R., Chen, Y., Goodman, M. B., Chalfie, M. The DEG/ENaC protein MEC-10 regulates the transduction channel complex in Caenorhabditis elegans touch receptor neurons. J. Neurosci. 31 (35), 12695-12704 (2011).
- Lockhead, D., et al. The tubulin repertoire of Caenorhabditis elegans sensory neurons and its context-dependent role in process outgrowth. Mol. Biol. Cell. 27 (23), 3717-3728 (2016).
- Goodman, M. B., Ernstrom, G. G., Chelur, D. S., O'hagan, R., Yao, C. A., Chalfie, M. MEC-2 regulates C. elegans DEG/ENaC channels needed for mechanosensation. Nature. 415 (6875), 1039-1042 (2002).
- Nekimken, A., Fehlauer, H., Kim, A., Goodman, M., Pruitt, B. L., Krieg, M. Pneumatic stimulation of C. elegans mechanoreceptor neurons in a microfluidic trap. Lab Chip. , (2017).
- Brower, K., White, A. K., Fordyce, P. M. Multi-step Variable Height Photolithography for Valved Multilayer Microfluidic Devices. J. Vis. Exp. (119), e55276 (2017).
- Jenkins, G. Rapid prototyping of PDMS devices using SU-8 lithography. Methods Mol. Biol. 949 (1), 153-168 (2013).
- Faustino, V., Catarino, S. O., Lima, R., Minas, G. Biomedical microfluidic devices by using low-cost fabrication techniques: A review. J. Biomech. 49 (11), 2280-2292 (2016).
- Xia, Y., Whitesides, G. M. SOFT LITHOGRAPHY. Annu. Rev. Mater. Sci. 28 (1), 153-184 (1998).
- Chen, T. -. W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Cho, Y., Porto, D., Hwang, H., Grundy, L., Schafer, W. R., Lu, H. Automated and controlled mechanical stimulation and functional imaging in vivo in C. elegans. Lab Chip. , (2017).
- Edwards, S. L., et al. A novel molecular solution for ultraviolet light detection in Caenorhabditis elegans. PLoS Biol. 6 (8), 1715-1729 (2008).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Cerón, J. Basic Caenorhabditis elegans methods: synchronization and observation. J. Vis. Exp. (64), e4019 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat. Methods. 9 (7), 676-682 (2012).
- Cho, Y., Porto, D. A., Hwang, H., Grundy, L. J. High-Throughput Controlled Mechanical Stimulation and Functional Imaging In Vivo. BiorXiv. , (2017).
- Petzold, B. C., Park, S. -. J., Mazzochette, E. A., Goodman, M. B., Pruitt, B. L. MEMS-based force-clamp analysis of the role of body stiffness in C. elegans touch sensation. Integr. Biol. (Camb). 5 (6), 853-864 (2013).
- Nekimken, A. L., Mazzochette, E. A., Goodman, M. B., Pruitt, B. L. Forces applied during classical touch assays for Caenorhabditis elegans. PLoS One. 12 (5), e0178080 (2017).
- Chronis, N., Zimmer, M., Bargmann, C. I. Microfluidics for in vivo imaging of neuronal and behavioral activity in Caenorhabditis elegans. Nat. Methods. 4 (9), 727-731 (2007).
- Gilleland, C. L., Rohde, C. B., Zeng, F., Yanik, M. F. Microfluidic immobilization of physiologically active Caenorhabditis elegans. Nat. Protoc. 5 (12), 1888-1902 (2010).
- Chuang, H. -. S., Raizen, D. M., Lamb, A., Dabbish, N., Bau, H. H. Dielectrophoresis of Caenorhabditis elegans. Lab Chip. 11 (4), 599 (2011).
- Christensen, A. M., Chang-Yen, D. A., Gale, B. K. Characterization of interconnects used in PDMS microfluidic systems. J. Micromechanics Microengineering. 15 (5), 928-934 (2005).
- Gilpin, W., Uppaluri, S., Brangwynne, C. P. Worms under Pressure: Bulk Mechanical Properties of C. elegans Are Independent of the Cuticle. Biophys. J. 108 (8), 1887-1898 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon