Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un análisis basado en la citometría de flujo para identificar compuestos que alteran la Unión del ligando de quimiocina CXC marcada con fluorescencia 12 al CXC Chemokine Receptor 4
En este artículo
Resumen
Un flujo de ensayo de enlace celular basado en la citometría se describe que se utiliza principalmente como una herramienta de detección para identificar compuestos que inhiben el atascamiento de un ligand de quimiocina CXC fluorescencia etiquetado 12 (CXCL12) para el receptor de quimiocina CXC 4 (CXCR4).
Resumen
Orientación farmacológica de receptores acoplados a proteína G (GPCRs) es de gran importancia para la salud humana, como mediada de GPCR señalización disfuncional contribuye a la progresión de muchas enfermedades. El par de ligando/receptor ligando de quimiocina CXC 12 (CXCL12) / receptor de quimiocina CXC 4 (CXCR4) ha levantado gran interés clínico, por ejemplo como un objetivo potencial para el tratamiento de cáncer y enfermedades inflamatorias. Pequeñas moléculas como anticuerpos terapéuticos que específicamente objetivo CXCR4 e inhiben la función del receptor por lo tanto se consideran como valiosas herramientas farmacológicas. Aquí, se describe un análisis celular flujo base de citometría que permite la identificación de compuestos (por ejemplo, moléculas pequeñas) que derogar CXCL12 vinculante para CXCR4. Esencialmente, el análisis se basa en la competencia por el receptor vinculante entre una cantidad fija de fluorescencia etiquetada CXCL12, el agonista natural chemokine para CXCR4 y compuestos sin etiqueta. Por lo tanto, el indeseable uso de sondas marcadas radiactivamente se evita en este ensayo. Además, las células vivas se utilizan como la fuente del receptor (CXCR4) en lugar de preparaciones de membrana de la célula. Esto permite la fácil adaptación del ensayo a un formato de placa, lo que aumenta el rendimiento. Este ensayo ha demostrado ser un ensayo de descubrimiento valioso medicamento genérico para identificar compuestos dirigidos a CXCR4. El protocolo probablemente adaptables a otros GPCRs, al menos si fluorescencia etiquetadas ligandos están disponibles o pueden generarse. No se requiere conocimiento previo sobre las vías de señalización intracelulares que inducen en la activación de los GPCRs.
Introducción
Receptores acoplados a proteína G (GPCRs) son proteínas de la superficie celular que pueden ser activadas por ligandos extracelulares (por ejemplo, péptidos, hormonas proteicas, aminas), tal modo de regulación fisiológica y del desarrollo de procesos1. Cuando un agonista ocupa su bolsillo de unión de GPCR, el inducido cambio conformacional en la proteína del receptor promueve la Unión de proteínas intracelular asociado a receptor heterotriméricas G, de Gα/ PIB y Gβγ subunidades. El posterior intercambio de GTP para el PIB en los resultados de la subunidadα de G en la disociación de las subunidades de la proteína de G (Gα-GTP y Gβγ) que, a su vez, más inicia aguas abajo de señalización vías2,3. Cuando el Gα-GTP se convierte hidrolizado, la Asociación de laαde la G / PIB y Gβγ subunidades convertirá la proteína G a su reposo estado3,4. Distintos Existen tipos de proteínas G (Gs, Gentrada-salida, Gq, G12/13), que se clasifican basados en la similitud de secuencia con el de subunidadα G5. Todas estas proteínas G inducen definidas vías de señalización intracelular que subyacen a la respuesta biológica a la activación del receptor. Con posterioridad a la activación del receptor, kinasas GPCR (GRKs) fosforilan la cola intracelular de los GPCRs, promoviendo así la interacción con β-arrestins. Este proceso conduce a la terminación de la proteína G señalización, desensibilización e internalización de receptores6. Β-arrestins también forman parte de complejos multi-moleculares que gatillo señalización cascadas independientes de la proteína G señalización7.
GPCRs son entre las dianas moleculares más validados para la intervención terapéutica, como desregulados GPCR-mediada de señalización, por ejemplo debido a las mutaciones de la ganar-de-función en el gene del receptor o sobreexpresión del receptor, contribuye a la etiología de muchas enfermedades humanas8. Por lo tanto, los GPCRs representan una de las más importantes clases de drogas objetivos investigados por la industria farmacéutica8,9,10. Un ejemplo notable de un GPCR clínicamente relevante es el receptor de quimiocina CXC 4 (CXCR4), que puede ser activado por un ligando natural único, la CXC chemokine ligando 12 (CXCL12)11. Debido a su papel establecido como un co-receptor importante para virus de inmunodeficiencia humana 1 (VIH-1) entrada e infección en cúmulo de diferenciación 4 (CD4) positivo linfocitos T12, CXCR4 se investigó en primer lugar como objetivo de un fármaco antiviral. Interacción de CXCL12-CXCR4 en la médula más regula la retención y autoguiado hacia el blanco del vástago y del progenitor células13. También, dada su participación en muchos aspectos del cáncer Biología (p. ej., supervivencia de la célula del tumor, metástasis y angiogénesis tumorales)14 y varias otras enfermedades humanas (p. ej., enfermedades inflamatorias)15, CXCR4 levantado gran interés como un prometedor objetivo de descubrimiento de fármacos. AMD3100, una pequeña molécula que se dirige específicamente a CXCR4, fue descubierto inicialmente como un anti-VIH drogas candidato16 y sigue siendo uno de los más potentes antagonistas CXCR4 descritos hasta la fecha17. Su desarrollo como un fármaco antiviral, sin embargo, suspendió18. Esta molécula se utiliza actualmente como un agente de movilización de células madre durante el tratamiento de mieloma múltiple y linfoma pacientes18. Otras químicamente relacionado con pequeñas moléculas y productos biológicos que inhiben la función de CXCR4 con potencia diferente han sido descritas19.
Métodos de unión del receptor son herramientas valiosas en farmacología que permiten la identificación de compuestos (por ejemplo, moléculas pequeñas) que interactúan directamente con el GPCR de interés. Para llevar a cabo estudios de la Unión, no existe necesidad de conocimientos previos acerca de la propiedades de señalización intracelulares o funcionalidad de un determinado GPCR. Aunque esto puede considerarse una ventaja, implica que compuestos para que receptor pueden demostrarse vinculantes deben caracterizarse aún más mediante la evaluación de su potencial actividad de agonistas o antagonista. Esta actividad puede evaluarse mediante ensayos farmacológicos o biológicos relacionados con el GPCR bajo estudio. Depende de su perfil de actividad, las moléculas de unión del receptor podrían entonces potencialmente evolucionar para convertirse en nuevos compuestos para la investigación en los estudios preclínicos y clínicos. Las moléculas que se unen específicamente a un receptor con alta afinidad también pueden servir como andamios para generar herramientas terapéuticas o de diagnósticos, por ejemplo por radiolabeling para no invasiva en vivo la proyección de imagen del tumor las células20, o como potencial vehículos para la entrega específica de therapeutics21. En el caso de CXCR4, en vivo la proyección de imagen de células del tumor ya ha sido demostrada utilizando modelos de ratón en donde moléculas CXCR4-targeting etiquetadas permitieron la visualización de cáncer humano xenoinjertos20,22,23 .
En este informe, describimos un protocolo detallado para un ensayo de competencia vinculante que permite la identificación de pequeñas moléculas y productos biológicos que interfieren directamente con agonista (CXCL12) vinculante a CXCR4. El principio básico del ensayo es la competencia entre una cantidad fija de fluorescencia etiquetada ligando (CXCL12AF647, véase tabla de materiales y reactivos) y sin etiqueta compuestos por la Unión a los receptores proteína17, 24. la señal fluorescente específica de etiquetado ligando a las células expresan CXCR4 es luego analizada por citometría de flujo. Esta señal fluorescente se reducirá cuando moléculas pequeñas interrumpen la interacción entre CXCL12AF647 y CXCR4. El ensayo utiliza células vivas no manipulado que endógeno expresan CXCR4 (es decir, células de Jurkat). Por lo tanto, ninguna preparación de la membrana de la célula es necesaria, lo que hace el ensayo conveniente, rápido y compatible con el mayor rendimiento. Puesto que un ligando marcado fluorescente se utiliza, se evita la radiactividad.
Porque CXCL12 es el agonista natural para CXCR4, compuestos de pequeñas moléculas que interfieren con la CXCL12AF647 vinculantes en el análisis son capaces de interactuar con el sitio de unión del receptor de la orthosteric (es decir, el sitio de Unión ocupado por los naturales agonista). Moléculas que interactúan con sitios de unión del receptor topográficamente distintos desde el sitio de unión de orthosteric permanecen no detectadas, si no influyen atascamiento de CXCL12. Por ejemplo, moduladores alostéricos positivos y negativos, una categoría importante y emergente de GPCR dirigidos a moléculas que actúan en sitios de Unión alostérica25, serán potencialmente no recogidos con este ensayo. Además, si los compuestos identificaron con esta función de ensayo de enlace como agonistas o antagonistas de los receptores no se puede derivar. Investigación de los compuestos identificados en análisis adicionales farmacológicos o funcionales relacionadas con el receptor por lo tanto será necesario. Estos ensayos pueden incluir (combinación de) celular fluorescencia o luminiscencia-ensayos para la detección de segundos mensajeros (p. ej., Ca2 +, monofosfato de adenosina cíclico (campo)), fenotípicas o ensayos biológicos y β-arrestin Análisis de la contratación, la elección de la que depende de las propiedades específicas de señalización de lo GPCR en estudio. Por lo tanto, el análisis de Unión competitiva descrito en este documento principalmente sirve como un ensayo de prueba inicial que debe ser complementado con otros ensayos celulares para permitir una caracterización detallada de los compuestos con la potencia de unión del receptor.
Protocolo
1. mantenimiento del cultivo celular
Nota: Todos los pasos descritos en 1 y 2 se llevan a cabo en condiciones estériles en un gabinete de flujo laminar.
- Cultivar células en T75 frascos de cultivo a 37 ° C y 5% de CO2 en una incubadora humidificada.
Nota: En este ensayo, se utilizan células de Jurkat (es decir, linfocitos de T leucémicas humanos células que endógeno expresan CXCR417). Expresión de CXCR4 en la superficie de la célula debe ser evaluado a través de cultivo celular mediante citometría de flujo. Una descripción del procedimiento de citometría de flujo y reactivos para determinar niveles de expresión del receptor en la superficie celular, sin embargo, no es en el ámbito de este protocolo, pero ha sido descrito previamente17. - Dejar que las células de Jurkat crecen en suspensión hasta 80-85% confluency. Antes pases las células a un nuevo frasco, deje todos los reactivos a temperatura ambiente (RT).
- Añadir 20 mL de medio completo fresco (Medio RPMI-1640, 10% suero fetal bovino (FBS), glutamina 2 mM) a un matraz de cultivo T75 novela.
- Añadir 5 mL de suspensión de células de Jurkat del original frasco T75 (contiene 25 mL de la suspensión celular) a la novela T75 frasco de cultura. Incubar a 37 ° C y 5% de CO2 en una incubadora humidificada.
2. preparación de células Jurkat, CXCL12 y tampón de ensayo para el concurso de ensayo de enlace.
- Preparar una solución stock de CXCL12AF647 (20 μg/mL; véase tabla de materiales y reactivos) por disolver el Reactivo liofilizado (almacenado a-80 ° C, en la oscuridad) en agua ultrapura con 0,01% (volumen/volumen) de polisorbato 20. Tienda de solo usar alícuotas de esta solución stock a-80 ° C, protegido de la luz.
- Tampón de ensayo se preparan mediante la adición de 40 mL HEPES (concentración final 1 M, 20 mM) para equilibrada solución de 200 mL Hank de sal (HBSS, 10 x, sin fenol rojo y sin bicarbonato de sodio, 1 x concentración final). Agregar agua ultrapura para obtener un volumen final de 2 L. Añadir 4 g (0.2% peso/volumen) albúmina de suero bovino (BSA) y disolver la BSA vía magnética revolviendo. Por último, ajustar el pH a 7.4 (usar NaOH para esto) y filtrar la solución a través de 0,2 poros μm (véase tabla de materiales y reactivos) con un colector de vacío.
Nota: Se utilizará este tampón de ensayo en todas las etapas del protocolo adicionales. - Cuenta el número y la viabilidad de las células. Para ello, tomar una muestra de la suspensión de células y diluido en tampón fosfato salino (PBS).
Nota: Habitualmente utilizamos un analizador de viabilidad celular automatizado (véase tabla de materiales y reactivos) capaz de suspensiones celulares a concentraciones variables de conteo. Para el recuento celular, diluya 0.5 mL de la suspensión celular en 1,5 mL de PBS (las otras diluciones, por ejemplo, 0,1 mL en 1,9 mL de PBS, también son posibles). El uso de este método, que se basa en el método de exclusión de colorante azul de tripano, ha sido descrito previamente26. Varios otros dispositivos están comercialmente disponibles para contar el numero de celular y viabilidad que debería funcionar igualmente bien. - Recoger el número de células (es decir, ~ 24 x 106 células para ejecutar el ensayo con una placa de 96 pocillos completa) en un tubo estéril de 50 mL por centrifugación (véase tabla de materiales y reactivos para el tipo de centrífuga) a 400 x g durante 5 min a RT.
- Suavemente retirar el sobrenadante sin perturbar el sedimento celulares. Añadir el tampón de ensayo fresca (p. ej., 20 mL) y resuspender las células transfiriendo suavemente hacia arriba y hacia abajo.
- Centrifugar las células a 400 x g durante 5 min a TA.
- Retirar nuevamente el sobrenadante y resuspender el precipitado de células en tampon de ensayo fresca para obtener una densidad de 5 x 106 células/mL.
3. competencia vinculante ensayo
Nota: El ensayo del Enlace real competencia se realiza a temperatura ambiente y puede ser realizado bajo condiciones no estériles.
- Diluir los compuestos investigados en tampon de ensayo (véase 2.2) para obtener la concentración deseada. O bien preparar una concentración fija de compuesto para el cribado inicial (por ejemplo, 10 concentración final μm) o, alternativamente, una serie de dilución seriada de las concentraciones para una más detallada caracterización de los compuestos (por ejemplo, 1/3, 1/4 o 1 / 5 serie de diluciones a partir de 1 μm, concentración final). Tenga en cuenta que la solución compuesta en última instancia será 2 x diluido en el ensayo; por lo tanto, preparar un 2 x solución concentrada.
- Añada 100 μl de solución de compuesto (2 x concentrado) en un claro 96-bien redonda placa inferior (véase tabla de materiales y reactivos) según una endecha experimental previamente definida hacia fuera (por ejemplo, figura 1).
Nota: En esta etapa, se incluyen muestras de control positivo y negativo en el ensayo. En la muestra control negativo, se añade 100 μl de tampón de ensayo en vez de compuesto en los pocillos de la placa de 96 pocillos. Para la muestra control positivo, tampón de ensayo también se agrega durante este paso. Ver también figura 1 para un típico diseño experimental en el que se prueba una serie de diluciones de varios compuestos. - Añadir 50 μl de suspensión celular (véase 2.7; 0.25 x 106 células) de un reservorio de reactivo en la placa de 96 pozos utilizando una pipeta multicanal. Incubar la placa por 15 min a temperatura ambiente en la oscuridad.
- Añadir 50 μl de fluorescencia etiquetada CXCL12 (es decir, 100 ng/mL de CXCL12AF647 en tampón de ensayo, 4 x concentrado, concentración final de 25 ng/mL) de un reservorio de reactivo similar a los pozos de la placa de 96 pocillos. Incubar por 30 min a temperatura ambiente en la oscuridad.
Nota: Para las muestras control negativo, añadir tampón de ensayo en lugar de otro. Por lo tanto, la señal fluorescente detectada en las muestras control negativo corresponde a la señal de fondo autofluorescent (figura 1A). Para las muestras de control positivo, añadir 50 μL/pocillo de CXCL12AF647. Las muestras de control positivo dará la señal de fluorescencia máxima detectada, puesto que ningún potencial inhibición por preincubación con compuestos se incluyó (figura 1A). - Centrifugar la placa de 96 pozos a 400 x g durante 5 minutos a RT. Retire el sobrenadante de las células sedimentados por los bancos sobre la placa. Secar la placa en un tejido.
- Añadir 200 μL de tampón de ensayo fresco de un reservorio de reactivo a los pozos utilizando una pipeta multicanal. Proceder inmediatamente.
- Centrifugar la placa nuevamente por 5 min a 400 x g en RT. eliminar el sobrenadante por los bancos sobre la placa y secarla otra vez en el tejido.
- Suavemente Resuspender el precipitado de células en 200 μL de paraformaldehído al 1% disuelta en PBS. Este paso será fijar las células.
- Seguir el protocolo inmediatamente con la cuantificación de la fluorescencia mediante citometría de flujo.
4. Análisis de las muestras por citometría de flujo
CXCL12AF647 manchadas y resecos células ya están listas para ser analizados mediante citometría de flujo. Pueden utilizarse varios tipos de citómetros de flujo, pero tienen que estar equipados con el láser correcto (es decir, un láser rojo, excitación rango ~ 630 nm) para la excitación y filtros adecuados para la detección del fluoróforo (emisión filtros ~ 660 nm). Tienen que ser capaces de manejar muestras en un formato de placa de 96 pocillos. Ejemplos de dispositivos de citometría de flujo adecuado se dan en la tabla de materiales y reactivos.
- Puesta en marcha del dispositivo y abrir el software correspondiente (véase tabla de materiales y reactivos).
- Seleccione los siguientes parámetros celulares a visualizarse en formato dot blot: adelante scatter (FSC), lado scatter (SSC) y el canal de detección de fluoróforo (CXCL12AF647).
Nota: Con el parámetro FSC, las células son discriminadas en base a su tamaño, puesto que la absorción de la luz detectada es proporcional al diámetro de la célula. El parámetro de la SSC, mide la dispersión de la luz en un ángulo de 90°, proporciona información sobre el nivel de detalle de las células. - Elige una muestra (por ejemplo, una muestra de control negativo) para realizar el control de una población celular homogénea definida basándose en los parámetros FSC y SSC.
- Seleccione inyección automática de ~ 100 μl de células determinadas de esta muestra control negativo en el citómetro de flujo. Seleccione la opción "mezcla" antes de la inyección y utilizar un caudal de muestra de 1,5 μl/s.
- Ejecutar este ejemplo seleccionando "Datos de adquisición". Los parámetros para esta muestra de FSC y SSC aparecerá en la pantalla.
- Seleccione la herramienta bloquea el software. Basado en la visualización de blot dot FSC y SSC, predefinir una población celular homogénea y viable por compuerta. Para ello, crear un polígono (utilizando herramienta bloquea el software) que incluye las células homogéneamente distribuidas ("eventos") basadas en estas dos dimensiones.
Nota: El procedimiento bloquea pretende definir una población celular homogénea y viable que se utilizará para su posterior análisis. Compuerta se basa en el supuesto de que la mayoría de las células viables forma una población celular homogénea, basándose en los parámetros FSC y SSC. Al realizar este paso, agregados de células, detritos celulares y células muertas en gran medida pueden ser excluidas del análisis. Una ilustración del proceso bloquea se da en la figura 1B.
- Seleccione analizar 20.000 'eventos' (es decir, las células) por muestra.
Nota: Esto significa que para cada muestra, 20.000 células que caen dentro de la puerta predefinida serán finalmente analizadas. Adquisición de datos para cada muestra continuará hasta que se analiza este número de eventos. - Iniciar el funcionamiento (seleccione "datos de registro"). El dispositivo de citometría de flujo ahora a analizar todas las muestras una por una mediante el registro de la intensidad de fluorescencia media (IFM) para cada muestra. Esta IMF se corresponde con la señal fluorescente media correspondiente a las 20.000 células que caen dentro de la puerta previamente definida.
5. Análisis de datos
Utilizar la IMF obtenida para cada muestra para realizar todos los cálculos más. Análisis de los datos de la citometría de flujo puede realizarse por diversos paquetes de software disponibles en el mercado (véase tabla de materiales y reactivos).
- Para determinar el porcentaje de inhibición de la señal fluorescente vinculante, como resultado de preincubación compuesto, aplicar la siguiente fórmula:
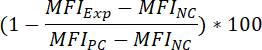
Donde:
MFIExp = IMF de la experimental (= Tratado compuesto) muestra
MFIPC = el MFI del control positivo
MFINC = IMF del control negativo - Para determinar el valor de IC50 de un compuesto (es decir, la concentración del compuesto que puede reducir la señal de enlace fluorescente en un 50%), aplicar la siguiente fórmula:
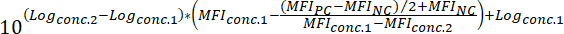
Donde:
Registroconc.2 = el log de la concentración del compuesto que resulta en menos del 50% de inhibición de la diferencia entre el valor de la IMF de la PC y NC
Registroconc.1 = el log de concentración de compuesto que resulta en más del 50% de inhibición de la diferencia entre el valor de la IMF de la PC y NC
Nota: Alternativamente, en caso de compuestos muy activos, una curva de dosis-respuesta que abarca varios registros de magnitud puede generarse en la IMF correspondiente a cada concentración probada de compuesto base. Mediante la aplicación de ajuste de curvas de regresión no lineal utilizando el software apropiado (véase tabla de materiales y reactivos), entonces se deduce valores de IC50 de las curvas generadas. En la figura 3se muestra un ejemplo de este enfoque de análisis.
Resultados
El flujo de trabajo general de la prueba de enlace se presenta en la figura 1A. Una ilustración del tipo de datos de citometría de flujo obtenidos para tipos diferentes de la muestra en un experimento estándar (es decir, el control negativo, control positivo y muestra experimental) se representa en la figura 1By un diseño de placa posible para llevar a cabo el ensayo en un formato de placa de 96 pocillos se da en la ...
Discusión
Comparado con otros tipos de ensayos de unión (es decir, enlace de saturación y experimentos de cinética obligatoria), ensayos de competencia vinculante son los más adecuados para fines de detección. De hecho, permiten evaluación de lotes grandes de compuestos sin etiqueta, por ejemplo pequeñas moléculas, marcando su capacidad para interferir con la Unión de una cantidad fija de un ligando del receptor etiquetados. Compuestos que se unen a otros sitios del receptor que el ligando marcado podrían perman...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores desean agradecer a Eric Fonteyn por asistencia técnica excelente. Este trabajo ha sido apoyado por el KU Leuven (subsidio no. PF/10/018), Fonds voor Wetenschappelijk Onderzoek (FWO, subsidio no. G.485.08) y la Fondation Dormeur Vaduz.
Materiales
| Name | Company | Catalog Number | Comments |
| BD FACSCanto II | Becton Dickinson | Not applicable | Flow cytometry device |
| BD FACSDIVA Software | |||
| BD FACSArray | Becton Dickinson | Not applicable | Flow cytometry device |
| BD FACSArray System Software | |||
| Graphpad Prism | Graphpad | software package used for nonlinear regression analysis in Figure 2 and Figure 3 | |
| FlowJo | FlowJo is now a wholly owned subsidiary of BD. | ||
| Vi-CELL | Beckman Coulter | Not applicable | cell viability analyzer |
| Sigma 3-18 KS | Sigma | Not applicable | centrifuge |
| AMD3100 | Sigma | A5602-5mg | specific CXCR4 antagonist |
| Maraviroc | Pfizer | antiretroviral drug, CCR5 antagonist, available for research at Selleckchem (cat#S2003), Sigma (cat#PZ0002) | |
| h-SDF1a (AF647) | ALMAC | CAF-11-B-01 | fluorescently labeled CXCL12, CXCL12AF647 |
| Fetal Bovine Serum (FBS) | Gibco (Life Technologies) | 10270-106 | |
| Bovine Serum Albumin (BSA) | Sigma | A1933-25G | |
| HBSS (10x), calcium, magnesium, no phenol red | Gibco (Life Technologies) | 14065-049 | |
| HEPES (1M) | Gibco (Life Technologies) | 15630-056 | |
| Dulbecco's Phosphate Buffered Saline (DPBS) | Gibco (Life Technologies) | 14190-094 | |
| Jurkat cells | ATCC | ||
| Reagent reservoir PP | Sigma | BR703411 | |
| Rapid flow filter: 0.2 µm aPES | Thermo Scientific | 566-0020 | |
| Sterilin microtiter plate, 96-well, U bottom, clear | Thermo Scientific | 611U96 | |
| Falcon tubes, 50ml | Greiner Bio-One | 227 261 | |
| Tissue culture flask (T75) | Corning | 353024 |
Referencias
- Fredriksson, R., Lagerstrom, M. C., Lundin, L. G., Schioth, H. B. The G-protein-coupled receptors in the human genome form five main families. Phylogenetic analysis, paralogon groups, and fingerprints. Mol Pharmacol. 63, 1256-1272 (2003).
- Milligan, G., Kostenis, E. Heterotrimeric G-proteins: A short history. Br J Pharmacol. 147 Suppl 1, S46-S55 (2006).
- Oldham, W. M., Hamm, H. E. Heterotrimeric G protein activation by G-protein-coupled receptors. Nat Rev Mol Cell Biol. 9, 60-71 (2008).
- Tuteja, N. Signaling through G protein coupled receptors. Plant Signal Behav. 4, 942-947 (2009).
- Neves, S. R., Ram, P. T., Iyengar, R. G protein pathways. Science. 296, 1636-1639 (2002).
- Gurevich, E. V., Tesmer, J. J., Mushegian, A., Gurevich, V. V. G protein-coupled receptor kinases: more than just kinases and not only for GPCRs. Pharmacol Ther. 133, 40-69 (2012).
- Smith, J. S., Rajagopal, S. The beta-arrestins: Multifunctional regulators of G protein-coupled receptors. J Biol Chem. 291, 8969-8977 (2016).
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Hopkins, A. L., Groom, C. R. The druggable genome. Nat Rev Drug Discov. 1, 727-730 (2002).
- Lappano, R., Maggiolini, M. G protein-coupled receptors: Novel targets for drug discovery in cancer. Nat Rev Drug Discov. 10, 47-60 (2011).
- Chatterjee, S., Behnam Azad, ., Nimmagadda, B., S, The intricate role of CXCR4 in cancer. Adv Cancer Res. 124, 31-82 (2014).
- Bleul, C. C., Farzan, M., Choe, H., Parolin, C., Clark-Lewis, I., Sodroski, J., Springer, T. A. The lymphocyte chemoattractant SDF-1 is a ligand for LESTR/fusin and blocks HIV-1 entry. Nature. 382, 829-833 (1996).
- Flomenberg, N., DiPersio, J., Calandra, G. Role of CXCR4 chemokine receptor blockade using AMD3100 for mobilization of autologous hematopoietic progenitor cells. Acta Haematol. 114, 198-205 (2005).
- Domanska, U. M., Kruizinga, R. C., Nagengast, W. B., Timmer-Bosscha, H., Huls, G., de Vries, E. G., Walenkamp, A. M. A review on CXCR4/CXCL12 axis in oncology: No place to hide. Eur J Cancer. 49, 219-230 (2013).
- Tsou, L. K., Huang, Y. H., Song, J. S., Ke, Y. Y., Huang, J. K., Shia, K. S. Harnessing CXCR4 antagonists in stem cell mobilization, HIV infection, ischemic diseases, and oncology. Med Res Rev. , (2017).
- Schols, D., Struyf, S., Van Damme, J., Este, J. A., Henson, G., De Clercq, E. Inhibition of T-tropic HIV strains by selective antagonization of the chemokine receptor CXCR4. J Exp Med. 186, 1383-1388 (1997).
- Van Hout, A., D'Huys, T., Oeyen, M., Schols, D., Van Loy, T. Comparison of cell-based assays for the identification and evaluation of competitive CXCR4 inhibitors. PLoS One. 12, e0176057 (2017).
- De Clercq, E. The AMD3100 story: the path to the discovery of a stem cell mobilizer (Mozobil). Biochem Pharmacol. 77, 1655-1664 (2009).
- Debnath, B., Xu, S., Grande, F., Garofalo, A., Neamati, N. Small molecule inhibitors of CXCR4. Theranostics. 3, 47-75 (2013).
- Woodard, L. E., Nimmagadda, S. CXCR4-based imaging agents. J Nucl Med. 52, 1665-1669 (2011).
- Wang, Y., Xie, Y., Oupicky, D. Potential of CXCR4/CXCL12 Chemokine Axis in Cancer Drug Delivery. Curr Pharmacol Rep. 2, 1-10 (2016).
- Nimmagadda, S., Pullambhatla, M., Stone, K., Green, G., Bhujwalla, Z. M., Pomper, M. G. Molecular imaging of CXCR4 receptor expression in human cancer xenografts with [64Cu]AMD3100 positron emission tomography. Cancer Res. 70, 3935-3944 (2010).
- De Silva, R. A., Peyre, K., Pullambhatla, M., Fox, J. J., Pomper, M. G., Nimmagadda, S. Imaging CXCR4 expression in human cancer xenografts: evaluation of monocyclam 64Cu-AMD3465. J Nucl Med. 52, 986-993 (2011).
- Hatse, S., Princen, K., Liekens, S., Vermeire, K., De Clercq, E., Schols, D. Fluorescent CXCL12AF647 as a novel probe for nonradioactive CXCL12/CXCR4 cellular interaction studies. Cytometry A. 61, 178-188 (2004).
- Wootten, D., Christopoulos, A., Sexton, P. M. Emerging paradigms in GPCR allostery: implications for drug discovery. Nat Rev Drug Discov. 12, 630-644 (2013).
- Louis, K. S., Siegel, A. C. Cell viability analysis using trypan blue: manual and automated methods. Methods Mol Biol. 740, 7-12 (2011).
- Perry, C. M. Maraviroc: a review of its use in the management of CCR5-tropic HIV-1 infection. Drugs. 70, 1189-1213 (2010).
- Moyle, G., DeJesus, E., Boffito, M., Wong, R. S., Gibney, C., Badel, K., MacFarland, R., Calandra, G., Bridger, G., Becker, S. Proof of activity with AMD11070, an orally bioavailable inhibitor of CXCR4-tropic HIV type 1. Clin Infect Dis. 48, 798-805 (2009).
- Balabanian, K., Lagane, B., Infantino, S., Chow, K. Y., Harriague, J., Moepps, B., Arenzana-Seisdedos, F., Thelen, M., Bachelerie, F. The chemokine SDF-1/CXCL12 binds to and signals through the orphan receptor RDC1 in T lymphocytes. J Biol Chem. 280, 35760-35766 (2005).
- Burns, J. M., Summers, B. C., Wang, Y., Melikian, A., Berahovich, R., Miao, Z., Penfold, M. E., Sunshine, M. J., Littman, D. R., Kuo, C. J., Wei, K., McMaster, B. E., Wright, K., Howard, M. C., Schall, T. J. A novel chemokine receptor for SDF-1 and I-TAC involved in cell survival, cell adhesion, and tumor development. J Exp Med. 203, 2201-2213 (2006).
- Stoddart, L. A., Kilpatrick, L. E., Briddon, S. J., Hill, S. J. Probing the pharmacology of G protein-coupled receptors with fluorescent ligands. Neuropharmacology. 98, 48-57 (2015).
- Vernall, A. J., Hill, S. J., Kellam, B. The evolving small-molecule fluorescent-conjugate toolbox for Class A GPCRs. Br J Pharmacol. 171, 1073-1084 (2014).
- Stoddart, L. A., White, C. W., Nguyen, K., Hill, S. J., Pfleger, K. D. Fluorescence- and bioluminescence-based approaches to study GPCR ligand binding. Br J Pharmacol. 173, 3028-3037 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados