É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um ensaio de baseados em citometria de fluxo para identificar compostos que interrompem a ligação do ligante de Chemokine fluorescente-etiquetadas CXC 12 a CXC Chemokine Receptor 4
Neste Artigo
Resumo
Um fluxo de ensaio de ligação celular baseados em citometria é descrito que é usado principalmente como uma ferramenta de triagem para identificar compostos que inibem a ligação de um ligante de chemokine CXC fluorescente etiquetada 12 (CXCL12) para o receptor do chemokine CXC 4 (CXCR4).
Resumo
Como alvo farmacológico dos receptores de acoplado à proteína G (GPCRs) é de grande importância para a saúde humana, como disfuncional GPCR mediada por sinalização contribui para a progressão de muitas doenças. O par ligante/receptor ligante de chemokine CXC 12 (CXCL12) / receptor do chemokine do CXC 4 (CXCR4) tem gerado interesse clínico significativo, por exemplo, como um alvo em potencial para o tratamento de câncer e doenças inflamatórias. Pequenas moléculas, bem como anticorpos terapêuticos que especificamente alvo CXCR4 e inibem a função do receptor, portanto, são considerados valiosas ferramentas farmacológicas. Aqui, um fluxo baseados em citometria celular ensaio que permite a identificação de compostos (por exemplo, pequenas moléculas) que revogará CXCL12 vinculação de CXCR4, é descrita. Essencialmente, o ensaio se baseia na competição para receptor vinculação entre uma quantidade fixa de CXCL12 fluorescente etiquetadas, o agonista chemokine natural para CXCR4 e compostos sem rótulo. Portanto, é evitar a utilização indesejável de sondas radioactivamente marcadas neste ensaio. Além disso, as células vivas são usadas como a fonte do receptor (CXCR4) ao invés de preparações de membrana celular. Isto permite uma fácil adaptação do ensaio para um formato de chapa, o que aumenta a taxa de transferência. Este ensaio tem demonstrado ser um ensaio de descoberta valiosa medicamento genérico para identificar compostos CXCR4-direcionamento. O protocolo pode provavelmente ser adaptado para outros GPCRs, pelo menos se fluorescente etiquetadas ligantes estão disponíveis ou podem ser gerados. Conhecimento prévio sobre as vias de sinalização intracelulares que são induzidas após a ativação destas GPCRs, não é necessário.
Introdução
G receptores (GPCRs) são proteínas de superfície celular que podem ser ativadas por ligantes extracelulares (por exemplo, peptídeos, hormônios proteicos, aminas), desse modo regular muitos fisiológicos e do desenvolvimento de processos1. Quando um agonista ocupa seu bolso de vinculação GPCR, a mudança conformacional induzida da proteína do receptor promove a ligação de proteínas intracelulares associadas do receptor via G, consistindo de Gα- PIB e Gβγ subunidades. A subsequente troca de GTP para PIB sobre os resultados de subunidadeα G na dissociação das subunidades de proteína G (Gα-GTP e Gβγ) que, por sua vez, ainda mais iniciará a jusante sinalizando caminhos2,3. Quando o Gα-GTP se torna hidrolisado, re-associação da Gα- PIB e Gβγ subunidades irão converter a proteína G volta em seu descanso de3,de estado4. Tipos distintos de proteínas G existem (Gs, G,i/o, Gq, G12/13), que são classificados com base na similaridade de sequência com o de subunidadeα G5. Todas estas proteínas G induzem definidas vias de sinalização intracelulares que fundamentam a resposta biológica à ativação do receptor. Na sequência de ativação do receptor, quinases GPCR (GRKs) fosforilar a cauda intracelular de GPCRs, promovendo a interação com β-arrestins. Este processo leva à rescisão da proteína G, sinalização,6, de dessensibilização e internalização do receptor. Β-arrestins também são parte dos complexos multi moleculares que gatilho sinalizando cascatas independentes da proteína G sinalização7.
GPCRs estão entre os alvos moleculares mais validados para a intervenção terapêutica, como desregulamentado GPCR mediada por sinalização, por exemplo devido a mutações de ganho de função no gene do receptor ou superexpressão do receptor, contribui para a etiologia de muitas doenças humanas8. Portanto, GPCRs representam uma das mais importantes classes de alvos de drogas investigados pela indústria farmacêutica8,9,10. Um exemplo notável de um GPCR clinicamente relevante é a CXC receptor do chemokine 4 (CXCR4), que pode ser ativado por um ligante natural único, o CXC chemokine ligante 12 (CXCL12)11. Devido ao seu papel estabelecido como um grande receptor de co para vírus de imunodeficiência humana 1 (HIV-1) entrada e infecção em cluster de diferenciação 4 (CD4) positivo linfócitos T12, CXCR4 primeiro foi investigada como alvo uma droga antiviral. CXCL12-CXCR4 interação na medula óssea mais regula a retenção e orientação de tronco e progenitor células13. Também, dado o seu envolvimento em muitos aspectos do câncer biologia (por exemplo, sobrevivência de células de tumor, metástase, relacionados ao tumor angiogênese)14 e várias outras doenças humanas (por exemplo, doenças inflamatórias)15, CXCR4 criou um interesse significativo como um alvo promissor para a descoberta de medicamentos. AMD3100, uma pequena molécula que se destina especificamente a CXCR4, descobriu-se, inicialmente, como uma droga de anti-HIV candidato16 e ainda é um dos mais potentes antagonistas CXCR4 descritos a data17. Seu desenvolvimento como uma droga antiviral foi, no entanto, descontinuado18. Atualmente, esta molécula é usada como um agente de mobilização de células-tronco durante o tratamento de pacientes de mieloma múltiplo e linfoma18. Várias outras moléculas pequenas quimicamente independentes e produtos biológicos que inibem a função de CXCR4 com potência variando foram descritos19.
Métodos de ligação do receptor são ferramentas valiosas em farmacologia que permitem a identificação de compostos (por exemplo, pequenas moléculas) que interagem diretamente com o GPCR de interesse. Para realizar estudos de ligação, não há nenhuma necessidade de conhecimento prévio sobre a funcionalidade de um determinado GPCR ou a propriedades de sinalização intracelulares. Embora isto pode ser considerado uma vantagem, sugere que compostos que receptor vinculação pode ser demonstrada precisam ainda mais ser caracterizada através da avaliação da sua potencial atividade agonística ou antagônica. Esta atividade pode ser avaliada usando ensaios farmacológicos, ou biológicos relacionados com o GPCR sob estudo. Dependente de seu perfil de atividade, moléculas de ligação do receptor podem então potencialmente evoluir para tornar-se os compostos de chumbo romance para investigação em estudos pré-clínicos e clínicos. As moléculas que se ligam especificamente a um receptor com alta afinidade também podem servir como andaimes para gerar ferramentas de diagnósticos ou terapêuticas, por exemplo, os radioativos para a imagem latente não-invasiva na vivo de células de tumor20, ou como potencial veículos para entrega alvo da terapêutica21. Em caso de CXCR4, imagem latente na vivo de células tumorais já foi demonstrado usando modelos de rato onde moléculas CXCR4-direcionamento rotuladas permitiu a visualização de câncer humano xenografts20,22,23 .
Neste relatório, descrevemos um protocolo detalhado para um ensaio de vinculação de competição que permite a identificação de moléculas pequenas e produtos biológicos que interferem diretamente com agonista (CXCL12) vinculativa de CXCR4. O princípio básico do ensaio é a concorrência entre uma quantidade fixa de ligante fluorescente etiquetada (CXCL12AF647, consulte tabela de materiais e reagentes) e unlabeled compostos para a ligação com o receptor da proteína17, 24. o sinal fluorescente específico do ligante rotulado vinculado a única células expressando CXCR4 é então analisado por citometria de fluxo. Este sinal fluorescente diminui quando as moléculas pequenas sem rótulo perturbam a interação entre CXCL12AF647 e CXCR4. O ensaio utiliza células de vida não-manipulada que expressam endogenamente CXCR4 (i.e., células Jurkat). Portanto, nenhuma preparação da membrana celular é necessária, o que torna o ensaio conveniente, rápido e compatível com maior throughput. Desde que é usado um ligante fluorescente etiquetado, radioatividade é evitada.
Porque CXCL12 é o agonista natural para CXCR4, compostos de pequenas moléculas que interferem com a vinculação de CXCL12AF647 no ensaio são susceptíveis de interagir com o sítio de ligação orthosteric do receptor (ou seja, o sítio de ligação ocupado pelo natural agonista). Moléculas que iria interagir com locais de ligação do receptor topograficamente distintas do local de ligação do orthosteric despercebido, se elas não influenciam a vinculação de CXCL12. Por exemplo, moduladores alostéricos positivos e negativos, uma categoria importante e emergente de GPCR como alvo moléculas atuando em sítios de ligação alostéricos25, serão potencialmente não apanhados com este ensaio. Além disso, se os compostos identificaram com esta função de ensaio de ligação como antagonistas dos receptores ou como agonistas não podem ser derivado. Investigação dos compostos identificados em adicionais ensaios relacionados ao receptor farmacológicos ou funcionais, portanto, serão necessária. Estes ensaios podem incluir (uma combinação de) celular fluorescência ou luminescência-com base em ensaios para a detecção de segundo mensageiros (por exemplo, Ca2 +, adenosina monofosfato cíclica (cAMP)), fenotípicas ou ensaios biológicos e β-arrestin ensaios de recrutamento, a escolha dos quais depende as propriedades específicas de sinalização do GPCR sob estudo. Portanto, o ensaio de ligação competitiva descrito neste documento principalmente serve como um ensaio de triagem inicial que precisa de ser complementada com outros ensaios cell-based para permitir uma caracterização aprofundada de compostos com potência de ligação do receptor.
Protocolo
1. a manutenção da cultura de pilha
Nota: Todos os passos descritos em 1 e 2 são realizados em condições estéreis em um fluxo laminar.
- Desenvolvem-se células em frascos de cultura T75 em 37 ° C e 5% CO2 em uma incubadora umidificada.
Nota: Neste ensaio, são utilizadas células Jurkat (isto é, células de humanas leucêmicas dos linfócitos T que expressam endogenamente CXCR417). Expressão do CXCR4 na superfície da célula deve ser avaliado ao longo do cultivo de células através da citometria de fluxo. Uma descrição do procedimento de citometria de fluxo e reagentes para determinar os níveis de expressão de receptores na superfície celular, no entanto, não é no âmbito do presente protocolo, mas tem sido descrito anteriormente a17. - Deixe as células Jurkat crescer em suspensão até atingirem 80-85% Confluencia. Antes de passagem as células para um balão de romance, permitir que todos os reagentes atingirem a temperatura (RT).
- Adicione 20 mL de meio fresco crescimento completo (meio RPMI-1640, 10% soro bovino fetal (FBS), glutamina 2mm) para um balão de cultura T75 romance.
- Adicione 5 mL de suspensão de células Jurkat do recipiente original T75 (contendo 25 mL de suspensão de células) para a novela T75 frasco de cultura. Incube a 37 ° C e 5% CO2 em uma incubadora umidificada.
2. preparação de células Jurkat, Buffer de ensaio e CXCL12 para a competição de vinculação do ensaio.
- Prepare uma solução stock de CXCL12AF647 (20 µ g/mL; ver tabela de materiais e reagentes) dissolvendo-se o reagente liofilizado (armazenado a-80 ° C, no escuro) em água ultrapura suplementado com 0,01% (volumétrica) de polissorbato 20. Loja única usar alíquotas desta solução a-80 ° C, protegido da luz.
- Prepare-se buffer de ensaio, adicionando 40 mL HEPES (concentração final 1 M, 20 mM) para equilibrada solução 200 mL do Hank salina (HBSS, 10 x, sem vermelho de fenol e sem bicarbonato de sódio, 1 x concentração final). Adicione água ultrapura para obter um volume final de 2 L. adicionar 4 g (0,2% peso/volume) albumina de soro bovino (BSA) e dissolver a BSA através do magnético mexendo. Finalmente, ajustar o pH para 7,4 (usar NaOH para isso) e filtrar a solução através de 0,2 µm poros (ver tabela de materiais e reagentes) usando um distribuidor de vácuo.
Nota: Esse buffer de ensaio será usado em todas as etapas adicionais do protocolo. - Conte o número e a viabilidade das células. Para isso, tirar uma amostra da suspensão celular e diluí-la em salina tamponada fosfato (PBS).
Nota: Usamos rotineiramente com um analisador de viabilidade celular automatizada (ver tabela de materiais e reagentes) capaz de contagem de suspensões celulares em diferentes concentrações. Para a contagem de células, dilua 0,5 mL de suspensão de células em 1,5 mL de PBS (outras diluições, por exemplo, 0,1 mL em 1,9 mL de PBS, também são possíveis). O uso desse método, que se baseia o método de exclusão do corante azul de Tripan, tem sido descrito anteriormente26. Vários outros dispositivos são comercialmente disponíveis para a contagem celular e viabilidade que deve funcionar igualmente bem. - Recolher o número desejado de células (ou seja, ~ 24 x 106 células para executar o ensaio com uma placa de 96 poços completa) em um tubo estéril 50 mL por centrifugação (veja tabela de materiais e reagentes para o tipo de centrífuga usada) a 400 x g durante 5 min à RT.
- Despeje delicadamente o sobrenadante sem perturbar o centrifugado. Adicionar o buffer de ensaio fresco (por exemplo, 20 mL) e ressuspender as células pipetando delicadamente acima e para baixo.
- Centrifugar as células novamente a 400 x g, durante 5 min à RT
- Despeje novamente o sobrenadante e ressuspender as células em buffer de ensaio fresco para obter uma densidade de 5 x 106 células/mL.
3. concorrência ensaio
Nota: O ensaio de ligação real concorrência é realizado a RT e pode ser realizado em condições não estéreis.
- Diluir os compostos sob investigação no buffer de ensaio (ver 2.2) para obter a concentração desejada. Quer preparar uma concentração fixa de composto para triagem inicial (por exemplo, 10 µM concentração final) ou, alternativamente, uma série de série de diluição das concentrações para mais detalhadas caracterização dos compostos (por exemplo, um 1/3, 1/4 ou 1 / 5 séries de diluição começando com 1 µM, concentração final). Tenha em mente que o composto solução irá tornar-se, finalmente, 2 x diluído no ensaio; Portanto, prepare uma solução concentrada de 2x.
- Dispensar 100 µ l de solução de composto (2x concentrado) num claro 96-poço redondo placa inferior (veja tabela de materiais e reagentes) de acordo com um leigo experimental pre-definido para fora (por exemplo, Figura 1).
Nota: Nesta fase, as amostras de controle negativo e positivo são incluídas no ensaio. Na amostra de controlo negativo, 100 µ l de buffer de ensaio é adicionado ao invés de composto aos poços da placa de 96 poços. Para a amostra de controlo positivo, buffer de ensaio também é adicionado durante esta etapa. Veja também a Figura 1 para um layout experimental típico em que uma série de diluições de diversos compostos é testada. - Adicionar 50 µ l de suspensão de células (ver 2.7; 0,25 x 106 células) de um reservatório de reagente na placa de 96 poços, com uma pipeta multicanal. Incube a placa por 15 min em RT no escuro.
- Adicionar 50 µ l de CXCL12 fluorescente etiquetadas (ou seja, 100 ng/mL de CXCL12AF647 no buffer de ensaio, 4 x concentrado, concentração final de 25 ng/mL) de um reservatório de reagente semelhantes aos poços da placa de 96 poços. Incube durante 30 min à RT no escuro.
Nota: Para as amostras de controlo negativo, adicione o buffer de ensaio em vez disso. Portanto, o sinal fluorescente detectado nas amostras de controlo negativo irá corresponder ao sinal de fundo de autofluorescent (figura 1A). Para as amostras de controlo positivo, adicione 50 µ l/poço de CXCL12AF647. As amostras de controlo positivo irão produzir o sinal de fluorescência máxima detectado, uma vez que nenhum potencial inibição por pré-incubação com compostos foi incluída (figura 1A). - Centrifugue a placa de 96 poços a 400 x g durante 5 min à RT. Remove o sobrenadante das células peletizadas lançando-se sobre a placa. Seca a placa em um tecido.
- Adicione 200 µ l de buffer de ensaio fresco de um reservatório de reagente nos poços utilizando uma pipeta multicanal. Proceda imediatamente.
- Centrifugue a placa novamente por 5 min a 400 x g em RT. Remove o sobrenadante por capotar a placa e secá-lo novamente no tecido.
- Delicadamente resuspenda o pellet de células em 200 µ l de 1% paraformaldeído dissolvido em PBS. Esta etapa irá corrigir as células.
- Continue o protocolo imediatamente com a quantificação da fluorescência por citometria de fluxo.
4. análise das amostras por citometria de fluxo
CXCL12AF647 manchado e células fixadas agora estão prontas para ser analisado usando citometria de fluxo. Vários tipos de citômetros podem ser usados, mas eles precisam ser equipados com o laser correto (ou seja, um laser vermelho, excitação gama ~ 630 nm) para excitação e filtros adequados para a deteção de fluoróforo (emissão filtros ~ 660 nm). Eles precisam ser capazes de lidar com amostras em um formato de placa de 96 poços. Exemplos de dispositivos de citometria de fluxo adequado são dadas na tabela de materiais e reagentes.
- Iniciar o dispositivo e abrir o software correspondente (ver tabela de materiais e reagentes).
- Selecione os seguintes parâmetros celulares sejam visualizadas em um formato de borrão ponto: encaminhar scatter (FSC), dispersão lateral (SSC) e o canal de deteção de fluoróforo (CXCL12AF647).
Nota: Com o parâmetro FSC, as células são discriminadas com base no seu tamanho, uma vez que a absorção da luz detectada é proporcional ao diâmetro da célula. O parâmetro SSC, medir o espalhamento de luz em um ângulo de 90°, fornece informações sobre a granularidade das células. - Escolha uma amostra (por exemplo, uma amostra de controlo negativo) para realizar a retenção de uma população celular homogênea definidos com base nos parâmetros de FSC e SSC.
- Selecione injeção automática de ~ 100 µ l de células fixadas nesta amostra de controlo negativo para o citômetro de fluxo. Selecione a opção "mistura" antes da injeção e usar uma taxa de fluxo da amostra de 1,5 µ l/s.
- Executar este exemplo, selecionando "Adquirir dados." O FSC e SSC parâmetros para esta amostra aparecerá na tela.
- Selecione a ferramenta associada do software. Baseia-se a visualização de borrão ponto FSC e SSC, pré-defina uma população homogénea e viável célula gating. Para fazer isso, criar um polígono (usando a ferramenta associada do software) que inclui as células única homogênea distribuídas ("eventos") baseia-se estas duas dimensões.
Nota: O procedimento associado visa definir uma população de células viáveis e homogênea que será usada para uma análise mais aprofundada. Gating baseia-se no pressuposto de que a maioria das células viáveis irá formar uma população homogênea de células com base em parâmetros FSC e SSC. Realizando este passo, restos celulares, células mortas e agregados de células podem amplamente ser excluídos uma análise mais aprofundada. Uma ilustração do processo associada é dada na figura 1B.
- Selecione para analisar 20.000 "eventos" (isto é, células únicas) por exemplo.
Nota: Isto significa que para cada amostra, 20.000 células que caem dentro do portão pré-definidos serão eventualmente analisadas. Aquisição de dados para cada amostra irá continuar até que este número de eventos é analisado. - Começa a correr (selecione "gravar dados"). O dispositivo de citometria de fluxo agora vai analisar todas as amostras, um por um, gravando-a intensidade de fluorescência média (IFM) para cada amostra. Esta IFM corresponde ao sinal fluorescente médio correspondente as 20.000 células que caem dentro do portão pré-definido.
5. análise de dados
Use o IFM obtido para cada amostra para realizar todos os cálculos mais. Análise dos dados de citometria de fluxo pode ser realizada por vários pacotes de software disponíveis no mercado (ver tabela de materiais e reagentes).
- Para determinar a porcentagem de inibição do sinal fluorescente de ligação, como resultado de pré-incubação composto, aplica a seguinte fórmula:
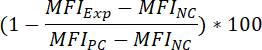
Onde:
IFMExp = IFM de experimental (= composto-tratados) amostra
IFMPC = IFM do controlo positivo
IFMNC = IFM do controlo negativo - Para determinar o valor de IC50 de um composto (ou seja, a concentração de compostos que podem reduzir o sinal fluorescente vinculação de 50%), aplica a seguinte fórmula:
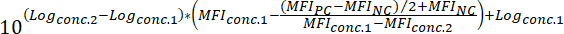
Onde:
Logconc.2 = o registo de uma concentração de composto que resulta em menos de 50% inibição da diferença entre o valor de IFM da NC e PC
Logconc.1 = o registo de uma concentração de composto que resulta em mais de 50% inibição da diferença entre o valor de IFM da NC e PC
Nota: Como alternativa, no caso de compostos altamente ativos, uma curva de dose-resposta, cobrindo vários registros de magnitude pode ser gerada baseia a IMF correspondentes a cada concentração testada de composto. Aplicando o encaixe de curva de regressão não-linear usando o software apropriado (ver tabela de materiais e reagentes), valores de IC50 então podem ser deduzidas a partir das curvas geradas. Um exemplo desta abordagem de análise é mostrado na Figura 3.
Resultados
O fluxo de trabalho geral do ensaio vinculação é apresentado na figura 1A. Uma ilustração do tipo de dados de citometria de fluxo obtidos para tipos diferentes de amostra em um experimento de padrão (ou seja, o controlo negativo, controle positivo e amostra experimental) está representada na figura 1Be um layout de placa possível para executar o ensaio em um formato de placa de 96 poços é dada na
Discussão
Ensaios obrigatórios de concorrência em comparação a outros tipos de ensaios obrigatórios (ou seja, a vinculação de saturação e experimentos de cinética de ligação), são mais adequados para fins de triagem. Na verdade, eles permitem avaliação de grandes lotes de compostos sem rótulo, por exemplo, pequenas moléculas, marcando sua capacidade de interferir com a ligação de uma quantidade fixa de um ligante-receptor rotulada. Compostos que se ligam a outros locais do receptor do que o ligante rotu...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Os autores gostaria de agradecer Eric Fonteyn excelente assistência técnica. Este trabalho foi apoiado pelo KU Leuven (conceder n. PF/10/018), Fonds voor Wetenschappelijk Onderzoek (FWO, conceder n. G.485.08) e a Fondation Dormeur Vaduz.
Materiais
| Name | Company | Catalog Number | Comments |
| BD FACSCanto II | Becton Dickinson | Not applicable | Flow cytometry device |
| BD FACSDIVA Software | |||
| BD FACSArray | Becton Dickinson | Not applicable | Flow cytometry device |
| BD FACSArray System Software | |||
| Graphpad Prism | Graphpad | software package used for nonlinear regression analysis in Figure 2 and Figure 3 | |
| FlowJo | FlowJo is now a wholly owned subsidiary of BD. | ||
| Vi-CELL | Beckman Coulter | Not applicable | cell viability analyzer |
| Sigma 3-18 KS | Sigma | Not applicable | centrifuge |
| AMD3100 | Sigma | A5602-5mg | specific CXCR4 antagonist |
| Maraviroc | Pfizer | antiretroviral drug, CCR5 antagonist, available for research at Selleckchem (cat#S2003), Sigma (cat#PZ0002) | |
| h-SDF1a (AF647) | ALMAC | CAF-11-B-01 | fluorescently labeled CXCL12, CXCL12AF647 |
| Fetal Bovine Serum (FBS) | Gibco (Life Technologies) | 10270-106 | |
| Bovine Serum Albumin (BSA) | Sigma | A1933-25G | |
| HBSS (10x), calcium, magnesium, no phenol red | Gibco (Life Technologies) | 14065-049 | |
| HEPES (1M) | Gibco (Life Technologies) | 15630-056 | |
| Dulbecco's Phosphate Buffered Saline (DPBS) | Gibco (Life Technologies) | 14190-094 | |
| Jurkat cells | ATCC | ||
| Reagent reservoir PP | Sigma | BR703411 | |
| Rapid flow filter: 0.2 µm aPES | Thermo Scientific | 566-0020 | |
| Sterilin microtiter plate, 96-well, U bottom, clear | Thermo Scientific | 611U96 | |
| Falcon tubes, 50ml | Greiner Bio-One | 227 261 | |
| Tissue culture flask (T75) | Corning | 353024 |
Referências
- Fredriksson, R., Lagerstrom, M. C., Lundin, L. G., Schioth, H. B. The G-protein-coupled receptors in the human genome form five main families. Phylogenetic analysis, paralogon groups, and fingerprints. Mol Pharmacol. 63, 1256-1272 (2003).
- Milligan, G., Kostenis, E. Heterotrimeric G-proteins: A short history. Br J Pharmacol. 147 Suppl 1, S46-S55 (2006).
- Oldham, W. M., Hamm, H. E. Heterotrimeric G protein activation by G-protein-coupled receptors. Nat Rev Mol Cell Biol. 9, 60-71 (2008).
- Tuteja, N. Signaling through G protein coupled receptors. Plant Signal Behav. 4, 942-947 (2009).
- Neves, S. R., Ram, P. T., Iyengar, R. G protein pathways. Science. 296, 1636-1639 (2002).
- Gurevich, E. V., Tesmer, J. J., Mushegian, A., Gurevich, V. V. G protein-coupled receptor kinases: more than just kinases and not only for GPCRs. Pharmacol Ther. 133, 40-69 (2012).
- Smith, J. S., Rajagopal, S. The beta-arrestins: Multifunctional regulators of G protein-coupled receptors. J Biol Chem. 291, 8969-8977 (2016).
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Hopkins, A. L., Groom, C. R. The druggable genome. Nat Rev Drug Discov. 1, 727-730 (2002).
- Lappano, R., Maggiolini, M. G protein-coupled receptors: Novel targets for drug discovery in cancer. Nat Rev Drug Discov. 10, 47-60 (2011).
- Chatterjee, S., Behnam Azad, ., Nimmagadda, B., S, The intricate role of CXCR4 in cancer. Adv Cancer Res. 124, 31-82 (2014).
- Bleul, C. C., Farzan, M., Choe, H., Parolin, C., Clark-Lewis, I., Sodroski, J., Springer, T. A. The lymphocyte chemoattractant SDF-1 is a ligand for LESTR/fusin and blocks HIV-1 entry. Nature. 382, 829-833 (1996).
- Flomenberg, N., DiPersio, J., Calandra, G. Role of CXCR4 chemokine receptor blockade using AMD3100 for mobilization of autologous hematopoietic progenitor cells. Acta Haematol. 114, 198-205 (2005).
- Domanska, U. M., Kruizinga, R. C., Nagengast, W. B., Timmer-Bosscha, H., Huls, G., de Vries, E. G., Walenkamp, A. M. A review on CXCR4/CXCL12 axis in oncology: No place to hide. Eur J Cancer. 49, 219-230 (2013).
- Tsou, L. K., Huang, Y. H., Song, J. S., Ke, Y. Y., Huang, J. K., Shia, K. S. Harnessing CXCR4 antagonists in stem cell mobilization, HIV infection, ischemic diseases, and oncology. Med Res Rev. , (2017).
- Schols, D., Struyf, S., Van Damme, J., Este, J. A., Henson, G., De Clercq, E. Inhibition of T-tropic HIV strains by selective antagonization of the chemokine receptor CXCR4. J Exp Med. 186, 1383-1388 (1997).
- Van Hout, A., D'Huys, T., Oeyen, M., Schols, D., Van Loy, T. Comparison of cell-based assays for the identification and evaluation of competitive CXCR4 inhibitors. PLoS One. 12, e0176057 (2017).
- De Clercq, E. The AMD3100 story: the path to the discovery of a stem cell mobilizer (Mozobil). Biochem Pharmacol. 77, 1655-1664 (2009).
- Debnath, B., Xu, S., Grande, F., Garofalo, A., Neamati, N. Small molecule inhibitors of CXCR4. Theranostics. 3, 47-75 (2013).
- Woodard, L. E., Nimmagadda, S. CXCR4-based imaging agents. J Nucl Med. 52, 1665-1669 (2011).
- Wang, Y., Xie, Y., Oupicky, D. Potential of CXCR4/CXCL12 Chemokine Axis in Cancer Drug Delivery. Curr Pharmacol Rep. 2, 1-10 (2016).
- Nimmagadda, S., Pullambhatla, M., Stone, K., Green, G., Bhujwalla, Z. M., Pomper, M. G. Molecular imaging of CXCR4 receptor expression in human cancer xenografts with [64Cu]AMD3100 positron emission tomography. Cancer Res. 70, 3935-3944 (2010).
- De Silva, R. A., Peyre, K., Pullambhatla, M., Fox, J. J., Pomper, M. G., Nimmagadda, S. Imaging CXCR4 expression in human cancer xenografts: evaluation of monocyclam 64Cu-AMD3465. J Nucl Med. 52, 986-993 (2011).
- Hatse, S., Princen, K., Liekens, S., Vermeire, K., De Clercq, E., Schols, D. Fluorescent CXCL12AF647 as a novel probe for nonradioactive CXCL12/CXCR4 cellular interaction studies. Cytometry A. 61, 178-188 (2004).
- Wootten, D., Christopoulos, A., Sexton, P. M. Emerging paradigms in GPCR allostery: implications for drug discovery. Nat Rev Drug Discov. 12, 630-644 (2013).
- Louis, K. S., Siegel, A. C. Cell viability analysis using trypan blue: manual and automated methods. Methods Mol Biol. 740, 7-12 (2011).
- Perry, C. M. Maraviroc: a review of its use in the management of CCR5-tropic HIV-1 infection. Drugs. 70, 1189-1213 (2010).
- Moyle, G., DeJesus, E., Boffito, M., Wong, R. S., Gibney, C., Badel, K., MacFarland, R., Calandra, G., Bridger, G., Becker, S. Proof of activity with AMD11070, an orally bioavailable inhibitor of CXCR4-tropic HIV type 1. Clin Infect Dis. 48, 798-805 (2009).
- Balabanian, K., Lagane, B., Infantino, S., Chow, K. Y., Harriague, J., Moepps, B., Arenzana-Seisdedos, F., Thelen, M., Bachelerie, F. The chemokine SDF-1/CXCL12 binds to and signals through the orphan receptor RDC1 in T lymphocytes. J Biol Chem. 280, 35760-35766 (2005).
- Burns, J. M., Summers, B. C., Wang, Y., Melikian, A., Berahovich, R., Miao, Z., Penfold, M. E., Sunshine, M. J., Littman, D. R., Kuo, C. J., Wei, K., McMaster, B. E., Wright, K., Howard, M. C., Schall, T. J. A novel chemokine receptor for SDF-1 and I-TAC involved in cell survival, cell adhesion, and tumor development. J Exp Med. 203, 2201-2213 (2006).
- Stoddart, L. A., Kilpatrick, L. E., Briddon, S. J., Hill, S. J. Probing the pharmacology of G protein-coupled receptors with fluorescent ligands. Neuropharmacology. 98, 48-57 (2015).
- Vernall, A. J., Hill, S. J., Kellam, B. The evolving small-molecule fluorescent-conjugate toolbox for Class A GPCRs. Br J Pharmacol. 171, 1073-1084 (2014).
- Stoddart, L. A., White, C. W., Nguyen, K., Hill, S. J., Pfleger, K. D. Fluorescence- and bioluminescence-based approaches to study GPCR ligand binding. Br J Pharmacol. 173, 3028-3037 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados