Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un test Flow Cytometry d’identifier des composés qui perturbent la fixation du Ligand de chimiokines CXC fluorescent marqué 12 au récepteur de chimiokines CXC 4
Dans cet article
Résumé
Une coulée d’essai de liaison cellulaire axée sur la cytométrie en flux est décrite qui est principalement utilisée comme outil de dépistage pour identifier les composés qui inhibent la fixation d’un ligand de chimiokines CXC fluorescent étiquetée 12 (CXCL12) pour les récepteurs de chimiokines CXC 4 (CXCR4).
Résumé
Cible pharmacologique des récepteurs couplés aux protéines G (RCPG) est d’une grande importance pour la santé humaine, comme dysfonctionnelle GPCR-mediated signaling contribue à la progression de nombreuses maladies. La paire de ligand/récepteur ligand de chimiokines CXC 12 (CXCL12) / récepteurs de chimiokines CXC 4 (CXCR4) a soulevé des intérêts cliniques significatifs, par exemple comme une cible potentielle pour le traitement du cancer et des maladies inflammatoires. Petites molécules ainsi que des anticorps thérapeutiques qui précisément ciblent CXCR4 et inhibent la fonction de récepteur sont donc considérés comme précieux outils pharmacologiques. Ici, un écoulement cytometry cellulaire test d’amplification qui permet l’identification de composés (par exemple, de petites molécules) qui abroge CXCL12 liaison à CXCR4, est décrite. Essentiellement, l’analyse se fonde sur la compétition pour récepteur liaison entre un montant fixe de CXCL12 fluorescent étiquetés, l’agoniste de chimiokine naturelle pour CXCR4 et composés sans étiquette. Par conséquent, l’utilisation indésirable de sondes marquées radioactivement est évitée dans cet essai. En outre, les cellules vivantes sont utilisés comme source de récepteur (CXCR4) au lieu de préparations de la membrane cellulaire. Cela permet une adaptation facile du dosage d’un format de plat, ce qui augmente le débit. Ce test s’est avéré être un test de découverte précieux médicament générique pour identifier les composés de ciblage CXCR4. Le protocole peut probablement être adapté aux autres RCPG, au moins si ligands fluorescent étiquetés sont disponibles ou peuvent être générés. Une connaissance préalable concernant les voies de signalisation intracellulaires qui sont induites lors de l’activation de ces RCPG, n’est pas nécessaire.
Introduction
Les récepteurs couplés aux protéines G (RCPG) sont des protéines de surface de cellules pouvant être activés par des ligands extracellulaires (p. ex., peptides, hormones protéiques, amines), donc réglementer beaucoup physiologique et le développement de procédés1. Quand un agoniste occupe sa poche de liaison de GPCR, le changement conformationnel induit dans la protéine réceptrice favorise la liaison d’intracellulaire récepteurs associés aux protéines G hétérotrimériques, consistant en Gα- PIB et sous-unitésβγ G. L’échange subséquent du GTP pour le PIB sur les résultats de sous-unitéα G dans la dissociation de les sous-unités de protéine G (Gα-GTP et Gβγ) qui, à son tour, va lancer plus loin en aval de signalisation des voies2,3. Lorsque le Gα-GTP est hydrolysé, re-association du Gα- PIB et sous-unitésβγ G convertira la protéine G dans son état au repos3,4. Différents types de protéines G existent (Gs, G,i/o, Gq, G12/13), qui sont classés par catégorie basée sur la similarité de séquence avec le G de sous-unitéα 5. Toutes ces protéines G induisent des voies de signalisation intracellulaire définis qui sous-tendent la réaction biologique à l’activation du récepteur. À la suite de l’activation des récepteurs, kinases GPCR (krg) phosphorylent la queue intracellulaire des RCPG, ce qui favorise une interaction avec le β-arrestins. Ce processus conduit à la résiliation de la signalisation, du récepteur de la désensibilisation et l’internalisation6G de protéines. Β-arrestins font également partie des complexes moléculaires multiples que le déclencheur de signalisation cascades indépendant de signalisation7G de protéines.
RCPG est parmi les cibles moléculaires plus validés pour une intervention thérapeutique, déréglementé GPCR signalisation médiée par, par exemple en raison du gain de fonction des mutations dans le gène du récepteur ou de la surexpression du récepteur, contribue à l’étiologie de la plupart 8de maladies humaines. Par conséquent, RCPG représente l’une des classes plus importantes de médicaments cibles étudiées par l’industrie pharmaceutique8,9,10. Un exemple notable d’un RCPG cliniquement pertinente est le récepteur de chimiokines CXC 4 (CXCR4), qui peut être activé par un ligand naturel unique, le CXC chemokine ligand 12 (CXCL12)11. En raison de son rôle établi comme un corécepteur majeur pour 1, virus de l’immunodéficience humaine (VIH-1) entrée et infection en cluster de différenciation 4 (CD4) positive T-lymphocytes12, CXCR4 a d’abord étudié comme une cible de médicament antiviral. Domiciliation de souches et progénitrices cellules13et CXCL12-CXCR4 interaction dans la moelle osseuse plus régule la rétention. Aussi, compte tenu de son implication dans nombreux aspects de la biologie (p. ex., la survie des cellules tumorales, métastase, angiogenèse tumorale liées) de cancer du14 et plusieurs autres maladies humaines (p. ex., les maladies inflammatoires)15, CXCR4 a soulevé beaucoup d’intérêt comme une cible prometteuse pour la découverte de médicaments. AMD3100, une petite molécule qui vise spécifiquement les CXCR4, initialement détectée comme un anti-VIH médicament candidat16 et demeure l’un des plus puissants antagonistes CXCR4 décrits à la date17. Son développement comme un médicament antiviral a toutefois abandonné18. Actuellement, cette molécule est utilisée comme agent de mobilisation des cellules souches pendant le traitement du myélome multiple et lymphome patients18. Plusieurs autres non chimiquement apparentés de petites molécules et des produits biologiques qui inhibent la fonction CXCR4 avec puissance variable ont été décrits19.
Méthodes de liaison du récepteur sont des outils précieux en pharmacologie qui permettent l’identification des composés (par exemple, de petites molécules) qui interagissent directement avec la GRCP d’intérêt. Afin d’effectuer des études de liaison, il n’y a aucun besoin de connaissances préalables concernant les propriétés de signalisation intracellulaires ou fonctionnalités d’un RCPG donnée. Bien que cela peut être considéré comme un avantage, elle implique que composés pour quel récepteur liaison peut être prouvé doivent être davantage caractérisé en évaluant leur potentielle activité agoniste ou antagoniste. Cette activité peut être évaluée à l’aide de tests pharmacologiques ou biologiques associés à la GRCP sous étude. Selon leur profil d’activité, les molécules de liaison du récepteur pourraient alors potentiellement évoluer pour devenir composés de plomb roman d’enquête dans les études précliniques et cliniques. Les molécules qui se lient spécifiquement à un récepteur à haute affinité peuvent aussi servir comme échafaudages à créer des outils thérapeutiques ou diagnostiques, par exemple par radiomarquage eux pour l’imagerie non invasive en vivo de cellules de tumeur20, ou comme potentiel véhicules pour l’administration ciblée d’agents thérapeutiques,21. Dans le cas de CXCR4, l’imagerie in vivo de cellules tumorales a déjà été démontrée à l’aide de modèles de souris dans lequel des molécules marquées CXCR4 ciblage a permis la visualisation du cancer humain xénogreffes20,22,23 .
Dans ce rapport, nous décrivons un protocole détaillé pour un test de fixation de concurrence qui permet l’identification de petites molécules et des produits biologiques qui interfèrent directement avec agoniste (CXCL12) contraignant à CXCR4. Le principe de base du dosage est la concurrence entre un montant fixe de ligand fluorescent étiqueté (CXCL12AF647, voir la Table des matières et réactifs) et non composés pour la liaison aux récepteurs protéiques17, 24. le signal fluorescent spécifique d’étiquetées ligand lié à des cellules individuelles exprimant le CXCR4 est ensuite analysé par cytométrie en flux. Ce signal fluorescent sera réduit lorsque non étiquetées de petites molécules perturbent l’interaction CXCL12AF647 / CXCR4. Le test utilise des cellules vivantes non manipulé endogène exprimant CXCR4 (c.-à-d., les cellules Jurkat). Par conséquent, aucune préparation de la membrane cellulaire n’est nécessaire, ce qui rend le test pratique, rapide et compatible avec un débit plus élevé. Comme un ligand fluorescent étiqueté est utilisé, la radioactivité est évitée.
CXCL12 étant l’agoniste naturel pour CXCR4, composés de petites molécules qui interfèrent avec la liaison CXCL12AF647 dans l’essai sont susceptibles d’interagir avec le site de liaison du récepteur orthosteric (c.-à-d., le site de liaison occupé par le naturel agoniste). Les molécules qui interagissent avec les sites de fixation de récepteurs topographiquement distincts de l’accepteur orthosteric ne sont pas détectés, si ils n’influencent pas la liaison de CXCL12. Par exemple, les MODULATEURS ALLOSTÉRIQUES positifs et négatifs, une catégorie importante et émergente des RCPG ciblant les molécules agissant sur les sites de liaison allostérique25, seront potentiellement pas ramassés avec ce test. En outre, si les composés identifiés avec cette fonction de test de liaison agonistes ou antagonistes des récepteurs ne peuvent pas être dérivés. Enquête des composés identifiés dans les essais pharmacologiques ou fonctionnelles supplémentaires axés sur le récepteur sera donc nécessaire. Ces tests pourraient inclure (une combinaison de) cellulaire fluorescence ou luminescence-tests pour la détection des seconds messagers (p. ex., Ca2 +, adénosine monophosphate cyclique (AMPc)), phénotypiques ou dosages biologiques et la β-arrestine tests de recrutement, le choix dont dépend les signalisation des propriétés spécifiques de la GRCP sous étude. Par conséquent, le test de liaison compétitive décrit dans les présentes principalement sert un test de dépistage initial qui doit être complétée par d’autres analyses pour permettre à une caractérisation approfondie des composés avec puissance de fixation de récepteurs cellulaires.
Protocole
1. maintien de la Culture cellulaire
Remarque : Toutes les étapes décrites aux points 1 et 2 sont effectués dans des conditions stériles dans une enceinte à flux laminaire.
- La croissance de cellules en T75 flacons de culture à 37 ° C et 5 % de CO2 dans un incubateur humidifié.
Remarque : Dans cet essai, Jurkat cellules (c'est-à-dire des lymphocytes T leucémiques humaines qui expriment endogène CXCR417) sont utilisés. Expression de CXCR4 à la surface de la cellule doit être évaluée tout au long de mise en culture cellulaire par cytométrie en flux. Une description de la procédure de cytométrie de flux et de réactifs pour déterminer les niveaux d’expression de récepteurs à la surface cellulaire est, cependant, pas dans le champ d’application du présent protocole, mais a été décrit précédemment17. - Laisser pousser en suspension jusqu'à ce qu’ils atteignent 80-85 % confluency les cellules Jurkat. Avant le passage des cellules dans une fiole de roman, permettent tous les réactifs à température ambiante (RT).
- Ajouter 20 mL de milieu frais complet (milieu RPMI-1640, 10 % sérum bovin fœtal (SVF), 2 mM de glutamine) dans une fiole de culture T75 roman.
- Ajouter 5 mL de suspension de cellules Jurkat du flacon original de T75 (contenant 25 mL de suspension cellulaire) dans le roman T75 flacon de culture. Incuber à 37 ° C et 5 % de CO2 dans un incubateur humidifié.
2. préparation des cellules Jurkat CXCL12 et du tampon d’essai pour la compétition Binding Assay.
- Préparer une solution de CXCL12AF647 (20 µg/mL ; voir Table des matières et réactifs) en dissolvant le réactif lyophilisé (stocké à-80 ° C, dans l’obscurité) dans de l’eau ultrapure additionné de 0,01 % (volume/volume) de Polysorbate 20. Stocker les aliquotes à usage unique de cette solution mère à-80 ° C, abri de la lumière.
- Préparer du tampon en ajoutant 40 mL HEPES (concentration finale de 1 M, 20 mM) de 200 mL Hank Balanced Salt solution (HBSS, 10 x, sans rouge de phénol et sans bicarbonate de sodium, 1 x concentration finale). Ajouter de l’eau ultrapure pour obtenir un volume final de 2 L. Ajouter 4 g (0,2 % poids/volume) l’albumine sérique bovine (BSA) et dissoudre la BSA via magnétique en remuant. Enfin, ajuster le pH à 7,4 (utiliser le NaOH pour cela) et filtrer la solution par le biais de 0,2 µm pores (voir Table des matières et réactifs) à l’aide d’une tubulure de vide.
Remarque : Ce tampon sera utilisé dans tous les autres mesures du protocole. - Compter le nombre et la viabilité des cellules. Pour ce faire, prélever un échantillon de la suspension cellulaire et le diluer dans une solution saline tamponnée au phosphate (PBS).
Remarque : Nous utilisons systématiquement un analyseur de viabilité cellulaire automatisée (voir Table des matières et réactifs) capable de compter les suspensions cellulaires à des concentrations variables. Pour le comptage de cellules, diluer 0,5 mL de suspension cellulaire dans 1,5 mL de PBS (autres dilutions, p. ex., 0,1 mL dans 1,9 mL PBS, sont également possibles). L’utilisation de cette méthode, qui repose sur la méthode d’exclusion du colorant bleu trypan, a été décrit précédemment26. Plusieurs autres dispositifs sont disponibles dans le commerce pour compter le nombre de cellules et de viabilité qui devrait fonctionner tout aussi bien. - Recueillir le nombre désiré de cellules (c.-à-d. ~ 24 x 106 cellules pour exécuter le test avec une plaque à 96 puits complete) dans un tube stérile de 50 mL par centrifugation (voir Table des matières et réactifs pour le type de centrifugeuse utilisée) à 400 x g pendant 5 min à RT.
- Doucement de décanter le liquide surnageant sans déranger le culot cellulaire. Ajouter frais de tampon (par exemple, 20 mL) et remettre en suspension les cellules par pipetage doucement verticalement.
- Centrifuger les cellules à nouveau à 400 x g pendant 5 min à température ambiante.
- Décanter le liquide surnageant à nouveau et Resuspendre le culot dans frais de tampon pour obtenir une densité de 5 x 106 cellules/mL.
3. essai de liaison de la concurrence
Remarque : L’essai de liaison réelle concurrence est effectuée à la RT et peut être effectuée dans des conditions non stériles.
- Diluer les composés incriminés dans du tampon (voir 2.2) pour obtenir la concentration désirée. Soit préparer une concentration fixe du composé pour le dépistage initial (par exemple, 10 concentration finale de µM) ou, alternativement, une série de dilutions de concentrations pour de plus amples caractérisation des composés (par exemple, un 1/3, 1/4 ou 1 / 5 série de dilutions à partir de 1 µM, concentration finale). N’oubliez pas que la solution composée deviendra à terme 2 x dilué lors de l’essai ; par conséquent, préparer un 2 x solution concentrée.
- Déposer 100 µL du composé en solution (2 x concentré) dans un clair 96 puits rond plaque de fond (voir Table des matières et réactifs) selon un prédéfinis expérimental prédisposer (p. ex., Figure 1).
Remarque : À ce stade, des échantillons témoins positifs et négatifs sont inclus dans l’essai. Dans l' échantillon témoin négatif, 100 µL de tampon est ajouté au lieu de composé dans les puits de la plaque à 96 puits. Pour l' échantillon de contrôle positif, le tampon est également ajouté au cours de cette étape. Voir aussi la Figure 1 pour une mise en page expérimentale typique dans lequel une série de dilutions de plusieurs composés est testée. - Ajouter 50 µL de la suspension cellulaire (voir 2.7 ; 0,25 x 106 cellules) provenant d’un réservoir de réactif dans la plaque 96 puits à l’aide d’une pipette multicanaux. Incuber les plaques pendant 15 minutes à la RT dans l’obscurité.
- Ajouter 50 µL de CXCL12 fluorescent étiquetés (c.-à-d. 100 ng/mL de CXCL12AF647 dans du tampon d’essai, 4 x concentré, concentration finale de 25 ng/mL) provenant d’un réservoir de réactif semblables aux puits de la plaque à 96 puits. Incuber 30 min à ta dans l’obscurité.
Remarque : Pour les échantillons de contrôle négatif, ajouter du tampon à la place. Par conséquent, le signal fluorescent détecté dans les échantillons de contrôle négatif correspondra à la signal de fond auto-fluorescente (Figure 1 a). Pour les échantillons de contrôle positif, ajouter 50 µL/puits de CXCL12AF647. Les échantillons de contrôle positif donnera le signal de fluorescence maximale détecté, puisque aucune inhibition éventuelle par incubation préalable avec des composés a été incluse (Figure 1 a). - Centrifuger la plaque 96 puits à 400 x g pendant 5 min à RT. Enlever le surnageant des cellules granulés en basculant sur la plaque. Sécher la plaque sur un mouchoir en papier.
- Ajouter 200 µL de tampon frais provenant d’un réservoir de réactif dans les puits à l’aide d’une pipette multicanaux. Procéder immédiatement.
- Centrifuger la plaque à nouveau pendant 5 min à 400 x g à RT. Enlever le surnageant en retournant sur la plaque et séchez-le à nouveau sur le tissu.
- Doucement Resuspendre le culot dans 200 µL de paraformaldéhyde à 1 % dissous dans du PBS. Cette étape va fixer les cellules.
- Continuer le protocole immédiatement avec la quantification de la fluorescence par cytométrie en flux.
4. analyse des échantillons par cytométrie en flux
CXCL12AF647 colorées et obsédé de cellules sont maintenant prêtes à être analysées à l’aide de cytométrie en flux. Plusieurs types de cytomètres peuvent être utilisés, mais ils ont besoin d’être équipés avec le laser correct (c'est-à-dire, un laser rouge, excitation entre ~ 630 nm) pour l’excitation et de filtres adaptés pour la détection de fluorophore (émission filtres ~ 660 nm). Ils doivent être capables de traiter des échantillons dans un format de plaque à 96 puits. Exemples de dispositifs de cytométrie en flux appropriés sont donnés dans Table des matières et réactifs.
- Démarrer l’appareil et ouvrez le logiciel correspondant (voir Table des matières et réactifs).
- Sélectionnez les paramètres cellulaires suivants pour être visualisé en format dot blot : transmettre scatter (FSC), diffusion latérale (SSC) et la chaîne de détection fluorophore (CXCL12AF647).
Remarque : Avec le paramètre FSC, les cellules sont discriminés selon leur taille, étant donné que l’absorption de la lumière détectée est proportionnelle au diamètre de la cellule. Le paramètre SSC, mesurer la diffusion de la lumière à un angle de 90°, fournit des informations sur la granularité des cellules. - Choisir un échantillon (par exemple, un échantillon témoin négatif) pour effectuer le blocage d’une population de cellules homogènes définie selon les paramètres FSC et SSC.
- Sélectionnez injection automatique de ~ 100 µL de cellules obsédés de cet échantillon de contrôle négatif dans le cytomètre en flux. Sélectionnez l’option « mixage » avant l’injection et utiliser un débit d’échantillon de 1,5 µL/s.
- Exécuter cet exemple, sélectionnez « Acquire Data. » Les paramètres FSC et SSC pour cet échantillon apparaîtra maintenant sur l’écran.
- Sélectionnez l’outil de blocage du logiciel. Basée sur la visualisation de la tache de dot FSC et SSC, prédéfinir une population cellulaire homogène et viable de blocage. Pour ce faire, créez un polygone (en utilisant l’outil de blocage du logiciel) qui comprend les cellules simples distribuées de façon homogène (« événements ») basés sur ces deux dimensions.
Remarque : La procédure de blocage a pour but de définir une population de cellules homogènes et viable qui sera utilisée pour une analyse plus approfondie. Gating repose sur l’hypothèse que la majorité des cellules viables forment une population de cellules homogènes selon les paramètres FSC et SSC. En effectuant cette étape, les débris cellulaires, les cellules mortes et agrégats cellulaires peuvent largement être exclus de l’analyse. Une illustration du processus d’injection est donnée dans la Figure 1 b.
- Sélectionnez cette option pour analyser les 20 000 « événements » (p. ex., les cellules individuelles) par exemple.
Remarque : Cela signifie que pour chaque échantillon, 20 000 cellules qui entrent dans le portail prédéfini seront éventuellement analysés. Acquisition de données pour chaque échantillon continuera jusqu'à ce que ce nombre d’événements est analysé. - Lancer l’exécution (sélectionnez « dossier Data »). Le dispositif de cytométrie de flux va maintenant analyser tous les échantillons par un en enregistrant l’intensité de la fluorescence moyenne (MFI) pour chaque échantillon. Cette IMF correspond au signal fluorescent moyens correspondant aux 20 000 cellules qui entrent dans le portail prédéfini.
5. analyse
L’IFM obtenu pour chaque échantillon permet d’effectuer tous les calculs supplémentaires. Analyse les données de cytométrie de flux peut être interprété par plusieurs logiciels disponibles dans le commerce (voir Table des matières et réactifs).
- Pour déterminer le pourcentage d’inhibition du signal fluorescent obligatoire, à la suite de pré-incubation composé, appliquez la formule suivante :
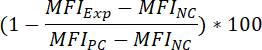
Où :
MFIExp = l’IMF de l’expérimental (= imprégnées d’insecticide composé) échantillon
MFIPC = l’IMF du contrôle positif
IFMNC = l’IMF du contrôle négatif - Pour déterminer la valeur50 d’un composé (c.-à-d., la concentration de composés qui peuvent réduire le signal fluorescent obligatoire de 50 %), appliquez la formule suivante :
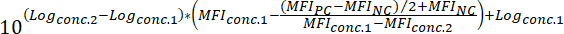
Où :
Logconc.2 = le journal d’une concentration du composé qui se traduit par moins de 50 % d’inhibition de la différence entre la valeur de l’IFM du PC et NC
Logconc.1 = le journal d’une concentration du composé qui se traduit par plus de 50 % d’inhibition de la différence entre la valeur de l’IFM du PC et NC
NOTE : Sinon, dans le cas de composés très actifs, une courbe dose-réponse couvrant plusieurs journaux de grandeur peut être générée basé sur l’IMF correspondant à chaque concentration d’essai du composé. En appliquant l’ajustement de courbe de régression non linéaire à l’aide des logiciels appropriés (voir Table des matières et réactifs), valeurs50 IC peuvent alors déduire les courbes générées. Un exemple de cette approche de l’analyse est illustré à la Figure 3.
Résultats
Le déroulement général de l’essai de liaison est présenté dans la Figure 1 a. Une illustration du type de données de cytométrie en flux obtenus pour des types d’échantillons différents dans une expérience standard (c'est-à-dire le contrôle négatif, contrôle positif et échantillon expérimental) est représentée dans la Figure 1 bet un schéma possible pour effectuer le dosage dans un format de plaque ...
Discussion
Par rapport à d’autres types d’essais de liaison (c.-à-d., liaison de saturation et des expériences de cinétique de fixation), analyses de liaison de compétition sont plus adaptés pour fins de présélection. En effet, ils permettent l’évaluation de grandes quantités de composés sans étiquette, par exemple de petites molécules, en marquant leur capacité d’interférer avec la fixation d’un montant fixe d’un ligand du récepteur marqué. Composés qui se lient à d’autres sites récepteur...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs aimeraient remercier Eric Fonteyn excellente assistance technique. Ce travail a été soutenu par la KU Leuven (subvention no. PF/10/018), Fonds voor Wetenschappelijk Onderzoek (FWO, grant no. G.485.08) et la Fondation Dormeur Vaduz.
matériels
| Name | Company | Catalog Number | Comments |
| BD FACSCanto II | Becton Dickinson | Not applicable | Flow cytometry device |
| BD FACSDIVA Software | |||
| BD FACSArray | Becton Dickinson | Not applicable | Flow cytometry device |
| BD FACSArray System Software | |||
| Graphpad Prism | Graphpad | software package used for nonlinear regression analysis in Figure 2 and Figure 3 | |
| FlowJo | FlowJo is now a wholly owned subsidiary of BD. | ||
| Vi-CELL | Beckman Coulter | Not applicable | cell viability analyzer |
| Sigma 3-18 KS | Sigma | Not applicable | centrifuge |
| AMD3100 | Sigma | A5602-5mg | specific CXCR4 antagonist |
| Maraviroc | Pfizer | antiretroviral drug, CCR5 antagonist, available for research at Selleckchem (cat#S2003), Sigma (cat#PZ0002) | |
| h-SDF1a (AF647) | ALMAC | CAF-11-B-01 | fluorescently labeled CXCL12, CXCL12AF647 |
| Fetal Bovine Serum (FBS) | Gibco (Life Technologies) | 10270-106 | |
| Bovine Serum Albumin (BSA) | Sigma | A1933-25G | |
| HBSS (10x), calcium, magnesium, no phenol red | Gibco (Life Technologies) | 14065-049 | |
| HEPES (1M) | Gibco (Life Technologies) | 15630-056 | |
| Dulbecco's Phosphate Buffered Saline (DPBS) | Gibco (Life Technologies) | 14190-094 | |
| Jurkat cells | ATCC | ||
| Reagent reservoir PP | Sigma | BR703411 | |
| Rapid flow filter: 0.2 µm aPES | Thermo Scientific | 566-0020 | |
| Sterilin microtiter plate, 96-well, U bottom, clear | Thermo Scientific | 611U96 | |
| Falcon tubes, 50ml | Greiner Bio-One | 227 261 | |
| Tissue culture flask (T75) | Corning | 353024 |
Références
- Fredriksson, R., Lagerstrom, M. C., Lundin, L. G., Schioth, H. B. The G-protein-coupled receptors in the human genome form five main families. Phylogenetic analysis, paralogon groups, and fingerprints. Mol Pharmacol. 63, 1256-1272 (2003).
- Milligan, G., Kostenis, E. Heterotrimeric G-proteins: A short history. Br J Pharmacol. 147 Suppl 1, S46-S55 (2006).
- Oldham, W. M., Hamm, H. E. Heterotrimeric G protein activation by G-protein-coupled receptors. Nat Rev Mol Cell Biol. 9, 60-71 (2008).
- Tuteja, N. Signaling through G protein coupled receptors. Plant Signal Behav. 4, 942-947 (2009).
- Neves, S. R., Ram, P. T., Iyengar, R. G protein pathways. Science. 296, 1636-1639 (2002).
- Gurevich, E. V., Tesmer, J. J., Mushegian, A., Gurevich, V. V. G protein-coupled receptor kinases: more than just kinases and not only for GPCRs. Pharmacol Ther. 133, 40-69 (2012).
- Smith, J. S., Rajagopal, S. The beta-arrestins: Multifunctional regulators of G protein-coupled receptors. J Biol Chem. 291, 8969-8977 (2016).
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Hopkins, A. L., Groom, C. R. The druggable genome. Nat Rev Drug Discov. 1, 727-730 (2002).
- Lappano, R., Maggiolini, M. G protein-coupled receptors: Novel targets for drug discovery in cancer. Nat Rev Drug Discov. 10, 47-60 (2011).
- Chatterjee, S., Behnam Azad, ., Nimmagadda, B., S, The intricate role of CXCR4 in cancer. Adv Cancer Res. 124, 31-82 (2014).
- Bleul, C. C., Farzan, M., Choe, H., Parolin, C., Clark-Lewis, I., Sodroski, J., Springer, T. A. The lymphocyte chemoattractant SDF-1 is a ligand for LESTR/fusin and blocks HIV-1 entry. Nature. 382, 829-833 (1996).
- Flomenberg, N., DiPersio, J., Calandra, G. Role of CXCR4 chemokine receptor blockade using AMD3100 for mobilization of autologous hematopoietic progenitor cells. Acta Haematol. 114, 198-205 (2005).
- Domanska, U. M., Kruizinga, R. C., Nagengast, W. B., Timmer-Bosscha, H., Huls, G., de Vries, E. G., Walenkamp, A. M. A review on CXCR4/CXCL12 axis in oncology: No place to hide. Eur J Cancer. 49, 219-230 (2013).
- Tsou, L. K., Huang, Y. H., Song, J. S., Ke, Y. Y., Huang, J. K., Shia, K. S. Harnessing CXCR4 antagonists in stem cell mobilization, HIV infection, ischemic diseases, and oncology. Med Res Rev. , (2017).
- Schols, D., Struyf, S., Van Damme, J., Este, J. A., Henson, G., De Clercq, E. Inhibition of T-tropic HIV strains by selective antagonization of the chemokine receptor CXCR4. J Exp Med. 186, 1383-1388 (1997).
- Van Hout, A., D'Huys, T., Oeyen, M., Schols, D., Van Loy, T. Comparison of cell-based assays for the identification and evaluation of competitive CXCR4 inhibitors. PLoS One. 12, e0176057 (2017).
- De Clercq, E. The AMD3100 story: the path to the discovery of a stem cell mobilizer (Mozobil). Biochem Pharmacol. 77, 1655-1664 (2009).
- Debnath, B., Xu, S., Grande, F., Garofalo, A., Neamati, N. Small molecule inhibitors of CXCR4. Theranostics. 3, 47-75 (2013).
- Woodard, L. E., Nimmagadda, S. CXCR4-based imaging agents. J Nucl Med. 52, 1665-1669 (2011).
- Wang, Y., Xie, Y., Oupicky, D. Potential of CXCR4/CXCL12 Chemokine Axis in Cancer Drug Delivery. Curr Pharmacol Rep. 2, 1-10 (2016).
- Nimmagadda, S., Pullambhatla, M., Stone, K., Green, G., Bhujwalla, Z. M., Pomper, M. G. Molecular imaging of CXCR4 receptor expression in human cancer xenografts with [64Cu]AMD3100 positron emission tomography. Cancer Res. 70, 3935-3944 (2010).
- De Silva, R. A., Peyre, K., Pullambhatla, M., Fox, J. J., Pomper, M. G., Nimmagadda, S. Imaging CXCR4 expression in human cancer xenografts: evaluation of monocyclam 64Cu-AMD3465. J Nucl Med. 52, 986-993 (2011).
- Hatse, S., Princen, K., Liekens, S., Vermeire, K., De Clercq, E., Schols, D. Fluorescent CXCL12AF647 as a novel probe for nonradioactive CXCL12/CXCR4 cellular interaction studies. Cytometry A. 61, 178-188 (2004).
- Wootten, D., Christopoulos, A., Sexton, P. M. Emerging paradigms in GPCR allostery: implications for drug discovery. Nat Rev Drug Discov. 12, 630-644 (2013).
- Louis, K. S., Siegel, A. C. Cell viability analysis using trypan blue: manual and automated methods. Methods Mol Biol. 740, 7-12 (2011).
- Perry, C. M. Maraviroc: a review of its use in the management of CCR5-tropic HIV-1 infection. Drugs. 70, 1189-1213 (2010).
- Moyle, G., DeJesus, E., Boffito, M., Wong, R. S., Gibney, C., Badel, K., MacFarland, R., Calandra, G., Bridger, G., Becker, S. Proof of activity with AMD11070, an orally bioavailable inhibitor of CXCR4-tropic HIV type 1. Clin Infect Dis. 48, 798-805 (2009).
- Balabanian, K., Lagane, B., Infantino, S., Chow, K. Y., Harriague, J., Moepps, B., Arenzana-Seisdedos, F., Thelen, M., Bachelerie, F. The chemokine SDF-1/CXCL12 binds to and signals through the orphan receptor RDC1 in T lymphocytes. J Biol Chem. 280, 35760-35766 (2005).
- Burns, J. M., Summers, B. C., Wang, Y., Melikian, A., Berahovich, R., Miao, Z., Penfold, M. E., Sunshine, M. J., Littman, D. R., Kuo, C. J., Wei, K., McMaster, B. E., Wright, K., Howard, M. C., Schall, T. J. A novel chemokine receptor for SDF-1 and I-TAC involved in cell survival, cell adhesion, and tumor development. J Exp Med. 203, 2201-2213 (2006).
- Stoddart, L. A., Kilpatrick, L. E., Briddon, S. J., Hill, S. J. Probing the pharmacology of G protein-coupled receptors with fluorescent ligands. Neuropharmacology. 98, 48-57 (2015).
- Vernall, A. J., Hill, S. J., Kellam, B. The evolving small-molecule fluorescent-conjugate toolbox for Class A GPCRs. Br J Pharmacol. 171, 1073-1084 (2014).
- Stoddart, L. A., White, C. W., Nguyen, K., Hill, S. J., Pfleger, K. D. Fluorescence- and bioluminescence-based approaches to study GPCR ligand binding. Br J Pharmacol. 173, 3028-3037 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon