Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein Flow Cytometry-basierte Assay, Verbindungen zu identifizieren, die Bindung von Eindringmittel beschriftet CXC-Chemokin-Liganden 12 an CXC-Chemokin-Rezeptor 4 stören
In diesem Artikel
Zusammenfassung
Ein Flow Cytometry-basierten zellulären Bindung Assay beschrieben wird, die in erster Linie als Screening-Instrument verwendet wird, um Verbindungen zu identifizieren, die die Bindung eines Eindringmittel beschrifteten CXC-Chemokin-Liganden 12 (CXCL12) an den CXC-Chemokin-Rezeptor 4 (CXCR4) hemmen.
Zusammenfassung
Pharmakologische Ausrichtung der G-Protein-gekoppelten Rezeptoren (GPCRs) ist von großer Bedeutung für die menschliche Gesundheit als dysfunktionale GPCR-vermittelten Signalisierung zu den Verlauf vieler Krankheiten beiträgt. Das Ligand-Rezeptor-paar CXC-Chemokin-Liganden 12 (CXCL12) / CXC-Chemokin-Rezeptor 4 (CXCR4) hat erhebliches klinische Interesse, zum Beispiel als ein potenzielles Ziel für die Behandlung von Krebs und entzündliche Erkrankungen erhöht. Kleine Moleküle sowie therapeutische Antikörper, die spezifisch CXCR4 und hemmen die Rezeptor-Funktion gelten daher als wertvolle pharmakologische Werkzeuge. Hier wird ein Flow Cytometry-basierten zellulären Assay, der Identifizierung von Verbindungen (z. B. kleine Moleküle) ermöglicht, die CXCL12 Bindung an CXCR4, aufzuheben beschrieben. Im wesentlichen stützt sich der Test auf den Wettbewerb für die Rezeptor-Bindung zwischen einen Festbetrag von Eindringmittel beschrifteten CXCL12, die natürliche Chemokin-Agonist für CXCR4 und unbeschriftete Verbindungen. Daher ist die unerwünschte Verwendung von radioaktiv markierte Sonden in diesem Assay vermieden. Darüber hinaus sind lebende Zellen als Quelle des Rezeptors (CXCR4) anstelle von Zellmembran Vorbereitungen verwendet. Dies ermöglicht eine einfache Anpassung des Assays zu einem Plattenformat, das den Durchsatz erhöht. Dieser Test hat sich gezeigt, zu einem wertvollen Generika Entdeckung Assay, CXCR4-targeting Verbindungen zu identifizieren. Das Protokoll kann wahrscheinlich andere GPCRs mindestens angepasst wenn Fluoreszent markierte Liganden zur Verfügung stehen oder erzeugt werden können. Vorkenntnisse über die intrazelluläre Signalwege, die durch die Aktivierung dieser GPCRs verursacht werden ist nicht erforderlich.
Einleitung
G-Protein-gekoppelten Rezeptoren (GPCRs) sind Zelle Oberflächenproteine, die von extrazellulären Liganden (z. B. Peptide, Proteinhormone, Amine) aktiviert werden können, dadurch regulieren viele physiologische und entwicklungspolitische Prozesse1. Wenn ein Agonist seine GPCR-Bindungstasche einnimmt, induzierte Konformationsänderung im Rezeptor-Protein fördert die Bindung von intrazellulären Rezeptor-assoziierten Heterotrimeric G Proteine, bestehend aus Gα- BIP und Gβγ Untereinheiten. Der anschließende Austausch von GTP für das BIP auf der Gα -Untereinheit resultiert die Dissoziation der G Protein Untereinheiten (α-GTP G und Gβγ), die wiederum weiter flussabwärts initiieren Signalisierung Wege2,3. Wenn die Gα-GTP hydrolysiert wird, erneute Vereinigung Gα- BIP und Gβγ Untereinheiten werden das G-Protein wieder in seiner ruhenden Zustand3,4umwandeln. Verschiedene Arten von G-Proteine vorhanden (GsGi/oGQ, G12/13), die kategorisiert sind, anhand der Reihenfolge Ähnlichkeit mit dem Gα -Untereinheit5. Alle diese G-Proteine induzieren definierte intrazellulären Signalwege, die die biologische Reaktion auf Rezeptor-Aktivierung zugrunde liegen. Im Anschluss an den Rezeptor-Aktivierung phosphorylieren GPCR Kinasen (GRKs) der intrazellulären Schweif von GPCRs, dadurch Förderung der Interaktion mit β-Arrestins. Dieser Prozess führt zur Beendigung des G Protein signaling, Rezeptor Desensibilisierung und Internalisierung6. Β-Arrestins sind ebenfalls Teil der Multi-molekulare komplexe, dass Trigger Signal unabhängig von G Protein signaling7Kaskaden.
GPCRs gehören zu der am besten validierten molekularen Zielstrukturen für eine therapeutische Intervention, da deregulierten GPCR-vermittelten Signalisierung, zum Beispiel durch Gain-of-Function-Mutationen in der Rezeptor-Gens oder Rezeptor-Überexpression, zur Ätiologie von vielen beiträgt Krankheiten des Menschen8. Daher sind GPCRs eines der wichtigsten Klassen von Angriffspunkte für Medikamente durch die Pharmaindustrie8,9,10untersucht. Ein bemerkenswertes Beispiel von einer klinisch relevanten GPCR ist der CXC-Chemokin-Rezeptor 4 (CXCR4), der durch eine einzige natürliche Liganden, der CXC Chemokin Liganden 12 (CXCL12)11aktiviert werden kann. Aufgrund der etablierten Rolle als eine große Ko-Rezeptor für Human Immunodeficiency Virus 1 (HIV-1) Eintrag und Infektion im Cluster der Differenzierung 4 (CD4) positiven T-Lymphozyten12, CXCR4 wurde zuerst als antivirale Medikament Ziel untersucht. CXCL12-CXCR4 Interaktion im Knochenmark weiter regelt die Aufbewahrung und Homing von Stamm- und Vorläuferzellen Zellen13. Auch sein Engagement in vielen Aspekten der Krebs-Biologie (z. B. Tumor Zelle überleben, Metastasen, Tumor-bezogene Angiogenese)14 und mehrere andere Krankheiten des Menschen (z. B. entzündliche Krankheiten)15, CXCR4 gegeben erhebliches Interesse als ein viel versprechendes Ziel für die Wirkstoffforschung angehoben. AMD3100, ein kleines Molekül, das richtet sich speziell an CXCR4, wurde zunächst als eine Anti-HIV-Medikament Kandidat16 entdeckt und ist immer noch eines der potentesten CXCR4-Antagonisten bis Datum17beschrieben. Seine Entwicklung als eine antivirale Medikament jedoch wurde eingestellt18. Derzeit ist dieses Molekül als Stammzell-Mobilisierung während der Behandlung des multiplen Myeloms und Lymphom Patienten18verwendet. Mehrere andere chemisch nicht verwandten kleine Moleküle und Biopharmazeutika, die CXCR4 Funktion mit unterschiedlicher Potenz hemmen wurden beschrieben19.
Rezeptor-Bindung-Methoden sind wertvolle Werkzeuge in der Pharmakologie, die es ermöglichen die Identifizierung von Verbindungen (z. B. kleine Moleküle), die direkte Interaktion mit der GPCR von Interesse. Um verbindliche Studien durchzuführen, ist ohne vorherige Kenntnis der intrazellulären Signal Eigenschaften und der Funktionsweise von einem bestimmten GPCR. Obgleich dies als Vorteil betrachtet werden kann, bedeutet es, dass Verbindungen für welche, die Rezeptor Bindung nachgewiesen werden kann weiter charakterisiert werden, durch die Auswertung ihrer potenziellen agonistischen und antagonistischen Aktivität müssen. Diese Aktivität kann mit pharmakologischen oder biologische Assays im Zusammenhang mit der GPCR unter Studie ausgewertet werden. Abhängig von ihrem Tätigkeitsprofil, verbindliche Rezeptormoleküle dann möglicherweise entstehen um neuartige Bleiverbindungen zur Untersuchung in präklinischen und klinischen Studien werden. Moleküle, die gezielt an einen Rezeptor mit hoher Affinität binden können auch als Gerüste therapeutische oder diagnostische Werkzeuge, zum Beispiel generieren, indem sie für die nicht-invasive in-Vivo Bildgebung von Tumor Zellen20, enzymatische oder Potenzial dienen. Fahrzeuge für gezielte Bereitstellung von Therapeutika21. Im Falle von CXCR4 in-Vivo Bildgebung von Tumorzellen bereits nachweislich mit Mausmodellen wobei beschrifteten CXCR4-targeting Moleküle die Visualisierung von Krebserkrankungen Xenotransplantate20,22,23 erlaubt .
In diesem Bericht beschreiben wir ein detailliertes Protokoll für einen Wettbewerb-Bindung-Assay, der es ermöglicht die Identifikation von kleinen Molekülen und Biologics, die direkt mit Agonist (CXCL12) verbindlich zu CXCR4 stören. Das grundlegende Prinzip des Tests ist der Wettbewerb zwischen einen Festbetrag von Eindringmittel markierten Liganden (CXCL12AF647, siehe Tabelle der Werkstoffe und Reagenzien) und stumme Verbindungen für die Bindung an den Rezeptor-Protein17, 24. das bestimmte Fluoreszenzsignal aus markierten Liganden gebunden, einzelne Zellen mit dem Ausdruck CXCR4 wird dann durch Durchflusszytometrie analysiert. Das Fluoreszenzsignal wird verringert, wenn unbeschriftete kleine Moleküle die Interaktion zwischen CXCL12AF647 und CXCR4 stören. Der Assay verwendet nicht manipulierten lebenden Zellen, die endogen CXCR4 ausdrücken (d. h. Jurkat-Zellen). Daher ist keine Zellmembran Vorbereitung erforderlich, wodurch der Assay, bequem, schnell und kompatibel mit erhöhter Durchsatz. Da eine Fluoreszent markierten Liganden verwendet wird, wird Radioaktivität vermieden.
Da CXCL12 den natürlichen Agonist für CXCR4 ist, dürften niedermolekulare Verbindungen, die CXCL12AF647 Bindung in der Probe stören die Interaktion mit der Orthosteric-Rezeptor-Bindungsstelle (d. h. die Bindungsstelle von natürlichen besetzt Agonist). Moleküle, die mit Rezeptor Bindungsstellen topographisch unterscheidet sich von der Orthosteric-Bindungsstelle interagieren würde bleiben unentdeckt, wenn sie die Bindung von CXCL12 keinen Einfluß haben. Zum Beispiel werden positive und negative allosterische Modulatoren, eine wichtige und neue Kategorie von GPCR targeting Moleküle auf allosterische Bindung Seiten25, möglicherweise nicht mit dieser Assay abgeholt. Darüber hinaus, ob die Verbindungen mit dieser Bindung-Assay-Funktion als Rezeptor-Antagonisten oder Agonisten identifiziert kann nicht abgeleitet werden. Untersuchung der identifizierten Verbindungen in zusätzliche pharmakologische oder funktionellen Rezeptor-bezogene Tests werden somit benötigt. Diese Tests beinhalten (eine Kombination aus) zellulären Fluoreszenz oder Lumineszenz basierende Assays zum Nachweis von sekundären Botenstoffen (z.B. Ca2 +, zyklische Adenosin Monophosphate (cAMP)), phänotypische oder biologische Assays und β-arrestin Rekrutierung-Assays, richtet sich die Wahl von denen auf die spezifischen Eigenschaften der Signalisierung von GPCR unter Studie. Daher dient die wettbewerbsfähige Bindung Assay hierin vor allem beschrieben als ein erstes Screening-Test, die mit anderen zellbasierte Assays ermöglichen eine detaillierte Charakterisierung der Verbindungen mit der Rezeptor-Bindung-Potenz ergänzt werden muss.
Protokoll
1. Wartung der Zellkultur
Hinweis: Alle unter 1 und 2 beschriebenen Schritte werden unter sterilen Bedingungen in einem Laminar-Flow Kabinett durchgeführt.
- Wachsen Sie Zellen in T75 Kulturflaschen bei 37 ° C und 5 % CO2 in einem befeuchteten Inkubator.
Hinweis: In diesem Test werden Jurkat Zellen (z.B. menschliche leukämischen T-Lymphozyten, die endogen CXCR417ausdrücken) verwendet. Ausdruck von CXCR4 an der Zelloberfläche sollten in der gesamten Zelle Kultivierung mittels Durchflusszytometrie ausgewertet werden. Eine Beschreibung der Flow Cytometry Verfahren und Reagenzien zu bestimmen Rezeptor Ausdruck Niveaus an der Zelloberfläche ist jedoch nicht in den Anwendungsbereich dieses Protokolls, sondern wurde beschriebenen zuvor17. - Lassen Sie Jurkat Zellen in Suspension wachsen, bis sie 80-85 erreichen % confluency. Lassen Sie bevor die Zellen in einem neuartigen Kolben Passagierung alle Reagenzien auf Raumtemperatur (RT).
- Ein neuartiger T75 Kultur Fläschchen 20 mL frische komplette Wachstumsmedium (RPMI-1640 Medium, 10 % fetalen bovine Serum (FBS), 2 mM Glutamin) hinzufügen.
- Der Roman T75 Kultur Flasche fügen Sie 5 mL Zellsuspension Jurkat aus der ursprünglichen T75-Flasche (mit 25 mL Zellsuspension hinzu). Inkubation bei 37 ° C und 5 % CO2 in einem befeuchteten Inkubator.
2. Vorbereitung von CXCL12, Testpuffer und Jurkat-Zellen für den Wettbewerb verbindlich Assay.
- Bereiten Sie eine Stammlösung von CXCL12AF647 (20 µg/mL; siehe Tabelle der Werkstoffe und Reagenzien) durch Auflösen der lyophilisierte Reagenz (bei-80 ° C im Dunkeln aufbewahrt) in Reinstwasser mit 0,01 % (Volumen/Volumen) Polysorbat 20 ergänzt. Bewahren Sie Einzelnutzung Aliquote aus dieser Stammlösung bei-80 ° C, vor Licht geschützt auf.
- Bereiten Sie Testpuffer durch Zugabe von 40 mL HEPES (1 M, 20 mM endgültige Konzentration), 200 mL Hank ausgewogen Salzlösung (HBSS, 10 X, ohne Phenol rot und Natriumbicarbonat, 1 x Endkonzentration). Hochreines Wasser hinzufügen zu einem Endvolumen von 2 L. hinzufügen 4 g (0,2 % Gewicht/Volumen) Rinderserumalbumin (BSA) erhalten, und lösen die BSA über magnetische rühren. Schließlich stellen Sie den pH-Wert auf 7,4 (NaOH dafür verwenden) und Filtern Sie die Lösung durch 0,2 µm-Poren (siehe Tabelle der Werkstoffe und Reagenzien) mit einem Vakuum Verteiler.
Hinweis: Dieser Assay-Puffer wird bei allen weiteren Schritten des Protokolls verwendet werden. - Zählen Sie die Anzahl und die Lebensfähigkeit der Zellen. Dazu nehmen Sie eine Probe der Zellsuspension und verdünnen Sie es in Phosphat gepufferte Kochsalzlösung (PBS).
Hinweis: Wir verwenden routinemäßig einen automatisierten Zelle Lebensfähigkeit Analyzer (siehe Tabelle der Werkstoffe und Reagenzien) in der Lage sind, zählen Zellsuspensionen in unterschiedlichen Konzentrationen. Verdünnen Sie für die Zellzählung 0,5 mL Zellsuspension in 1,5 mL PBS ( z. B. 0,1 mL in 1,9 mL PBS, andere Verdünnungen sind auch möglich). Die Verwendung dieser Methode, basierend auf der Trypan blau Ausgrenzung Färbemethode wurde zuvor26beschrieben. Mehrere andere Geräte sind im Handel erhältlich für die Zählung der Zellzahl und Lebensfähigkeit, die genauso gut funktionieren sollte. - Sammeln Sie die gewünschte Anzahl von Zellen (d. h. ~ 24 x 106 Zellen auf die Probe mit einem kompletten 96-Well-Platte laufen) in einer sterilen 50 mL Tube durch Zentrifugieren (siehe Tabelle von Materialien und Reagenzien für die Art der Zentrifuge verwendet) 400 X g für 5 min bei RT.
- Ohne zu stören die Zelle Pellets Gießen Sie sanft überstand ab. Fügen Sie frisches Testpuffer (z. B. 20 mL) und Aufschwemmen der Zellen durch sanft auf und ab pipettieren.
- Zentrifugieren Sie die Zellen wieder auf 400 X g für 5 min bei RT
- Wieder überstand abgießen und Aufschwemmen der Zelle Pellet in frischen Testpuffer, eine Dichte von 5 x 106 Zellen/mL zu erhalten.
3. Wettbewerb Bindung Assay
Hinweis: Der eigentliche Wettbewerb Bindung Test erfolgt bei RT und unter nicht sterilen Bedingungen durchgeführt werden kann.
- Die Verbindungen untersuchten in Testpuffer verdünnen (siehe 2.2), um die gewünschte Konzentration zu erhalten. Entweder einen festen Konzentration der Verbindung zur ersten Screening (z. B. 10 µM Endkonzentration) oder alternativ eine serielle Verdünnungsreihen Konzentrationen vorzubereiten, für nähere Charakterisierung der Verbindungen (z. B. 1/3, 1/4 oder 1 / 5 Verdünnungsreihen ab 1 µM, Endkonzentration). Denken Sie daran, das die zusammengesetzte Lösung letztlich 2 X in der Probe verdünnt werden. Deshalb bereiten Sie einen 2 x konzentrierte Lösung.
- 100 µL zusammengesetzte Lösung (2fach konzentriert) in eine klare 96-Well Runde Bodenplatte zu verzichten (siehe Tabelle der Werkstoffe und Reagenzien) nach einer vordefinierten experimentelle Anordnung (z. B. Abbildung 1).
Hinweis: In diesem Stadium sind negative und positive Kontrollproben in der Probe enthalten. In der negativen Stichprobewird 100 µL Testpuffer statt Verbindung in die Vertiefungen der 96-Well-Platte hinzugefügt. Für die positive Kontrollprobewird Testpuffer auch während dieses Schritts hinzugefügt. Siehe auch Abbildung 1 für ein typisches experimentellen Layout, in dem eine Verdünnungsreihe von mehreren Verbindungen getestet wird. - 50 µL Zellsuspension hinzufügen (siehe 2.7; 0,25 x 106 -Zellen) aus einem Reagenz-Reservoir in der 96-Well-Platte mit einer Mehrkanal-Pipette. Inkubieren Sie die Platte für 15 min bei RT im Dunkeln.
- Fügen Sie 50 µL Eindringmittel beschrifteten CXCL12 (d. h. 100 ng/mL von CXCL12AF647 in Testpuffer, 4-fach konzentriert, 25 ng/mL Endkonzentration) aus einem ähnlichen Reagenz Reservoir in den Vertiefungen der 96-Well-Platte. 30 min bei RT im Dunkeln inkubieren.
Hinweis: Für die negative Kontrollprobenfügen Sie Testpuffer stattdessen hinzu. Daher entspricht das Fluoreszenzsignal erkannt in der negativen Kontrollproben Autofluorescent Hintergrundsignal (Abbildung 1A). Fügen Sie für die Positivkontrolle Proben50 µL/Well von CXCL12AF647 hinzu. Die Positivkontrolle Proben ergibt die maximale Fluoreszenzsignal erkannt, da keine möglichen Hemmung durch Vorinkubation mit Verbindungen aufgenommen wurde (Abbildung 1A). - Zentrifugieren der 96-Well-Platte 400 X g für 5 min bei RT. Entfernen Sie den Überstand von der gebeizte Zellen durch Umklappen der Plattenrandes. Trocknen Sie die Platte auf einem Tuch.
- Der Brunnen mit einer Mehrkanal-Pipette fügen Sie 200 µL des frischen Testpuffer aus einem Reservoir Reagenz hinzu. Sofort gehen.
- Zentrifugieren Sie die Platte wieder für 5 min bei 400 X g bei RT entfernen der Überstand durch die Platte umdrehen und trocknen Sie es wieder auf das Gewebe.
- Sanft Aufschwemmen der Zelle Pellet in 200 µL 1 % Paraformaldehyd in PBS aufgelöst. Dieser Schritt wird die Zellen beheben.
- Das Protokoll sofort mit der Quantifizierung der Fluoreszenz durch Durchflusszytometrie fortgesetzt werden.
4. Analyse der Proben durch Durchflusszytometrie
CXCL12AF647 gebeizt und fixierten Zellen können nun mittels Durchflusszytometrie analysiert werden. Verschiedene Arten von fließen Cytometers können verwendet werden, aber sie müssen mit dem richtigen Laser (d. h., ein roter Laser, Erregung Palette ~ 630 nm) für Anregung und geeignete Filter für Fluorophor-Erkennung ausgerüstet werden (Emission Filter ~ 660 nm). Sie müssen in der Lage, Proben in einem 96-Well-Platte-Format sein. Beispiele für geeignete Flow Cytometry Geräte sind in der Tabelle der Werkstoffe und Reagenzienangegeben.
- Starten Sie das Gerät und die entsprechende Software zu öffnen (siehe Tabelle der Werkstoffe und Reagenzien).
- Wählen Sie die folgenden zellulären Parameter in einem Dot-Blot-Format dargestellt werden: forward Scatter (FSC), Side Scatter (SSC) und der Fluorophor (CXCL12AF647) Erkennung Kanal.
Hinweis: Mit dem FSC-Parameter werden Zellen diskriminiert basierend auf ihrer Größe, da die erkannten Lichtabsorption proportional zum Durchmesser der Zelle ist. Der SSC-Parameter, Messung der Lichtstreuung in einem 90° Winkel, informiert über die Granularität der Zellen. - Wählen Sie eine Probe (z. B. eine negative Kontrollprobe) gating einer definierten homogene Zellpopulation, basierend auf den FSC und SSC Parametern durchführen.
- Wählen Sie automatische Injektion von ~ 100 µL der fixierten Zellen aus dieser negativen Kontrollprobe in das Durchflusszytometer. Wählen Sie die Option "mischen" vor der Injektion und einen Probe-Durchfluss von 1,5 µL/s.
- Führen Sie dieses Beispiel durch Auswahl von "Acquire" Daten." Der FSC und SSC Parameter für dieses Beispiel werden nun auf dem Bildschirm angezeigt.
- Wählen Sie die Software gating Werkzeug. Basierend auf der FSC und SSC Dot-Blot-Visualisierung, eine homogene und tragfähige Zellpopulation durch Anspritzung vordefinieren. Hierzu erstellen Sie ein Polygon (mit der Software-gating-Tool), das homogen verteilten Einzelzellen ("Events") basierend auf diese beiden Dimensionen umfasst.
Hinweis: Das gating Verfahren zielt darauf ab, eine homogene und tragfähige Zellpopulation zu definieren, die für die weitere Analyse verwendet wird. Anspritzung beruht auf der Annahme, dass die Mehrheit der lebensfähigen Zellen eine homogene Zellpopulation bilden, basierend auf den FSC und SSC-Parametern. Mit diesem Schritt können Zelltrümmer, abgestorbene Zellen und Zellen Aggregate weitgehend von der weiteren Analyse ausgeschlossen. Eine Illustration des gating-Prozesses wird in Abbildung 1 bangegeben.
- Wählen Sie 20.000 "Ereignisse" (d. h. Einzelzellen) pro Probe zu analysieren.
Hinweis: Dies bedeutet, dass für jede Probe 20.000 Zellen, die in den vordefinierten Tor fallen schließlich analysiert werden. Datenerfassung für jede Probe wird fortgesetzt, bis diese Anzahl der Ereignisse analysiert wird. - Start "" ausführen (Wählen Sie "Record Data"). Die Flow-Zytometrie-Gerät wird nun alle eins nach dem anderen Proben durch die Aufnahme der mittlere Fluoreszenzintensität (MFI) für jede Probe. Dieses MFI entspricht der mittleren Fluoreszenzsignal entspricht 20.000 Zellen, die in den vordefinierten Tor fallen.
(5) Datenanalyse
Verwenden Sie die MFI erhalten für jede Probe für alle weiteren Berechnungen. Analyse der Flow Cytometry Daten erfolgt durch mehrere handelsübliche Software-Pakete (siehe Tabelle der Werkstoffe und Reagenzien).
- Um festzustellen, die prozentuale Hemmung der fluoreszierende Bindung Signal, als Folge der zusammengesetzten Vorinkubation gelten Sie die folgende Formel:
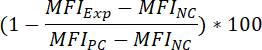
Wo:
MFIExp = MFI des experimentellen (= Verbindung behandelt) Probe
MFIPC = MFI der Positivkontrolle
MFI-NC = die MFI die negativ-Kontrolle - Um den IC50 Wert einer Verbindung (d. h. die Konzentration der Substanz, die das fluoreszierende Bindung Signal um 50 % reduzieren können) zu ermitteln, wenden Sie die folgende Formel an:
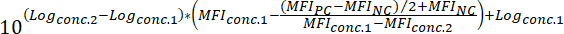
Wo:
Logconc.2 = das Protokoll eines Zusammenschlusses der Verbindung, die Ergebnisse in weniger als 50 % Hemmung der Differenz zwischen der MFI-Wert Ihres PCs und NC
Logconc.1 = das Protokoll eines Zusammenschlusses der Verbindung, die Ergebnisse in mehr als 50 % Hemmung der Differenz zwischen der MFI-Wert Ihres PCs und NC
Hinweis: Alternativ kann im Falle einer hochaktiven Verbindungen, eine Dosis-Wirkungs-Kurve über mehrere Protokolle Größenordnung generiert werden anhand der MFI entspricht jede getestete Konzentration der Verbindung. Durch die Anwendung nicht-lineare Regression Kurvenanpassung mittels geeigneter Software (siehe Tabelle der Werkstoffe und Reagenzien), IC50 Werte aus der generierten Kurven dann ableiten. Ein Beispiel für diese Analyseansatz ist in Abbildung 3dargestellt.
Ergebnisse
Die allgemeine Workflow des Assays Bindung ist in Figur 1Avorgestellt. Eine Illustration des Typs der Flow Cytometry Daten für unterschiedliche Probentypen in ein standard-Experiment (d. h. negative Kontrolle, positive Kontrolle und experimentelle Probe) ist in Abbildung 1 bund eine mögliche Platte Layout durchführen dargestellt. der Test in einem 96-Well-Platte-Format wird in Abbildung 1...
Diskussion
Im Vergleich zu anderen Arten der Bindung-Assays (z. B. Sättigung Bindung und kinetische verbindliche Experimente), sind Wettbewerb-Bindung-Assays am besten geeignet zu screening-Zwecken. In der Tat, ermöglichen sie Bewertung von Großserien unbeschriftete Verbindungen, zum Beispiel kleine Moleküle durch scoring ihre Fähigkeit, die Bindung eines Festbetrags eines beschrifteten Rezeptor Liganden stören. Verbindungen, die an andere Rezeptoren als die markierten Liganden binden könnte im Test unentdeckt zu bl...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Die Autoren möchten Eric Fonteyn für ausgezeichnete technische Unterstützung zu danken. Diese Arbeit wurde unterstützt von der KU Leuven (keine zu gewähren. PF/10/018), Fonds Voor Wetenschappelijk Onderzoek-(FWO, Nein zu gewähren. G.485.08) und der Fondation Dormeur Vaduz.
Materialien
| Name | Company | Catalog Number | Comments |
| BD FACSCanto II | Becton Dickinson | Not applicable | Flow cytometry device |
| BD FACSDIVA Software | |||
| BD FACSArray | Becton Dickinson | Not applicable | Flow cytometry device |
| BD FACSArray System Software | |||
| Graphpad Prism | Graphpad | software package used for nonlinear regression analysis in Figure 2 and Figure 3 | |
| FlowJo | FlowJo is now a wholly owned subsidiary of BD. | ||
| Vi-CELL | Beckman Coulter | Not applicable | cell viability analyzer |
| Sigma 3-18 KS | Sigma | Not applicable | centrifuge |
| AMD3100 | Sigma | A5602-5mg | specific CXCR4 antagonist |
| Maraviroc | Pfizer | antiretroviral drug, CCR5 antagonist, available for research at Selleckchem (cat#S2003), Sigma (cat#PZ0002) | |
| h-SDF1a (AF647) | ALMAC | CAF-11-B-01 | fluorescently labeled CXCL12, CXCL12AF647 |
| Fetal Bovine Serum (FBS) | Gibco (Life Technologies) | 10270-106 | |
| Bovine Serum Albumin (BSA) | Sigma | A1933-25G | |
| HBSS (10x), calcium, magnesium, no phenol red | Gibco (Life Technologies) | 14065-049 | |
| HEPES (1M) | Gibco (Life Technologies) | 15630-056 | |
| Dulbecco's Phosphate Buffered Saline (DPBS) | Gibco (Life Technologies) | 14190-094 | |
| Jurkat cells | ATCC | ||
| Reagent reservoir PP | Sigma | BR703411 | |
| Rapid flow filter: 0.2 µm aPES | Thermo Scientific | 566-0020 | |
| Sterilin microtiter plate, 96-well, U bottom, clear | Thermo Scientific | 611U96 | |
| Falcon tubes, 50ml | Greiner Bio-One | 227 261 | |
| Tissue culture flask (T75) | Corning | 353024 |
Referenzen
- Fredriksson, R., Lagerstrom, M. C., Lundin, L. G., Schioth, H. B. The G-protein-coupled receptors in the human genome form five main families. Phylogenetic analysis, paralogon groups, and fingerprints. Mol Pharmacol. 63, 1256-1272 (2003).
- Milligan, G., Kostenis, E. Heterotrimeric G-proteins: A short history. Br J Pharmacol. 147 Suppl 1, S46-S55 (2006).
- Oldham, W. M., Hamm, H. E. Heterotrimeric G protein activation by G-protein-coupled receptors. Nat Rev Mol Cell Biol. 9, 60-71 (2008).
- Tuteja, N. Signaling through G protein coupled receptors. Plant Signal Behav. 4, 942-947 (2009).
- Neves, S. R., Ram, P. T., Iyengar, R. G protein pathways. Science. 296, 1636-1639 (2002).
- Gurevich, E. V., Tesmer, J. J., Mushegian, A., Gurevich, V. V. G protein-coupled receptor kinases: more than just kinases and not only for GPCRs. Pharmacol Ther. 133, 40-69 (2012).
- Smith, J. S., Rajagopal, S. The beta-arrestins: Multifunctional regulators of G protein-coupled receptors. J Biol Chem. 291, 8969-8977 (2016).
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Hopkins, A. L., Groom, C. R. The druggable genome. Nat Rev Drug Discov. 1, 727-730 (2002).
- Lappano, R., Maggiolini, M. G protein-coupled receptors: Novel targets for drug discovery in cancer. Nat Rev Drug Discov. 10, 47-60 (2011).
- Chatterjee, S., Behnam Azad, ., Nimmagadda, B., S, The intricate role of CXCR4 in cancer. Adv Cancer Res. 124, 31-82 (2014).
- Bleul, C. C., Farzan, M., Choe, H., Parolin, C., Clark-Lewis, I., Sodroski, J., Springer, T. A. The lymphocyte chemoattractant SDF-1 is a ligand for LESTR/fusin and blocks HIV-1 entry. Nature. 382, 829-833 (1996).
- Flomenberg, N., DiPersio, J., Calandra, G. Role of CXCR4 chemokine receptor blockade using AMD3100 for mobilization of autologous hematopoietic progenitor cells. Acta Haematol. 114, 198-205 (2005).
- Domanska, U. M., Kruizinga, R. C., Nagengast, W. B., Timmer-Bosscha, H., Huls, G., de Vries, E. G., Walenkamp, A. M. A review on CXCR4/CXCL12 axis in oncology: No place to hide. Eur J Cancer. 49, 219-230 (2013).
- Tsou, L. K., Huang, Y. H., Song, J. S., Ke, Y. Y., Huang, J. K., Shia, K. S. Harnessing CXCR4 antagonists in stem cell mobilization, HIV infection, ischemic diseases, and oncology. Med Res Rev. , (2017).
- Schols, D., Struyf, S., Van Damme, J., Este, J. A., Henson, G., De Clercq, E. Inhibition of T-tropic HIV strains by selective antagonization of the chemokine receptor CXCR4. J Exp Med. 186, 1383-1388 (1997).
- Van Hout, A., D'Huys, T., Oeyen, M., Schols, D., Van Loy, T. Comparison of cell-based assays for the identification and evaluation of competitive CXCR4 inhibitors. PLoS One. 12, e0176057 (2017).
- De Clercq, E. The AMD3100 story: the path to the discovery of a stem cell mobilizer (Mozobil). Biochem Pharmacol. 77, 1655-1664 (2009).
- Debnath, B., Xu, S., Grande, F., Garofalo, A., Neamati, N. Small molecule inhibitors of CXCR4. Theranostics. 3, 47-75 (2013).
- Woodard, L. E., Nimmagadda, S. CXCR4-based imaging agents. J Nucl Med. 52, 1665-1669 (2011).
- Wang, Y., Xie, Y., Oupicky, D. Potential of CXCR4/CXCL12 Chemokine Axis in Cancer Drug Delivery. Curr Pharmacol Rep. 2, 1-10 (2016).
- Nimmagadda, S., Pullambhatla, M., Stone, K., Green, G., Bhujwalla, Z. M., Pomper, M. G. Molecular imaging of CXCR4 receptor expression in human cancer xenografts with [64Cu]AMD3100 positron emission tomography. Cancer Res. 70, 3935-3944 (2010).
- De Silva, R. A., Peyre, K., Pullambhatla, M., Fox, J. J., Pomper, M. G., Nimmagadda, S. Imaging CXCR4 expression in human cancer xenografts: evaluation of monocyclam 64Cu-AMD3465. J Nucl Med. 52, 986-993 (2011).
- Hatse, S., Princen, K., Liekens, S., Vermeire, K., De Clercq, E., Schols, D. Fluorescent CXCL12AF647 as a novel probe for nonradioactive CXCL12/CXCR4 cellular interaction studies. Cytometry A. 61, 178-188 (2004).
- Wootten, D., Christopoulos, A., Sexton, P. M. Emerging paradigms in GPCR allostery: implications for drug discovery. Nat Rev Drug Discov. 12, 630-644 (2013).
- Louis, K. S., Siegel, A. C. Cell viability analysis using trypan blue: manual and automated methods. Methods Mol Biol. 740, 7-12 (2011).
- Perry, C. M. Maraviroc: a review of its use in the management of CCR5-tropic HIV-1 infection. Drugs. 70, 1189-1213 (2010).
- Moyle, G., DeJesus, E., Boffito, M., Wong, R. S., Gibney, C., Badel, K., MacFarland, R., Calandra, G., Bridger, G., Becker, S. Proof of activity with AMD11070, an orally bioavailable inhibitor of CXCR4-tropic HIV type 1. Clin Infect Dis. 48, 798-805 (2009).
- Balabanian, K., Lagane, B., Infantino, S., Chow, K. Y., Harriague, J., Moepps, B., Arenzana-Seisdedos, F., Thelen, M., Bachelerie, F. The chemokine SDF-1/CXCL12 binds to and signals through the orphan receptor RDC1 in T lymphocytes. J Biol Chem. 280, 35760-35766 (2005).
- Burns, J. M., Summers, B. C., Wang, Y., Melikian, A., Berahovich, R., Miao, Z., Penfold, M. E., Sunshine, M. J., Littman, D. R., Kuo, C. J., Wei, K., McMaster, B. E., Wright, K., Howard, M. C., Schall, T. J. A novel chemokine receptor for SDF-1 and I-TAC involved in cell survival, cell adhesion, and tumor development. J Exp Med. 203, 2201-2213 (2006).
- Stoddart, L. A., Kilpatrick, L. E., Briddon, S. J., Hill, S. J. Probing the pharmacology of G protein-coupled receptors with fluorescent ligands. Neuropharmacology. 98, 48-57 (2015).
- Vernall, A. J., Hill, S. J., Kellam, B. The evolving small-molecule fluorescent-conjugate toolbox for Class A GPCRs. Br J Pharmacol. 171, 1073-1084 (2014).
- Stoddart, L. A., White, C. W., Nguyen, K., Hill, S. J., Pfleger, K. D. Fluorescence- and bioluminescence-based approaches to study GPCR ligand binding. Br J Pharmacol. 173, 3028-3037 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten