Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Analizar el tamaño, forma y direccionalidad de las redes de astrocitos acoplados
En este artículo
Resumen
Aquí presentamos un protocolo para evaluar la organización de redes astrocytic. El método descrito minimiza el sesgo para proporcionar medidas descriptivas de estas redes como recuento de células, el tamaño, zona y posición dentro de un núcleo. Anisotropía es evaluada con un análisis vectorial.
Resumen
Se ha vuelto cada vez más claro que los astrocitos modulan la función neuronal a nivel sináptico y unicelulares, sino también en el nivel de red. Los astrocitos están fuertemente conectados entre sí mediante uniones comunicantes y acoplamiento a través de estas uniones es dinámica y altamente regulada. Un concepto emergente es que funciones astrocíticos son especializadas y adaptadas a las funciones del circuito neuronal que se asocian. Por lo tanto, se necesitan métodos para medir diversos parámetros de redes astrocytic mejor describir las reglas que rigen la comunicación y el acoplamiento y entiendo sus funciones.
Aquí, usando el software de análisis de imagen (por ej., ImageJFIJI), se describe un método para analizar imágenes confocales de redes astrocytic reveladas por el acoplador de tinte. Estos métodos permiten 1) una detección automatizada y objetiva de las células marcadas, 2) cálculo del tamaño de la red, 3) cálculo de la orientación preferencial de tinte extendido dentro de la red y 4) reposición de la red dentro del área de interés .
Este análisis puede usarse para caracterizar astrocytic redes de un área en particular, comparar redes de diferentes áreas asociadas a diferentes funciones o comparar redes obtenidas en condiciones diferentes que tienen diferentes efectos en el acoplamiento. Estas observaciones pueden llevar a consideraciones funcionales importantes. Por ejemplo, se analizan las redes astrocíticos de un núcleo del trigémino, donde previamente hemos demostrado que acoplamiento astrocytic es esencial para la capacidad de las neuronas cambiar sus patrones de disparo de tónica a estallido rítmico1. Midiendo el tamaño, el parto y la orientación preferencial de redes astrocytic en este núcleo, podemos construir hipótesis sobre dominios funcionales que circunscriben. Varios estudios sugieren que varias otras áreas del cerebro, incluyendo corteza de barril, oliva superior lateral, glomérulos olfativos los núcleos sensoriales en el tálamo y la corteza visual, por nombrar algunos, pueden beneficiarse de un análisis similar.
Introducción
Muchos estudios han descrito cómo el diálogo astrositos neurona a nivel subcelular o sináptico puede tener implicaciones en las funciones neuronales y transmisión sináptica. Está bien establecido que los astrocitos son sensibles a los que rodean la actividad neuronal; de hecho, tienen receptores para muchos neurotransmisores como glutamato, GABA, acetilcolina y ATP (ver comentarios anteriormente publicados2,3,4). A cambio, astrocytic procesos elementos sinápticas ensheath y actividad neuronal influencia allí y en los sitios extrasinápticos regulación de la homeostasis iónica extracelular y soltando varios factores o transmisores tales como glutamato, D-serina y ATP 5 , 6 , 7.
Ha surgido la idea de que los astrocitos también pueden modular la función neuronal a nivel de red, con pruebas de que astrocytic acoplamiento espacial está regulada y corresponde a la segmentación neuronal en áreas caracterizadas por un claro anatómica compartimentalización (como zonas con representaciones sensoriales), indicando que los astrocitos se pareja otros astrocitos que sirven la misma función en lugar de sólo los que están cerca. En la oliva superior lateral, por ejemplo, redes más astrocíticos se orientan ortogonalmente al eje tonotópica8, mientras que en los glomérulos de corteza o olfactoty de barril, comunicación entre astrocitos es mucho más fuerte dentro de barriles o glomérulos y más débil entre los adyacentes unos9,10. En ambos casos, las redes astrocíticos se orientan hacia el centro de la glomerule o barril9,10.
Recientemente demostramos que actividad astrocytic modula la leña neuronal por la disminución de la concentración extracelular Ca2 + ([Ca2 +]e), presumiblemente a través de la liberación de S100β, Ca2 +-binding proteína11. Este efecto, que fue demostrado en una población de neuronas rhythmogenic del trigémino en la parte dorsal del sensorial principal del trigémino núcleo (NVsnpr, se cree que juegan un papel importante en la generación de movimientos de masticación), resulta del hecho de que rítmico disparo de estas neuronas depende de una persistente Na+ actual que es promovida por disminuciones de [Ca2 +]e11,12. Rítmico disparo de estas neuronas puede sacaron "fisiológico" por el estímulo de sus entradas o disminución artificial de la [Ca2 +]e. Demostramos más que acoplamiento astrocytic fue requerida para leña rítmica neuronal1. Esto levantó la posibilidad de que redes astrocytic pueden formar dominios funcionales circunscritos donde la actividad neuronal puede ser sincronizada y coordinada. Para evaluar esta hipótesis, primero teníamos que desarrollar un método para documentar rigurosamente la organización de estas redes en NVsnpr.
Estudios previos en redes astrocytic han descrito sobre todo el grado de acoplamiento en términos de número de la célula y la densidad y área cubierta. Tentativas para evaluar la forma de redes astrocytic y la dirección de acoplamiento de tinte en su mayoría fueron realizadas comparando el tamaño de las redes a lo largo de dos ejes (x e y) en el barril corteza9, hipocampo13,14, 15, barreloid campos del tálamo16, oliva superior lateral8, glomérulos olfativos10y14de la corteza. Los métodos aquí descritos permiten imparciales recuentos de células marcadas en una red y una estimación del área que cubren. También hemos desarrollado herramientas para definir la orientación preferida de acoplamiento dentro de una red y evaluar si la orientación preferida es hacia el centro del núcleo o en una dirección diferente. En comparación con los métodos utilizados anteriormente, este protocolo proporciona un medio para describir la organización y orientación de astrocytic redes en estructuras como el núcleo sensorial principal del trigémino dorsal que no tienen una clara anatómica conocida compartimentalización. En los estudios mencionados, la orientación de la red se describe como una relación a la forma de la estructura que ya está documentada (por ej., la barreloid en el tálamo, barriles en la corteza, las capas en el hipocampo y corteza, glomérulos en el bulbo olfatorio, etcetera). Además, análisis vectorial permite comparaciones de orientaciones reveladas bajo diferentes condiciones de acoplamiento. Para analizar si estos parámetros cambiaron según la posición de la red dentro del núcleo, también se desarrolló un método para cada red en referencia a los límites del núcleo. Estas herramientas pueden ser fácilmente adaptadas a otras áreas para investigar redes de células acopladas.
Protocolo
Todos los procedimientos de morada por el Reglamento de los institutos canadienses de investigación en salud y fueron aprobados por el Comité de uso y cuidado Animal de la Universidad de Montreal.
1. preparación de rodajas de cerebro de rata
- Preparar 1 L de una solución de sacarosa (tabla 1) y 1 L de líquido cerebral espinal artificial estándar (aCSF) (tabla 2).
- La burbuja de la solución de sacarosa con una mezcla de 95% O2 y 5% CO2 (carbogen) por 30 min antes de colocar de-80 ° C durante 30 minutos, hasta que la solución esté fría pero no totalmente congelado. Utilizar esta sacarosa helada como el búfer de corte para el corte del cerebro. Mantenga el hielo una vez que se retira del congelador.
- La burbuja de la aCSF con carbogen a través de todo el experimento. Utilice esta solución para el almacenamiento de la rebanada y la perfusión buffer en el cual se realizará la sujeción de parche y de biocitina de astrocitos. Preparar una recuperación de la rebanada con cámara para depositar las rebanadas de cerebro tan pronto como se cortan.
Nota: La cámara de retención recuperación rebanada podría ser por encargo y consiste esencialmente en un pequeño pozo con una malla en la parte inferior, suspendida en un pozo más grande que se llena con aCSF, en el cual se inserta un tubo para llevar carbogen desde la parte inferior. - Uso de 15 a 21 días de edad ratas Sprague-Dawley sin ningún sesgo de sexo o cepa específica. Anestesiar los animales con isoflurano (1 mL de isoflurano en una cámara de inducción de la anestesia). Verificar la profundidad de la anestesia pellizcando suavemente la pata trasera o la cola del animal.
- Decapitar a la rata con una guillotina, corte su cráneo con unas tijeras y quitar rápidamente el cerebro del cráneo con una espátula plana.
- Sumergir el cerebro en la helada solución de sacarosa de 30 s y transferencia que (con solución) un petri dish. Con una cuchilla de afeitar, retirar las porciones anterior y posterior a la zona para ser seccionados, si cortar rebanadas en el plano transversal.
- El bloque restante del tejido de cerebro en su parte rostral del pegamento y proceder a cortar rebanadas de cerebro (350 μm de espesor) en el medio de sacarosa con un vibratome. Luego, se transfieren las rebanadas recogidas en una recuperación sosteniendo la cámara llenado de aCSF carbogenated a temperatura ambiente (RT) hasta que estén listos para ser usados (permiten al menos 1 hora para la recuperación).
Nota: Los acontecimientos recientes en el campo permitan incubación prolongada hasta 24 h de rebanadas cerebrales in vitro condiciones17.
2. sulforodamina 101 (SR-101) etiquetado de astrocitos
- Calentar previamente un baño de agua a 34 ° C y colocar dos cámaras de incubación de la rebanada en él. Una de las cámaras de incubación de la rebanada se llenan de una solución de la aCSF que contiene 1 μM SR-101 y el otro sólo aCSF.
- Incubar las rebanadas en la cámara de incubación con el 1 μm SR-101 para 20 minutos y luego de la transferencia en la segunda incubación de cámara para enjuagar el exceso SR-101 del tejido. Dejar incubar durante 20 min o más a 34 ° C, mantenga entonces la cámara de incubación conteniendo las láminas a temperatura ambiente hasta que necesita18.
3. astrositos remendar y rellenar de biocitina
- Seleccione un sector y colocar en la cámara de grabación del microscopio. Parche de astrocitos, usar electrodos con una resistencia de 4-6MΩ cuando se llena con una solución a base de gluconato de potasio (ver tabla 3).
- Bajo orientación visual y con un micromanipulador, dirija el electrodo de registro hacia un SR-101-labeled astrositos como se muestra en la figura 1. Evitar células situadas en la superficie de la rodaja ya que son más propensos a dañarse o han perdido conexiones a células vecinas.
- Para evitar fugas de biocitina en el tejido, reducir al mínimo la presión positiva a la pipeta de parche y aplicarlo sólo cuando está cerca de los astrositos que serán parcheado (0.1-0.4 mL en una jeringa de 1 mL).
- Antes de parchear, ajuste la compensación de la pipeta y su capacitancia. Corregir para potenciales de Unión líquida, como críticos a los experimentos19comandos de voltaje preciso y exacto.
- Mueva la pipeta lo suficientemente cerca a los astrositos observar depresión causada por la presión positiva. Luego, retirar la presión positiva y lentamente aplique algo de presión negativa. Sujete los astrositos a -70 mV cuando el sello alcanza 100 MΩ. Espere hasta que el sello alcanza 1-3 GΩ. Continúe aplicando presión negativa hasta romper en la célula. Tenga cuidado mientras aplica presión negativa, ya que los astrocitos son muy frágiles.
- Evaluar las propiedades electrofisiológicas de la célula parcheada. En modo de pinza de voltaje, realizar un protocolo de corriente-voltaje de células completas con un comando de voltaje de la rampa de 600 ms de duración oscilan entre -120 y + 110 mV. En el modo de la abrazadera de la corriente, realizar un protocolo de paso IV en el que la inyección de 1000 ms pasos actual de 100 pA de -1 a 1 nA, la frecuencia de muestreo de las grabaciones de celulares es de 10 kHz.
Nota: Los astrocitos muestran un perfil lineal de corriente-voltaje sin ningún tipo de rectificación (como se muestra en la figura 1B) y ningún potencial de acción, disparando sobre la despolarización de la membrana (figura 1). El potencial de membrana de reposo (RMP) debe ser estable y positivo - 60mV. En algunas áreas del cerebro, el RMP de astrocitos es hyperpolarized más que en el NVsnpr. - Permiten la biocitina difundir dentro de los astrositos durante 30 minutos mientras se realiza un paso Protocolo IV cada 5 min.
- Retraer y separar la pipeta del parche con cuidado sin dañar los astrositos parcheado e inmediatamente Observe el desplazamiento de la pipeta antes de sacarlo de la sala de grabación. Reste ese valor de las grabaciones de potencial de membrana.
- Deje la rebanada del cerebro para descansar en la sala de grabación para un mínimo de 15 minutos (además de los 30 min de la inyección) para permitir la difusión del trazalíneas de astrositos parcheado a toda la red de células acopladas.
Nota: Para descubrir una red de células acopladas, sólo una sola celda debe ser parchada en el núcleo de interés. Si múltiples intentos se requieren para lograr un éxito parche, deseche el caso y tratar de parche en el lado contralateral o en otro segmento del cerebro. - Hacer una marca o una incisión en el tejido para identificar de qué lado de la rebanada es hacia arriba y transferir el segmento primero a una placa Petri conteniendo aCSF normal, luego en una solución de paraformaldehído al 4% (PFA). Incubar la rebanada a 4 ° C durante la noche.
PRECAUCIÓN: El PFA es extremadamente dañino. Usar equipo de protección en una campana de ventilación. Asegúrese de que todos los materiales (pinceles, tubos, etc.) que han estado en contacto con la PFA no tienen contacto con la recuperación que sostiene la cámara, cámara de incubación u otros materiales utilizados para la grabación del tejido fresco.
4. biocitina revelación
- Revelación con streptavidina fluorescente
- Lavado el cerebro rodajas 2 x 10 min en la salina de tampón de fosfato de 0,1 M (PBS) a TA. A continuación, revelan la biocitina por incubación de los cortes de cerebro con streptavidina conjugado con un fluoróforo en dilución 1/200 en PBS con detergente no iónico al 4% durante 4 horas a TA.
- Lavar las rodajas de cerebro 3 x 10 min en PBS de 0,1 M a RT y Monte las secciones sobre portaobjetos de vidrio con un medio de montaje acuoso. Coloque los lados que se inyectaron, cubreobjetos de las diapositivas y sellar con esmalte de uñas.
- Revelación con DAB
Nota: DAB (3,3 diaminobenzidina) revelación puede utilizarse cuando no se puede utilizar la fluorescencia. En este caso, método de solo revelación debe emplearse para hacer comparaciones.- Lavar las rodajas de cerebro 3 x 10 min en PBS a RT incuban las rebanadas en PBS + H2O2 0.5% durante 1 hora a TA.
- Enjuagar las rodajas 3 x 10 min en PBS a TA. Luego, incubar las rebanadas en una solución de tinción de complejo de avidina-biotina compuesta por PBS y 0,1% detergente no iónico + avidina-biotina peroxidasa complejo estándar tinción los reactivos del kit en 1/100 de 24 horas a TA.
- Enjuagar las rodajas 3 x 10 min en PBS a temperatura ambiente, incuban en una solución de PBS DAB 0.05% por 20 min a temperatura ambiente y transferencia en una solución de PBS + DAB 0.05% + H2O2 0.5%.
PRECAUCIÓN: DAB es extremadamente dañino. Usar equipo de protección en una campana de ventilación. La reacción de color se detiene cuando se transfieren las rebanadas en PBS. - Enjuagar las rodajas de 5 x 10 min en PBS a temperatura ambiente, las secciones de montaje en portaobjetos (hacia los lados que fueron inyectados hacia arriba) y deje secar sobre un portaobjetos secado Banco de noche a 34 ° C.
- Sumerja el portaobjetos durante 1 minuto en varios baños de alcohol al 70%, 95% y 100% y al final con un baño de xileno durante 1 minuto.
- Montar el portaobjetos con resina sintética basado en tolueno medio de montaje. Coloque el cubreobjetos sobre el portaobjetos y sellar con barniz de uñas.
5. proyección de imagen de la red
- Visualizar las redes astrocytic usando un microscopio de barrido confocal equipado con 20 X y 4 objetivos de X (o un objetivo apropiado para visualizar el núcleo entero donde se encuentra la red) y un láser para detectar el fluoróforo (en este caso, Alexa-594 es utilizado).
- Utilice el aumento de X 20 para hacer una pila de z de la red de células marcadas. Utilice una resolución de 800 x 800 píxeles y exploración velocidad de 12.5 μs/pixel.
Nota: Para toda la red de la imagen, son generalmente requiere múltiples pilas, y el número de pilas necesita ser ajustado para cada red. La resolución y exploración de la velocidad puede cambiarse, pero asegúrese de utilizar la misma configuración a todos los datos de la imagen. - Utilice los 4 aumentos para tomar imágenes de la red y la región de interés.
Nota: El 4 X la proyección de imagen se utiliza para determinar la posición de la red dentro del núcleo de interés. Siempre la imagen del mismo campo en luz transmitida. Esta imagen será útil si no puede determinar la frontera del núcleo de la imagen fluorescente confocal.
6. Análisis de la imagen
- Preparación de datos
- Utilizar el software ImageJFIJI (descargar en https://fiji.sc/). Abra el archivo y haga clic en aceptar en la ventana de "Opciones de importación de formatos de Bio".
- Para redefinir un z-stack que contiene sólo las rebanadas ópticas necesarias para la final z-pila (figura 2A), haga clic en el botón de la 'Pila' en la barra de herramientas (para encontrar, primero seleccione el stk | Z proyecto). Seleccione "Máxima intensidad" en el tipo de proyección ajuste (figura 2A). Guarde el archivo y asígnele el nombre "pila de archivo".
- Si el archivo de imagen contiene varios canales, dividirlo para conservar sólo el canal con la proyección de imagen de red astrocytic (imagen | Color | Dividir canales).
- Compruebe la configuración del pixel de la imagen [imagen | Propiedades | Pixel (con "1" para la dimensión de píxel)].
- Utilice la herramienta de fondo de restar (proceso | Restar el fondo) para quitar el fondo del etiquetado de biocitina. Utilice la función de vista previa para ajustar el radio de bola rodante, que generalmente está fijado en 50 píxeles (figura 2B).
- Utilice la herramienta de quitar outliers (proceso | Ruido | Quitar Outliers) si es necesario tras el paso del fondo restar. Seleccione a "Brillante" en el ajuste "Que afloramientos" (figura 2). Utilice la función de vista previa para ajustar el radio y umbral. Tenga cuidado con esta herramienta, ya que pueden difuminar los datos, como se muestra en la Figura 2D.
Nota: Esta herramienta quita las pequeñas manchas causadas por depósitos inespecíficos de streptavidina (como se indica por las flechas blancas de la figura 2B y 2C antes y después del tratamiento, respectivamente). - Ajuste el umbral (imagen | Ajustar | Umbral). Seleccione el modo "Default" y "B & W" (Figura 2E). Haga clic en "Aplicar".
Nota: Ajuste automático se puede utilizar, pero ajuste manual con las dos barras de control deslizante es preferido. El objetivo de este paso es reducir el ruido al máximo sin perder las células marcadas. - Convertir la imagen en una imagen binaria con la herramienta de proceso binario (proceso | Binario | Hacer el binario) como se muestra en la figura 2F. Guarde el archivo como un archivo TIFF y asígnele el nombre "binario".
- Conteo de células
- Compruebe el ajuste de la función de medida (analizar | Sistema de medición). Seleccione la opción "Centroide".
- Utilice la herramienta de "Analizar las partículas" en el "archivo binario" (Figura 3A) producido en el paso anterior (analizar | Analizar partículas) (figura 3 a la izquierda). Seleccione "Contornos" en la configuración de "Mostrar" (figura 3). Esto genera un nuevo archivo que muestra el resultado de la detección (figura 3B).
- Jugar con los parámetros: tamaño (para detectar sólo células, utilizar valores entre 30 y 6000) y circularidad (para determinar un intervalo de entre 0 a 1, en el que "1" define un círculo perfecto y "0" una forma al azar) para la detección (figura 3, parte a la izquierda). Ejecutar la detección haciendo clic en "OK".
Nota: Aparecerán dos tablas después de la detección: 1) una tabla titulada "Resumen" que proporciona el número de células detectadas y 2) una tabla titulada "Resultados" (figura 3, parte derecha) que proporciona las coordenadas x y y de cada célula. - Los valores de copiar y pegar en una aplicación de hoja de cálculo. Guardar esta tabla bajo el nombre "detección tabla". Un archivo con una trama de células detectadas también aparecerá (figura 3B). Guarde este archivo como un archivo TIFF bajo el nombre "archivo de detección".
- Si se detecta un grupo de células de 2 o más con etiquetadas en la red como una sola celda con la herramienta de analizar partículas porque están demasiado cerca entre sí, utilice la herramienta de Cuenca (proceso | Binario | Cuencas hidrográficas) en la imagen binaria antes de aplicar el análisis de partículas de la herramienta y rehacer los pasos de las partículas de analizar.
Nota: La herramienta cuenca crea una delimitación de 1 píxel de ancho entre células extremadamente cercanas. Una puede plug-in del contador celular se utiliza cuando es inequívoco y puede llevarse a cabo manualmente.
- Medición de área de red astrocytic
- Para medir el área de las redes, utilizar el archivo de detección mediante imagen J.
- Utilice la herramienta de selección de Polígono (izquierda haga clic en el botón en la barra de herramientas para seleccionar) para trazar un polígono que une todas las celdas ubicado en la periferia externa de la red (Figura 4A). Haga clic para comenzar a trazar el polígono y haga clic para cerrar.
Nota: Este polígono se definirá como una región de interés (ROI) y su superficie será medida para determinar el área superficial de la red. - Abrir la ventana de sistema de medición (análisis | Sistema de medición) y seleccione la opción "Area". Abra el administrador de ROI (analizar | Herramientas | ROI Manager) (Figura 4B). A continuación, añadir el polígono trazado en el ROI Manager haciendo clic en '' agregar '' (Figura 4B) y ejecutar la medición haciendo clic en '' medida '' en el ROI Manager.
Nota: La medida del área aparecerá en una tabla y se expresa en píxeles. No te olvides de convertir este valor con el factor de conversión para el microscopio utilizado para obtener un valor en micras2.
- Determinación del vector de dirección principal
- Determinación de la celda parcheada
- Abra el archivo de pila en ImageJFIJI e identificar la célula parcheada en el archivo de pila basada en su intensidad más fuerte de etiquetado (figura 4). A continuación, abra el archivo denominado "tabla de detección" en una aplicación de hoja de cálculo y encontrar el número asociado a la celda parcheada y sus coordenadas correspondientes.
- Si no es posible determinar con precisión la célula parcheada, rodean la zona donde el depósito de biocitina es más denso en la red imagen de las células acopladas, utilizando la herramienta polígono en ImageJFIJI, y se refieren a esta posición como la de la célula parcheada (figura 4).
- Utilice el administrador de ROI (analizar | Herramientas | ROI Manager). Luego, dibujar un ROI en la localización celular parcheado y agregarlo al ROI Manager (consulte la Figura 4B).
- Configurar una medición (análisis | Sistema de medición) y seleccione la opción "Centroide".
- En el ROI Manager, haga clic en "Medida" para obtener las coordenadas del centroide del área trazado. Utilizar estas coordenadas como punto de referencia para esta red específica.
- Traducción referencial
- Calcular las coordenadas de cada celda en referencia a los astrositos parches usando la siguiente fórmula:

Con: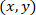 como las coordenadas de una celda determinada;
como las coordenadas de una celda determinada; 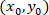 como las coordenadas de la celda parcheada o el punto referencial de la red; y
como las coordenadas de la celda parcheada o el punto referencial de la red; y 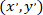 como las coordenadas de una celda determinada en el nuevo referencial.
como las coordenadas de una celda determinada en el nuevo referencial.
Nota: Expresar las coordenadas de cada celda en referencia a los astrositos parcheado es un paso importante en el cálculo de vectores de los astrositos parcheado. Tenga cuidado cuando use ImageJFIJI referencial para cualquier imagen se encuentra en la esquina superior izquierda de la imagen.
- Calcular las coordenadas de cada celda en referencia a los astrositos parches usando la siguiente fórmula:
- Determinación del vector principal de la orientación preferencial de
- Calcular las coordenadas del vector principal de orientación preferencial con la siguiente fórmula:
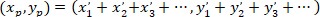
Con: (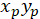 ) como las coordenadas del vector principal de la orientación preferencial; y
) como las coordenadas del vector principal de la orientación preferencial; y 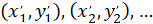 como las coordenadas de cada célula de la red obtenida con el parcheado de la célula como referencial.
como las coordenadas de cada célula de la red obtenida con el parcheado de la célula como referencial.
Nota: Para cada celda de la red, un vector se determina en relación con las coordenadas de los astrositos parcheado. El principal vector de orientación preferencial de la red es la suma de estos vectores. - Dividir la longitud del vector principal (proporcionado por las coordenadas obtenidas mediante la fórmula anterior) por el número de células en la red menos uno (ya que las coordenadas de la celda parcheada no se incluyen), para normalizar los valores y poder realizar comparaciones entre redes. Una vista esquemática de este análisis se presenta en la figura 5.
- Calcular las coordenadas del vector principal de orientación preferencial con la siguiente fórmula:
- Determinación de la celda parcheada
- Colocación de las redes analizadas en el núcleo de interés
- Alineación de 20 X y 4 X imágenes
- Para determinar la posición de cada red en el núcleo de interés (NVsnpr), utilice la imagen de X 4. Abre la imagen de X 4 con un editor de imágenes vectoriales.
- Seleccione la imagen de X 4 y modificar su tamaño, multiplicando por 5. La ventana de dimensión se encuentra en la parte derecha de la barra horizontal superior. Por ejemplo, para una imagen de 4 X que ha sido muestreada en 800 x 800 píxeles, cambiar la resolución de muestreo de 4000 x 4000 píxeles (para establecer la unidad de trabajo en píxeles, vaya a "Configuración de documento" en la pestaña de archivo: archivo | Configuración de documento). Exporte el archivo en formato TIFF y nombre "tamaño 4 X".
- Alinee la esquina superior izquierda de la imagen con la esquina inferior izquierda del documento de Adobe Illustrator.
Nota: El software proporciona coordenadas de un punto referencial de la esquina inferior izquierda. - Abre la imagen de X 20 de la red. Utilizar el '' archivo binario '' o '' archivo de detección '' porque son más fáciles de alinear. Entonces, jugar con la herramienta de opacidad en el '' archivo binario '' de la imagen de X 20 para alinear TI con el nuevo tamaño 4 X imagen.
Nota: La herramienta de opacidad está en la barra de herramientas horizontal superior. - Cuando la alineación es, seleccione mantener la herramienta pipeta para que aparezca la herramienta de medida y seleccione en la barra de herramientas izquierda.
- Uso la herramienta de medida para hacer clic en la esquina superior izquierda de la imagen de X 20 para obtener las coordenadas de los 20 X referencial del punto sobre el tamaño 4 X imagen.
Nota: Estas coordenadas, que se denominan el 20 X referencial (20XR en la figura 5), será útil para expresar la posición de cada red en un dibujo esquemático del núcleo.
- Normalización del núcleo de interés
Nota: Para resumir los datos, se utiliza una normalización del núcleo (NVsnpr) como un rectángulo. Los pasos se describen a continuación.- Abierto 4 X tamaño archivo en ImageJFIJI y usar la herramienta polígono para rodear el núcleo (NVsnpr). Usar la imagen con luz transmitida, si los bordes del núcleo no son capaces de verse; en cuyo caso, no olvide cambiar primero.
- Abra el administrador de ROI y añadir el ROI dibujado. En"sistema", seleccione la opción '' rectángulo delimitador ''.
Nota: La opción de "Rectángulo delimitador" calcula el rectángulo más pequeño alrededor del núcleo dibujado. - Haga clic en "Medida".
Nota: Aparecerá una tabla con BX y BY, las coordenadas de la esquina superior izquierda del rectángulo: '' rectángulo posición '' y proporciona la anchura y la altura. BX y BY son las coordenadas del rectángulo delimitador referencial que se conocen como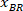 y
y 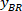 .
.
- Expresión de cada posición de la red en el núcleo normalizado
- Expresar las coordenadas de cada célula con el 4 X referencial (cuadrado negro en la figura 5) utilizando la siguiente fórmula:

Dónde: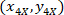 son las coordenadas de la celda con el 4 X referenciales;
son las coordenadas de la celda con el 4 X referenciales; 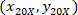 son las coordenadas de la celda con 20 X referencial (cuadrado naranja en la figura 5); y
son las coordenadas de la celda con 20 X referencial (cuadrado naranja en la figura 5); y 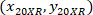 son las coordenadas de los 20 X punto referencial en la imagen de X 4 (20XR).
son las coordenadas de los 20 X punto referencial en la imagen de X 4 (20XR). - Expresar las coordenadas de la celda en el rectángulo de delimitación referencial (azul en la figura 5) utilizando la siguiente fórmula:
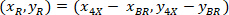
Dónde: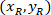 son la célula coordina en el rectángulo de delimitación referencial;
son la célula coordina en el rectángulo de delimitación referencial; 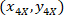 son las coordenadas de celda de 4 X referenciales; y
son las coordenadas de celda de 4 X referenciales; y  son las coordenadas del rectángulo delimitador referenciales en la imagen de X 4.
son las coordenadas del rectángulo delimitador referenciales en la imagen de X 4. - Transformar las coordenadas de la celda en el rectángulo de delimitación referencial el porcentaje de la anchura y altura del rectángulo delimitador que se utiliza la siguiente fórmula:
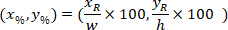
Dónde: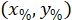 son la célula coordina en el porcentaje de la anchura y la altura del rectángulo delimitador;
son la célula coordina en el porcentaje de la anchura y la altura del rectángulo delimitador; 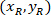 son la célula coordina en el rectángulo de delimitación referencial;
son la célula coordina en el rectángulo de delimitación referencial;  es el ancho del rectángulo delimitador medido por encima en el protocolo; y
es el ancho del rectángulo delimitador medido por encima en el protocolo; y 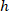 se mide la altura del rectángulo delimitador por encima en el protocolo.
se mide la altura del rectángulo delimitador por encima en el protocolo. - Para representar todas las redes en la misma figura, tener en cuenta la orientación de la rebanada (lado izquierdo o derecho). Para estandarizar los datos, la referencia se aplica en el lado izquierdo de la rebanada. Transferir las coordenadas de la red de la derecha a la izquierda aplicando la siguiente fórmula a coordenadas x:
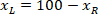
Dónde: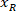 es la coordenada x del lado derecho de la rodaja; y
es la coordenada x del lado derecho de la rodaja; y 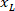 es la nueva coordenada x expresada en el referencial en el lado izquierdo de la rebanada.
es la nueva coordenada x expresada en el referencial en el lado izquierdo de la rebanada.
Nota: Como alternativa, espejo de la imagen en ImageJFIJI antes del análisis (imagen | Transformar | Voltear horizontalmente). - Para expresar las coordenadas del vector principal de la orientación preferencial, siga los mismos pasos con las coordenadas de la celda (6.5.3.1 a 6.5.3.4 los pasos).
Nota: La expresión de las coordenadas en porcentajes permite una recopilación de los datos como una sola parcela en la que el núcleo (NVsnpr) está diseñado como un rectángulo.
- Expresar las coordenadas de cada célula con el 4 X referencial (cuadrado negro en la figura 5) utilizando la siguiente fórmula:
- Alineación de 20 X y 4 X imágenes
- Estudio de la diferencia angular del principal vector de dirección preferencial
Nota: La diferencia angular de una red astrocítico se utiliza para determinar si su orientación preferencial es hacia el centro del núcleo de interés. Para calcular la diferencia angular (α en la figura 5), que es el ángulo entre el vector principal de la dirección preferencial de la red (línea de PD, rojo en la figura 5) y la línea que une P y C, se aplica el teorema de Al-Kashi en el triángulo PDC (véase inserción de Métodos complementariosy figura 5 ).- En primer lugar, calcular las longitudes diferentes mediante la aplicación del teorema de Pitágoras en un uso referencial cartesiano la siguiente ecuación:
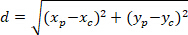
Dónde: las coordenadas de P y C son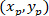 y
y  , respectivamente, en el rectángulo de delimitación referencial (calculado anteriormente).
, respectivamente, en el rectángulo de delimitación referencial (calculado anteriormente). - Determinar la diferencia angular en radianes utilizando la siguiente fórmula:
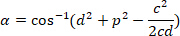
- Convertir la diferencia angular en grados (por ejemplo, con los grados de función en el software de Excel).
- Compilar todas las diferencias angulares mediante la representación en gráficos de barras verticales (figura 7 y 7 D) y determinar si existe una orientación preferencial de redes astrocytic.
- En primer lugar, calcular las longitudes diferentes mediante la aplicación del teorema de Pitágoras en un uso referencial cartesiano la siguiente ecuación:
Resultados
Acoplamiento entre las células del cerebro no es estático sino más bien dinámica regulada por muchos factores. Los métodos descritos fueron desarrollados para analizar redes astrocytic reveladas bajo diferentes condiciones y entender su organización en NVsnpr. Estos resultados han sido ya publicados1. Realizamos la biocitina relleno de astrocitos individuales en la parte dorsal de la NVsnpr en tres condiciones diferentes: en reposo (en condiciones control en ...
Discusión
Existe un número de métodos electrofisiológicos para evaluar funcional acoplamiento entre astrocitos23,24. Sin embargo, estos métodos no proporcionan información sobre el arreglo anatómico astrocytic redes. Varios estudios ya han demostrado que "colorante o trazador-acoplamiento", como aquí, se produce sólo en una fracción de junto a las células que se detectan por métodos electrofisiológicos25,26
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo es financiado por los institutos canadienses de investigación en salud, número de subvención o premio: 14392.
Materiales
| Name | Company | Catalog Number | Comments |
| NaCl | Fisher Chemicals | S671-3 | |
| KCl | Fisher Chemicals | P217-500 | |
| KH2PO4 | Fisher Chemicals | P285-500 | |
| MgSO4 | Fisher Chemicals | M65-500 | |
| NaHCO3 | Fisher Chemicals | S233-500 | |
| C6H12O6 Dextrose anhydrous | Fisher Chemicals | D16-500 | |
| CaCl2 dihydrated | Sigma | C70-500 | |
| Sucrose | Sigma | S9378 | |
| D-gluconic acid potassium salt | Sigma | G45001 | |
| MgCl2 anhydrous | Sigma | M8266 | |
| HEPES | Sigma | H3375 | |
| EGTA | Sigma | E4378 | |
| ATPTris Salt | Sigma | A9062 | |
| GTPTris Salt | Sigma | G9002 | |
| Biocytin | Sigma | B4261 | |
| Carbenoxolone disodium salt | Sigma | C4790 | |
| avidin-biotin complex : ABC kit | Vestor laboratories | PK-4000 | |
| Streptavidine-alexa 594 | Molecular Probes | S11227 | |
| Triton | Fisher Chemicals | BP151-500 | |
| Xylene | Fisher Chemicals | X5-1 | |
| Aqueous mounting medium 1 : Fluoromount-G | SouthernBiotech | 0100-01 | |
| Toluen-based synthetic resin mounting medium : Permount | Fisher Chemicals | SP15-100 | |
| Slide Drying Bench | Fisherbrand | 11-474-470 | |
| Vibratome | Leica | VT 1000S | |
| Microscope cover glass | Fisherbrand | 12-544A | |
| Microscope slide ColorFrost | Fisherbrand | 12-550-413 | |
| PFA | Fisherchemicals | 04042-500 | |
| Olympus FluoView FV 1000 Confocal microscope | Olympus | ||
| 40X water-immersion lens | Olympus | LUMPLFLN40XW | |
| 20X water-immersion lens | Olympus | XLUMPLFL20XW | |
| 4X water-immersion lens | Olympus | XLFLUOR4X/340 | |
| Micropipette puller | Sutter Instrument | P97 | |
| Micromanipulator | Sutter Instrument | MP 225 | |
| Camera CCD | Sony | CX-ST50 | |
| Black and white monitor | Sony | SSM-125 | |
| Digidata | Molecular devices | 1322A | |
| Patch Clamp amplifier | Axon instrument | Mulitclamp 700A | |
| Electrophysiology acquisition software | Molecular devices | pClamp 8 | |
| Electrophysiology analysis software | Molecular devices | Clampfit 8 | |
| Imaging analysis software | ImageJFIJI | Open source software. FIJI version including plug in package. | |
| Vector image editor | Adobe | Illustrator CS4 | |
| Spreadsheet application | Microsoft Office | Excel 2010 |
Referencias
- Condamine, S., Lavoie, R., Verdier, D., Kolta, A. Functional rhythmogenic domains defined by astrocytic networks in the trigeminal main sensory nucleus. Glia. 66 (2), 311-326 (2018).
- Verkhratsky, A., Orkand, R. K., Kettenmann, H. Glial calcium: homeostasis and signaling function. Physiological Review. 78 (1), 99-141 (1998).
- Christensen, R. K., Petersen, A. V., Perrier, J. F. How do glial cells contribute to motor control?. Current Pharmaceutical Design. 19 (24), 4385-4399 (2013).
- Verkhratsky, A., Steinhauser, C. Ion channels in glial cells. Brain Research Review. 32 (2-3), 380-412 (2000).
- Harada, K., Kamiya, T., Tsuboi, T. Gliotransmitter Release from Astrocytes: Functional, Developmental, and Pathological Implications in the Brain. Frontiers Neuroscience. 9, 499 (2015).
- Montero, T. D., Orellana, J. A. Hemichannels: new pathways for gliotransmitter release. Neuroscience. 286, 45-59 (2015).
- Araque, A., et al. Gliotransmitters travel in time and space. Neuron. 81 (4), 728-739 (2014).
- Augustin, V., et al. Functional anisotropic panglial networks in the lateral superior olive. Glia. 64 (11), 1892-1911 (2016).
- Houades, V., Koulakoff, A., Ezan, P., Seif, I., Giaume, C. Gap junction-mediated astrocytic networks in the mouse barrel cortex. Journal of Neuroscience. 28 (20), 5207-5217 (2008).
- Roux, L., Benchenane, K., Rothstein, J. D., Bonvento, G., Giaume, C. Plasticity of astroglial networks in olfactory glomeruli. Proceedings of the National Academy of Science of the United State of America. 108 (45), 18442-18446 (2011).
- Morquette, P., et al. An astrocyte-dependent mechanism for neuronal rhythmogenesis. Nature Neuroscience. 18 (6), 844-854 (2015).
- Brocard, F., Verdier, D., Arsenault, I., Lund, J. P., Kolta, A. Emergence of intrinsic bursting in trigeminal sensory neurons parallels the acquisition of mastication in weanling rats. Journal of Neurophysiology. 96 (5), 2410-2424 (2006).
- Anders, S., et al. Spatial properties of astrocyte gap junction coupling in the rat hippocampus. Philosophical Transactions of the Royal Society of London. Series B, Biological Science. 369 (1654), (2014).
- Houades, V., et al. Shapes of astrocyte networks in the juvenile brain. Neuron Glia Biology. 2 (1), 3-14 (2006).
- Rouach, N., Koulakoff, A., Abudara, V., Willecke, K., Giaume, C. Astroglial metabolic networks sustain hippocampal synaptic transmission. Science. 322 (5907), 1551-1555 (2008).
- Claus, L., et al. Barreloid Borders and Neuronal Activity Shape Panglial Gap Junction-Coupled Networks in the Mouse Thalamus. Cerebral Cortex. 28 (1), 213-222 (2018).
- Cameron, M. A., et al. Prolonged Incubation of Acute Neuronal Tissue for Electrophysiology and Calcium-imaging. Journal of Visualized Experiments. (120), (2017).
- Kafitz, K. W., Meier, S. D., Stephan, J., Rose, C. R. Developmental profile and properties of sulforhodamine 101--Labeled glial cells in acute brain slices of rat hippocampus. Journal of Neuroscience Methods. 169 (1), 84-92 (2008).
- Neher, E. Correction for liquid junction potentials in patch clamp experiments. Methods in Enzymology. , 123-131 (1992).
- Giaume, C., Leybaert, L., Naus, C. C., Saez, J. C. Connexin and pannexin hemichannels in brain glial cells: properties, pharmacology, and roles. Frontiers in Pharmacology. 4, 88 (2013).
- Torres, A., et al. Extracellular Ca(2)(+) acts as a mediator of communication from neurons to glia. Science Signaling. 5 (208), ra8 (2012).
- Ye, Z. C., Wyeth, M. S., Baltan-Tekkok, S., Ransom, B. R. Functional hemichannels in astrocytes: a novel mechanism of glutamate release. Journal of Neuroscience. 23 (9), 3588-3596 (2003).
- Ma, B., et al. Gap junction coupling confers isopotentiality on astrocyte syncytium. Glia. 64 (2), 214-226 (2016).
- Meme, W., Vandecasteele, M., Giaume, C., Venance, L. Electrical coupling between hippocampal astrocytes in rat brain slices. Neuroscience Research. 63 (4), 236-243 (2009).
- Ransom, B. R., Kettenmann, H. Electrical coupling, without dye coupling, between mammalian astrocytes and oligodendrocytes in cell culture. Glia. 3 (4), 258-266 (1990).
- Audesirk, G., Audesirk, T., Bowsher, P. Variability and frequent failure of lucifer yellow to pass between two electrically coupled neurons in Lymnaea stagnalis. Journal of Neurobiology. 13 (4), 369-375 (1982).
- Ewadinger, N., Syed, N., Lukowiak, K., Bulloch, A. Differential Tracer Coupling between Pairs of Identified Neurones of the Mollusc Lymnaea Stagnalis. Journal of Experimental Biology. 192 (1), 291-297 (1994).
- Griemsmann, S., et al. Characterization of Panglial Gap Junction Networks in the Thalamus, Neocortex, and Hippocampus Reveals a Unique Population of Glial Cells. Cerebral Cortex. 25 (10), 3420-3433 (2015).
- Kuwajima, T., et al. ClearT: a detergent- and solvent-free clearing method for neuronal and non-neuronal tissue. Development. 140 (6), 1364-1368 (2013).
- Gourine, A. V., et al. Astrocytes control breathing through pH-dependent release of ATP. Science. 329 (5991), 571-575 (2010).
- Forsberg, D., Ringstedt, T., Herlenius, E. Astrocytes release prostaglandin E2 to modify respiratory network activity. eLife. 6, (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados