È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analizzando le dimensioni, la forma e la direzionalità delle reti dei Astrocytes accoppiato
In questo articolo
Riepilogo
Qui presentiamo un protocollo per valutare l'organizzazione di reti astrocytic. Il metodo descritto riduce al minimo la distorsione per fornire misure descrittivi di queste reti come il conteggio delle cellule, dimensioni, area e posizione all'interno di un nucleo. Anisotropia è valutato con un'analisi vettoriale.
Abstract
È diventato sempre più chiaro che gli astrociti modulano la funzione di un neurone non solo a livello sinaptico e cella singola, ma anche a livello di rete. Gli astrociti sono fortemente collegati tra loro attraverso giunzioni di gap e accoppiamento attraverso queste giunzioni è dinamico e altamente regolato. Un concetto emergente è che funzioni astrocytic sono specializzate e adattate per le funzioni del circuito neuronale a cui sono associati. Pertanto, metodi per misurare i vari parametri delle reti astrocytic sono necessari per meglio descrivere le regole che disciplinano la loro comunicazione e l'accoppiamento e per capire meglio le loro funzioni.
Qui, utilizzando il software di analisi di immagine (ad es., ImageJFIJI), descriviamo un metodo per analizzare immagini confocal di reti astrocytic rivelati da tintura-accoppiamento. Questi metodi consentono di 1) un rilevamento automatizzato e imparziale delle cellule con etichettate, 2) calcolo della dimensione della rete, 3) calcolo dell'orientamento preferenziale della tintura diffuse all'interno della rete e 4) riposizionamento della rete all'interno dell'area di interesse .
Questa analisi consente di caratterizzare astrocytic reti di una particolare area, confrontare reti di diverse aree associate a funzioni diverse o reti ottenute in condizioni diverse che hanno effetti diversi sull'accoppiamento. Queste osservazioni potrebbero portare a importanti considerazioni funzionali. Per esempio, analizziamo le reti astrocytic di un nucleo trigeminale, dove precedentemente abbiamo indicato che accoppiamento astrocytic è essenziale per la capacità dei neuroni di passare loro schemi di attivazione da tonico per rottura ritmica1. Misurando le dimensioni, confinamento e orientamento preferenziale delle reti astrocytic in questo nucleo, possiamo costruire ipotesi sui domini funzionali che circoscrivono. Parecchi studi suggeriscono che diverse altre aree cerebrali, tra cui la corteccia barile, oliva superiore laterale, glomeruli olfattivi e sensoriali nuclei nel talamo e corteccia visiva, per citarne alcuni, possono beneficiare di una simile analisi.
Introduzione
Molti studi hanno descritto come il dialogo di neuroni-astrociti a livello sub-cellulare o sinaptico può avere implicazioni in funzioni neuronali e trasmissione sinaptica. È affermato che gli astrociti sono sensibili ai circostanti attività neuronale; Infatti, essi hanno recettori per neurotrasmettitori molti tra cui glutammato, GABA, acetilcolina e ATP (Vedi recensioni precedentemente pubblicate2,3,4). In cambio, astrocitari elabora elementi sinaptica Sheath e attività neuronale influenza entrambe c'e presso siti extrasynaptic regolazione dell'omeostasi ionica extracellulare e rilasciando diversi fattori o trasmettitori quali glutammato, D-serina e ATP 5 , 6 , 7.
L'idea che gli astrociti possono anche modulare la funzione di un neurone a livello di rete è emerso, con evidenza che accoppiamento astrocytic spazialmente è regolata e corrisponde a un neurone segmentazione in aree caratterizzate da un chiaro anatomico compartimentazione (come le zone con le rappresentazioni sensoriali), che indica che gli astrociti saranno accoppiare ad altri astrociti che servono la stessa funzione, piuttosto che solo quelli che sono nelle vicinanze. Nell'oliva superiore laterale, ad esempio, reti più astrocytic sono orientati ortogonalmente al tonotopico asse8, considerando che nei glomeruli barile corteccia o olfactoty, è molto più forte all'interno di botti o glomeruli comunicazione tra astrociti e più debole tra adiacente quelli9,10. In entrambi i casi, le reti astrocytic sono orientate verso il centro del glomerulo o canna9,10.
Recentemente abbiamo dimostrato che attività astrocytic modula il firing neuronale diminuendo la concentrazione di Ca extracellulare2 + ([Ca2 +]e), presumibilmente attraverso il rilascio di S100β, a Ca2 +-binding proteina11. Questo effetto, che è stato dimostrato in una popolazione di neuroni trigeminali rhythmogenic nella parte dorsale della sensitiva del trigeminal principale nucleo (NVsnpr, pensato per svolgere un ruolo importante nella generazione di movimenti masticatori), deriva dal fatto che ritmica infornamento in questi neuroni dipende da un Na persistente+ attuale che è promosso da diminuzioni della [Ca2 +]t11,12. Ritmica infornamento in questi neuroni può essere suscitata "fisiologicamente" da stimolazione dei loro ingressi o artificiale diminuzione della [Ca2 +]e. Abbiamo ulteriormente dimostrato che astrocytic accoppiamento è stato richiesto per firing neuronale ritmica1. Ciò ha sollevato la possibilità che reti astrocytic possono formare circoscritte domini funzionali dove le attività di un neurone può essere sincronizzato e coordinato. Per valutare questa ipotesi, abbiamo prima bisogno di sviluppare un metodo per documentare rigorosamente l'organizzazione di queste reti all'interno di NVsnpr.
Gli studi precedenti sulle reti astrocytic sono descritti principalmente nella misura dell'accoppiamento in termini di numero delle cellule e la densità e l'area coperta. Tentativi di valutare la forma delle reti astrocytic e la direzione della tintura-accoppiamento per la maggior parte sono stati eseguiti confrontando le dimensioni delle reti lungo due assi (x e y) nella canna corteccia9, ippocampo13,14, 15, barreloid campi il talamo16, laterale superiore oliva8, glomeruli olfattivi10e corteccia14. I metodi descritti qui abilitare imparziali conteggi delle cellule con etichettate in una rete e una stima dell'area che coprono. Abbiamo inoltre sviluppato strumenti per definire l'orientamento preferito di accoppiamento all'interno di una rete e per valutare se l'orientamento preferito è verso il centro del nucleo o in una direzione diversa. Rispetto ai metodi precedenti, questo protocollo fornisce un mezzo per descrivere l'organizzazione e l'orientamento delle reti astrocytic in strutture come il nucleo sensitivo principale del trigemino dorsale che non dispongono di un conosciuto Chiara anatomico compartimentalizzazione. Negli studi di cui sopra, l'orientamento della rete è descritto come una relazione tra la forma della struttura stessa che è già documentata (ad es., strati di barreloid nel talamo, barili nella corteccia, nell'ippocampo e nella corteccia, glomeruli in il bulbo olfattivo, ecc.). Inoltre, analisi vettoriale consente comparazioni di orientamenti ha rivelati in diverse condizioni di accoppiamento. Per analizzare se questi parametri cambiati secondo la posizione della rete all'interno del nucleo, abbiamo anche sviluppato un metodo per sostituire ogni rete in riferimento i confini del nucleo. Questi strumenti possono essere facilmente adattati ad altre aree per le reti d'istruttore delle cellule accoppiate.
Protocollo
Tutte le procedure di dimora secondo le regole di ricerca canadese istituti di salute e sono state approvate dal comitato di uso e cura degli animali University di Montreal.
1. preparazione di fettine di cervello di ratto
- Preparare 1 L di una soluzione a base di saccarosio (tabella 1) e 1 L di fluido cerebrale-spinale artificiale standard (aCSF) (tabella 2).
- Bolla la soluzione basata su saccarosio con un mix di 95% O2 e 5% CO2 (carbogen) per 30 min prima di posizionarlo a-80 ° C per circa 30 min, fino a quando la soluzione è freddo ma non completamente congelato. Utilizzare questo saccarosio gelida come il buffer di taglio per il taglio del cervello. Una volta che viene rimosso dal freezer, tenerlo sul ghiaccio.
- Bolla aCSF con carbogen attraverso l'intero esperimento. Utilizzare questa soluzione per l'archiviazione di fetta e come irrorante buffer in cui verrà eseguite la patch di bloccaggio e biocitina-riempimento degli astrociti. Preparare un recupero di fetta holding camera per depositare le fette di cervello, non appena vengono tagliati.
Nota: La camera di detenzione di recupero fetta potrebbe essere su ordine e consiste essenzialmente in un piccolo pozzo con una maglia in basso sospeso in un ben più grande che viene riempito con aCSF, in cui viene inserito un tubo per portare carbogen dal basso. - Utilizzare ratti Sprague-Dawley 15 - a 21-giorno-vecchio senza pregiudizi o sesso-ceppo-specifici. Anestetizzare l'animale con isoflurano (1 mL di isoflurano in una camera di induzione di anestesia). Controllare la profondità dell'anestesia pinzando delicatamente la zampa posteriore o coda dell'animale.
- Decapitare il ratto usando una ghigliottina, tagliare il suo cranio con le forbici e rimuovere rapidamente il cervello dal cranio con una spatola piatta.
- Immergere il cervello nella soluzione di saccarosio-base ghiacciata per circa 30 s e trasferimento (con la soluzione) che un petri piatto. Con una lama di rasoio, rimuovere le parti anteriore e posteriore nella zona per essere sezionato, se taglio fette nel piano trasversa.
- Incollare il blocco restante del tessuto cerebrale sul suo lato rostrale e procedere con il taglio fette del cervello (350 µm di spessore) a base di saccarosio medio utilizzando un vibratomo. Quindi, trasferire le fette raccolte in un recupero della holding camera piena di carbogenated aCSF a temperatura ambiente (TA) fino a quando non sono pronti per essere utilizzati (consentire almeno 1 ora per il recupero).
Nota: Recenti sviluppi nel campo consentono l'incubazione prolungata fino a 24 h di fettine di cervello in vitro condizioni17.
2. solforodamina 101 (SR-101) etichettatura degli astrociti
- Pre-riscaldare un bagno di acqua a 34 ° C e inserire due alloggiamenti di incubazione fetta in esso. Riempire una delle camere di incubazione fetta con una soluzione di aCSF contenente 1 μM SR-101 e riempire l'altro solo con aCSF.
- Incubare le fette nella camera di incubazione contenente 1 µM SR-101 per 20 minuti e poi il trasferimento nella seconda incubazione dell'alloggiamento da risciacquare SR-101 in eccesso dal tessuto. Lasciare Incubare per 20 minuti o più a 34 ° C, poi tenere la camera di incubazione contenente le fette a RT fino a quando essi sono necessari18.
3. astrocyte Patching e biocitina riempimento
- Selezionare una fetta e posizionarlo nella camera di registrazione del microscopio. A patch astrociti, utilizzare elettrodi con una resistenza di 4-6MΩ quando riempito con una soluzione a base di gluconato di potassio (Vedi tabella 3).
- Sotto guida visiva e utilizzando un micromanipolatore, diretto l'elettrodo di registrazione verso un Astrocita SR-101-etichettato come mostrato in Figura 1. Evitare le cellule situate sulla superficie della fetta dal momento che sono più probabilità di essere danneggiato o che hanno perso le connessioni per le cellule vicine.
- Per evitare perdite di biocitina nel tessuto, ridurre al minimo la pressione positiva aggiunta per la pipetta patch e applicarlo solo quando è vicino l'Astrocita che sarà implementata (0.1-0.4 mL in una siringa da 1 mL).
- Prima dell'applicazione di patch, regolare l'offset della pipetta e la sua capacità. Correggere per potenziali di giunzione liquida, come tensione accurati e precisi comandi sono critici al esperimenti19.
- Spostare la pipetta abbastanza vicino per l'Astrocita osservare depressione causata dalla pressione positiva. Rimuovere la pressione positiva, quindi applicare lentamente qualche pressione negativa. Morsetto del astrocyte a -70 mV quando il sigillo raggiunge 100 MΩ. Attendere fino a quando la guarnizione raggiunge 1-3 GΩ. Continuare ad applicare una pressione negativa fino a rompersi in cella. Prestare attenzione durante l'applicazione di pressione negativa, dato che gli astrociti sono molto fragili.
- Valutare le proprietà elettrofisiologiche della cella con patch. In modalità tensione-morsetto, eseguire un protocollo di corrente-tensione della intero-cellula con un comando di tensione di rampa 600 ms di durata che vanno da -120 a + 110 mV. In modalità corrente-morsetto, è necessario eseguire un protocollo di fase IV in cui l'iniezione di 1000 ms passi corrente di 100 pA da -1 a 1 nA, la frequenza di campionamento delle registrazioni della intero-cellula è 10 kHz.
Nota: Gli astrociti mostrano un profilo di corrente-tensione lineare senza alcun tipo di rettifica (come mostrato in Figura 1B) e nessun potenziale d'azione sparando su di depolarizzazione della membrana (Figura 1). Il potenziale di membrana di riposo (RMP) dovrebbe essere stabile e non positivo a - 60mV. In alcune zone del cervello, RMP degli astrociti è più hyperpolarized rispetto alla NVsnpr. - Consentire la biocitina diffondere all'interno del astrocyte per 30 min durante l'esecuzione di un passo protocollo IV ogni 5 min.
- Ritrarre e staccare la pipetta patch accuratamente senza danneggiare l'Astrocita patchato e immediatamente prendere nota dell'offset della pipetta prima di portarlo fuori dalla camera di registrazione. Sottrarre tale valore dalle registrazioni di potenziale di membrana.
- Lasciare la fetta di cervello di riposare nella camera di registrazione per un minimo di 15 min (oltre i 30 min per iniezione) per consentire la diffusione dell'elemento tracciante dall'Astrocita patchato per l'intera rete delle cellule accoppiate.
Nota: Per rivelare una rete di cellule accoppiate, solo una singola cella dovrebbe essere corretto nel nucleo di interesse. Se i tentativi multipli sono necessari per conseguire un successo patch, scartare il caso e cercare di patch sul lato controlaterale o in un'altra fetta di cervello. - Fare un segno o un incisione nel tessuto per identificare quale parte della fetta è rivolto verso l'alto e trasferire la fetta prima di una capsula di Petri contenente aCSF normale, quindi in una soluzione di paraformaldeide al 4% (PFA). Incubare la fetta a 4 ° C durante la notte.
Attenzione: PFA è estremamente dannoso. Utilizzare dispositivi di protezione in un cappuccio ventilato. Garantire che tutti i materiali (spazzole, tubi, ecc.) che sono stati a contatto con la PFA non contattare il recupero della holding camera, camera di incubazione o altri materiali utilizzati per la registrazione il tessuto fresco.
4. biocitina rivelazione
- Rivelazione con fluorescente streptavidine
- Lavaggio del cervello a fette 2 x 10 min in Salina 0,1 M di tampone fosfato (PBS) a TA. Poi, rivelano la biocitina incubando le fette di cervello con streptavidine coniugata con un fluoroforo alla diluizione di 1/200 in PBS con 4% detergente non ionico per 4 h a TA.
- Lavare le fette di cervello 3 x 10 min in 0.1 M PBS a RT e montare le sezioni sulle lastre di vetro usando un mezzo di montaggio acquoso. Posizionare i lati che sono stati iniettati verso l'alto, vetrino coprioggetti le diapositive e sigillare con smalto per unghie.
- Rivelazione con DAB
Nota: DAB (3,3-diaminobenzidina) rivelazione può essere utilizzato quando fluorescenza non può essere utilizzato. In questo caso, solo una rivelazione metodo dovrebbe essere impiegato per fare paragoni.- Lavare le fette di cervello 3 x 10 min in PBS a TA. Incubare le fette in PBS + H2O2 0,5% per 1 ora a TA.
- Sciacquare le fette 3 x 10 min in PBS a TA. Quindi, incubare le fette in una soluzione di macchiatura complesso avidina-biotina composto di PBS e detergente non ionico 0,1% + avidina-biotina perossidasi complessi standard colorazione reagenti del kit a 1/100 per 24 ore a TA.
- Sciacquare le fette 3 x 10 min in PBS a RT, li Incubare in una soluzione di PBS + DAB 0.05% per 20 min a RT e trasferirli in una soluzione di PBS + DAB 0,05% + H2O2 0,5%.
Attenzione: DAB è estremamente dannoso. Utilizzare dispositivi di protezione in un cappuccio ventilato. La reazione al colore è interrotta quando le fette sono trasferite in PBS. - Sciacquare le fette 5 x 10 min in PBS a RT, montare le sezioni sulle lastre di vetro (affrontare i lati che sono stati iniettati verso l'alto) e lasciarli asciugare su una diapositiva essiccazione panchina durante la notte a 34 ° C.
- Immergere i vetrini per 1 min in diversi bagni di alcool al 70%, 95% e 100% e fine con un bagno di xilene per 1 min.
- Montare i vetrini utilizzando una resina sintetica a base di toluene, mezzo di montaggio. Posizionare i vetrini coprioggetti sugli scivoli e sigillare con smalto per unghie.
5. rete Imaging
- Visualizzare le reti astrocytic utilizzando un microscopio confocale dotato X 20 e 4 X obiettivi (o un obiettivo appropriato per visualizzare l'intero nucleo in cui si trova la rete) e un laser per rilevare il fluoroforo (in questo caso, Alexa-594 è utilizzato).
- Utilizzare l'ingrandimento X 20 per rendere uno z-stack della rete delle cellule con etichettate. Utilizzare una risoluzione di 800 x 800 pixel e scansione velocità di 12,5 μs/pixel.
Nota: Al fine di tutta la rete di immagine, più pile sono di solito necessari, e il numero di stack deve essere regolata per ogni rete. La velocità di risoluzione e di scansione può essere cambiata, ma assicuratevi di utilizzare le stesse impostazioni per tutti i dati di immagine. - Utilizzare l'ingrandimento 4x per prendere le immagini della rete e della regione di interesse.
Nota: La 4 X imaging viene utilizzato per determinare la posizione di rete all'interno del nucleo di interesse. Sempre immagine lo stesso campo in luce trasmessa. Questa immagine sarà utile se non è possibile determinare il bordo del nucleo nell'immagine fluorescente confocal.
6. image Analysis
- Preparazione dei dati
- Utilizzare il software ImageJFIJI (scaricarlo a https://fiji.sc/). Aprire il file e fare clic su OK nella finestra "Opzioni di importazione di Bio-Formats".
- Per ridefinire uno z-stack che conterrà solo le sezioni ottiche necessarie per lo z-stack finale (Figura 2A), fare clic sulla manopola "Stack" nella barra degli strumenti (per trovare, in primo luogo selezionare stk | Progetto di Z). Selezionare "Max intensità" nell'impostazione del tipo di proiezione (Figura 2A). Salvare il file e il nome "file di stack".
- Se il file di immagine contiene diversi canali, dividerlo per conservare solo il canale con l'imaging di rete astrocitari (immagine | Colore | Dividere i canali).
- Controllare le impostazioni di pixel dell'immagine [immagine | Proprietà | Pixel (con "1" per dimensione in pixel)].
- Utilizzare lo strumento di sottrazione sfondo (processo | Sottrarre sfondo) per rimuovere lo sfondo di biocitina etichettatura. Utilizzare la funzione di anteprima per impostare il raggio di rotolamento sfere, che è generalmente fissato a 50 pixel (Figura 2B).
- Utilizzare lo strumento di rimozione outlier (processo | Rumore | Rimuovere i valori erratici) se necessario seguendo il passo di sfondo Sottrai. Selezionare "Bright" in ambito "Che Outliers" (Figura 2). Utilizzare la funzione di anteprima per impostare il raggio e soglia. Stare attenti con questo strumento, dal momento che esso può offuscare i dati, come mostrato in Figura 2D.
Nota: Questo strumento rimuove le piccole macchie causate da depositi aspecifici di streptavidine in (come mostrato dalle frecce in Figura 2B e 2C bianche prima e dopo il trattamento, rispettivamente). - Regolare la soglia (immagine | Regolare | Soglia). Selezionare la modalità di "Default" e "B & W" (Figura 2E). Fare clic su "applica".
Nota: Regolazione automatica può essere utilizzato, ma la regolazione manuale con le due barre di scorrimento è preferito. L'obiettivo di questa fase è quello di ridurre il rumore al massimo senza perdere tutte le celle con etichettate. - Convertire l'immagine in un'immagine binaria con lo strumento binario processo (processo | Binario | Rendere il binario) come mostrato in Figura 2F. Salvare il file come un file TIFF e denominarlo "file binario".
- Conta cellulare
- Controllare l'impostazione della funzione di misura (Analyze | Impostare la misurazione). Selezionare l'opzione "Baricentro".
- Utilizzare lo strumento di "Analizzare particelle" sul "file binario" (Figura 3A) prodotta nel passaggio precedente (Analyze | Analizzare particelle) (Figura 3 a sinistra). Selezionare "Contorni è" l'impostazione "Visualizza" (Figura 3). Questo genera un nuovo file che mostra il risultato della rilevazione (Figura 3B).
- Giocare con i parametri: dimensione (per rilevare solo le celle, utilizzare valori compresi tra 30 e 6000) e circolarità (per determinare un intervallo tra 0 e 1, in cui "1" definisce un cerchio perfetto e "0" una forma casuale) per perfezionare la rilevazione (Figura 3, parte a sinistra). Eseguire il rilevamento facendo clic su "OK".
Nota: Due tabelle verranno visualizzati dopo la rilevazione: 1) una tavola dal titolo "Riepilogo" che fornisce il numero di cellule rilevate e 2) una tavola dal titolo "Risultati" (Figura 3, a destra) che fornisce le coordinate x e y di ogni cella. - Copiare i valori e incollarli in un foglio elettronico. Salvare la tabella sotto il nome "rilevamento tavolo". Un file con una trama di cellule rilevate verrà visualizzati anche (Figura 3B). Salvare questo file come un file TIFF sotto il nome di "file di rilevamento".
- Se un gruppo di 2 o più con etichetta celle nella rete vengono rilevato come una singola cella con lo strumento di analisi particelle perché sono troppo vicino a vicenda, utilizzare lo strumento di spartiacque (processo | Binario | Spartiacque) sull'immagine binaria prima di applicare le particelle analizza tool e rifare la procedura di particelle analizza.
Nota: Lo strumento di spartiacque crea una delimitazione dei pari a 1 pixel tra le celle estremamente stretta. Una lattina di plug-in contatore delle cellule essere utilizzato quando l'etichettatura è inequivocabile e possa essere eseguita manualmente.
- Astrocitari rete area di misurazione
- Per misurare l'area della superficie delle reti, utilizzare il file di rilevamento utilizzando Image J.
- Utilizzare lo strumento di selezione poligono (click sinistro sul pulsante nella barra degli strumenti per selezionarlo) per tracciare un poligono che collega tutte le celle si trova nella periferia esterna della rete (Figura 4A). Click sinistro per iniziare l'analisi del poligono e fare clic con il pulsante destro per chiuderla.
Nota: Questo poligono viene definito come una regione di interesse (ROI) e la sua superficie sarà misurata per determinare l'area della superficie della rete. - Aprire la finestra di impostazione misura (Analyze | Impostare la misura) e selezionare l'opzione "Area". Aprire il gestore di ROI (analizzare | Strumenti | Manager di ROI) (Figura 4B). Quindi, aggiungere il poligono tracciato in ROI Manager facendo clic su 'Aggiungi ' (Figura 4B) ed eseguire la misurazione facendo clic su ' misura ' in gestione il ROI.
Nota: La misura dello spazio verrà visualizzata in una tabella ed essere espressa in pixel. Non dimenticate di convertire questo valore con il fattore di conversione per il microscopio utilizzato per ottenere un valore in μm2.
- Determinazione del vettore direzione principale
- Determinazione della cella con patch
- Aprire il file dello stack in ImageJFIJI e identificare la cella con patch in file dello stack basato su sua più forte intensità d'etichettatura (Figura 4). Aprire il file denominato "tabella di rilevamento" in un'applicazione di foglio di calcolo, quindi trovare il numero associato alla cella con patch e le sue coordinate corrispondenti.
- Se non è possibile determinare con precisione la cella con patch, circondano la zona dove è più denso nella rete imaged di cellule accoppiate, utilizzando lo strumento poligono nel ImageJFIJI, il deposito di biocitina e fare riferimento a questa posizione come quello della cellula patchata (Figura 4).
- Utilizzare il gestore di ROI (analizzare | Strumenti | Manager di ROI). Quindi, disegnare un ROI nella posizione cella patchato e aggiungerlo al ROI Manager (Vedi Figura 4B).
- Impostare una misura (Analyze | Impostare la misura) e selezionare l'opzione "Baricentro".
- In ROI Manager, fare clic su "Misura" per ottenere le coordinate del centroide dell'area tracciata. Utilizzare queste coordinate come punto di riferimento per questa specifica rete.
- Traduzione referenziale
- Calcolare le coordinate di ogni cella in riferimento l'Astrocita patchato utilizzando la seguente formula:

Con: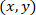 come le coordinate per una determinata cella;
come le coordinate per una determinata cella; 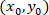 come le coordinate della cella con patch (o il punto referenziale della rete); e
come le coordinate della cella con patch (o il punto referenziale della rete); e 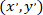 come le coordinate per una determinata cella nel nuovo referenziale.
come le coordinate per una determinata cella nel nuovo referenziale.
Nota: Esprimere le coordinate di ogni cella in riferimento l'Astrocita patchato è un passo importante nel calcolo vettori dall'Astrocita patchato. Fare attenzione quando si utilizza ImageJFIJI che il referenziale per qualsiasi immagine si trova nell'angolo superiore sinistro dell'immagine.
- Calcolare le coordinate di ogni cella in riferimento l'Astrocita patchato utilizzando la seguente formula:
- Determinazione del vettore principale di orientamento preferenziale
- Calcolare le coordinate del vettore principale di orientamento preferenziale con la seguente formula:
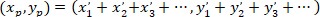
Con: (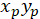 ) come le coordinate del vettore principale di orientamento preferenziale; e
) come le coordinate del vettore principale di orientamento preferenziale; e 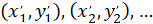 come le coordinate di ogni cella della rete ottenuta con la patch cell come referenziale.
come le coordinate di ogni cella della rete ottenuta con la patch cell come referenziale.
Nota: Per ogni cella della rete, un vettore è determinato rispetto alle coordinate dell'Astrocita patchato. Il principale vettore di orientamento preferenziale della rete è la somma di tutti questi vettori. - Dividere la lunghezza del vettore principale (fornito da delle coordinate ottenute da utilizzando la formula di cui sopra) per il numero di celle della rete meno uno (poiché le coordinate della cella con patch non sono incluse), per normalizzare i valori e abilitare i confronti tra reti. Una vista schematica di questa analisi è presentata nella Figura 5.
- Calcolare le coordinate del vettore principale di orientamento preferenziale con la seguente formula:
- Determinazione della cella con patch
- Posizionamento delle reti analizzati nel nucleo di interesse
- Allineamento di 20 volte e 4 X immagini
- Per determinare la posizione di ciascuna rete nel nucleo di interesse (NVsnpr), utilizzare l'immagine di X 4. Aprire l'immagine X 4 con un editor di immagini vettoriali.
- Selezionare l'immagine di X 4 e modificare le sue dimensioni moltiplicandolo per 5. La finestra di dimensione si trova nella parte destra della barra orizzontale superiore. Per esempio, per un'immagine di X 4 che è stata campionata a 800 x 800 pixel, modificare la risoluzione di campionamento a 4000 x 4000 pixel (per impostare l'unità di lavoro in pixel, vai a "Impostazioni documento" in scheda file: File | Impostazione del documento). Esportare il file in formato TIFF e nome che "4 X ridimensionata".
- Allineare l'angolo superiore sinistro dell'immagine con l'angolo inferiore sinistro del documento Adobe Illustrator.
Nota: Il software fornisce le coordinate di un punto referenziale nell'angolo inferiore sinistro. - Aprire l'immagine X 20 della rete. Utilizzare il ' binario ' o ' rilevamento file ' perché sono più facili da allineare. Poi, giocare con lo strumento opacità sul 'file binario ' dell'immagine X 20 per allinearlo con il re-sized 4x immagine.
Nota: Lo strumento opacità è nella barra degli strumenti orizzontale superiore. - Quando l'allineamento è, selezionare e tenere l'utensile di pipetta così appare lo strumento misura e selezionarlo nella barra di sinistra.
- Utilizzare lo strumento di misura, fare clic sull'angolo superiore sinistro dell'immagine X 20 per ottenere le coordinate del 20 X referenziale punto sul ridimensionata 4x immagine.
Nota: Queste coordinate, che sono indicate come i 20 X referenziale (20XR nella Figura 5), sarà utile per esprimere la posizione di ciascuna rete su un disegno schematico del nucleo.
- Normalizzazione del nucleo di interesse
Nota: Per riassumere i dati, viene utilizzata una normalizzazione del nucleo (NVsnpr) come un rettangolo. I passaggi sono descritti di seguito.- Aprire il file ridimensionato a 4x in ImageJFIJI e utilizzare lo strumento poligono per circondano il nucleo (NVsnpr). Utilizzare l'immagine con luce trasmessa, se i confini del nucleo non sono in grado di essere visto; in questo caso, ricordarsi di ridimensionarla prima.
- Aprire il gestore di ROI e aggiungere il ROI disegnato. "Impostare le misure", selezionare l'opzione ' Rectangle di delimitazione '.
Nota: L'opzione "Rettangolo di delimitazione" calcola il rettangolo più piccolo intorno al nucleo disegnato. - Fare clic su "Misura".
Nota: Viene visualizzata una tabella con BX e BY, le coordinate dell'angolo superiore sinistro del rettangolo: 'Rettangolo posizione ' e fornisce la larghezza e l'altezza. BX e BY sono le coordinate del rettangolo di delimitazione referenziale che si riferiscono a come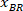 e
e 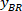 .
.
- Espressione di ogni posizione di rete nel nucleo normalizzato
- Esprimere le coordinate di ogni cella con 4 X referenziale (quadrato nero in Figura 5) utilizzando la seguente formula:

Dove: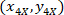 sono le coordinate di cella con il 4x referenziale;
sono le coordinate di cella con il 4x referenziale; 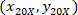 sono le coordinate di cella con 20 X referenziale (quadrato arancione nella Figura 5); e
sono le coordinate di cella con 20 X referenziale (quadrato arancione nella Figura 5); e 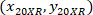 sono le coordinate del 20 X punto referenziale nell'immagine X 4 (20XR).
sono le coordinate del 20 X punto referenziale nell'immagine X 4 (20XR). - Esprimere le coordinate delle celle nel rettangolo di delimitazione referenziale (blu nella Figura 5) utilizzando la seguente formula:
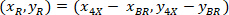
Dove: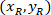 sono la cella coordinate nel rettangolo di delimitazione referenziale;
sono la cella coordinate nel rettangolo di delimitazione referenziale; 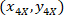 sono le coordinate di cella in 4 X referenziale; e
sono le coordinate di cella in 4 X referenziale; e  sono le coordinate del rettangolo di delimitazione referenziale nell'immagine X 4.
sono le coordinate del rettangolo di delimitazione referenziale nell'immagine X 4. - Trasformare le coordinate di cella nel rettangolo di delimitazione referenziale per la percentuale della larghezza e altezza del rettangolo di delimitazione, utilizzando la seguente formula:
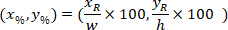
Dove: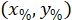 sono la cella coordinate nella percentuale della larghezza e altezza del rettangolo di delimitazione;
sono la cella coordinate nella percentuale della larghezza e altezza del rettangolo di delimitazione; 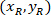 sono la cella coordinate nel rettangolo di delimitazione referenziale;
sono la cella coordinate nel rettangolo di delimitazione referenziale;  è la larghezza del rettangolo di delimitazione, misurata di sopra del protocollo; e
è la larghezza del rettangolo di delimitazione, misurata di sopra del protocollo; e 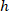 è l'altezza del rettangolo di delimitazione misurata di sopra del protocollo.
è l'altezza del rettangolo di delimitazione misurata di sopra del protocollo. - Per rappresentare tutte le reti sulla stessa figura, prendere in considerazione l'orientamento della fetta (lato destro o sinistro). Per standardizzare i dati, il referenziale viene applicata sul lato sinistro della fetta. Trasferire le coordinate di rete del lato destro al lato sinistro applicando la seguente formula solo alle coordinate x:
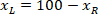
Dove: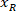 è la coordinata x sul lato destro della sezione; e
è la coordinata x sul lato destro della sezione; e 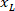 è la nuova coordinata x espressa nel referenziale sul lato sinistro della fetta.
è la nuova coordinata x espressa nel referenziale sul lato sinistro della fetta.
Nota: In alternativa, specchio l'immagine in ImageJFIJI prima dell'analisi (immagine | Trasformare | Rifletti orizzontalmente). - Per esprimere le coordinate del vettore principale di orientamento preferenziale, seguire la stessa procedura con le coordinate delle celle (passaggi 6.5.3.1 a 6.5.3.4).
Nota: L'espressione delle coordinate in percentuali permette una raccolta di dati come un unico appezzamento in cui il nucleo (NVsnpr) è stato progettato come un rettangolo.
- Esprimere le coordinate di ogni cella con 4 X referenziale (quadrato nero in Figura 5) utilizzando la seguente formula:
- Allineamento di 20 volte e 4 X immagini
- Studio della differenza angolare del principale vettore di direzione preferenziale
Nota: La differenza angolare di una rete astrocytic viene utilizzata per determinare se l'orientamento preferenziale è verso il centro del nucleo di interesse. Per calcolare la differenza angolare (α nella Figura 5), che è l'angolo tra il vettore principale di direzione preferenziale della rete (linea PD, rosso nella Figura 5) e la linea che collega P a C, applicare il teorema di Al-Kashi nel triangolo PDC (Vedi inserto di Figura 5 e Metodi supplementari).- In primo luogo, calcolare le lunghezze differenti usando l'applicazione del teorema di Pitagora in un cartesiani referenziale utilizzando la seguente equazione:
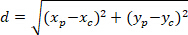
Dove: le coordinate di P e C sono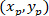 e
e  , rispettivamente, nel rettangolo di delimitazione referenziale (calcolato in precedenza).
, rispettivamente, nel rettangolo di delimitazione referenziale (calcolato in precedenza). - Determinare la differenza angolare in radianti utilizzando la seguente formula:
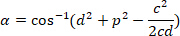
- Convertire la differenza angolare in gradi (ad es., con la funzione gradi nel software Excel).
- Compilare tutte le differenze angolari tracciando loro nei grafici a barre verticali (Figura 7 e 7D) e determinare se c'è un orientamento preferenziale delle reti astrocytic.
- In primo luogo, calcolare le lunghezze differenti usando l'applicazione del teorema di Pitagora in un cartesiani referenziale utilizzando la seguente equazione:
Risultati
Accoppiamento tra le cellule del cervello non è statico ma piuttosto dinamicamente regolato da molti fattori. I metodi descritti sono stati sviluppati per analizzare reti astrocytic ha rivelati in condizioni diverse e a capire la loro organizzazione in NVsnpr. Questi risultati sono stati già pubblicati1. Abbiamo effettuato biocitina riempimento dei astrocytes singolo nella parte dorsale del NVsnpr in tre diverse condizioni: a riposo (in condizioni di controllo in...
Discussione
Esiste un numero di metodi elettrofisiologici per valutare funzionale accoppiamento tra astrociti23,24. Tuttavia, questi metodi non forniscono informazioni circa la disposizione anatomica delle reti astrocytic. Una serie di studi hanno già dimostrato che "tintura - o tracciante-accoppiamento", come fatto qui, si verifica solo in una frazione di accoppiato le cellule che vengono rilevate da metodi elettrofisiologici25,
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è finanziato da istituti canadesi di ricerca sanitaria, Grant/premio numero: 14392.
Materiali
| Name | Company | Catalog Number | Comments |
| NaCl | Fisher Chemicals | S671-3 | |
| KCl | Fisher Chemicals | P217-500 | |
| KH2PO4 | Fisher Chemicals | P285-500 | |
| MgSO4 | Fisher Chemicals | M65-500 | |
| NaHCO3 | Fisher Chemicals | S233-500 | |
| C6H12O6 Dextrose anhydrous | Fisher Chemicals | D16-500 | |
| CaCl2 dihydrated | Sigma | C70-500 | |
| Sucrose | Sigma | S9378 | |
| D-gluconic acid potassium salt | Sigma | G45001 | |
| MgCl2 anhydrous | Sigma | M8266 | |
| HEPES | Sigma | H3375 | |
| EGTA | Sigma | E4378 | |
| ATPTris Salt | Sigma | A9062 | |
| GTPTris Salt | Sigma | G9002 | |
| Biocytin | Sigma | B4261 | |
| Carbenoxolone disodium salt | Sigma | C4790 | |
| avidin-biotin complex : ABC kit | Vestor laboratories | PK-4000 | |
| Streptavidine-alexa 594 | Molecular Probes | S11227 | |
| Triton | Fisher Chemicals | BP151-500 | |
| Xylene | Fisher Chemicals | X5-1 | |
| Aqueous mounting medium 1 : Fluoromount-G | SouthernBiotech | 0100-01 | |
| Toluen-based synthetic resin mounting medium : Permount | Fisher Chemicals | SP15-100 | |
| Slide Drying Bench | Fisherbrand | 11-474-470 | |
| Vibratome | Leica | VT 1000S | |
| Microscope cover glass | Fisherbrand | 12-544A | |
| Microscope slide ColorFrost | Fisherbrand | 12-550-413 | |
| PFA | Fisherchemicals | 04042-500 | |
| Olympus FluoView FV 1000 Confocal microscope | Olympus | ||
| 40X water-immersion lens | Olympus | LUMPLFLN40XW | |
| 20X water-immersion lens | Olympus | XLUMPLFL20XW | |
| 4X water-immersion lens | Olympus | XLFLUOR4X/340 | |
| Micropipette puller | Sutter Instrument | P97 | |
| Micromanipulator | Sutter Instrument | MP 225 | |
| Camera CCD | Sony | CX-ST50 | |
| Black and white monitor | Sony | SSM-125 | |
| Digidata | Molecular devices | 1322A | |
| Patch Clamp amplifier | Axon instrument | Mulitclamp 700A | |
| Electrophysiology acquisition software | Molecular devices | pClamp 8 | |
| Electrophysiology analysis software | Molecular devices | Clampfit 8 | |
| Imaging analysis software | ImageJFIJI | Open source software. FIJI version including plug in package. | |
| Vector image editor | Adobe | Illustrator CS4 | |
| Spreadsheet application | Microsoft Office | Excel 2010 |
Riferimenti
- Condamine, S., Lavoie, R., Verdier, D., Kolta, A. Functional rhythmogenic domains defined by astrocytic networks in the trigeminal main sensory nucleus. Glia. 66 (2), 311-326 (2018).
- Verkhratsky, A., Orkand, R. K., Kettenmann, H. Glial calcium: homeostasis and signaling function. Physiological Review. 78 (1), 99-141 (1998).
- Christensen, R. K., Petersen, A. V., Perrier, J. F. How do glial cells contribute to motor control?. Current Pharmaceutical Design. 19 (24), 4385-4399 (2013).
- Verkhratsky, A., Steinhauser, C. Ion channels in glial cells. Brain Research Review. 32 (2-3), 380-412 (2000).
- Harada, K., Kamiya, T., Tsuboi, T. Gliotransmitter Release from Astrocytes: Functional, Developmental, and Pathological Implications in the Brain. Frontiers Neuroscience. 9, 499 (2015).
- Montero, T. D., Orellana, J. A. Hemichannels: new pathways for gliotransmitter release. Neuroscience. 286, 45-59 (2015).
- Araque, A., et al. Gliotransmitters travel in time and space. Neuron. 81 (4), 728-739 (2014).
- Augustin, V., et al. Functional anisotropic panglial networks in the lateral superior olive. Glia. 64 (11), 1892-1911 (2016).
- Houades, V., Koulakoff, A., Ezan, P., Seif, I., Giaume, C. Gap junction-mediated astrocytic networks in the mouse barrel cortex. Journal of Neuroscience. 28 (20), 5207-5217 (2008).
- Roux, L., Benchenane, K., Rothstein, J. D., Bonvento, G., Giaume, C. Plasticity of astroglial networks in olfactory glomeruli. Proceedings of the National Academy of Science of the United State of America. 108 (45), 18442-18446 (2011).
- Morquette, P., et al. An astrocyte-dependent mechanism for neuronal rhythmogenesis. Nature Neuroscience. 18 (6), 844-854 (2015).
- Brocard, F., Verdier, D., Arsenault, I., Lund, J. P., Kolta, A. Emergence of intrinsic bursting in trigeminal sensory neurons parallels the acquisition of mastication in weanling rats. Journal of Neurophysiology. 96 (5), 2410-2424 (2006).
- Anders, S., et al. Spatial properties of astrocyte gap junction coupling in the rat hippocampus. Philosophical Transactions of the Royal Society of London. Series B, Biological Science. 369 (1654), (2014).
- Houades, V., et al. Shapes of astrocyte networks in the juvenile brain. Neuron Glia Biology. 2 (1), 3-14 (2006).
- Rouach, N., Koulakoff, A., Abudara, V., Willecke, K., Giaume, C. Astroglial metabolic networks sustain hippocampal synaptic transmission. Science. 322 (5907), 1551-1555 (2008).
- Claus, L., et al. Barreloid Borders and Neuronal Activity Shape Panglial Gap Junction-Coupled Networks in the Mouse Thalamus. Cerebral Cortex. 28 (1), 213-222 (2018).
- Cameron, M. A., et al. Prolonged Incubation of Acute Neuronal Tissue for Electrophysiology and Calcium-imaging. Journal of Visualized Experiments. (120), (2017).
- Kafitz, K. W., Meier, S. D., Stephan, J., Rose, C. R. Developmental profile and properties of sulforhodamine 101--Labeled glial cells in acute brain slices of rat hippocampus. Journal of Neuroscience Methods. 169 (1), 84-92 (2008).
- Neher, E. Correction for liquid junction potentials in patch clamp experiments. Methods in Enzymology. , 123-131 (1992).
- Giaume, C., Leybaert, L., Naus, C. C., Saez, J. C. Connexin and pannexin hemichannels in brain glial cells: properties, pharmacology, and roles. Frontiers in Pharmacology. 4, 88 (2013).
- Torres, A., et al. Extracellular Ca(2)(+) acts as a mediator of communication from neurons to glia. Science Signaling. 5 (208), ra8 (2012).
- Ye, Z. C., Wyeth, M. S., Baltan-Tekkok, S., Ransom, B. R. Functional hemichannels in astrocytes: a novel mechanism of glutamate release. Journal of Neuroscience. 23 (9), 3588-3596 (2003).
- Ma, B., et al. Gap junction coupling confers isopotentiality on astrocyte syncytium. Glia. 64 (2), 214-226 (2016).
- Meme, W., Vandecasteele, M., Giaume, C., Venance, L. Electrical coupling between hippocampal astrocytes in rat brain slices. Neuroscience Research. 63 (4), 236-243 (2009).
- Ransom, B. R., Kettenmann, H. Electrical coupling, without dye coupling, between mammalian astrocytes and oligodendrocytes in cell culture. Glia. 3 (4), 258-266 (1990).
- Audesirk, G., Audesirk, T., Bowsher, P. Variability and frequent failure of lucifer yellow to pass between two electrically coupled neurons in Lymnaea stagnalis. Journal of Neurobiology. 13 (4), 369-375 (1982).
- Ewadinger, N., Syed, N., Lukowiak, K., Bulloch, A. Differential Tracer Coupling between Pairs of Identified Neurones of the Mollusc Lymnaea Stagnalis. Journal of Experimental Biology. 192 (1), 291-297 (1994).
- Griemsmann, S., et al. Characterization of Panglial Gap Junction Networks in the Thalamus, Neocortex, and Hippocampus Reveals a Unique Population of Glial Cells. Cerebral Cortex. 25 (10), 3420-3433 (2015).
- Kuwajima, T., et al. ClearT: a detergent- and solvent-free clearing method for neuronal and non-neuronal tissue. Development. 140 (6), 1364-1368 (2013).
- Gourine, A. V., et al. Astrocytes control breathing through pH-dependent release of ATP. Science. 329 (5991), 571-575 (2010).
- Forsberg, D., Ringstedt, T., Herlenius, E. Astrocytes release prostaglandin E2 to modify respiratory network activity. eLife. 6, (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon