Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Analyse der Größe, Form und Direktionalität von Netzwerken von gekoppelten Astrozyten
In diesem Artikel
Zusammenfassung
Hier präsentieren wir ein Protokoll, um die Organisation der astrocytic Netzwerke zu beurteilen. Das beschriebene Verfahren minimiert Vorspannung um beschreibende Maßnahmen dieser Netze z. B. Zellzahl, Größe, Umgebung und Position innerhalb eines Kerns zu bieten. Anisotropie ist mit einer vektoriellen Analyse bewertet.
Zusammenfassung
Es ist immer deutlicher geworden, dass Astrozyten neuronale Funktion nicht nur auf der synaptischen und einzellige Ebene, sondern auch auf der Netzwerkebene modulieren. Astrozyten sind stark miteinander verbunden durch Gap Junctions und Kupplung durch diese Kreuzungen ist dynamisch und stark reguliert. Eine aufstrebende Konzept ist, dass astrocytic Funktionen spezialisiert und an die Funktionen der neuronalen Schaltung angepasst, mit dem sie verbunden sind. Daher sind Methoden zur Messung verschiedener Parameter der astrocytic Netze, die Regeln für ihre Kommunikation und Kupplung besser zu beschreiben und um ihre Funktionen zu verstehen benötigt.
Hier, unter Verwendung der Bildanalyse-Software (zB., ImageJFIJI), wir beschreiben eine Methode zur Analyse konfokale Bilder astrocytic Netzwerke von Farbstoff-Kupplung offenbart. Diese Methoden ermöglichen (1) eine automatisierte und unvoreingenommene Erkennung von markierten Zellen (2) Berechnung der Größe des Netzes, (3) Berechnung der bevorzugte Ausrichtung der Farbstoff innerhalb des Netzwerks zu verbreiten, und (4) Neupositionierung des Netzes innerhalb des Bereichs des Interesses .
Diese Analyse kann zur astrocytic Netzwerke eines bestimmten Gebietes zu charakterisieren, Netzwerke verschiedener Bereiche, die verschiedene Funktionen zugeordnet, oder vergleichen Netzwerke unter verschiedenen Bedingungen, die unterschiedliche Auswirkungen auf Kupplung haben gewonnen. Diese Beobachtungen führen zu wichtigen funktionalen Überlegungen. Zum Beispiel analysieren wir die astrocytic Netzwerken aus einem trigeminus Kern, wo wir zuvor gezeigt haben, daß astrocytic Kupplung entscheidend für die Fähigkeit von Nervenzellen, ihre impulsmuster von Tonika auf rhythmische platzen1wechseln. Durch die Messung der Größe, Entbindung und bevorzugte Ausrichtung der astrocytic Netzwerke in diesem Kern, können wir Hypothesen über funktionelle Domänen erstellen, die sie umschreiben. Mehrere Studien deuten darauf hin, dass mehrere andere Hirnareale, einschließlich Barrel Cortex, seitliche überlegene Olive, olfaktorische Glomeruli und sensorischen Kerne in den Thalamus und visuellen Kortex, um nur einige zu nennen eine ähnliche Analyse profitieren können.
Einleitung
Viele Studien haben beschrieben, wie der Neuron-Astrozyten Dialog eine Ebene der subzellulären oder synaptic in neuronalen Funktionen und synaptische Übertragung auswirken kann. Es ist allgemein bekannt, dass die Astrozyten empfindlich zu den umliegenden neuronalen Aktivität; in der Tat haben sie Rezeptoren für viele Neurotransmitter wie Glutamat, GABA, Acetylcholin und ATP (siehe zuvor veröffentlichten Bewertungen2,3,4). Im Gegenzug verarbeitet astrocytic Ensheath synaptischen Elemente und Einfluss neuronaler Aktivität dort und an extrasynaptic Standorten durch Regulierung der extrazellulären Ionischen Homöostase und Freigabe mehrere Faktoren oder Transmitter wie Glutamat, D-Serin und ATP 5 , 6 , 7.
Die Idee, dass Astrozyten auch neuronale Funktion auf der Netzwerkebene modulieren können hat, Beweise gezeigt, dass astrocytic Kupplung räumlich geregelt ist und neuronalen Segmentierung in Bereichen zeichnet sich durch eine klare anatomische entspricht Abschottung (wie Bereiche mit sensorischen Darstellungen), darauf hinweist, dass Astrozyten koppeln werden an andere Astrozyten dienen die gleiche Funktion anstatt nur diejenigen, die ganz in der Nähe sind. In der seitlichen überlegene Olive orientieren sich zum Beispiel am meisten astrocytic Netzwerke orthogonal zu der tonotopen Achse8, während in den Lauf Kortex oder Olfactoty Glomeruli, Kommunikation zwischen Astrozyten in Fässern oder Glomeruli viel stärker ist und schwächeren zwischen benachbarten,9,10. In beiden Fällen orientieren sich die astrocytic Netzwerke an das Zentrum der Nierenbeckenentzündung oder Lauf9,10.
Wir zeigten kürzlich, dass astrocytic Aktivität neuronale feuern moduliert durch eine Verringerung der Konzentration der extrazellulären Ca2 + ([Ca2 +]e), vermutlich durch die Freisetzung von S100β, Ca2 +-bindende Protein11. Dieser Effekt, der in einer Population von trigeminus Rhythmogenic Neuronen im dorsalen Teil des Trigeminus sensorischen Main Kern (NVsnpr zeigte, gedacht, um eine wichtige Rolle bei der Erzeugung von physiologischen Bewegungen), ergibt sich daraus, dass rhythmische feuern in diesen Neuronen hängt eine persistente Na+ aktuelle, die durch die Verluste von [Ca2 +]e11,12gefördert wird. Rhythmische feuern in diese Neuronen kann "physiologisch" durch Stimulation der Eingaben oder künstliche Verringerung der [Ca2 +]eausgelöst. Weiter zeigten wir, dass astrocytic Kupplung für neuronale rhythmische feuern1erforderlich war. Dies erhöht die Möglichkeit, dass astrocytic Netzwerke umschriebene Funktionsbereiche bilden können, wo neuronaler Aktivität synchronisiert und koordiniert werden können. Um diese Hypothese zu beurteilen, mussten wir zuerst eine Methode um die Organisation dieser Netze innerhalb NVsnpr rigoros zu dokumentieren zu entwickeln.
Frühere Studien zu astrocytic Netzwerke haben vor allem das Ausmaß der Kupplung in Bezug auf die Anzahl von Zellen und die Dichte und die Fläche beschrieben. Versuche, die Form der astrocytic Netzwerke und die Richtung der Farbstoff-Kupplung zu bewerten waren meist durch einen Vergleich der Größe der Netze auf zwei Achsen (X und y) im Fass Kortex9, Hippocampus13,14, durchgeführt 15, Barreloid Felder der Thalamus16, seitliche überlegene Olive8, olfaktorische Glomeruli10und Kortex14. Die hier beschriebenen Methoden ermöglichen unvoreingenommene Grafen von markierten Zellen in einem Netzwerk und eine Abschätzung des Bereichs, den sie abdecken. Wir haben auch Werkzeuge, die bevorzugte Ausrichtung der Kupplung innerhalb eines Netzwerkes zu definieren und zu prüfen, ob die bevorzugte Ausrichtung in Richtung der Mitte des Kerns oder in eine andere Richtung entwickelt. Im Vergleich zu bisher verwendeten Methoden bietet dieses Protokolls ein Mittel zur Beschreibung der Organisation und Ausrichtung der astrocytic Netze in Strukturen wie der dorsalen trigeminus wichtigsten sensorischen Kern, die nicht über eine klare anatomische bekannt Abschottung. In den oben genannten Studien, die Netzwerk-Ausrichtung wird beschrieben als eine Beziehung auf die Form der Struktur selbst die bereits dokumentiert ist (zB., die Barreloid in den Thalamus, Fässer in der Hirnrinde "layers" im Hippocampus und Cortex, Glomeruli in die Riechkolben usw.). Darüber hinaus ermöglicht vektoriellen Analyse für Vergleiche der Kupplung Orientierungen unter verschiedenen Bedingungen aufgedeckt. Um zu analysieren, ob diese Parameter entsprechend der Position des Netzes innerhalb des Zellkerns geändert, entwickelten wir auch eine Methode, um jedes Netzwerk in Bezug auf die Grenzen des Kerns zu ersetzen. Diese Werkzeuge können leicht auf andere Bereiche für Ermittlungsbehörden Netzwerke von gekoppelten Zellen angepasst werden.
Protokoll
Alle Verfahren blieb durch die Canadian Institutes of Health Research-Regeln und wurden von der Université de Montréal, Tierpflege und Use Committee genehmigt.
1. Vorbereitung der Ratte Gehirnscheiben
- Bereiten Sie 1 L einer Saccharose-basierten Lösung (Tabelle 1) und 1 L standard künstliches Gehirn-Rückenmark-Flüssigkeit (ACFS) (Tabelle 2).
- Blase die Saccharose-basierte Lösung mit einer Mischung aus 95 % O2 und 5 % CO2 (Carbogen) für 30 min vor der Platzierung bei-80 ° C für ca. 30 min, bis die Lösung kalt, aber nicht vollständig gefroren ist. Verwenden Sie dieses eiskalte Saccharose als Zuschnitt Puffer für das Schneiden des Gehirns. Halten Sie es auf Eis, nachdem es aus dem Gefrierfach entfernt wurde.
- Blase der ACFS mit Carbogen durch das gesamte Experiment. Verwenden Sie diese Lösung für die Lagerung der Scheibe sowie die perfusing Puffer in dem Patch-Spann- und Biocytin-Füllung der Astrozyten ausgeführt werden. Bereiten Sie eine Stück Erholung hält die Kammer die Gehirnscheiben hinterlegen, sobald sie geschnitten werden.
Hinweis: Die aufnahmekammer Stück Erholung nach Maß sein könnte und besteht im Wesentlichen aus einem kleinen Brunnen mit einem Netz an der Unterseite ausgesetzt in einem größeren gut, die ACFS voller ist, in denen ein Rohr eingefügt wird, um Carbogen von unten zu bringen. - 15 bis 21 Tage alte Sprague-Dawley Ratten geschlechtsspezifische oder Stamm unvoreingenommen zu verwenden. Betäuben Sie das Tier mit Isofluran (1 mL von Isofluran in einer Narkose Induktion Kammer). Prüfen Sie die Tiefe der Narkose durch sanft Kneifen der Hinterpfote oder Endstück des Tieres.
- Enthaupten Sie die Ratte mit einer Guillotine, geschnitten Sie der Schädel mit einer Schere und entfernen Sie schnell das Gehirn aus dem Schädel mit einem flachen Spatel.
- Tauchen Sie das Gehirn in die eiskalte Saccharose-basierte Lösung für über 30 s und Transfer (mit Lösung) damit ein Petri dish. Mit einer Rasierklinge entfernen Sie die Teile, die Front- und Seitenzahnbereich in das Gebiet um geschnitten werden, wenn die Querebene schneiden zerschneiden.
- Kleben Sie die restlichen Block des Gehirngewebes rostral seinerseits und im Saccharose-basierte Medium mit einem Vibratome Gehirnscheiben (350 µm dick) schneiden. Übertragen Sie anschließend die gesammelten Scheiben in eine Erholung hält Kammer gefüllt mit Carbogenated ACFS bei Raumtemperatur (RT), bis sie bereit sind, verwendet werden (mindestens 1 Stunde für die Wiederherstellung zu ermöglichen).
Hinweis: Aktuelle Entwicklungen im Bereich ermöglichen längere Inkubationszeit bis zu 24 h Gehirnscheiben in in-vitro- Bedingungen17.
2. Sulforhodamine 101 (SR 101) Kennzeichnung der Astrozyten
- Ein Wasserbad auf 34 ° C vorheizen und zwei Stück Inkubation Kammern entgegenbringen. Füllen eines Slice Inkubation Kammern mit einer Lösung aus ACFS 1 μM mit SR-101, und füllen Sie die anderen nur mit ACFS.
- Inkubieren Sie die Scheiben in der Inkubation Kammer mit 1 µM SR-101 für 20 min und übertragen Sie dann in der zweiten Inkubation Kammer um zu spülen überschüssige SR-101 aus dem Gewebe. Lassen Sie es 20 min inkubieren oder mehr bei 34 ° C, halten Sie die Inkubation-Kammer, die die Scheiben bei RT bis sie sind18benötigt.
(3) Astrozyten Patchen und Biocytin Füllung
- Wählen Sie ein Stück und legen Sie sie in der Aufnahme Kammer des Mikroskops. Patch-Astrozyten, verwenden Sie Elektroden mit einem Widerstand von 4-6MΩ mit einer Kalium-Gluconat-basierten Lösung gefüllt (siehe Tabelle 3).
- Unter visuelle Führung und mit einem Mikromanipulator direkte Aufnahme Elektrode in Richtung einer SR 101 beschriftet Astrozyten wie in Abbildung 1dargestellt. Vermeiden Sie Zellen an der Oberfläche des Segments sind eher beschädigt werden oder verloren haben Verbindungen zu benachbarten Zellen.
- Zur Vermeidung von Leckagen von Biocytin im Gewebe minimieren den positiven-Luftdruck hinzugefügt, um die Patch-Pipette und wenden Sie es nur, wenn es in der Nähe der Astrozyten, die gepatcht werden (0,1-0,4 mL in einer Spritze 1 ml).
- Vor dem Patchen, stellen Sie den Offset von der Pipette und seine Kapazität. Korrigieren Sie für flüssige Verzweigung Potenziale, so genau und präzise Spannung Befehle sind entscheidend für die Experimente19.
- Bewegen Sie die Pipette nahe genug, um die Astrozyten, Depression, die durch den positiv-Luftdruck zu beobachten. Dann entfernen Sie den positiven-Luftdruck und langsam einige negativen Druck. Klemmen Sie die Astrozyten zu-70 mV erreicht die Dichtung 100 MΩ. Warten Sie, bis die Dichtung 1-3 GΩ erreicht. Unterdruck bis Einbruch in die Zelle gelten weiterhin. Seien Sie vorsichtig bei der Anwendung von Unterdruck, da Astrozyten sehr zerbrechlich sind.
- Die elektrophysiologischen Eigenschaften der gepatchten Zelle zu beurteilen. In Voltage-Clamp Modus durchführen eine ganze Zelle Strom-Spannungs-Protokoll mit einer Rampe Spannung Befehl von 600 ms Dauer von-120 bis + 110 mV. Führen Sie im Strom-Clamp-Modus ein Schritt IV-Protokoll, in dem die Injektion von 1000 ms aktuelle Schritte von 100 pA von-1 bis 1 nA, die Sampling-Rate der gesamten Zelle Aufnahmen 10 kHz ist.
Hinweis: Astrozyten zeigen einen lineare Strom-Spannungs-Profil ohne jegliche Korrektur (siehe Abbildung 1 b) und kein Aktionspotential abfeuern auf Membran-Depolarisation (Abbildung 1). Die Membran-Ruhepotential (RMP) sollte stabil und nicht positiv - 60mV sein. In einigen Bereichen des Gehirns ist das RMP von Astrozyten mehr hyperpolarisierten als in der NVsnpr. - Erlauben Sie die Biocytin, innerhalb der Astrozyten für 30 min zu verbreiten, während der Durchführung eines Schritt IV Protokoll alle 5 min..
- Einfahren Sie und lösen Sie die Patch-Pipette vorsichtig ohne Beschädigung der gepatchten Astrozyten und sofort notieren Sie des Versatzes der Pipette vor der Einnahme von es aus der Aufnahme-Kammer. Subtrahieren Sie diesen Wert aus der Membran möglichen Aufnahmen.
- Lassen Sie das Gehirn Slice in der Aufnahme-Kammer für ein Minimum von 15 min (zusätzlich 30 min für die Injektion) ruhen, um Ausbreitung des Tracers aus der gepatchten Astrozyten im gesamten Netzwerk gekoppelten Zellen zu ermöglichen.
Hinweis: Um ein Netzwerk von gekoppelten Zellen sichtbar zu machen, sollte nur eine einzelne Zelle im Zellkern von Interesse gepatcht werden. Wenn mehrere Versuche erforderlich, um einen erfolgreichen Patch zu erreichen sind, verwerfen des Falls und versuchen, auf der kontralateralen Seite oder in einem anderen Gehirn-Scheibe zu patchen. - Machen Sie eine Markierung oder Schnitt in das Gewebe zu erkennen, welche Seite der Scheibe nach oben zeigt, und übertragen Sie die Scheibe zuerst auf eine Petrischale mit normalen ACFS, dann in eine Lösung von 4 % Paraformaldehyd (PFA). Über Nacht inkubieren Sie die Scheibe bei 4 ° C.
Achtung: PFA ist extrem schädlich. Verwenden Sie Schutzausrüstung in einem gelüfteten Abzug. Stellen Sie sicher, dass alle Materialien (Pinsel, Rohre, etc.), die in Kontakt mit PFA wurden die Erholung hält Kammer, Inkubation Kammer oder andere Materialien, die für die Aufnahme der frischen Gewebe nicht kontaktieren.
4. Biocytin Offenbarung
- Offenbarung mit fluoreszierenden streptavidine
- Das Gehirn waschen Scheiben 2 x 10 min in 0,1 M Phosphat-Puffer Kochsalzlösung (PBS) bei RT Dann zeigen Sie die Biocytin durch Inkubation der Gehirnscheiben mit Streptavidine konjugiert zu einem Fluorophor bei 1/200 Verdünnung mit PBS-Puffer mit 4 % nicht-ionische Reinigungsmittel für 4 h bei RT
- Waschen Sie die Gehirnscheiben 3 x 10 min in 0,1 M PBS bei RT und montieren Sie die Abschnitte auf Glas-Objektträger mit einer wässrigen Eindeckmedium. Legen Sie den Seiten, die nach oben, Deckglas injiziert wurden die Folien und versiegeln sie mit Nagellack.
- Offenbarung mit DAB
Hinweis: DAB (3,3-Diaminobenzidine) Offenbarung kann verwendet werden, wenn Fluoreszenz verwendet werden kann. In diesem Fall sollte nur eine Offenbarung Methode für Vergleiche eingesetzt werden.- Waschen die Gehirnscheiben 3 x 10 min in PBS bei RT inkubieren der Scheiben in PBS + H2O2 0,5 % für 1 Stunde bei RT
- Spülen Sie die Scheiben 3 x 10 min in PBS bei RT Dann inkubieren Sie die Scheiben in einen Avidin-Biotin Komplex Färbelösung bestehend aus PBS und 0,1 % nicht-ionische Waschmittel + Avidin-Biotin Komplex Peroxidase standard Färbung Kit Reagenzien auf 1/100 für 24 Stunden bei RT
- Spülen Sie die Scheiben 3 x 10 min in PBS bei RT, brüten sie in einer Lösung von PBS + 0,05 % für 20 min bei RT, DAB und übertragen Sie sie in eine Lösung von PBS + DAB 0,05 % + H2O2 0,5 %.
Achtung: DAB ist extrem schädlich. Verwenden Sie Schutzausrüstung in einem gelüfteten Abzug. Die Farbreaktion wird gestoppt, wenn die Scheiben mit PBS-Puffer übertragen werden. - Spülen Sie die Scheiben 5 x 10 min in PBS bei RT, montieren Sie die Abschnitte auf Objektträgern (Gesicht der Seiten, die nach oben injiziert wurden) und auf einer Folie Trocknen Bank über Nacht bei 34 ° c trocknen lassen
- Tauchen Sie den Objektträger 1 min. in mehreren Alkohol Bädern zu 70 %, 95 % und 100 % und am Ende mit einem Bad von Xylol für 1 min.
- Montieren Sie die Glas-Objektträger mit einem Toluol-basierte Kunstharz Montage Medium. Die Folien Deckgläsern aufsetzen und mit Nagellack versiegeln.
5. Netzwerk-Imaging
- Visualisieren der astrocytic Netze mit einem Scan confocal Mikroskop ausgestattet mit 20 X und 4 X Objektive (oder eine entsprechende Ziel, den gesamten Kern zu visualisieren, wo befindet sich das Netzwerk) und ein Laser, der Fluorophor zu erkennen (in diesem Fall ist Alexa-594 verwendet).
- Verwenden Sie die 20 X Vergrößerung eine Z-Stapel des Netzes der markierten Zellen. Verwenden Sie eine Auflösung von 800 x 800 Pixel und scan-Geschwindigkeit von 12,5 μs/Pixel.
Hinweis: Um das gesamte Netzwerk Bild, mehrere Stacks sind in der Regel erforderlich, und die Anzahl der Stapel für das jeweilige Netzwerk angepasst werden muss. Die Auflösung und Scan-Geschwindigkeit kann geändert werden, aber achten Sie darauf, die gleichen Einstellungen verwenden, um alle Daten zu Bild. - Verwenden Sie die 4 X Vergrößerung, Bilder des Netzwerks und des Interessenbereichs zu nehmen.
Hinweis: Die 4 X Bildgebung dient zur Bestimmung der Netzwerkposition innerhalb des Zellkerns von Interesse. Das Bild immer des gleichen Feldes im Durchlicht. Dieses Bild wird nützlich sein, wenn Sie nicht die Grenze des Kerns in die konfokale Fluoreszenz Bild ermitteln können.
6. die Bildanalyse
- Aufbereitung der Daten
- Verwenden Sie die Software ImageJFIJI (Laden Sie es auf https://fiji.sc/). Öffnen Sie die Datei und klicken Sie auf "OK" im Fenster "Bio-Formate-Import-Optionen".
- Um eine Z-Stapel neu zu definieren, die nur die optische Scheiben benötigt für den letzten Z-Stapel (Abbildung 2A) enthalten soll, klicken Sie auf den "Stack" Knopf in der Werkzeugleiste (zu finden, wählen zuerst Stk | Z-Projekt). Wählen Sie "Max Intensität" in den Projektionstyp festlegen (Abbildung 2A). Speichern Sie die Datei und nennen Sie es "Stapel-Datei".
- Wenn die bildgebende Datei mehrere Kanäle enthält, teilen Sie es um nur den Kanal mit der astrocytic Netzwerk-Bildgebung zu sparen (Bild | Farbe | Kanäle aufgeteilt).
- Überprüfen Sie die Pixeleinstellungen des Bildes [Bild | Eigenschaften | Pixel (mit "1" für Pixel Dimension)].
- Verwenden Sie das Tool subtrahieren Hintergrund (Prozess | Subtrahieren Sie Hintergrund), den Hintergrund der Biocytin Kennzeichnung zu entfernen. Verwenden Sie die Vorschau-Funktion, den rollenden Kugel Radius festzulegen, die in der Regel auf 50 Pixel (Abb. 2 b) festgelegt ist.
- Verwenden Sie das Werkzeug entfernen Ausreißer (Prozess | Lärm | Entfernen Sie Ausreißer) wenn es nach dem subtrahieren Hintergrund Schritt erforderlich ist. Wählen Sie "Bright" in der Einstellung "Die Ausreißer" (Abbildung 2). Verwenden Sie die Vorschau-Funktion, um den Radius und Schwellenwert festzulegen. Seien Sie vorsichtig mit diesem Tool, da es die Daten verwischen kann, wie in Abb. 2Ddargestellt.
Hinweis: Dieses Tool entfernt die kleinen Flecken verursacht unspezifische Ablagerung von Streptavidine (dargestellt durch die weißen Pfeile in Abbildung 2 b und 2 C vor und nach der Behandlung, beziehungsweise). - Passen Sie den Schwellenwert (Bild | Anpassen | Schwelle). Wählen Sie "Standard" und "B & W" Modus (Abb. 2E). Klicken Sie auf "Anwenden".
Hinweis: Auto-Einstellung kann verwendet werden, aber manuelle Anpassung mit zwei Schieberegler wird bevorzugt. Ziel dieses Schrittes ist es, Lärm bis zum Maximum zu reduzieren, ohne alle markierten Zellen. - Konvertieren Sie das Bild in ein binäres Bild mit dem binären Prozess (Prozess | Binäre | Stellen Sie binär) wie in Abbildung 2Fgezeigt. Speichern Sie die Datei als TIFF-Datei, und nennen Sie es "binäre Datei".
- Zellzählung
- Überprüfen Sie die Einstellung für die Messfunktion (Analyze | Set-Messung). Wählen Sie die Option "Schwerpunkt".
- Verwenden Sie das Tool "Analysieren Partikel" auf die "binäre Datei" (Abbildung 3A) produziert im vorherigen Schritt (Analyze | Analysieren Sie Teilchen) (Abbildung 3 auf der linken Seite). Wählen Sie "Umrisse" in die "Show" Einstellung (Abbildung 3). Dies erzeugt eine neue Datei, die das Ergebnis der Erkennung (Abb. 3 b) zeigt.
- Spielen Sie mit den Parametern: Größe (nur Zellen zu erkennen, verwenden Sie Werte zwischen 30 und 6000) und Zirkularität (um zu bestimmen, ein Intervall zwischen 0 und 1, wobei "1" definiert einen perfekten Kreis und "0" eine zufällige Form), die Erkennung (Abbildung 3, Links Teil) zu verfeinern. Führen Sie die Erkennung durch Klicken auf "OK".
Hinweis: Zwei Tabellen erscheinen nach der Aufdeckung: (1) eine Tabelle mit dem Titel "Zusammenfassung", die die Anzahl der gefundenen Zellen liefert, und (2) eine Tabelle mit dem Titel "Ergebnisse" (Abbildung 3, rechten Teil), bietet die x und y-Koordinaten der einzelnen Zellen. - Kopieren Sie die Werte und fügen Sie sie in ein Tabellenkalkulationsprogramm. Speichern Sie diese Tabelle unter dem Namen "Erkennung Tisch". Eine Datei mit einem Grundstück von erkannten Zellen erscheinen auch (Abb. 3 b). Speichern Sie diese Datei als TIFF-Datei unter dem Namen "Erkennung File".
- Wenn eine Gruppe von 2 oder mehr markierten Zellen im Netzwerk erkannt werden, da eine einzelne Zelle mit dem Analyse-Partikel-Tool weil sie sind zu nah beieinander, verwenden Sie das Tool Wasserscheide (Prozess | Binäre | Wasserscheide) auf das binäre Bild vor der Anwendung die Analyze-Partikel-tool, und wiederholen Sie die Analyze-Partikel-Schritte.
Hinweis: Die Wasserscheide-Tool erstellt eine Abgrenzung von 1 Pixel breit zwischen den extrem engen Zellen. Eine Zelle Zähler plug-in kann verwendet werden, wenn die Kennzeichnung ist eindeutig und kann manuell durchgeführt werden.
- Messfläche astrocytic Netzwerk
- Um die Fläche der Netze zu messen, verwenden Sie die Erkennung-Datei mit Bild J.
- Verwenden der Polygon-Auswahl-Werkzeug (linker Mausklick auf die Schaltfläche in der Symbolleiste zur Auswahl) zu verfolgen ein Polygon, das alle Zellen verbindet befindet sich in der äußeren Peripherie des Netzes (Abb. 4A). Links klicken Sie, um die Verfolgung des Polygons zu beginnen, und klicken Sie rechts um ihn zu schließen.
Hinweis: Dieses Polygon wird definiert als einer Region of Interest (ROI) und seine Oberfläche wird gemessen werden, um die Fläche des Netzwerks zu bestimmen. - Öffnen Sie das Fenster Set Messung (Analyze | Messung eingestellt) und wählen Sie die Option "Bereich". Öffnen Sie den ROI-Manager (analysieren | Werkzeuge | ROI-Manager) (Abbildung 4 b). Dann fügen Sie das vektorisierte Polygon im ROI-Manager durch Klicken auf "Hinzufügen" (Abbildung 4 b) und führen Sie die Messung durch Klicken auf "Maß '' im ROI-Manager.
Hinweis: Die Flächenmessung wird in einer Tabelle angezeigt und werden in Pixel angegeben. Vergessen Sie nicht, diesen Wert mit der Umrechnungsfaktor für das Mikroskop verwendet, um einen Wert in μm2zu konvertieren.
- Bestimmung der wichtigsten Richtungsvektor
- Bestimmung der gepatchten Zelle
- Öffnen Sie die Stapel-Datei in ImageJFIJI und identifizieren Sie die gepatchte Zelle in der Stapel-Datei basierend auf seiner stärkeren Kennzeichnung Intensität (Abbildung 4). Dann öffnen Sie die Datei mit dem Namen "Erkennung Tisch" in einer Tabellenkalkulationsanwendung und finden Sie die Nummer auf die gepatchte Zelle und die entsprechenden Koordinaten.
- Wenn nicht in der Lage, die gepatchte Zelle genau zu bestimmen, umgeben das Gebiet, wo die Kaution in Höhe von Biocytin Dichter in der abgebildeten Netzwerk gekoppelten Zellen, mit dem Polygon-Werkzeug in ImageJFIJI, und beziehen sich auf diese Position wie die gepatchte Zelle (Abbildung 4).
- Verwenden Sie den ROI-Manager (analysieren | Werkzeuge | ROI-Manager). Dann ziehen Sie einen ROI in die gepatchte Zellenposition und fügen Sie es zum ROI-Manager (siehe Abbildung 4 b).
- Legen Sie eine Messung (Analyze | Messung eingestellt) und wählen Sie die Option "Schwerpunkt".
- Klicken Sie im ROI-Manager auf "Messen", um die Koordinaten der Schwerpunkt des aufgenommenen Bereichs zu erhalten. Verwenden Sie diese Koordinaten als Referenzpunkt für dieses spezifische Netzwerk.
- Referentielle Übersetzung
- Berechnen Sie die Koordinaten der einzelnen Zellen in Bezug auf die gepatchte Astrozyten, mithilfe der folgenden Formel:

Mit: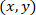 als die Koordinaten für eine bestimmte Zelle;
als die Koordinaten für eine bestimmte Zelle; 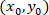 als die Koordinaten der gepatchten Zelle (oder der referenziellen Punkt des Netzes); und
als die Koordinaten der gepatchten Zelle (oder der referenziellen Punkt des Netzes); und 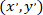 als die Koordinaten für eine bestimmte Zelle in der neuen referenzielle.
als die Koordinaten für eine bestimmte Zelle in der neuen referenzielle.
Hinweis: Die Koordinaten der einzelnen Zellen in Bezug auf die gepatchte Astrozyten auszudrücken ist ein wichtiger Schritt bei der Berechnung von Vektoren aus der gepatchten Astrozyten. Seien Sie vorsichtig, wenn Sie ImageJFIJI verwenden, die die referenzielle für jedes Bild befindet sich in der oberen linken Ecke des Bildes.
- Berechnen Sie die Koordinaten der einzelnen Zellen in Bezug auf die gepatchte Astrozyten, mithilfe der folgenden Formel:
- Bestimmung der wichtigste Vektor der bevorzugte Ausrichtung
- Berechnen Sie die Koordinaten der wichtigsten Vektor der bevorzugte Ausrichtung mit der folgenden Formel:
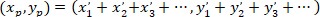
Mit: (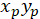 ) als die Koordinaten der wichtigsten Vektor der bevorzugte Ausrichtung; und
) als die Koordinaten der wichtigsten Vektor der bevorzugte Ausrichtung; und 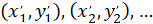 als die Koordinaten der einzelnen Zellen des Netzwerks erhalten mit der gepatchten Zelle als referenzielle.
als die Koordinaten der einzelnen Zellen des Netzwerks erhalten mit der gepatchten Zelle als referenzielle.
Hinweis: Für jede Zelle im Netzwerk wird ein Vektor relativ zu den Koordinaten der gepatchten Astrozyten bestimmt. Der wichtigste Vektor der bevorzugte Ausrichtung des Netzwerks ist die Summe aller dieser Vektoren. - Teilen Sie die Länge der wichtigste Vektor (bereitgestellt durch die Koordinaten mit Hilfe der oben genannten Formel entnommen) durch die Anzahl der Zellen in das Netzwerk minus eins (da die Koordinaten der gepatchten Zelle nicht enthalten sind), um die Werte zu normalisieren und ermöglichen Vergleiche zwischen Netzwerken. Eine schematische Darstellung dieser Analyse ist in Abbildung 5dargestellt.
- Berechnen Sie die Koordinaten der wichtigsten Vektor der bevorzugte Ausrichtung mit der folgenden Formel:
- Bestimmung der gepatchten Zelle
- Platzierung der analysierten Netzwerke im Zellkern von Interesse
- Ausrichtung der 20 X und 4 X Bilder
- Um die Position der einzelnen Netzwerke im Zellkern von Interesse (NVsnpr) zu bestimmen, verwenden Sie das 4 X Bild. 4 X Bild mit einem Vektor-Bild-Editor zu öffnen.
- Wählen Sie 4 X Bild und ändern Sie seine Größe mit 5 multipliziert. Die Dimension-Fenster befindet sich im rechten Teil der oberen horizontalen Symbolleiste. Zum Beispiel für einen 4 X Bild, das bei 800 x 800 Pixel abgetastet wurden, ändern die Abtastauflösung auf 4000 x 4000 Pixel (um das Arbeitsgerät in Pixel festzulegen, gehen Sie zu "Dokument einrichten" unter der Registerkarte "Datei": Datei | Dokument einrichten). Exportieren Sie die Datei im TIFF-Format und Name es 4 X "verkleinert".
- Richten Sie die linke obere Ecke des Bildes mit der linken unteren Ecke des Adobe Illustrator-Dokument.
Hinweis: Die Software bietet Koordinaten von einem referenzielle Punkt unten links. - Öffnen Sie das 20 X-Bild des Netzwerks. Verwenden Sie '' binäre Datei '' oder '' Erkennung Datei '', weil sie leichter zu richten sind. Dann spielen mit dem Deckkraft-Werkzeug auf die '' binäre Datei '' 20 X-Image mit der Größe 4 X ausrichten Bild.
Hinweis: Die Deckkraft-Tool ist in der oberen horizontalen Symbolleiste. - Wenn die Ausrichtung ist, wählen halten Sie das Pipette-Werkzeug zu, so dass das Messwerkzeug erscheint und wählen sie in der linken Seitenleiste.
- Verwenden das Werkzeug "Messen", klicken Sie auf der linken oberen Ecke des der 20 X Bild, um die Koordinaten der 20 X referenzielle verweisen auf die Größe 4 X Bild.
Hinweis: Diese Koordinaten, die als die 20 X referentielle (20XR in Abbildung 5) bezeichnet werden, ist nützlich für die Position der einzelnen Netzwerke auf eine schematische Zeichnung des Kerns zum Ausdruck zu bringen.
- Normalisierung des Kerns von Interesse
Hinweis: Um die Daten zusammenzufassen, ist eine Normalisierung des Kerns (NVsnpr) als Rechteck verwendet. Die Schritte sind im folgenden beschrieben.- Öffnen Sie 4 X Größe Datei in ImageJFIJI und verwenden Sie das Polygon-Werkzeug, um den Kern (NVsnpr) umgeben. Verwenden Sie das Bild mit Durchlicht, wenn die Grenzen des Kerns nicht gesehen werden können; in diesem Fall, denken Sie daran, es zuerst die Größe.
- Öffnen Sie den ROI-Manager, und fügen Sie die gezogenen ROI. "Set Measurement" wählen Sie die Option '' Bounding Rectangle''.
Hinweis: Die Option "Bounding Rectangle" berechnet das kleinste Rechteck um den gezeichneten Kern. - Klicken Sie auf "Messen".
Hinweis: Erscheint eine Tabelle mit BX und BY, die Koordinaten der oberen linken Ecke des Rechtecks: '' Rechteck Position'' und bietet die Breite und Höhe. BX und BY sind die Koordinaten des umschließenden Rechtecks referenzielle als bezeichneten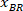 und
und 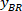 .
.
- Ausdruck jeder Netzwerk-Position in der normalisierten Kern
- Express die Koordinaten der einzelnen Zellen mit dem 4 X referentielle (Schwarzes Quadrat in Abbildung 5) mithilfe der folgenden Formel:

Wo: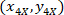 sind die Zellenkoordinaten mit dem 4 X referenzielle;
sind die Zellenkoordinaten mit dem 4 X referenzielle; 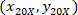 sind die Zellkoordinaten mit 20 X referentielle (orangefarbenes Quadrat in Abbildung 5); und
sind die Zellkoordinaten mit 20 X referentielle (orangefarbenes Quadrat in Abbildung 5); und 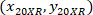 sind die Koordinaten der 20 X referenzielle Punkt im 4 X Bild (20XR).
sind die Koordinaten der 20 X referenzielle Punkt im 4 X Bild (20XR). - Express die Zellkoordinaten in das umschließende Rechteck referentielle (blau in Abbildung 5) mithilfe der folgenden Formel:
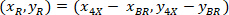
Wo: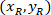 sind die Zelle das umschließende Rechteck referenzielle koordiniert;
sind die Zelle das umschließende Rechteck referenzielle koordiniert; 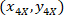 sind die Zellkoordinaten in der 4 X referenzielle; und
sind die Zellkoordinaten in der 4 X referenzielle; und  sind die Koordinaten des umschließenden Rechtecks referenzielle im 4 X Bild.
sind die Koordinaten des umschließenden Rechtecks referenzielle im 4 X Bild. - Transformieren Sie die Zellkoordinaten in das umschließende Rechteck referentielle, der Prozentsatz der Breite und Höhe des umschließenden Rechtecks mithilfe der folgenden Formel:
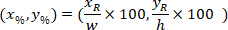
Wo: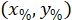 sind die Zelle in den Prozentsatz der Breite und Höhe des umschließenden Rechtecks koordiniert;
sind die Zelle in den Prozentsatz der Breite und Höhe des umschließenden Rechtecks koordiniert; 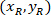 sind die Zelle das umschließende Rechteck referenzielle koordiniert;
sind die Zelle das umschließende Rechteck referenzielle koordiniert;  ist die Breite des umschließenden Rechtecks gemessen oberhalb des Protokolls; und
ist die Breite des umschließenden Rechtecks gemessen oberhalb des Protokolls; und 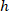 ist die Höhe des umschließenden Rechtecks gemessen oben im Protokoll.
ist die Höhe des umschließenden Rechtecks gemessen oben im Protokoll. - Um die Netze auf der gleichen Figur darzustellen, berücksichtigen Sie die Ausrichtung des Segments (links oder rechts). Um die Daten zu standardisieren, die referenzielle auf der linken Seite der Scheibe anliegt. Übertragen Sie das Koordinatennetz der rechten Seite auf der linken Seite anhand der folgenden Formel nur für X-Koordinaten:
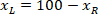
Wo: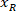 ist die X-Koordinate auf der rechten Seite der Scheibe; und
ist die X-Koordinate auf der rechten Seite der Scheibe; und 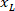 ist die neue X-Koordinate auf der linken Seite der Scheibe in die referenzielle ausgedrückt.
ist die neue X-Koordinate auf der linken Seite der Scheibe in die referenzielle ausgedrückt.
Hinweis: Alternativ, spiegeln das Bild im ImageJFIJI vor der Analyse (Bild | Transformieren | Horizontal spiegeln Sie). - Die Koordinaten der wichtigsten Vektor der bevorzugte Ausrichtung zum Ausdruck bringen, die gleichen Schritte mit den Zellkoordinaten (Schritte 6.5.3.1, 6.5.3(4)).
Hinweis: Der Ausdruck der Koordinaten in Prozenten ermöglicht eine Zusammenstellung der Daten als einem einzigen Grundstück, in dem der Kern (NVsnpr) als ein Rechteck ausgebildet ist.
- Express die Koordinaten der einzelnen Zellen mit dem 4 X referentielle (Schwarzes Quadrat in Abbildung 5) mithilfe der folgenden Formel:
- Ausrichtung der 20 X und 4 X Bilder
- Studie über die Winkeldifferenz der wichtigste Vektor der Vorzugsrichtung
Hinweis: Die Winkeldifferenz eines astrocytic Netzwerks wird verwendet, um festzustellen, ob die bevorzugte Ausrichtung in Richtung der Mitte des Kerns von Interesse ist. Berechnen Sie die Winkeldifferenz (α in Abbildung 5), ist der Winkel zwischen der wichtigste Vektor der Vorzugsrichtung des Netzes (PD, rote Linie in Abbildung 5) und die Verbindungslinie zwischen P, C, gelten Sie die Al-Kashi-Theorem in das Dreieck PDC (siehe INSET Abbildung 5 und Ergänzende Methoden).- Berechnen Sie zunächst, die unterschiedlichen Längen der Anwendung des Satzes von Pythagoras in eine kartesische referenzielle mit folgender Gleichung:
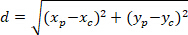
Wo: die Koordinaten von P und C sind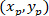 und
und  , bzw. in das umschließende Rechteck referentielle (oben berechneten).
, bzw. in das umschließende Rechteck referentielle (oben berechneten). - Bestimmen Sie die Winkeldifferenz in Bogenmaß mithilfe der folgenden Formel:
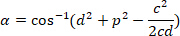
- Die Winkeldifferenz in Grad (z. B. mit der Grad-Funktion in Excel Software) zu konvertieren.
- Kompilieren Sie die eckige Unterschiede durch plotten sie in vertikale Balkendiagramme (Abbildung 7 und 7 D) und festzustellen Sie, ob eine bevorzugte Ausrichtung der astrocytic Netzwerke.
- Berechnen Sie zunächst, die unterschiedlichen Längen der Anwendung des Satzes von Pythagoras in eine kartesische referenzielle mit folgender Gleichung:
Ergebnisse
Kopplung zwischen Zellen im Gehirn ist nicht statisch, sondern eher dynamisch geregelten von vielen Faktoren ab. Beschriebenen Methoden wurden entwickelt, um astrocytic Netzwerke offenbart unter verschiedenen Bedingungen zu analysieren und zu verstehen, ihre Organisation in NVsnpr. Diese Ergebnisse wurden bereits veröffentlichten1. Wir Biocytin Füllung der einzelnen Astrozyten im dorsalen Teil des NVsnpr in drei verschiedenen Bedingungen durchgeführt: im Ruhezus...
Diskussion
Es gibt eine Reihe von elektrophysiologischen Methoden um funktionelle Kopplung zwischen Astrozyten23,24zu beurteilen. Diese Methoden bieten jedoch keine Informationen über die anatomische Anordnung der astrocytic Netzwerke. Eine Reihe von Studien haben bereits gezeigt, dass "Farbstoff - oder Tracer-Kupplung", wie hier, nur in einem Bruchteil der auftritt verbunden Zellen, die durch elektrophysiologische Methoden25,
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wird finanziert durch Canadian Institutes of Health Research, Grant/Prämiennummer: 14392.
Materialien
| Name | Company | Catalog Number | Comments |
| NaCl | Fisher Chemicals | S671-3 | |
| KCl | Fisher Chemicals | P217-500 | |
| KH2PO4 | Fisher Chemicals | P285-500 | |
| MgSO4 | Fisher Chemicals | M65-500 | |
| NaHCO3 | Fisher Chemicals | S233-500 | |
| C6H12O6 Dextrose anhydrous | Fisher Chemicals | D16-500 | |
| CaCl2 dihydrated | Sigma | C70-500 | |
| Sucrose | Sigma | S9378 | |
| D-gluconic acid potassium salt | Sigma | G45001 | |
| MgCl2 anhydrous | Sigma | M8266 | |
| HEPES | Sigma | H3375 | |
| EGTA | Sigma | E4378 | |
| ATPTris Salt | Sigma | A9062 | |
| GTPTris Salt | Sigma | G9002 | |
| Biocytin | Sigma | B4261 | |
| Carbenoxolone disodium salt | Sigma | C4790 | |
| avidin-biotin complex : ABC kit | Vestor laboratories | PK-4000 | |
| Streptavidine-alexa 594 | Molecular Probes | S11227 | |
| Triton | Fisher Chemicals | BP151-500 | |
| Xylene | Fisher Chemicals | X5-1 | |
| Aqueous mounting medium 1 : Fluoromount-G | SouthernBiotech | 0100-01 | |
| Toluen-based synthetic resin mounting medium : Permount | Fisher Chemicals | SP15-100 | |
| Slide Drying Bench | Fisherbrand | 11-474-470 | |
| Vibratome | Leica | VT 1000S | |
| Microscope cover glass | Fisherbrand | 12-544A | |
| Microscope slide ColorFrost | Fisherbrand | 12-550-413 | |
| PFA | Fisherchemicals | 04042-500 | |
| Olympus FluoView FV 1000 Confocal microscope | Olympus | ||
| 40X water-immersion lens | Olympus | LUMPLFLN40XW | |
| 20X water-immersion lens | Olympus | XLUMPLFL20XW | |
| 4X water-immersion lens | Olympus | XLFLUOR4X/340 | |
| Micropipette puller | Sutter Instrument | P97 | |
| Micromanipulator | Sutter Instrument | MP 225 | |
| Camera CCD | Sony | CX-ST50 | |
| Black and white monitor | Sony | SSM-125 | |
| Digidata | Molecular devices | 1322A | |
| Patch Clamp amplifier | Axon instrument | Mulitclamp 700A | |
| Electrophysiology acquisition software | Molecular devices | pClamp 8 | |
| Electrophysiology analysis software | Molecular devices | Clampfit 8 | |
| Imaging analysis software | ImageJFIJI | Open source software. FIJI version including plug in package. | |
| Vector image editor | Adobe | Illustrator CS4 | |
| Spreadsheet application | Microsoft Office | Excel 2010 |
Referenzen
- Condamine, S., Lavoie, R., Verdier, D., Kolta, A. Functional rhythmogenic domains defined by astrocytic networks in the trigeminal main sensory nucleus. Glia. 66 (2), 311-326 (2018).
- Verkhratsky, A., Orkand, R. K., Kettenmann, H. Glial calcium: homeostasis and signaling function. Physiological Review. 78 (1), 99-141 (1998).
- Christensen, R. K., Petersen, A. V., Perrier, J. F. How do glial cells contribute to motor control?. Current Pharmaceutical Design. 19 (24), 4385-4399 (2013).
- Verkhratsky, A., Steinhauser, C. Ion channels in glial cells. Brain Research Review. 32 (2-3), 380-412 (2000).
- Harada, K., Kamiya, T., Tsuboi, T. Gliotransmitter Release from Astrocytes: Functional, Developmental, and Pathological Implications in the Brain. Frontiers Neuroscience. 9, 499 (2015).
- Montero, T. D., Orellana, J. A. Hemichannels: new pathways for gliotransmitter release. Neuroscience. 286, 45-59 (2015).
- Araque, A., et al. Gliotransmitters travel in time and space. Neuron. 81 (4), 728-739 (2014).
- Augustin, V., et al. Functional anisotropic panglial networks in the lateral superior olive. Glia. 64 (11), 1892-1911 (2016).
- Houades, V., Koulakoff, A., Ezan, P., Seif, I., Giaume, C. Gap junction-mediated astrocytic networks in the mouse barrel cortex. Journal of Neuroscience. 28 (20), 5207-5217 (2008).
- Roux, L., Benchenane, K., Rothstein, J. D., Bonvento, G., Giaume, C. Plasticity of astroglial networks in olfactory glomeruli. Proceedings of the National Academy of Science of the United State of America. 108 (45), 18442-18446 (2011).
- Morquette, P., et al. An astrocyte-dependent mechanism for neuronal rhythmogenesis. Nature Neuroscience. 18 (6), 844-854 (2015).
- Brocard, F., Verdier, D., Arsenault, I., Lund, J. P., Kolta, A. Emergence of intrinsic bursting in trigeminal sensory neurons parallels the acquisition of mastication in weanling rats. Journal of Neurophysiology. 96 (5), 2410-2424 (2006).
- Anders, S., et al. Spatial properties of astrocyte gap junction coupling in the rat hippocampus. Philosophical Transactions of the Royal Society of London. Series B, Biological Science. 369 (1654), (2014).
- Houades, V., et al. Shapes of astrocyte networks in the juvenile brain. Neuron Glia Biology. 2 (1), 3-14 (2006).
- Rouach, N., Koulakoff, A., Abudara, V., Willecke, K., Giaume, C. Astroglial metabolic networks sustain hippocampal synaptic transmission. Science. 322 (5907), 1551-1555 (2008).
- Claus, L., et al. Barreloid Borders and Neuronal Activity Shape Panglial Gap Junction-Coupled Networks in the Mouse Thalamus. Cerebral Cortex. 28 (1), 213-222 (2018).
- Cameron, M. A., et al. Prolonged Incubation of Acute Neuronal Tissue for Electrophysiology and Calcium-imaging. Journal of Visualized Experiments. (120), (2017).
- Kafitz, K. W., Meier, S. D., Stephan, J., Rose, C. R. Developmental profile and properties of sulforhodamine 101--Labeled glial cells in acute brain slices of rat hippocampus. Journal of Neuroscience Methods. 169 (1), 84-92 (2008).
- Neher, E. Correction for liquid junction potentials in patch clamp experiments. Methods in Enzymology. , 123-131 (1992).
- Giaume, C., Leybaert, L., Naus, C. C., Saez, J. C. Connexin and pannexin hemichannels in brain glial cells: properties, pharmacology, and roles. Frontiers in Pharmacology. 4, 88 (2013).
- Torres, A., et al. Extracellular Ca(2)(+) acts as a mediator of communication from neurons to glia. Science Signaling. 5 (208), ra8 (2012).
- Ye, Z. C., Wyeth, M. S., Baltan-Tekkok, S., Ransom, B. R. Functional hemichannels in astrocytes: a novel mechanism of glutamate release. Journal of Neuroscience. 23 (9), 3588-3596 (2003).
- Ma, B., et al. Gap junction coupling confers isopotentiality on astrocyte syncytium. Glia. 64 (2), 214-226 (2016).
- Meme, W., Vandecasteele, M., Giaume, C., Venance, L. Electrical coupling between hippocampal astrocytes in rat brain slices. Neuroscience Research. 63 (4), 236-243 (2009).
- Ransom, B. R., Kettenmann, H. Electrical coupling, without dye coupling, between mammalian astrocytes and oligodendrocytes in cell culture. Glia. 3 (4), 258-266 (1990).
- Audesirk, G., Audesirk, T., Bowsher, P. Variability and frequent failure of lucifer yellow to pass between two electrically coupled neurons in Lymnaea stagnalis. Journal of Neurobiology. 13 (4), 369-375 (1982).
- Ewadinger, N., Syed, N., Lukowiak, K., Bulloch, A. Differential Tracer Coupling between Pairs of Identified Neurones of the Mollusc Lymnaea Stagnalis. Journal of Experimental Biology. 192 (1), 291-297 (1994).
- Griemsmann, S., et al. Characterization of Panglial Gap Junction Networks in the Thalamus, Neocortex, and Hippocampus Reveals a Unique Population of Glial Cells. Cerebral Cortex. 25 (10), 3420-3433 (2015).
- Kuwajima, T., et al. ClearT: a detergent- and solvent-free clearing method for neuronal and non-neuronal tissue. Development. 140 (6), 1364-1368 (2013).
- Gourine, A. V., et al. Astrocytes control breathing through pH-dependent release of ATP. Science. 329 (5991), 571-575 (2010).
- Forsberg, D., Ringstedt, T., Herlenius, E. Astrocytes release prostaglandin E2 to modify respiratory network activity. eLife. 6, (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten