É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Analisando o tamanho, forma e direcionalidade das redes de astrócitos acoplados
Neste Artigo
Resumo
Aqui nós apresentamos um protocolo para avaliar a organização de redes hamartomas. O método descrito minimiza o viés para fornecer medidas descritivas dessas redes tais como a contagem de células, tamanho, área e posição dentro de um núcleo. Anisotropia é avaliada com uma análise vectorial.
Resumo
Tornou-se cada vez mais claro que os astrócitos modulam função neuronal não só a nível sináptico e célula única, mas também no nível da rede. Astrócitos são fortemente ligados uns aos outros através de junções e acoplamento através destes cruzamentos é dinâmico e altamente regulamentado. Um conceito emergente é que funções hamartomas são especializadas e adaptadas para as funções do circuito neuronal com o qual eles estão associados. Portanto, métodos para medir vários parâmetros das redes hamartomas são necessários para melhor descrever as regras que regem a sua comunicação e acoplamento e a entender melhor as suas funções.
Aqui, usando o software de análise de imagem (ex., ImageJFIJI), descrevemos um método para analisar imagens confocal de redes hamartomas reveladas pela tintura-acoplamento. Estes métodos permitem 1) uma detecção automatizada e imparcial de pilhas etiquetadas, 2) cálculo do tamanho da rede, 3) cálculo da orientação preferencial da tintura espalhar dentro da rede e 4) reposicionamento da rede dentro da área de interesse .
Esta análise pode ser usada para caracterizar redes hamartomas de uma determinada área, comparar as redes de diferentes áreas associadas a diferentes funções ou comparar redes obtidas em condições diferentes que têm efeitos diferentes sobre o acoplamento. Estas observações podem levar a importantes considerações funcionais. Por exemplo, analisamos as redes hamartomas de um núcleo trigeminal, onde anteriormente mostramos que hamartomas acoplamento é essencial para a capacidade dos neurônios mudar seus padrões de fuzilamento de tónico para ruptura rítmica1. Ao medir o tamanho, confinamento e orientação preferencial das redes hamartomas esse núcleo, podemos construir hipóteses sobre domínios funcionais que eles delimitam. Vários estudos sugerem que várias outras áreas do cérebro, incluindo o córtex de barril, oliva superior lateral, glomérulos olfativos e núcleos sensoriais no tálamo e no córtex visual, para citar alguns, podem beneficiar de uma análise semelhante.
Introdução
Muitos estudos têm descrito como o diálogo do neurônio-astrocyte em um nível celular sub ou sináptico pode ter implicações em funções neuronais e a transmissão sináptica. Está bem estabelecido que astrócitos são sensíveis ao redor de atividade neuronal; na verdade, eles possuem receptores para muitos neurotransmissores incluindo glutamato, GABA, acetilcolina e ATP (ver comentários publicados anteriormente2,3,4). Em troca, hamartomas processa sináptica elementos ensheath e influência de atividade neuronal ambos lá e em locais extrasynaptic regulação homeostase iónica extracelular e soltando vários fatores ou transmissores, tais como o glutamato, D-serina e ATP 5 , 6 , 7.
Surgiu a ideia de que astrócitos também podem modular a função neuronal no nível da rede, com evidências de que o acoplamento hamartomas espacialmente é regulamentado e corresponde à segmentação neuronal em áreas caracterizadas por uma clara anatômica compartimentação (como áreas com representações sensoriais), indicando que astrócitos combinar para outros astrócitos, servindo a mesma função, em vez de apenas aqueles que estão por perto. Na oliva superior lateral, por exemplo, redes mais hamartomas são orientadas ortogonalmente ao tonotopic eixo8, Considerando que em glomérulos barril córtex ou olfactoty, a comunicação entre os astrócitos é muito mais forte dentro de barris ou glomérulos e mais fraco entre adjacentes uns9,10. Em ambos os casos, as redes hamartomas são orientadas em direção ao centro do glomerule ou barril9,10.
Recentemente mostramos que a atividade hamartomas modula disparo neuronal, diminuindo a concentração de extracelular Ca2 + ([Ca2 +]e), presumivelmente através da liberação de S100β, uma Ca2 +-ligação proteína11. Este efeito, o que foi demonstrado em uma população de neurônios rhythmogenic trigeminal na parte dorsal das principais trigeminal sensorial núcleo (NVsnpr, pensado para desempenhar um papel importante na geração de movimentos de mastigação), resulta do fato de que fuzilamento rítmico nestes neurônios depende de um persistente at+ atual que é promovido por diminuições de [Ca2 +]11,e12. Fuzilamento rítmico nestes neurônios pode ser eliciado "fisiologicamente" estimulação de suas entradas ou diminuição artificial da [Ca2 +]e. Nós revelou ainda que hamartomas acoplamento foi necessário para acionamento rítmico neuronal1. Isso levantou a possibilidade que as redes hamartomas podem formar circunscritos domínios funcionais, onde a atividade neuronal pode ser sincronizada e coordenada. Para avaliar esta hipótese, precisamos primeiro desenvolver um método para documentar rigorosamente a organização destas redes dentro de NVsnpr.
Estudos anteriores sobre redes hamartomas principalmente descreveu a extensão do acoplamento em termos de número de célula e a densidade e a área coberta. As tentativas de avaliar a forma de redes hamartomas e a direção da tintura-acoplamento principalmente foram realizadas comparando o tamanho das redes ao longo de dois eixos (x e y) do barril córtex9, hipocampo13,14, 15, barreloid campos do tálamo16, oliva superior lateral8, glomérulos olfativos10e o córtex14. Os métodos descritos aqui permitem imparciais acusações de pilhas etiquetadas em uma rede e uma estimativa da área que eles cobrem. Também desenvolvemos ferramentas para definir a orientação preferencial do acoplamento dentro de uma rede e avaliar se a orientação preferencial é em direção ao centro do núcleo, ou em uma direção diferente. Em comparação com métodos utilizados anteriormente, este protocolo fornece um meio para descrever a organização e a orientação das redes hamartomas em estruturas como o núcleo sensorial principal trigeminal dorsal que não têm um conhecido clara anatômico compartimentalização. Nos estudos acima, a orientação de rede é descrita como uma relação com a forma da estrutura em si que já está documentada (ex., o barreloid no tálamo, barris no córtex, camadas no hipocampo e córtex, glomérulos em o bulbo olfatório, etc.). Além disso, análise vectorial permite comparações de orientações reveladas sob diferentes condições de acoplamento. Para analisar se estes parâmetros alterados de acordo com a posição da rede dentro do núcleo, nós também desenvolvemos um método para substituir cada rede em referência os limites do núcleo. Estas ferramentas podem ser facilmente adaptadas para outras áreas para investigar redes de células acopladas.
Protocolo
Todos os procedimentos de morada pelas regras institutos canadenses de pesquisa em saúde e foram aprovados pelo Comitê de uso e cuidados com animais da Universidade de Montreal.
1. preparação de fatias de cérebro de rato
- Prepare 1 L de uma solução baseada em sacarose (tabela 1) e 1 litro de líquido de cerebral-espinhal artificial padrão (aCSF) (tabela 2).
- Bolha a solução de sacarose com uma mistura de 95% O2 e 5% CO2 (carbogênio) por 30 min antes de colocá-lo em-80 ° C por cerca de 30 min, até que a solução é frio, mas não totalmente congelada. Use este sacarose gelada como o buffer de corte para o corte do cérebro. Mantê-lo no gelo, uma vez que ele é removido do freezer.
- Bolha a aCSF com carbogênio através de todo o experimento. Use esta solução para armazenamento de fatia e como o buffer perfusing em que o remendo-aperto e biocytin-enchimento de astrócitos serão executadas. Prepare uma fatia recuperação segurando a câmara para depositar as fatias do cérebro, tão logo eles são cortados.
Nota: A câmara de exploração de recuperação fatia poderia ser feitos sob medida e consiste essencialmente de um pequeno poço com uma malha na parte inferior suspendido em um bem maior que é preenchido com aCSF, no qual um tubo é inserido para trazer carbogen do fundo. - Use ratos Sprague-Dawley de 15 a 21 dias de idade sem qualquer preconceito de sexo ou estirpe específica. Anestesia o animal com isoflurano (1 mL de isoflurano em uma câmara de indução da anestesia). Verifique a profundidade da anestesia beliscando suavemente a pata posterior ou cauda do animal.
- Decapitar o rato usando uma guilhotina, corte seu crânio com uma tesoura e rapidamente Retire o cérebro o crânio com uma espátula plana.
- Mergulhe o cérebro na solução baseada em sacarose gelada para sobre 30 s e transferência-o (com a solução) para uma petri dish. Com uma lâmina de barbear, remover as partes que são anterior e posterior à área a ser seccionado, se corte fatias no plano transversal.
- Cole o bloco restante do tecido cerebral na parte rostral e inicie o corte fatias de cérebro (350 µm de espessura) no meio de sacarose-baseado usando um vibratome. Então, transfira as fatias coletadas em uma recuperação segurando câmara cheia de carbogenated aCSF à temperatura ambiente (RT) até que estejam prontos para serem usados (Aguarde pelo menos 1 hora para recuperação).
Nota: Desenvolvimentos recentes no campo permitem incubação prolongada até 24 h de fatias de cérebro no in vitro as condições de17.
2. sulforhodamina 101 (SR-101) rotulagem de astrócitos
- Pré-aquecer o banho de água a 34 ° C e coloque duas câmaras de incubação fatia nele. Preencher uma das câmaras de incubação de fatia com uma solução de aCSF contendo 1 μM SR-101 e preencher o outro apenas com aCSF.
- Incubar as fatias na câmara de incubação contendo a 1 µM SR-101 para 20 min e depois transferência-los para a segunda incubação de câmara de lavar SR-101 em excesso do tecido. Deixe incubar por 20 min ou mais a 34 ° C, então mantenha a câmara de incubação contendo as fatias no RT até que eles são necessários18.
3. astrocyte patches e enchimento de Biocytin
- Selecione uma fatia e colocá-lo na câmara de gravação do microscópio. Para patch astrócitos, usar eletrodos com uma resistência de 4-6MΩ, quando preenchido com uma solução de gluconato de potássio-base (ver quadro 3).
- Sob orientação visual e usando um micromanipulador, direcionar o eléctrodo de gravação para um astrocyte SR-101-rotulado como mostrado na Figura 1. Evite células localizadas na superfície da fatia, uma vez que eles são mais propensos a ser danificado ou perdido conexões às células vizinhas.
- Para evitar fugas de biocytin no tecido, minimizar a pressão positiva adicionada para a pipeta de remendo e aplicá-lo apenas quando está perto da astrocyte que será remendado (0,1-0,4 mL em uma seringa de 1 mL).
- Antes da aplicação de patches, ajuste o deslocamento da pipeta e da sua capacidade. Corrigir para potenciais de junção líquida, como precisas e comandos precisos de tensão são fundamentais para as experiências de19.
- Mova a pipeta perto o suficiente para o astrocyte para observar a depressão causada pela pressão positiva. Em seguida, retire a pressão positiva e lentamente aplicar alguma pressão negativa. Fixar o astrocyte de -70 mV quando o selo atinge 100 MΩ. Espere até que o selo atinge GΩ 1-3. Continue a aplicar pressão negativa até invadir a célula. Tenha cuidado ao aplicar pressão negativa, uma vez que os astrócitos são muito frágeis.
- Avalie as propriedades eletrofisiológicas da célula remendada. No modo de tensão-braçadeira, executar um protocolo de corrente-tensão de célula inteira com um comando de tensão de rampa de duração de 600 ms, variando de -120 a 110 mV. Em modo de corrente-braçadeira, execute um protocolo passo IV, em que a injeção de 1000 ms atual passos do pA 100 -1 para 1 nd, a taxa de amostragem de gravações a célula inteira é 10 kHz.
Nota: Astrócitos mostram um perfil linear de corrente-tensão sem qualquer tipo de retificação (como mostrado na figura 1B) e sem potencial de ação, atirando em cima de despolarização da membrana (Figura 1). O potencial de membrana de repouso (RMP) deve ser estável e não positivo a - 60mV. Em algumas áreas do cérebro, o RMP de astrócitos é mais hyperpolarized do que no NVsnpr. - Permitir que o biocytin difundir dentro do astrocyte por 30 min, durante a execução de um passo protocolo IV cada 5 min.
- Retraia e desanexar a pipeta de remendo cuidadosamente sem danificar o astrocyte remendado e imediatamente tomar nota do deslocamento da pipeta antes de tomá-lo fora da câmara de gravação. Subtrai esse valor das gravações de potenciais de membrana.
- Embora a fatia do cérebro descansar em câmara de gravação para um mínimo de 15 min (além de 30 min para injeção) para permitir a propagação do traçador do astrocyte remendado para toda a rede de células acopladas.
Nota: Para revelar uma rede de células acopladas, apenas uma única célula deve ser corrigida no núcleo de interesse. Se várias tentativas são necessárias para alcançar um sucesso patch, descartar o caso e tentar corrigir no lado contralateral, ou em outra fatia do cérebro. - Fazer uma marca ou incisão no tecido para identificar qual lado da fatia é virada para cima e transferir a fatia primeiro para uma placa de Petri contendo aCSF normal, então em uma solução de paraformaldeído 4% (PFA). Incube a fatia em 4 ° C durante a noite.
Cuidado: PFA é extremamente prejudicial. Use equipamento de proteção em uma campânula ventilada. Certifique-se de que todos os materiais (pincéis, tubos, etc.) que estiveram em contacto com o PFA não entre em contato com a recuperação, mantendo a câmara, câmara de incubação ou outros materiais utilizados para a gravação do tecido fresco.
4. Biocytin revelação
- Revelação com streptavidine fluorescente
- Lavar o cérebro fatias 2 x 10 min em soro de tampão de fosfato 0,1 M (PBS) no RT Em seguida, revelar o biocytin incubando-se as fatias de cérebro com streptavidine conjugada com um fluoróforo na diluição de 1/200 em PBS com detergente não-iônico de 4% para 4h no RT
- Lave as fatias de cérebro 3 x 10 min em PBS 0,1 M a RT e montar as seções em lâminas de vidro usando um meio de montagem aquoso. Coloque os lados que foram injetados para cima, lamela os slides e selá-los com verniz das unhas.
- Revelação com DAB
Nota: Revelação DAB (3,3-diaminobenzidina) pode ser usada quando a fluorescência não pode ser usada. Neste caso, apenas uma revelação método deve ser empregado para fazer comparações.- Lave as fatias de cérebro 3 x 10 min em PBS em RT incubam as fatias em PBS + H2O2 0,5% durante 1 hora a RT
- Lave as fatias 3 x 10 min em PBS em RT Em seguida, incubar as fatias em uma solução coloração complexo avidina-biotina, composto de PBS e 0,1% de detergente não-iônico + avidina-biotina peroxidase complexo padrão coloração reagentes em 1/100, durante 24 horas a RT
- Lave as fatias 3 x 10 min em PBS em RT, incube-os em uma solução de PBS + DAB 0.05% por 20 min na RT e transferi-los para uma solução de PBS + DAB 0.05% + H2O2 0,5%.
Cuidado: DAB é extremamente prejudicial. Use equipamento de proteção em uma campânula ventilada. A reação de cor é interrompida quando as fatias são transferidas em PBS. - Lave as fatias de 5 x 10 min em PBS em RT, as seções de montagem em lâminas de vidro (encarar os lados que foram injetados para cima) e deixe-os secar em um slide de secagem banco durante a noite a 34 ° C.
- Submergi as lâminas de vidro para 1 min em vários banhos de álcool no 70%, 95% e 100% e no final com um banho de xilol por 1 min.
- Monte as lâminas de vidro usando uma resina sintética baseada em tolueno, meio de montagem. Coloque as lâminas lamelas e selá-los com verniz das unhas.
5. rede Imaging
- Visualizar as redes hamartomas usando um microscópio confocal equipado com 20 X e 4 objectivos de X (ou um objectivo adequado para visualizar o núcleo inteiro onde se encontra a rede) e um laser para detectar o fluoróforo (neste caso, Alexa-594 é usado).
- Use a ampliação X 20 para fazer uma z-pilha da rede de pilhas etiquetadas. Usar uma resolução de 800 x 800 pixels e velocidade de 12,5 μs/pixel de digitalização.
Nota: Para toda a rede de imagem, pilhas múltiplas são geralmente necessárias, e o número de pilhas precisa ser ajustado para cada rede. A velocidade de resolução e varredura pode ser mudada, mas certifique-se de usar as mesmas configurações para todos os dados de imagem. - Use a ampliação de 4x para tirar imagens da rede e região de interesse.
Nota: A imagem de 4x é usado para determinar a posição de rede dentro do núcleo de interesse. Sempre o mesmo campo em contra-luz da imagem. Esta imagem será útil se você não pode determinar a fronteira do núcleo na imagem fluorescente confocal.
6. análise de imagens
- Preparação de dados
- Usar o software ImageJFIJI (download em https://fiji.sc/). Abra o arquivo e clique em Okey na janela "Opções de importação de Bio-formatos".
- Para redefinir um z-pilha que conterá somente as fatias ópticas necessárias para a z-pilha final (Figura 2A), clique no botão "Pilha" na barra de ferramentas (para encontrar, primeiro selecione o stk | Projeto de Z). Selecione "Max intensidade" no tipo de projeção configuração (Figura 2A). Salve o arquivo e nomeie-a "pilha de arquivo".
- Se o arquivo de imagem contém vários canais, dividi-lo para conservar apenas o canal com a imagem de rede hamartomas (imagem | Cor | Separação de canais).
- Verifique as configurações de pixel da imagem [imagem | Propriedades | Pixel (com "1" para a dimensão de pixel)].
- Use a ferramenta de plano de fundo de subtrair (processo | Subtrair o fundo) para remover o fundo de biocytin de rotulagem. Use a função preview para definir o raio de bola rolando, que geralmente é fixado em 50 pixels (Figura 2B).
- Use a ferramenta de outliers remover (processo | Ruído | Remover Outliers) se for necessário após a etapa de plano de fundo de subtrair. Selecione "Bright" no cenário "Que Outliers" (Figura 2). Use a função preview para definir o raio e limiar. Cuidado com esta ferramenta, já que ele pode borrar os dados, conforme mostrado na Figura 2D.
Nota: Esta ferramenta remove as pequenas manchas causadas por depósitos inespecíficos de streptavidine (como indicado pelas setas na Figura 2B e 2C brancas antes e após o tratamento, respectivamente). - Ajustar o limite (imagem | Ajustar | Limiar). Selecione o modo "Padrão" e "B & W" (Figura 2E). Clique em "Aplicar".
Nota: Ajuste automático pode ser usado, mas ajuste manual com as duas barras de controle deslizante é preferível. O objetivo desta etapa é reduzir o ruído ao máximo sem perder quaisquer pilhas etiquetadas. - Converter a imagem em uma imagem binária com a ferramenta de processo binário (processo | Binário | Faça binário) como mostrado na Figura 2F. Salve o arquivo como um arquivo TIFF e denomine "arquivo binário".
- Contagem de células
- Verifique a configuração da função de medida (Analyze | Conjunto de medição). Selecione a opção "Baricentro".
- Use a ferramenta "Analisar partículas" no arquivo"binário" (Figura 3A) produzidos na etapa anterior (Analyze | Analisar a partícula) (Figura 3 , à esquerda). Selecione "Contornos" no cenário do "Show" (Figura 3). Isso gera um novo arquivo que mostra o resultado da detecção (Figura 3B).
- Jogar com os parâmetros: tamanho (para detectar apenas células, usar valores entre 30 e 6000) e circularidade (para determinar um intervalo entre 0 e 1, em que "1" define um círculo perfeito e "0" uma forma aleatória) para refinar a deteção (Figura 3, deixada a porção). Execute a deteção clicando em "Okey".
Nota: Duas tabelas aparecerá após a detecção: 1) um quadro intitulado "Resumo" que fornece o número de células detectadas e 2) uma mesa intitulada "Resultados" (Figura 3, certa porção) que fornece as coordenadas x e y de cada célula. - Copiar os valores e colá-los em um aplicativo de planilha. Salve esta tabela sob a nome "deteção tabela". Um arquivo com uma trama de células detectadas também será exibido (Figura 3B). Salve este arquivo como um arquivo TIFF sob o nome "arquivo de detecção".
- Se um grupo de células de 2 ou mais rotulados na rede é detectado como uma única célula com a ferramenta de partículas de analisar porque eles são muito perto um do outro, use a ferramenta de bacias hidrográficas (processo | Binário | Bacia hidrográfica) sobre a imagem binária antes de aplicar as partículas de analisar a ferramenta e refazer os passos de partículas de analisar.
Nota: A ferramenta de bacia hidrográfica cria uma delimitação de 1 pixel de largura entre células extremamente estreita. Uma lata de plug-in de contador de célula ser usado quando a rotulagem é inequívoco e pode ser realizado manualmente.
- Área de rede hamartomas de medição
- Para medir a área de superfície das redes, use o arquivo de detecção usando imagem J.
- Use a ferramenta de seleção de polígono (à esquerda, clique no botão na barra de ferramentas para selecioná-lo) para rastrear um polígono que conecta todas as células localizado na periferia externa da rede (Figura 4A). Clique com o botão esquerdo para iniciar o rastreamento do polígono e clique com o botão direito para fechá-lo.
Nota: Este polígono será definido como uma região de interesse (ROI) e sua superfície será medida para determinar a área de superfície da rede. - Abra a janela de medição conjunto (Analyze | Conjunto de medição) e selecione a opção "Área". Abra o Gerenciador de ROI (analisar | Ferramentas | ROI Manager) (Figura 4B). Em seguida, adicionar o polígono rastreado no ROI Manager clicando em 'Adicionar ' (Figura 4B) e executar a medição clicando em ' medida ' no Gerenciador de ROI.
Nota: A medição de área será exibido em uma tabela e ser expressa em pixels. Não esqueça de converter esse valor com o fator de conversão para o microscópio usado para obter um valor em μm2.
- Determinação do vetor direção principal
- Determinação da célula remendada
- Abra o arquivo de pilha no ImageJFIJI e identificar a célula remendada no arquivo pilha com base em sua forte intensidade de rotulagem (Figura 4). Em seguida, abra o arquivo chamado "mesa da deteção" em um aplicativo de planilha e encontrar o número de associados para a célula remendada e suas coordenadas correspondentes.
- Se não for possível determinar com precisão a célula remendada, cercar a área onde o depósito de biocytin é mais denso na rede imagem de células acopladas, usando a ferramenta polígono em ImageJFIJI, e referir-se a esta posição como que da célula remendada (Figura 4).
- Use o Gerenciador de ROI (analisar | Ferramentas | ROI Manager). Em seguida, desenhe um ROI na localidade de célula remendado e adicioná-lo para o ROI Manager (veja Figura 4B).
- Definir uma medição (Analyze | Conjunto de medição) e selecione a opção "Baricentro".
- No Gerenciador de ROI, clique na "Medida" para obter as coordenadas do centroide da área rastreada. Use estas coordenadas como ponto de referência para esta rede específica.
- Tradução de referencial
- Calcule as coordenadas de cada célula em referência a remendado astrocyte usando a seguinte fórmula:

Com: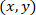 como as coordenadas para uma determinada célula;
como as coordenadas para uma determinada célula; 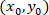 como as coordenadas da célula remendada (ou o ponto referencial da rede); e
como as coordenadas da célula remendada (ou o ponto referencial da rede); e 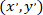 como as coordenadas para uma determinada célula no novo referencial.
como as coordenadas para uma determinada célula no novo referencial.
Nota: Expressar as coordenadas de cada célula em referência a astrocyte remendado é um passo importante no cálculo de vetores da astrocyte remendado. Tenha cuidado ao usar o ImageJFIJI que o referencial para qualquer imagem está localizado no canto superior esquerdo da imagem.
- Calcule as coordenadas de cada célula em referência a remendado astrocyte usando a seguinte fórmula:
- Determinação do principal vetor de orientação preferencial
- Calcule as coordenadas do principal vetor de orientação preferencial com a seguinte fórmula:
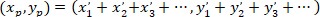
Com: (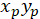 ) como as coordenadas do principal vetor de orientação preferencial; e
) como as coordenadas do principal vetor de orientação preferencial; e 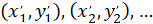 como as coordenadas de cada célula da rede obtida com o remendado de célula como referencial.
como as coordenadas de cada célula da rede obtida com o remendado de célula como referencial.
Nota: Para cada célula na rede, um vetor é determinado em relação às coordenadas do astrocyte remendado. O principal vetor de orientação preferencial da rede é a soma de todos estes vetores. - Divida o comprimento do vetor principal (fornecido pelas coordenadas obtidas usando a fórmula acima) pelo número de células na rede menos um (desde que as coordenadas da célula remendada não estão incluídas), para normalizar os valores e permitir comparações entre redes. Uma visão esquemática desta análise é apresentada na Figura 5.
- Calcule as coordenadas do principal vetor de orientação preferencial com a seguinte fórmula:
- Determinação da célula remendada
- Colocação das redes analisadas no núcleo de interesse
- Alinhamento de 20 X e 4 imagens X
- Para determinar a posição de cada rede no núcleo de interesse (NVsnpr), use a imagem de X 4. Abra a imagem de X 4 com um editor de imagens vetoriais.
- Selecione a imagem de X 4 e modificar seu tamanho, multiplicando-o por 5. A janela de dimensão está localizada na parte direita da barra de ferramentas horizontal superior. Por exemplo, para uma imagem X 4 que tem sido amostradas em 800 x 800 pixels, alterar a resolução de amostragem para 4000 x 4000 pixels (para definir a unidade de trabalho em pixels, vá para "Instalação do documento" sob a aba de arquivo: arquivo | ' Configurar documento '). Exporte o arquivo no formato TIFF e nome "redimensionado 4 X".
- Alinhe o canto superior esquerdo da imagem com o canto inferior esquerdo do documento Adobe Illustrator.
Nota: O software fornece as coordenadas de um ponto referencial do canto inferior esquerdo. - Abra a imagem de X 20 da rede. Use o ' arquivo binário ' ou ' arquivo de detecção ' porque eles são mais fáceis de alinhar. Então, jogar com a ferramenta de opacidade na 'arquivo binário ' da imagem X 20 para alinhá-lo com o re-tamanho 4 X imagem.
Nota: A ferramenta de opacidade é na barra de ferramentas horizontal superior. - Quando o alinhamento, selecione e segure a ferramenta de pipeta para a ferramenta ' medida ' aparece e selecione-o na barra de ferramentas à esquerda.
- Usar a ferramenta ' medida ' clique no canto superior esquerdo da imagem X 20 para obter as coordenadas do 20 X referencial aponte para o redimensionada 4 X imagem.
Nota: Estas coordenadas, que são referidas como os 20 X referencial (20XR na Figura 5), será útil para expressar a posição de cada rede em um desenho esquemático do núcleo.
- Normalização do núcleo de interesse
Nota: Para resumir os dados, é usada uma normalização do núcleo (NVsnpr) como um retângulo. Os passos são descritos abaixo.- Abra o arquivo redimensionado de 4x em ImageJFIJI e usar a ferramenta polígono para cercar o núcleo (NVsnpr). Usar a imagem com luz transmitida, se as bordas do núcleo não são capazes de ser visto; Nesse caso, lembre-se de redimensioná-la primeiro.
- Abra o Gerenciador de ROI e adicionar o ROI desenhado. Em "Conjunto de medição", selecione a opção ' retângulo delimitador '.
Nota: A opção "Bounding Rectangle" calcula o menor retângulo ao redor do núcleo desenhado. - Clique em "Medida".
Nota: Uma tabela é exibida com BX e BY, as coordenadas do canto superior esquerdo do retângulo: ' retângulo posição ' e fornece a largura e altura. BX e BY são as coordenadas do retângulo delimitador referencial que são referidas como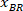 e
e 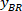 .
.
- Expressão de cada posição de rede no núcleo normalizado
- Expressar as coordenadas de cada célula com o 4 X referencial (quadrado preto na Figura 5) usando a seguinte fórmula:

Onde: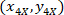 são as coordenadas de célula com o 4 X referenciais;
são as coordenadas de célula com o 4 X referenciais; 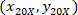 são as coordenadas da célula com o 20 X referencial (quadrado laranja na Figura 5); e
são as coordenadas da célula com o 20 X referencial (quadrado laranja na Figura 5); e 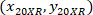 são as coordenadas dos 20 X ponto referencial na imagem 4 X (20XR).
são as coordenadas dos 20 X ponto referencial na imagem 4 X (20XR). - Expressar as coordenadas de célula no retângulo delimitador referencial (azul na Figura 5) usando a seguinte fórmula:
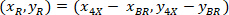
Onde: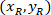 são coordenadas de célula no retângulo delimitador referencial;
são coordenadas de célula no retângulo delimitador referencial; 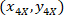 são as coordenadas de célula no 4 X referenciais; e
são as coordenadas de célula no 4 X referenciais; e  são as coordenadas do retângulo delimitador referencial na imagem X 4.
são as coordenadas do retângulo delimitador referencial na imagem X 4. - Transforme as coordenadas de célula no retângulo delimitador referencial para a porcentagem de largura e altura do retângulo delimitador usando a seguinte fórmula:
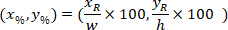
Onde: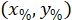 são a célula coordena a porcentagem da largura e altura do retângulo delimitador;
são a célula coordena a porcentagem da largura e altura do retângulo delimitador; 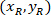 são coordenadas de célula no retângulo delimitador referencial;
são coordenadas de célula no retângulo delimitador referencial;  é a largura do retângulo delimitador medido acima no protocolo; e
é a largura do retângulo delimitador medido acima no protocolo; e 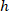 é a altura do retângulo delimitador medida acima no protocolo.
é a altura do retângulo delimitador medida acima no protocolo. - Para representar todas as redes na mesma figura, leve em conta a orientação da fatia (lado esquerdo ou direito). Para padronizar os dados, o referencial é aplicado no lado esquerdo da fatia. Transfira as coordenadas de rede do lado direito para o lado esquerdo, aplicando a fórmula a seguir somente a coordenadas x:
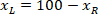
Onde: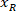 é a coordenada x do lado direito da fatia; e
é a coordenada x do lado direito da fatia; e 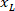 é o novo x-coordenada expressado no referencial no lado esquerdo da fatia.
é o novo x-coordenada expressado no referencial no lado esquerdo da fatia.
Nota: Como alternativa, a foto no ImageJFIJI antes da análise do espelho (imagem | Transformar | Inverta horizontalmente). - Para expressar as coordenadas do principal vetor de orientação preferencial, siga os mesmos passos com as coordenadas de célula (etapas 6.5.3.1 para 6.5.3.4).
Nota: A expressão das coordenadas em percentagens permite uma compilação dos dados como um enredo único, no qual o núcleo (NVsnpr) é projetado como um retângulo.
- Expressar as coordenadas de cada célula com o 4 X referencial (quadrado preto na Figura 5) usando a seguinte fórmula:
- Alinhamento de 20 X e 4 imagens X
- Estudo da diferença angular do principal vetor de direção preferencial
Nota: A diferença angular de uma rede de hamartomas é usada para determinar se sua orientação preferencial é em direção ao centro do núcleo de interesse. Para calcular a diferença angular (α na Figura 5), que é o ângulo entre o principal vetor de direção preferencial da rede (linha PD, vermelho na Figura 5) e a linha que conecta P c, aplicar o teorema de Al-Kashi no triângulo PDC (ver baixo-relevo de Métodos complementarese Figura 5 ).- Em primeiro lugar, calcule os comprimentos diferentes usando a aplicação do teorema de Pitágoras em um cartesiano referencial utilizando a seguinte equação:
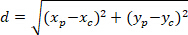
Onde: as coordenadas de P e C são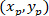 e
e  , respectivamente, no retângulo delimitador referencial (calculado acima).
, respectivamente, no retângulo delimitador referencial (calculado acima). - Determine a diferença angular em radianos usando a seguinte fórmula:
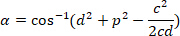
- Converta a diferença angular em graus (por exemplo, com a função de graus no software Excel).
- Compilar todas as diferenças angulares traçando-os em gráficos de barras verticais (Figura 7 e 7D) e determinar se existe uma orientação preferencial de redes hamartomas.
- Em primeiro lugar, calcule os comprimentos diferentes usando a aplicação do teorema de Pitágoras em um cartesiano referencial utilizando a seguinte equação:
Resultados
Acoplamento entre as células do cérebro não é estático, mas prefiro dinamicamente regulamentado por muitos fatores. Os métodos descritos foram desenvolvidos para redes hamartomas reveladas sob diferentes condições de analisar e entender sua organização em NVsnpr. Estes resultados foram já publicados1. Biocytin enchimento de astrócitos único foram realizadas na parte dorsal do NVsnpr em três condições diferentes: em repouso (em controlar as condiçõ...
Discussão
Existe um número de métodos eletrofisiológicos para avaliar funcional acoplamento entre astrócitos23,24. No entanto, esses métodos não fornecem informações sobre o arranjo anatômico das redes hamartomas. Vários estudos já mostraram que "corante ou marcador-acoplamento", como feito aqui, ocorre apenas em uma fração de juntamente com as células que são detectadas por métodos eletrofisiológicos25,...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho é financiado por institutos canadenses de pesquisa saúde, Grant/prêmio número: 14392.
Materiais
| Name | Company | Catalog Number | Comments |
| NaCl | Fisher Chemicals | S671-3 | |
| KCl | Fisher Chemicals | P217-500 | |
| KH2PO4 | Fisher Chemicals | P285-500 | |
| MgSO4 | Fisher Chemicals | M65-500 | |
| NaHCO3 | Fisher Chemicals | S233-500 | |
| C6H12O6 Dextrose anhydrous | Fisher Chemicals | D16-500 | |
| CaCl2 dihydrated | Sigma | C70-500 | |
| Sucrose | Sigma | S9378 | |
| D-gluconic acid potassium salt | Sigma | G45001 | |
| MgCl2 anhydrous | Sigma | M8266 | |
| HEPES | Sigma | H3375 | |
| EGTA | Sigma | E4378 | |
| ATPTris Salt | Sigma | A9062 | |
| GTPTris Salt | Sigma | G9002 | |
| Biocytin | Sigma | B4261 | |
| Carbenoxolone disodium salt | Sigma | C4790 | |
| avidin-biotin complex : ABC kit | Vestor laboratories | PK-4000 | |
| Streptavidine-alexa 594 | Molecular Probes | S11227 | |
| Triton | Fisher Chemicals | BP151-500 | |
| Xylene | Fisher Chemicals | X5-1 | |
| Aqueous mounting medium 1 : Fluoromount-G | SouthernBiotech | 0100-01 | |
| Toluen-based synthetic resin mounting medium : Permount | Fisher Chemicals | SP15-100 | |
| Slide Drying Bench | Fisherbrand | 11-474-470 | |
| Vibratome | Leica | VT 1000S | |
| Microscope cover glass | Fisherbrand | 12-544A | |
| Microscope slide ColorFrost | Fisherbrand | 12-550-413 | |
| PFA | Fisherchemicals | 04042-500 | |
| Olympus FluoView FV 1000 Confocal microscope | Olympus | ||
| 40X water-immersion lens | Olympus | LUMPLFLN40XW | |
| 20X water-immersion lens | Olympus | XLUMPLFL20XW | |
| 4X water-immersion lens | Olympus | XLFLUOR4X/340 | |
| Micropipette puller | Sutter Instrument | P97 | |
| Micromanipulator | Sutter Instrument | MP 225 | |
| Camera CCD | Sony | CX-ST50 | |
| Black and white monitor | Sony | SSM-125 | |
| Digidata | Molecular devices | 1322A | |
| Patch Clamp amplifier | Axon instrument | Mulitclamp 700A | |
| Electrophysiology acquisition software | Molecular devices | pClamp 8 | |
| Electrophysiology analysis software | Molecular devices | Clampfit 8 | |
| Imaging analysis software | ImageJFIJI | Open source software. FIJI version including plug in package. | |
| Vector image editor | Adobe | Illustrator CS4 | |
| Spreadsheet application | Microsoft Office | Excel 2010 |
Referências
- Condamine, S., Lavoie, R., Verdier, D., Kolta, A. Functional rhythmogenic domains defined by astrocytic networks in the trigeminal main sensory nucleus. Glia. 66 (2), 311-326 (2018).
- Verkhratsky, A., Orkand, R. K., Kettenmann, H. Glial calcium: homeostasis and signaling function. Physiological Review. 78 (1), 99-141 (1998).
- Christensen, R. K., Petersen, A. V., Perrier, J. F. How do glial cells contribute to motor control?. Current Pharmaceutical Design. 19 (24), 4385-4399 (2013).
- Verkhratsky, A., Steinhauser, C. Ion channels in glial cells. Brain Research Review. 32 (2-3), 380-412 (2000).
- Harada, K., Kamiya, T., Tsuboi, T. Gliotransmitter Release from Astrocytes: Functional, Developmental, and Pathological Implications in the Brain. Frontiers Neuroscience. 9, 499 (2015).
- Montero, T. D., Orellana, J. A. Hemichannels: new pathways for gliotransmitter release. Neuroscience. 286, 45-59 (2015).
- Araque, A., et al. Gliotransmitters travel in time and space. Neuron. 81 (4), 728-739 (2014).
- Augustin, V., et al. Functional anisotropic panglial networks in the lateral superior olive. Glia. 64 (11), 1892-1911 (2016).
- Houades, V., Koulakoff, A., Ezan, P., Seif, I., Giaume, C. Gap junction-mediated astrocytic networks in the mouse barrel cortex. Journal of Neuroscience. 28 (20), 5207-5217 (2008).
- Roux, L., Benchenane, K., Rothstein, J. D., Bonvento, G., Giaume, C. Plasticity of astroglial networks in olfactory glomeruli. Proceedings of the National Academy of Science of the United State of America. 108 (45), 18442-18446 (2011).
- Morquette, P., et al. An astrocyte-dependent mechanism for neuronal rhythmogenesis. Nature Neuroscience. 18 (6), 844-854 (2015).
- Brocard, F., Verdier, D., Arsenault, I., Lund, J. P., Kolta, A. Emergence of intrinsic bursting in trigeminal sensory neurons parallels the acquisition of mastication in weanling rats. Journal of Neurophysiology. 96 (5), 2410-2424 (2006).
- Anders, S., et al. Spatial properties of astrocyte gap junction coupling in the rat hippocampus. Philosophical Transactions of the Royal Society of London. Series B, Biological Science. 369 (1654), (2014).
- Houades, V., et al. Shapes of astrocyte networks in the juvenile brain. Neuron Glia Biology. 2 (1), 3-14 (2006).
- Rouach, N., Koulakoff, A., Abudara, V., Willecke, K., Giaume, C. Astroglial metabolic networks sustain hippocampal synaptic transmission. Science. 322 (5907), 1551-1555 (2008).
- Claus, L., et al. Barreloid Borders and Neuronal Activity Shape Panglial Gap Junction-Coupled Networks in the Mouse Thalamus. Cerebral Cortex. 28 (1), 213-222 (2018).
- Cameron, M. A., et al. Prolonged Incubation of Acute Neuronal Tissue for Electrophysiology and Calcium-imaging. Journal of Visualized Experiments. (120), (2017).
- Kafitz, K. W., Meier, S. D., Stephan, J., Rose, C. R. Developmental profile and properties of sulforhodamine 101--Labeled glial cells in acute brain slices of rat hippocampus. Journal of Neuroscience Methods. 169 (1), 84-92 (2008).
- Neher, E. Correction for liquid junction potentials in patch clamp experiments. Methods in Enzymology. , 123-131 (1992).
- Giaume, C., Leybaert, L., Naus, C. C., Saez, J. C. Connexin and pannexin hemichannels in brain glial cells: properties, pharmacology, and roles. Frontiers in Pharmacology. 4, 88 (2013).
- Torres, A., et al. Extracellular Ca(2)(+) acts as a mediator of communication from neurons to glia. Science Signaling. 5 (208), ra8 (2012).
- Ye, Z. C., Wyeth, M. S., Baltan-Tekkok, S., Ransom, B. R. Functional hemichannels in astrocytes: a novel mechanism of glutamate release. Journal of Neuroscience. 23 (9), 3588-3596 (2003).
- Ma, B., et al. Gap junction coupling confers isopotentiality on astrocyte syncytium. Glia. 64 (2), 214-226 (2016).
- Meme, W., Vandecasteele, M., Giaume, C., Venance, L. Electrical coupling between hippocampal astrocytes in rat brain slices. Neuroscience Research. 63 (4), 236-243 (2009).
- Ransom, B. R., Kettenmann, H. Electrical coupling, without dye coupling, between mammalian astrocytes and oligodendrocytes in cell culture. Glia. 3 (4), 258-266 (1990).
- Audesirk, G., Audesirk, T., Bowsher, P. Variability and frequent failure of lucifer yellow to pass between two electrically coupled neurons in Lymnaea stagnalis. Journal of Neurobiology. 13 (4), 369-375 (1982).
- Ewadinger, N., Syed, N., Lukowiak, K., Bulloch, A. Differential Tracer Coupling between Pairs of Identified Neurones of the Mollusc Lymnaea Stagnalis. Journal of Experimental Biology. 192 (1), 291-297 (1994).
- Griemsmann, S., et al. Characterization of Panglial Gap Junction Networks in the Thalamus, Neocortex, and Hippocampus Reveals a Unique Population of Glial Cells. Cerebral Cortex. 25 (10), 3420-3433 (2015).
- Kuwajima, T., et al. ClearT: a detergent- and solvent-free clearing method for neuronal and non-neuronal tissue. Development. 140 (6), 1364-1368 (2013).
- Gourine, A. V., et al. Astrocytes control breathing through pH-dependent release of ATP. Science. 329 (5991), 571-575 (2010).
- Forsberg, D., Ringstedt, T., Herlenius, E. Astrocytes release prostaglandin E2 to modify respiratory network activity. eLife. 6, (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados