Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Visualización de dinámicas diffusionales de nanorods de oro en la membrana celular mediante microscopía de campo oscuro de nanopartícula única
En este artículo
Resumen
Aquí, mostramos el uso de microscopía tradicional de campo oscuro para monitorear la dinámica de los nanorods de oro (AuNRs) en la membrana celular. La ubicación y orientación de los AuNR únicos se detectan utilizando ImageJ y MATLAB, y los estados difusivos de los AUNR se caracterizan por un solo análisis de seguimiento de partículas.
Resumen
El análisis de la dinámica diffusional de las nanopartículas en la membrana celular desempeña un papel importante en la mejor comprensión del proceso de absorción celular y proporciona una base teórica para el diseño racional de la administración de nanomedicación. El análisis de seguimiento de partículas únicas (SPT) podría sondear la posición y orientación de nanopartículas individuales en la membrana celular, y revelar sus estados traslacionales y rotacionales. Aquí, mostramos cómo utilizar la microscopía tradicional de campo oscuro para monitorear la dinámica de los nanorods de oro (AuNRs) en la membrana celular viva. También mostramos cómo extraer la ubicación y orientación de auNRs utilizando ImageJ y MATLAB, y cómo caracterizar los estados difusos de auNRs. El análisis estadístico de cientos de partículas muestra que un solo AuNRs realiza movimiento browniano en la superficie de la membrana celular U87 MG. Sin embargo, el análisis individual de larga trayectoria muestra que los AUNR tienen dos tipos de valores de movimiento claramente diferentes en la membrana, a saber, el transporte de largo alcance y el confinamiento de área limitada. Nuestros métodos SPT se pueden utilizar potencialmente para estudiar la difusión de partículas superficiales o intracelulares en diferentes células biológicas y pueden convertirse en una poderosa herramienta para investigaciones de mecanismos celulares complejos.
Introducción
La dinámica de las nanopartículas (NPs) en la membrana está estrechamente asociada con el proceso de absorción celular, que es esencial para la comprensión de las funciones celulares, infecciones virales o bacterianas y el desarrollo de sistemas de administración nanomédica artificial1,2. La técnica de seguimiento de partículas únicas (SPT) es una herramienta robusta para caracterizar los comportamientos heterogéneos de los NPs3,4. En general, la membrana celular es fluida, lo que significa que los componentes como proteínas y lípidos pueden moverse lateralmente en el plano de membrana plasmática5,6,7. La complejidad espaciotemporal de la organización y estructura de la membrana puede conducir a la heterogeneidad espaciotemporal de la interacción entre los NPs y la membrana. Por lo tanto, la visualización directa del movimiento de los NPs en la membrana requiere una alta resolución espacial y temporal.
Microscopía de seguimiento de partículas únicas que monitorea la localización de partículas individuales en células vivas con una resolución espacial de decenas de nanómetros y una resolución de tiempo de milisegundos ha sido bien desarrollada para estudiar los NPs o moléculas de membrana dinámica8,9. Las técnicas de imágenes microscópicas basadas en fluorescencia se han convertido en valiosas herramientas para observar NPs/moléculas en entornos celulares vivos9,10,11,12. Por ejemplo, la microscopía de fluorescencia de reflexión interna total, que muestra capas delgadas (~100 nm) de la muestra en la interfaz de sustrato/solución con una alta resolución espaciotemporal ha sido ampliamente utilizada en estudios de moléculas de membrana dinámica13,14. Sin embargo, las desventajas inherentes de los fluoróforos individuales, como la baja intensidad y el fotobleaching irreversible rápido reducen la precisión y la duración del seguimiento13. Por lo tanto, los NPs plásmidos no fluorescentes, que reemplazan a las sondas fluorescentes, han atraído cada vez más atención en estudios de imágenes a largo plazo debido a sus características ópticas únicas15. Basándose en las señales de dispersión de las sondas np plásmidas, se han utilizado varios tipos de tecnologías ópticas de imágenes microscópicas para estudiar el mecanismo de procesos biológicos, como microscopía de campo oscuro (DFM)16,microscopía de dispersión interferométrica (iSCAT)17 y microscopía de contraste de interferencia diferencial (DICM)18. Además, la dinámica de movimiento y rotación de auNRs se puede obtener utilizando DFM y DICM18,19,20,21,22. Normalmente, en un experimento SPT, el microscopio óptico registra el movimiento del objeto y, a continuación, se analiza mediante métodos de análisis SPT3. Las trayectorias resueltas en el tiempo y los ángulos de orientación generados por los NPs individuales son normalmente estocásticos y heterogéneos, por lo que es necesario presentar abundante información dinámica con varios métodos de análisis.
Aquí, proporcionamos un protocolo integrado que monitorea la dinámica de auNRs en la membrana celular utilizando DFM, extrae la ubicación y orientación de auNRs con ImageJ y MATLAB y caracteriza la difusión de auNRs con métodos de análisis SPT. Como demostración, aquí mostramos cómo utilizar el protocolo SPT para visualizar dinámicas de AUNRs no modificados (CTAB-AuNRs, sintetizados por molécula de bromuro de amonio de cetiltrimetillammonium como agente protector) en la membrana celular U87 MG. Se ha demostrado que los TTAB-AUNR pueden adsorberar proteínas en el entorno biológico, moverse sobre la membrana celular y luego entrar en las células2,20,22. Células de MG U87 es el tumor más común y más maligno del sistema nervioso central, y sus receptores de membrana se expresan anormalmente. Los receptores de membrana pueden interactuar con proteínas en auNRs, que influyen en la dinámica de auNRs. Nuestro protocolo es generalmente aplicable a otros experimentos SPT en el campo de la biología.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Cultivo celular
- Preparar un medio completo para las células MG U87 añadiendo suero bovino fetal (concentración final 10%) y penicilina-estreptomicina (concentración final 1%) al medio esencial mínimo (MEM). Utilice un plato de cultivo celular de plástico para la subcultura celular.
- Células de paso de 2 a 3 veces a la semana.
- Retire el medio de cultivo y enjuague la capa celular con la solución salina tamponada por fosfato (D-PBS) de Dulbecco 2 ~ 3 veces cuando confluente (80% ~ 90%).
- Agregue de 1.0 a 2.0 ml de solución Trypsin-EDTA al plato de cultivo celular y observe las células bajo un microscopio invertido hasta que las células se vuelvan redondas (3 ~ 5 min).
- Añadir 3,0 ml de medio completo preparado y dispersar las células pipeteando suavemente.
- Agregue la suspensión celular (1 ml) a un nuevo plato de cultivo con el medio celular fresco (3 ml) y vuelva a usar las células.
- Mantener las células a 37 °C y 5% CO2 en una atmósfera humidificada.
2. Preparación de diapositivas de microscopio
NOTA: Células U87 MG de tercera a décima generación con alta actividad se utilizan en experimentos SPT.
- Esterilizar 22 mm × 22 mm de cubiertas ya limpiadas con solución Piranha sumergiéndose en etanol (99,9%).
- Utilice fórceps para eliminar el deslizamiento de la cubierta de la solución de etanol (paso 2.1) y esterilizar quemando etanol en la llama. Una vez que se queme todo el etanol, coloque los cubremas en un plato de cultivo celular de plástico (35 x 10 mm) lleno de 2 ml de medio celular (sin rojo fenol).
- Agregue 50 μL de la suspensión celular del paso 1.2.3 en el control de cubiertas y empuje suavemente el plato hacia adelante y hacia atrás y hacia la izquierda y la derecha para distribuir uniformemente las células. Colóquelo en una atmósfera humidificada.
- Cuando las células U87 MG en el deslizamiento de cubierta alcancen entre un 20% y un 40% de confluencia (~12 h), agregue 20 μL de CTAB-AuNRs (138 pM) en el plato y ex disperse. Incubar en ambiente humidificado durante 5 min.
- Añadir 100 μL del medio de cultivo (sin rojo fenol) del plato en el paso 2.4 en la ranura de la diapositiva de vidrio ranurado(Figura 1)que está precleaned con solución Piranha.
- Saque el tubo de cobertura del plato e invertida en la parte superior de la ranura de la diapositiva de vidrio del microscopio(Figura 1). Sellar con esmalte de uñas, dejar que se seque y colocarlo en el escenario para realizar experimentos SPT.
3. Realización de experimentos de seguimiento de partículas individuales con microscopía de campo oscuro (Figura 1).
- Coloque una gota de aceite en el condensador de campo oscuro sumergido en aceite (NA 1.43-1.20) y gire la perilla para hacer que el condensador entre en contacto con el tobogán de vidrio.
- Coloque una gota de aceite en la parte superior del vidrio de la cubierta y gire la perilla de enfoque para hacer que el objetivo de inmersión de aceite de 60x (NA 0.7–1.25) toque el aceite.
- Encienda la fuente de luz y gire ligeramente la perilla de enfoque para enfocar el plano de imágenes.
NOTA: En el campo de visión, el fondo es negro, las celdas son brillantes y los CTAB-AuNRs (relación de aspecto~2:1, Figura 2)son pequeños puntos de dispersión de color (rojo, amarillo o verde). - Capture la luz de dispersión de muestras mediante una cámara CMOS de color. Haga clic en"Icono de cámara"en el software para grabar y exportar el formato TIFF para guardar imágenes.
4. Adquisición de datos
- Extraer una sola trayectoria a largo plazo
- Opere como se describe en la Figura 3 para convertir las imágenes de campo oscuro de la serie temporal del modo "Color RGB" al modo "8 bits". En la imagen J haga clic en Imagen | Tipo | 8 bits. Para ajustar el contraste, haga clic en Imagen | Ajustar | Brillo | Contraste.
- Seleccione una partícula de destino y corte los fondos de la serie temporal boxeando y eliminando el fondo con"Ctrl+X".
- Abra la ventana detección de partículas y enlace de partículas haciendo clic en"Plugins | Rastreador de partículas | clásico Rastreador de partículas".
- Establezca Radio en 6, Corte en 0 y Percentil en 0.01%.
NOTA: Para detectar la partícula, ajuste por encima de tres parámetros con la ayuda de Vista previa. Asegúrese de que el radio sea ligeramente mayor que la partícula objetivo y más pequeño que la separación entre partículas más pequeña. Percentil es el límite más bajo de distribución de intensidad que para ser partículas candidatas. - Establezca el Rango de enlace en 5 y Desplazamiento en 10.
NOTA: Para vincular la partícula entre fotogramas adyacentes consecutivos, ajuste los dos parámetros anteriores. El desplazamiento es el píxel máximo que una partícula puede moverse entre dos fotogramas siguientes, y rango de vínculos es el número de fotogramas consecutivos a tener en cuenta al determinar la mejor coincidencia correspondiente. - Haga clic en "Aceptar" para abrir la ventana Resultados de ParticleTracker para ver los resultados.
- Haga clic en "Visualizar todas las trayectorias" para inspeccionar las trayectorias generadas.
- Haga clic en el menú "Volver avincular partículas"en la parte superior para volver a vincular las partículas detectadas con diferentes parámetros de rango de enlace y percentil, si la trayectoria generada por el software no coincide con la trayectoria móvil del AuNR.
- Haga clic en"Guardar informe completo"para guardar los resultados si la trayectoria generada por el software y la trayectoria móvil del AuNR coinciden.
NOTA: En la figura 4se muestra un ejemplo de extracción de una sola trayectoria a largo plazo con la imagen J.
- Extracción de trayectorias múltiples
NOTA: En la figura 5se muestra un ejemplo de extracción de múltiples trayectorias con Fiji. Hay varias etapas, y cada etapa constituye un paso en el proceso de seguimiento. El resultado de cada paso se muestra inmediatamente, lo que permite al usuario volver a la configuración de reajuste cuando la salida no es satisfactoria.- Opere como se describe en la Figura 3 para convertir las imágenes de campo oscuro de la serie temporal del modo "Color RGB" al modo "8 bits". En la ruta imagen J haga clic en "Imagen | Tipo | 8 bits". Para ajustar su contraste, haga clic en "Imagen | Ajustar | Brillo | Contraste".
- Abra el panel de inicio haciendo clic en "Plugins | Seguimiento | TrackMate". Haga clic en "Siguiente".
- Elija el"detector loG"en el menú desplegable y haga clic en "Siguiente".
- Ajuste los parámetros del panel de configuración del detector. Establezca El diámetro estimado del blob en 10, establezca Umbral en 0 y seleccione"Realizar localización de subpíxeles".
- Haga clic en "Siguiente" para abrir el panel de filtrado de puntos inicial.
NOTA: Se pueden ajustar varios parámetros para optimizar aún más los puntos de destino. En este ejemplo, no se ajustaron otros parámetros. - Haga clic en "Siguiente" y seleccione "Visualización de HyperStack" en el menú desplegable.
- Haga clic en "Siguiente" para abrir el panel Filtrado de puntos. Establezca la calidad por encima de 1,88, X por encima de 38,86, Y por encima de 56,54.
- Haga clic en "Siguiente" y seleccione "Simple LAP tracker" en el menú desplegable. Configure el rastreador LAP simple ajustando tres parámetros, es decir, la distancia máxima de vinculación = 15, distancia máxima de cierre de huecos = 15 y espacio de fotogramas máximo de cierre de brecha = 5.
NOTA: La distancia máxima de vinculación es el desplazamiento máximo de un punto entre dos fotogramas. La distancia máxima de cierre de huecos es el desplazamiento máximo de dos segmentos. El espacio máximo de fotogramas de cierre de huecos es el marco más grande entre dos puntos que se va a cerrar. - Haga clic en "Siguiente" para realizar un seguimiento. Cuando haya terminado, continúe haciendo clic en "Siguiente".
- Establezca filtros en pistas, como el conjunto Número de puntos en la pista por encima de 300.
- Siga haciendo clic en "Siguiente" hasta que se abra el panel de guardado final. Seleccione"Exportar pistas al archivo XML"en el menú desplegable y, a continuación, haga clic en"Ejecutar"para guardarlo en formato csv.
- Valores de R/G
NOTA: Las intensidades de dispersión de los AuNRs dirigidos en los canales R y G se obtienen a partir de imágenes de campo oscuro de color mediante un código escrito en MATLAB (https://github.com/fenggeqd/JOVE-2020/tree/master/RGandPolarangle), y el principio de extracción se presenta en la Figura 6.- Utilice la función de xycoordination.m para encontrar la coordenada de píxeles centrales del AuNR en cada fotograma según la coordenada x-y (extraída por ImageJ/Fiji).
- Utilice la función de RGextraction.m para delimitar una matriz de 3 x 3 píxeles, extraer los 9 valores de intensidad de dispersión de los canales R o G y calcular un valor medio (μ, definido como R o G).
NOTA: La matriz de 3 x 3 píxeles se centra en las coordenadas de píxel obtenidas del paso 4.3.1.
- Ángulo polar
NOTA: El ángulo polar es el ángulo entre la longitud del AuNR y el eje óptico (como se muestra en la Figura 1),que puede reflejar la dinámica de rotación espacial (eje Z) del AuNR.- Utilice la función polarangle.m para calcular los ángulos polares (ḥ) mediante el método diferencial de doble canal22,
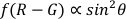 .
.
- Utilice la función polarangle.m para calcular los ángulos polares (ḥ) mediante el método diferencial de doble canal22,
5. Análisis de datos
NOTA: Un marco de análisis de datos sistemático y sólido es esencial para el rendimiento y la eficiencia de los métodos de análisis SPT. Se utiliza el software personalizado escrito en MATLAB (https://github.com/fenggeqd/JOVE-2020/tree/master/Analysis_parameters). Se utiliza un software de gráficos y análisis (consulte Tabla de materiales)para dibujar las gráficas.
- Parámetros de análisis
- Utilice scripts de csv_data_extract_dis_vel_ss.m y csv_data_MSD.m para calcular parámetros dinámicos según las fórmulas que se muestran en la Tabla 1.
NOTA: Estos parámetros se utilizan para analizar la dinámica de auNRs y constan de tres partes. (1) Parámetros relacionados con la trayectoria: desplazamiento, tamaño del paso, velocidad, radio de giro (Rg)y ángulo de giro (Ta); 2) Parámetros de MSD: coeficiente de difusión (Dt)y exponente de difusión anormal (α); y (3) Parámetros relacionados con la rotación: ángulo polar y labilidad rotacional (σ).
- Utilice scripts de csv_data_extract_dis_vel_ss.m y csv_data_MSD.m para calcular parámetros dinámicos según las fórmulas que se muestran en la Tabla 1.
| Medidas | Definición | Significado físico | |
| Desplazamiento | 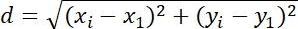 | Cambios en la posición de los objetos | |
| Tamaño del paso | 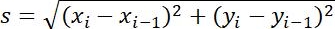 | Distancia entre dos puntos adyacentes | |
| Velocidad |  | Velocidad del movimiento de los objetos | |
| Rg | 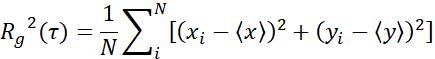 | Rango móvil de objetos en un intervalo de tiempo específico | |
| Ta | 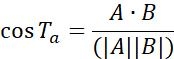 | Dirección de movimiento de objetos entre dos puntos adyacentes | |
| Msd | 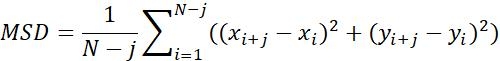 | Distancia móvil media de los objetos en un intervalo de tiempo específico | |
| Dt | 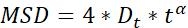 | Capacidad de difusión de objetos | |
| Α | 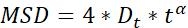 | Difusión normal (α~1) | |
| Ángulo polar |  | Información de orientación 3D de objetos | |
| Σ |  | Grado de dispersión del conjunto de datos de ángulo polar | |
Tabla 1: Tres tipos de parámetros utilizados para el análisis. Estos incluyen parámetros relacionados con la trayectoria (desplazamiento, tamaño del paso, velocidad, Rg y Ta),parámetros MSD (MSD, Dt y α) y parámetros relacionados con la rotación (ángulo polar y labilidad rotacional).
- Análisis visual de la trayectoria
NOTA: La visualización de la trayectoria puede presentar intuitivamente la heterogeneidad espaciotemporal del movimiento de partículas y la distribución de trayectoria (coordenada) de parámetros dinámicos, como la trayectoria de mapeo de tiempo, la trayectoria de mapeo Rg- y Dt- y la trayectoria de mapeo de ángulo polar. Las trayectorias cartográficas se dibujaron utilizando el software de gráficos y análisis.- Establezca la coordenada x como X, y coordinar como Y, y el tiempo (Rg,Dt,ángulo polar) como Z.
- Haga clic en "Trazar | Parcela de dispersión | Trazado de mapeo de color".
- Agregue la barra de colores.
- Análisis de MSD
NOTA: La actividad de movimiento y el modo de movimiento de las partículas se pueden obtener mediante el análisis MSD23. Cuanto mayor sea el Dt,más activo será el movimiento de difusión de las partículas. cuando α~1, las partículas realizan movimiento de difusión normal, de lo contrario realizan un movimiento de difusión anormal.- Cifras de MSD-τ
- Establezca el intervalo de tiempo (τ) como X, datos MSD como Y.
- Haga clic en "Trazar | Parcela de dispersión"
- Ajuste los datos haciendo clic en "Analizar | Ajuste | Ajuste de curva no lineal (Función: Allometricl)".
- FIGURA MSD-τ doble logarítmica
NOTA: La figura msd-τ de doble logarítmica, cuya pendiente es α y la intercepción es Dt, puede presentar intuitivamente el movimiento de partículas.- Establezca el intervalo de tiempo logarítmico como X y el MSD logarítmico como Y.
- Haga clic en "Trazar | Parcela de dispersión".
- Ajuste los datos haciendo clic en "Analizar | Ajuste | Ajuste lineal de curvas".
- Figura D-τ (MSD/4t)
NOTA: D=MSD/4τ, es una función de tiempo τ y factor de anomalía α, y la figura D-τ muestra directamente el cambio de coeficiente de difusión con el tiempo. Cuando D aumenta con el tiempo, α es mayor que 1 y las partículas hacen movimiento de superdiffusión.- Establezca el intervalo de tiempo logarítmico (τ) como X y el logarítmico D como Y.
- Haga clic en "Trazar | Parcela de dispersión".
- Ajuste los datos haciendo clic en "Analizar | Ajuste | Ajuste de curva no lineal (Función: "Allometricl")".
NOTA: Cuanto más cortas sean las trayectorias, mayor será la imprecisión de las estimaciones de difusión. En general, se obtuvieron Dt y α a través del análisis MSD-τ de tiempo de intervalo largo (> 30 τ). Sin embargo, el ajuste simple y áspero suavizará los detalles del movimiento. Por lo tanto, el análisis MSD-τ de tiempo de intervalo corto (< 10 τ) debe realizarse para analizar el comportamiento de movimiento de las partículas en poco tiempo.
- Cifras de MSD-τ
- Análisis estadístico
- Análisis estadístico de partículas múltiples
NOTA: El análisis estadístico de partículas múltiples puede reflejar el estado de movimiento de las partículas en una región espacial, lo que indica indirectamente el entorno de heterogeneidad espacial. Por ejemplo, si el histograma de Dt exhibe una distribución a gran escala o una distribución multi-picos, significa que las actividades de movimiento de las partículas son heterogéneas.- Establezca parámetros dinámicos (como Dt,Rg,desplazamiento máximo) como Y.
- Haga clic en "Trazar | Histograma".
- Haga doble clic en el histograma y establezca el tamaño de la división o el número de divisiones. Haga clic en "Aplicar".
- Análisis estadístico de partículas únicas
NOTA: El análisis estadístico de partículas individuales puede mostrar el comportamiento de movimiento de partículas individuales, lo que también refleja indirectamente la heterogeneidad espaciotemporal del entorno circundante.- Múltiples marcos
- Calcule los parámetros dinámicos (como Ta,tamaño de paso, ángulo polar) de todos los fotogramas de una sola trayectoria larga, y copie en la tabla Origin y establezca como Y.
- Haga clic en "Trazar | Histograma".
- Haga doble clic en el histograma y establezca el tamaño de la división o el número de divisiones. Haga clic en "Aplicar".
NOTA: Si tanto el tamaño de Ta como el de paso presentan una distribución de valor pequeño, las partículas realizan un movimiento de super-difusión en pequeño paso.
- Ventanas móviles
- Calcule parámetros dinámicos (como Rg,Dt)de todos los fotogramas de una sola trayectoria larga a través del método de ventana móvil (11 fotogramas), y copie en la tabla Origin y establezca como Y.
- Haga clic en "Trazar | Histograma".
- Haga doble clic en el histograma y establezca el tamaño de la división o el número de divisiones. Haga clic en "Aplicar".
- Múltiples marcos
- Análisis estadístico de partículas múltiples
- Análisis de series temporales
NOTA: El análisis estadístico puede revelar el estado de movimiento de los NPs, y el análisis de la serie temporal puede presentar el comportamiento del movimiento como un suplemento. Combinando varios parámetros de series temporales, puede discriminar el comportamiento de movimiento de los NPs en los niveles temporal y espacial.- Establezca la hora como X, parámetros de series temporales como Y (como desplazamiento, ángulo polar y Rg).
- Haga clic en "Trazar | Diagrama de varios cristales | Parcela apilada | Línea + Símbolo".
Access restricted. Please log in or start a trial to view this content.
Resultados
En el protocolo, se utilizaron los CTAB-AuNRs no modificados de 40 x 85 nm. Como se muestra en la Figura 2B,su máximo plásmido longitudinal es ~650 nm (región roja) y la resonancia transversal es de 520 nm (región verde). Las literaturas anteriores han revelado que las propiedades ópticas (como la intensidad LSPR) de los AuNRs plásmidos cambiarán significativamente con su diámetro20,22. En la Figura 2C,
Access restricted. Please log in or start a trial to view this content.
Discusión
El protocolo presentado se utiliza para estudiar la dinámica de los AUNRs en la membrana celular. El protocolo consta de cuatro partes, incluyendo imágenes microscópicas, extracción de datos, métodos de cálculo de parámetros dinámicos y análisis de datos, y cada parte es flexible y universal. Por lo tanto, hay muchas aplicaciones futuras posibles, por ejemplo, estudiando el movimiento de moléculas de membrana vinculadas a NP en la membrana, dinámica de endocitosis de receptores etiquetados np, análisis dinám...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por la Fundación Nacional de Ciencias Naturales de China con números de subvención de 21425519, 91853105 y 21621003.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| CTAB coated gold nanorods(CTAB-AuNRs) | Nanoseedz | NR-40-650 | 85 nm * 40 nm |
| Color CMOS camera | Olympus | DP74 | Japan |
| Coverslips | Citoglas | z10212222C | 22*22 mm |
| Dark-field microscopy | Nikon | 80i | upright microscope |
| Fetal bovine serum (FBS) | Gibco | 10099141 | |
| Fiji | National Institutes of Health | 2.0.0-rc-69/1.52 p | a distribution of ImageJ |
| Grooved glass slide | Sail brand | 7103 | Single concave |
| Image J | National Institutes of Health | 1.52 j | |
| MATLAB | MathWorks | R2019b | |
| MATLAB Code | https://github.com/fenggeqd/JOVE-2020 | ||
| Minimum essential medium (MEM) | Gibco | 10-010-CVR | with phenol red |
| Minimum essential medium (MEM) | Gibco | 51200038 | no phenol red |
| Origin | OriginLab | Origin Pro 2018C | |
| Penicillin-streptomycin | Gibco | 15140122 | |
| Plastic cell culture dishes | Falcon | 353002 | |
| Plastic cell culture dishes | Falcon | 353001 | 35*10 mm |
| U87 MG cell | American Type Culture Collection | ATCC HTB-14 | a human primary glioblastoma cell line |
Referencias
- Rees, P., Wills, J. W., Brown, M. R., Barnes, C. M., Summers, H. D. The origin of heterogeneous nanoparticle uptake by cells. Nature Communication. 10 (1), 2341(2019).
- Behzadi, S., et al. Cellular uptake of nanoparticles: journey inside the cell. Chemical Society Reviews. 46 (14), 4218-4244 (2017).
- Shen, H., et al. Single Particle Tracking: From Theory to Biophysical Applications. Chemical Reviews. 117 (11), 7331-7376 (2017).
- Saxton, M. J. Single-particle tracking: connecting the dots. Nature Methods. 5 (8), 671-672 (2008).
- Kusumi, A., et al. Dynamic organizing principles of the plasma membrane that regulate signal transduction: commemorating the fortieth anniversary of Singer and Nicolson's fluid-mosaic model. Annual Review of Cell and Developmental Biology. 28, 215-250 (2012).
- Jacobson, K., Liu, P., Lagerholm, B. C. The Lateral Organization and Mobility of Plasma Membrane Components. Cell. 177 (4), 806-819 (2019).
- Sezgin, E., Levental, I., Mayor, S., Eggeling, C. The mystery of membrane organization: composition, regulation and roles of lipid rafts. Nature Reviews Molecular Cell Biology. 18 (6), 361-374 (2017).
- von Diezmann, A., Shechtman, Y., Moerner, W. E. Three-Dimensional Localization of Single Molecules for Super-Resolution Imaging and Single-Particle Tracking. Chemical Reviews. 117 (11), 7244-7275 (2017).
- Rosenberg, J., Huang, J. Visualizing Surface T-Cell Receptor Dynamics Four-Dimensionally Using Lattice Light-Sheet Microscopy. Journal of Visualized Experiments. (155), e59914(2020).
- Kusumi, A., Tsunoyama, T. A., Hirosawa, K. M., Kasai, R. S., Fujiwara, T. K. Tracking single molecules at work in living cells. Nature Chemical Biology. 10 (7), 524-532 (2014).
- Rocha, J. M., Gahlmann, A. Single-Molecule Tracking Microscopy - A Tool for Determining the Diffusive States of Cytosolic Molecules. Journal of Visualized Experiments. (151), e59387(2019).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing protein-DNA interactions in live bacterial cells using photoactivated single-molecule tracking. Journal of Visualized Experiments. (85), e511177(2014).
- Pinaud, F., Clarke, S., Sittner, A., Dahan, M. Probing cellular events, one quantum dot at a time. Nature Methods. 7 (4), 275-285 (2010).
- Mehidi, A., et al. Transient Activations of Rac1 at the Lamellipodium Tip Trigger Membrane Protrusion. Current Biology. 29 (17), 2852-2866 (2019).
- Ye, Z., Wang, X., Xiao, L. Single-Particle Tracking with Scattering-Based Optical Microscopy. Analytical Chemistry. 91 (24), 15327-15334 (2019).
- Pan, Q., Zhao, H., Lin, X., He, Y. Spatiotemporal Heterogeneity of Reactions in Solution Observed with High-Speed Single-Nanorod Rotational Sensing. Angewandte Chemie. International Ed. in English. 58 (25), 8389-8393 (2019).
- Taylor, R. W., et al. Interferometric scattering microscopy reveals microsecond nanoscopic protein motion on a live cell membrane. Nature Photonics. 13 (7), 480-487 (2019).
- Chen, K., et al. Characteristic rotational behaviors of rod-shaped cargo revealed by automated five-dimensional single particle tracking. Nature Communication. 8 (1), 887(2017).
- Xu, D., He, Y., Yeung, E. S. Y. Direct observation of the orientation dynamics of single protein-coated nanoparticles at liquid/solid interfaces. Angewandte Chemie. International Ed. in English. 53 (27), 6951-6955 (2014).
- Lehui, X., Yan, H., Edward, S. Y. Three Dimensional Orientational Imaging of Nanoparticles with Darkfield Microscopy. Analytical Chemistry. 82, 5268-5274 (2010).
- Ge, F., Xue, J., Wang, Z., Xiong, B., He, Y. Real-time observation of dynamic heterogeneity of gold nanorods on plasma membrane with darkfield microscopy. Science China Chemistry. 62, 1072-1081 (2019).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Janczura, J., Weron, A. Ergodicity testing for anomalous diffusion: small sample statistics. The Journal of Chemical Physics. 142 (14), 144103(2015).
- Kim, D. H., et al. Single particle tracking-based reaction progress kinetic analysis reveals a series of molecular mechanisms of cetuximab-induced EGFR processes in a single living cell. Chemical Science. 8 (7), 4823-4832 (2017).
- Kurzthaler, C., et al. Probing the Spatiotemporal Dynamics of Catalytic Janus Particles with Single-Particle Tracking and Differential Dynamic Microscopy. Physical Review Letters. 121 (7), 078001(2018).
- Lin, X., Pan, Q., He, Y. In situ detection of protein corona on single particle by rotational diffusivity. Nanoscale. 11 (39), 18367-18374 (2019).
- Wei, L., et al. Sub-diffraction-limit localization imaging of a plasmonic nanoparticle pair with wavelength-resolved dark-field microscopy. Nanoscale. 9 (25), 8747-8755 (2017).
- Cheng, X., Dai, D., Xu, D., He, Y., Yeung, E. S. Subdiffraction-limited plasmonic imaging with anisotropic metal nanoparticles. Analytical Chemistry. 86 (5), 2303-2307 (2014).
- Belyy, V., et al. PhotoGate microscopy to track single molecules in crowded environments. Nature Communication. 8, 13978(2017).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados