Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Visualisation de la dynamique diffusionnelle des nanorods d’or sur la membrane cellulaire à l’aide d’une microscopie darkfield nanoparticule unique
Dans cet article
Résumé
Ici, nous montrons l’utilisation de la microscopie traditionnelle en champ sombre pour surveiller la dynamique des nanorods d’or (AuNRs) sur la membrane cellulaire. L’emplacement et l’orientation des aunrs simples sont détectés à l’aide d’ImageJ et de MATLAB, et les états diffusifs des AuNRs sont caractérisés par une analyse unique du suivi des particules.
Résumé
L’analyse de la dynamique de diffusion des nanoparticules sur la membrane cellulaire joue un rôle important dans une meilleure compréhension du processus d’absorption cellulaire et fournit une base théorique pour la conception rationnelle de la livraison de nanomédication. L’analyse du suivi unique des particules (SPT) pourrait sonder la position et l’orientation des nanoparticules individuelles sur la membrane cellulaire et révéler leurs états translationnels et de rotation. Ici, nous montrons comment utiliser la microscopie traditionnelle en champ sombre pour surveiller la dynamique des nanorods d’or (AuNRs) sur la membrane cellulaire vivante. Nous montrons également comment extraire l’emplacement et l’orientation des AuNR à l’aide d’ImageJ et matlab, et comment caractériser les états diffusifs des AuNRs. L’analyse statistique de centaines de particules montre qu’un seul AuNRs effectue le mouvement brownien à la surface de la membrane cellulaire U87 MG. Toutefois, l’analyse individuelle à longue trajectoire montre que les AuNR ont deux types d’états de mouvement distinctement différents sur la membrane, à savoir le transport à longue portée et l’enfermement à zone limitée. Nos méthodes de SPT peuvent être potentiellement utilisées pour étudier la diffusion des particules de surface ou intracellulaires dans différentes cellules biologiques et peuvent devenir un outil puissant pour l’étude de mécanismes cellulaires complexes.
Introduction
La dynamique des nanoparticules (PN) sur la membrane est étroitement associée au processus d’absorption cellulaire, qui est essentiel à la compréhension des fonctions cellulaires, des infections virales ou bactériennes et au développement de systèmes artificiels de livraison nanomédicale1,2. La technique de suivi des particules simples (SPT) est un outil robuste pour caractériser les comportements hétérogènes desPN 3,4. En général, la membrane cellulaire est fluide, ce qui signifie que les composants tels que les protéines et les lipides peuvent se déplacer latéralement dans le plan de la membraneplasmatique 5,6,7. La complexité spatiotemporal de l’organisation et de la structure de membrane peut mener à l’hétérogénéité spatiotemporal de l’interaction entre NPs et membrane. Par conséquent, la visualisation directe du mouvement des PN sur la membrane nécessite à la fois une résolution spatiale et temporelle élevée.
La microscopie de suivi des particules individuelles qui surveille la localisation des particules individuelles dans les cellules vivantes avec une résolution spatiale de dizaines de nanomètres et une résolution de temps de millisecondes a été bien développée pour étudier les SNP ou la dynamique des molécules membranaires8,9. Les techniques d’imagerie microscopique à base de fluorescence sont devenues des outils précieux pour l’observation des PN/molécules dans le milieu cellulairevivant 9,10,11,12. Par exemple, la microscopie totale de fluorescence de réflexion interne, qui images couches minces (~100 nm) de l’échantillon à l’interface substrat/solution avec une résolution spatiotemporal élevée a été employée couramment dans des études de la dynamique de molécules de membrane13,14. Cependant, les inconvénients inhérents des fluorophores simples, tels que la faible intensité et le photobleaching irréversible rapide réduisent la précision et la durée du suivi13. Par conséquent, les PN plasmoniques non fluorescents, qui remplacent les sondes fluorescentes, ont attiré de plus en plus l’attention dans les études d’imagerie à long terme en raison de leurs caractéristiques optiques uniques15. Sur la base des signaux de diffusion des sondes plasmoniques de PN, plusieurs types de technologies optiques d’imagerie microscopique ont été utilisés pour étudier le mécanisme des processus biologiques, tels que la microscopie des champs sombres (DFM)16,la microscopie interférométrique de diffusion (iSCAT)17 et la microscopie différentielle de contraste d’interférence (DICM)18. En outre, la dynamique de mouvement et de rotation des AuNRs peut être obtenue à l’aide de DFM et DICM18,19,20,21,22. En règle générale, dans une expérience SPT, le mouvement de l’objet est enregistré par le microscope optique, puis analysé par les méthodes d’analyse SPT3. Les trajectoires et les angles d’orientation résolus dans le temps générés par les PN individuels sont normalement stochastiques et hétérogènes, il est donc nécessaire de présenter des informations dynamiques abondantes avec diverses méthodes d’analyse.
Ici, nous fournissons un protocole intégré qui surveille la dynamique des aunrs sur la membrane cellulaire à l’aide de DFM, extrait l’emplacement et l’orientation des AuNRs avec ImageJ et MATLAB et caractérise la diffusion des AuNRs avec des méthodes d’analyse SPT. En démonstration, nous montrons ici comment utiliser le protocole SPT pour visualiser la dynamique des AuNRs non modifiés (CTAB-AuNRs, synthétisés par la molécule de bromure d’ammonium de cetyltriméthylammonium comme agent protecteur) sur la membrane cellulaire U87 MG. Il a été démontré que les CTAB-AuNRs peuvent adsorb protéines dans l’environnement biologique, se déplacer sur la membrane cellulaire, puis entrer dansles cellules 2,20,22. La cellule U87 MG est la tumeur la plus commune et la plus maligne du système nerveux central, et ses récepteurs membranaires sont anormalement exprimés. Les récepteurs membranaires peuvent interagir avec les protéines des AuNR, qui influencent la dynamique des AuNRs. Notre protocole s’applique généralement à d’autres expériences SPT dans le domaine de la biologie.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Culture cellulaire
- Préparer un milieu complet pour les cellules U87 MG en ajoutant le sérum bovin fœtal (concentration finale 10%) et pénicilline-streptomycine (concentration finale 1%) au milieu essentiel minimum (MEM). Utilisez un plat de culture cellulaire en plastique pour la sous-culture cellulaire.
- Cellules de passage 2 à 3 fois par semaine.
- Retirez le milieu de culture et rincez la couche cellulaire avec la solution saline tamponnée de phosphate (D-PBS) de Dulbecco 2~3 fois lorsqu’elle est confluente (80%~90%).
- Ajouter 1,0 à 2,0 mL de solution Trypsin-EDTA au plat de culture cellulaire et observer les cellules au microscope inversé jusqu’à ce que les cellules deviennent rondes (3~5 min).
- Ajouter 3,0 mL de milieu complet préparé et disperser les cellules en pipetting doucement.
- Ajouter la suspension cellulaire (1 mL) au nouveau plat de culture avec un milieu cellulaire frais (3 mL) et resuspendre les cellules.
- Maintenir les cellules à 37 °C et 5 % de CO2 dans une atmosphère humidifiée.
2. Préparation de glissière de microscope
REMARQUE : Les cellules U87 MG de troisième à dixième génération à forte activité sont utilisées dans les expériences SPT.
- Stériliser 22 mm × couvercles de 22 mm déjà nettoyés avec la solution Piranha en plongeant dans l’éthanol (99,9%).
- Utilisez des forceps pour enlever le bordereau de couverture de la solution d’éthanol (étape 2.1) et stériliser en brûlant de l’éthanol sur la flamme. Une fois que tout l’éthanol est brûlé, placez les couvercles dans un plat de culture cellulaire en plastique (35 x 10 mm) rempli de 2 mL de milieu cellulaire (sans rouge phénol).
- Ajouter 50 μL de la suspension cellulaire de l’étape 1.2.3 sur le coverslip et pousser doucement le plat d’avant en arrière et à gauche et à droite pour répartir uniformément les cellules. Placez-le dans une atmosphère humidifiée.
- Lorsque les cellules U87 MG sur le coverslip atteignent 20%-40% confluency (~12 h), ajouter 20 μL de CTAB-AuNRs (138 pM) dans le plat et se disperser. Incuber dans une atmosphère humidifiée pendant 5 min.
- Ajouter 100 μL du milieu de culture (pas de rouge phénol) du plat à l’étape 2.4 dans la rainure de la glissière en verre rainuré (figure 1) qui est préchauré avec la solution Piranha.
- Sortez le coverslip du plat et inversé sur le dessus de la rainure de la glissière en verre microscope (Figure 1). Sceller avec du vernis à ongles, le laisser sécher et le placer sur la scène pour effectuer des expériences SPT.
3. Effectuer des expériences de suivi des particules simples avec la microscopie darkfield (Figure 1).
- Placez une goutte d’huile sur le condenseur darkfield immergé dans l’huile (NA 1.43-1.20) et tournez le bouton pour faire entrer le condenseur en contact avec la glissière en verre.
- Mettez une goutte d’huile sur le dessus du verre de couverture et tournez le bouton de focalisation pour faire l’objectif d’immersion d’huile 60x (NA 0.7-1.25) toucher l’huile.
- Allumez la source lumineuse et tournez légèrement le bouton de mise au point pour concentrer le plan d’imagerie.
REMARQUE : Dans le champ de vision, l’arrière-plan est noir, les cellules sont brillantes et les CTAB-AuNRs (rapport d’aspect~2:1, figure 2)sont de petites taches de diffusion colorées (rouges, jaunes ou vertes). - Capturez la lumière de diffusion d’échantillon par un appareil-photo de CMOS de couleur. Cliquez sur "Icône caméra" dans le logiciel pour enregistrer et exporter le format TIFF pour enregistrer des images.
4. Acquisition de données
- Extraire une trajectoire unique à long terme
- Fonctionnez comme décrit dans la figure 3 pour convertir les images en champ sombre de la série de temps du mode « couleur RVB » en mode « 8 bits ». Dans l’image J cliquez sur Image | Type | 8 bits. Pour ajuster le contraste, cliquez sur Image | Ajuster | Luminosité | Contraste.
- Sélectionnez une particule cible et coupez les arrière-plans des séries de temps en boxe et en supprimant l’arrière-plan avec "Ctrl+X« .
- Ouvrez la fenêtre de détection des particules et de liaison des particules en cliquant surle " Plugins | Particle Tracker Classic | Particle Tracker« .
- Réglez le rayon à 6, le seuil à 0 et le percentile à 0,01 %.
REMARQUE : Pour détecter la particule, ajustez au-dessus de trois paramètres à l’aide de Preview. Assurez-vous que le rayon est légèrement plus grand que la particule ciblée et plus petit que la plus petite séparation interparticules. Le percentile est la limite inférieure de distribution de l’intensité qui est celle des particules candidats. - Réglez la plage de liaison à 5 et le déplacement à 10.
REMARQUE : Pour relier la particule entre les cadres adjacents consécutifs, ajustez les deux paramètres ci-dessus. Le déplacement est le maximum de pixels qu’une particule peut déplacer entre deux images qui réussissent, et Link Range est le nombre d’images consécutives à considérer lors de la détermination de la meilleure correspondance correspondante. - Cliquez sur" OK" pour ouvrir la fenêtre Résultats ParticleTracker pour voir les résultats.
- Cliquez sur" Visualiser toutes les trajectoires" pour inspecter les trajectoires générées.
- Cliquez sur le menu "Particules de relink" en haut pour reconnecter les particules détectées avec différentes plages de liaison et paramètres percentiles, si la trajectoire générée par le logiciel ne correspond pas à la trajectoire mobile de l’AuNR.
- Cliquez sur "Enregistrer le rapport complet" pour enregistrer les résultats si la trajectoire générée par le logiciel et la trajectoire mobile de l’AuNR sont appariées.
REMARQUE : Un exemple d’extraction d’une trajectoire unique à long terme avec l’image J est indiqué dans la figure 4.
- Extraction de multi-trajectoires
REMARQUE : Un exemple d’extraction de multi-trajectoires avec fidji est montré dans la figure 5. Il y a plusieurs étapes, et chaque étape constitue une étape dans le processus de suivi. Le résultat de chaque étape est affiché immédiatement, ce qui permet à l’utilisateur de revenir aux paramètres de réajustement lorsque la sortie n’est pas satisfaisante.- Fonctionnez comme décrit dans la figure 3 pour convertir les images en champ sombre de la série de temps du mode « couleur RVB » en mode « 8 bits ». Dans la voie Image J cliquez sur "Image | Type | 8 bits« . Pour ajuster leur contraste, cliquez sur "Image | Ajuster | Luminosité | Contraste ».
- Ouvrez le panneau de départ en cliquant sur" Plugins | Suivi | TrackMate« . Cliquez sur "Suivant« .
- Choisissez le "détecteur LoG" dans le menu drop-down et cliquez sur "Suivant« .
- Ajustez les paramètres du panneau de configuration du détecteur. Réglez le diamètre estimé de l’blob à 10, réglez seuil à 0, et sélectionnez "Do Sub-pixel Localization« .
- Cliquez sur "Suivant" pour ouvrir le panneau de filtrage initial.
REMARQUE : Plusieurs paramètres peuvent être ajustés pour optimiser davantage les points cibles. Dans cet exemple, aucun autre paramètre n’a été ajusté. - Cliquez sur" Suivant" et sélectionnez "Displayer HyperStack" dans le menu drop-down.
- Cliquez sur "Suivant" pour ouvrir le panneau de filtrage Spot. Réglez la qualité au-dessus de 1,88, X au-dessus de 38,86, Y au-dessus de 56,54.
- Cliquez sur" Suivant" et sélectionnez " SimpleLAP tracker" dans le menu drop-down. Configurez le tracker LAP simple en ajustant trois paramètres, c’est-à-dire la distance maximale de liaison = 15, la distance maximale de fermeture des écarts = 15 et l’écart de trame max de fermeture de gap = 5.
REMARQUE : La distance de liaison-max est le déplacement maximum d’un point entre deux images. La distance maximale de fermeture des écarts est le déplacement maximal de deux segments. L’écart de trame max de fermeture d’écart est le plus grand cadre entre deux points à combler. - Cliquez sur "Suivant" pour suivre. Une fois terminé, continuer à cliquer sur "Suivant« .
- Réglez les filtres sur les pistes, comme définir le nombre de taches en piste au-dessus de 300.
- Continuez à cliquer sur "Suivant" jusqu’à ce que le panneau d’arrêt final s’ouvre. Sélectionnez "Export tracks to XML file" à partir du menu drop-down, puis cliquez sur "Exécuter" pour économiser en format csv.
- Valeurs R/G
REMARQUE : Les intensités de diffusion des aunrs ciblés dans les canaux R et G sont obtenues à partir d’images couleur darkfield à l’aide d’un code écrit dans MATLAB (https://github.com/fenggeqd/JOVE-2020/tree/master/RGandPolarangle), et le principe d’extraction est présenté à la figure 6.- Utilisez la fonction de xycoordination.m pour trouver la coordonnée du pixel central de l’AuNR dans chaque image selon la coordonnée x-y (extraite par ImageJ/Fiji).
- Utilisez la fonction de RGextraction.m pour délimiter une matrice de 3 x 3 pixels, extraire les 9 valeurs d’intensité de diffusion des canaux R ou G et calculer une valeur moyenne (μ, définie comme R ou G).
REMARQUE : La matrice de 3 x 3 pixels est centrée sur les coordonnées pixel obtenues à partir de l’étape 4.3.1.
- Angle polaire
REMARQUE : L’angle polaire est l’angle entre la longitude de l’AuNR et l’axe optique (comme le montre la figure 1),qui peut refléter la dynamique de rotation spatiale (axe Z) de l’AuNR.- Utilisez la fonction de polarangle.m pour calculer les angles polaires (θ) par la méthode différentielle à double canal22,
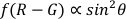 .
.
- Utilisez la fonction de polarangle.m pour calculer les angles polaires (θ) par la méthode différentielle à double canal22,
5. Analyse des données
REMARQUE : Un cadre d’analyse systématique et robuste des données est essentiel à la performance et à l’efficacité des méthodes d’analyse SPT. Le logiciel personnalisé écrit dans MATLAB est utilisé (https://github.com/fenggeqd/JOVE-2020/tree/master/Analysis_parameters). Un logiciel de graphique et d’analyse (voir tableau des matériaux)est utilisé pour dessiner les parcelles.
- Paramètres d’analyse
- Utilisez des scripts de csv_data_extract_dis_vel_ss.m et csv_data_MSD.m pour calculer les paramètres dynamiques selon les formules indiquées dans le tableau 1.
REMARQUE : Ces paramètres sont utilisés pour analyser la dynamique des AuNR et se composent de trois parties. (1) Paramètres liés à la trajectoire : déplacement, taille de l’étape, vitesse, rayon de gyration (Rg)et angle de rotation (Ta); (2) Paramètres MSD : coefficient de diffusion (Dt)et exposant de diffusion anormal (α); et (3) Paramètres liés à la rotation : angle polaire et labilité de rotation (σ).
- Utilisez des scripts de csv_data_extract_dis_vel_ss.m et csv_data_MSD.m pour calculer les paramètres dynamiques selon les formules indiquées dans le tableau 1.
| Mesures | Définition | Signification physique | |
| Déplacement | 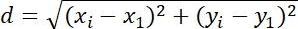 | Changements de position des objets | |
| Taille de l’étape | 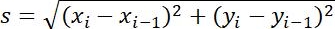 | Distance entre deux points adjacents | |
| Vitesse |  | Vitesse du mouvement des objets | |
| Rg | 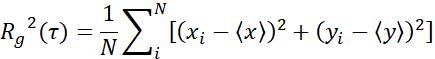 | Déplacement de la gamme d’objets dans un intervalle de temps spécifique | |
| Ta | 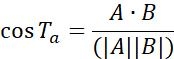 | Direction de mouvement des objets entre deux points adjacents | |
| Msd | 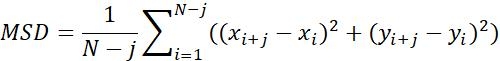 | Distance mobile moyenne des objets dans un intervalle de temps spécifique | |
| Dt | 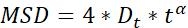 | Capacité de diffusion des objets | |
| α | 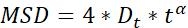 | Diffusion normale (α~1) | |
| Angle polaire |  | Informations d’orientation 3D des objets | |
| Σ |  | Degré de dispersion de l’ensemble de données sur l’angle polaire | |
Tableau 1 : Trois types de paramètres utilisés pour l’analyse. Il s’agit notamment des paramètres liés à la trajectoire (déplacement, taille de l’étape, vitesse, Rg et Ta),des paramètres MSD (MSD, Dt et α) et des paramètres liés à la rotation (angle polaire et labilité de rotation).
- Analyse visuelle de la trajectoire
REMARQUE : La visualisation de trajectoire peut présenter intuitivement l’hétérogénéité spatiotemporale du mouvement des particules et la répartition de la trajectoire (coordonnée) des paramètres dynamiques, tels que la trajectoire de cartographie du temps,la trajectoire de cartographieR g - et Dt,et la trajectoire de cartographie à angle polaire. Des trajectoires cartographiques ont été dessinées à l’aide du logiciel de graphique et d’analyse.- Définir x coordonner comme X, y coordonner comme Y, et le temps (Rg, Dt, angle polaire) comme Z.
- Cliquez sur "Tracer | Disperser l’intrigue | Parcelle de cartographie des couleurs« .
- Ajouter la barre de couleur.
- Analyse msd
REMARQUE : L’activité de mouvement et le mode de mouvement des particules peuvent être obtenus par l’analyse MSD23. Plus le Dtest grand, plus le mouvement de diffusion des particules est actif. lorsqu α~1, les particules effectuent un mouvement de diffusion normal, sinon elles effectuent un mouvement de diffusion anormal.- Chiffres MSD-τ
- Définissez l’intervalle de temps (τ) comme X, données MSD comme Y.
- Cliquez sur "Tracer | Scatter parcelle»
- Adaptez les données en cliquant sur" Analyser | Raccord | Raccord de courbe non lignellaire (Fonction: Allometricl)« .
- MSD-τ chiffre double-logarithmic
REMARQUE: MSD-τ double-logarithmic figure, dont la pente est α et intercepter est Dt, peut intuitivement présenter le mouvement des particules.- Définissez l’intervalle de temps logarithmique comme X, et msd logarithmic comme Y.
- Cliquez sur "Tracer | Scatter parcelle« .
- Adaptez les données en cliquant sur" Analyser | Raccord | Montage de courbe linéaire« .
- Figure D-τ (MSD/4t)
REMARQUE : D=MSD/4τ, est fonction du facteur de temps et d’anomalie α, et le chiffre D-τ montre directement le changement du coefficient de diffusion avec le temps. Lorsque D augmente avec le temps, α est supérieur à 1 et les particules ne superdiffusion mouvement.- Définissez l’intervalle de temps logarithmique (τ) comme X, et logarithmic D comme Y.
- Cliquez sur "Tracer | Scatter parcelle« .
- Adaptez les données en cliquant sur" Analyser | Raccord | Raccord de courbe non lignellaire (Fonction : " Allometricl « )« .
REMARQUE : Plus les trajectoires sont courtes, plus l’inexactitude des estimations de diffusion est élevée. En général, Dt et α par l’analyse MSD-τ du temps d’intervalle long (> 30 τ) ont été obtenus. Toutefois, un raccord simple et rugueux lissera les détails du mouvement. Par conséquent, l’analyse MSD-τ du temps d’intervalle court (< 10 τ) devrait être effectuée pour analyser le comportement de mouvement des particules dans un court laps de temps.
- Chiffres MSD-τ
- Analyse statistique
- Analyse statistique multiparticules
REMARQUE : L’analyse statistique multiparticules peut refléter l’état de mouvement des particules dans une région spatiale, ce qui indique indirectement l’environnement d’hétérogénéité spatiale. Par exemple, si l’histogramme de Dt présente une distribution à grande échelle ou une distribution à plusieurs pics, cela signifie que les activités de mouvement des particules sont hétérogènes.- Définissez des paramètres dynamiques (tels que Dt,Rg,déplacement maximum) comme Y.
- Cliquez sur "Tracer | Histogramme« .
- Double clic sur l’histogramme, et définir la taille de la division ou le nombre de divisions. Cliquez sur "Appliquer« .
- Analyse statistique à particule unique
REMARQUE : L’analyse statistique des particules individuelles peut montrer le comportement de mouvement des particules individuelles, qui reflète également indirectement l’hétérogénéité spatiotemporale de l’environnement environnant.- Cadres multiples
- Calculez les paramètres dynamiques (tels que Ta,taille d’étape, angle polaire) de tous les cadres d’une seule longue trajectoire, et copiez à la table Origin et définissez comme Y.
- Cliquez sur "Tracer | Histogramme« .
- Double clic sur l’histogramme, et définir la taille de la division ou le nombre de divisions. Cliquez sur "Appliquer« .
REMARQUE : Si la taillet a et la taille des pas présentent une petite distribution de valeur, les particules font un mouvement de super-diffusion en petits pas.
- Fenêtres mobiles
- Calculez les paramètres dynamiques (tels que Rg,Dt)de tous les cadres d’une seule longue trajectoire grâce à la méthode de fenêtre mobile (11 images), et copiez à la table Origin et définissez comme Y.
- Cliquez sur "Tracer | Histogramme« .
- Double clic sur l’histogramme, et définir la taille de la division ou le nombre de divisions. Cliquez sur "Appliquer« .
- Cadres multiples
- Analyse statistique multiparticules
- Analyse des séries de temps
REMARQUE : L’analyse statistique peut révéler l’état de mouvement des PN, et l’analyse des séries de temps peut présenter le comportement de mouvement comme un supplément. Combinant plusieurs paramètres de séries temporelles, il peut discriminer le comportement de mouvement des PN aux niveaux temporel et spatial.- Définissez l’heure comme X, les paramètres de la série de temps comme Y (tels que le déplacement, l’angle polaire et Rg).
- Cliquez sur "Tracer | Diagramme multi-volets | Terrain empilé | Ligne + Symbole« .
Access restricted. Please log in or start a trial to view this content.
Résultats
Dans le protocole, les 40 x 85 nm CTAB-AuNRs non modifiés ont été utilisés. Comme le montre la figure 2B,son maximum plasmonique longitudinal est d’environ 650 nm (région rouge) et la résonance transversale est de 520 nm (région verte). Les littératures précédentes ont indiqué que les propriétés optiques (telles que l’intensité de LSPR) des AuNRs plasmoniques changeront sensiblement avec leurdiamètre 20,22. Dans
Access restricted. Please log in or start a trial to view this content.
Discussion
Le protocole présenté est utilisé pour étudier la dynamique des AuNRs sur la membrane cellulaire. Le protocole se compose de quatre parties, y compris l’imagerie microscopique, l’extraction de données, le calcul des paramètres dynamiques et les méthodes d’analyse des données, et chaque partie est flexible et universelle. Par conséquent, il existe de nombreuses applications futures possibles, par exemple, l’étude du mouvement des molécules membranaires liées à la PN sur la membrane, la dynamique endoc...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ces travaux ont été soutenus par la National Natural Science Foundation of China avec des subventions de 21425519, 91853105 et 21621003.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| CTAB coated gold nanorods(CTAB-AuNRs) | Nanoseedz | NR-40-650 | 85 nm * 40 nm |
| Color CMOS camera | Olympus | DP74 | Japan |
| Coverslips | Citoglas | z10212222C | 22*22 mm |
| Dark-field microscopy | Nikon | 80i | upright microscope |
| Fetal bovine serum (FBS) | Gibco | 10099141 | |
| Fiji | National Institutes of Health | 2.0.0-rc-69/1.52 p | a distribution of ImageJ |
| Grooved glass slide | Sail brand | 7103 | Single concave |
| Image J | National Institutes of Health | 1.52 j | |
| MATLAB | MathWorks | R2019b | |
| MATLAB Code | https://github.com/fenggeqd/JOVE-2020 | ||
| Minimum essential medium (MEM) | Gibco | 10-010-CVR | with phenol red |
| Minimum essential medium (MEM) | Gibco | 51200038 | no phenol red |
| Origin | OriginLab | Origin Pro 2018C | |
| Penicillin-streptomycin | Gibco | 15140122 | |
| Plastic cell culture dishes | Falcon | 353002 | |
| Plastic cell culture dishes | Falcon | 353001 | 35*10 mm |
| U87 MG cell | American Type Culture Collection | ATCC HTB-14 | a human primary glioblastoma cell line |
Références
- Rees, P., Wills, J. W., Brown, M. R., Barnes, C. M., Summers, H. D. The origin of heterogeneous nanoparticle uptake by cells. Nature Communication. 10 (1), 2341(2019).
- Behzadi, S., et al. Cellular uptake of nanoparticles: journey inside the cell. Chemical Society Reviews. 46 (14), 4218-4244 (2017).
- Shen, H., et al. Single Particle Tracking: From Theory to Biophysical Applications. Chemical Reviews. 117 (11), 7331-7376 (2017).
- Saxton, M. J. Single-particle tracking: connecting the dots. Nature Methods. 5 (8), 671-672 (2008).
- Kusumi, A., et al. Dynamic organizing principles of the plasma membrane that regulate signal transduction: commemorating the fortieth anniversary of Singer and Nicolson's fluid-mosaic model. Annual Review of Cell and Developmental Biology. 28, 215-250 (2012).
- Jacobson, K., Liu, P., Lagerholm, B. C. The Lateral Organization and Mobility of Plasma Membrane Components. Cell. 177 (4), 806-819 (2019).
- Sezgin, E., Levental, I., Mayor, S., Eggeling, C. The mystery of membrane organization: composition, regulation and roles of lipid rafts. Nature Reviews Molecular Cell Biology. 18 (6), 361-374 (2017).
- von Diezmann, A., Shechtman, Y., Moerner, W. E. Three-Dimensional Localization of Single Molecules for Super-Resolution Imaging and Single-Particle Tracking. Chemical Reviews. 117 (11), 7244-7275 (2017).
- Rosenberg, J., Huang, J. Visualizing Surface T-Cell Receptor Dynamics Four-Dimensionally Using Lattice Light-Sheet Microscopy. Journal of Visualized Experiments. (155), e59914(2020).
- Kusumi, A., Tsunoyama, T. A., Hirosawa, K. M., Kasai, R. S., Fujiwara, T. K. Tracking single molecules at work in living cells. Nature Chemical Biology. 10 (7), 524-532 (2014).
- Rocha, J. M., Gahlmann, A. Single-Molecule Tracking Microscopy - A Tool for Determining the Diffusive States of Cytosolic Molecules. Journal of Visualized Experiments. (151), e59387(2019).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing protein-DNA interactions in live bacterial cells using photoactivated single-molecule tracking. Journal of Visualized Experiments. (85), e511177(2014).
- Pinaud, F., Clarke, S., Sittner, A., Dahan, M. Probing cellular events, one quantum dot at a time. Nature Methods. 7 (4), 275-285 (2010).
- Mehidi, A., et al. Transient Activations of Rac1 at the Lamellipodium Tip Trigger Membrane Protrusion. Current Biology. 29 (17), 2852-2866 (2019).
- Ye, Z., Wang, X., Xiao, L. Single-Particle Tracking with Scattering-Based Optical Microscopy. Analytical Chemistry. 91 (24), 15327-15334 (2019).
- Pan, Q., Zhao, H., Lin, X., He, Y. Spatiotemporal Heterogeneity of Reactions in Solution Observed with High-Speed Single-Nanorod Rotational Sensing. Angewandte Chemie. International Ed. in English. 58 (25), 8389-8393 (2019).
- Taylor, R. W., et al. Interferometric scattering microscopy reveals microsecond nanoscopic protein motion on a live cell membrane. Nature Photonics. 13 (7), 480-487 (2019).
- Chen, K., et al. Characteristic rotational behaviors of rod-shaped cargo revealed by automated five-dimensional single particle tracking. Nature Communication. 8 (1), 887(2017).
- Xu, D., He, Y., Yeung, E. S. Y. Direct observation of the orientation dynamics of single protein-coated nanoparticles at liquid/solid interfaces. Angewandte Chemie. International Ed. in English. 53 (27), 6951-6955 (2014).
- Lehui, X., Yan, H., Edward, S. Y. Three Dimensional Orientational Imaging of Nanoparticles with Darkfield Microscopy. Analytical Chemistry. 82, 5268-5274 (2010).
- Ge, F., Xue, J., Wang, Z., Xiong, B., He, Y. Real-time observation of dynamic heterogeneity of gold nanorods on plasma membrane with darkfield microscopy. Science China Chemistry. 62, 1072-1081 (2019).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Janczura, J., Weron, A. Ergodicity testing for anomalous diffusion: small sample statistics. The Journal of Chemical Physics. 142 (14), 144103(2015).
- Kim, D. H., et al. Single particle tracking-based reaction progress kinetic analysis reveals a series of molecular mechanisms of cetuximab-induced EGFR processes in a single living cell. Chemical Science. 8 (7), 4823-4832 (2017).
- Kurzthaler, C., et al. Probing the Spatiotemporal Dynamics of Catalytic Janus Particles with Single-Particle Tracking and Differential Dynamic Microscopy. Physical Review Letters. 121 (7), 078001(2018).
- Lin, X., Pan, Q., He, Y. In situ detection of protein corona on single particle by rotational diffusivity. Nanoscale. 11 (39), 18367-18374 (2019).
- Wei, L., et al. Sub-diffraction-limit localization imaging of a plasmonic nanoparticle pair with wavelength-resolved dark-field microscopy. Nanoscale. 9 (25), 8747-8755 (2017).
- Cheng, X., Dai, D., Xu, D., He, Y., Yeung, E. S. Subdiffraction-limited plasmonic imaging with anisotropic metal nanoparticles. Analytical Chemistry. 86 (5), 2303-2307 (2014).
- Belyy, V., et al. PhotoGate microscopy to track single molecules in crowded environments. Nature Communication. 8, 13978(2017).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon