A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הדמיה של דינמיקה דיפוזיונית של ננו-ירדס זהב על קרום התא באמצעות מיקרוסקופיה אחת של ננו-חלקיקים בדארקפילד
In This Article
Summary
כאן, אנו מראים את השימוש במיקרוסקופיה מסורתית של שדה כהה כדי לפקח על הדינמיקה של nanorods זהב (AuNRs) על קרום התא. המיקום והכיוון של AuNRs יחיד מזוהים באמצעות ImageJ ו- MATLAB, והמדינות המפוזרות של AuNRs מאופיינים בניתוח מעקב אחר חלקיקים בודדים.
Abstract
ניתוח הדינמיקה הדיפוזיונית של חלקיקים על קרום התא ממלא תפקיד משמעותי בהבנה טובה יותר של תהליך הספיגה התאית ומספק בסיס תיאורטי לעיצוב הרציונלי של אספקת ננו-רפואה. ניתוח מעקב חלקיקים יחיד (SPT) יכול לחקור את המיקום והכיוון של חלקיקים בודדים על קרום התא, ולחשוף את מצבי התרגום והסיבוב שלהם. כאן, אנו מראים כיצד להשתמש מיקרוסקופיה שדה כהה מסורתי כדי לפקח על הדינמיקה של nanorods זהב (AuNRs) על קרום תא חי. אנו גם מראים כיצד לחלץ את המיקום והכיוון של AuNRs באמצעות ImageJ ו- MATLAB, וכיצד לאפיין את המצבים המפוזרים של AuNRs. ניתוח סטטיסטי של מאות חלקיקים מראים כי AuNRs יחיד לבצע תנועה בראונית על פני השטח של קרום התא U87 MG. עם זאת, ניתוח מסלול ארוך בודדים מראה כי AuNRs יש שני סוגים שונים במובהק של מצבי תנועה על הממברנה, כלומר תחבורה ארוכת טווח וכליאה באזור מוגבל. שיטות SPT שלנו יכול לשמש פוטנציאל לחקור את פני השטח או דיפוזיה חלקיקים תאיים בתאים ביולוגיים שונים והוא יכול להיות כלי רב עוצמה לחקירות של מנגנונים תאיים מורכבים.
Introduction
הדינמיקה של חלקיקים (NPs) על הממברנה קשורה קשר הדוק עם תהליך ספיגת הסלולר, אשר חיוני להבנת תפקודי התא, זיהומים ויראליים או חיידקיים ופיתוח של מערכות משלוח ננו רפואי מלאכותי1,2. טכניקת מעקב חלקיקים בודדים (SPT) היא כלי חזק לאפיון ההתנהגויות ההטרוגניות של NPs3,4. באופן כללי, קרום התא הוא נוזלי, כלומר הרכיבים כגון חלבונים ושומנים יכולים לנוע לרוחב במישור קרום הפלזמה5,6,7. המורכבות המרחבית של ארגון ומבנה הממברנה עלולה להוביל להטרוגניות מרחבית של האינטראקציה בין NPs לקרום. לפיכך, הדמיה ישירה של התנועה של NPs על הממברנה דורשת רזולוציה מרחבית וטמפורלית גבוהה.
מיקרוסקופ מעקב חלקיקים יחיד המנטר לוקליזציה של חלקיקים בודדים בתאים חיים עם רזולוציה מרחבית של עשרות ננומטר ורזולוציית זמן של אלפיות שנייה פותחה היטב כדי לחקור את NPs או מולקולות קרוםדינמיקה 8,9. טכניקות הדמיה מיקרוסקופיות מבוססות פלואורסצנטיות הפכו לכלים יקרי ערך להתבוננות ב- NPs /מולקולות בסביבת תאיםחיים 9,10,11,12. לדוגמה, מיקרוסקופ פלואורסצנטי השתקפות פנימית הכוללת, אשר תמונות שכבות דקות (~ 100 ננומטר) של המדגם בממשק מצע / פתרון עם רזולוציה spatiotemporal גבוהה כבר בשימוש נרחב במחקרים של מולקולות קרוםדינמיקה 13,14. עם זאת, החסרונות הטבועים בפלואורופורים בודדים, כגון אינטנסיביות נמוכה וצילום מהיר ובלתי הפיך מפחיתים את הדיוק ומשך המעקב13. לכן, NPs פלסמוניים שאינם פלואורסצנטיים, אשר מחליפים את הבדיקות הפלואורסצנטיות, משכו יותר ויותר תשומת לב במחקרי הדמיה ארוכי טווח בשל המאפיינים האופטיים הייחודיים שלהם15. בהתבסס על אותות הפיזור של בדיקות NP פלסמוניות, נעשה שימוש במספר סוגים של טכנולוגיות הדמיה מיקרוסקופיות אופטיות כדי לחקור את המנגנון של תהליכים ביולוגיים, כגון מיקרוסקופיה שדה כהה (DFM)16, פיזור אינטרפרומטרי (iSCAT) מיקרוסקופיה17 ומיקרוסקופיית ניגוד הפרעה דיפרנציאלית (DICM)18. בנוסף, ניתן להשיג את דינמיקת התנועה והסיבוב של AuNRs באמצעות DFM ו DICM18,19,20,21,22. בדרך כלל, בניסוי SPT, התנועה של האובייקט נרשמת על ידי המיקרוסקופ האופטי, ולאחר מכן מנותחת על ידי שיטות ניתוח SPT3. המסלולים שנפתרו בזמן וזוויות האוריינטציה שנוצרו על ידי NPs בודדים הם בדרך כלל סטוכסטיים והטרוגניים, ולכן יש צורך להציג מידע דינמי בשפע עם שיטות ניתוח שונות.
כאן, אנו מספקים פרוטוקול משולב המנטר את הדינמיקה של AuNRs על קרום התא באמצעות DFM, מחלץ את המיקום והכיוון של AuNRs עם ImageJ ו- MATLAB ומאפיין את הדיפוזיה של AuNRs עם שיטות ניתוח SPT. כהדגמה, אנו מראים כאן כיצד להשתמש בפרוטוקול SPT כדי לדמיין את הדינמיקה של AuNRs ללא שינוי (CTAB-AuNRs, מסונתז על ידי מולקולת אמוניום ברומיד cetyltrimethylammonium כסוכן מגן) על קרום התא U87 MG. הוכח כי CTAB-AuNRs יכול לתסלח חלבונים בסביבה ביולוגית, לנוע על קרום התא ולאחר מכן להזין תאים2,20,22. תא U87 MG הוא הגידול הנפוץ והממאיר ביותר של מערכת העצבים המרכזית, קולטני הממברנה שלו באים לידי ביטוי באופן חריג. קולטני הממברנה יכולים לקיים אינטראקציה עם חלבונים על AuNRs, אשר משפיעים על הדינמיקה של AuNRs. הפרוטוקול שלנו ישים בדרך כלל לניסויי SPT אחרים בתחום הביולוגיה.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. תרבות התאים
- הכן מדיום מלא עבור תאי U87 MG על ידי הוספת סרום שור עוברי (ריכוז סופי 10%) ופניצילין-סטרפטומיצין (ריכוז סופי 1%) למדיום החיוני המינימלי (MEM). השתמש צלחת תרבות תא פלסטיק עבור תת תרבות תאים.
- תאי מעבר 2 עד 3 פעמים בשבוע.
- הסר את מדיום התרבות ושטוף את שכבת התא באמצעות מלוחי הפוספט (D-PBS) של D-PBS 2~ 3 פעמים כאשר הם משולבים (80%~ 90%).
- הוסף 1.0 כדי 2.0 מ"ל של פתרון טריפסין-EDTA לצלחת תרבות התא ולהתבונן תאים תחת מיקרוסקופ הפוך עד התאים הופכים עגולים (3 ~ 5 דקות).
- מוסיפים 3.0 מ"ל של מדיום שלם מוכן ומפזרים את התאים על ידי צינור בעדינות.
- הוסף השעיית תא (1 מ"ל) לצלחת תרבות חדשה עם מדיום תא טרי (3 מ"ל) ו לשימוש חוזר בתאים.
- לשמור על התאים ב 37 °C (69 °F) ו 5% CO2 באטמוספירה לחה.
2. הכנת שקופיות מיקרוסקופ
הערה: U87 תאי MG של הדור השלישי עד העשירי עם פעילות גבוהה משמשים בניסויי SPT.
- לעקר 22 מ"מ × 22 מ"מ כיסויים כבר ניקה עם פתרון פיראנה על ידי טבילה באתנול (99.9%).
- השתמש מלקחיים להוציא את כיסוי להחליק מתמיסת אתנול (שלב 2.1) לעקר על ידי שריפת אתנול על הלהבה. לאחר שכל האתנול נשרף, מניחים מכסה בצלחת תרבית תאי פלסטיק (35 x 10 מ"מ) במילוי 2 מ"ל של מדיום התא (ללא פנול אדום).
- הוסף 50 μL של השעיית התא משלב 1.2.3 על כריכה בעדינות לדחוף את המנה קדימה ואחורה ושמאלה וימינה כדי להפיץ את התאים באופן שווה. מניחים אותו באווירה לחה.
- כאשר תאי U87 MG על מכסה להגיע 20%-40% מפגש (~ 12 שעות), להוסיף 20 μL של CTAB-AuNRs (138 pM) לתוך המנה ולפזר. דגירה באווירה לחה במשך 5 דקות.
- הוסיפו 100 μL של מדיום התרבות (ללא פנול אדום) מהצלחת בשלב 2.4 לתוך החריץ של מגלשת הזכוכית המחורצת (איור 1) אשר precleaned עם פתרון פיראנה.
- הוציאו את הכיסוי מהמנה והיפוכו בחלק העליון של החריץ של מגלשת הזכוכית המיקרוסקופית(איור 1). לאטום עם לק, לתת לו להתייבש ומניחים אותו על הבמה כדי לבצע ניסויים SPT.
3. ביצוע ניסויים במעקב אחר חלקיקים בודדים באמצעות מיקרוסקופיה של שדה אפל(איור 1).
- מניחים טיפת שמן על מנבא שדה כהה שקוע בשמן (NA 1.43-1.20) ומסובבים את הידית כדי לגרום למבני ליצור קשר עם מגלשת הזכוכית.
- שים טיפת שמן על החלק העליון של זכוכית הכיסוי ולהפוך את הידית התמקדות כדי להפוך את המטרה טבילה שמן 60x (NA 0.7–1.25) לגעת בשמן.
- הפעל את מקור האור וסובב מעט את ידית המיקוד כדי למקד את מישור ההדמיה.
הערה: בשדה התצוגה, הרקע שחור, התאים בהירים ונקודות הפיזור CTAB-AuNRs (יחס גובה-רוחב~ 2:1, איור 2)הן נקודות פיזור בצבעים קטנים (אדום, צהוב או ירוק). - לכוד אור פיזור לדוגמה באמצעות מצלמת CMOS צבעונית. לחץ על "סמל מצלמה" בתוכנה כדי להקליט ולייצא תבנית TIFF לשמירת תמונות.
4. רכישת נתונים
- חילוץ מסלול יחיד לטווח ארוך
- פעלו כמתואר באיור 3 כדי להמיר את תמונות השדה הכהה מסדרת הזמן ממצב "צבע RGB" למצב "8 סיביות". בתמונה J לחץ על תמונה | הקלד | 8 סיביות. להתאמת החדות, לחצו על 'תמונה' | כוונון | | בהירות ניגודיות.
- בחר חלקיק יעד וקטע את הרקעים של סידרת הזמן על-ידי איגרוף ומחיקת הרקע באמצעות "Ctrl+X".
- פתח את החלון זיהוי חלקיקים וקישור חלקיקים על-ידי לחיצה על "תוספים | גשש חלקיקים קלאסי | גשש חלקיקים".
- הגדר את Radius ל- 6, ניתוק ל- 0 ואת אחוזון ל- 0.01%.
הערה: כדי לזהות את החלקיק, התאם מעל שלושה פרמטרים בעזרת תצוגה מקדימה. ודאו שהרדיוס מעט גדול יותר מהחלקיק הייעודי וקטן יותר מהפרדת החלקיקים הקטנה ביותר. אחוזון הוא הגבול התחתון של התפלגות אינטנסיביות כי להיות חלקיקי מועמד. - הגדר את טווח הקישורים ל- 5 ואת ההעתקה ל- 10.
הערה: כדי לקשר את החלקיק בין מסגרות סמוכות רצופות, התאם את שני הפרמטרים לעיל. הזזה היא הפיקסלים המרביים שחלקיק יכול לעבור בין שתי מסגרות עוקבות, וטווח קישורים הוא מספר המסגרות העוקבות שיש לקחת בחשבון בעת קביעת ההתאמה המתאימה ביותר. - לחץ על "אישור" כדי לפתוח את החלון תוצאות של ParticleTracker כדי לראות את התוצאות.
- לחץ על "דמיין את כל המסלולים" כדי לבדוק את המסלולים שנוצרו.
- לחץ על תפריט "קשר מחדש חלקיקים" בחלק העליון כדי לקשר מחדש את החלקיקים שזוהו עם טווח קישורים ופרמטרים שונים של אחוזון, אם המסלול שנוצר על-ידי התוכנה אינו תואם את המסלול הנע של AuNR.
- לחץ על "שמור דוח מלא" כדי לשמור תוצאות אם התוכנה יצרה מסלול ואת המסלול הנע של AuNR תואמים.
הערה: דוגמה לחילוץ מסלול יחיד לטווח ארוך עם Image J מוצגת באיור 4.
- חילוץ מסלולים מרובים
הערה: דוגמה לחילוץ מסלולים מרובים עם פיג'י מוצגת באיור 5. ישנם מספר שלבים, וכל שלב מהווה שלב בתהליך המעקב. התוצאה של כל שלב מוצגת באופן מיידי, דבר המאפשר למשתמש לחזור כדי להתאים מחדש את ההגדרות כאשר הפלט אינו משביע רצון.- פעלו כמתואר באיור 3 כדי להמיר את תמונות השדה הכהה מסדרת הזמן ממצב "צבע RGB" למצב "8 סיביות". במסלול תמונה J לחץ על "תמונה | הקלד | 8 סיביות". כדי להתאים את החדות שלהם, לחץ על "תמונה | כוונון | | בהירות ניגודיות".
- פתח את לוח ההתחלה על-ידי לחיצה על "תוספים | | מעקב שותף למסלול". לחץ על "הבא".
- בחר את "גלאי LoG" מהתפריט הנפתח ולחץ על "הבא".
- התאם פרמטרים של לוח קביעת התצורה של הגלאי. הגדר קוטר כתם משוער ל- 10, הגדר את סף ל- 0 ובחר באפשרות "בצע לוקליזציה של תת-פיקסלים".
- לחץ על "הבא" כדי לפתוח את החלונית הראשונית לסינון ספוט.
הערה: ניתן להתאים מספר פרמטרים כדי למטב עוד יותר את נקודות היעד. בדוגמה זו, לא הותאמו פרמטרים אחרים. - לחץ על "הבא" ובחר "מציג HyperStack" מהתפריט הנפתח.
- לחץ על "הבא" כדי לפתוח את החלונית 'סינון ספוט'. הגדר את האיכות מעל 1.88, X מעל 38.86, Y מעל 56.54.
- לחץ על "הבא" ובחר "עוקב LAP פשוט" מהתפריט הנפתח. קבע את התצורה של עוקב LAP הפשוט על-ידי התאמת שלושה פרמטרים, כלומר, המרחק המרבי המקשר = 15, מרחק מרבי לסגירת פער = 15 ו- Gap-סגירת מרווח מסגרת מרבי = 5.
הערה: מרחק קישור-מקסימום הוא ההעתקה המרבית של נקודה בין שתי מסגרות. מרחק מרבי של סגירת פערים הוא ההעתקה המרבית של שני מקטעים. פער מסגרת מרבי סוגר פערים הוא המסגרת הגדולה ביותר בין שתי נקודות שיש לגשר עליהן. - לחץ על "הבא" כדי לעקוב אחר. בסיום, המשך ללחוץ על "הבא".
- הגדר מסננים ברצועות, כגון הגדרת מספר הנקודות ברצועה מעל 300.
- המשך ללחוץ על "הבא" עד לפתיחת לוח השמירה הסופי. בחר באפשרות "יצא רצועות לקובץ XML" מהתפריט הנפתח ולאחר מכן לחץ על "ביצוע" כדי לשמור בתבנית csv.
- ערכי R/G
הערה: עוצמות פיזור של AuNRs ממוקדים בערוצי R ו- G מתקבלות מתמונות צבע של שדה כהה באמצעות קוד שנכתב ב- MATLAB (https://github.com/fenggeqd/JOVE-2020/tree/master/RGandPolarangle), ועיקרון החילוץ מוצג באיור 6.- השתמש בפונקציה של xycoordination.m כדי למצוא את קואורדינטת הפיקסלים המרכזית של AuNR בכל מסגרת בהתאם לקואורדינטת x-y (שחולצה על-ידי ImageJ/fiji).
- השתמש בפונקציה RGextraction.m כדי לתחום מטריצה של 3 x 3 פיקסלים, לחלץ את 9 ערכי עוצמת הפיזור של ערוצי R או G ולחשב ערך ממוצע (μ, המוגדר כ- R או G).
הערה: מטריצת 3 x 3 פיקסלים ממורכזת בקואורדינטות הפיקסלים המתקבלות משלב 4.3.1.
- זווית קוטבית
הערה: זווית הקוטב היא הזווית בין קו האורך של ה- AuNR לבין הציר האופטי (כפי שמוצג באיור 1), שיכולה לשקף את דינמיקת הסיבוב המרחבית (ציר Z) של ה- AuNR.- השתמש בפונקציה של polarangle.m כדי לחשב את זוויות הקוטב (θ) בשיטת הדיפרנציאל הדו-ערוצית22,
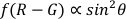 .
.
- השתמש בפונקציה של polarangle.m כדי לחשב את זוויות הקוטב (θ) בשיטת הדיפרנציאל הדו-ערוצית22,
5. ניתוח נתונים
הערה: מסגרת ניתוח נתונים שיטתית וחזקה חיונית לביצועים וליעילות של שיטות ניתוח SPT. התוכנה המותאמת אישית שנכתבה ב- MATLAB משמשת (https://github.com/fenggeqd/JOVE-2020/tree/master/Analysis_parameters). תוכנת גרף וניתוח (ראה טבלת חומרים)משמשת לציור החלקות.
- פרמטרי ניתוח
- השתמש בקבצי Script של csv_data_extract_dis_vel_ss.m ו- csv_data_MSD.m כדי לחשב פרמטרים דינאמיים בהתאם לנוסחאות המוצגות בטבלה 1.
הערה: פרמטרים אלה משמשים לניתוח הדינמיקה של AuNRs ומורכבים משלושה חלקים. (1) פרמטרים הקשורים למסלול:תזוזה,גודל צעד, מהירות, רדיוס של gyration (Rg),וזווית סיבוב (T a ); (2) פרמטרי MSD: מקדם דיפוזיה (Dt)ומעריך דיפוזיה חריג (α); ו-(3) פרמטרים הקשורים לסיבוב: זווית קוטב ויכולת סיבובית (σ).
- השתמש בקבצי Script של csv_data_extract_dis_vel_ss.m ו- csv_data_MSD.m כדי לחשב פרמטרים דינאמיים בהתאם לנוסחאות המוצגות בטבלה 1.
| אמצעי | הגדרה | משמעות פיזית | |
| הזחה | 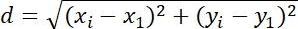 | שינויים במיקום האובייקטים | |
| גודל שלב | 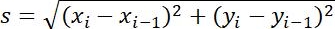 | מרחק בין שתי נקודות סמוכות | |
| מהירות |  | מהירות תנועת אובייקטים | |
| Rg | 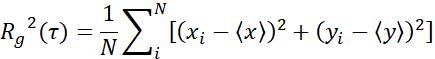 | הזזת טווח אובייקטים במרווח זמן ספציפי | |
| Ta | 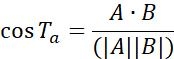 | כיוון תנועה של אובייקטים בין שתי נקודות סמוכות | |
| MSD | 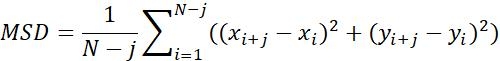 | מרחק נע ממוצע של אובייקטים במרווח זמן מסוים | |
| Dt | 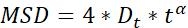 | יכולת דיפוזיה של אובייקטים | |
| α | 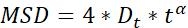 | דיפוזיה רגילה (α ~ 1) | |
| זווית קוטבית |  | מידע כיוון תלת-ממדי של אובייקטים | |
| σ |  | מידת הפיזור של ערכת הנתונים של זווית הקוטב | |
טבלה 1: שלושה סוגים של פרמטרים המשמשים לניתוח. אלה כוללים פרמטרים הקשורים למסלול (הזחה, גודל צעד, מהירות, Rg ו- Ta),פרמטרי MSD (MSD, Dt ו- α) ופרמטרים הקשורים לסיבוב (זווית קוטבית ויכולת סיבובית).
- ניתוח חזותי של מסלול
הערה: הדמיית מסלול יכולה להציג באופן אינטואיטיבי את ההטרוגניות המרחבית של תנועת החלקיקים ואת התפלגות המסלול (קואורדינטות) של פרמטרים דינמיים, כגון מסלול מיפוי זמן, מסלול מיפויR-ו- Dt,ומסלול מיפוי זווית קוטבית. מסלולי מיפוי צוירו באמצעות תוכנת הגרפים והניתוח.- הגדר קואורדינטת x כ- X, קואורדינטת y כ- Y וזמן (Rg, Dt, זווית קוטב) כ- Z.
- לחץ על "התווה | | עלילת פיזור התוויית מיפוי צבעים".
- הוסף את סרגל הצבע.
- ניתוח MSD
הערה: פעילות תנועה ומצב תנועה של החלקיקים ניתן להשיג על ידי ניתוח MSD23. ככל שה-Dtגדול יותר , כך תנועת הדיפוזיה של החלקיקים פעילה יותר. כאשר α ~ 1, חלקיקים לעשות תנועת דיפוזיה נורמלית, אחרת הם מבצעים תנועת דיפוזיה חריגה.- MSD-τ דמויות
- הגדר מרווח זמן (τ) כ- X, נתוני MSD כ- Y.
- לחץ על "התווה | עלילת פיזור"
- התאם נתונים על-ידי לחיצה על "נתח | התאמת | התאמת עקומה לא ליניארית (פונקציה: Allometricl)".
- דמות לוגריתמית כפולה של MSD-τ
הערה: MSD-τ דמות לוגריתמית כפולה, אשר השיפוע שלה הוא α ליירט הוא Dt, יכול להציג באופן אינטואיטיבי תנועת חלקיקים.- הגדר מרווח זמן לוגריתמי כ- X ו- MSD לוגריתמי כ- Y.
- לחץ על "התווה | חלקת פיזור".
- התאם נתונים על-ידי לחיצה על "נתח | התאמת | התאמת עקומה ליניארית".
- איור D-τ (MSD/4t)
הערה: D= MSD/4τ, היא פונקציה של זמן τ וגורם אנומליה α, ואת הנתון D-τ מראה ישירות את השינוי של מקדם דיפוזיה עם הזמן. כאשר D עולה עם הזמן, α גדול מ 1 וחלקיקים לעשות תנועה superdiffusion.- הגדר מרווח זמן לוגריתמי (τ) כ- X ו- D לוגריתמי כ- Y.
- לחץ על "התווה | חלקת פיזור".
- התאם נתונים על-ידי לחיצה על "נתח | התאמת | התאמת עקומה לא ליניארית (פונקציה: "Allometricl")".
הערה: ככל שהמסלולים קצרים יותר, כך אי הדיוק של הערכות הדיפוזיה גבוה יותר. באופן כללי, Dt ו- α באמצעות ניתוח MSD-τ של זמן מרווח זמן ארוך (> 30 τ) הושגו. עם זאת, התאמה פשוטה ומחוספסת תחליק את פרטי התנועה. לפיכך, ניתוח MSD-τ של זמן מרווח זמן קצר (< 10 τ) צריך להתבצע כדי לנתח את התנהגות התנועה של חלקיקים בזמן קצר.
- MSD-τ דמויות
- ניתוח סטטיסטי
- ניתוח סטטיסטי מרובה חלקיקים
הערה: ניתוח סטטיסטי מרובה חלקיקים יכול לשקף את מצב התנועה של חלקיקים באזור מרחבי, אשר בעקיפין מציין את סביבת ההטרוגניות המרחבית. לדוגמה, אם ההיסטוגרמה של Dt מציגה התפלגות בקנה מידה גדול או התפלגות של פסגות מרובות, זה אומר שפעילות התנועה של חלקיקים היא הטרוגנית.- הגדר פרמטרים דינאמיים (כגון Dt, Rg, תזוזה מרבית) כ- Y.
- לחץ על "התווה | היסטוגרמה".
- לחץ פעמיים על ההיסטוגרמה והגדר את גודל החלוקה או את מספר החטיבות. לחץ על "החל".
- ניתוח סטטיסטי של חלקיק יחיד
הערה: הניתוח הסטטיסטי של חלקיקים בודדים יכול להראות את התנהגות התנועה של חלקיקים בודדים, אשר גם בעקיפין משקף את ההטרוגניות spatiotemporal של הסביבה שמסביב.- מסגרות מרובות
- חשב פרמטרים דינמיים (כגון Ta, גודל צעד, זווית קוטבית) של כל המסגרות של מסלול ארוך יחיד, והעתק לטבלת המקור והגדר כ- Y.
- לחץ על "התווה | היסטוגרמה".
- לחץ פעמיים על ההיסטוגרמה והגדר את גודל החלוקה או את מספר החטיבות. לחץ על "החל".
הערה: אם גם Ta וגם גודל צעד מציגים התפלגות ערך קטנה, החלקיקים עושים תנועת דיפוזיה סופר צעד קטן.
- הזזת חלונות
- חשב פרמטרים דינמיים (כגון Rg, Dt) של כל המסגרות בעלות מסלול ארוך יחיד באמצעות שיטת חלון נע (11 מסגרות), והעתק לטבלת המקור והגדר כ- Y.
- לחץ על "התווה | היסטוגרמה".
- לחץ פעמיים על ההיסטוגרמה והגדר את גודל החלוקה או את מספר החטיבות. לחץ על "החל".
- מסגרות מרובות
- ניתוח סטטיסטי מרובה חלקיקים
- ניתוח סדרות זמן
הערה: ניתוח סטטיסטי יכול לחשוף את מצב התנועה של NPs, וניתוח סדרת זמן יכול להציג את התנהגות התנועה כתוספת. שילוב של מספר פרמטרים של סדרות זמן, הוא יכול להפלות את התנהגות התנועה של NPs ברמות הזמניות והמרחביות.- הגדר זמן כ- X, פרמטרים מסדרת זמן כ- Y (כגון תזוזה, זווית קוטב ו- Rg).
- לחץ על "התווה | | דיאגרמות מרובות חלונית | התוויית נתונים מוערמת שורה + סימן".
Access restricted. Please log in or start a trial to view this content.
תוצאות
בפרוטוקול נעשה שימוש ב- CTAB-AuNRs ללא שינוי של 40 x 85 ננומטר. כפי שמוצג באיור 2B, המקסימום הפלסמוני האורך שלו הוא ~ 650 ננומטר (אזור אדום) ותהודה רוחבית היא ב 520 ננומטר (אזור ירוק). ספרות קודמת חשפה כי המאפיינים האופטיים (כגון עוצמת LSPR) של AuNRs פלסמוני ישתנו באופן משמעותי עם קוטרם
Access restricted. Please log in or start a trial to view this content.
Discussion
הפרוטוקול המוצג משמש כדי ללמוד את הדינמיקה של AuNRs על קרום התא. הפרוטוקול מורכב מארבעה חלקים, כולל הדמיה מיקרוסקופית, חילוץ נתונים, חישוב פרמטרים דינמיים ושיטות ניתוח נתונים, וכל חלק הוא גמיש ואוניברסלי. לכן, ישנם יישומים עתידיים אפשריים רבים, למשל, לימוד תנועה של מולקולות קרום הקשורות NP על ?...
Access restricted. Please log in or start a trial to view this content.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי הקרן הלאומית למדעי הטבע של סין עם מספרי מענקים של 21425519, 91853105 ו 21621003.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| CTAB coated gold nanorods(CTAB-AuNRs) | Nanoseedz | NR-40-650 | 85 nm * 40 nm |
| Color CMOS camera | Olympus | DP74 | Japan |
| Coverslips | Citoglas | z10212222C | 22*22 mm |
| Dark-field microscopy | Nikon | 80i | upright microscope |
| Fetal bovine serum (FBS) | Gibco | 10099141 | |
| Fiji | National Institutes of Health | 2.0.0-rc-69/1.52 p | a distribution of ImageJ |
| Grooved glass slide | Sail brand | 7103 | Single concave |
| Image J | National Institutes of Health | 1.52 j | |
| MATLAB | MathWorks | R2019b | |
| MATLAB Code | https://github.com/fenggeqd/JOVE-2020 | ||
| Minimum essential medium (MEM) | Gibco | 10-010-CVR | with phenol red |
| Minimum essential medium (MEM) | Gibco | 51200038 | no phenol red |
| Origin | OriginLab | Origin Pro 2018C | |
| Penicillin-streptomycin | Gibco | 15140122 | |
| Plastic cell culture dishes | Falcon | 353002 | |
| Plastic cell culture dishes | Falcon | 353001 | 35*10 mm |
| U87 MG cell | American Type Culture Collection | ATCC HTB-14 | a human primary glioblastoma cell line |
References
- Rees, P., Wills, J. W., Brown, M. R., Barnes, C. M., Summers, H. D. The origin of heterogeneous nanoparticle uptake by cells. Nature Communication. 10 (1), 2341(2019).
- Behzadi, S., et al. Cellular uptake of nanoparticles: journey inside the cell. Chemical Society Reviews. 46 (14), 4218-4244 (2017).
- Shen, H., et al. Single Particle Tracking: From Theory to Biophysical Applications. Chemical Reviews. 117 (11), 7331-7376 (2017).
- Saxton, M. J. Single-particle tracking: connecting the dots. Nature Methods. 5 (8), 671-672 (2008).
- Kusumi, A., et al. Dynamic organizing principles of the plasma membrane that regulate signal transduction: commemorating the fortieth anniversary of Singer and Nicolson's fluid-mosaic model. Annual Review of Cell and Developmental Biology. 28, 215-250 (2012).
- Jacobson, K., Liu, P., Lagerholm, B. C. The Lateral Organization and Mobility of Plasma Membrane Components. Cell. 177 (4), 806-819 (2019).
- Sezgin, E., Levental, I., Mayor, S., Eggeling, C. The mystery of membrane organization: composition, regulation and roles of lipid rafts. Nature Reviews Molecular Cell Biology. 18 (6), 361-374 (2017).
- von Diezmann, A., Shechtman, Y., Moerner, W. E. Three-Dimensional Localization of Single Molecules for Super-Resolution Imaging and Single-Particle Tracking. Chemical Reviews. 117 (11), 7244-7275 (2017).
- Rosenberg, J., Huang, J. Visualizing Surface T-Cell Receptor Dynamics Four-Dimensionally Using Lattice Light-Sheet Microscopy. Journal of Visualized Experiments. (155), e59914(2020).
- Kusumi, A., Tsunoyama, T. A., Hirosawa, K. M., Kasai, R. S., Fujiwara, T. K. Tracking single molecules at work in living cells. Nature Chemical Biology. 10 (7), 524-532 (2014).
- Rocha, J. M., Gahlmann, A. Single-Molecule Tracking Microscopy - A Tool for Determining the Diffusive States of Cytosolic Molecules. Journal of Visualized Experiments. (151), e59387(2019).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing protein-DNA interactions in live bacterial cells using photoactivated single-molecule tracking. Journal of Visualized Experiments. (85), e511177(2014).
- Pinaud, F., Clarke, S., Sittner, A., Dahan, M. Probing cellular events, one quantum dot at a time. Nature Methods. 7 (4), 275-285 (2010).
- Mehidi, A., et al. Transient Activations of Rac1 at the Lamellipodium Tip Trigger Membrane Protrusion. Current Biology. 29 (17), 2852-2866 (2019).
- Ye, Z., Wang, X., Xiao, L. Single-Particle Tracking with Scattering-Based Optical Microscopy. Analytical Chemistry. 91 (24), 15327-15334 (2019).
- Pan, Q., Zhao, H., Lin, X., He, Y. Spatiotemporal Heterogeneity of Reactions in Solution Observed with High-Speed Single-Nanorod Rotational Sensing. Angewandte Chemie. International Ed. in English. 58 (25), 8389-8393 (2019).
- Taylor, R. W., et al. Interferometric scattering microscopy reveals microsecond nanoscopic protein motion on a live cell membrane. Nature Photonics. 13 (7), 480-487 (2019).
- Chen, K., et al. Characteristic rotational behaviors of rod-shaped cargo revealed by automated five-dimensional single particle tracking. Nature Communication. 8 (1), 887(2017).
- Xu, D., He, Y., Yeung, E. S. Y. Direct observation of the orientation dynamics of single protein-coated nanoparticles at liquid/solid interfaces. Angewandte Chemie. International Ed. in English. 53 (27), 6951-6955 (2014).
- Lehui, X., Yan, H., Edward, S. Y. Three Dimensional Orientational Imaging of Nanoparticles with Darkfield Microscopy. Analytical Chemistry. 82, 5268-5274 (2010).
- Ge, F., Xue, J., Wang, Z., Xiong, B., He, Y. Real-time observation of dynamic heterogeneity of gold nanorods on plasma membrane with darkfield microscopy. Science China Chemistry. 62, 1072-1081 (2019).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Janczura, J., Weron, A. Ergodicity testing for anomalous diffusion: small sample statistics. The Journal of Chemical Physics. 142 (14), 144103(2015).
- Kim, D. H., et al. Single particle tracking-based reaction progress kinetic analysis reveals a series of molecular mechanisms of cetuximab-induced EGFR processes in a single living cell. Chemical Science. 8 (7), 4823-4832 (2017).
- Kurzthaler, C., et al. Probing the Spatiotemporal Dynamics of Catalytic Janus Particles with Single-Particle Tracking and Differential Dynamic Microscopy. Physical Review Letters. 121 (7), 078001(2018).
- Lin, X., Pan, Q., He, Y. In situ detection of protein corona on single particle by rotational diffusivity. Nanoscale. 11 (39), 18367-18374 (2019).
- Wei, L., et al. Sub-diffraction-limit localization imaging of a plasmonic nanoparticle pair with wavelength-resolved dark-field microscopy. Nanoscale. 9 (25), 8747-8755 (2017).
- Cheng, X., Dai, D., Xu, D., He, Y., Yeung, E. S. Subdiffraction-limited plasmonic imaging with anisotropic metal nanoparticles. Analytical Chemistry. 86 (5), 2303-2307 (2014).
- Belyy, V., et al. PhotoGate microscopy to track single molecules in crowded environments. Nature Communication. 8, 13978(2017).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved