Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Tek Nanopartikül Darkfield Mikroskopisi Kullanarak Hücre Zarında Altın Nanorodların Difüzyonel Dinamiklerini Görselleştirme
Bu Makalede
Özet
Burada, hücre zarındaki altın nanorodların (AuNR) dinamiklerini izlemek için geleneksel karanlık alan mikroskopisinin kullanımını gösteriyoruz. Tek AuNR'lerin konumu ve yönü ImageJ ve MATLAB kullanılarak algılanır ve AuNR'lerin difüzif durumları tek parçacık izleme analizi ile karakterize edilir.
Özet
Hücre zarı üzerindeki nanopartiküllerin difüzyonel dinamiklerinin analiz etmek, hücresel alım sürecinin daha iyi anlaşılmasında önemli bir rol oynar ve nano-tıp teslimatının rasyonel tasarımı için teorik bir temel sağlar. Tek parçacık izleme (SPT) analizi, hücre zarı üzerindeki bireysel nanopartiküllerin konumunu ve yönünü araştırabilir ve çevirisel ve dönme durumlarını ortaya getirebilir. Burada, canlı hücre zarında altın nanorodların (AuNR) dinamiklerini izlemek için geleneksel karanlık alan mikroskopisinin nasıl kullanılacağını gösteriyoruz. Ayrıca ImageJ ve MATLAB kullanarak AuNR'lerin konumunun ve yönünün nasıl çıkarılacağını ve AuNR'lerin difüzör durumlarının nasıl karakterize edeceğini gösteriyoruz. Yüzlerce parçacığın istatistiksel analizi, tek AuNR'lerin U87 MG hücre zarının yüzeyinde Brownian hareketi gerçekleştirdiğini göstermektedir. Bununla birlikte, bireysel uzun yörünge analizi, AuNR'ların membran üzerinde iki belirgin şekilde farklı hareket durumlarının olduğunu göstermektedir, yani uzun menzilli taşıma ve sınırlı alan hapsi. SPT yöntemlerimiz potansiyel olarak farklı biyolojik hücrelerdeki yüzey veya hücre içi parçacık difüzyonunu incelemek için kullanılabilir ve karmaşık hücresel mekanizmaların araştırılması için güçlü bir araç haline gelebilir.
Giriş
Membran üzerindeki nanopartiküllerin (NP) dinamikleri, hücre fonksiyonlarının, viral veya bakteriyel enfeksiyonların anlaşılması ve yapay nanomedikal iletim sistemlerinin geliştirilmesi için gerekli olan hücresel alım süreci ile yakından ilişkilidir1,2. Tek parçacık izleme (SPT) tekniği, NPs3,4'ünheterojen davranışlarını karakterize etmek için sağlam bir araçtır. Genel olarak, hücre zarı akışkandır, bu da proteinler ve lipitler gibi bileşenlerin plazma membran düzleminde yanal olarak hareket edebileceği anlamına gelir5,6,7. Membran organizasyonu ve yapısının mekansal karmaşıklığı, NP'ler ve membran arasındaki etkileşimin mekansal heterojenliğine yol açabilir. Bu nedenle, NP'lerin membran üzerindeki hareketinin doğrudan görselleştirilmesi hem yüksek uzamsal hem de zamansal çözünürlük gerektirir.
Canlı hücrelerdeki tek tek parçacıkların lokalizasyonunu onlarca nanometre uzamsal çözünürlük ve milisaniyelik bir zaman çözünürlüğü ile izleyen tek parçacık izleme mikroskopisi, NP'leri veya membran molekülleri dinamiklerini incelemek için iyi geliştirilmiştir8,9. Floresan bazlı mikroskobik görüntüleme teknikleri canlı hücre ortamında NP/molekülleri gözlemlemek için değerli araçlar haline gelmiştir9,10,11,12. Örneğin, numunenin ince katmanlarını (~100 nm) yüksek patiotemporal çözünürlüğe sahip substrat/çözelti arayüzünde gösteren toplam iç yansıma floresan mikroskopisi, membran molekülleri dinamikleri13,14çalışmalarında yaygın olarak kullanılmıştır. Bununla birlikte, düşük yoğunluklu ve hızlı geri dönüşü olmayan fotobleaching gibi tek floroforların doğal dezavantajları izleme doğruluğunu ve süresini azaltır13. Bu nedenle, floresan probların yerini alan floresan olmayan plazmonik NP'ler, benzersiz optik özellikleri nedeniyle uzun süreli görüntüleme çalışmalarında giderek daha fazla dikkat çekmiştir15. Plazmonik NP problarının saçılma sinyallerine dayanarak, karanlık alan mikroskopisi (DFM)16,interferometrik saçılma (iSCAT) mikroskopisi 17 ve diferansiyel girişim kontrastı mikroskopisi(DICM) 18 gibi biyolojik süreçlerin mekanizmasını incelemek için çeşitli optik mikroskobik görüntüleme teknolojileri kullanılmıştır. Ek olarak, AuNR'ların hareket ve dönme dinamiği DFM ve DICM18 , 19,20,21,22kullanılarak elde edilebilir. Tipik olarak, bir SPT deneyinde, nesnenin hareketi optik mikroskop tarafından kaydedilir ve daha sonra SPT analiz yöntemleri ile analiz edilir3. Bireysel NP'ler tarafından oluşturulan zaman çözümlenmiş yörüngeler ve oryantasyonsal açılar normalde stokastik ve heterojendir, bu nedenle çeşitli analiz yöntemleriyle bol dinamik bilgi sunmak gerekir.
Burada, DFM kullanarak Hücre zarı üzerindeki AuNR'lerin dinamiklerini izleyen, ImageJ ve MATLAB ile AuNR'lerin konumunu ve yönünü çıkaran ve AuNR'lerin difüzyonunu SPT analiz yöntemleriyle karakterize eden entegre bir protokol sunuyoruz. Bir gösteri olarak, U87 MG hücre zarında değiştirilmemiş AuNR'lerin (Cetyltrimethylammonium amonyum bromür molekülü tarafından koruyucu ajan olarak sentezlenen CTAB-AuNRs) dinamiklerini görselleştirmek için SPT protokolünün nasıl kullanılacağını burada gösteriyoruz. CTAB-AuNR'lerin biyolojik ortamda proteinleri adsorbe edebileceği, hücre zarı üzerinde hareket edebileceği ve daha sonra 2,20,22hücrelerine girebileceği gösterilmiştir. U87 MG hücre merkezi sinir sisteminin en sık görülen ve en kötü huylu tümörüdür ve membran reseptörleri anormal bir şekilde ifade edilir. Membran reseptörleri, AuNR'lerin dinamiklerini etkileyen AuNR'lerdeki proteinlerle etkileşime girebilir. Protokolümüz genellikle biyoloji alanındaki diğer SPT deneyleri için geçerlidir.
Access restricted. Please log in or start a trial to view this content.
Protokol
1. Hücre kültürü
- Fetal sığır serumu ekleyerek U87 MG hücreleri için komple ortam hazırlayın (son konsantrasyon% 10) ve penisilin-streptomisin (son konsantrasyon %1) minimum temel ortama (MEM). Hücreler alt kültürü için plastik hücre kültürü çanağı kullanın.
- Pasaj hücreleri haftada 2 ila 3 kez.
- Kültür ortamını çıkarın ve hücre tabakasını Dulbecco'nun fosfat tamponlu saliniyle (D-PBS) birleştiğinde 2 ~3 kez durulayın (%80~%90).
- Hücre kültürü çanağına 1,0 ila 2,0 mL Trypsin-EDTA çözeltisi ekleyin ve hücreler yuvarlaklaşana kadar ters bir mikroskop altında hücreleri gözlemleyin (3 ~ 5 dk).
- 3.0 mL hazırlanmış tam orta ekleyin ve hücreleri hafifçe pipetleme ile dağıtın.
- Taze hücre ortamı (3 mL) ile yeni kültür yemeğine hücre süspansiyonu (1 mL) ekleyin ve hücreleri yeniden biriktirin.
- Hücreleri nemli bir atmosferde 37 °C ve%5 CO 2'de sakla.
2. Mikroskop slayt hazırlığı
NOT: SPT deneylerinde üçüncü ila onuncu nesil U87 MG hücreleri yüksek aktivite ile kullanılmaktadır.
- Etanol (%99,9) içine daldırarak Piranha çözeltisi ile önceden temizlenmiş 22 mm × 22 mm kapak örtülerini sterilize edin.
- Kapak fişini etanol çözeltisinden çıkarmak için forseps kullanın (adım 2.1) ve alev üzerinde etanol yakarak sterilize edin. Tüm etanol yakıldıktan sonra, kapakları 2 mL hücre ortamı (fenol kırmızısı yok) ile doldurulmuş plastik bir hücre kültürü kabına (35 x 10 mm) yerleştirin.
- Kapakçık üzerindeki 1.2.3 adımından 50 μL hücre süspansiyonu ekleyin ve hücreleri eşit bir şekilde dağıtmak için kabı ileri geri ve sola ve sağa doğru hafifçe itin. Nemli bir atmosfere yerleştirin.
- Kapak üzerindeki U87 MG hücreleri % 20-% 40 izdiah (~12 h) ulaştığında, yemeğe 20 μL CTAB-AuNR (138 pM) ekleyin ve dağılın. 5 dakika boyunca nemli atmosferde kuluçkaya yaslanın.
- Piranha çözeltisi ile önceden toplanan yivli cam kaydırağın (Şekil 1) oluğuna 2.4 adımda tabaktan100μL kültür ortamını (fenol kırmızısı yok) ekleyin.
- Kapak sapını tabaktan çıkar ve mikroskop cam kaydırağının oluğunun üstüne ters çevirin (Şekil 1). Oje ile kapatın, kurumaya bırakın ve SPT deneyleri yapmak için sahneye yerleştirin.
3. Darkfield mikroskopisi ile tek parçacık izleme deneyleri yapmak (Şekil 1).
- Yağa batırılmış karanlık alan kondenserine (NA 1.43-1.20) bir damla yağ yerleştirin ve kondenserin cam kaydırağa temas etmesini sağlamak için düğmeyi çevirin.
- Kapak camının üstüne bir damla yağ koyun ve 60x yağ daldırma hedefini (NA 0.7–1.25) yağa dokunmak için odaklama düğmesini çevirin.
- Işık kaynağını açın ve görüntüleme düzlemini odaklamak için odaklama düğmesini hafifçe çevirin.
NOT: Görüş alanında arka plan siyah, hücreler parlak ve CTAB-AuNR'ler (en boy oranı~2:1, Şekil 2)küçük renkli (kırmızı, sarı veya yeşil) saçılma noktalarıdır. - Renkli bir CMOS kamera ile örnek saçılma ışığını yakalayın. Görüntüleri kaydetmek için TIFF formatını kaydetmek ve dışa vermek için yazılımdaki "Kamera simgesi" ni tıklatın.
4. Veri toplama
- Tek uzun vadeli yörüngeyi ayıkla
- Zaman serisi koyu alan görüntülerini "RGB renk" modundan "8 bit" moduna dönüştürmek için Şekil 3'te açıklandığı gibi çalıştırın. Resim J'de Resim | | yazın 8 bit. Karşıtlığı ayarlamak için Görüntü | | ayarlama Parlaklık | Karşıtlık.
- Bir hedef parçacık seçin ve arka planı "Ctrl+X" ile kutulayıp silerek zaman serisi arka planlarını kesin.
- "Eklentiler | Parçacık İzleyici Klasik | Parçacık İzleyici".
- Yarıçapı 6' ya, Kesmeyi 0' a ve Yüzdelik'i %0,01 olarak ayarlayın.
NOT: Parçacığı algılamak için Önizleme yardımıyla üç parametrenin üzerinde ayarlayın. Yarıçapın hedeflenen parçacıktan biraz daha büyük ve en küçük parçacıklar arası ayırmadan daha küçük olduğundan emin olun. Yüzdebirlik, aday parçacıklar olan yoğunluk dağılımının alt sınırıdır. - Bağlantı Aralığını 5'e ve Yer Değiştirme'yi 10 olarak ayarlayın.
NOT: Parçacığı ardışık bitişik çerçeveler arasında bağlamak için yukarıdaki iki parametreyi ayarlayın. Öteleme, bir parçacığın iki sonraki kare arasında hareket edebileceği maksimum pikseldir ve Bağlantı Aralığı, en iyi karşılık gelen eşleşmeyi belirlerken dikkate alınması gereken ardışık kare sayısıdır. - Sonuçları görmek için ParticleTracker Sonuçları penceresini açmak için "Tamam" ı tıklatın.
- Oluşturulan yörüngeleri incelemek için "Tüm Yörüngeleri Görselleştir" i tıklatın.
- Yazılım tarafından oluşturulan yörünge AuNR'nin hareketli yörüngesiyle eşleşmiyorsa, algılanan parçacıkları farklı bağlantı aralığı ve yüzdebirlik parametrelerle yeniden bağlamak için üstteki "Parçacıkları Yeniden Bağla" menüsünü tıklatın.
- Yazılım tarafından oluşturulan yörünge ve AuNR'nin hareketli yörüngesi eşleşiyorsa sonuçları kaydetmek için "Tam Raporu Kaydet" i tıklatın.
NOT: Resim J ile tek bir uzun vadeli yörünge çıkarma örneği Şekil 4'tegösterilmiştir.
- Çok yörüngeli ayıklama
NOT: Fiji ile çok yörüngeli çıkarma örneği Şekil 5'te gösterilmiştir. Birkaç aşama vardır ve her aşama izleme sürecinde bir adım oluşturur. Her adımın sonucu hemen görüntülenir, bu da çıktı tatmin edici olmadığında kullanıcının yeniden ayar ayarlarına geri dönmesini sağlar.- Zaman serisi koyu alan görüntülerini "RGB renk" modundan "8 bit" moduna dönüştürmek için Şekil 3'te açıklandığı gibi çalıştırın. Resim J yolunda "Resim | | yazın 8 bit". Kontrastlarını ayarlamak için "Resim | | ayarlama Parlaklık | Kontrast".
- "Eklentiler | İzleme | TrackMate". "İleri" yi tıklatın.
- Açılır menüden "LoG dedektörü" nü seçin ve " İleri "yitıklayın.
- Dedektör konfigürasyon panelinin parametrelerini ayarlayın. Tahmini blob çapı 10 olarak ayarlayın, Eşiği 0 olarak ayarlayın ve "Alt Piksel Yerelleştirmesi Yap" ı seçin.
- İlk nokta filtreleme panelini açmak için "İleri" yi tıklatın.
NOT: Hedef noktaları daha da optimize etmek için çeşitli parametreler ayarlanabilir. Bu örnekte, başka hiçbir parametre ayarlandı. - "İleri" yi tıklatın ve açılır menüden "HyperStack displayer" ı seçin.
- Spot filtreleme panelini açmak için "İleri" yi tıklatın. Kaliteyi 1,88'in, X'i 38,86'nın, Y'yi 56,54'ün üzerine ayarlayın.
- "İleri" yi tıklayın ve açılır menüden "Basit LAP izleyici" yi seçin. Üç parametreyi, yani Bağlama maksimum mesafesini = 15, Boşluk kapatma maksimum mesafesini = 15 ve Boşluk kapatma maksimum çerçeve boşluğu = 5'i ayarlayarak basit LAP izleyicisini yapılandırın.
NOT: Bağlama-maksimum mesafe, bir noktanın iki kare arasındaki maksimum yer değiştirmesidir. Boşluk kapatma maksimum mesafesi, iki segmentin maksimum deplasmanıdır. Boşluk kapatan maksimum çerçeve boşluğu, köprülenecek iki nokta arasındaki en büyük çerçevedir. - İzlemek için "İleri" yi tıklatın. İşiniz bittiğinde, "İleri" yi tıklatmaya devam edin.
- Parçalarda filtreler ayarlayın, örneğin 300'ün üzerindeki parçadaki nokta sayısını ayarlayın.
- Son kaydetme paneli açılana kadar "İleri" yi tıklatarak devam edin. Açılan menüden "Parçaları XML dosyasına aktar" ı seçin ve csv biçiminde kaydetmek için " Yürüt "ütıklatın.
- R/G değerleri
NOT: R ve G kanallarında hedeflenen AuNR'lerin saçılma yoğunlukları MATLAB (https://github.com/fenggeqd/JOVE-2020/tree/master/RGandPolarangle) ile yazılmış bir kod kullanılarak renkli koyu alan görüntülerinden elde edilir ve ekstraksiyon prensibi Şekil 6'dasunulmuştur.- X-y koordinatı (ImageJ/Fiji tarafından ayıklanır) göre her karede AuNR'nin orta piksel koordinatını bulmak için xycoordination.m işlevini kullanın.
- RGextraction işlevini kullanın.m 3 x 3 piksel matrisi sınırlandırmak, R veya G kanallarının 9 saçılma yoğunluklu değerlerini ayıklamak ve ortalama bir değer hesaplamak için (μ, R veya G olarak tanımlanır).
NOT: 3 x 3 piksel matrisi, 4.3.1 adımından elde edilen piksel koordinatları üzerinde ortalanır.
- Kutup açısı
NOT: Kutup açısı, AuNR'nin boylamı ile AuNR'nin uzamsal (Z ekseni) dönme dinamiklerini yansıtabilen optik eksen (Şekil 1'degösterildiği gibi) arasındaki açıdır.- Polarangle.m işlevini kullanarak kutup açılarını (φ) çift kanallı diferansiyel yöntemiyle hesaplamakiçin 22,
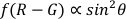 .
.
- Polarangle.m işlevini kullanarak kutup açılarını (φ) çift kanallı diferansiyel yöntemiyle hesaplamakiçin 22,
5. Veri analizi
NOT: SPT analiz yöntemlerinin performansı ve verimliliği için sistematik ve sağlam bir veri analizi çerçevesi gereklidir. MATLAB ile yazılmış özel yazılım kullanılır (https://github.com/fenggeqd/JOVE-2020/tree/master/Analysis_parameters). Çizimleri çizmek için bir grafik ve analiz yazılımı (bkz. Malzeme Tablosu)kullanılır.
- Analiz parametreleri
- Dinamik parametreleri Tablo 1'degösterilen formüllere göre hesaplamak için csv_data_extract_dis_vel_ss.m ve csv_data_MSD.m komut dosyalarını kullanın.
NOT: Bu parametreler AuNR'lerin dinamiklerini analiz etmek için kullanılır ve üç bölümden oluşur. (1) Yörünge ile ilgili parametreler: yer değiştirme, adım boyutu, hız, gyrasyon yarıçapı (Rg)ve dönüş açısı (Ta); (2) MSD parametreleri: difüzyon katsayısı (Dt)ve anormal difüzyon üssü (α); ve (3) Döndürme ile ilgili parametreler: kutup açısı ve dönme yeteneği (σ).
- Dinamik parametreleri Tablo 1'degösterilen formüllere göre hesaplamak için csv_data_extract_dis_vel_ss.m ve csv_data_MSD.m komut dosyalarını kullanın.
| Önlem -ler | Tanımı | Fiziksel anlam | |
| Deplasman | 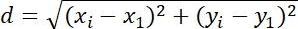 | Nesnelerin konumundaki değişiklikler | |
| Adım boyutu | 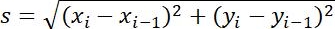 | bitişik iki nokta arasındaki mesafe | |
| Hız |  | Nesnelerin hareket hızı | |
| Rg | 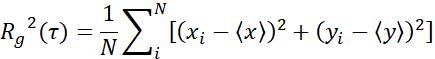 | Nesnelerin aralığını belirli bir zaman aralığında taşıma | |
| Ta | 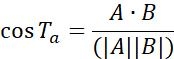 | nesnelerin iki bitişik nokta arasındaki hareket yönü | |
| Msd | 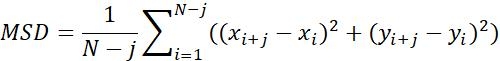 | Belirli bir zaman aralığındaki nesnelerin ortalama hareket mesafesi | |
| Dt | 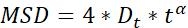 | Nesnelerin difüzyon yeteneği | |
| Α | 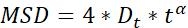 | Normal difüzyon (α~1) | |
| Kutup açısı |  | Nesnelerin 3D yönlendirme bilgileri | |
| Σ |  | Kutup açısı veri kümesinin dağılım derecesi | |
Tablo 1: Analiz için kullanılan üç tür parametre. Bunlar arasında yörünge ile ilgili parametreler (yer değiştirme, adım boyutu, hız, Rg ve Ta),MSD parametreleri (MSD, Dt ve α) ve döndürme ile ilgili parametreler (kutup açısı ve dönme kabiliyeti) saydır.
- Yörüngenin görsel analizi
NOT: Yörünge görselleştirme, parçacık hareketinin mekansal heterojenliğini ve zaman haritalama yörüngesi, Rg- ve Dt-haritalama yörüngesi ve kutup açısı haritalama yörüngesi gibi dinamik parametrelerin yörünge (koordinat) dağılımını sezgisel olarak sunabilir. Haritalama yörüngeleri grafik ve analiz yazılımı kullanılarak çizilmiştir.- x koordinatı X, y koordinatı Y ve zaman (Rg, Dt, kutup açısı) Z olarak ayarlayın.
- Tıklayın "Çizim | Saçılma arsa | Renk eşleme Çizimi".
- Renk çubuğunu ekleyin.
- MSD analizi
NOT: Parçacıkların hareket aktivitesi ve hareket modu MSD analizi ile elde edilebilir23. Dtne kadar büyükse, parçacıkların difüzyon hareketi o kadar aktif olur. α ~ 1 olduğunda, parçacıklar normal difüzyon hareketi yaparlar, aksi takdirde anormal difüzyon hareketi gerçekleştirirler.- MSD-ν rakamları
- Zaman aralığını (φ) X, MSD verilerini Y olarak ayarlayın.
- Tıklayın "Çizim | Saçılma arsası"
- "Analiz | Uygun | Doğrusal olmayan eğri montajı (fonksiyon: alometrik)".
- MSD-ν çift logaritmik şekil
NOT: Eğimi α ve kesişimi Dtolan MSD-ν çift logaritmik figür, sezgisel olarak parçacık hareketi sunabilir.- Logaritmik zaman aralığını X, logaritmik MSD'yi Y olarak ayarlayın.
- Tıklayın "Çizim | Saçılma arsa".
- "Analiz | Uygun | Doğrusal eğri uydurma".
- D-ν rakamı (MSD/4t)
NOT: D=MSD/4τ, α zaman ve anomali faktörünün bir fonksiyonudur ve D-ν şekli doğrudan difüzyon katsayısının zamanla değişimini gösterir. D zamanla arttığında, α 1'den büyüktür ve parçacıklar süper difüzyon hareketi yapar.- Logaritmik zaman aralığını (δ) X, logaritmik D'yi Y olarak ayarlayın.
- Tıklayın "Çizim | Saçılma arsa".
- "Analiz | Uygun | Doğrusal olmayan eğri montajı (İşlev: "Allometrik")".
NOT: Yörüngeler ne kadar kısa olursa, difüzyon tahminlerinin yanlışlığı o kadar yüksek olur. Genel olarak, uzun aralık süresinin MSD-ν analizi yoluyla Dt ve α elde edildi (> 30 φ). Ancak, basit ve kaba montaj hareket ayrıntılarını yumuşatacaktır. Bu nedenle, parçacıkların hareket davranışını kısa sürede analiz etmek için kısa aralık süresinin (< 10 δ) MSD-ν analizi yapılmalıdır.
- MSD-ν rakamları
- İstatistiksel analiz
- Çok parçacıklı istatistiksel analiz
NOT: Çok parçacıklı istatistiksel analiz, uzamsal bir bölgedeki parçacıkların hareket durumunu yansıtabilir ve bu da dolaylı olarak mekansal heterojenlik ortamını gösterir. Örneğin, Dt histogramı büyük ölçekli bir dağılım veya çok tepeli bir dağılım sergiliyorsa, parçacıkların hareket aktivitelerinin heterojen olduğu anlamına gelir.- Dinamik parametreleri (Dt, Rg, maksimum yer değiştirme gibi) Y olarak ayarlayın.
- Tıklayın "Çizim | Histogram".
- Histogramı çift tıklatın ve bölme boyutunu veya bölüm sayısını ayarlayın. "Uygula" yı tıklatın.
- Tek parçacıklı istatistiksel analiz
NOT: Tek parçacıkların istatistiksel analizi, çevrenin mekanotemporal heterojenliğini de dolaylı olarak yansıtan bireysel parçacıkların hareket davranışını gösterebilir.- Birden çok kare
- Tek bir uzun yörüngenin tüm karelerinin dinamik parametrelerini (Ta, adım boyutu, kutup açısı gibi) hesaplayın ve Origin tablosuna kopyalayıp Y olarak ayarlayın.
- Tıklayın "Çizim | Histogram".
- Histogramı çift tıklatın ve bölme boyutunu veya bölüm sayısını ayarlayın. "Uygula" yı tıklatın.
NOT: Hem Ta hem de adım boyutu küçük bir değer dağılımı sergiliyorsa, parçacıklar küçük adımlı bir süper difüzyon hareketi yapar.
- Pencereleri taşıma
- Hareketli pencere yöntemiyle (11 kare) tek uzun yörüngenin tüm karelerinin dinamik parametrelerini (Rg, Dtgibi) hesaplayın ve Origin tablosuna kopyalayıp Y olarak ayarlayın.
- Tıklayın "Çizim | Histogram".
- Histogramı çift tıklatın ve bölme boyutunu veya bölüm sayısını ayarlayın. "Uygula" yı tıklatın.
- Birden çok kare
- Çok parçacıklı istatistiksel analiz
- Zaman serisi analizi
NOT: İstatistiksel analiz NP'lerin hareket durumunu ortaya alabilir ve zaman serisi analizi hareket davranışını ek olarak sunabilir. Birkaç zaman serisi parametresini birleştirerek, NP'lerin hareket davranışını zamansal ve uzamsal düzeylerde ayırt edebilir.- Zamanı X, zaman serisi parametrelerini Y (yer değiştirme, kutup açısı ve R g gibi) olarakayarlayın.
- Tıklayın "Çizim | Çok bölmeli diyagram | Yığılmış çizim | Çizgi + Sembol".
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
Protokolde değiştirilmemiş 40 x 85 nm CTAB-AuNR kullanıldı. Şekil 2B'degösterildiği gibi, boyuna plazmonik maksimum değeri ~650 nm 'dir (kırmızı bölge) ve enine rezonansı 520 nm'dedir (yeşil bölge). Önceki literatürler plazmonik AuNR'lerin optik özelliklerinin (LSPR yoğunluğu gibi) çapları20,22ile önemli ölçüde değişeceğini ortaya koydu. Şekil 2C'de, U87 hücre zarında CTA...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Sunulan protokol, AuNR'lerin hücre zarı üzerindeki dinamiklerini incelemek için kullanılır. Mikroskobik görüntüleme, veri çıkarma, dinamik parametre hesaplama ve veri analizi yöntemleri olmak üzere dört bölümden oluşan protokolde her bölüm esnek ve evrenseldir. Bu nedenle, örneğin, NP bağlantılı membran moleküllerinin membran üzerindeki hareketini, NP etiketli reseptörlerin endositoz dinamiklerini, hücre içi NP'lerin dinamik analizini ve mikrotübüller boyunca vezikül kaplı NPs taşımacı...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Bu çalışma Çin Ulusal Doğa Bilimleri Vakfı tarafından 21425519, 91853105 ve 21621003 hibe numaralarıyla desteklendi.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| CTAB coated gold nanorods(CTAB-AuNRs) | Nanoseedz | NR-40-650 | 85 nm * 40 nm |
| Color CMOS camera | Olympus | DP74 | Japan |
| Coverslips | Citoglas | z10212222C | 22*22 mm |
| Dark-field microscopy | Nikon | 80i | upright microscope |
| Fetal bovine serum (FBS) | Gibco | 10099141 | |
| Fiji | National Institutes of Health | 2.0.0-rc-69/1.52 p | a distribution of ImageJ |
| Grooved glass slide | Sail brand | 7103 | Single concave |
| Image J | National Institutes of Health | 1.52 j | |
| MATLAB | MathWorks | R2019b | |
| MATLAB Code | https://github.com/fenggeqd/JOVE-2020 | ||
| Minimum essential medium (MEM) | Gibco | 10-010-CVR | with phenol red |
| Minimum essential medium (MEM) | Gibco | 51200038 | no phenol red |
| Origin | OriginLab | Origin Pro 2018C | |
| Penicillin-streptomycin | Gibco | 15140122 | |
| Plastic cell culture dishes | Falcon | 353002 | |
| Plastic cell culture dishes | Falcon | 353001 | 35*10 mm |
| U87 MG cell | American Type Culture Collection | ATCC HTB-14 | a human primary glioblastoma cell line |
Referanslar
- Rees, P., Wills, J. W., Brown, M. R., Barnes, C. M., Summers, H. D. The origin of heterogeneous nanoparticle uptake by cells. Nature Communication. 10 (1), 2341(2019).
- Behzadi, S., et al. Cellular uptake of nanoparticles: journey inside the cell. Chemical Society Reviews. 46 (14), 4218-4244 (2017).
- Shen, H., et al. Single Particle Tracking: From Theory to Biophysical Applications. Chemical Reviews. 117 (11), 7331-7376 (2017).
- Saxton, M. J. Single-particle tracking: connecting the dots. Nature Methods. 5 (8), 671-672 (2008).
- Kusumi, A., et al. Dynamic organizing principles of the plasma membrane that regulate signal transduction: commemorating the fortieth anniversary of Singer and Nicolson's fluid-mosaic model. Annual Review of Cell and Developmental Biology. 28, 215-250 (2012).
- Jacobson, K., Liu, P., Lagerholm, B. C. The Lateral Organization and Mobility of Plasma Membrane Components. Cell. 177 (4), 806-819 (2019).
- Sezgin, E., Levental, I., Mayor, S., Eggeling, C. The mystery of membrane organization: composition, regulation and roles of lipid rafts. Nature Reviews Molecular Cell Biology. 18 (6), 361-374 (2017).
- von Diezmann, A., Shechtman, Y., Moerner, W. E. Three-Dimensional Localization of Single Molecules for Super-Resolution Imaging and Single-Particle Tracking. Chemical Reviews. 117 (11), 7244-7275 (2017).
- Rosenberg, J., Huang, J. Visualizing Surface T-Cell Receptor Dynamics Four-Dimensionally Using Lattice Light-Sheet Microscopy. Journal of Visualized Experiments. (155), e59914(2020).
- Kusumi, A., Tsunoyama, T. A., Hirosawa, K. M., Kasai, R. S., Fujiwara, T. K. Tracking single molecules at work in living cells. Nature Chemical Biology. 10 (7), 524-532 (2014).
- Rocha, J. M., Gahlmann, A. Single-Molecule Tracking Microscopy - A Tool for Determining the Diffusive States of Cytosolic Molecules. Journal of Visualized Experiments. (151), e59387(2019).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing protein-DNA interactions in live bacterial cells using photoactivated single-molecule tracking. Journal of Visualized Experiments. (85), e511177(2014).
- Pinaud, F., Clarke, S., Sittner, A., Dahan, M. Probing cellular events, one quantum dot at a time. Nature Methods. 7 (4), 275-285 (2010).
- Mehidi, A., et al. Transient Activations of Rac1 at the Lamellipodium Tip Trigger Membrane Protrusion. Current Biology. 29 (17), 2852-2866 (2019).
- Ye, Z., Wang, X., Xiao, L. Single-Particle Tracking with Scattering-Based Optical Microscopy. Analytical Chemistry. 91 (24), 15327-15334 (2019).
- Pan, Q., Zhao, H., Lin, X., He, Y. Spatiotemporal Heterogeneity of Reactions in Solution Observed with High-Speed Single-Nanorod Rotational Sensing. Angewandte Chemie. International Ed. in English. 58 (25), 8389-8393 (2019).
- Taylor, R. W., et al. Interferometric scattering microscopy reveals microsecond nanoscopic protein motion on a live cell membrane. Nature Photonics. 13 (7), 480-487 (2019).
- Chen, K., et al. Characteristic rotational behaviors of rod-shaped cargo revealed by automated five-dimensional single particle tracking. Nature Communication. 8 (1), 887(2017).
- Xu, D., He, Y., Yeung, E. S. Y. Direct observation of the orientation dynamics of single protein-coated nanoparticles at liquid/solid interfaces. Angewandte Chemie. International Ed. in English. 53 (27), 6951-6955 (2014).
- Lehui, X., Yan, H., Edward, S. Y. Three Dimensional Orientational Imaging of Nanoparticles with Darkfield Microscopy. Analytical Chemistry. 82, 5268-5274 (2010).
- Ge, F., Xue, J., Wang, Z., Xiong, B., He, Y. Real-time observation of dynamic heterogeneity of gold nanorods on plasma membrane with darkfield microscopy. Science China Chemistry. 62, 1072-1081 (2019).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Janczura, J., Weron, A. Ergodicity testing for anomalous diffusion: small sample statistics. The Journal of Chemical Physics. 142 (14), 144103(2015).
- Kim, D. H., et al. Single particle tracking-based reaction progress kinetic analysis reveals a series of molecular mechanisms of cetuximab-induced EGFR processes in a single living cell. Chemical Science. 8 (7), 4823-4832 (2017).
- Kurzthaler, C., et al. Probing the Spatiotemporal Dynamics of Catalytic Janus Particles with Single-Particle Tracking and Differential Dynamic Microscopy. Physical Review Letters. 121 (7), 078001(2018).
- Lin, X., Pan, Q., He, Y. In situ detection of protein corona on single particle by rotational diffusivity. Nanoscale. 11 (39), 18367-18374 (2019).
- Wei, L., et al. Sub-diffraction-limit localization imaging of a plasmonic nanoparticle pair with wavelength-resolved dark-field microscopy. Nanoscale. 9 (25), 8747-8755 (2017).
- Cheng, X., Dai, D., Xu, D., He, Y., Yeung, E. S. Subdiffraction-limited plasmonic imaging with anisotropic metal nanoparticles. Analytical Chemistry. 86 (5), 2303-2307 (2014).
- Belyy, V., et al. PhotoGate microscopy to track single molecules in crowded environments. Nature Communication. 8, 13978(2017).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır