このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
単一ナノ粒子ダークフィールド顕微鏡を用いた細胞膜上の金ナノロッドの拡散ダイナミクスの可視化
要約
ここでは、従来の暗視野顕微鏡を用いて、細胞膜上の金ナノロッド(AuNRs)のダイナミクスを監視する方法を示す。単一の AuNR の位置と向きは ImageJ および MATLAB を使用して検出され、AuNRs の拡散状態は単一の粒子追跡解析によって特徴付けられる。
要約
細胞膜上のナノ粒子の拡散ダイナミクスを分析することは、細胞取り込みプロセスをよりよく理解する上で重要な役割を果たし、ナノメディシンデリバリーの合理的設計の理論的根拠となる。単一粒子追跡(SPT)解析は、細胞膜上の個々のナノ粒子の位置と向きを調査し、その並進状態と回転状態を明らかにすることができる。ここでは、従来の暗視野顕微鏡を用いて、生細胞膜上の金ナノロッド(AuNRs)のダイナミクスを監視する方法を示す。また、ImageJ と MATLAB を使用して AuNRs の位置と方向を抽出する方法と、AuNRs の拡散状態を特徴付ける方法についても説明します。数百個の粒子の統計解析は、単一のAuNRsがU87 MG細胞膜の表面でブラウン運動を行っていることを示しています。しかし、個々の長い軌道分析は、AuNRsが膜上の動き状態の2つの明確に異なるタイプ、すなわち長距離輸送および限られた領域の閉じ込めを有することを示す。当社のSPT法は、異なる生物学的細胞における表面または細胞内粒子拡散を研究するために使用される可能性があり、複雑な細胞機構の調査のための強力なツールとなり得ます。
概要
膜上のナノ粒子(NPs)のダイナミクスは、細胞の取り込みプロセスと密接に関連しており、細胞機能、ウイルスまたは細菌感染、および人工ナノ医療デリバリーシステム1,2の開発の理解に不可欠である。単一粒子追跡(SPT)技術は、NP3,4の異種挙動を特徴付けるための堅牢なツールです。一般に、細胞膜は流体であり、タンパク質や脂質などの成分が、形質膜面5、6、7内で横に移動できることを意味する。膜組織と構造の時空間的な複雑さは、NPと膜の相互作用の時空間的不均一性をもたらす可能性がある。したがって、膜上のNPの動きを直接可視化するには、高い空間解像度と時間的解像度の両方が必要です。
数十ナノメートルの空間分解能とミリ秒の時間分解能を有する生細胞における個々の粒子の局在を監視する単一粒子追跡顕微鏡は、NPまたは膜分子ダイナミクス8,9を研究するために十分に開発されている。蛍光ベースの顕微鏡イメージング技術は、生細胞環境9、10、11、12の中でNPs/分子を観察するための貴重なツールとなっています。例えば、全内部反射蛍光顕微鏡検査は、高い時空間分解能を有する基板/溶液界面で試料の薄層(〜100nm)を画像化する、膜分子ダイナミクス13,14の研究において広く用いられている。しかし、低強度や急速な不可逆的な光の漂白のような単一の蛍光ホルの固有の欠点は、トラッキング13の精度と持続時間を低下させる。したがって、蛍光プローブに代わる非蛍光プラズモニックNPは、そのユニークな光学特性15による長期イメージング研究においてますます注目を集めている。プラズモニックNPプローブの散乱信号に基づいて、暗視野顕微鏡(DFM)16、インターフェログメトリック散乱(iSCAT)顕微鏡法17、差動干渉差影顕微鏡(DICM)18などの生物学的プロセスのメカニズムを研究するために、数種類の光学顕微鏡イメージング技術が使用されている。さらに、DFM および DICM 18、19、 20、21、22を使用して、AuNRs のモーションと回転の動きが得られます。典型的には、SPT実験において、対象物の運動は光学顕微鏡によって記録され、次いでSPT分析方法3によって分析される。個々のNPによって生成される時間分解軌道と向きの角度は、通常、確率的で異種的であるため、さまざまな解析方法で豊富な動的情報を提示する必要があります。
ここでは、DFMを用いて細胞膜上のAuNRsのダイナミクスを監視し、ImageJおよびMATLABでAuNRsの位置と向きを抽出し、SPT分析法によるAuNRsの拡散を特徴付ける統合プロトコルを提供します。このデモンストレーションとして、U87 MG細胞膜上の非修飾AuNRs(CTAB-AuNRs、セチルトリメチルアンモニウムアンモニウム臭化アンモニウム分子によって合成された)のダイナミクスを可視化する方法を示す。CTAB-AuNRsは、生物学的環境でタンパク質を吸引し、細胞膜上を移動し、細胞2、20、22に入ることができることが実証されている。U87 MG細胞は中枢神経系の最も一般的で最も悪性の腫瘍であり、その膜受容体は異常に発現する。膜受容体は、AuNRsのタンパク質と相互作用し、AuNRsのダイナミクスに影響を与えます。我々のプロトコルは、一般に生物学の分野で他のSPT実験に適用されます。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. 細胞培養
- ウシ胎児血清(最終濃度10%)を添加してU87 MG細胞用の培地を調製ペニシリンストレプトマイシン(最終濃度1%)最小必須培地(MEM)に。細胞サブカルチャーにはプラスチック細胞培養皿を使用してください。
- パッセージ細胞は週に2〜3回である。
- 培地を取り出し、コンフルエント(80〜90%)の場合、Dulbeccoのリン酸緩衝生理食塩水(D-PBS)で細胞層を2〜3回リンスします。
- 細胞培養皿に1.0〜2.0mLのトリプシン-EDTA溶液を加え、細胞が丸くなるまで反転顕微鏡で細胞を観察します(3〜5分)。
- 調製した完全な培地の3.0 mLを加え、穏やかにピペット処理して細胞を分散させます。
- 新鮮な細胞培地(3 mL)で新しい培養皿に細胞懸濁液(1mL)を加え、細胞を再懸濁させる。
- 加湿雰囲気で37°C、5%CO2で細胞を維持します。
2. 顕微鏡のスライドの準備
注:高活性の3代から10代のU87 MG細胞はSPT実験で使用されています。
- 22mm×22mmのカバーリップをエタノールに浸してピラニア溶液で洗浄済み(99.9%)滅菌します。
- 鉗子を使用してエタノール溶液からカバースリップを取り出し(ステップ2.1)、炎上でエタノールを燃焼させることで滅菌します。すべてのエタノールが焼かれると、2 mLの細胞培地(フェノールレッドなし)で満たされたプラスチック細胞培養皿(35 x 10 mm)にカバースリップを入れます。
- ステップ1.2.3のセルサスペンションを50μLのカバースリップに加え、皿を前後左右に軽く押して細胞を均等に分配します。加湿した雰囲気の中に置きます。
- カバースリップ上のU87 MG細胞が20%~40%の合流率(〜12時間)に達したら、20 μLのCTAB-AuWR(138 pM)を皿に加えて分散させます。加湿雰囲気で5分間インキュベートします。
- 工程2.4の皿から、ピラニア溶液で予洗浄された溝ガラススライド(図1)の溝に、100μLの培養培地(フェノールレッドなし)を加えます。
- カバースリップを皿から取り出し、顕微鏡ガラススライドの溝の上に反転する(図1)。マニキュアで密封し、乾燥させてステージに置いてSPT実験を行います。
3. 暗視野顕微鏡による単一粒子追跡実験の実行 (図 1)。
- 油浸しダークフィールドコンデンサー(NA 1.43-1.20)に油を一滴置き、ノブを回してコンデンサーがガラススライドに接触させます。
- カバーガラスの上部にオイルを一滴入れ、焦点を合わせるノブを回して60倍の油浸漬目的(NA 0.7-1.25)が油に触れるようにします。
- 光源をオンにし、焦点を合わせるノブを少し回して、イメージング平面に焦点を合わせます。
注: 視野では、背景は黒、セルは明るく、CTAB-AuWR(縦横比〜2:1、 図 2)は小さな色(赤、黄、または緑)の散乱スポットです。 - カラー CMOS カメラでサンプル散乱光をキャプチャします。TIFF 形式を記録して書き出して画像を保存するには、ソフトウェアの [カメラ アイコン]をクリックします。
4. データ取得
- 単一の長期軌道を抽出する
- 図 3に示すように動作して、時系列の暗視野画像を"RGB カラー" モードから "8 ビット" モードに変換します。イメージ J で [イメージ] をクリック|タイプ|8ビット。コントラストを調整するには、[画像] ||を調整する明るさ|コントラスト.
- ターゲット パーティクルを選択し、"Ctrl+X"で背景をボックス化して削除することで、時系列の背景をカットします。
- 「プラグイン」をクリックして、パーティクル検出とパーティクルのリンクウィンドウを開く|パーティクル トラッカー クラシック |パーティクルトラッカー".
- [半径]を 6 に、カットオフを 0 に、パーセンタイルを 0.01% に設定します。
注: パーティクルを検出するには、プレビューの助けを借りて、3 つのパラメータの上に調整します。半径がターゲットパーティクルよりもわずかに大きく、最小のパーティクル間分離よりも小さいようにします。パーセンタイルは、候補粒子となる強度分布の下限です。 - [リンク範囲]を 5 に設定し、[変位]を 10 に設定します。
注: 連続する隣接フレーム間でパーティクルをリンクするには、上記の 2 つのパラメータを調整します。変位は、パーティクルが後続の 2 つのフレーム間を移動できる最大ピクセル数で、リンク範囲は、最適な対応する一致を決定する際に考慮する連続フレームの数です。 - [OK] をクリックして[パーティクルトラッカー結果]ウィンドウを開き、結果を確認します。
- 生成された軌道を検査するには、「すべての軌道を視覚化」をクリックします。
- ソフトウェアによって生成された軌道が AuNR の移動軌道と一致しない場合は、上部の [パーティクルを再リンク] メニューをクリックして、検出されたパーティクルを異なるリンク範囲とパーセンタイル パラメータで再リンクします。
- ソフトウェアが生成した軌道と AuNR の移動軌道が一致した場合に結果を保存するには、「完全レポートを保存」をクリックします。
注: イメージ J を使用して単一の長期軌道を抽出する例を 図 4に示します。
- マルチ軌道の抽出
注: フィジーでのマルチ軌道抽出の例を 図 5に示します。いくつかの段階があり、各ステージは追跡プロセスのステップを構成します。各ステップの結果は直ちに表示され、出力が不十分な場合に再び設定に戻ることができます。- 図 3に示すように動作して、時系列の暗視野画像を"RGB カラー" モードから "8 ビット" モードに変換します。イメージ J のパスウェイで「 イメージ |タイプ|8ビット"。コントラストを調整するには、「画像||を調整する明るさ|コントラスト」 .
- 「プラグイン」をクリックしてスタートパネルを 開| 追跡|トラックメイト".「次へ」をクリックします。
- ドロップダウン メニューから[LoG 検出器] を選択し、[次へ] をクリックします。
- 検出器構成パネルのパラメータを調整します。[推定ブロブ直径] を 10 に設定し、[しきい値] を 0 に設定し、[サブピクセルローカリゼーションを行う] を選択します。
- [次へ] をクリックして、初期スポット フィルタリング パネルを開きます。
注: いくつかのパラメータを調整して、目標点をさらに最適化することができます。この例では、他のパラメーターは調整されませんでした。 - [次へ] をクリックし、ドロップダウン メニューから [ハイパースタック ディスプレイ] を選択します。
- [次へ] をクリックしてスポット フィルタリング パネルを開きます。品質を1.88以上、Xを38.86以上、Yを56.54以上に設定します。
- [次へ] をクリックし、ドロップダウン メニューから [シンプル LAP トラッカー] を選択します。3 つのパラメータ、つまり[最大距離をリンク] = 15、ギャップを閉じる最大距離 = 15、ギャップクローズ最大フレーム ギャップ = 5 を調整して、シンプルな LAP トラッカーを設定します。
注: リンク最大距離は、2 つのフレーム間のポイントの最大変位です。ギャップを閉じる最大距離は、2 つのセグメントの最大変位です。ギャップを閉じる最大フレームギャップは、ブリッジされる 2 つのポイント間の最大フレームです。 - [次へ] をクリックして追跡します。終了したら、続けて [次へ] をクリックします。
- 300 を超えるトラックのスポット数を設定するなど、トラックにフィルターを設定します。
- 最後の保存パネルが開くまで、"Next"をクリックし続けます。ドロップダウン メニューから [トラックを XML ファイルにエクスポート] を選択し、[実行] をクリックして CSV 形式で保存します。
- R/G 値
注: R および G チャネルのターゲット AuNR の散乱強度は、MATLAB (https://github.com/fenggeqd/JOVE-2020/tree/master/RGandPolarangle) で記述されたコードを使用してカラー ダークフィールド イメージから取得され、抽出原理を 図 6に示します。- xy コーディネーションの関数を使用して.m X-y 座標 (ImageJ/Fiji によって抽出された) に従って各フレームの AuNR の中心ピクセル座標を見つけます。
- RGextract.m関数を使用して、3 x 3ピクセルの行列を区切り、RまたはGチャンネルの9つの散乱強度値を抽出し、平均値(μ、RまたはGとして定義)を計算します。
注: 3 x 3 ピクセルの行列は、ステップ 4.3.1 から取得したピクセル座標の中央に配置されます。
- 極角
注: 極角度は、AuNR の経度と光軸 ( 図 1に示すように) の間の角度で、AuNR の空間(Z 軸)回転ダイナミクスを反映できます。- 極角の関数を使用して.m二重チャネル差動法22によって極角角(θ)を計算
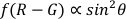 する。
する。
- 極角の関数を使用して.m二重チャネル差動法22によって極角角(θ)を計算
5. データ分析
注: SPT 分析手法のパフォーマンスと効率を実現するには、体系的で堅牢なデータ分析フレームワークが不可欠です。MATLAB で書かれたカスタム ソフトウェアが使用されます (https://github.com/fenggeqd/JOVE-2020/tree/master/Analysis_parameters)。グラフおよび解析ソフトウェア( 材料表を参照)は、プロットの描画に使用されます。
- 分析パラメーター
- csv_data_extract_dis_vel_ss.mとcsv_data_MSD.mのスクリプトを使用して、表 1に示す数式に従って動的パラメータを計算します。
注: これらのパラメータは、AuNRsのダイナミクスを分析するために使用され、3つの部分で構成されています。(1) 軌道関連パラメータ:変位、歩数サイズ、速度、旋回半径(Rg)、旋回角(Ta);(2) MSD パラメータ: 拡散係数 (Dt) および異常拡散指数 (α);そして(3)回転関連のパラメータ:極角および回転のlability(σ)。
- csv_data_extract_dis_vel_ss.mとcsv_data_MSD.mのスクリプトを使用して、表 1に示す数式に従って動的パラメータを計算します。
| 対策 | 定義 | 物理的な意味 | |
| 変位 | 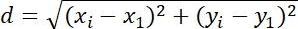 | オブジェクトの位置の変更 | |
| ステップサイズ | 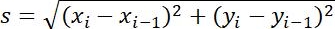 | 2 つの隣接する点間の距離 | |
| 速度 |  | オブジェクトの動きの速度 | |
| Rg | 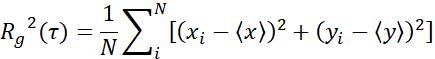 | 特定の時間間隔でのオブジェクトの移動範囲 | |
| Ta | 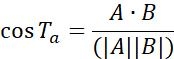 | 2 つの隣接する点間のオブジェクトのモーション方向 | |
| Msd | 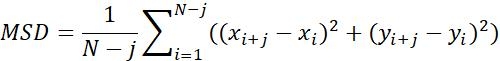 | 特定の時間間隔におけるオブジェクトの平均移動距離 | |
| Dt | 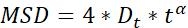 | オブジェクトの拡散能力 | |
| α | 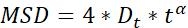 | 正常拡散(α〜1) | |
| 極角 |  | オブジェクトの 3D 方向情報 | |
| Σ |  | 極角データセットの分散度 | |
表1:分析に使用する3種類のパラメータこれには、軌道関連パラメータ(変位、歩幅、速度、RgおよびT a)、MSDパラメータ(MSD、Dtおよびα)、回転関連パラメータ(極角および回転可分性)が含まれます。
- 軌跡の視覚解析
注: 軌道ビジュアライゼーションは、時間マッピング軌道、Rg-およびDt-マッピング軌道、極角マッピング軌道などの動的パラメータの、粒子運動の時空間的不均一性と軌道(座標)分布を直感的に提示することができます。マッピング軌跡は、グラフ作成および解析ソフトウェアを用いて描かれた。- X 座標を X、y 座標を Y、時間 (Rg、Dt、極角度) を Z として設定します。
- [プロット|] をクリックします。散布図|カラーマッピングプロット".
- カラーバーを追加します。
- MSD 分析
注: パーティクルのモーション アクティビティとモーション モードは、MSD 解析23で取得できます。Dtが大きいほど、パーティクルの拡散運動が活発になります。α〜1の場合、粒子は正常な拡散運動を行い、そうでなければ異常な拡散運動を行う。- MSD-τフィギュア
- 時間間隔 (τ) を X、MSD データを Y として設定します。
- [プロット|] をクリックします。散布図"
- [分析] をクリックしてデータを調整|フィッティング|非線形曲線フィッティング(関数:同値)」。
- MSD-τ二重対数図
注: MSD-τ 二対数の図は、その傾きがαで、切片が Dtで、直感的にパーティクルの動きを表すことができます。- 対数時間間隔を X、対数 MSD を Y として設定します。
- [プロット|] をクリックします。散布図".
- [分析] をクリックしてデータを調整|フィッティング|線形曲線フィッティング"。
- D-τフィギュア(MSD/4t)
注: D=MSD/4τ は時間 τ と異常因子α関数で、D-τ は時間とともに拡散係数の変化を直接示します。時間とともにDが増加すると、αは1より大きくなり、粒子はスーパーディフュージョン運動を行います。- 対数時間間隔 (τ) を X、対数 D を Y に設定します。
- [プロット|] をクリックします。散布図".
- [分析] をクリックしてデータを調整|フィッティング|非線形曲線フィッティング(関数: "アロメトリックル")"。
注: 軌道が短いほど、拡散推定の不正確さが高くなります。一般に、長い間隔時間(>30 τ)のMSD-τ分析を通してDt およびαが得られた。ただし、シンプルでラフなフィッティングは、モーションの詳細を滑らかにします。したがって、短時間で粒子の動きの挙動を解析するために、短い間隔時間(<10 τ)のMSD-τ分析を行う必要があります。
- MSD-τフィギュア
- 統計分析
- マルチ粒子統計解析
注: マルチパーティクル統計解析では、空間領域内のパーティクルのモーション状態を反映することができ、空間的不均一性環境を間接的に示します。例えば、Dt のヒストグラムが大規模分布またはマルチピーク分布を示す場合、粒子の運動活動が不均一であることを意味する。- 動的パラメータ(Dt、Rg、最大変位など)を Y に設定します。
- [プロット|] をクリックします。ヒストグラム".
- ヒストグラムをダブルクリックし、分割サイズまたは分割数を設定します。「適用」をクリックします。
- 単粒子統計解析
注: 単一粒子の統計解析では、個々のパーティクルの動きの挙動を示すことができます。- 複数のフレーム
- 1 つの長い軌道のすべてのフレームの動的パラメータ(Ta、ステップ サイズ、極角度など)を計算し、[原点]テーブルにコピーして Y に設定します。
- [プロット|] をクリックします。ヒストグラム".
- ヒストグラムをダブルクリックし、分割サイズまたは分割数を設定します。「適用」をクリックします。
注: Ta とステップ サイズの両方が小さな値分布を示す場合、パーティクルは小さいステップの超拡散モーションを行います。
- ウィンドウの移動
- 移動窓法(11フレーム)を通じて単一長軌道のすべてのフレームの動的パラメータ(Rg、Dtなど)を計算し、原点テーブルにコピーしてYに設定します。
- [プロット|] をクリックします。ヒストグラム".
- ヒストグラムをダブルクリックし、分割サイズまたは分割数を設定します。「適用」をクリックします。
- 複数のフレーム
- マルチ粒子統計解析
- 時系列分析
注: 統計解析では、NP のモーション状態を明らかにすることができ、時系列解析は、モーション動作を補足として示すことができます。複数の時系列パラメータを組み合わせることで、時間レベルと空間レベルでの AP のモーション動作を判別できます。- 時間を X、時系列パラメータを Y (変位、極角度、Rgなど) として設定します。
- [プロット|] をクリックします。マルチペイン ダイアグラム |積み上げプロット|ライン + シンボル".
Access restricted. Please log in or start a trial to view this content.
結果
プロトコルでは、変更されていない 40 x 85 nm CTAB-AuNRs が使用されました。図2Bに示すように、その縦断プラズモニック最大値は〜650nm(赤色領域)であり、横共鳴は520nm(緑色領域)である。以前の文献では、プラズモニックオーナーの光学特性(LSPR強度など)は、直径20,22で大きく変化することが明らかとなった。図2C<...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
提示されたプロトコルは細胞膜のAuNRsの動態を研究するために使用される。このプロトコルは、顕微鏡イメージング、データ抽出、動的パラメータ計算、データ分析方法など4つの部分で構成され、各部分は柔軟で普遍的です。したがって、膜上のNP結合膜分子の動き、NP標識受容体のエンドサイトーシスダイナミクス、微小管に沿った細胞内NPおよび小胞被覆NPの動的分析など、将来の応用が?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者らは開示するものは何もない。
謝辞
この研究は、中国国立自然科学財団の助成番号21425519、91853105、21621003によって支援されました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| CTAB coated gold nanorods(CTAB-AuNRs) | Nanoseedz | NR-40-650 | 85 nm * 40 nm |
| Color CMOS camera | Olympus | DP74 | Japan |
| Coverslips | Citoglas | z10212222C | 22*22 mm |
| Dark-field microscopy | Nikon | 80i | upright microscope |
| Fetal bovine serum (FBS) | Gibco | 10099141 | |
| Fiji | National Institutes of Health | 2.0.0-rc-69/1.52 p | a distribution of ImageJ |
| Grooved glass slide | Sail brand | 7103 | Single concave |
| Image J | National Institutes of Health | 1.52 j | |
| MATLAB | MathWorks | R2019b | |
| MATLAB Code | https://github.com/fenggeqd/JOVE-2020 | ||
| Minimum essential medium (MEM) | Gibco | 10-010-CVR | with phenol red |
| Minimum essential medium (MEM) | Gibco | 51200038 | no phenol red |
| Origin | OriginLab | Origin Pro 2018C | |
| Penicillin-streptomycin | Gibco | 15140122 | |
| Plastic cell culture dishes | Falcon | 353002 | |
| Plastic cell culture dishes | Falcon | 353001 | 35*10 mm |
| U87 MG cell | American Type Culture Collection | ATCC HTB-14 | a human primary glioblastoma cell line |
参考文献
- Rees, P., Wills, J. W., Brown, M. R., Barnes, C. M., Summers, H. D. The origin of heterogeneous nanoparticle uptake by cells. Nature Communication. 10 (1), 2341(2019).
- Behzadi, S., et al. Cellular uptake of nanoparticles: journey inside the cell. Chemical Society Reviews. 46 (14), 4218-4244 (2017).
- Shen, H., et al. Single Particle Tracking: From Theory to Biophysical Applications. Chemical Reviews. 117 (11), 7331-7376 (2017).
- Saxton, M. J. Single-particle tracking: connecting the dots. Nature Methods. 5 (8), 671-672 (2008).
- Kusumi, A., et al. Dynamic organizing principles of the plasma membrane that regulate signal transduction: commemorating the fortieth anniversary of Singer and Nicolson's fluid-mosaic model. Annual Review of Cell and Developmental Biology. 28, 215-250 (2012).
- Jacobson, K., Liu, P., Lagerholm, B. C. The Lateral Organization and Mobility of Plasma Membrane Components. Cell. 177 (4), 806-819 (2019).
- Sezgin, E., Levental, I., Mayor, S., Eggeling, C. The mystery of membrane organization: composition, regulation and roles of lipid rafts. Nature Reviews Molecular Cell Biology. 18 (6), 361-374 (2017).
- von Diezmann, A., Shechtman, Y., Moerner, W. E. Three-Dimensional Localization of Single Molecules for Super-Resolution Imaging and Single-Particle Tracking. Chemical Reviews. 117 (11), 7244-7275 (2017).
- Rosenberg, J., Huang, J. Visualizing Surface T-Cell Receptor Dynamics Four-Dimensionally Using Lattice Light-Sheet Microscopy. Journal of Visualized Experiments. (155), e59914(2020).
- Kusumi, A., Tsunoyama, T. A., Hirosawa, K. M., Kasai, R. S., Fujiwara, T. K. Tracking single molecules at work in living cells. Nature Chemical Biology. 10 (7), 524-532 (2014).
- Rocha, J. M., Gahlmann, A. Single-Molecule Tracking Microscopy - A Tool for Determining the Diffusive States of Cytosolic Molecules. Journal of Visualized Experiments. (151), e59387(2019).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing protein-DNA interactions in live bacterial cells using photoactivated single-molecule tracking. Journal of Visualized Experiments. (85), e511177(2014).
- Pinaud, F., Clarke, S., Sittner, A., Dahan, M. Probing cellular events, one quantum dot at a time. Nature Methods. 7 (4), 275-285 (2010).
- Mehidi, A., et al. Transient Activations of Rac1 at the Lamellipodium Tip Trigger Membrane Protrusion. Current Biology. 29 (17), 2852-2866 (2019).
- Ye, Z., Wang, X., Xiao, L. Single-Particle Tracking with Scattering-Based Optical Microscopy. Analytical Chemistry. 91 (24), 15327-15334 (2019).
- Pan, Q., Zhao, H., Lin, X., He, Y. Spatiotemporal Heterogeneity of Reactions in Solution Observed with High-Speed Single-Nanorod Rotational Sensing. Angewandte Chemie. International Ed. in English. 58 (25), 8389-8393 (2019).
- Taylor, R. W., et al. Interferometric scattering microscopy reveals microsecond nanoscopic protein motion on a live cell membrane. Nature Photonics. 13 (7), 480-487 (2019).
- Chen, K., et al. Characteristic rotational behaviors of rod-shaped cargo revealed by automated five-dimensional single particle tracking. Nature Communication. 8 (1), 887(2017).
- Xu, D., He, Y., Yeung, E. S. Y. Direct observation of the orientation dynamics of single protein-coated nanoparticles at liquid/solid interfaces. Angewandte Chemie. International Ed. in English. 53 (27), 6951-6955 (2014).
- Lehui, X., Yan, H., Edward, S. Y. Three Dimensional Orientational Imaging of Nanoparticles with Darkfield Microscopy. Analytical Chemistry. 82, 5268-5274 (2010).
- Ge, F., Xue, J., Wang, Z., Xiong, B., He, Y. Real-time observation of dynamic heterogeneity of gold nanorods on plasma membrane with darkfield microscopy. Science China Chemistry. 62, 1072-1081 (2019).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Janczura, J., Weron, A. Ergodicity testing for anomalous diffusion: small sample statistics. The Journal of Chemical Physics. 142 (14), 144103(2015).
- Kim, D. H., et al. Single particle tracking-based reaction progress kinetic analysis reveals a series of molecular mechanisms of cetuximab-induced EGFR processes in a single living cell. Chemical Science. 8 (7), 4823-4832 (2017).
- Kurzthaler, C., et al. Probing the Spatiotemporal Dynamics of Catalytic Janus Particles with Single-Particle Tracking and Differential Dynamic Microscopy. Physical Review Letters. 121 (7), 078001(2018).
- Lin, X., Pan, Q., He, Y. In situ detection of protein corona on single particle by rotational diffusivity. Nanoscale. 11 (39), 18367-18374 (2019).
- Wei, L., et al. Sub-diffraction-limit localization imaging of a plasmonic nanoparticle pair with wavelength-resolved dark-field microscopy. Nanoscale. 9 (25), 8747-8755 (2017).
- Cheng, X., Dai, D., Xu, D., He, Y., Yeung, E. S. Subdiffraction-limited plasmonic imaging with anisotropic metal nanoparticles. Analytical Chemistry. 86 (5), 2303-2307 (2014).
- Belyy, V., et al. PhotoGate microscopy to track single molecules in crowded environments. Nature Communication. 8, 13978(2017).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved