É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Visualizando dinâmica difusional de nanorods de ouro na membrana celular usando microscopia de campo escuro de nanopartícula única
Neste Artigo
Resumo
Aqui, mostramos o uso da microscopia tradicional de campo escuro para monitorar a dinâmica dos nanorods de ouro (AuNRs) na membrana celular. A localização e orientação de AuNRs únicos são detectadas usando ImageJ e MATLAB, e os estados difusivos de AuNRs são caracterizados por uma análise de rastreamento de partículas únicas.
Resumo
Analisar a dinâmica difusiva das nanopartículas na membrana celular desempenha um papel significativo na melhor compreensão do processo de absorção celular e fornece uma base teórica para o desenho racional da entrega de nanoma medicamentos. A análise de rastreamento de partículas únicas (SPT) poderia sondar a posição e orientação de nanopartículas individuais na membrana celular, e revelar seus estados translacionais e rotacionais. Aqui, mostramos como usar a microscopia tradicional de campo escuro para monitorar a dinâmica dos nanorods de ouro (AuNRs) na membrana celular viva. Também mostramos como extrair a localização e orientação dos AuNRs utilizando ImageJ e MATLAB, e como caracterizar os estados difusivos de AuNRs. A análise estatística de centenas de partículas mostra que um único AuNRs realiza movimento browniano na superfície da membrana celular U87 MG. No entanto, a análise individual de longa trajetória mostra que os AuNRs têm dois tipos distintamente diferentes de estados de movimento na membrana, ou seja, transporte de longo alcance e confinamento de áreas limitadas. Nossos métodos SPT podem ser potencialmente usados para estudar a difusão de partículas superficiais ou intracelulares em diferentes células biológicas e podem se tornar uma ferramenta poderosa para investigações de mecanismos celulares complexos.
Introdução
A dinâmica das nanopartículas (NPs) na membrana está intimamente associada ao processo de captação celular, essencial para a compreensão das funções celulares, infecções virais ou bacterianas e o desenvolvimento de sistemas de entrega nanomédicas artificiais1,2. A técnica de rastreamento de partículas únicas (SPT) é uma ferramenta robusta para caracterizar os comportamentos heterogêneos das NPs3,4. Em geral, a membrana celular é fluidica, o que significa que os componentes como proteínas e lipídios podem se mover lateralmente no plano da membrana plasmática5,6,7. A complexidade espiotemporal da organização e estrutura da membrana pode levar à heterogeneidade espiotemporal da interação entre NPs e membrana. Assim, a visualização direta do movimento de NPs na membrana requer alta resolução espacial e temporal.
Microscopia de rastreamento de partículas únicas que monitora a localização de partículas individuais em células vivas com uma resolução espacial de dezenas de nanômetros e uma resolução temporal de milissegundos foi bem desenvolvida para estudar as NPs ou moléculas de membrana dinâmica8,9. Técnicas de imagem microscópica baseadas em fluorescência tornaram-se ferramentas valiosas para observar NPs/moléculas no ambiente celular vivo9,10,11,12. Por exemplo, a microscopia total de fluorescência de reflexão interna, que imagens de camadas finas (~100 nm) da amostra na interface substrato/solução com alta resolução espátula tem sido amplamente utilizada em estudos da dinâmica das moléculas de membrana13,14. No entanto, as desvantagens inerentes aos fluoroforos únicos, como baixa intensidade e fotobleaching irreversível rápido reduzem a precisão e a duração do rastreamento13. Portanto, NPs plasmônicos não fluorescentes, que substituem as sondas fluorescentes, têm atraído cada vez mais atenção em estudos de imagem de longo prazo devido às suas características ópticas únicas15. Com base nos sinais de dispersão de sondas NP plasmônicas, vários tipos de tecnologias ópticas de imagem microscópica têm sido utilizadas para estudar o mecanismo de processos biológicos, como microscopia de campo escuro (DFM)16,microscopia de dispersão interferométrica (iSCAT)17 e microscopia de contraste de interferência diferencial (DICM)18. Além disso, a dinâmica de movimento e rotação de AuNRs pode ser obtida utilizando DFM e DICM18,19,20,21,22. Normalmente, em um experimento SPT, o movimento do objeto é registrado pelo microscópio óptico e, em seguida, analisado pelos métodos de análise SPT3. As trajetórias e ângulos orientacionais gerados por NPs individuais são normalmente estocásticos e heterogêneos, por isso é necessário apresentar informações dinâmicas abundantes com vários métodos de análise.
Aqui, fornecemos um protocolo integrado que monitora a dinâmica dos AuNRs na membrana celular utilizando DFM, extrai a localização e orientação dos AuNRs com ImageJ e MATLAB e caracteriza a difusão de AuNRs com métodos de análise SPT. Como demonstração, mostramos aqui como usar o protocolo SPT para visualizar dinâmicas de AuNRs não modificados (CTAB-AuNRs, sintetizados pela molécula de brometo de amônio cetililomina como agente protetor) na membrana celular U87 MG. Foi demonstrado que os CTAB-AuNRs podem adsorb proteínas em ambiente biológico, mover-se na membrana celular e, emseguida,entrar nas células2,20,22. A célula U87 MG é o tumor mais comum e mais maligno do sistema nervoso central, e seus receptores de membrana são anormalmente expressos. Os receptores de membrana podem interagir com proteínas em AuNRs, que influenciam a dinâmica dos AuNRs. Nosso protocolo é geralmente aplicável a outros experimentos SPT no campo da biologia.
Protocolo
1. Cultura celular
- Prepare o meio completo para células U87 MG adicionando soro bovino fetal (concentração final 10%) e penicilina-estreptomicina (concentração final 1%) ao meio mínimo essencial (MEM). Use prato de cultura de células plásticas para subcultura celular.
- Células de passagem de 2 a 3 vezes por semana.
- Remova o meio de cultura e enxágue a camada celular com o soro fisco tamponado de Fosfato de Dulbecco (D-PBS) 2~3 vezes quando confluente (80%~90%).
- Adicione 1.0 a 2,0 mL de solução Trypsin-EDTA ao prato de cultura celular e observe as células sob um microscópio invertido até que as células se tornem redondas (3~5 min).
- Adicione 3,0 mL de meio completo preparado e disperse as células por pipetação suave.
- Adicione suspensão celular (1 mL) ao novo prato de cultura com meio de célula fresca (3 mL) e resuspense as células.
- Mantenha as células a 37 °C e 5% de CO2 em uma atmosfera umidificada.
2. Preparação de slides de microscópio
NOTA: Células U87 MG de terceira a décima geração com alta atividade são usadas em experimentos SPT.
- Esterilizar 22 mm × tampas de 22 mm já limpas com solução Piranha, imersão em etanol (99,9%).
- Use fórceps para retirar o deslizamento de cobertura da solução de etanol (etapa 2.1) e esterilizar queimando etanol na chama. Uma vez que todo o etanol é queimado, coloque tampas em um prato de cultura de células plásticas (35 x 10 mm) preenchido com 2 mL de meio celular (sem vermelho fenol).
- Adicione 50 μL da suspensão da célula a partir da etapa 1.2.3 na tampa e empurre suavemente o prato para frente e para trás e para a esquerda e para a direita para distribuir uniformemente as células. Coloque-o em uma atmosfera umidificada.
- Quando as células U87 MG no deslizamento de cobertura atingirem 20%-40% de confluência (~12 h), adicione 20 μL de CTAB-AuNRs (138 pM) no prato e disperse. Incubar em atmosfera umidificada por 5 minutos.
- Adicione 100 μL do meio de cultura (sem fenol vermelho) do prato na etapa 2.4 no sulco do escorregador de vidro ranhurado(Figura 1)que é pré-recolhido com solução Piranha.
- Retire a tampa do prato e invertida na parte superior da ranhura do deslizamento de vidro do microscópio(Figura 1). Sele com esmalte, deixe secar e coloque-o no palco para realizar experimentos SPT.
3. Realizando experimentos de rastreamento de partículas únicas com microscopia de campo escuro(Figura 1).
- Coloque uma gota de óleo no condensador de campo escuro imerso em óleo (NA 1.43-1.20) e gire o botão para fazer com que o condensador entre em contato com o deslizamento de vidro.
- Coloque uma gota de óleo na parte superior do vidro de cobertura e gire o botão de foco para fazer o objetivo de imersão de óleo de 60x (NA 0,7-1,25) tocar o óleo.
- Acenda a fonte de luz e gire ligeiramente o botão de foco para focar o plano de imagem.
NOTA: No campo de visão, o fundo é preto, as células são brilhantes, e os CTAB-AuNRs (proporção ~2:1, Figura 2) são pequenos pontos de dispersão coloridos (vermelho, amarelo ou verde). - Capture a luz de dispersão de amostras por uma câmera CMOS colorida. Clique em "Ícone de câmera" no software para gravar e exportar o formato TIFF para salvar imagens.
4. Aquisição de dados
- Extrair trajetória única a longo prazo
- Opere conforme descrito na Figura 3 para converter as imagens de campo escuro da série temporal do modo "cor RGB" para o modo "8 bits". Na imagem J clique em Imagem | Tipo | 8 bits. Para ajustar o contraste, clique em Imagem | Ajuste | | de brilho Contraste.
- Selecione uma partícula de destino e corte os fundos da série de tempo, boxeando e excluindo o fundo com "Ctrl+X".
- Abra a janela de detecção de partículas e de linkículas clicando nos "Plugins | | Clássico do Rastreador de Partículas Rastreador de partículas".
- Definir raio para 6, cortar para 0 e Percentile para 0,01%.
NOTA: Para detectar a partícula, ajuste acima de três parâmetros com o auxílio da Visualização. Certifique-se de que o raio é ligeiramente maior que a partícula alvo e menor do que a menor separação entre partículas. Percentil é o limite mais baixo de distribuição de intensidade que para ser partículas candidatas. - Defina a faixa de link para 5 e deslocamento para 10.
NOTA: Para ligar a partícula entre quadros adjacentes consecutivos, ajuste os dois parâmetros acima. Deslocamento é o máximo de pixels que uma partícula pode mover entre dois quadros sucessores, e Link Range é o número de quadros consecutivos a considerar ao determinar a melhor correspondência correspondente. - Clique em "OK" para abrir a janela Resultados do ParticleTracker para ver os resultados.
- Clique em "Visualize Todas as Trajetórias" para inspecionar as trajetórias geradas.
- Clique em "Relink Particles" no topo para reconectar as partículas detectadas com diferentes intervalos de link e parâmetros percentil, se a trajetória gerada pelo software não corresponder à trajetória móvel do AuNR.
- Clique em "Salvar relatório completo" para salvar resultados se a trajetória gerada pelo software e a trajetória móvel do AuNR forem combinadas.
NOTA: Um exemplo de extração de uma trajetória única de longo prazo com a Imagem J é mostrado na Figura 4.
- Extração de multi-trajetórias
NOTA: Um exemplo de extração de multi-trajetórias com Fiji é mostrado na Figura 5. Existem várias etapas, e cada etapa constitui um passo no processo de rastreamento. O resultado de cada etapa é exibido imediatamente, o que permite ao usuário voltar às configurações de reajuste quando a saída é insatisfatória.- Opere conforme descrito na Figura 3 para converter as imagens de campo escuro da série temporal do modo "cor RGB" para o modo "8 bits". Na via imagem J clique em "Imagem | Tipo | 8 bits". Para ajustar seu contraste, clique em "Imagem | Ajuste | | de brilho Contraste".
- Abra o painel de partida clicando em "Plugins | Rastreando | TrackMate". Clique em "Next".
- Escolha o "detector log" no menu suspenso e clique em "Next".
- Ajuste os parâmetros do painel de configuração do detector. Defina o diâmetro estimado da bolha para 10, defina Limiar para 0 e Selecione "Do Sub-pixel Localização".
- Clique em "Next" para abrir o painel de filtragem do ponto inicial.
NOTA: Vários parâmetros podem ser ajustados para otimizar ainda mais os pontos-alvo. Neste exemplo, nenhum outro parâmetro foi ajustado. - Clique em "Next" e selecione "HyperStack displayer" no menu suspenso.
- Clique em "Next" para abrir o painel de filtragem Spot. Definir a Qualidade acima de 1,88, X acima de 38,86, Y acima de 56,54.
- Clique em "Next" e selecione "Simple LAP tracker" no menu suspenso. Configure o rastreador LAP simples ajustando três parâmetros, ou seja, a distância máxima de Linking = 15, distância máxima de fechamento de lacuna = 15 e gap-closing max frame gap = 5.
NOTA: A distância de ligação-máxima é o deslocamento máximo de um ponto entre dois quadros. A distância máxima de fechamento de lacunas é o deslocamento máximo de dois segmentos. Gap-closing max frame gap gap é o maior quadro entre dois pontos a serem superados. - Clique em "Next" para acompanhar. Quando terminar, continue a clicar em "Next".
- Defina filtros nas faixas, como definir Número de pontos na faixa acima de 300.
- Continue clicando em "Next" até que o painel de salvamento final seja aberto. Selecione "Exportar faixas para arquivo XML" no menu suspenso e, em seguida, clique em "Executar" para salvar no formato csv.
- Valores de R/G
NOTA: As intensidades de dispersão de AuNRs direcionados nos canais R e G são obtidas a partir de imagens de campo escuro coloridas usando um código escrito no MATLAB (https://github.com/fenggeqd/JOVE-2020/tree/master/RGandPolarangle), e o princípio de extração é apresentado na Figura 6.- Use a função de xicoordinação.m para encontrar a coordenada de pixel central do AuNR em cada quadro de acordo com a coordenada x-y (extraída por ImageJ/Fiji).
- Use a função de RGextraction.m para delimitar uma matriz de 3 x 3 pixels, extrair os 9 valores de intensidade de dispersão dos canais R ou G e calcular um valor médio (μ, definido como R ou G).
NOTA: A matriz de 3 x 3 pixels está centrada nas coordenadas de pixels obtidas a partir da etapa 4.3.1.
- Ângulo polar
NOTA: O ângulo polar é o ângulo entre a longitude do AuNR e o eixo óptico (como mostrado na Figura 1), que pode refletir a dinâmica de rotação espacial (eixo Z) do AuNR.- Use a função de polarangle.m para calcular os ângulos polares (φ) pelo método diferencial de dois canais22,
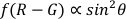 .
.
- Use a função de polarangle.m para calcular os ângulos polares (φ) pelo método diferencial de dois canais22,
5. Análise de dados
NOTA: Uma estrutura sistemática e robusta de análise de dados é essencial para o desempenho e eficiência dos métodos de análise SPT. O software personalizado escrito no MATLAB é usado (https://github.com/fenggeqd/JOVE-2020/tree/master/Analysis_parameters). Um software de grafia e análise (ver Tabela de Materiais) é usado para desenhar as parcelas.
- Parâmetros de análise
- Use scripts de csv_data_extract_dis_vel_ss.m e csv_data_MSD.m para calcular parâmetros dinâmicos de acordo com fórmulas mostradas na Tabela 1.
NOTA: Estes parâmetros são utilizados para analisar a dinâmica dos AuNRs e consistem em três partes. (1) Parâmetros relacionados à trajetória: deslocamento, tamanho do passo, velocidade, raio de giro (Rg),e ângulo de giro (Ta); (2) Parâmetros de MSD: coeficiente de difusão (Dt) e expoente de difusão anormal (α); e (3) Parâmetros relacionados à rotação: ângulo polar e capacidade rotacional (σ).
- Use scripts de csv_data_extract_dis_vel_ss.m e csv_data_MSD.m para calcular parâmetros dinâmicos de acordo com fórmulas mostradas na Tabela 1.
| Medidas | Definição | Significado físico | |
| Deslocamento | 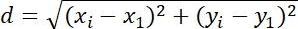 | Mudanças na posição dos objetos | |
| Tamanho do passo | 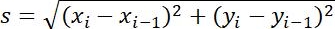 | Distância entre dois pontos adjacentes | |
| Velocidade |  | Velocidade do movimento dos objetos | |
| Rg | 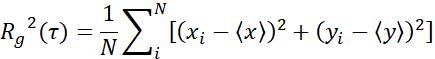 | Alcance de movimento de objetos em um intervalo de tempo específico | |
| Ta | 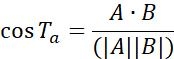 | Direção de movimento de objetos entre dois pontos adjacentes | |
| Msd | 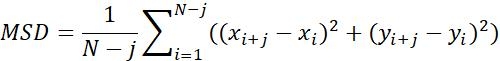 | Distância móvel média de objetos em um intervalo de tempo específico | |
| Dt | 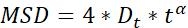 | Capacidade de difusão de objetos | |
| Α | 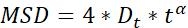 | Difusão normal (α~1) | |
| Ângulo polar |  | Informações de orientação 3D de objetos | |
| Σ |  | Grau de dispersão do conjunto de dados do ângulo polar | |
Tabela 1: Três tipos de parâmetros utilizados para análise. Estes incluem parâmetros relacionados à trajetória (deslocamento, tamanho do passo, velocidade, Rg e Ta),parâmetros MSD (MSD, Dt e α) e parâmetros relacionados à rotação (ângulo polar e capacidade rotacional).
- Análise visual da trajetória
NOTA: A visualização da trajetória pode apresentar intuitivamente a heterogeneidade espiotemporal do movimento das partículas e a distribuição de trajetória (coordenada) de parâmetros dinâmicos, como trajetória de mapeamento do tempo, trajetória de mapeamento de Rg- e Dte trajetória de mapeamento de ângulos polares. As trajetórias de mapeamento foram desenhadas utilizando-se o software de grafação e análise.- Definir x coordenada como X, y coordenar como Y, e tempo (Rg, Dt, ângulo polar) como Z.
- Clique em "Plot | | de parcela de dispersão Gráfico de mapeamento de cores".
- Adicione a barra de cor.
- Análise msd
NOTA: A atividade de movimento e o modo de movimento das partículas podem ser obtidos pela análise MSD23. Quanto maior o Dt,mais ativo é o movimento de difusão das partículas. quando α~1, as partículas fazem movimento de difusão normal, caso contrário, realizam movimento de difusão anormal.- Números do MSD-τ
- Defina o intervalo de tempo (τ) como X, dados MSD como Y.
- Clique em "Plot | Gráfico de dispersão"
- Ajuste os dados clicando em "Analisar | Montagem | Encaixe da curva não linear (Função: Aométrico)".
- MSD-τ figura logarítmica dupla
NOTA: A figura trâmica dupla-logarítmica msd-τ, cuja inclinação é α e interceptação é Dt, pode apresentar intuitivamente movimento de partículas.- Defina o intervalo de tempo logarítmico como X e o MSD logarítmico como Y.
- Clique em "Plot | Dispersar enredo".
- Ajuste os dados clicando em "Analisar | Montagem | Ajuste de curva linear".
- Figura D-τ (MSD/4t)
NOTA: D=MSD/4τ, é uma função do fator de tempo e fator de anomalia α, e a figura D-τ mostra diretamente a alteração do coeficiente de difusão com o tempo. Quando D aumenta com o tempo, α é maior que 1 e as partículas fazem movimento de superdiffusão.- Defina intervalo de tempo logarítmico (τ) como X e logarítmico D como Y.
- Clique em "Plot | Dispersar enredo".
- Ajuste os dados clicando em "Analisar | Montagem | Encaixe de curva não linear (Função: "Allometricl")".
NOTA: Quanto menor as trajetórias, maior a imprecisão das estimativas de difusão. Em geral, foram obtidas dt e α através da análise MSD-τ de longo intervalo (> 30 τ). No entanto, o ajuste simples e áspero irá suavizar os detalhes de movimento. Assim, deve ser realizada a análise do MSD-τ de curto tempo de intervalo (< 10 τ) para analisar o comportamento de movimento das partículas em pouco tempo.
- Números do MSD-τ
- Análise estatística
- Análise estatística de partículas multipartidárias
NOTA: A análise estatística de partículas multipartidárias pode refletir o estado de movimento das partículas em uma região espacial, o que indica indiretamente o ambiente de heterogeneidade espacial. Por exemplo, se o histograma de Dt exibe uma distribuição em larga escala ou distribuição de vários picos, significa que as atividades de movimento das partículas são heterogêneas.- Definir parâmetros dinâmicos (como Dt, Rg, deslocamento máximo) como Y.
- Clique em "Plot | Histograma".
- Clique duas vezes no histograma e defina o tamanho da divisão ou o número de divisões. Clique em "Aplicar".
- Análise estatística de partículas únicas
NOTA: A análise estatística de partículas únicas pode mostrar o comportamento de movimento de partículas individuais, o que também reflete indiretamente a heterogeneidade espiotemporal do ambiente circundante.- Múltiplos quadros
- Calcule parâmetros dinâmicos (como Ta, tamanho do passo, ângulo polar) de todos os quadros de trajetória longa única, e copie para a tabela Origin e definido como Y.
- Clique em "Plot | Histograma".
- Clique duas vezes no histograma e defina o tamanho da divisão ou o número de divisões. Clique em "Aplicar".
NOTA: Se o tamanhode um e o tamanho do passo apresentarem uma distribuição de pequeno valor, as partículas fazem um movimento de super-difusão em pequeno passo.
- Janelas móveis
- Calcule parâmetros dinâmicos (como Rg, Dt)de todos os quadros de trajetória longa única através do método de janela móvel (11 quadros), e copie para a tabela Origin e definido como Y.
- Clique em "Plot | Histograma".
- Clique duas vezes no histograma e defina o tamanho da divisão ou o número de divisões. Clique em "Aplicar".
- Múltiplos quadros
- Análise estatística de partículas multipartidárias
- Análise de séries temporentas
NOTA: A análise estatística pode revelar o estado de movimento dos NPs, e a análise de séries temporânicos pode apresentar o comportamento de movimento como um suplemento. Combinando vários parâmetros de séries temporais, pode discriminar o comportamento de movimento dos NPs nos níveis temporal e espacial.- Definir o tempo como X, parâmetros de séries temporais como Y (como deslocamento, ângulo polar e Rg).
- Clique em "Plot | Diagrama multi-painel | | de parcelas empilhadas Linha + Símbolo".
Resultados
No protocolo, foram utilizados os CTAB-AuNRs não modificados de 40 x 85 nm. Como mostrado na Figura 2B, seu máximo plasmônico longitudinal em é de ~650 nm (região vermelha) e a ressonância transversal está em 520 nm (região verde). Literaturas anteriores revelaram que as propriedades ópticas (como a intensidade de LSPR) de AuNRs plasmônicos mudarão significativamente com seu diâmetro20,22. Na Figura 2...
Discussão
O protocolo apresentado é utilizado para estudar a dinâmica dos AuNRs na membrana celular. O protocolo consiste em quatro partes, incluindo imagem microscópica, extração de dados, cálculo de parâmetros dinâmicos e métodos de análise de dados, e cada parte é flexível e universal. Portanto, existem muitas aplicações futuras possíveis, por exemplo, estudando o movimento de moléculas de membrana ligadas ao NP na membrana, dinâmicas de endocitose de receptores rotulados por NP, análise dinâmica de NPs intra...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado pela Fundação Nacional de Ciências Naturais da China com números de bolsas de 21425519, 91853105 e 21621003.
Materiais
| Name | Company | Catalog Number | Comments |
| CTAB coated gold nanorods(CTAB-AuNRs) | Nanoseedz | NR-40-650 | 85 nm * 40 nm |
| Color CMOS camera | Olympus | DP74 | Japan |
| Coverslips | Citoglas | z10212222C | 22*22 mm |
| Dark-field microscopy | Nikon | 80i | upright microscope |
| Fetal bovine serum (FBS) | Gibco | 10099141 | |
| Fiji | National Institutes of Health | 2.0.0-rc-69/1.52 p | a distribution of ImageJ |
| Grooved glass slide | Sail brand | 7103 | Single concave |
| Image J | National Institutes of Health | 1.52 j | |
| MATLAB | MathWorks | R2019b | |
| MATLAB Code | https://github.com/fenggeqd/JOVE-2020 | ||
| Minimum essential medium (MEM) | Gibco | 10-010-CVR | with phenol red |
| Minimum essential medium (MEM) | Gibco | 51200038 | no phenol red |
| Origin | OriginLab | Origin Pro 2018C | |
| Penicillin-streptomycin | Gibco | 15140122 | |
| Plastic cell culture dishes | Falcon | 353002 | |
| Plastic cell culture dishes | Falcon | 353001 | 35*10 mm |
| U87 MG cell | American Type Culture Collection | ATCC HTB-14 | a human primary glioblastoma cell line |
Referências
- Rees, P., Wills, J. W., Brown, M. R., Barnes, C. M., Summers, H. D. The origin of heterogeneous nanoparticle uptake by cells. Nature Communication. 10 (1), 2341 (2019).
- Behzadi, S., et al. Cellular uptake of nanoparticles: journey inside the cell. Chemical Society Reviews. 46 (14), 4218-4244 (2017).
- Shen, H., et al. Single Particle Tracking: From Theory to Biophysical Applications. Chemical Reviews. 117 (11), 7331-7376 (2017).
- Saxton, M. J. Single-particle tracking: connecting the dots. Nature Methods. 5 (8), 671-672 (2008).
- Kusumi, A., et al. Dynamic organizing principles of the plasma membrane that regulate signal transduction: commemorating the fortieth anniversary of Singer and Nicolson's fluid-mosaic model. Annual Review of Cell and Developmental Biology. 28, 215-250 (2012).
- Jacobson, K., Liu, P., Lagerholm, B. C. The Lateral Organization and Mobility of Plasma Membrane Components. Cell. 177 (4), 806-819 (2019).
- Sezgin, E., Levental, I., Mayor, S., Eggeling, C. The mystery of membrane organization: composition, regulation and roles of lipid rafts. Nature Reviews Molecular Cell Biology. 18 (6), 361-374 (2017).
- von Diezmann, A., Shechtman, Y., Moerner, W. E. Three-Dimensional Localization of Single Molecules for Super-Resolution Imaging and Single-Particle Tracking. Chemical Reviews. 117 (11), 7244-7275 (2017).
- Rosenberg, J., Huang, J. Visualizing Surface T-Cell Receptor Dynamics Four-Dimensionally Using Lattice Light-Sheet Microscopy. Journal of Visualized Experiments. (155), e59914 (2020).
- Kusumi, A., Tsunoyama, T. A., Hirosawa, K. M., Kasai, R. S., Fujiwara, T. K. Tracking single molecules at work in living cells. Nature Chemical Biology. 10 (7), 524-532 (2014).
- Rocha, J. M., Gahlmann, A. Single-Molecule Tracking Microscopy - A Tool for Determining the Diffusive States of Cytosolic Molecules. Journal of Visualized Experiments. (151), e59387 (2019).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing protein-DNA interactions in live bacterial cells using photoactivated single-molecule tracking. Journal of Visualized Experiments. (85), e511177 (2014).
- Pinaud, F., Clarke, S., Sittner, A., Dahan, M. Probing cellular events, one quantum dot at a time. Nature Methods. 7 (4), 275-285 (2010).
- Mehidi, A., et al. Transient Activations of Rac1 at the Lamellipodium Tip Trigger Membrane Protrusion. Current Biology. 29 (17), 2852-2866 (2019).
- Ye, Z., Wang, X., Xiao, L. Single-Particle Tracking with Scattering-Based Optical Microscopy. Analytical Chemistry. 91 (24), 15327-15334 (2019).
- Pan, Q., Zhao, H., Lin, X., He, Y. Spatiotemporal Heterogeneity of Reactions in Solution Observed with High-Speed Single-Nanorod Rotational Sensing. Angewandte Chemie. International Ed. in English. 58 (25), 8389-8393 (2019).
- Taylor, R. W., et al. Interferometric scattering microscopy reveals microsecond nanoscopic protein motion on a live cell membrane. Nature Photonics. 13 (7), 480-487 (2019).
- Chen, K., et al. Characteristic rotational behaviors of rod-shaped cargo revealed by automated five-dimensional single particle tracking. Nature Communication. 8 (1), 887 (2017).
- Xu, D., He, Y., Yeung, E. S. Y. Direct observation of the orientation dynamics of single protein-coated nanoparticles at liquid/solid interfaces. Angewandte Chemie. International Ed. in English. 53 (27), 6951-6955 (2014).
- Lehui, X., Yan, H., Edward, S. Y. Three Dimensional Orientational Imaging of Nanoparticles with Darkfield Microscopy. Analytical Chemistry. 82, 5268-5274 (2010).
- Ge, F., Xue, J., Wang, Z., Xiong, B., He, Y. Real-time observation of dynamic heterogeneity of gold nanorods on plasma membrane with darkfield microscopy. Science China Chemistry. 62, 1072-1081 (2019).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Janczura, J., Weron, A. Ergodicity testing for anomalous diffusion: small sample statistics. The Journal of Chemical Physics. 142 (14), 144103 (2015).
- Kim, D. H., et al. Single particle tracking-based reaction progress kinetic analysis reveals a series of molecular mechanisms of cetuximab-induced EGFR processes in a single living cell. Chemical Science. 8 (7), 4823-4832 (2017).
- Kurzthaler, C., et al. Probing the Spatiotemporal Dynamics of Catalytic Janus Particles with Single-Particle Tracking and Differential Dynamic Microscopy. Physical Review Letters. 121 (7), 078001 (2018).
- Lin, X., Pan, Q., He, Y. In situ detection of protein corona on single particle by rotational diffusivity. Nanoscale. 11 (39), 18367-18374 (2019).
- Wei, L., et al. Sub-diffraction-limit localization imaging of a plasmonic nanoparticle pair with wavelength-resolved dark-field microscopy. Nanoscale. 9 (25), 8747-8755 (2017).
- Cheng, X., Dai, D., Xu, D., He, Y., Yeung, E. S. Subdiffraction-limited plasmonic imaging with anisotropic metal nanoparticles. Analytical Chemistry. 86 (5), 2303-2307 (2014).
- Belyy, V., et al. PhotoGate microscopy to track single molecules in crowded environments. Nature Communication. 8, 13978 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados