Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Visualisierung der Diffusionsdynamik von Gold-Nanostäben auf der Zellmembran mit Single Nanoparticle Darkfield-Mikroskopie
In diesem Artikel
Zusammenfassung
Hier zeigen wir den Einsatz der traditionellen Dunkelfeldmikroskopie zur Überwachung der Dynamik von Gold-Nanostäben (AuNRs) auf der Zellmembran. Die Position und Ausrichtung einzelner AuNRs wird mit ImageJ und MATLAB erkannt, und die diffusiven Zustände von AuNRs sind durch eine einzelfaktnverfolgte Analyse gekennzeichnet.
Zusammenfassung
Die Analyse der Diffusionsdynamik von Nanopartikeln auf der Zellmembran spielt eine wichtige Rolle für ein besseres Verständnis des zellulären Aufnahmeprozesses und bietet eine theoretische Grundlage für die rationale Gestaltung der Nanomedizin-Entbindung. Die Einzelpartikelverfolgungsanalyse (SPT) könnte die Position und Ausrichtung einzelner Nanopartikel auf der Zellmembran untersuchen und deren Translations- und Rotationszustände aufzeigen. Hier zeigen wir, wie sie die traditionelle Dunkelfeldmikroskopie nutzt, um die Dynamik von Gold-Nanostäben (AuNRs) auf der lebenden Zellmembran zu überwachen. Wir zeigen auch, wie Sie die Position und Ausrichtung von AuNRs mit ImageJ und MATLAB extrahieren und die diffusiven Zustände von AuNRs charakterisieren. Die statistische Analyse von Hunderten von Partikeln zeigt, dass einzelne AuNRs Brownsche Bewegungen auf der Oberfläche der U87 MG-Zellmembran durchführen. Die individuelle Langzeitanalyse zeigt jedoch, dass AuNRs zwei deutlich unterschiedliche Arten von Bewegungszuständen auf der Membran aufweisen, nämlich Langstreckentransport und ein begrenztes Gebiet. Unsere SPT-Methoden können potenziell verwendet werden, um die Oberflächen- oder intrazelluläre Partikeldiffusion in verschiedenen biologischen Zellen zu untersuchen und kann zu einem leistungsfähigen Werkzeug für Untersuchungen komplexer zellulärer Mechanismen werden.
Einleitung
Die Dynamik von Nanopartikeln (NPs) auf der Membran ist eng mit dem zellulären Aufnahmeprozess verbunden, der für das Verständnis von Zellfunktionen, viralen oder bakteriellen Infektionen und der Entwicklung künstlicher nanomedizinischer Abgabesysteme1,2wesentlich ist. Die Single-Partikel-Tracking-Technik (SPT) ist ein robustes Werkzeug zur Charakterisierung des heterogenen Verhaltens von NPs3,4. Im Allgemeinen ist die Zellmembran flüssig, was bedeutet, dass sich die Komponenten wie Proteine und Lipide seitlich in der Plasmamembranebene5,6,7bewegen können. Die räumlich zeitliche Komplexität der Membranorganisation und -struktur kann zu einer raumzeitlichen Heterogenität der Interaktion zwischen NPs und Membran führen. Daher erfordert die direkte Visualisierung der Bewegung von NPs auf der Membran sowohl eine hohe räumliche als auch zeitliche Auflösung.
Die Einzelpartikel-Tracking-Mikroskopie, die die Lokalisierung einzelner Teilchen in lebenden Zellen mit einer räumlichen Auflösung von zig Nanometern und einer Zeitauflösung von Millisekunden überwacht, wurde gut entwickelt, um die Dynamik von NPs oder Membranmolekülen zu untersuchen8,9. Fluoreszenzbasierte mikroskopische Bildgebungstechniken sind zu wertvollen Werkzeugen für die Beobachtung von NPs/Molekülen in lebenden Zellumgebungen9,10,11,12. Zum Beispiel wurde die gesamte interne Reflexionsfluoreszenzmikroskopie, die dünne Schichten (ca. 100 nm) der Probe an der Substrat-/Lösungsschnittstelle mit einer hohen raumzeitlichen Auflösung abbildt, in Studien der Membranmoleküldynamik13,14weit verbreitet eingesetzt. Die inhärenten Nachteile einzelner Fluorophore, wie niedrige Intensität und schnelle irreversible Photobleichung, reduzieren jedoch die Genauigkeit und Dauer der Verfolgung13. Daher haben nichtfluoreszierende Plasmononische NPs, die die fluoreszierenden Sonden ersetzen, aufgrund ihrer einzigartigen optischen Eigenschaften immer mehr Aufmerksamkeit in Langzeit-Bildgebungsstudien erregt15. Basierend auf den Streusignalen von plasmonischen NP-Sonden wurden verschiedene Arten von optischen mikroskopischen Bildgebungstechnologien verwendet, um den Mechanismus biologischer Prozesse zu untersuchen, wie z. B. Die Dunkelfeldmikroskopie (DFM)16, die interferometrische Streuung (iSCAT) Mikroskopie17 und die Differentialinterferenzkontrastmikroskopie (DICM)18. Darüber hinaus kann die Bewegungs- und Rotationsdynamik von AuNRs mit DFM und DICM18,19,20,21,22erhalten werden. In der Regel wird in einem SPT-Experiment die Bewegung des Objekts vom optischen Mikroskop aufgezeichnet und dann mit SPT-Analysemethoden3analysiert. Die zeitaufgelösten Bahnen und Orientierungswinkel, die von einzelnen NPs erzeugt werden, sind normalerweise stochastisch und heterogen, so dass es notwendig ist, reichlich dynamische Informationen mit verschiedenen Analysemethoden darzustellen.
Hier bieten wir ein integriertes Protokoll, das die Dynamik von AuNRs auf Zellmembran mit DFM überwacht, die Position und Ausrichtung von AuNRs mit ImageJ und MATLAB extrahiert und die Diffusion von AuNRs mit SPT-Analysemethoden charakterisiert. Als Demonstration zeigen wir hier, wie man das SPT-Protokoll verwendet, um die Dynamik von unveränderten AuNRs (CTAB-AuNRs, synthetisiert durch Cetyltrimethylammoniumammoniumbromidmolekül als Schutzmittel) auf der U87 MG-Zellmembran zu visualisieren. Es wurde gezeigt, dass CTAB-AuNRs Proteine in biologischer Umgebung adsorbieren, sich auf der Zellmembran bewegen und dann in Zellen2,20,22eindringen können. U87 MG-Zelle ist der häufigste und bösartigste Tumor des zentralen Nervensystems, und seine Membranrezeptoren sind ungewöhnlich exprimiert. Die Membranrezeptoren können mit Proteinen auf AuNRs interagieren, die die Dynamik von AuNRs beeinflussen. Unser Protokoll ist allgemein auf andere SPT-Experimente auf dem Gebiet der Biologie anwendbar.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Zellkultur
- Bereiten Sie ein komplettes Medium für U87 MG-Zellen vor, indem Sie fetales Rinderserum hinzufügen (Endkonzentration 10%) und Penicillin-Streptomycin (Endkonzentration 1%) auf das minimale wesentliche Medium (MEM). Verwenden Sie Kunststoff-Zellkulturschale für Zellen Subkultur.
- Durchgangszellen 2 bis 3 Mal pro Woche.
- Entfernen Sie das Kulturmedium und spülen Sie die Zellschicht mit der phosphatgepufferten Saline (D-PBS) von Dulbecco 2-3 mal, wenn sie konfluent ist (80 % bis 90 %).
- Fügen Sie der Zellkulturschale 1,0 bis 2,0 ml Trypsin-EDTA-Lösung hinzu und beobachten Sie Zellen unter einem invertierten Mikroskop, bis sich die Zellen drehen (3 x 5 min).
- Fügen Sie 3,0 ml vorbereitetes komplettes Medium hinzu und dispergieren Sie die Zellen durch sanftepipettieren.
- Fügen Sie der Zellsuspension (1 ml) eine neue Kulturschale mit frischem Zellmedium (3 ml) hinzu und setzen Sie die Zellen wieder auf.
- Halten Sie die Zellen bei 37 °C und 5%CO2 in einer befeuchteten Atmosphäre.
2. Mikroskop-Dia-Vorbereitung
HINWEIS: In SPT-Experimenten werden U87 MG-Zellen der dritten bis zehnten Generation mit hoher Aktivität verwendet.
- 22 mm × 22 mm Abdeckungen sterilisieren, die bereits mit Piranha-Lösung gereinigt wurden, indem sie in Ethanol (99,9%) eintauchen.
- Verwenden Sie Zangen, um den Deckelschlupf aus der Ethanollösung (Schritt 2.1) zu nehmen und durch Verbrennen von Ethanol auf der Flamme zu sterilisieren. Sobald alle Ethanol verbrannt sind, legen Sie Deckellipsen in eine Kunststoffzellkulturschale (35 x 10 mm) gefüllt mit 2 ml Zellmedium (kein Phenolrot).
- Fügen Sie 50 l der Zellsuspension ab Schritt 1.2.3 auf den Deckelschlupf und drücken Sie die Schale vorsichtig hin und her und links und rechts, um die Zellen gleichmäßig zu verteilen. Legen Sie es in eine befeuchtete Atmosphäre.
- Wenn U87 MG-Zellen auf dem Deckblatt 20 %–40 % Konfluenz erreichen (ca. 12 h), fügen Sie 20 L CTAB-AuNRs (138 pM) in die Schale ein und dispergieren Sie sie. In kubierter Atmosphäre für 5 min inkubieren.
- Fügen Sie 100 l des Kulturmediums (kein Phenolrot) von der Schale in Schritt 2.4 in die Nut des gerillten Glasschlittens(Abbildung 1) ein, der mit Piranha-Lösung vorgereinigt wird.
- Nehmen Sie den Deckelausrutsche aus der Schale und invertiert auf der Oberseite der Nut des Mikroskop-Glasschlittens (Abbildung 1). Mit Nagellack versiegeln, trocknen lassen und auf die Bühne stellen, um SPT-Experimente durchzuführen.
3. Durchführung von Einzelpartikel-Tracking-Experimenten mit Dunkelfeldmikroskopie (Abbildung 1).
- Legen Sie einen Tropfen Öl auf den ölgetauchten Dunkelfeldkondensator (NA 1.43-1.20) und drehen Sie den Knopf, um den Kondensator mit der Glasrutsche in Berührung zu bringen.
- Legen Sie einen Tropfen Öl auf die Oberseite des Deckglases und drehen Sie den Fokussierknopf, um das 60x Öl-Immersionsobjektiv (NA 0.7–1.25) das Öl berühren zu lassen.
- Schalten Sie die Lichtquelle ein, und drehen Sie den Fokussierknopf leicht, um die Bildebene zu fokussieren.
HINWEIS: Im Sichtfeld ist der Hintergrund schwarz, die Zellen hell und die CTAB-AuNRs (Seitenverhältnis 2:1, Abbildung 2) sind kleine farblich gefärbte (rote, gelbe oder grüne) Streustellen. - Erfassen Sie Dasabstreuen von Licht durch eine Farb-CMOS-Kamera. Klicken Sie in der Software auf"Kamerasymbol", um das TIFF-Format aufzuzeichnen und zu exportieren, um Bilder zu speichern.
4. Datenerfassung
- Extrahieren Sie einzelne langfristige Flugbahn
- Arbeiten Sie wie in Abbildung 3 beschrieben, um die Bilder der Zeitreihe im Dunkelfeld aus dem Modus "RGB-Farbe" in den Modus "8 Bit" zu konvertieren. Klicken Sie im Bild J auf Bild | Typ | 8 Bit. Um den Kontrast anzupassen, klicken Sie auf Bild | Anpassen | Helligkeits-| Kontrast.
- Wählen Sie ein Zielteilchen aus und schneiden Sie die Zeitreihenhintergründe ab, indem Sie den Hintergrund mitStrg+Xboxing und löschen.
- Öffnen Sie das Partikelerkennungs- und Partikelverknüpfungsfenster, indem Sie auf die "Plugins | Partikel-Tracker Klassische | Partikel-Tracker".
- Setzen Sie Radius auf 6, Cutoff auf 0 und Perzentil auf 0,01 %.
HINWEIS: Um das Partikel zu erkennen, passen Sie mit Hilfe von Preview über drei Parameter an. Stellen Sie sicher, dass der Radius etwas größer als das Zielpartikel und kleiner als die kleinste Trennpunkttrennung ist. Perzentil ist die untere Grenze der Intensitätsverteilung, die Kandidatenpartikel sein sollen. - Legen Sie den Linkbereich auf 5 und der Verschiebungshub auf 10 fest.
ANMERKUNG: Um das Partikel zwischen aufeinander folgenden benachbarten Frames zu verknüpfen, passen Sie die beiden oben genannten Parameter an. Verschiebung ist das Maximum an Pixeln, die ein Partikel zwischen zwei nachfolgenden Frames verschieben kann, und LinkBereich ist die Anzahl der aufeinander folgenden Frames, die bei der Bestimmung der besten entsprechenden Übereinstimmung berücksichtigt werden sollen. - Klicken Sie auf "OK", um das ParticleTracker-Ergebnisfenster zu öffnen, um die Ergebnisse anzuzeigen.
- Klicken Sie auf "Alle Flugbahnen visualisieren", um die generierten Flugbahnen zu überprüfen.
- Klicken Sie oben auf das Menü"Partikel neu verknüpfen", um die erkannten Partikel mit unterschiedlichen Linkbereichs- und Perzentilparametern erneut zu verknüpfen, wenn die von der Software generierte Flugbahn nicht mit der bewegten Flugbahn des AuNR übereinstimmt.
- Klicken Sie auf "Vollständiger Bericht speichern", um Ergebnisse zu speichern, wenn die von der Software generierte Flugbahn und die Bewegungsbahn des AuNR übereinstimmen.
HINWEIS: Ein Beispiel für das Extrahieren einzelner Langzeitbahnen mit Bild J ist in Abbildung 4dargestellt.
- Extrahieren von Multitraflugbahnen
HINWEIS: Ein Beispiel für das Extrahieren von Multi-Trajekrien mit Fidschi ist in Abbildung 5dargestellt. Es gibt mehrere Phasen, und jede Phase stellt einen Schritt im Tracking-Prozess dar. Das Ergebnis jedes Schritts wird sofort angezeigt, sodass der Benutzer wieder einstellungen anpassen kann, wenn die Ausgabe unbefriedigend ist.- Arbeiten Sie wie in Abbildung 3 beschrieben, um die Bilder der Zeitreihe im Dunkelfeld aus dem Modus "RGB-Farbe" in den Modus "8 Bit" zu konvertieren. Klicken Sie im Bild-J-Pfad auf "Bild | Typ | 8 Bit". Um den Kontrast anzupassen, klicken Sie auf "Bild | Anpassen | Helligkeits-| Kontrast".
- Öffnen Sie das Startfenster, indem Sie auf"Plugins | Tracking-| TrackMate". Klicken Sie auf "Weiter".
- Wählen Sie den "LoG-Detektor" aus dem Dropdown-Menü und klicken Sie auf"Weiter".
- Passen Sie die Parameter des Detektorkonfigurationspanels an. Legen Sie den geschätzten Blobdurchmesser auf 10 fest, legen Sie Schwellenwert auf 0 fest, und wählen Sie "Do Sub-Pixel Localization" aus.
- Klicken Sie auf"Weiter", um das anfängliche Spotfilterfeld zu öffnen.
HINWEIS: Mehrere Parameter können angepasst werden, um die Zielpunkte weiter zu optimieren. In diesem Beispiel wurden keine anderen Parameter angepasst. - Klicken Sie im Dropdown-Menü auf "Weiter" und wählen Sie "HyperStack-Displayer".
- Klicken Sie auf"Weiter", um das Spot-Filterfeld zu öffnen. Stellen Sie die Qualität über 1,88, X über 38,86, Y über 56,54 ein.
- Klicken Sie im Dropdown-Menü auf "Weiter" und wählen Sie "Simple LAP tracker". Konfigurieren Sie den einfachen LAP-Tracker, indem Sie drei Parameter anpassen, d. h. den Linking max Distance = 15, Gap-closing max distance = 15 und Gap-closing max frame gap = 5.
HINWEIS: Linking-max Abstand ist die maximale Verschiebung eines Punkts zwischen zwei Frames. Der lückenschließende maximale Abstand ist die maximale Verschiebung von zwei Segmenten. Lückenschließende maximale Rahmenlücke ist der größte Rahmen zwischen zwei Zubrücken. - Klicken Sie auf "Weiter", um nachzuverfolgen. Wenn Sie fertig sind, klicken Sie weiter auf"Weiter".
- Legen Sie Filter auf Spuren fest, z. B. Anzahl der Spots in Spur über 300.
- Klicken Sie weiterhin auf"Weiter", bis das endgültige Speicherfeld geöffnet ist. Wählen Sie"Tracks in XML-Datei exportieren" aus dem Dropdown-Menü aus, und klicken Sie dann auf "Ausführen", um im csv-Format zu speichern.
- R/G-Werte
HINWEIS: Streuintensitäten von gezielten AuNRs in den R- und G-Kanälen werden aus Farbdunkelfeldbildern mithilfe eines in MATLAB (https://github.com/fenggeqd/JOVE-2020/tree/master/RGandPolarangle) geschriebenen Codes ermittelt, und das Extraktionsprinzip ist in Abbildung 6dargestellt.- Verwenden Sie die Funktion der xycoordination.m, um die mittlere Pixelkoordinate des AuNR in jedem Frame entsprechend der x-y-Koordinate (extrahiert durch ImageJ/Fiji) zu finden.
- Verwenden Sie die Funktion der RGextraction.m, um eine 3 x 3 Pixel Matrix abzugrenzen, die 9 Streuintensitätswerte von R- oder G-Kanälen zu extrahieren und einen Durchschnittswert zu berechnen (μ, definiert als R oder G).
HINWEIS: Die Matrix von 3 x 3 Pixeln wird auf den Pixelkoordinaten zentriert, die aus Schritt 4.3.1 erhalten wurden.
- Polarwinkel
HINWEIS: Der Polarwinkel ist der Winkel zwischen dem Längengrad des AuNR und der optischen Achse (wie in Abbildung 1dargestellt), der die räumliche (Z-Achse) Rotationsdynamik des AuNR widerspiegeln kann.- Verwenden Sie die Funktion von polarangle.m, um die Polarwinkel () nach der dual-kanaligen Differentialmethode22,
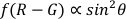 zu berechnen.
zu berechnen.
- Verwenden Sie die Funktion von polarangle.m, um die Polarwinkel () nach der dual-kanaligen Differentialmethode22,
5. Datenanalyse
HINWEIS: Ein systematisches und robustes Datenanalyse-Framework ist für die Leistung und Effizienz von SPT-Analysemethoden unerlässlich. Die in MATLAB geschriebene benutzerdefinierte Software wird verwendet (https://github.com/fenggeqd/JOVE-2020/tree/master/Analysis_parameters). Zum Zeichnen der Diagramme wird eine Grafik- und Analysesoftware (siehe Materialtabelle)verwendet.
- Analyseparameter
- Verwenden Sie Skripte csv_data_extract_dis_vel_ss.m und csv_data_MSD.m, um dynamische Parameter gemäß den in Tabelle 1dargestellten Formeln zu berechnen.
HINWEIS: Diese Parameter werden verwendet, um die Dynamik von AuNRs zu analysieren und bestehen aus drei Teilen. (1) Trajektorie-bezogene Parameter: Verschiebung, Schrittgröße, Geschwindigkeit, Kreiselradius (Rg) und Drehwinkel (Ta); (2) MSD-Parameter: Diffusionskoeffizient (Dt) und abnormaler Diffusionsexponent (α); und (3) Rotationsparameter: Polarwinkel und Drehlability (σ).
- Verwenden Sie Skripte csv_data_extract_dis_vel_ss.m und csv_data_MSD.m, um dynamische Parameter gemäß den in Tabelle 1dargestellten Formeln zu berechnen.
| Maßnahmen | Definition | Physikalische Bedeutung | |
| Verschiebung | 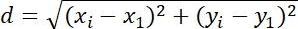 | Positionsänderungen von Objekten | |
| Schrittgröße | 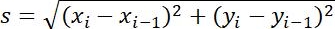 | Abstand zwischen zwei benachbarten Punkten | |
| Geschwindigkeit |  | Geschwindigkeit der Objektbewegung | |
| Rg | 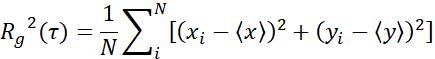 | Verschieben des Objektbereichs in einem bestimmten Zeitintervall | |
| Ta | 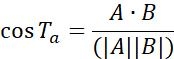 | Bewegungsrichtung von Objekten zwischen zwei benachbarten Punkten | |
| Msd | 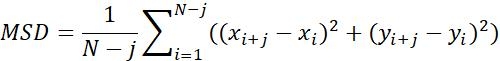 | Durchschnittliche bewegungsweite von Objekten in einem bestimmten Zeitintervall | |
| Dt | 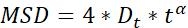 | Diffusionsfähigkeit von Objekten | |
| Α | 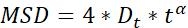 | Normale Diffusion (α 1) | |
| Polarwinkel |  | 3D-Orientierungsinformationen von Objekten | |
| Σ |  | Streugrad des Polarwinkel-Datensatzes | |
Tabelle 1: Drei Arten von Parametern, die für die Analyse verwendet werden. Dazu gehören bahnbezogene Parameter (Verschiebung, Schrittgröße, Geschwindigkeit, Rg und Ta), MSD-Parameter (MSD, Dt und α) und rotationsbezogene Parameter (Polarwinkel und Drehbarkeit).
- Visuelle Analyse der Flugbahn
HINWEIS: Die Flugbahnvisualisierung kann intuitiv die raumzeitliche Heterogenität der Partikelbewegung und die Flugbahnverteilung (Koordinaten) dynamischer Parameter darstellen, wie z. B. Zeitmapping-Trajektorie, Rg- und Dt-Mapping-Trajektorie und Polarwinkel-Mapping-Trajektorie. Mapping-Trajektorien wurden mit der Graphik- und Analysesoftware gezeichnet.- Legen Sie x-Koordinate als X, y-Koordinate als Y und Zeit (Rg, Dt, Polarwinkel) als Z fest.
- Klicken Sie auf "Plot | Streudiagramm | Color-mapping Plot".
- Fügen Sie die Farbleiste hinzu.
- MSD-Analyse
HINWEIS: Bewegungsaktivität und Bewegungsmodus der Partikel können durch MSD-Analyse23erreicht werden. Je größer der Dt,desto aktiver ist die Diffusionsbewegung der Teilchen. Wenn α 1, Partikel normale Diffusionsbewegung durchführen, sonst führen sie abnormale Diffusionsbewegung aus.- MSD-Zahlen
- Legen Sie das Zeitintervall () als X fest, MSD-Daten als Y.
- Klicken Sie auf "Plot | Streudiagramm"
- Anpassen von Daten durch Klicken auf "Analysieren | Fitting | Nichtlineare Kurvenanpassung (Funktion: Allometricl)".
- MSD-- - Doppellogarithmische Figur
ANMERKUNG: Die MSD---Doppellogarithmische Figur, deren Neigung α ist und abfangen Dtist, kann intuitiv Partikelbewegungen darstellen.- Legen Sie das logarithmische Zeitintervall als X und die logarithmische MSD als Y fest.
- Klicken Sie auf "Plot | Streudiagramm".
- Anpassen von Daten durch Klicken auf "Analysieren | Fitting | Lineare Kurvenanpassung".
- D-Nr. (MSD/4t)
ANMERKUNG: D=MSD/4, ist eine Funktion des Zeit- und Anomaliefaktors α, und die D-Zahl zeigt direkt die Veränderung des Diffusionskoeffizienten mit der Zeit an. Wenn D mit der Zeit zunimmt, ist α größer als 1 und Partikel machen Superdiffusionsbewegung.- Legen Sie das logarithmische Zeitintervall () als X und das logarithmische D als Y fest.
- Klicken Sie auf "Plot | Streudiagramm".
- Anpassen von Daten durch Klicken auf "Analysieren | Fitting | Nichtlineare Kurvenanpassung (Funktion: "Allometricl")".
ANMERKUNG: Je kürzer die Flugbahnen, desto höher ist die Ungenauigkeit der Diffusionsschätzungen. Im Allgemeinen wurden Dt und α durch MSD-Analyse der langen Intervallzeit (> 30) ermittelt. Einfache und grobe Passform glättet jedoch Bewegungsdetails. Daher sollte eine MSD--Analyse der kurzen Intervallzeit (< 10) durchgeführt werden, um das Bewegungsverhalten von Teilchen in kurzer Zeit zu analysieren.
- MSD-Zahlen
- Statistische Auswertung
- Multi-Partikel-Statistik
HINWEIS: Die statistische Analyse mehrerer Partikel kann den Bewegungszustand von Partikeln in einem räumlichen Bereich widerspiegeln, was indirekt die räumliche Heterogenitätsumgebung anzeigt. Wenn beispielsweise das Histogramm von Dt eine großflächige Oder Multipeaks-Verteilung aufweist, bedeutet dies, dass die Bewegungsaktivitäten von Teilchen heterogen sind.- Legen Sie dynamische Parameter (z. B. Dt, Rg, max Verschiebung) als Y fest.
- Klicken Sie auf "Plot | Histogramm".
- Doppelklicken Sie auf das Histogramm, und legen Sie die Divisionsgröße oder die Anzahl der Divisionen fest. Klicken Sie auf "Bewerben".
- Statistische Einzelpartikelanalyse
HINWEIS: Die statistische Analyse einzelner Teilchen kann das Bewegungsverhalten einzelner Teilchen zeigen, was auch indirekt die räumlich-zeitliche Heterogenität der Umgebung widerspiegelt.- Mehrere Frames
- Berechnen Sie dynamische Parameter (z. B. Ta, Schrittgröße, Polarwinkel) aller Frames einer einzelnen langen Flugbahn, und kopieren Sie in die Origin-Tabelle und legen Sie als Y fest.
- Klicken Sie auf "Plot | Histogramm".
- Doppelklicken Sie auf das Histogramm, und legen Sie die Divisionsgröße oder die Anzahl der Divisionen fest. Klicken Sie auf "Bewerben".
HINWEIS: Wenn sowohl Ta als auch Schrittgröße eine kleine Werteverteilung aufweisen, machen die Partikel eine kleine Superdiffusionsbewegung.
- Verschieben von Fenstern
- Berechnen Sie dynamische Parameter (z. B. Rg, Dt) aller Frames einer einzelnen langen Flugbahn durch die Moving-Window-Methode (11 Frames), und kopieren Sie in die Origin-Tabelle und legen Sie als Y fest.
- Klicken Sie auf "Plot | Histogramm".
- Doppelklicken Sie auf das Histogramm, und legen Sie die Divisionsgröße oder die Anzahl der Divisionen fest. Klicken Sie auf "Bewerben".
- Mehrere Frames
- Multi-Partikel-Statistik
- Zeitreihenanalyse
HINWEIS: Statistische Analysen können den Bewegungszustand von NPs aufdecken, und Zeitreihenanalysen können das Bewegungsverhalten als Ergänzung darstellen. Durch die Kombination mehrerer Zeitreihenparameter kann das Bewegungsverhalten von NPs auf zeitlicher und räumlicher Ebene diskriminiert werden.- Legen Sie die Zeit als X fest, Zeitreihenparameter als Y (z. B. Verschiebung, Polarwinkel und Rg).
- Klicken Sie auf "Plot | Multi-Fenster-Diagramm | Gestapeltes Plot-| Zeile + Symbol".
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Im Protokoll wurden die unveränderten 40 x 85 nm CTAB-AuNRs verwendet. Wie in Abbildung 2Bdargestellt, liegt sein Längsplasmamaximum bei 650 nm (roter Bereich) und die Querresonanz bei 520 nm (grüner Bereich). Frühere Literaturen haben gezeigt, dass sich die optischen Eigenschaften (wie die LSPR-Intensität) von plasmonischen AuNRs mit ihrem Durchmesser20,22signifikant verändern werden. In Abbildung 2C
Access restricted. Please log in or start a trial to view this content.
Diskussion
Das vorgestellte Protokoll wird verwendet, um die Dynamik von AuNRs auf Zellmembran zu untersuchen. Das Protokoll besteht aus vier Teilen, einschließlich mikroskopischer Bildgebung, Datenextraktion, Berechnung dynamischer Parameter und Datenanalysemethoden, und jedes Teil ist flexibel und universell. Daher gibt es viele mögliche zukünftige Anwendungen, zum Beispiel die Untersuchung der Bewegung von NP-verknüpften Membranmolekülen auf der Membran, endozytose Dynamik von NP-markierten Rezeptoren, dynamische Analyse vo...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde von der National Natural Science Foundation of China mit den Fördernummern 21425519, 91853105 und 21621003 unterstützt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| CTAB coated gold nanorods(CTAB-AuNRs) | Nanoseedz | NR-40-650 | 85 nm * 40 nm |
| Color CMOS camera | Olympus | DP74 | Japan |
| Coverslips | Citoglas | z10212222C | 22*22 mm |
| Dark-field microscopy | Nikon | 80i | upright microscope |
| Fetal bovine serum (FBS) | Gibco | 10099141 | |
| Fiji | National Institutes of Health | 2.0.0-rc-69/1.52 p | a distribution of ImageJ |
| Grooved glass slide | Sail brand | 7103 | Single concave |
| Image J | National Institutes of Health | 1.52 j | |
| MATLAB | MathWorks | R2019b | |
| MATLAB Code | https://github.com/fenggeqd/JOVE-2020 | ||
| Minimum essential medium (MEM) | Gibco | 10-010-CVR | with phenol red |
| Minimum essential medium (MEM) | Gibco | 51200038 | no phenol red |
| Origin | OriginLab | Origin Pro 2018C | |
| Penicillin-streptomycin | Gibco | 15140122 | |
| Plastic cell culture dishes | Falcon | 353002 | |
| Plastic cell culture dishes | Falcon | 353001 | 35*10 mm |
| U87 MG cell | American Type Culture Collection | ATCC HTB-14 | a human primary glioblastoma cell line |
Referenzen
- Rees, P., Wills, J. W., Brown, M. R., Barnes, C. M., Summers, H. D. The origin of heterogeneous nanoparticle uptake by cells. Nature Communication. 10 (1), 2341(2019).
- Behzadi, S., et al. Cellular uptake of nanoparticles: journey inside the cell. Chemical Society Reviews. 46 (14), 4218-4244 (2017).
- Shen, H., et al. Single Particle Tracking: From Theory to Biophysical Applications. Chemical Reviews. 117 (11), 7331-7376 (2017).
- Saxton, M. J. Single-particle tracking: connecting the dots. Nature Methods. 5 (8), 671-672 (2008).
- Kusumi, A., et al. Dynamic organizing principles of the plasma membrane that regulate signal transduction: commemorating the fortieth anniversary of Singer and Nicolson's fluid-mosaic model. Annual Review of Cell and Developmental Biology. 28, 215-250 (2012).
- Jacobson, K., Liu, P., Lagerholm, B. C. The Lateral Organization and Mobility of Plasma Membrane Components. Cell. 177 (4), 806-819 (2019).
- Sezgin, E., Levental, I., Mayor, S., Eggeling, C. The mystery of membrane organization: composition, regulation and roles of lipid rafts. Nature Reviews Molecular Cell Biology. 18 (6), 361-374 (2017).
- von Diezmann, A., Shechtman, Y., Moerner, W. E. Three-Dimensional Localization of Single Molecules for Super-Resolution Imaging and Single-Particle Tracking. Chemical Reviews. 117 (11), 7244-7275 (2017).
- Rosenberg, J., Huang, J. Visualizing Surface T-Cell Receptor Dynamics Four-Dimensionally Using Lattice Light-Sheet Microscopy. Journal of Visualized Experiments. (155), e59914(2020).
- Kusumi, A., Tsunoyama, T. A., Hirosawa, K. M., Kasai, R. S., Fujiwara, T. K. Tracking single molecules at work in living cells. Nature Chemical Biology. 10 (7), 524-532 (2014).
- Rocha, J. M., Gahlmann, A. Single-Molecule Tracking Microscopy - A Tool for Determining the Diffusive States of Cytosolic Molecules. Journal of Visualized Experiments. (151), e59387(2019).
- Uphoff, S., Sherratt, D. J., Kapanidis, A. N. Visualizing protein-DNA interactions in live bacterial cells using photoactivated single-molecule tracking. Journal of Visualized Experiments. (85), e511177(2014).
- Pinaud, F., Clarke, S., Sittner, A., Dahan, M. Probing cellular events, one quantum dot at a time. Nature Methods. 7 (4), 275-285 (2010).
- Mehidi, A., et al. Transient Activations of Rac1 at the Lamellipodium Tip Trigger Membrane Protrusion. Current Biology. 29 (17), 2852-2866 (2019).
- Ye, Z., Wang, X., Xiao, L. Single-Particle Tracking with Scattering-Based Optical Microscopy. Analytical Chemistry. 91 (24), 15327-15334 (2019).
- Pan, Q., Zhao, H., Lin, X., He, Y. Spatiotemporal Heterogeneity of Reactions in Solution Observed with High-Speed Single-Nanorod Rotational Sensing. Angewandte Chemie. International Ed. in English. 58 (25), 8389-8393 (2019).
- Taylor, R. W., et al. Interferometric scattering microscopy reveals microsecond nanoscopic protein motion on a live cell membrane. Nature Photonics. 13 (7), 480-487 (2019).
- Chen, K., et al. Characteristic rotational behaviors of rod-shaped cargo revealed by automated five-dimensional single particle tracking. Nature Communication. 8 (1), 887(2017).
- Xu, D., He, Y., Yeung, E. S. Y. Direct observation of the orientation dynamics of single protein-coated nanoparticles at liquid/solid interfaces. Angewandte Chemie. International Ed. in English. 53 (27), 6951-6955 (2014).
- Lehui, X., Yan, H., Edward, S. Y. Three Dimensional Orientational Imaging of Nanoparticles with Darkfield Microscopy. Analytical Chemistry. 82, 5268-5274 (2010).
- Ge, F., Xue, J., Wang, Z., Xiong, B., He, Y. Real-time observation of dynamic heterogeneity of gold nanorods on plasma membrane with darkfield microscopy. Science China Chemistry. 62, 1072-1081 (2019).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Janczura, J., Weron, A. Ergodicity testing for anomalous diffusion: small sample statistics. The Journal of Chemical Physics. 142 (14), 144103(2015).
- Kim, D. H., et al. Single particle tracking-based reaction progress kinetic analysis reveals a series of molecular mechanisms of cetuximab-induced EGFR processes in a single living cell. Chemical Science. 8 (7), 4823-4832 (2017).
- Kurzthaler, C., et al. Probing the Spatiotemporal Dynamics of Catalytic Janus Particles with Single-Particle Tracking and Differential Dynamic Microscopy. Physical Review Letters. 121 (7), 078001(2018).
- Lin, X., Pan, Q., He, Y. In situ detection of protein corona on single particle by rotational diffusivity. Nanoscale. 11 (39), 18367-18374 (2019).
- Wei, L., et al. Sub-diffraction-limit localization imaging of a plasmonic nanoparticle pair with wavelength-resolved dark-field microscopy. Nanoscale. 9 (25), 8747-8755 (2017).
- Cheng, X., Dai, D., Xu, D., He, Y., Yeung, E. S. Subdiffraction-limited plasmonic imaging with anisotropic metal nanoparticles. Analytical Chemistry. 86 (5), 2303-2307 (2014).
- Belyy, V., et al. PhotoGate microscopy to track single molecules in crowded environments. Nature Communication. 8, 13978(2017).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten