Method Article
Análisis de la dinámica de la actina, el acoplamiento del embrague y la fuerza de tracción para el avance del cono de crecimiento
En este artículo
Resumen
Para avanzar, los conos de crecimiento deben ejercer fuerzas de tracción contra el entorno externo. La generación de fuerzas de tracción depende de la dinámica de la actina y el acoplamiento del embrague. El presente estudio describe métodos para analizar la dinámica de la actina, el acoplamiento del embrague y las fuerzas de tracción para el avance del cono de crecimiento.
Resumen
Para establecer redes funcionales, las neuronas deben migrar a sus destinos apropiados y luego extender los axones hacia sus células objetivo. Estos procesos dependen de los avances de los conos de crecimiento que se encuentran en las puntas de las neuritas. Los conos de crecimiento axonal generan fuerzas motrices al detectar su microambiente local y modular la dinámica citoesquelética y el acoplamiento actina-adhesión (acoplamiento de embrague). Décadas de investigación han llevado a la identificación de moléculas de guía, sus receptores y cascadas de señalización aguas abajo para regular la migración neuronal y la guía axonal; sin embargo, las maquinarias moleculares requeridas para generar fuerzas para impulsar el avance del cono de crecimiento y la navegación apenas están comenzando a dilucidarse. En el borde delantero de los conos de crecimiento neuronal, los filamentos de actina experimentan un flujo retrógrado, que es impulsado por la polimerización de actina y la contracción de actomiosina. Un acoplamiento de embrague entre el flujo retrógrado de F-actina y el sustrato adhesivo genera fuerzas de tracción para el avance del cono de crecimiento. El presente estudio describe un protocolo detallado para monitorear el flujo retrógrado de F-actina mediante imágenes de una sola mota. Es importante destacar que, cuando se combina con un marcador de actina F Lifeact, esta técnica puede cuantificar 1) la tasa de polimerización de F-actina y 2) la eficiencia de acoplamiento del embrague entre el flujo retrógrado de F-actina y el sustrato adhesivo. Ambas son variables críticas para generar fuerzas para el avance y la navegación del cono de crecimiento. Además, el presente estudio describe un protocolo detallado de microscopía de fuerza de tracción, que puede cuantificar 3) la fuerza de tracción generada por los conos de crecimiento. Por lo tanto, al acoplar los análisis de imágenes de una sola mancha y microscopía de fuerza de tracción, los investigadores pueden monitorear la mecánica molecular subyacente al avance y la navegación del cono de crecimiento.
Introducción
En el cerebro de vertebrados en desarrollo, las neuronas se someten a migraciones elaboradamente organizadas y proyectan axones hacia socios sinápticos apropiados para establecer redes neuronales funcionales1,2,3. Los conos de crecimiento, que son estructuras sensoriales y móviles ubicadas en la punta de las neuritas, determinan la velocidad y la dirección de la migración neuronal y el crecimiento del axón3,4,5. Dado que las neuronas están rodeadas de ambientes apretados, los conos de crecimiento deben ejercer fuerzas contra su entorno para avanzar6,7. Para comprender los mecanismos subyacentes a la migración neuronal y la guía axonal, los análisis de la mecánica molecular para el avance del cono de crecimiento son esenciales.
Décadas de análisis han revelado que la fuerza de tracción para impulsar el avance del cono de crecimiento es generada por el mecanismo de 'embrague'; se cree que este mecanismo funciona no solo en el cono de crecimiento axonal, sino también en el cono de crecimiento del proceso principal de las neuronas migratorias8,9,10,11,12. Es decir, los filamentos de actina (F-actinas) en los conos de crecimiento polimerizan en el borde de ataque y se despolimerizan proximalmente, expulsando la membrana de vanguardia13,14,15. La fuerza resultante, junto con la contracción de la actomiosina, induce el movimiento hacia atrás de las F-actinas llamado flujo retrógrado7,11,16,17,18,19,20,21. Las moléculas de adhesión de embrague y célula median el acoplamiento mecánico entre el flujo retrógrado de F-actina y el sustrato adhesivo y transmiten la fuerza del flujo de F-actina sobre el sustrato, generando así fuerza de tracción para el avance del cono de crecimiento7,8,9,11,12,22 . Al mismo tiempo, el acoplamiento actina-sustrato reduce la velocidad de flujo F-actina y convierte la polimerización de actina en la fuerza para sobresalir de la membrana de vanguardia9,10.
Los conos de crecimiento axonales detectan señales químicas locales y las transducen en una fuerza motriz direccional para la navegación del cono de crecimiento3,23,24,25. Por ejemplo, una molécula de guía axónica netrin-1 estimula su receptor eliminado en el cáncer colorrectal (DCC) y activa las proteínas de unión a rho guanosina trifosfato (GTP) proteína de control de división celular 42 (Cdc42) y el sustrato 1 de toxina botulínica C3 relacionada con Ras (Rac1), y su quinasa 1 activada por quinasa p21 aguas abajo (Pak1)26. Cdc42 y Rac1 promueven 1) la polimerización de actina, y Pak1 fosforila una molécula de embrague shootin122,26. Shootin1 interactúa con el flujo retrógrado de F-actina a través de una proteína cortactina de unión a actina27. Shootin1 también interactúa con la molécula de adhesión celular L1 (L1-CAM)20,24. La fosforilación de Shootin1 aumenta las afinidades de unión para cortactina y L1-CAM, y mejora el acoplamiento de embrague 2) mediado por shootin124,27. Dentro del cono de crecimiento, las activaciones asimétricas de la polimerización de actina y el acoplamiento del embrague aumentan 3) la fuerza de tracción en el lado de la fuente netrin-1, generando así una fuerza motriz direccional para el giro del cono de crecimiento (Figura 1)24. La investigación intensiva en las últimas décadas con respecto a la migración neuronal y la guía de axones ha mejorado la comprensión de las moléculas de guía, sus receptores y las cascadas de señalización aguas abajo asociadas2,10,28,29,30. Sin embargo, las maquinarias moleculares para generar fuerzas para el avance del cono de crecimiento apenas están comenzando a dilucidarse; esto puede atribuirse al uso limitado de los protocolos para los análisis mecanobiológicos.
El presente estudio describe un protocolo detallado para el monitoreo del flujo retrógrado de F-actina mediante imágenes de una sola mota16,18. El monitoreo del flujo retrógrado de F-actina se ha realizado ampliamente utilizando microscopía de superresolución, microscopía confocal de disco giratorio y microscopía de fluorescencia de reflexión de interferencia total (TIRF)25,31,32,33,34,35,36,37,38 . El protocolo del presente estudio, sin embargo, utiliza un microscopio de epifluorescencia estándar y, por lo tanto, es fácilmente adoptable11,16,18,20,22,23,24,27,39,40,41,42. Cuando se combina con el etiquetado de F-actina de Lifeact43, la imagen de una sola mota permite cuantificar la tasa de polimerización de actina y la eficiencia de acoplamiento del embrague entre el flujo retrógrado de F-actina y el sustrato adhesivo39,42. El presente estudio describe además un protocolo detallado de microscopía de fuerza de tracción utilizando un gel de poliacrilamida (PAA) incrustado en perla fluorescente11,22,23,24,27,39,41,42,44. Este método detecta y cuantifica la fuerza de tracción bajo el cono de crecimiento mediante el monitoreo de los movimientos de perlas inducidos por la fuerza44,45. Se proporciona un código de análisis de fuerza de tracción de código abierto y se explica en detalle el método para cuantificar la fuerza de tracción durante la migración del cono de crecimiento. Con la ayuda de imágenes de una sola mancha y microscopía de fuerza de tracción, se facilitará la comprensión de la mecánica molecular subyacente a la migración y navegación del cono de crecimiento. Estas técnicas también son aplicables para analizar la mecánica molecular subyacente al agrandamiento de la columna dendrítica, que se sabe que es importante en el aprendizaje y la memoria42.
Protocolo
Todos los experimentos con animales de laboratorio se realizaron con el Comité Institucional de Cuidado y Uso de Animales del Instituto de Ciencia y Tecnología de Nara. Los investigadores deben seguir las directrices establecidas por sus comités reguladores de animales institucionales y nacionales para el cuidado y uso de animales de laboratorio.
1. Preparación de soluciones y medios
- Preparar las soluciones y los medios de cultivo como se resume en el Archivo Suplementario 1
2. Preparación de sustratos recubiertos de poli-D-lisina (PDL)/laminina
- Cubra los platos con fondo de vidrio de 14 mm de diámetro con 100 μg/ml de PDL, disueltos en solución salina tamponada con fosfato (PBS, pH 7.4), e incube durante la noche en una incubadora humidificada a 37 °C.
NOTA: No seque las superficies de vidrio después de este paso - Retire la solución PDL y pipetee 1 ml de PBS en el vidrio durante 10 s tres veces.
- Cubra los platos con 5 μg/ml de laminina, disuelto en PBS, e incube durante la noche en una incubadora humidificada a 37 °C.
- Retire la solución de laminina y pipete 1 ml de PBS en el vidrio durante 10 s tres veces.
- Retire el PBS y coloque 0,5 ml del medio neurobasal sobre las superficies de vidrio. Guarde los platos en una incubadora humidificada a 37 °C.
NOTA: Los platos recubiertos de PDL/laminina se pueden almacenar durante 2-3 días en una incubadora humidificada a 37 °C.
3. Disección y disociación del hipocampo
- Eutanasia a una ratón embarazada por luxación cervical.
NOTA: El presente estudio utiliza ratones disponibles comercialmente (ver Tabla de Materiales). Los investigadores deben usar ratones que sean criados y tratados humanamente en un ambiente esterilizado. - Diseccionar embrionarios de ratón del día 16 (E16) y colocarlos en hielo.
- En una campana de flujo laminar, diseccione los cerebros con tijeras y colóquelos en un plato estéril de 10 cm que contenga 10 ml de solución de disección helada.
- Con un microscopio estereoscópico de disección, despegue cuidadosamente las meninges de los hemisferios cerebrales y luego diseccione los hipocampos con fórceps.
- Transfiera el hipocampo a 5 ml de solución de digestión helada en un tubo de 15 ml. Incubar el hipocampo en un baño de agua durante 20 min a 37 °C.
- Retire la solución de digestión y agregue 3 ml de solución de disección.
- Pipetee suavemente el hipocampo cuatro veces con una pipeta Pasteur.
- Incubar el hipocampo en un baño de agua durante 20 min a 37 °C.
- Transfiera la suspensión celular a un tubo centrífugo de 50 ml. Añadir 3 ml de nueva solución de disección al tejido no disociado.
- Repita los pasos 3.7-3.9 hasta que el hipocampo esté completamente disociado.
- Elimine los agregados flotantes de ADN de la suspensión celular girando y aspirando con una pipeta Pasteur. Centrifugar la suspensión celular a 180 x g durante 20 min a 4 °C.
NOTA: El ADN derivado de células dañadas perturbará el proceso de centrifugación. Si las neuronas permanecen en el sobrenadante, centrífuga la suspensión celular nuevamente después de eliminar cuidadosamente el ADN. - Medio neurobasal equilibrante que contiene 10% de suero fetal bovino (FBS), penicilina (100 U/mL) y estreptomicina (100 μg/mL) en una incubadora humidificada a 37 °C con 5% de CO2.
4. Transfección y cultivo de neuronas
- Retire el sobrenadante y agregue 5 ml de PBS helado en el tubo de 50 ml. Resuspend el pellet celular mediante pipeteo suave.
- Transfiera 10 μL de la suspensión celular a un tubo de microcentrífuga, agregue 10 μL de solución de azul de tripano al 0,5% y luego cuente el número de células con un hemocitómetro.
- Centrifugar la suspensión celular en el tubo de 50 ml a 180 x g durante 20 min a 4 °C.
- Mientras tanto, la alícuota para la transfección, 1 μg de pFN21A-HaloTag-actina y 3 μg de pmNeonGreen-N1-Lifeact por 1 x 106 células, en un tubo de microcentrífuga.
- Después de la centrifugación, retire el sobrenadante y agregue 100 μL de medio de electroporación (proporcionado por el kit de transfección) en el tubo de 50 ml. Resuspendir el pellet celular mediante pipeteo.
- Mezcle la suspensión celular con la solución de ADN alicitada y transfiera la mezcla a una cubeta (proporcionada por el kit de transfección).
NOTA: Dado que las burbujas de aire perturban la electroporación, retírelas de la suspensión de la celda. - Inserte la cubeta en el aparato de electroporación (Tabla de Materiales) y realice la electroporación utilizando el programa O-005.
- Agregue inmediatamente 1 ml del medio neurobasal precalentado y equilibrado que contiene 10% de FBS, penicilina (100 U / ml) y estreptomicina (100 μg / ml) a la cubeta.
- Transfiera la suspensión celular a un tubo centrífugo de 15 ml utilizando una pipeta de plástico (proporcionada por el kit de transfección).
- Transfiera 10 μL de la suspensión celular a un microtubo, agregue 10 μL de solución de azul de tripano al 0,5% y luego cuente el número de células con un hemocitómetro.
- Saque los platos con fondo de vidrio recubiertos de PDL / laminina de la incubadora y retire el medio neurobasal.
- Pipetear 0,5 mL de la suspensión celular que contiene 2,0 x 105 células por plato e incubar en una incubadora humidificada a 37 °C con 5% de CO2 durante 3 h.
- Reemplace el medio con 0.5 ml de medio neurobasal que contenga un suplemento de B-27 al 2%, glutamina (1 mM), penicilina (100 U / ml) y estreptomicina (100 μg / ml). Cultiva las neuronas en una incubadora humidificada durante 3 días a 37 °C con un 5% de CO2.
5. Imágenes de una sola mota en los conos de crecimiento neuronal
- El día in vitro (DIV) 3, tratar las neuronas con ligando tetrametil-rodamina (TMR) a una dilución de dilución 1:2000 en medio de cultivo. Mantener las neuronas durante 1 h a 37 °C con un 5% de CO2.
- Lave el ligando TMR tres veces con PBS precalentado.
- Retire el PBS y pipetee 0.5 ml del medio L-15 de Leibovitz calentado que contiene un suplemento de B-27 al 2%, glutamina (1 mM), penicilina (100 U / ml) y estreptomicina (100 μg / ml).
- Mantener las neuronas durante 1 h a 37 °C.
- Encienda un microscopio de epifluorescencia y ajuste la incubadora de la parte superior del escenario a 37 °C.
NOTA: El presente protocolo utiliza un microscopio de epifluorescencia equipado con una cámara semiconductora complementaria de óxido metálico, una lente de objetivo de inmersión en aceite de 100x/1.40 NA, una lámpara de mercurio y un software de adquisición de imágenes (ver Tabla de Materiales). Para este análisis también se pueden utilizar otros microscopios de epifluorescencia con especificaciones equivalentes. - Coloque las neuronas tratadas con ligando TMR en el plato con fondo de vidrio en la incubadora de la parte superior de la etapa calentada.
- Establezca los parámetros de adquisición de imágenes de la siguiente manera: tiempo de exposición, 500 ms para los canales fluorescentes Lifeact y HaloTag-actina; binning, 1 x 1 (0,065 μm × 0,065 μm por píxel); intervalo de tiempo, 3 s; duración, 50 fotogramas.
- Seleccione un cono de crecimiento que exprese fuertemente Lifeact y exprese débilmente HaloTag-actina.
NOTA: La expresión de Lifeact debe ser lo suficientemente fuerte como para visualizar la morfología del cono de crecimiento. La expresión de HaloTag-actina debe ser débil e insuficiente para ser detectada (Figura 2A). Los niveles de expresión de proteínas fluorescentes se pueden ajustar variando la cantidad de ADN para la transfección. - Cierre el campo del diafragma para iluminar un área mínima que incluya el cono de crecimiento (Figura 2B).
NOTA: La iluminación de un área mínima reduce la señal de fondo y aumenta la relación señal de fluorescencia a ruido (S / N), lo que permite la detección de manchas de actina (Figura 2B). Para aumentar aún más la relación S / N, se recomienda iluminar el cono de crecimiento sin reducir la luz mediante filtros de densidad neutra. - Adquirir imágenes de lapso de tiempo (Archivo suplementario 2). Guardar como una imagen de pila de lapso de tiempo multicanal (formato tiff).
6. Cuantificaciones de la velocidad de flujo de F-actina y la tasa de polimerización utilizando un software de procesamiento y análisis de imágenes Fiji
NOTA: Consulte el Archivo Suplementario 3 para obtener datos de práctica para cuantificar la velocidad del flujo de F-actina y la tasa de polimerización de actina.
- Abra la imagen de la pila de lapso de tiempo multicanal en Fiji.
- Seleccione Analizar > Establecer escala y establezca el tamaño de píxel de las imágenes.
- Encuentre una mota de actina que fluya retrógradamente durante un mínimo de cinco marcos de tiempo dentro de un haz de F-actina en filopodia o lamellipodia.
- Seleccione Imagen > Transformar > Rotar y ajuste el ángulo de las imágenes para que el haz de F-actina apunte hacia arriba (Figura 3A).
- Haga clic en Rectángulo en la barra de herramientas y dibuje un cuadro que incluya manchas de actina y la punta del paquete de actina F (Figura 3B, 1 y 2).
NOTA: Las señales de las manchas de halotag-actina son débiles. Además, las señales de Lifeact en las puntas distales del cono de crecimiento son débiles y débiles (Figura 2B) porque las puntas distales del cono de crecimiento son delgadas en comparación con la parte proximal. Los investigadores deben mejorar las señales para optimizar las cuantificaciones de la velocidad del flujo de F-actina y la tasa de polimerización (Figura 3B). Aunque la señal de Lifeact en el cono de crecimiento proximal se satura, esto no perturbará la determinación del extremo distal de las F-actinas (Figura 3E, línea amarilla). - Seleccione Imagen > Duplicar e introduzca los cinco marcos de tiempo (Figura 3B, 3-5).
- Seleccione Image > Stacks > Make Montage e introduzca los parámetros: Columns, 5; Filas, 1; Factor de escala, 1.
- Haga clic en Aceptar. El montaje de la imagen aparecerá en la pantalla.
- Seleccione Imagen > color > Dividir canales. Esto separa los dos canales fluorescentes.
- Cuantificación de la velocidad de flujo de F-actina.
- Seleccione Analizar > Establecer medidas y elija Área y rectángulo delimitador.
- Haga clic en Óvalo en la barra de herramientas y dibuje un círculo en una mancha de actina (Figura 3C, 1 y 2).
- Seleccione Imagen > superposición > Agregar selección. El círculo se superpone en el montaje de la imagen (Figura 3C, 3 y 4).
NOTA: El color de la superposición se puede cambiar en Editar > Opciones > Colores. - Repita la superposición de círculos para las manchas restantes (Figura 3C, 5).
NOTA: Para determinar con precisión los centros de las motas de actina, los investigadores deben superponer círculos en las manchas. - Haga clic en Recto en la barra de herramientas y dibuje una línea que vincule los centros de los círculos (Figura 3D, 1 y 2).
- Seleccione Analizar > medir. El resultado aparecerá en la pantalla (Figura 3D, 3).
NOTA: La altura mostrada en el resultado indica la distancia de translocación de la mancha de actina durante la observación (Figura 3D, 3). - Calcule la velocidad del flujo de F-actina dividiendo la distancia de translocación por el tiempo de observación.
- Cuantificación de la tasa de polimerización de F-actina.
- Dibuje una línea que vincule las puntas del fibrado de F-actina (Figura 3E, 1).
- Seleccione Analizar > medir. El resultado aparecerá en la pantalla (Figura 3E, 2).
NOTA: La altura en el resultado indica la longitud de extensión del haz de F-actina durante la observación (Figura 3E, 2). - Calcule la tasa de extensión de F-actina dividiendo la longitud de la extensión por el tiempo de observación.
- Calcule la tasa de polimerización de F-actina como la suma de la velocidad de flujo de F-actina y la tasa de extensión (Figura 3F).
7. Preparación de un gel PAA para microscopía de fuerza de tracción y cultivos de neuronas
- Preparación de geles PAA necesarios para la microscopía de fuerza de tracción (Figura 4)
- Pipete 0,5 ml de NaOH (100 mM), disuelto en H2O destilado, en platos con fondo de vidrio de 27 mm de diámetro, e incube durante 15 min a temperatura ambiente (RT).
- Agregue 50 μL de 3-aminopropiltrimetoxisilano (APTMS) en la solución de NaOH en los platos y mezcle las soluciones mediante pipeteo suave, e incube durante 15 min en RT.
- Lave los platos con fondo de vidrio con H2O destilado y séquelos.
- Pipetear 0,5 ml de solución de glutaraldehído al 0,5%, disuelto en PBS, en el plato con fondo de vidrio tratado con APTMS, e incubar durante 30 min a RT.
- Lavar los platos con fondo de vidrio con H2O destilado y secar.
- Preparar una solución que contenga H2O destilado (412 μL), solución de acrilamida al 30% (p/v) (63 μL), solución de bisacrilamida al 2,5% (p/v) (6 μL) y solución de microesfera modificada con carboxilato (perla fluorescente) (20 μL).
NOTA: Vórtice la solución de perla fluorescente para disociar las perlas agregadas antes de agregarlas a la solución de acrilamida. - Desgasifica la solución en una cámara de vacío durante 30 min a RT.
- A esta solución, agregue 1 μL de persulfato de amonio (APS) al 10% (p/v), disuelto en H2O destilado, y 1 μL de N,N,N',N'-tetrametilendiamina (TEMED), y mezcle mediante pipeteo suave.
- Coloque una cubierta de vidrio (18 mm de diámetro) en la tapa de un tubo centrífugo de 15 ml.
- Agregue inmediatamente 25 μL de la solución a la cubierta y luego coloque suavemente un plato con fondo de vidrio invertido en el estuche. Deje que la acrilamida polimerice durante 1 h en RT.
- Aplique H2O destilado en la funda y quítelo del gel con una aguja de jeringa con la punta doblada.
- Retire el H2O y agregue sulfo-SANPAH (1 mM), disuelto en PBS, al gel.
- En condiciones estériles, exponga los geles a la luz ultravioleta durante 5 min. Lave el gel tres veces con PBS durante 15 minutos por lavado.
- Determinación de la rigidez del gel con un método de indentación de microesferas (Figura 5A)44.
- Transfiera una microesfera que se especifica para el diámetro y la densidad, al gel y luego colóquela en la etapa de muestra de un microscopio confocal de escaneo láser.
- Concéntrese en la superficie del gel y registre la posición z (Figura 5B).
- Del mismo modo, concéntrese en la parte inferior de la microesfera y registre la posición z (Figura 5C).
- Calcule la profundidad de la hendidura determinada por la diferencia entre las posiciones z de la superficie del gel y el fondo de la microesfera.
- Calcule el módulo E de Young del gel mediante:
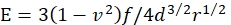
donde f es el peso corregido de flotabilidad de la microesfera, d es la profundidad de indentación del gel, r es el radio de la microesfera y v es la relación de Poisson (cuyo valor es 0,3, como se determinó anteriormente46).
- Cubra los geles con PDL y laminina como se describe en la sección 2.
- Como se describe en las secciones 3 y 4, diseccionar embriones de ratón E16, disociar hipocampos y transfectar con 5 μg de pEGFP-C1 por 1,0 x 106 células. Semilla 2.0 x 105 células en geles recubiertos de PDL/laminina.
NOTA: Para el análisis de la fuerza de tracción, las imágenes microscópicas de las células marcadas fluorescentemente son necesarias para determinar el área del cono de crecimiento.
8. Microscopía de fuerza de tracción en conos de crecimiento neuronal
- En DIV3, reemplace el medio de cultivo con 0.5 ml de medio L-15 calentado de Leibovitz que contiene un suplemento de B-27 al 2%, glutamina (1 mM), penicilina (100 U / ml) y estreptomicina (100 μg / ml). Mantener las neuronas durante 1 h a 37 °C.
- Encienda un microscopio confocal de barrido láser y ajuste la incubadora de la parte superior del escenario a 37 ° C.
NOTA: El presente estudio utiliza un microscopio confocal de barrido láser equipado con una lente de objetivo de inmersión en agua 63x/1.2 NA y un software de adquisición y análisis de imágenes (ver Tabla de Materiales). - Coloque las neuronas en el plato con fondo de vidrio en la incubadora de la parte superior del escenario precalentada.
- Establezca los parámetros de adquisición de imágenes de la siguiente manera: tamaño de escaneo, 512 × 512 píxeles; área de escaneo, zoom de 1.5-3x; velocidad de escaneo, ~1 s por fotograma; longitud de onda del láser, 561 nm (la longitud de onda de excitación para las perlas fluorescentes) y 488 nm (la longitud de onda de excitación para la proteína fluorescente verde mejorada (EGFP)); intervalo de tiempo, 3 s; duración, 50 fotogramas.
- Para visualizar la morfología del cono de crecimiento, seleccione un cono de crecimiento que exprese fuertemente EGFP.
- Concéntrese en la superficie del gel y adquiera imágenes de lapso de tiempo (Figura 6A y Archivo suplementario 4).
NOTA: Para el análisis de tracción, utilice dos canales fluorescentes (para perlas fluorescentes y EGFP) y un canal de campo brillante (se recomienda capturar imágenes de contraste de interferencia diferencial (DIC) o de contraste de fase). - Utilice un software de procesamiento de imágenes (por ejemplo, Fiji) para producir pilas de imágenes RGB de lapso de tiempo de un solo canal y guarde estas imágenes como archivos tiff.
NOTA: Es fundamental que los investigadores también guarden los datos sin procesar. - Aplique 100 μL de dodecil sulfato de sodio (SDS) al 10% (p/v), disuelto en H2O destilado, al plato con fondo de vidrio para relajar el sustrato del gel liberando neuronas del sustrato. Incubar el plato durante 5 min a 37°C para estabilizar la temperatura.
NOTA: La imagen de las perlas en un sustrato no tensado se utiliza para hacer referencia a las posiciones originales de las perlas durante el análisis de la fuerza de tracción (Figura 6C). La aplicación de la solución SDS altera las posiciones xyz debido al cambio térmico en la incubadora y la relajación de la deformación inducida por la célula. Los investigadores, por lo tanto, necesitan corregir el plano focal y la posición x-y. - Concéntrese en la superficie del gel y adquiera una imagen de las perlas en el sustrato sin tensión (Figura 6B).
- Produzca una imagen RGB de un solo canal de las perlas en el sustrato sin restricciones y guarde esta imagen como un archivo tiff.
- Corrección de la posición x-y de la imagen de la cuenta en el sustrato no tensado utilizando Fiji
- En Fiji, abra la imagen de la cuenta en el sustrato sin restricciones y la imagen de la pila de lapso de tiempo de las cuentas fluorescentes.
- Seleccione Pilas de > de imágenes > Herramientas > Concatenar. Aparecerá un cuadro de diálogo en la pantalla (Figura 7A, 1).
- Seleccione la imagen de perla en el sustrato sin tensión y la imagen de pila de lapso de tiempo de cuentas fluorescentes en Image1 e Image2, respectivamente (Figura 7A, 2).
- Haga clic en Aceptar. La imagen de perla en el sustrato no entrenado se agrega a la imagen de pila de lapso de tiempo (Figura 7A, 3).
- Usando la barra de desplazamiento (Figura 7B, 1, marco rojo), muestre el segundo marco de la imagen de la pila (flecha roja).
- Seleccione Plugins > StackReg (Figura 7B, 2). Aparecerá un cuadro de diálogo en la pantalla (Figura 7B, 3).
- Seleccione Cuerpo rígido en la lista desplegable (Figura 7B, 3, marco rojo) y haga clic en Aceptar. La corrección de la posición x-y comienza entonces.
- Utilice la barra de desplazamiento para mostrar el primer fotograma de la imagen de pila corregida por posición x-y.
- Seleccione Imagen > Duplicar (Figura 7C, 1). Aparecerá un cuadro de diálogo en la pantalla (Figura 7B, 2).
- Introduzca 1 en el rango, anule la selección de Duplicar pila (figura 7C, 2) y haga clic en Aceptar. La imagen de perla corregida por la posición x-y en el sustrato sin tensión aparecerá en la pantalla (Figura 7C, 3). Guarde esta imagen como un archivo tiff.
- Cuantificación de la fuerza de tracción
NOTA: El método descrito aquí para el análisis de la fuerza de tracción utiliza MATLAB y dos cajas de herramientas de MATLAB, 'Image Processing Toolbox' y 'Parallel Computing Toolbox'. Los investigadores deberán instalarlos antes del análisis. El código de análisis de la fuerza de tracción se desarrolló en base a la versión 2018a de MATLAB. Por lo tanto, se debe utilizar la versión 2018a (o posterior) de MATLAB para el análisis. Los algoritmos utilizados para el análisis de la fuerza de tracción fueron descritos anteriormente22.- Descargue el código de análisis de fuerza de tracción TFM2021 del Archivo Suplementario 5. Abrir TFM 2021 en MATLAB
- Abra main.m en TFM2021 y ejecútelo. Aparecerá una interfaz gráfica de usuario (GUI) en la pantalla (Figura 8A).
- Haga clic en Cargar imagen de sustrato sin tensión y seleccione la imagen de cuenta de posición corregida x-y en sustrato sin forzar.
- Haga clic en Cargar imágenes de cuentas fluorescentes y seleccione la imagen de pila de lapso de tiempo de cuentas.
- Haga clic en Cargar imágenes de campo brillante y seleccione la imagen de pila de lapso de tiempo de campo brillante.
- Haga clic en Cargar imágenes GFP y seleccione la imagen de pila de lapso de tiempo de EGFP.
- Seleccione GFP en la lista desplegable de la GUI (Figura 8A, cuadro rojo).
- Haga clic en ROI para especificar la región rectangular de interés (ROI), incluido el cono de crecimiento, haciendo clic en dos puntos en la imagen de celda que se muestra en la GUI (Figura 8B).
- Haga clic en el botón Guardar en la GUI (Figura 8A, flecha roja). Las imágenes de pila seleccionadas, junto con el ROI, se guardarán en un archivo .mat (archivo de formato MATLAB).
- Haga clic en Detectar spot. Aparecerá un cuadro de diálogo en la pantalla.
- Introduzca un valor (normalmente 50-150) en el cuadro de diálogo para determinar un umbral para la detección de cuentas. Al hacer clic en Aceptar , se inicia el cálculo.
- Después de finalizar el cálculo, haga clic en Trazar pista para ampliar la región seleccionada en el paso 8.12.8 y mostrar las cuentas detectadas como puntos blancos (Figura 8C, 1).
NOTA: Confirme si la detección de cuentas (puntos blancos) se superpone con las cuentas fluorescentes. La detección de cuentas también puede detectar ruido de fondo. Para reducir esta interferencia externa, cambie el valor umbral en Spot detect. Además, seleccione manualmente los puntos correctos en el paso 8.12.13. - Haga clic en Seleccionar cuentas y demarque una región poligonal que incluya los puntos correctos debajo del cono de crecimiento. Pulse Intro en el teclado. Los puntos blancos dentro de la región poligonal cambiarán a un color rojo (Figura 8C, 2).
- Haga clic en Estimar fuerza en la GUI. Luego, ingrese valores para los siguientes parámetros: tamaño de píxel, μm / píxel; Módulo de Young, el valor calculado en el paso 7.2.5; Ratio de Poisson, 0,3.
- Ejecutar fuerza de estimación para iniciar el cálculo. El software guardará los resultados del cálculo en un archivo de formato de hoja de cálculo automáticamente.
NOTA: La hoja de cálculo muestra el componente x, el componente y la magnitud de la fuerza en cada marco de tiempo (Figura 8D). Consulte el Archivo Suplementario 6 para obtener datos de práctica para cuantificar la fuerza de tracción.
Resultados
Imágenes de una sola mota para cuantificar la tasa de polimerización de actina y la eficiencia de acoplamiento del embrague
Una expresión de Lifeact alta permite la visualización de F-actina dentro del cono de crecimiento; una expresión baja de HaloTag-actina permite el monitoreo del flujo retrógrado de F-actina (Figura 3, Archivo Suplementario 2). El rastreo de las manchas de actina permite medir la velocidad del flujo de F-actina (Figura 3C, D). Dado que el acoplamiento mecánico del flujo retrógrado de F-actina y el sustrato adhesivo reduce la velocidad de flujo de F-actina, la eficiencia de acoplamiento del embrague se puede estimar a partir de la velocidad. Además, el etiquetado de F-actina con Lifeact ayuda a la visualización de la extensión de F-actina y es útil para cuantificar la tasa de polimerización de actina (Figura 3E, F).
Análisis cuantitativo de la fuerza de tracción
El estricto cumplimiento de la metodología presentada aquí revelará los movimientos de las perlas fluorescentes bajo el cono de crecimiento (Figura 6C, Archivo Suplementario 4). El flujo retrógrado de F-actina sobre el sustrato genera fuerza de tracción haciendo que las perlas fluorescentes se muevan hacia atrás debajo del cono de crecimiento. El código de análisis de fuerza de tracción estima la fuerza de tracción a partir del desplazamiento de perlas fluorescentes y expresa la fuerza de tracción calculada como vector de fuerza. La dirección y la magnitud de la fuerza de tracción se determinan a partir de las componentes x e y del vector de fuerza (Figura 8C, D). El cuadro rojo de la Figura 8D representa las componentes x e y de un vector de fuerza; La Figura 8C muestra el cono de crecimiento correspondiente. En términos de coordenadas x-y, el vector apunta a -93.8° contra el eje x; esta orientación se dirige hacia la parte posterior del cono de crecimiento (Figura 8C). La magnitud de la fuerza de tracción F se calculó de la siguiente manera:
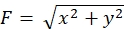
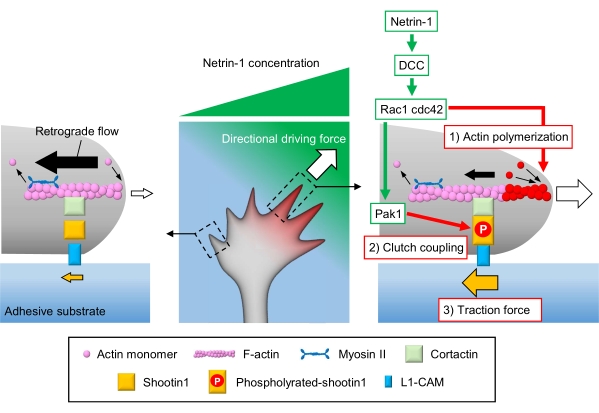
Figura 1: Una maquinaria de cono de crecimiento para la generación de fuerza y la navegación de conos de crecimiento. Un gradiente quimioatrayente netrin-1 induce la estimulación asimétrica de su receptor DCC en un cono de crecimiento axonal. Esto activa Rac1 y Cdc42, y su quinasa aguas abajo Pak1. Rac1 y Cdc42 promueven (1) la polimerización de actina, mientras que Pak1 fosforila shootin1, mejorando el acoplamiento de embrague mediado por shootin1 (2). La activación asimétrica de la dinámica de la actina y el acoplamiento del embrague dentro del cono de crecimiento aumenta (3) la fuerza de tracción en el lado de la fuente netrin-1, generando así una fuerza motriz direccional para la atracción del cono de crecimiento. Los protocolos presentados aquí permiten la cuantificación de las variables clave (1)-(3) para la navegación del cono de crecimiento. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Imágenes de fluorescencia de un cono de crecimiento neuronal debajo de un diafragma completamente abierto y estrecho. El cono de crecimiento expresa Lifeact y HaloTag-actina. (A) Una alta expresión de Lifeact permite la visualización de la morfología del cono de crecimiento. Por otro lado, los niveles de expresión de HaloTag-actina son muy bajos, con señales tenues cuando el diafragma está completamente abierto. (B) Cuando el diafragma se estrecha adecuadamente, las señales de fondo disminuyen y aparecen manchas de actina única en el cono de crecimiento. Barras de escala: 5 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Pasos para cuantificar la velocidad del flujo de F-actina y la tasa de polimerización de actina utilizando un software de procesamiento y análisis de imágenes Fiji. (A) Ajuste el ángulo de la imagen para el análisis. (1) Busque y seleccione una mancha de actina (punta de flecha) que fluya en un haz de actina F. (2) Seleccione Imagen > Transformar > Rotar. (3) Establezca el ángulo de modo que el fibrado de actina F se dirija hacia arriba. (4) Se cambiará el ángulo de la imagen. B) Demarcar la región, incluidas las motas de actina y el haz de actinas F. (1) Haga clic en Rectángulo en la barra de herramientas. (2) Delinear una región en la imagen. El brillo y el contraste de la imagen se incrementan para permitir una visualización clara de la mancha de actina y la punta del haz de F-actina. (3) Seleccione Imagen > Duplicar. (4) Ingrese los cinco marcos de tiempo que muestran el flujo de motas de actina en el fibrado de actina F. (5) La imagen de pila seleccionada aparecerá en la pantalla. (C) Círculos superpuestos en la mota de actina. (1) Haga clic en Óvalo en la barra de herramientas. (2) Dibuja un círculo en una mota de actina. (3) Seleccione Imagen > superposición > Agregar selección. (4) El círculo está superpuesto. 5) Repita la superposición de los círculos en las manchas de actina restantes. (D) Medir la distancia de translocación de las motas de actina durante los cinco marcos de tiempo. (1) Haga clic en Recto en la barra de herramientas. (2) Dibuja una línea que vincule los centros de los círculos. (3) Seleccione Analizar > medir. El resultado, indicado por el parámetro Altura (cuadro rojo), retransmite la distancia de translocación de la mancha de actina. (E) Medir el cambio en la longitud de la protuberancia de la F-actina durante los cinco marcos de tiempo. (1) Dibuje una línea que vincule las puntas de la protuberancia de la F-actina. (2) Seleccione Analizar > medir. El resultado (cuadro rojo), Altura, indica la longitud de extensión del haz de F-actina. (F) La tasa de polimerización de actina se calcula a partir de la suma de la velocidad de flujo de F-actina y la tasa de extensión. Véase también el Expediente Complementario 2. El Archivo Suplementario 3 ayuda a los investigadores a practicar la metodología descrita anteriormente. Haga clic aquí para ver una versión más grande de esta figura.
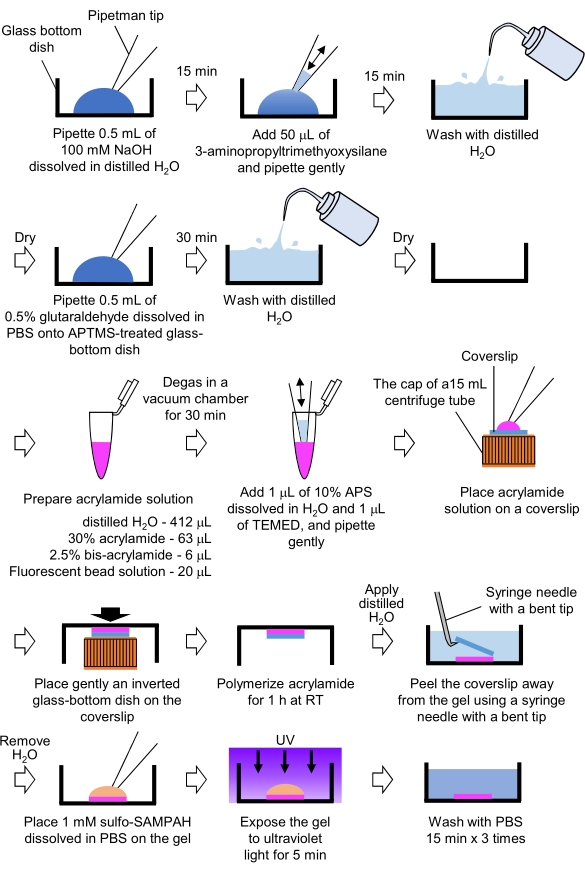
Figura 4: Pasos para la preparación del gel PAA. Consulte el paso 7.1 para obtener una descripción detallada. Haga clic aquí para ver una versión más grande de esta figura.
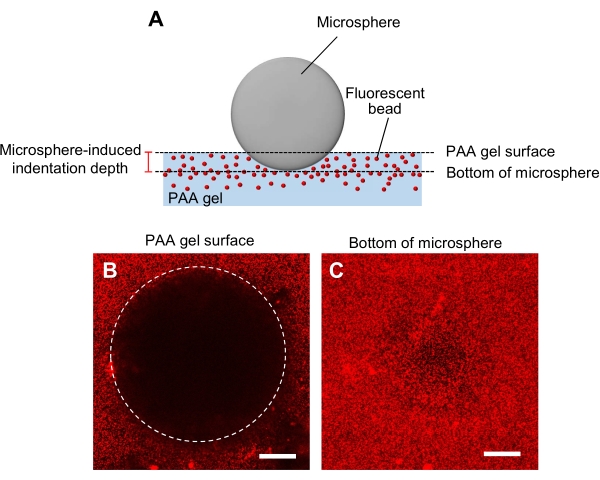
Figura 5: Determinación de la rigidez del gel PAA. (A) Un método de indentación de microesferas. Cuando se coloca una microesfera sobre un gel PAA incrustado en perla fluorescente, el peso de la microesfera causa una hendidura en el gel. La profundidad de indentación se calcula restando las posiciones z de la superficie del gel PAA del fondo de las imágenes de fluorescencia de la microesfera (B, C) de un gel PAA sangrado por una microesfera y que contiene perlas fluorescentes. Se utilizó un microscopio confocal de barrido láser para capturar imágenes de la superficie del gel (B) y el fondo de la microesfera (C). Las señales de las perlas fluorescentes no son visibles en la superficie del gel en la región con sangría (B, círculo). Sin embargo, se pueden observar en el fondo de la microesfera. Barras de escala: 50 μm. Haga clic aquí para ver una versión más grande de esta figura.
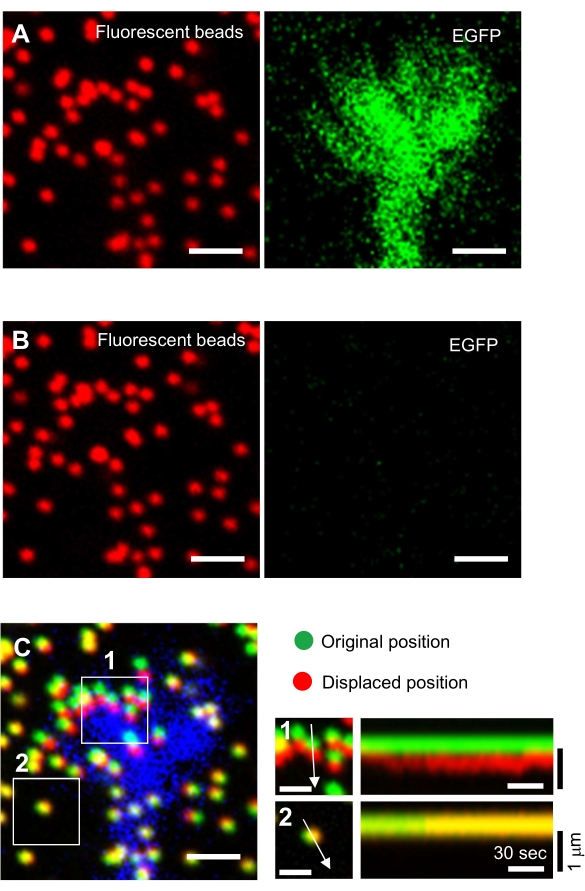
Figura 6: Mapeo de fuerza de un cono de crecimiento neuronal. (A-C) Imágenes de fluorescencia de perlas incrustadas en un gel PAA y un cono de crecimiento neuronal visualizado con EGFP. Después de la adquisición de imágenes de lapso de tiempo (A), la neurona se liberó del sustrato del gel mediante la aplicación de solución SDS, y se capturó una imagen de las perlas en el sustrato no entrenado (B). (C) La imagen de las cuentas en el sustrato no tensado muestra las cuentas en sus posiciones originales (verde) y desplazadas (rojo). La señal EGFP del cono de crecimiento se muestra en azul. Los kymographs (derecha) muestran los movimientos de las cuentas a intervalos de 3 s durante una duración de 147 s, indicados por las flechas en las áreas en caja 1 y 2. La cuenta en el área 2 es una cuenta de referencia. Barras de escala: 2 μm para (A), (B) y (C, izquierda), y 1 μm para (C, centro). Véase también el Expediente Suplementario 4. Haga clic aquí para ver una versión más grande de esta figura.
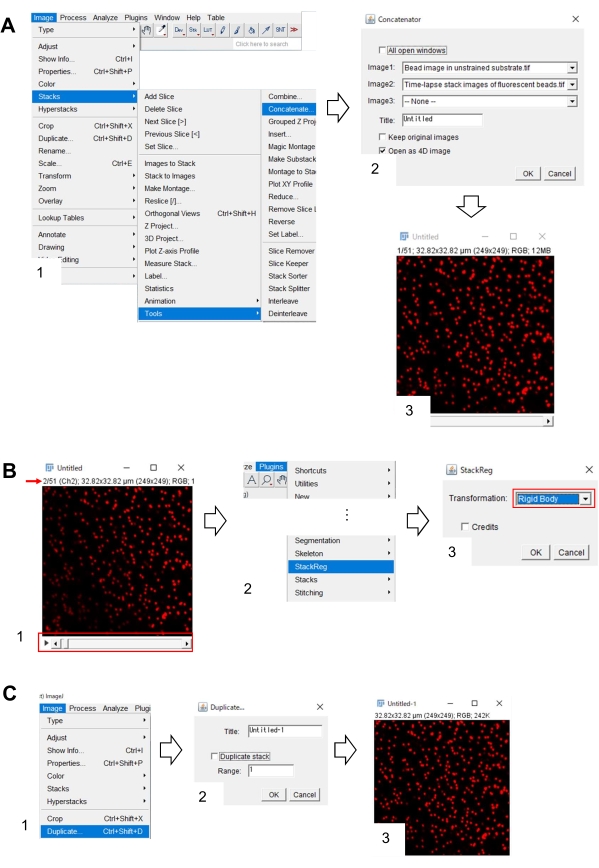
Figura 7: Pasos para corregir la posición x-y de la imagen de la cuenta en sustrato no entrenado usando Fiji. (A) Concatenar la imagen de la cuenta en sustrato no forzado con la imagen de pila de lapso de tiempo de cuentas fluorescentes. (1) Seleccione Pilas de > de imágenes > herramientas > Concatenar. (2) Seleccione la imagen de cuentas en sustrato no forzado y la imagen de pila de lapso de tiempo de cuentas fluorescentes en Image1 e Image2, respectivamente. Haga clic en Aceptar. (3) La imagen de perla en el sustrato no entrenado se agrega a la imagen de pila de lapso de tiempo. (B) Corrija la posición x-y de la imagen de la cuenta fluorescente. (1) Utilice la barra de desplazamiento (marco rojo) para seleccionar el segundo fotograma (flecha roja) en la pila de imágenes. (2) Seleccione Plugins > StackReg. (3) Seleccione Cuerpo rígido en la lista desplegable (marco rojo) y haga clic en Aceptar. Comenzará la corrección de la posición x-y. (C) Guarde la imagen de cuenta corregida por la posición x-y. Después de seleccionar el primer fotograma de la imagen de pila corregida por posición x-y, (1) seleccione Imagen > Duplicar. (2) ingrese 1 en el Rango y anule la selección de Duplicar pila. Luego haga clic en Aceptar. (3) la imagen de la cuenta corregida por la posición x-y en el sustrato sin tensión aparecerá en la pantalla. Guarde esta imagen como un archivo tiff. El Archivo Suplementario 6 ayuda a los investigadores a practicar la metodología descrita anteriormente. Haga clic aquí para ver una versión más grande de esta figura.
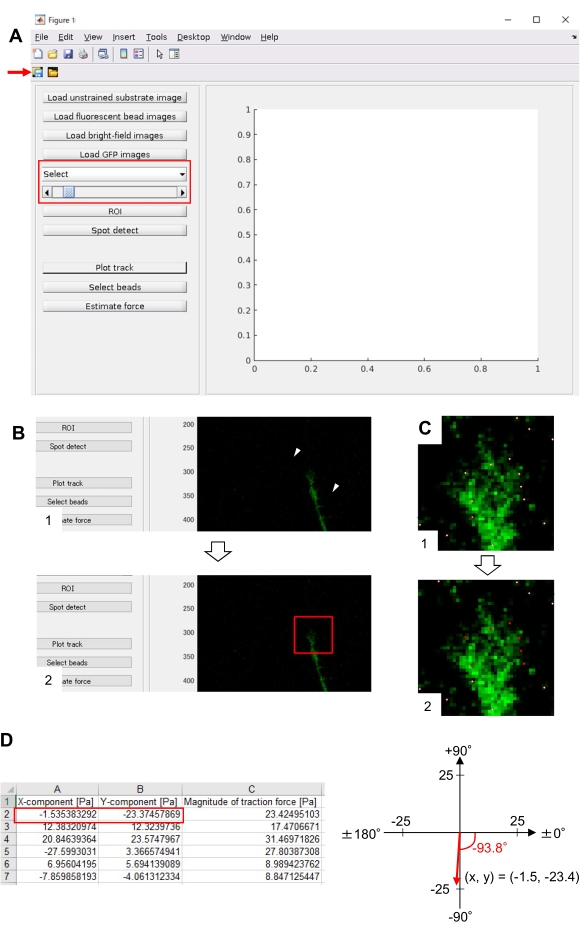
Figura 8: Análisis de la fuerza de tracción bajo un cono de crecimiento neuronal utilizando un código de análisis de fuerza de tracción de código abierto. (A) GUI para analizar la fuerza de tracción. Las imágenes de lapso de tiempo seleccionadas en la GUI se pueden confirmar desde la lista desplegable y con el control deslizante indicado (cuadro rojo). (B) Seleccione una región que incluya el cono de crecimiento. (1) Haga clic en ROI en la GUI. Con el cursor del ratón, especifique dos puntos (puntas de flecha) en la imagen de celda. (2) Aparecerá un cuadro rojo en la imagen de la celda. Dos clics determinan las ubicaciones de dos esquinas. (C) Seleccione las cuentas detectadas (puntos blancos) debajo del cono de crecimiento. (1) En la GUI, haga clic en Seleccionar cuentas y demarque una región poligonal que incluya el cono de crecimiento haciendo clic. Presione la tecla Intro . (2) Los puntos blancos dentro de la región poligonal cambiarán a un color rojo. (D) Resultados calculados de la dirección y magnitud de la fuerza de tracción. El cuadro rojo de la hoja de cálculo representa los componentes x e y del vector de fuerza, estimados mediante análisis de fuerza de tracción. En la coordenada x-y en el panel derecho, el vector de fuerza generado por el cono de crecimiento apunta a -93.8° contra el eje x; esta orientación se dirige hacia la parte posterior del cono de crecimiento en (C). El Archivo Suplementario 6 ayuda a los investigadores a practicar la metodología descrita anteriormente. Haga clic aquí para ver una versión más grande de esta figura.
Archivo suplementario 1: Recetas de soluciones y medios utilizados en este estudio. Consulte el texto para un uso detallado. Haga clic aquí para descargar este archivo.
Archivo suplementario 2: Imágenes de fluorescencia de Lifeact e imágenes de manchas fluorescentes de HaloTag-actina en un cono de crecimiento nervioso. Lifeact (verde) y HaloTag-actina (magenta). Las imágenes se adquirieron cada 3 s para una duración total de 147 s. Barra de escala: 2 μm. Consulte también la Figura 3. Haga clic aquí para descargar este archivo.
Archivo suplementario 3: Datos de práctica para cuantificar la velocidad del flujo de F-actina y la tasa de polimerización de actina. Una imagen de pila de lapso de tiempo multicanal de Lifeact (verde) y HaloTag-actina (magenta). Véase también la Figura 3. Haga clic aquí para descargar este archivo.
Archivo suplementario 4: Un video de mapeo de fuerza que detecta la fuerza de tracción en un cono de crecimiento neuronal. Las posiciones originales (verde) y desplazadas (rojo) de las cuentas. La señal EGFP en el cono de crecimiento se muestra en azul. Las imágenes se adquirieron cada 3 s durante 147 s. Barra de escala: 2 μm. Véase también la Figura 6. Haga clic aquí para descargar este archivo.
Expediente suplementario 5: Código de análisis de la fuerza de tracción. Consulte el paso 8.12 para obtener un uso detallado. Haga clic aquí para descargar este archivo.
Expediente suplementario 6: Datos de práctica para cuantificar la fuerza de tracción. Una imagen RGB de un solo canal de las perlas en sustrato sin tensión e imágenes RGB de pila de lapso de tiempo de un solo canal de cuentas fluorescentes bajo un cono de crecimiento, EGFP y campo brillante. Véanse también las figuras 7 y 8. Haga clic aquí para descargar este archivo.
Discusión
Los protocolos descritos en este estudio utilizan materiales disponibles comercialmente y equipos de microscopía que se encuentran rutinariamente en todos los laboratorios, institutos y universidades. Por lo tanto, los investigadores pueden adoptar fácilmente la actual microscopía de fuerza de tracción y imágenes de una sola mancha en sus estudios.
Las imágenes moteadas pueden analizar la polimerización de actina y el acoplamiento del embrague. Además, las imágenes moteadas pueden monitorear el flujo retrógrado de moléculas de embrague como shootin1 y cortactina, que interactúan con el flujo retrógrado de F-actina. Mediante el uso de un microscopio TIRF, también se puede monitorizar el flujo retrógrado de la molécula de adhesión celular L1-CAM23,41; L1-CAM se somete a comportamientos de agarre y deslizamiento que reflejan la eficiencia del acoplamiento del embrague23,41. Aunque el presente estudio emplea el sistema TMR-HaloTag para la obtención de imágenes moteadas, otras proteínas fluorescentes, como la EGFP y la proteína fluorescente roja monomérica, también están disponibles en el análisis16,18,20,23,24,27,39. Los elementos esenciales para visualizar las manchas de actina son un bajo nivel de expresión de actina fluorescente y la iluminación de un área mínima (Figura 2). En este protocolo, las señales Lifeact y HaloTag-actina se adquieren secuencialmente. Debido a que el flujo retrógrado de actina es relativamente lento (4,5 ± 0,1 μm/min)24, el análisis del flujo retrógrado de F-actina y la polimerización de actina no se ven afectados por la adquisición secuencial de imágenes de diferentes canales fluorescentes (intervalo de ~1 s). Lifeact es un marcador de actina F ampliamente utilizado, pero puede competir con las proteínas de unión a actina47. De mayor importancia, Lifeact puede alterar la dinámica de la actina, afectando así a las estructuras de la F-actina y a la morfología celular47,48,49.
La microscopía de fuerza de tracción puede detectar fuerzas para impulsar el avance del cono de crecimiento. Al montar las neuronas dentro de una matriz extracelular, los investigadores también pueden analizar las fuerzas generadas en un entorno semi-3D11. Las imágenes de alto aumento son importantes para la cuantificación precisa de la fuerza de tracción porque los conos de crecimiento generan fuerzas de tracción débiles7. Aunque también se utilizan otros métodos con nanopilares o biosensores sensibles al estrés para medir la fuerza de tracción50,51, el método basado en gel PAA es altamente adaptable y permite ajustar la rigidez del sustrato variando las concentraciones de acrilamida y bisacrilamida41,44,52. En este protocolo, el gel PAA se prepara a una concentración final de 3,75% de acrilamida y 0,03% de bis-acrilamida; El módulo de Young es ~270 Pa22 y esta rigidez está dentro del rango del tejido cerebral (100-10,000 Pa)53,54,55. Debido al grosor del gel PAA (~100 μm), este método limita el uso de lentes de alto aumento durante la microscopía. Para obtener imágenes de gran aumento, los investigadores deben usar la función de zoom en un microscopio confocal de barrido láser.
En conclusión, la actual imagen de motas y la microscopía de fuerza de tracción permiten análisis cuantitativos de los eventos clave en las generaciones de fuerza. Esta información será invaluable para mejorar la comprensión de los mecanismos que subyacen al avance y la navegación del cono de crecimiento.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Esta investigación fue apoyada en parte por AMED bajo el número de subvención 21gm0810011h0005 (N.I. y Y.S.), JSPS KAKENHI (JP19H03223, N.I.) y JSPS Grants-in-Aid for Early-Career Scientists (JP19K16258, T.M.), la Osaka Medical Research Foundation for Incurable Diseases (T.M.), y NAIST Next Generation Interdisciplinary Research Project (Y.S.).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.5% trypan blue stain solution | Nacalai | 29853-34 | |
| 3-aminopropyltrimethyoxysilane | Sigma | 281778-100ML | |
| Acrylamide monomer | Nacalai | 00809-85 | |
| Ammonium persulphate | Cytiva | 17-1311-1 | |
| Axio Observer Z1 | Zeiss | 431007-9902-000 | Epi-fluorescence microscope (single speckle imaging) |
| B-27 supplement (50x) | Thermo Fisher Scientific | 17504-044 | |
| Bovine serum albmine | Sigma | A7906-10G | |
| C-Apochromat 63x/1.2 W Corr | Zeiss | 421787-9970-799 | Objective lens (traction force microscopy) |
| Coverslip (diameter 18 mm) | Matsunami | C018001 | |
| D-glucose | Nacalai | 16806-25 | |
| DNaseI | Sigma | DN25-100MG | |
| Fetal bovine serum | Thermo Fisher Scientific | 10270-106 | |
| Fiji | Open source software package | https://imagej.net/software/fiji/ | |
| FluoSpheres carboxylate-modified 0.2 mm, red (580/605), 2% solid | Thermo Fisher Scientific | F8810 | carboxylate-modified microspheres |
| Glass bottom dish (14 mm diameter) | Matsunami | D1130H | |
| Glass bottom dish (27 mm diameter) | Matsunami | D1140H | |
| Glutaraldehyde solution | Sigma | G5882-10X10ML | |
| HaloTag TMR ligand | Promega | G8251 | |
| HBO103 W/2 | Osram | 4050300382128 | Mercury lamp (single speckle imaging) |
| Image Processing Toolbox | MathWork | https://www.mathworks.com/products/image.html | |
| Laminin solution from mouse EHS tumor | Wako | 120-05751 | |
| Leibovitz’s L-15 medium | Thermo Fisher Scientific | 11415064 | |
| L-glutamine | Nacalai | 16919-42 | |
| LSM710 | Zeiss | N/A | Conforcal laser microscope (traction force microscopy) |
| MATLAB2018a | MathWork | https://www.mathworks.com/products/new_products/release2018a.html | |
| Mouse C57BL/6 | Japan SLC | N/A | |
| Mouse neuron nucleofector kit | Lonza | VPG-1001 | |
| N,N,N’,N’-tetramethylethylenediamine (TEMED) | Nacalai | 33401-72 | |
| N,N’-methylenebisacrylamide | Nacalai | 22402-02 | |
| Neurobasal medium | Thermo Fisher Scientific | 21103-049 | |
| Nucleofector I | Amaxa | AAD-1001 | Electroporation apparatus |
| ORCA Flash 4.0 V2 | Hamamatsu | C11440-22CU | CMOS camera (single speckle imaging) |
| Papain | Nacalai | 26036-34 | |
| Parallel Computing Toolbox | MathWork | https://www.mathworks.com/products/parallel-computing.html | |
| pEGFP-C1 | Clontech | 1528177 | |
| Penicillin-streptomycin (100x) | Nacalai Tesque | 26253-84 | |
| pFN21A-HaloTag-actin | (Minegishi et al., 2018) | N/A | |
| Phosphate buffered saline (PBS) pH 7.4 (10x) | Thermo Fisher Scientific | 70011-044 | |
| Plan-Apochromat 100x/1.4 Oil | Zeiss | 420790-9901-000 | Objective lens (single speckle imaging) |
| pmNeonGreen-N1-Lifeact | (Kastian et al., 2021) | N/A | |
| Poly-D-lysine hydrobromide | Sigma | P6407-5MG | |
| Slulfo-SAMPHA | Thermo Fisher Scientific | 22589 | |
| Sodium dodecyl sulfate | Nacalai | 08933-05 | |
| Sodium hydrate (NaOH) | Nacalai | 31511-05 | |
| Steel ball | Sako tekkou | N/A | Microshpere to determin PAA gel rigidity. 0.6 mm diameter, 7.87 g/cm3. |
| ZEN2009 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (traction force microscopy) |
| ZEN2012 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (single speckle imaging) |
Referencias
- TessierLavigne, M., Goodman, C. S. The molecular biology of axon guidance. Science. 274, 1123-1133 (1996).
- Marin, O., Valiente, M., Ge, X., Tsai, L. H. Guiding neuronal cell migrations. Cold Spring Harbor Perspectives in Biology. 2, 001834 (2010).
- Vitriol, E. A., Zheng, J. Q. Growth cone travel in space and time: the cellular ensemble of cytoskeleton, adhesion, and membrane. Neuron. 73 (6), 1068-1081 (2012).
- Cooper, J. A. Cell biology in neuroscience: mechanisms of cell migration in the nervous system. Journal of Cell Biology. 202, 725-734 (2013).
- Dupraz, S., et al. RhoA controls axon extension independent of specification in the developing brain. Current Biology. 29, 3874-3886 (2019).
- Suter, D. M., Miller, K. E. The emerging role of forces in axonal elongation. Progress in Neurobiology. 94, 91-101 (2011).
- Franze, K. Integrating chemistry and mechanics: the forces driving axon growth. Annual Review of Cell and Developmental Biology. 36, 61-83 (2020).
- Mitchison, T., Kirschner, M. Cytoskeletal dynamics and nerve growth. Neuron. 1, 761-772 (1988).
- Suter, D. M., Forscher, P. Substrate-cytoskeletal coupling as a mechanism for the regulation of growth cone motility and guidance. Journal of Neurobiology. 44, 97-113 (2000).
- Lowery, L. A., Van Vactor, D. The trip of the tip: understanding the growth cone machinery. Nature Reviews Molecular Cell Biology. 10, 332-343 (2009).
- Minegishi, T., et al. Shootin1b mediates a mechanical clutch to produce force for neuronal migration. Cell Reports. 25, 624-639 (2018).
- Minegishi, T., Inagaki, N. Forces to drive neuronal migration steps. Frontiers in Cell and Developmental Biology. 8, 863 (2020).
- Forscher, P., Smith, S. J. Actions of cytochalasins on the organization of actin filaments and microtubules in a neuronal growth cone. Journal of Cell Biology. 107, 1505-1516 (1988).
- Suter, D. M., Forscher, P. An emerging link between cytoskeletal dynamics and cell adhesion molecules in growth cone guidance. Current Opinion in Neurobiology. 8, 106-116 (1998).
- Pollard, T. D., Borisy, G. G. Cellular motility driven by assembly and disassembly of actin filaments. Cell. 112, 453-465 (2003).
- Waterman-Storer, C. M., Desai, A., Bulinski, J. C., Salmon, E. D. Fluorescent speckle microscopy, a method to visualize the dynamics of protein assemblies in living cells. Current Biology. 8, 1227-1230 (1998).
- Katoh, K., Hammar, K., Smith, P. J., Oldenbourg, R. Birefringence imaging directly reveals architectural dynamics of filamentous actin in living growth cones. Molecular Biology of the Cell. 10, 197-210 (1999).
- Watanabe, N., Mitchison, T. J. Single-molecule speckle analysis of actin filament turnover in lamellipodia. Science. 295, 1083-1086 (2002).
- Medeiros, N. A., Burnette, D. T., Forscher, P. Myosin II functions in actin-bundle turnover in neuronal growth cones. Nature Cell Biology. 8, 215-226 (2006).
- Shimada, T., et al. Shootin1 interacts with actin retrograde flow and L1-CAM to promote axon outgrowth. Journal of Cell Biology. 181, 817-829 (2008).
- He, M., Zhang, Z. H., Guan, C. B., Xia, D., Yuan, X. B. Leading tip drives soma translocation via forward F-actin flow during neuronal migration. Jounal of Neuroscience. 30, 10885-10898 (2010).
- Toriyama, M., Kozawa, S., Sakumura, Y., Inagaki, N. Conversion of a signal into forces for axon outgrowth through Pak1-mediated shootin1 phosphorylation. Current Biology. 23, 529-534 (2013).
- Abe, K., et al. Grip and slip of L1-CAM on adhesive substrates direct growth cone haptotaxis. Proceedings of the National Academy of Sciences of the United States of America. 115, 2764-2769 (2018).
- Baba, K., et al. Gradient-reading and mechano-effector machinery for netrin-1-induced axon guidance. Elife. 7, 34593 (2018).
- Nichol, R. I., Hagen, K. M., Lumbard, D. C., Dent, E. W., Gomez, T. M. Guidance of axons by local coupling of retrograde flow to point contact adhesions. Journal of Neuroscience. 36, 2267-2282 (2016).
- Shekarabi, M., et al. Deleted in colorectal cancer binding netrin-1 mediates cell substrate adhesion and recruits Cdc42, Rac1, Pak1, and N-WASP into an intracellular signaling complex that promotes growth cone expansion. Journal of Neuroscience. 25, 3132-3141 (2005).
- Kubo, Y., et al. Shootin1-cortactin interaction mediates signal-force transduction for axon outgrowth. Journal of Cell Biology. 210, 663-676 (2015).
- Huber, A. B., Kolodkin, A. L., Ginty, D. D., Cloutier, J. F. Signaling at the growth cone: ligand-receptor complexes and the control of axon growth and guidance. Annual Review of Neuroscience. 26, 509-563 (2003).
- Kolodkin, A. L., Tessier-Lavigne, M. Mechanisms and molecules of neuronal wiring: a primer. Cold Spring Harbor Perspectives in Biology. 3, 001727 (2011).
- Ayala, R., Shu, T., Tsai, L. H. Trekking across the brain: the journey of neuronal migration. Cell. 128, 29-43 (2007).
- Tatavarty, V., Kim, E. J., Rodionov, V., Yu, J. Investigating sub-spine actin dynamics in rat hippocampal neurons with super-resolution optical imaging. PLoS One. 4, 7724 (2009).
- Frost, N. A., Shroff, H., Kong, H., Betzig, E., Blanpied, T. A. Single-molecule discrimination of discrete perisynaptic and distributed sites of actin filament assembly within dendritic spines. Neuron. 67, 86-99 (2010).
- Chazeau, A., et al. Nanoscale segregation of actin nucleation and elongation factors determines dendritic spine protrusion. EMBO Journal. 33, 2745-2764 (2014).
- Garcia, M., et al. Two-tiered coupling between flowing actin and immobilized N-cadherin/catenin complexes in neuronal growth cones. Proceedings of the National Academy of Sciences of the United States of America. 112, 6997-7002 (2015).
- Swaminathan, V., et al. Actin retrograde flow actively aligns and orients ligand-engaged integrins in focal adhesions. Proceedings of the National Academy of Sciences of the United States of America. 114, 10648-10653 (2017).
- Tsai, T. Y., et al. Efficient front-rear coupling in neutrophil chemotaxis by dynamic myosin II localization. Developmental Cell. 49, 189-205 (2019).
- Zhang, X. F., et al. Regulation of axon growth by myosin II-dependent mechanocatalysis of cofilin activity. Journal of Cell Biology. 218 (7), 2329-2349 (2019).
- Reversat, A., et al. Cellular locomotion using environmental topography. Nature. 582, 582-585 (2020).
- Katsuno, H., et al. Actin migration driven by directional assembly and disassembly of membrane-anchored actin filaments. Cell Reports. 12, 648-660 (2015).
- Urasaki, A., et al. Shootins mediate collective cell migration and organogenesis of the zebrafish posterior lateral line system. Scientific Reports. 9, 12156 (2019).
- Abe, K., et al. Mechanosensitive axon outgrowth mediated by L1-laminin clutch interface. Biophysical Journal. 120, 3566-3576 (2021).
- Kastian, R. F., et al. Shootin1a-mediated actin-adhesion coupling generates force to trigger structural plasticity of dendritic spines. Cell Reports. 35, 109130 (2021).
- Riedl, J., et al. Lifeact: a versatile marker to visualize F-actin. Nature Methods. 5, 605-607 (2008).
- Chan, C. E., Odde, D. J. Traction dynamics of filopodia on compliant substrates. Science. 322, 1687-1691 (2008).
- Wang, Y. L., Pelham, R. J. Preparation of a flexible, porous polyacrylamide substrate for mechanical studies of cultured cells. Methods in Enzymology. 298, 489-496 (1998).
- Li, Y., Hu, Z., Li, C. New method for measuring poisson's ratio in polymer gels. Journal of Applied Polymer Science. 50, 1107-1111 (1993).
- Belyy, A., Merino, F., Sitsel, O., Raunser, S. Structure of the Lifeact-F-actin complex. PLoS Biology. 18, 3000925 (2020).
- Flores, L. R., Keeling, M. C., Zhang, X., Sliogeryte, K., Gavara, N. Lifeact-GFP alters F-actin organization, cellular morphology and biophysical behaviour. Scientific Reports. 9, 3241 (2019).
- Kumari, A., Kesarwani, S., Javoor, M. G., Vinothkumar, K. R., Sirajuddin, M. Structural insights into actin filament recognition by commonly used cellular actin markers. EMBO Journal. 39, 104006 (2020).
- du Roure, O., et al. Force mapping in epithelial cell migration. Proceedings of the National Academy of Sciences of the United States of America. 102, 2390-2395 (2005).
- Grashoff, C., et al. Measuring mechanical tension across vinculin reveals regulation of focal adhesion dynamics. Nature. 466, 263-266 (2010).
- Koch, D., Rosoff, W. J., Jiang, J., Geller, H. M., Urbach, J. S. Strength in the periphery: growth cone biomechanics and substrate rigidity response in peripheral and central nervous system neurons. Biophysical Journal. 102 (3), 452-460 (2012).
- Barnes, J. M., Przybyla, L., Weaver, V. M. Tissue mechanics regulate brain development, homeostasis and disease. Journal of Cell Science. 130, 71-82 (2017).
- Moore, S. W., Roca-Cusachs, P., Sheetz, M. P. Stretchy proteins on stretchy substrates: the important elements of integrin-mediated rigidity sensing. Developmental Cell. 19, 194-206 (2010).
- Tyler, W. J. The mechanobiology of brain function. Nature Reviews Neuroscience. 13, 867-878 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados