Method Article
Analysen von Aktindynamik, Kupplungskupplung und Zugkraft für den Wachstumskegelvorschub
In diesem Artikel
Zusammenfassung
Um voranzukommen, müssen Wachstumskegel Zugkräfte gegen die äußere Umgebung ausüben. Die Erzeugung von Zugkräften ist abhängig von der Aktindynamik und der Kupplungskupplung. Die vorliegende Studie beschreibt Methoden zur Analyse der Aktindynamik, der Kupplungskupplung und der Zugkräfte für den Wachstumskegelvorschub.
Zusammenfassung
Um funktionelle Netzwerke aufzubauen, müssen Neuronen zu ihren entsprechenden Zielen wandern und dann Axone in Richtung ihrer Zielzellen ausdehnen. Diese Prozesse hängen von den Fortschritten der Wachstumskegel ab, die sich an den Spitzen der Neuriten befinden. Axonale Wachstumskegel erzeugen treibende Kräfte, indem sie ihre lokale Mikroumgebung wahrnehmen und die Zytoskelettdynamik und Aktinadhäsionskopplung (Kupplungskopplung) modulieren. Jahrzehntelange Forschung hat zur Identifizierung von Leitmolekülen, ihren Rezeptoren und nachgeschalteten Signalkaskaden zur Regulierung der neuronalen Migration und der axonalen Führung geführt; Die molekularen Maschinen, die zur Erzeugung von Kräften erforderlich sind, um den Wachstumskegelvorschub und die Navigation voranzutreiben, werden jedoch gerade erst aufgeklärt. An der Vorderkante neuronaler Wachstumskegel durchlaufen Aktinfilamente einen retrograden Fluss, der durch Aktinpolymerisation und Actomyosinkontraktion angetrieben wird. Eine Kupplungskopplung zwischen F-Aktin retrogradem Flow und Klebesubstrat erzeugt Zugkräfte für den Wachstumskegelvorschub. Die vorliegende Studie beschreibt ein detailliertes Protokoll zur Überwachung des retrograden F-Aktinflusses durch Single-Speckle-Bildgebung. Wichtig ist, dass diese Technik in Kombination mit einem F-Aktin-Marker Lifeact 1) die F-Aktin-Polymerisationsrate und 2) die Kupplungskopplungseffizienz zwischen dem retrograden F-Aktin-Fluss und dem Klebstoffsubstrat quantifizieren kann. Beides sind kritische Variablen für die Erzeugung von Kräften für den Wachstumskegelvorschub und die Navigation. Darüber hinaus beschreibt die vorliegende Studie ein detailliertes Protokoll der Zugkraftmikroskopie, das 3) die von Wachstumskegeln erzeugte Zugkraft quantifizieren kann. Durch die Kopplung der Analysen der Einzelfleckenbildgebung und der Zugkraftmikroskopie können die Forscher die molekulare Mechanik überwachen, die dem Wachstumskegelfortschritt und der Navigation zugrunde liegt.
Einleitung
Im sich entwickelnden Wirbeltiergehirn durchlaufen Neuronen aufwendig organisierte Migrationen und projizieren Axone in Richtung geeigneter synaptischer Partner, um funktionelle neuronale Netzwerke aufzubauen1,2,3. Wachstumskegel, die sensorische und bewegliche Strukturen an der Spitze von Neuriten sind, bestimmen die Geschwindigkeit und Richtung der neuronalen Migration und des Axonauswuchses3,4,5. Da Neuronen von dicht gedrängten Umgebungen umgeben sind, müssen Wachstumszapfen Kräfte gegen ihre Umgebung ausüben, um sich vorwärts zu bewegen6,7. Um die Mechanismen zu verstehen, die der neuronalen Migration und der axonalen Führung zugrunde liegen, sind Analysen der molekularen Mechanik für den Wachstumskegelfortschritt unerlässlich.
Jahrzehntelange Analysen haben ergeben, dass die Zugkraft zur Förderung des Wachstumskegels durch den "Kupplungsmechanismus" erzeugt wird. Es wird angenommen, dass dieser Mechanismus nicht nur im axonalen Wachstumskegel funktioniert, sondern auch im führenden Prozesswachstumskegel wandernder Neuronen8,9,10,11,12. Aktinfilamente (F-Aktine) in Wachstumskegeln polymerisieren nämlich an der vorderen Kante und depolymerisieren proximal, wodurch die vordere Membran herausgedrückt wird13,14,15. Die resultierende Kraft induziert in Verbindung mit der Actomyosinkontraktion eine Rückwärtsbewegung von F-Aktinen, die als retrograder Fluss bezeichnet werden7,11,16,17,18,19,20,21. Kupplungs- und Zelladhäsionsmoleküle vermitteln die mechanische Kopplung zwischen F-Aktin retrogradem Fluss und dem Klebstoffsubstrat und übertragen die Kraft des F-Aktinflusses auf das Substrat, wodurch Zugkraft für den Wachstumskegelfortschritt erzeugt wird7,8,9,11,12,22 . Gleichzeitig reduziert die Aktin-Substrat-Kopplung die F-Aktin-Strömungsgeschwindigkeit und wandelt die Aktinpolymerisation in die Kraft um, die die vordere Membran hervorsteht9,10.
Axonale Wachstumskegel spüren lokale chemische Hinweise und wandeln sie in eine richtungsweisende treibende Kraft für die Wachstumskegelnavigation um3,23,24,25. Zum Beispiel stimuliert ein Axon-Leitmolekül Netrin-1 seinen Rezeptor, der bei Darmkrebs (DCC) gelöscht wurde, und aktiviert die Rho-Guanosintriphosphat (GTP)-bindenden Proteine Zellteilungskontrollprotein 42 (Cdc42) und Ras-assoziiertes C3-Botulinumtoxinsubstrat 1 (Rac1) und ihre nachgeschaltete Kinase p21-aktivierte Kinase 1 (Pak1) 26. Cdc42 und Rac1 fördern 1) Aktinpolymerisation, und Pak1 phosphoryliert ein Kupplungsmolekül Shootin122,26. Shootin1 interagiert mit dem retrograden F-Aktin-Fluss über ein Aktin-bindendes Protein Cortactin27. Shootin1 interagiert auch mit dem L1-Zelladhäsionsmolekül (L1-CAM)20,24. Die Shootin1-Phosphorylierung erhöht die Bindungsaffinitäten für Cortactin und L1-CAM und verbessert die Shootin1-vermittelte 2)-Kupplungskopplung24,27. Innerhalb des Wachstumskegels erhöhen asymmetrische Aktivierungen der Aktinpolymerisation und der Kupplungskopplung 3) die Zugkraft auf der Seite der Netrin-1-Quelle und erzeugen dadurch eine gerichtete Antriebskraft für die Wachstumskegeldrehung (Abbildung 1)24. Die intensive Forschung der letzten Jahrzehnte in Bezug auf neuronale Migration und Axonführung hat das Verständnis von Leitmolekülen, ihren Rezeptoren und den damit verbundenen nachgeschalteten Signalkaskaden verbessert2,10,28,29,30. Die molekularen Maschinen zur Erzeugung von Kräften für den Wachstumskegelfortschritt beginnen jedoch gerade erst, aufgeklärt zu werden; dies kann auf die begrenzte Verwendung der Protokolle für mechanobiologische Analysen zurückgeführt werden.
Die vorliegende Studie beschreibt ein detailliertes Protokoll zur Überwachung des retrograden F-Aktin-Flusses durch Single-Speckle-Bildgebung16,18. Die Überwachung des retrograden F-Aktin-Flusses wurde ausgiebig mit hochauflösender Mikroskopie, Spinnscheiben-Konfokalmikroskopie und TIRF-Mikroskopie (Total Interference Reflection Fluorescence) durchgeführt25,31,32,33,34,35,36,37,38 . Das Protokoll in der vorliegenden Studie verwendet jedoch ein Standard-Epifluoreszenzmikroskop und ist daher leicht zu übernehmen11,16,18,20,22,23,24,27,39,40,41,42. In Kombination mit der F-Aktin-Markierung durch Lifeact43 ermöglicht die Single-Speckle-Bildgebung eine Quantifizierung der Aktinpolymerisationsrate und der Kupplungskopplungseffizienz zwischen dem retrograden F-Aktin-Fluss und dem Klebstoffsubstrat39,42. Die vorliegende Studie beschreibt ferner ein detailliertes Protokoll der Zugkraftmikroskopie unter Verwendung eines fluoreszierenden, in Die Perle eingebetteten Polyacrylamid (PAA)-Gels11,22,23,24,27,39,41,42,44. Diese Methode erkennt und quantifiziert die Zugkraft unter dem Wachstumskegel durch Überwachung der kraftinduzierten Perlenbewegungen44,45. Ein Open-Source-Zugkraftanalysecode wird bereitgestellt, und die Methode zur Quantifizierung der Zugkraft während der Wachstumskegelmigration wird ausführlich erläutert. Mit Hilfe der Einzelfleckenbildgebung und der Zugkraftmikroskopie wird das Verständnis der molekularen Mechanik, die der Migration und Navigation von Wachstumskegeln zugrunde liegt, erleichtert. Diese Techniken sind auch zur Analyse der molekularen Mechanik anwendbar, die der dendritischen Wirbelsäulenvergrößerung zugrunde liegt, von der bekannt ist, dass sie für das Lernen und das Gedächtnis wichtig ist42.
Protokoll
Alle Experimente mit Labortieren wurden mit dem Institutional Animal Care and Use Committee des Nara Institute of Science and Technology durchgeführt. Die Prüfer sollten die von ihren institutionellen und nationalen Tierregulierungsausschüssen festgelegten Leitlinien für die Pflege und Verwendung von Versuchstieren befolgen.
1. Vorbereitung von Lösungen und Medien
- Vorbereiten der Lösungen und Nährmedien wie in Ergänzungsdatei 1 zusammengefasst
2. Herstellung von Poly-D-Lysin (PDL)/Laminin-beschichteten Substraten
- Glasbodenschalen mit einem Durchmesser von 14 mm mit 100 μg/ml PDL, gelöst in phosphatgepufferter Kochsalzlösung (PBS, pH 7,4), beschichten und über Nacht in einem befeuchteten Inkubator bei 37 °C inkubieren.
HINWEIS: Trocknen Sie die Glasflächen nach diesem Schritt nicht aus - Entfernen Sie die PDL-Lösung und pipettieren Sie 1 mL PBS dreimal für 10 s auf das Glas.
- Das Geschirr mit 5 μg/ml Laminin, gelöst in PBS, bestreichen und über Nacht in einem befeuchteten Inkubator bei 37 °C inkubieren.
- Entfernen Sie die Lamininlösung und pipettieren Sie 1 ml PBS dreimal für 10 s auf das Glas.
- Entfernen Sie das PBS und legen Sie 0,5 ml des neurobasalen Mediums auf die Glasoberflächen. Lagern Sie das Geschirr in einem befeuchteten Inkubator bei 37 °C.
HINWEIS: PDL/Laminin-beschichtetes Geschirr kann 2-3 Tage in einem befeuchteten Inkubator bei 37 °C gelagert werden.
3. Dissektion und Dissoziation des Hippocampus
- Euthanasie eine schwangere Maus durch zervikale Luxation.
HINWEIS: Die vorliegende Studie verwendet kommerziell erhältliche Mäuse (siehe Materialtabelle). Forscher sollten Mäuse verwenden, die in einer sterilisierten Umgebung gezüchtet und human behandelt werden. - Sezieren Sie embryonale Tag-16 (E16) Mausembryonen und legen Sie sie auf Eis.
- Sezieren Sie in einer Laminar-Flow-Haube die Gehirne mit einer Schere und legen Sie sie auf eine sterile 10-cm-Schale mit 10 ml eiskalter Dissektionslösung.
- Ziehen Sie mit einem Sezierstereomikroskop vorsichtig die Hirnhäute der Gehirnhälften ab und sezieren Sie dann die Hippocampi mit einer Pinzette.
- Übertragen Sie die Hippocampi in 5 ml eiskalte Verdauungslösung in einem 15 ml Röhrchen. Inkubieren Sie die Hippocampi in einem Wasserbad für 20 min bei 37 °C.
- Entfernen Sie die Aufschlusslösung und fügen Sie 3 ml Dissektionslösung hinzu.
- Die Hippocampi viermal vorsichtig mit einer Pasteurpipette pipettieren.
- Inkubieren Sie die Hippocampi in einem Wasserbad für 20 min bei 37 °C.
- Die Zellsuspension wird in ein 50-ml-Zentrifugenröhrchen überführt. 3 ml neue Dissektionslösung in das undissoziierte Gewebe geben.
- Wiederholen Sie die Schritte 3.7-3.9, bis die Hippocampi vollständig dissoziiert sind.
- Entfernen Sie die schwimmenden DNA-Aggregate aus der Zellsuspension, indem Sie sie mit einer Pasteur-Pipette schwenken und absaugen. Zentrifugieren Sie die Zellsuspension bei 180 x g für 20 min bei 4 °C.
HINWEIS: DNA, die von beschädigten Zellen stammt, stört den Zentrifugationsprozess. Wenn Neuronen im Überstand verbleiben, zentrifugieren Sie die Zellsuspension nach vorsichtiger Entfernung der DNA erneut. - Equilibriertes neurobasales Medium mit 10% fetalem Rinderserum (FBS), Penicillin (100 U/ml) und Streptomycin (100 μg/ml) in einem befeuchteten Inkubator bei 37 °C mit 5 % CO2.
4. Transfektion und Kultivierung von Neuronen
- Entfernen Sie den Überstand und geben Sie 5 ml eiskaltes PBS in das 50 ml Rohr. Resuspendieren Sie das Zellpellet durch schonendes Pipettieren.
- Übertragen Sie 10 μL der Zellsuspension in ein Mikrozentrifugenröhrchen, fügen Sie 10 μL 0,5% ige Trypanblaulösung hinzu und zählen Sie dann die Anzahl der Zellen mit einem Hämozytometer.
- Zentrifugieren Sie die Zellsuspension im 50-ml-Röhrchen bei 180 x g für 20 min bei 4 °C.
- In der Zwischenzeit aliquot für die Transfektion, 1 μg pFN21A-HaloTag-Aktin und 3 μg pmNeonGreen-N1-Lifeact pro 1 x 106 Zellen, in ein Mikrozentrifugenröhrchen.
- Entfernen Sie nach der Zentrifugation den Überstand und geben Sie 100 μL Elektroporationsmedium (bereitgestellt durch Transfektionskit) in das 50-ml-Röhrchen. Resuspendieren Sie das Zellpellet durch Pipettieren.
- Mischen Sie die Zellsuspension mit der aliquotenden DNA-Lösung und übertragen Sie die Mischung auf eine Küvette (bereitgestellt durch das Transfektionskit).
HINWEIS: Da Luftblasen die Elektroporation stören, entfernen Sie sie aus der Zellsuspension. - Setzen Sie die Küvette in die Elektroporationsvorrichtung (Materialtabelle) ein und führen Sie die Elektroporation mit dem Programm O-005 durch.
- Geben Sie sofort 1 ml des vorgewärmten und äquilibrierten neurobasalen Mediums, das 10% FBS, Penicillin (100 U / ml) und Streptomycin (100 μg / ml) enthält, in die Küvette.
- Übertragen Sie die Zellsuspension mit einer Kunststoffpipette (bereitgestellt durch das Transfektionskit) in ein 15-ml-Zentrifugenröhrchen.
- Übertragen Sie 10 μL der Zellsuspension in eine Mikroröhre, fügen Sie 10 μL 0,5% ige Trypanblaulösung hinzu und zählen Sie dann die Anzahl der Zellen mit einem Hämozytometer.
- Nehmen Sie PDL/Laminin-beschichtete Glasbodenschalen aus dem Inkubator und entfernen Sie das neurobasale Medium.
- 0,5 ml der Zellsuspension mit 2,0 x 105 Zellen pro Schale werden pipettiert und in einem befeuchteten Inkubator bei 37 °C mit 5% CO2 für 3 h inkubiert.
- Ersetzen Sie das Medium durch 0,5 ml neurobasisales Medium, das 2% B-27-Präparat, Glutamin (1 mM), Penicillin (100 U / ml) und Streptomycin (100 μg / ml) enthält. Kulturieren Sie die Neuronen in einem befeuchteten Inkubator für 3 Tage bei 37 °C mit 5% CO2.
5. Einzelfleckenbildgebung an neuronalen Wachstumskegeln
- Behandeln Sie die Neuronen am Tag in vitro (DIV) 3 mit Tetramethyl-Rhodamin (TMR)-Liganden bei einer Verdünnung von 1:2000 Verdünnung im Kulturmedium. Halten Sie die Neuronen für 1 h bei 37 °C mit 5% CO2 aufrecht.
- Waschen Sie den TMR-Liganden dreimal mit vorgewärmtem PBS.
- Entfernen Sie das PBS und pipettieren Sie 0,5 ml erwärmtes Leibovitz-L-15-Medium mit 2% B-27-Präparat, Glutamin (1 mM), Penicillin (100 U / ml) und Streptomycin (100 μg / ml).
- Halten Sie Neuronen für 1 h bei 37 ° C aufrecht.
- Schalten Sie ein Epifluoreszenzmikroskop ein und stellen Sie den Stage-Top-Inkubator auf 37 °C ein.
HINWEIS: Das vorliegende Protokoll verwendet ein Epifluoreszenzmikroskop, das mit einer komplementären Metalloxid-Halbleiterkamera, einer 100x/1,40 NA Ölimmersionsobjektivlinse, einer Quecksilberlampe und einer Bilderfassungssoftware ausgestattet ist (siehe Materialtabelle). Für diese Analyse können auch andere Epifluoreszenzmikroskope mit gleichwertigen Spezifikationen verwendet werden. - Platzieren Sie die mit TMR-Liganden behandelten Neuronen in der Glasbodenschale auf dem erwärmten Stage-Top-Inkubator.
- Stellen Sie die Bildaufnahmeparameter wie folgt ein: Belichtungszeit, 500 ms für Lifeact- und HaloTag-Aktin-Fluoreszenzkanäle; Binning, 1 x 1 (0,065 μm × 0,065 μm pro Pixel); Zeitintervall, 3 s; Dauer, 50 Frames.
- Wählen Sie einen Wachstumskegel, der Lifeact stark und HaloTag-Actin schwach exprimiert.
HINWEIS: Die Lifeact-Expression sollte stark genug sein, um die Morphologie des Wachstumskegels zu visualisieren. Die HaloTag-Aktin-Expression sollte schwach sein, nicht ausreichend, um nachgewiesen zu werden (Abbildung 2A). Die Spiegel der fluoreszierenden Proteinexpressionen können durch Variieren der DNA-Menge für die Transfektion angepasst werden. - Schließen Sie das Feld der Membran, um einen Mindestbereich zu beleuchten, der den Wachstumskegel enthält (Abbildung 2B).
HINWEIS: Die Beleuchtung einer Minimalfläche reduziert das Hintergrundsignal und erhöht das Verhältnis von Fluoreszenzsignal zu Rauschen (S/N), wodurch die Erkennung von Aktinflecken ermöglicht wird (Abbildung 2B). Um das S/N-Verhältnis weiter zu erhöhen, wird empfohlen, den Wachstumskegel ohne Reduzierung des Lichts durch Neutraldichtefilter zu beleuchten. - Erfassen Sie Zeitrafferbilder (Ergänzende Datei 2). Speichern sie als Mehrkanal-Zeitraffer-Stack-Bild (TIFF-Format).
6. Quantifizierungen der F-Aktin-Strömungsgeschwindigkeit und Polymerisationsrate mit einer Bildverarbeitungs- und Analysesoftware Fidschi
HINWEIS: Praxisdaten zur Quantifizierung der F-Aktin-Strömungsgeschwindigkeit und der Aktinpolymerisationsrate finden Sie in der Ergänzungsdatei 3 .
- Öffnen Sie das Mehrkanal-Zeitraffer-Stack-Image auf Fidschi.
- Wählen Sie Analysieren > Maßstab festlegen und legen Sie die Pixelgröße der Bilder fest.
- Finden Sie einen Aktinfleck, der retrograd für mindestens fünf Zeitrahmen innerhalb eines F-Aktin-Bündels in Filopodien oder Lamellipodien fließt.
- Wählen Sie Bild > Transformieren > Drehen aus, und passen Sie den Winkel der Bilder so an, dass das F-Aktinbündel nach oben zeigt (Abbildung 3A).
- Klicken Sie in der Symbolleiste auf Rechteck und zeichnen Sie ein Feld mit Aktinflecken und der Spitze des F-Aktinbündels (Abbildung 3B, 1 und 2).
HINWEIS: Die Signale von HaloTag-Aktin-Sprenkeln sind schwach. Darüber hinaus sind Lifeact-Signale an den distalen Spitzen des Wachstumskegels schwach und schwach (Abbildung 2B), da die distalen Spitzen des Wachstumskegels im Vergleich zum proximalen Teil dünn sind. Die Forscher sollten die Signale verbessern, um die Quantifizierungen der F-Aktin-Strömungsgeschwindigkeit und der Polymerisationsrate zu optimieren (Abbildung 3B). Obwohl das Lifeact-Signal am proximalen Wachstumskegel gesättigt wird, stört dies die Bestimmung des distalen Endes von F-Aktinen nicht (Abbildung 3E, gelbe Linie). - Wählen Sie Bild > Duplizieren aus, und geben Sie die fünf Zeitrahmen ein (Abbildung 3B, 3-5).
- Wählen Sie Image > Stacks > Make Montage und geben Sie die folgenden Parameter ein: Spalten, 5; Reihen, 1; Skalierungsfaktor, 1.
- Klicken Sie auf OK. Die Bildmontage erscheint auf dem Bildschirm.
- Wählen Sie Bild > Farbe > Geteilte Kanäle. Dadurch werden die beiden Fluoreszenzkanäle getrennt.
- Quantifizierung der F-Aktin-Strömungsgeschwindigkeit.
- Wählen Sie Analysieren > Messwerte festlegen und dann Fläche und Begrenzungsrechteck aus.
- Klicken Sie in der Symbolleiste auf Oval und zeichnen Sie einen Kreis auf einem Aktinfleck (Abbildung 3C, 1 und 2).
- Wählen Sie Bild > Überlagerung > Auswahl hinzufügen. Der Kreis wird auf der Bildmontage überlagert (Abbildung 3C, 3 und 4).
HINWEIS: Die Farbe des Overlays kann unter Bearbeiten > Optionen > Farben geändert werden. - Wiederholen Sie die Überlagerung der Kreise für die verbleibenden Flecken (Abbildung 3C, 5).
HINWEIS: Um die Zentren des Aktinfleckens genau zu bestimmen, sollten die Ermittler Kreise auf den Flecken überlagern. - Klicken Sie in der Symbolleiste auf Gerade , und zeichnen Sie eine Linie, die die Mittelpunkte der Kreise verbindet (Abbildung 3D, 1 und 2).
- Wählen Sie > Measure analysieren aus. Das Ergebnis wird auf dem Bildschirm angezeigt (Abbildung 3D, 3).
HINWEIS: Die im Ergebnis angezeigte Höhe gibt den Translokationsabstand des Aktinflecks während der Beobachtung an (Abbildung 3D, 3). - Berechnen Sie die F-Aktin-Strömungsgeschwindigkeit, indem Sie den Translokationsabstand durch die Beobachtungszeit dividieren.
- Quantifizierung der F-Aktin-Polymerisationsrate.
- Zeichnen Sie eine Linie, die die Spitzen des F-Aktinbündels verbindet (Abbildung 3E, 1).
- Wählen Sie > Measure analysieren aus. Das Ergebnis wird auf dem Bildschirm angezeigt (Abbildung 3E, 2).
ANMERKUNG: Die Höhe im Ergebnis gibt die Ausdehnungslänge des F-Aktinbündels während der Beobachtung an (Abbildung 3E, 2). - Berechnen Sie die F-Aktin-Extensionsrate, indem Sie die Extensionslänge durch die Beobachtungszeit dividieren.
- Berechnen Sie die F-Aktin-Polymerisationsrate als Summe der F-Aktin-Strömungsgeschwindigkeit und der Ausdehnungsrate (Abbildung 3F).
7. Herstellung eines PAA-Gels für Zugkraftmikroskopie und Neuronenkulturen
- Herstellung von PAA-Gelen, die für die Zugkraftmikroskopie erforderlich sind (Abbildung 4)
- 0,5 mL NaOH (100 mM), gelöst in destilliertem H2O, auf Glasbodenschalen mit 27 mm Durchmesser pipettieren und 15 min bei Raumtemperatur (RT) inkubieren.
- 50 μL 3-Aminopropyltrimethyoxysilan (APTMS) in NaOH-Lösung auf den Schalen geben und die Lösungen durch sanftes Pipettieren mischen und 15 min bei RT inkubieren.
- Das Glasbodengeschirr mit destilliertem H2O waschen und trocknen.
- 0,5 ml 0,5% ige Glutaraldehydlösung, gelöst in PBS, auf die APTMS-behandelte Glasbodenschale pipettieren und 30 min bei RT inkubieren.
- Das Glasbodengeschirr mit destilliertem H2O abwaschen und trocknen.
- Es wird eine Lösung hergestellt, die destillierte H2O (412 μL), 30% (w/v) Acrylamidlösung (63 μL), 2,5% (w/v) Bisacrylamidlösung (6 μL) und carboxylatmodifizierte Mikrokugellösung (fluoreszierende Perle) (20 μL) enthält.
HINWEIS: Wirbeln Sie die fluoreszierende Perlenlösung vor, um aggregierte Kügelchen zu dissoziieren, bevor Sie sie der Acrylamidlösung hinzufügen. - Entgasen Sie die Lösung in einer Vakuumkammer für 30 min bei RT.
- Zu dieser Lösung werden 1 μL 10% (w/v) Ammoniumpersulfat (APS), gelöst in destilliertem H2O, und 1 μL N,N,N',N'-Tetramethylethylendiamin (TEMED) zugegeben und durch schonendes Pipettieren vermischt.
- Legen Sie einen Glasdecker (18 mm Durchmesser) auf die Kappe eines 15 ml Zentrifugenröhrchens.
- Sofort 25 μL der Lösung in den Deckglas geben und dann vorsichtig eine umgekehrte Glasbodenschale auf den Deckglas legen. Lassen Sie das Acrylamid für 1 h bei RT polymerisieren.
- Tragen Sie destilliertes H2O auf das Deckglas auf und schälen Sie es mit einer Spritzennadel mit einer gebogenen Spitze vom Gel ab.
- Entfernen Sie das H2O und geben Sie Sulfo-SANPAH (1 mM), gelöst in PBS, in das Gel.
- Unter sterilen Bedingungen die Gele 5 min ultraviolettem Licht aussetzen. Waschen Sie das Gel dreimal mit PBS für 15 min pro Wäsche.
- Bestimmung der Gelsteifigkeit mit einem Mikrokugel-Eindringverfahren (Abbildung 5A)44.
- Übertragen Sie eine Mikrokugel, die für durchmesser und dichte spezifiziert ist, auf das Gel und legen Sie sie dann auf die Probenbühne eines konfokalen Laserscanning-Mikroskops.
- Konzentrieren Sie sich auf die Geloberfläche und zeichnen Sie die Z-Position auf (Abbildung 5B).
- Konzentrieren Sie sich in ähnlicher Weise auf den Boden der Mikrosphäre und zeichnen Sie die z-Position auf (Abbildung 5C).
- Berechnen Sie die Eindringtiefe, die durch die Differenz zwischen den Z-Positionen der Geloberfläche und dem Boden der Mikrosphäre bestimmt wird.
- Berechnen Sie den Elastizitätsmodul E des Gels durch:
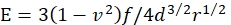
wobei f das auftriebskorrigierte Gewicht der Mikrosphäre, d die Eindringtiefe des Gels, r der Radius der Mikrosphäre und v das Poisson-Verhältnis ist (dessen Wert 0,3 ist, wie zuvor bestimmt46).
- Beschichten Sie die Gele mit PDL und Laminin, wie in Abschnitt 2 beschrieben.
- Wie in den Abschnitten 3 und 4 beschrieben, sezieren Sie E16-Mausembryonen, dissoziieren Sie Hippocampi und transfizieren Sie mit 5 μg pEGFP-C1 pro 1,0 x 106 Zellen. Säen Sie 2,0 x 105 Zellen auf PDL/Laminin-beschichtete Gele.
HINWEIS: Für die Zugkraftanalyse ist eine mikroskopische Bildgebung von fluoreszenzmarkierten Zellen zur Bestimmung der Wachstumskegelfläche erforderlich.
8. Zugkraftmikroskopie an neuronalen Wachstumskegeln
- Ersetzen Sie bei DIV3 das Kulturmedium durch 0,5 ml erwärmtes Leibovitz-L-15-Medium, das 2% B-27-Präparat, Glutamin (1 mM), Penicillin (100 U / ml) und Streptomycin (100 μg / ml) enthält. Halten Sie die Neuronen für 1 h bei 37 ° C aufrecht.
- Schalten Sie ein konfokales Laserscanning-Mikroskop ein und stellen Sie den Stage-Top-Inkubator auf 37 °C ein.
HINWEIS: Die vorliegende Studie verwendet ein konfokales Laserscanning-Mikroskop, das mit einer 63x/1,2 NA Wasserimmersionsobjektivlinse und einer Bilderfassungs- und Analysesoftware ausgestattet ist (siehe Materialtabelle). - Legen Sie die Neuronen in die Glasbodenschale auf den vorgewärmten Stage-Top-Inkubator.
- Stellen Sie die Bildaufnahmeparameter wie folgt ein: Scangröße, 512 × 512 Pixel; Scanbereich, 1,5-3-facher Zoom; Scangeschwindigkeit, ~1 s pro Frame; Laserwellenlänge, 561 nm (die Anregungswellenlänge für die Fluoreszenzperlen) und 488 nm (die Anregungswellenlänge für das verstärkte grün fluoreszierende Protein (EGFP)); Zeitintervall, 3 s; Dauer, 50 Frames.
- Um die Morphologie des Wachstumskegels zu visualisieren, wählen Sie einen Wachstumskegel, der EGFP stark exprimiert.
- Konzentrieren Sie sich auf die Geloberfläche und erfassen Sie Zeitrafferbilder (Abbildung 6A und Ergänzungsdatei 4).
HINWEIS: Verwenden Sie für die Traktionsanalyse zwei Fluoreszenzkanäle (für Fluoreszenzperlen und EGFP) und einen Hellfeldkanal (die Erfassung von differentiellem Interferenzkontrast (DIC) oder Phasenkontrastbildern wird empfohlen). - Verwenden Sie eine Bildverarbeitungssoftware (z. B. Fidschi), um einkanalige Zeitraffer-RGB-Bildstapel zu erstellen und diese Bilder als TIFF-Dateien zu speichern.
HINWEIS: Es ist wichtig, dass die Ermittler auch die Rohdaten speichern. - Tragen Sie 100 μL 10% (w/v) Natriumdodecylsulfat (SDS), gelöst in destilliertem H2O, auf die Glasbodenschale auf, um das Gelsubstrat zu entspannen, indem Neuronen aus dem Substrat freigesetzt werden. Die Schale 5 min bei 37°C inkubieren, um die Temperatur zu stabilisieren.
HINWEIS: Das Bild der Kügelchen in einem ungespannten Substrat wird verwendet, um die ursprünglichen Wulstpositionen während der Zugkraftanalyse zu referenzieren (Abbildung 6C). Die Anwendung von SDS-Lösung verändert die xyz-Positionen aufgrund der thermischen Änderung im Inkubator und der Relaxation der zellinduzierten Verformung. Die Ermittler müssen daher die Brennebene und die x-y-Position korrigieren. - Konzentrieren Sie sich auf die Geloberfläche und erfassen Sie ein Bild der Perlen im ungespannten Substrat (Abbildung 6B).
- Erzeugen Sie ein einkanaliges RGB-Bild der Perlen im ungespannten Substrat und speichern Sie dieses Bild als TIFF-Datei.
- Korrektur der x-y-Position des Perlenbildes im ungespannten Substrat mit Fidschi
- Öffnen Sie in Fidschi das Perlenbild im ungespannten Substrat und das Zeitraffer-Stapelbild von fluoreszierenden Perlen.
- Wählen Sie Bild > Stapel > Werkzeuge > verketten. Auf dem Bildschirm wird ein Dialogfeld angezeigt (Abbildung 7A, 1).
- Wählen Sie das Perlenbild im ungespannten Substrat und das Zeitraffer-Stapelbild der fluoreszierenden Kügelchen in Bild 1 bzw. Bild2 aus (Abbildung 7A, 2).
- Klicken Sie auf OK. Das Perlenbild im ungespannten Substrat wird dem Zeitraffer-Stapelbild hinzugefügt (Abbildung 7A, 3).
- Zeigen Sie mithilfe der Bildlaufleiste (Abbildung 7B, 1, roter Rahmen) den zweiten Frame des Stapelbilds (roter Pfeil) an.
- Wählen Sie Plugins > StackReg aus (Abbildung 7B, 2). Auf dem Bildschirm wird ein Dialogfeld angezeigt (Abbildung 7B, 3).
- Wählen Sie starre Karosserie aus der Dropdown-Liste (Abbildung 7B, 3, roter Rahmen) und klicken Sie auf OK. Die Korrektur der x-y-Position beginnt dann.
- Verwenden Sie die Bildlaufleiste, um das erste Bild des positionskorrigierten x-y-Stapelbilds anzuzeigen.
- Wählen Sie Bild > Duplikat aus (Abbildung 7C, 1). Auf dem Bildschirm wird ein Dialogfeld angezeigt (Abbildung 7B, 2).
- Geben Sie 1 in den Bereich ein, deaktivieren Sie Duplicate Stack (Stack duplizieren ) (Abbildung 7C, 2), und klicken Sie auf OK. Das x-y-positionskorrigierte Perlenbild im ungespannten Substrat wird auf dem Bildschirm angezeigt (Abbildung 7C, 3). Speichern Sie dieses Bild als TIFF-Datei.
- Quantifizierung der Zugkraft
HINWEIS: Die hier beschriebene Methode zur Zugkraftanalyse verwendet MATLAB und zwei MATLAB-Toolboxen, 'Image Processing Toolbox' und 'Parallel Computing Toolbox'. Die Ermittler müssen sie vor der Analyse installieren. Der Zugkraftanalysecode wurde auf Basis von MATLAB Version 2018a entwickelt. Daher muss MATLAB Version 2018a (oder höher) für die Analyse verwendet werden. Die für die Zugkraftanalyse verwendeten Algorithmen wurden zuvor beschrieben22.- Laden Sie den Zugkraftanalysecode TFM2021 aus Supplemental File 5 herunter. TfM 2021 in MATLAB öffnen
- Öffnen Sie main.m in TFM2021 und führen Sie es aus. Auf dem Bildschirm wird eine grafische Benutzeroberfläche (GUI) angezeigt (Abbildung 8A).
- Klicken Sie auf Unbelastetes Substratbild laden und wählen Sie das x-y-positionskorrigierte Perlenbild im unbelasteten Substrat aus.
- Klicken Sie auf Fluoreszierende Perlenbilder laden und wählen Sie das Zeitraffer-Stapelbild der Perlen aus.
- Klicken Sie auf Hellfeldbilder laden und wählen Sie das Zeitraffer-Stapelbild von Hellfeld aus.
- Klicken Sie auf GFP-Bilder laden und wählen Sie das Zeitraffer-Stack-Bild von EGFP aus.
- Wählen Sie GFP aus der Dropdown-Liste auf der GUI aus (Abbildung 8A, rotes Feld).
- Klicken Sie auf ROI , um die rechteckige Region of Interest (ROI), einschließlich des Wachstumskegels, anzugeben, indem Sie auf zwei Punkte auf dem zellenbild klicken, das auf der GUI angezeigt wird (Abbildung 8B).
- Klicken Sie auf der GUI auf die Schaltfläche Speichern (Abbildung 8A, roter Pfeil). Die ausgewählten Stack-Images werden zusammen mit dem ROI in einer .mat-Datei (MATLAB-Formatdatei) gespeichert.
- Klicken Sie auf Spot-Erkennung. Auf dem Bildschirm wird ein Dialogfeld angezeigt.
- Geben Sie einen Wert (normalerweise 50-150) in das Dialogfeld ein, um einen Schwellenwert für die Perlenerkennung zu bestimmen. Durch Klicken auf OK wird die Berechnung gestartet.
- Klicken Sie nach Abschluss der Berechnung auf Spur plotten , um den in Schritt 8.12.8 ausgewählten Bereich zu vergrößern und die erkannten Perlen als weiße Punkte anzuzeigen (Abbildung 8C, 1).
HINWEIS: Überprüfen Sie, ob sich die Perlendetektion (weiße Punkte) mit den fluoreszierenden Kügelchen überschneidet. Die Perlenerkennung kann auch Hintergrundgeräusche erkennen. Um diese externen Störungen zu reduzieren, ändern Sie den Schwellenwert bei Spot-Erkennung. Wählen Sie außerdem in Schritt 8.12.13 manuell die richtigen Punkte aus. - Klicken Sie auf Perlen auswählen und markieren Sie einen polygonalen Bereich, der die richtigen Punkte unter dem Wachstumskegel enthält. Drücken Sie die Eingabetaste auf der Tastatur. Die weißen Punkte innerhalb des polygonalen Bereichs ändern sich in eine rote Farbe (Abbildung 8C, 2).
- Klicken Sie in der GUI auf Kraft schätzen . Dann Eingabewerte für die folgenden Parameter: Pixelgröße, μm/Pixel; Elastizitätsmodul, der bei Schritt 7.2.5 berechnete Wert; Poisson-Verhältnis, 0,3.
- Ausführen von Estimate force , um die Berechnung zu initiieren. Die Software speichert die Berechnungsergebnisse automatisch in einer Datei im Tabellenkalkulationsformat.
HINWEIS: Die Tabelle zeigt die x-Komponente, die y-Komponente und die Kraftstärke in jedem Zeitrahmen an (Abbildung 8D). In Ergänzungsdatei 6 finden Sie Übungsdaten zur Quantifizierung der Zugkraft.
Ergebnisse
Single-Speckle-Bildgebung zur Quantifizierung der Aktinpolymerisationsrate und der Kupplungskupplungseffizienz
Eine hohe Lifeact-Expression ermöglicht die Visualisierung von F-Aktin innerhalb des Wachstumskegels; Eine niedrige HaloTag-Aktin-Expression ermöglicht die Überwachung des retrograden F-Aktin-Flusses (Abbildung 3, Ergänzende Datei 2). Die Verfolgung der Aktinflecken ermöglicht die Messung der F-Aktin-Strömungsgeschwindigkeit (Abbildung 3C,D). Da die mechanische Kopplung von F-Aktin retrograder Strömung und dem Klebesubstrat die F-Aktin-Strömungsgeschwindigkeit reduziert, kann die Kupplungskupplungseffizienz aus der Geschwindigkeit abgeschätzt werden. Darüber hinaus hilft die F-Aktin-Markierung mit Lifeact bei der Visualisierung der F-Aktin-Extension und ist nützlich für die Quantifizierung der Aktinpolymerisationsrate (Abbildung 3E,F).
Quantitative Analyse der Zugkraft
Die strikte Einhaltung der hier vorgestellten Methodik zeigt die Bewegungen der fluoreszierenden Kügelchen unter dem Wachstumskegel (Abbildung 6C, Supplemental File 4). Die retrograde F-Aktin-Strömung auf das Substrat erzeugt eine Zugkraft, wodurch sich die fluoreszierenden Kügelchen unter dem Wachstumskegel nach hinten bewegen. Der Zugkraftanalysecode schätzt die Zugkraft aus der Fluoreszenzperlenverschiebung und drückt die berechnete Zugkraft als Kraftvektor aus. Richtung und Größe der Zugkraft werden aus den x- und y-Komponenten des Kraftvektors bestimmt (Bild 8C,D). Das rote Kästchen in Abbildung 8D stellt die x- und y-Komponenten eines Kraftvektors dar. Abbildung 8C zeigt den entsprechenden Wachstumskegel. In Bezug auf x-y-Koordinaten zeigt der Vektor auf -93,8° gegenüber der x-Achse; diese Ausrichtung ist auf die Rückseite des Wachstumskegels gerichtet (Abbildung 8C). Die Größe der Zugkraft F wurde wie folgt berechnet:
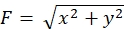
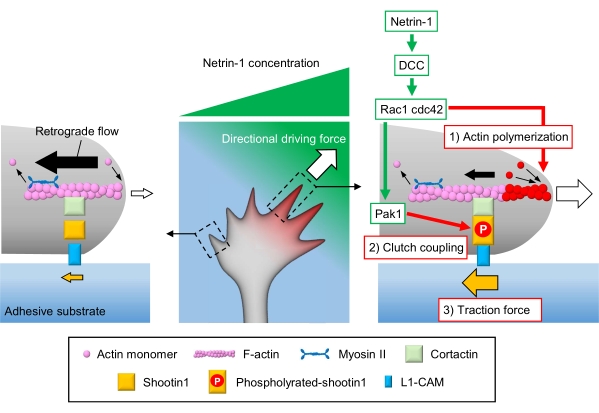
Abbildung 1: Eine Wachstumskegelmaschinerie zur Krafterzeugung und Wachstumskegelnavigation. Ein netrin-1 chemoattraktiver Gradient induziert eine asymmetrische Stimulation seines Rezeptors DCC auf einem axonalen Wachstumskegel. Dies aktiviert Rac1 und Cdc42 und ihre nachgeschaltete Kinase Pak1. Rac1 und Cdc42 fördern (1) die Aktinpolymerisation, während Pak1 Shootin1 phosphoryliert und die Shootin1-vermittelte (2) Kupplungskopplung verstärkt. Die asymmetrische Aktivierung der Aktindynamik und der Kupplungskopplung innerhalb des Wachstumskegels erhöht (3) die Zugkraft auf der Seite der Netrin-1-Quelle und erzeugt dadurch eine gerichtete Antriebskraft für die Wachstumskegelanziehung. Die hier vorgestellten Protokolle erlauben die Quantifizierung der Schlüsselvariablen (1)-(3) für die Wachstumskegelnavigation. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Fluoreszenzbilder eines neuronalen Wachstumskegels unter einem vollständig geöffneten und verengten Zwerchfell. Der Wachstumskegel drückt Lifeact und HaloTag-Aktin aus. (A) Eine hohe Lifeact-Expression ermöglicht die Visualisierung der Wachstumskegelmorphologie. Auf der anderen Seite sind die HaloTag-Aktin-Expressionsniveaus sehr niedrig, mit schwachen Signalen, wenn die Membran vollständig geöffnet ist. (B) Wenn das Zwerchfell angemessen verengt ist, nehmen die Hintergrundsignale ab und einzelne Aktinflecken erscheinen im Wachstumskegel. Maßstabsbalken: 5 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Schritte zur Quantifizierung der F-Aktin-F-Aktin-Strömungsgeschwindigkeit und Aktinpolymerisationsrate mit einer Bildverarbeitungs- und Analysesoftware Fidschi. (A) Passen Sie den Winkel des Bildes für die Analyse an. (1) Suchen und wählen Sie einen Aktinfleck (Pfeilspitze), der in einem F-Aktinbündel fließt. (2) Wählen Sie Bild > Transformieren > Drehen. (3) Stellen Sie den Winkel so ein, dass das F-Aktinbündel nach oben gerichtet ist. (4) Der Winkel des Bildes wird geändert. (B) Abgrenzung der Region, einschließlich des Aktinflecks und des F-Aktinbündels. (1) Klicken Sie in der Symbolleiste auf Rechteck. (2) Beschreiben Sie einen Bereich auf dem Bild. Die Helligkeit und der Kontrast des Bildes werden erhöht, um eine klare Visualisierung des Aktinflecks und der Spitze des F-Aktinbündels zu ermöglichen. (3) Wählen Sie Bild > Duplizieren. (4) Geben Sie die fünf Zeitrahmen ein, die den Aktin-Speckle-Fluss im F-Aktin-Bündel zeigen. (5) Das ausgewählte Stapelbild erscheint auf dem Bildschirm. (C) Überlagern Sie Kreise auf dem Aktinfleck. (1) Klicken Sie in der Symbolleiste auf Oval. (2) Zeichne einen Kreis auf einen Aktinfleck. (3) Wählen Sie Bild > Überlagerung > Auswahl hinzufügen. (4) Der Kreis wird überlagert. 5) Wiederholen Sie das Überlagern der Kreise auf den verbleibenden Aktinsprenkeln. (D) Messen Sie den Translokationsabstand von Aktinsprenkeln während der fünf Zeitrahmen. (1) Klicken Sie in der Symbolleiste auf Gerade. (2) Zeichne eine Linie, die die Mittelpunkte der Kreise verbindet. (3) Wählen Sie > Measure analysieren aus. Das Ergebnis, angezeigt durch den Parameter Höhe (rote Box), das den Aktin-Speckle-Translokationsabstand weitergibt. (E) Messen Sie die Änderung der Länge des F-Aktin-Vorsprungs während der fünf Zeitrahmen. (1) Zeichnen Sie eine Linie, die die Spitzen des F-Aktin-Vorsprungs verbindet. (2) Wählen Sie > Measure analysieren aus. Das Ergebnis (rotes Feld), Höhe, gibt die Verlängerungslänge des F-Aktinbündels an. (F) Die Aktinpolymerisationsrate wird aus der Summe der F-Aktin-Strömungsgeschwindigkeit und der Ausdehnungsrate berechnet. Siehe auch Ergänzende Datei 2. Ergänzende Akte 3 hilft den Ermittlern, die oben beschriebene Methodik zu üben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
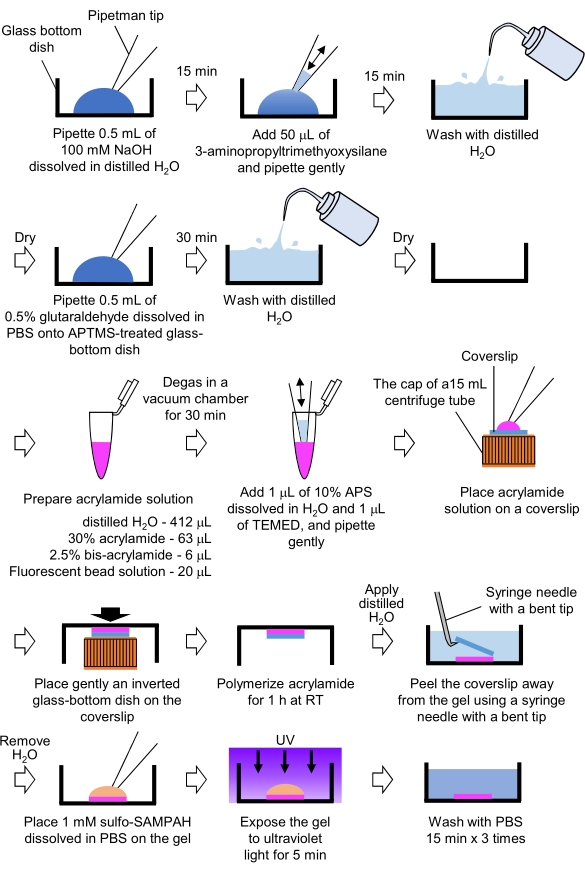
Abbildung 4: Schritte für die PAA-Gel-Zubereitung. Eine detaillierte Beschreibung finden Sie in Schritt 7.1. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
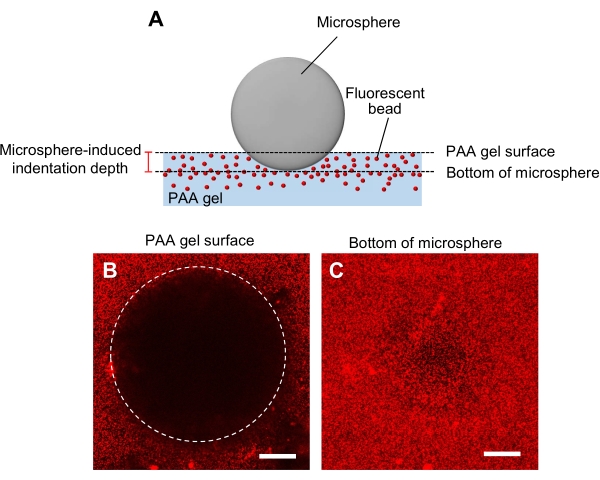
Abbildung 5: Bestimmung der PAA-Gelsteifigkeit. (A) Ein Mikrosphären-Eindringverfahren. Wenn eine Mikrokugel auf ein fluoreszierendes, in eine Perle eingebettetes PAA-Gel gelegt wird, verursacht das Gewicht der Mikrokugel eine Vertiefung im Gel. Die Eindringtiefe wird berechnet, indem die z-Positionen der PAA-Geloberfläche vom Boden der Mikrosphäre (B, C) subtrahiert werden Fluoreszenzbilder eines PAA-Gels, das von einer Mikrokugel eingerückt wird und fluoreszierende Kügelchen enthält. Ein konfokales Laserscanning-Mikroskop wurde verwendet, um Bilder der Geloberfläche (B) und des Bodens der Mikrosphäre (C) aufzunehmen. Signale der fluoreszierenden Kügelchen sind an der Geloberfläche im eingerückten Bereich (B, Kreis) nicht sichtbar. Sie können jedoch am Boden der Mikrosphäre beobachtet werden. Maßstabsbalken: 50 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
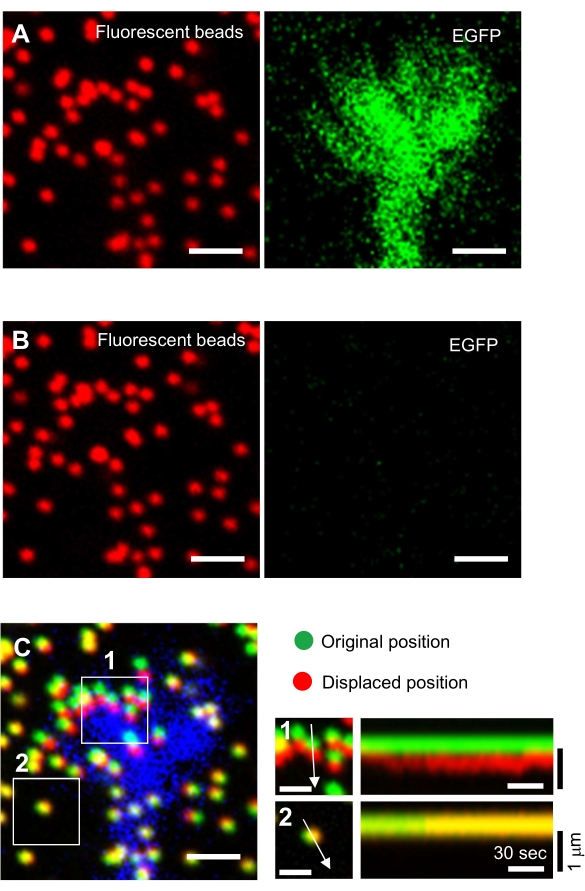
Abbildung 6: Kraftkartierung eines neuronalen Wachstumskegels. (A-C) Fluoreszenzbilder von Perlen, die in ein PAA-Gel und einen neuronalen Wachstumskegel eingebettet sind, visualisiert mit EGFP. Nach der Aufnahme von Zeitrafferbildern (A) wurde das Neuron durch Auftragen von SDS-Lösung aus dem Gelsubstrat freigesetzt und ein Bild der Kügelchen im ungespannten Substrat aufgenommen (B). (C) Das Bild der Perlen im ungespannten Substrat zeigt die Kügelchen in ihrer ursprünglichen (grün) und verschobenen (rot) Position. Das EGFP-Signal des Wachstumskegels ist blau dargestellt. Kymographen (rechts) zeigen die Bewegungen der Perlen in Abständen von 3 s für eine Dauer von 147 s, gekennzeichnet durch die Pfeile in den Feldern 1 und 2. Die Perle in Bereich 2 ist eine Referenzperle. Maßstabsbalken: 2 μm für (A), (B) und (C, links) und 1 μm für (C, Mitte). Siehe auch Ergänzende Datei 4. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
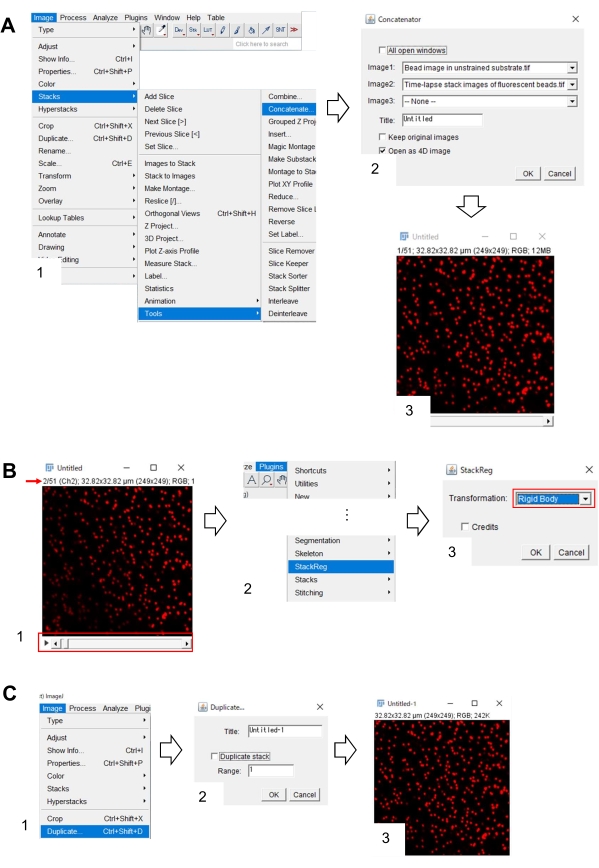
Abbildung 7: Schritte zur Korrektur der x-y-Position des Perlenbildes in unbelastetem Substrat unter Verwendung von Fidschi. (A) Verketten Sie das Perlenbild in ungespanntem Substrat mit dem Zeitraffer-Stapelbild fluoreszierender Perlen. (1) Wählen Sie Bild > Stapel > Werkzeuge > verketten. (2) Wählen Sie das Bild der Perlen in ungespanntem Substrat und das Zeitraffer-Stapelbild der fluoreszierenden Perlen in Bild 1 bzw. Bild2 aus. Klicken Sie auf OK. (3) Das Perlenbild im ungespannten Substrat wird dem Zeitraffer-Stapelbild hinzugefügt. (B) Korrigieren Sie die x-y-Position des fluoreszierenden Perlenbildes. (1) Verwenden Sie die Bildlaufleiste (roter Rahmen), um den zweiten Frame (roter Pfeil) im Bildstapel auszuwählen. (2) Wählen Sie Plugins > StackReg. (3) Wählen Sie Starrkörper aus der Dropdown-Liste (roter Rahmen) und klicken Sie auf OK. Die Korrektur der x-y-Position beginnt. (C) Speichern Sie das x-y-positionskorrigierte Perlenbild. Nachdem Sie das erste Bild des positionskorrigierten x-y-Stapelbilds ausgewählt haben, (1) wählen Sie Bild > Duplizieren. (2) Geben Sie 1 in den Bereich ein und deaktivieren Sie Stack duplizieren. Klicken Sie dann auf OK. (3) Das x-y-positionskorrigierte Perlenbild im ungespannten Substrat erscheint auf dem Bildschirm. Speichern Sie dieses Bild als TIFF-Datei. Ergänzende Datei 6 hilft den Ermittlern, die oben beschriebene Methodik zu üben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
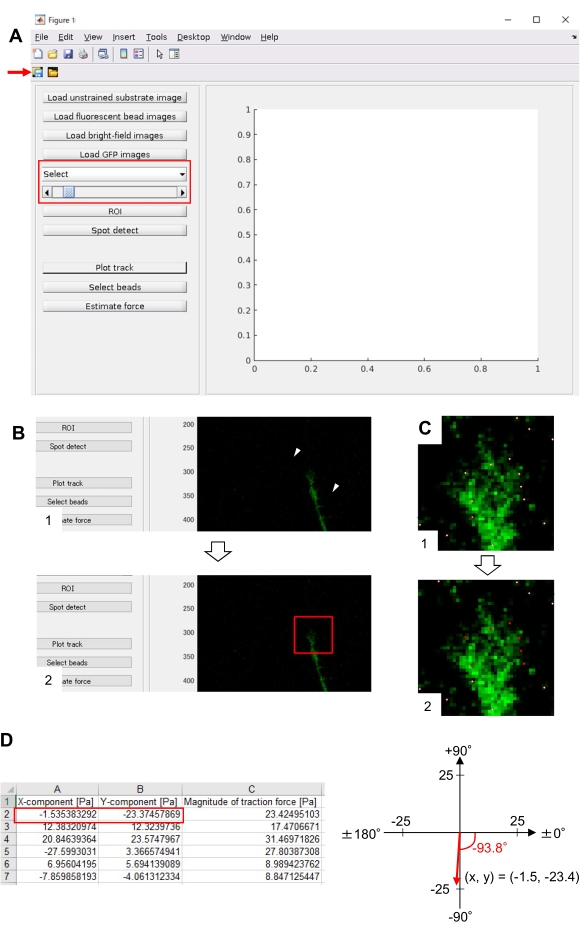
Abbildung 8: Analyse der Zugkraft unter einem neuronalen Wachstumskegel unter Verwendung eines Open-Source-Codes zur Traktionskraftanalyse. (A) GUI zur Analyse der Zugkraft. Zeitrafferbilder, die auf der GUI ausgewählt wurden, können aus der Dropdown-Liste und mit dem schieberegler (rote Box) bestätigt werden. (B) Wählen Sie eine Region aus, die den Wachstumskegel enthält. (1) Klicken Sie in der GUI auf ROI . Geben Sie mit dem Mauszeiger zwei Punkte (Pfeilspitzen) auf dem Zellenbild an. (2) Ein rotes Kästchen erscheint auf dem Zellenbild. Zwei Klicks bestimmen die Positionen von zwei Ecken. (C) Wählen Sie die erkannten Perlen (weiße Punkte) unter dem Wachstumskegel aus. (1) Klicken Sie in der GUI auf Perlen auswählen und markieren Sie einen polygonalen Bereich, der den Wachstumskegel enthält, indem Sie darauf klicken. Drücken Sie die Eingabetaste . (2) Die weißen Punkte innerhalb des polygonalen Bereichs ändern sich in eine rote Farbe. (D) Berechnete Ergebnisse der Richtung und des Umfangs der Zugkraft. Das rote Kästchen in der Tabelle stellt die x- und y-Komponenten des Kraftvektors dar, die durch die Zugkraftanalyse geschätzt werden. Auf der x-y-Koordinate im rechten Bereich zeigt der vom Wachstumskegel erzeugte Kraftvektor auf -93,8° gegen die x-Achse; diese Orientierung ist auf die Rückseite des Wachstumskegels in (C) gerichtet. Ergänzende Datei 6 hilft den Ermittlern, die oben beschriebene Methodik zu üben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Datei 1: Rezepte von Lösungen und Medien, die in dieser Studie verwendet werden. Siehe Text für eine detaillierte Verwendung. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: Fluoreszenzbildgebung von Lifeact und fluoreszierende Speckle-Bildgebung von HaloTag-Aktin in einem Nervenwachstumskegel. Lifeact (grün) und HaloTag-Aktin (Magenta). Die Bilder wurden alle 3 s für eine Gesamtdauer von 147 s aufgenommen. Maßstabsleiste: 2 μm. Siehe auch Abbildung 3. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: Praxisdaten zur Quantifizierung der F-Aktin-Strömungsgeschwindigkeit und der Aktinpolymerisationsrate. Ein Mehrkanal-Zeitraffer-Stack-Bild von Lifeact (grün) und HaloTag-Aktin (Magenta). Siehe auch Abbildung 3. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 4: Ein Force-Mapping-Video, das die Zugkraft an einem neuronalen Wachstumskegel erkennt. Die ursprünglichen (grün) und verschobenen (rot) Positionen der Perlen. Das EGFP-Signal im Wachstumskegel ist blau dargestellt. Bilder wurden alle 3 s für 147 s aufgenommen. Maßstabsleiste: 2 μm. Siehe auch Abbildung 6. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 5: Zugkraftanalysecode. Eine detaillierte Verwendung finden Sie in Schritt 8.12. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 6: Praxisdaten zur Quantifizierung der Zugkraft. Ein einkanaliges RGB-Bild der Perlen in ungespanntem Substrat und einkanaliger Zeitraffer-Stack RGB-Bilder von fluoreszierenden Perlen unter einem Wachstumskegel, EGFP und Hellfeld. Siehe auch Abbildungen 7 und 8. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die in dieser Studie beschriebenen Protokolle verwenden kommerziell verfügbare Materialien und Mikroskopiegeräte, die routinemäßig in allen Labors, Instituten und Universitäten zu finden sind. Daher können Forscher die vorliegende Einzelfleckenbildgebung und Zugkraftmikroskopie leicht in ihre Studien einbeziehen.
Die Speckle-Bildgebung kann Aktinpolymerisation und Kupplungskopplung analysieren. Darüber hinaus kann die Speckle-Bildgebung den retrograden Fluss von Kupplungsmolekülen wie Shootin1 und Cortactin überwachen, die mit dem retrograden F-Aktin-Fluss interagieren. Mit einem TIRF-Mikroskop kann auch der retrograde Fluss des Zelladhäsionsmoleküls L1-CAM überwacht werden23,41; L1-CAM erfährt ein Griff- und Schlupfverhalten, das die Effizienz der Kupplungskupplung widerspiegelt23,41. Obwohl die vorliegende Studie das TMR-HaloTag-System für die Speckle-Bildgebung verwendet, sind auch andere fluoreszierende Proteine wie EGFP und monomeres rot fluoreszierendes Protein in der Analyse verfügbar16,18,20,23,24,27,39. Die Grundlagen für die Visualisierung von Aktinflecken sind ein niedriges Expressionsniveau von fluoreszierendem Aktin und die Beleuchtung einer minimalen Fläche (Abbildung 2). In diesem Protokoll werden Lifeact- und HaloTag-Aktinsignale nacheinander erfasst. Da der retrograde Aktinfluss relativ langsam ist (4,5 ± 0,1 μm/min)24, wird die Analyse des retrograden F-Aktinflusses und der Aktinpolymerisation durch die sequentielle Bildaufnahme verschiedener Fluoreszenzkanäle (~1 s-Intervall) nicht beeinflusst. Lifeact ist ein weit verbreiteter F-Aktin-Marker, kann aber mit Aktin-bindenden Proteinen konkurrieren47. Von weiterer Bedeutung ist, dass Lifeact die Aktindynamik verändern kann, wodurch F-Aktinstrukturen und die Zellmorphologie beeinflusst werden47,48,49.
Die Zugkraftmikroskopie kann Kräfte erkennen, um den Wachstumskegelvorschub voranzutreiben. Durch die Montage der Neuronen in einer extrazellulären Matrix können die Forscher auch Kräfte analysieren, die in einer Semi-3D-Umgebung erzeugt werden11. Die Bildgebung mit hoher Vergrößerung ist wichtig für die genaue Quantifizierung der Zugkraft, da Wachstumskegel schwache Zugkräfte erzeugen7. Obwohl auch andere Methoden mit Nanosäulen oder spannungsempfindlichen Biosensoren zur Messung der Zugkraft verwendet werden50,51, ist die PAA-Gel-basierte Methode sehr anpassungsfähig und ermöglicht die Einstellung der Substratsteifigkeit durch Variation der Konzentrationen von Acrylamid und Bisacrylamid41,44,52. In diesem Protokoll wird das PAA-Gel in einer Endkonzentration von 3,75% Acrylamid und 0,03% Bisacrylamid hergestellt; Der Elastizitätsmodul beträgt ~ 270 Pa22 und diese Steifigkeit liegt im Bereich des Hirngewebes (100-10.000 Pa) 53,54,55. Aufgrund der Dicke des PAA-Gels (~100 μm) schränkt diese Methode die Verwendung von Linsen mit hoher Vergrößerung während der Mikroskopie ein. Um Bilder mit hoher Vergrößerung zu erhalten, sollten Forscher die Zoomfunktion in einem konfokalen Laserscanning-Mikroskop verwenden.
Zusammenfassend lässt sich sagen, dass die vorliegende Speckle-Bildgebung und die Zugkraftmikroskopie quantitative Analysen der Schlüsselereignisse in der Krafterzeugung ermöglichen. Diese Informationen werden von unschätzbarem Wert sein, um das Verständnis der Mechanismen zu verbessern, die dem Fortschritt und der Navigation des Wachstumskegels zugrunde liegen.
Offenlegungen
Die Autoren haben nichts preiszugeben.
Danksagungen
Diese Forschung wurde teilweise von AMED unter der Fördernummer 21gm0810011h0005 (N.I. und Y.S.), JSPS KAKENHI (JP19H03223, N.I.) und JSPS Grants-in-Aid for Early-Career Scientists (JP19K16258, T.M.), der Osaka Medical Research Foundation for Incurable Diseases (T.M.) und dem NAIST Next Generation Interdisciplinary Research Project (Y.S.) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.5% trypan blue stain solution | Nacalai | 29853-34 | |
| 3-aminopropyltrimethyoxysilane | Sigma | 281778-100ML | |
| Acrylamide monomer | Nacalai | 00809-85 | |
| Ammonium persulphate | Cytiva | 17-1311-1 | |
| Axio Observer Z1 | Zeiss | 431007-9902-000 | Epi-fluorescence microscope (single speckle imaging) |
| B-27 supplement (50x) | Thermo Fisher Scientific | 17504-044 | |
| Bovine serum albmine | Sigma | A7906-10G | |
| C-Apochromat 63x/1.2 W Corr | Zeiss | 421787-9970-799 | Objective lens (traction force microscopy) |
| Coverslip (diameter 18 mm) | Matsunami | C018001 | |
| D-glucose | Nacalai | 16806-25 | |
| DNaseI | Sigma | DN25-100MG | |
| Fetal bovine serum | Thermo Fisher Scientific | 10270-106 | |
| Fiji | Open source software package | https://imagej.net/software/fiji/ | |
| FluoSpheres carboxylate-modified 0.2 mm, red (580/605), 2% solid | Thermo Fisher Scientific | F8810 | carboxylate-modified microspheres |
| Glass bottom dish (14 mm diameter) | Matsunami | D1130H | |
| Glass bottom dish (27 mm diameter) | Matsunami | D1140H | |
| Glutaraldehyde solution | Sigma | G5882-10X10ML | |
| HaloTag TMR ligand | Promega | G8251 | |
| HBO103 W/2 | Osram | 4050300382128 | Mercury lamp (single speckle imaging) |
| Image Processing Toolbox | MathWork | https://www.mathworks.com/products/image.html | |
| Laminin solution from mouse EHS tumor | Wako | 120-05751 | |
| Leibovitz’s L-15 medium | Thermo Fisher Scientific | 11415064 | |
| L-glutamine | Nacalai | 16919-42 | |
| LSM710 | Zeiss | N/A | Conforcal laser microscope (traction force microscopy) |
| MATLAB2018a | MathWork | https://www.mathworks.com/products/new_products/release2018a.html | |
| Mouse C57BL/6 | Japan SLC | N/A | |
| Mouse neuron nucleofector kit | Lonza | VPG-1001 | |
| N,N,N’,N’-tetramethylethylenediamine (TEMED) | Nacalai | 33401-72 | |
| N,N’-methylenebisacrylamide | Nacalai | 22402-02 | |
| Neurobasal medium | Thermo Fisher Scientific | 21103-049 | |
| Nucleofector I | Amaxa | AAD-1001 | Electroporation apparatus |
| ORCA Flash 4.0 V2 | Hamamatsu | C11440-22CU | CMOS camera (single speckle imaging) |
| Papain | Nacalai | 26036-34 | |
| Parallel Computing Toolbox | MathWork | https://www.mathworks.com/products/parallel-computing.html | |
| pEGFP-C1 | Clontech | 1528177 | |
| Penicillin-streptomycin (100x) | Nacalai Tesque | 26253-84 | |
| pFN21A-HaloTag-actin | (Minegishi et al., 2018) | N/A | |
| Phosphate buffered saline (PBS) pH 7.4 (10x) | Thermo Fisher Scientific | 70011-044 | |
| Plan-Apochromat 100x/1.4 Oil | Zeiss | 420790-9901-000 | Objective lens (single speckle imaging) |
| pmNeonGreen-N1-Lifeact | (Kastian et al., 2021) | N/A | |
| Poly-D-lysine hydrobromide | Sigma | P6407-5MG | |
| Slulfo-SAMPHA | Thermo Fisher Scientific | 22589 | |
| Sodium dodecyl sulfate | Nacalai | 08933-05 | |
| Sodium hydrate (NaOH) | Nacalai | 31511-05 | |
| Steel ball | Sako tekkou | N/A | Microshpere to determin PAA gel rigidity. 0.6 mm diameter, 7.87 g/cm3. |
| ZEN2009 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (traction force microscopy) |
| ZEN2012 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (single speckle imaging) |
Referenzen
- TessierLavigne, M., Goodman, C. S. The molecular biology of axon guidance. Science. 274, 1123-1133 (1996).
- Marin, O., Valiente, M., Ge, X., Tsai, L. H. Guiding neuronal cell migrations. Cold Spring Harbor Perspectives in Biology. 2, 001834 (2010).
- Vitriol, E. A., Zheng, J. Q. Growth cone travel in space and time: the cellular ensemble of cytoskeleton, adhesion, and membrane. Neuron. 73 (6), 1068-1081 (2012).
- Cooper, J. A. Cell biology in neuroscience: mechanisms of cell migration in the nervous system. Journal of Cell Biology. 202, 725-734 (2013).
- Dupraz, S., et al. RhoA controls axon extension independent of specification in the developing brain. Current Biology. 29, 3874-3886 (2019).
- Suter, D. M., Miller, K. E. The emerging role of forces in axonal elongation. Progress in Neurobiology. 94, 91-101 (2011).
- Franze, K. Integrating chemistry and mechanics: the forces driving axon growth. Annual Review of Cell and Developmental Biology. 36, 61-83 (2020).
- Mitchison, T., Kirschner, M. Cytoskeletal dynamics and nerve growth. Neuron. 1, 761-772 (1988).
- Suter, D. M., Forscher, P. Substrate-cytoskeletal coupling as a mechanism for the regulation of growth cone motility and guidance. Journal of Neurobiology. 44, 97-113 (2000).
- Lowery, L. A., Van Vactor, D. The trip of the tip: understanding the growth cone machinery. Nature Reviews Molecular Cell Biology. 10, 332-343 (2009).
- Minegishi, T., et al. Shootin1b mediates a mechanical clutch to produce force for neuronal migration. Cell Reports. 25, 624-639 (2018).
- Minegishi, T., Inagaki, N. Forces to drive neuronal migration steps. Frontiers in Cell and Developmental Biology. 8, 863 (2020).
- Forscher, P., Smith, S. J. Actions of cytochalasins on the organization of actin filaments and microtubules in a neuronal growth cone. Journal of Cell Biology. 107, 1505-1516 (1988).
- Suter, D. M., Forscher, P. An emerging link between cytoskeletal dynamics and cell adhesion molecules in growth cone guidance. Current Opinion in Neurobiology. 8, 106-116 (1998).
- Pollard, T. D., Borisy, G. G. Cellular motility driven by assembly and disassembly of actin filaments. Cell. 112, 453-465 (2003).
- Waterman-Storer, C. M., Desai, A., Bulinski, J. C., Salmon, E. D. Fluorescent speckle microscopy, a method to visualize the dynamics of protein assemblies in living cells. Current Biology. 8, 1227-1230 (1998).
- Katoh, K., Hammar, K., Smith, P. J., Oldenbourg, R. Birefringence imaging directly reveals architectural dynamics of filamentous actin in living growth cones. Molecular Biology of the Cell. 10, 197-210 (1999).
- Watanabe, N., Mitchison, T. J. Single-molecule speckle analysis of actin filament turnover in lamellipodia. Science. 295, 1083-1086 (2002).
- Medeiros, N. A., Burnette, D. T., Forscher, P. Myosin II functions in actin-bundle turnover in neuronal growth cones. Nature Cell Biology. 8, 215-226 (2006).
- Shimada, T., et al. Shootin1 interacts with actin retrograde flow and L1-CAM to promote axon outgrowth. Journal of Cell Biology. 181, 817-829 (2008).
- He, M., Zhang, Z. H., Guan, C. B., Xia, D., Yuan, X. B. Leading tip drives soma translocation via forward F-actin flow during neuronal migration. Jounal of Neuroscience. 30, 10885-10898 (2010).
- Toriyama, M., Kozawa, S., Sakumura, Y., Inagaki, N. Conversion of a signal into forces for axon outgrowth through Pak1-mediated shootin1 phosphorylation. Current Biology. 23, 529-534 (2013).
- Abe, K., et al. Grip and slip of L1-CAM on adhesive substrates direct growth cone haptotaxis. Proceedings of the National Academy of Sciences of the United States of America. 115, 2764-2769 (2018).
- Baba, K., et al. Gradient-reading and mechano-effector machinery for netrin-1-induced axon guidance. Elife. 7, 34593 (2018).
- Nichol, R. I., Hagen, K. M., Lumbard, D. C., Dent, E. W., Gomez, T. M. Guidance of axons by local coupling of retrograde flow to point contact adhesions. Journal of Neuroscience. 36, 2267-2282 (2016).
- Shekarabi, M., et al. Deleted in colorectal cancer binding netrin-1 mediates cell substrate adhesion and recruits Cdc42, Rac1, Pak1, and N-WASP into an intracellular signaling complex that promotes growth cone expansion. Journal of Neuroscience. 25, 3132-3141 (2005).
- Kubo, Y., et al. Shootin1-cortactin interaction mediates signal-force transduction for axon outgrowth. Journal of Cell Biology. 210, 663-676 (2015).
- Huber, A. B., Kolodkin, A. L., Ginty, D. D., Cloutier, J. F. Signaling at the growth cone: ligand-receptor complexes and the control of axon growth and guidance. Annual Review of Neuroscience. 26, 509-563 (2003).
- Kolodkin, A. L., Tessier-Lavigne, M. Mechanisms and molecules of neuronal wiring: a primer. Cold Spring Harbor Perspectives in Biology. 3, 001727 (2011).
- Ayala, R., Shu, T., Tsai, L. H. Trekking across the brain: the journey of neuronal migration. Cell. 128, 29-43 (2007).
- Tatavarty, V., Kim, E. J., Rodionov, V., Yu, J. Investigating sub-spine actin dynamics in rat hippocampal neurons with super-resolution optical imaging. PLoS One. 4, 7724 (2009).
- Frost, N. A., Shroff, H., Kong, H., Betzig, E., Blanpied, T. A. Single-molecule discrimination of discrete perisynaptic and distributed sites of actin filament assembly within dendritic spines. Neuron. 67, 86-99 (2010).
- Chazeau, A., et al. Nanoscale segregation of actin nucleation and elongation factors determines dendritic spine protrusion. EMBO Journal. 33, 2745-2764 (2014).
- Garcia, M., et al. Two-tiered coupling between flowing actin and immobilized N-cadherin/catenin complexes in neuronal growth cones. Proceedings of the National Academy of Sciences of the United States of America. 112, 6997-7002 (2015).
- Swaminathan, V., et al. Actin retrograde flow actively aligns and orients ligand-engaged integrins in focal adhesions. Proceedings of the National Academy of Sciences of the United States of America. 114, 10648-10653 (2017).
- Tsai, T. Y., et al. Efficient front-rear coupling in neutrophil chemotaxis by dynamic myosin II localization. Developmental Cell. 49, 189-205 (2019).
- Zhang, X. F., et al. Regulation of axon growth by myosin II-dependent mechanocatalysis of cofilin activity. Journal of Cell Biology. 218 (7), 2329-2349 (2019).
- Reversat, A., et al. Cellular locomotion using environmental topography. Nature. 582, 582-585 (2020).
- Katsuno, H., et al. Actin migration driven by directional assembly and disassembly of membrane-anchored actin filaments. Cell Reports. 12, 648-660 (2015).
- Urasaki, A., et al. Shootins mediate collective cell migration and organogenesis of the zebrafish posterior lateral line system. Scientific Reports. 9, 12156 (2019).
- Abe, K., et al. Mechanosensitive axon outgrowth mediated by L1-laminin clutch interface. Biophysical Journal. 120, 3566-3576 (2021).
- Kastian, R. F., et al. Shootin1a-mediated actin-adhesion coupling generates force to trigger structural plasticity of dendritic spines. Cell Reports. 35, 109130 (2021).
- Riedl, J., et al. Lifeact: a versatile marker to visualize F-actin. Nature Methods. 5, 605-607 (2008).
- Chan, C. E., Odde, D. J. Traction dynamics of filopodia on compliant substrates. Science. 322, 1687-1691 (2008).
- Wang, Y. L., Pelham, R. J. Preparation of a flexible, porous polyacrylamide substrate for mechanical studies of cultured cells. Methods in Enzymology. 298, 489-496 (1998).
- Li, Y., Hu, Z., Li, C. New method for measuring poisson's ratio in polymer gels. Journal of Applied Polymer Science. 50, 1107-1111 (1993).
- Belyy, A., Merino, F., Sitsel, O., Raunser, S. Structure of the Lifeact-F-actin complex. PLoS Biology. 18, 3000925 (2020).
- Flores, L. R., Keeling, M. C., Zhang, X., Sliogeryte, K., Gavara, N. Lifeact-GFP alters F-actin organization, cellular morphology and biophysical behaviour. Scientific Reports. 9, 3241 (2019).
- Kumari, A., Kesarwani, S., Javoor, M. G., Vinothkumar, K. R., Sirajuddin, M. Structural insights into actin filament recognition by commonly used cellular actin markers. EMBO Journal. 39, 104006 (2020).
- du Roure, O., et al. Force mapping in epithelial cell migration. Proceedings of the National Academy of Sciences of the United States of America. 102, 2390-2395 (2005).
- Grashoff, C., et al. Measuring mechanical tension across vinculin reveals regulation of focal adhesion dynamics. Nature. 466, 263-266 (2010).
- Koch, D., Rosoff, W. J., Jiang, J., Geller, H. M., Urbach, J. S. Strength in the periphery: growth cone biomechanics and substrate rigidity response in peripheral and central nervous system neurons. Biophysical Journal. 102 (3), 452-460 (2012).
- Barnes, J. M., Przybyla, L., Weaver, V. M. Tissue mechanics regulate brain development, homeostasis and disease. Journal of Cell Science. 130, 71-82 (2017).
- Moore, S. W., Roca-Cusachs, P., Sheetz, M. P. Stretchy proteins on stretchy substrates: the important elements of integrin-mediated rigidity sensing. Developmental Cell. 19, 194-206 (2010).
- Tyler, W. J. The mechanobiology of brain function. Nature Reviews Neuroscience. 13, 867-878 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten