Method Article
Analyses de la dynamique de l’actine, de l’accouplement d’embrayage et de la force de traction pour l’avancement du cône de croissance
Dans cet article
Résumé
Pour avancer, les cônes de croissance doivent exercer des forces de traction contre l’environnement extérieur. La génération de forces de traction dépend de la dynamique de l’actine et de l’accouplement de l’embrayage. La présente étude décrit des méthodes d’analyse de la dynamique de l’actine, du couplage d’embrayage et des forces de traction pour l’avancement du cône de croissance.
Résumé
Pour établir des réseaux fonctionnels, les neurones doivent migrer vers leurs destinations appropriées, puis étendre les axones vers leurs cellules cibles. Ces processus dépendent des progrès des cônes de croissance situés à l’extrémité des neurites. Les cônes de croissance axonale génèrent des forces motrices en détectant leur microenvironnement local et en modulant la dynamique du cytosquelette et le couplage actine-adhérence (couplage embrayage). Des décennies de recherche ont conduit à l’identification de molécules de guidage, de leurs récepteurs et de cascades de signalisation en aval pour réguler la migration neuronale et le guidage axonal; cependant, les mécanismes moléculaires nécessaires pour générer des forces pour stimuler l’avancement et la navigation du cône de croissance commencent tout juste à être élucidés. Au bord d’attaque des cônes de croissance neuronale, les filaments d’actine subissent un écoulement rétrograde, qui est alimenté par la polymérisation de l’actine et la contraction de l’actomyosine. Un couplage d’embrayage entre l’écoulement rétrograde de F-actine et le substrat adhésif génère des forces de traction pour l’avancement du cône de croissance. La présente étude décrit un protocole détaillé pour la surveillance de l’écoulement rétrograde de la F-actine par imagerie à moucheture unique. Il est important de noter que lorsqu’elle est combinée avec un marqueur de F-actine Lifeact, cette technique peut quantifier 1) le taux de polymérisation de la F-actine et 2) l’efficacité du couplage d’embrayage entre l’écoulement rétrograde de la F-actine et le substrat adhésif. Les deux sont des variables critiques pour générer des forces pour l’avance et la navigation du cône de croissance. En outre, la présente étude décrit un protocole détaillé de microscopie à force de traction, qui peut quantifier 3) la force de traction générée par les cônes de croissance. Ainsi, en couplant les analyses de l’imagerie à une seule moucheture et de la microscopie à force de traction, les chercheurs peuvent surveiller la mécanique moléculaire sous-jacente à l’avancement et à la navigation des cônes de croissance.
Introduction
Dans le cerveau des vertébrés en développement, les neurones subissent des migrations soigneusement organisées et projettent des axones vers des partenaires synaptiques appropriés pour établir des réseaux neuronaux fonctionnels1,2,3. Les cônes de croissance, qui sont des structures sensorielles et mobiles situées à l’extrémité des neurites, déterminent la vitesse et la direction de la migration neuronale et de l’excroissance axonale3,4,5. Étant donné que les neurones sont entourés d’environnements serrés, les cônes de croissance doivent exercer des forces contre leur environnement pour avancer6,7. Pour comprendre les mécanismes sous-jacents à la migration neuronale et au guidage axonal, des analyses de la mécanique moléculaire pour l’avancement du cône de croissance sont essentielles.
Des décennies d’analyse ont révélé que la force de traction pour stimuler l’avance du cône de croissance est générée par le mécanisme d’embrayage; On pense que ce mécanisme fonctionne non seulement dans le cône de croissance axonal, mais aussi dans le cône de croissance du processus principal des neurones migrateurs8,9,10,11,12. À savoir, les filaments d’actine (F-actines) dans les cônes de croissance polymérisent au bord d’attaque et se dépolymérent proximalement, poussant la membrane de bord d’attaque13,14,15. La force résultante, en conjonction avec la contraction de l’actomyosine, induit un mouvement vers l’arrière des F-actines appelé écoulement rétrograde7,11,16,17,18,19,20,21. Les molécules d’adhésion de l’embrayage et de la cellule interviennent dans le couplage mécanique entre l’écoulement rétrograde de la F-actine et le substrat adhésif et transmettent la force de l’écoulement de la F-actine sur le substrat, générant ainsi une force de traction pour l’avance du cône de croissance7,8,9,11,12,22 . Parallèlement, le couplage actine-substrat réduit la vitesse d’écoulement de la F-actine et convertit la polymérisation de l’actine en force pour faire saillie la membrane de pointe9,10.
Les cônes de croissance axonale détectent les signaux chimiques locaux et les transduisent en une force motrice directionnelle pour la navigation des cônes de croissance3,23,24,25. Par exemple, une molécule de guidage axonale nétrine-1 stimule son récepteur supprimé dans le cancer colorectal (DCC) et active la protéine de contrôle de la division cellulaire 42 (Cdc42) et le substrat 1 de la toxine botulique C3 liée à Ras (Rac1), ainsi que leur kinase 1 activée par la kinase p21 en aval (Pak1)26. Cdc42 et Rac1 favorisent 1) la polymérisation de l’actine, et Pak1 phosphoryle une molécule d’embrayage shootin122,26. Shootin1 interagit avec le flux rétrograde de F-actine via une protéine de liaison à l’actine cortactine27. Shootin1 interagit également avec la molécule d’adhésion cellulaire L1 (L1-CAM)20,24. La phosphorylation de Shootin1 augmente les affinités de liaison pour la cortactine et la L1-CAM, et améliore le couplage d’embrayage médié par la shootin1 24,27. Dans le cône de croissance, les activations asymétriques de la polymérisation de l’actine et du couplage d’embrayage augmentent 3) la force de traction sur le côté de la source de nétrine 1, générant ainsi une force motrice directionnelle pour le tournage du cône de croissance (Figure 1)24. Des recherches intensives au cours des dernières décennies en ce qui concerne la migration neuronale et le guidage axonal ont amélioré la compréhension des molécules de guidage, de leurs récepteurs et des cascades de signalisation en aval associées2,10,28,29,30. Cependant, les mécanismes moléculaires pour générer des forces pour l’avancement du cône de croissance commencent tout juste à être élucidés; cela peut être attribué à l’utilisation limitée des protocoles pour les analyses mécanobiologiques.
La présente étude décrit un protocole détaillé pour la surveillance de l’écoulement rétrograde de la F-actine par imagerie à moucheture unique16,18. La surveillance de l’écoulement rétrograde de la F-actine a été largement réalisée à l’aide de la microscopie à super-résolution, de la microscopie confocale à disque rotatif et de la microscopie à fluorescence par réflexion par interférence totale (TIRF)25,31,32,33,34,35,36,37,38 . Le protocole de la présente étude, cependant, utilise un microscope à épifluorescence standard et est donc facilement adoptable11,16,18,20,22,23,24,27,39,40,41,42. Lorsqu’elle est combinée au marquage F-actine par Lifeact43, l’imagerie à tache unique permet de quantifier le taux de polymérisation de l’actine et l’efficacité du couplage d’embrayage entre le flux rétrograde de F-actine et le substrat adhésif39,42. La présente étude décrit en outre un protocole détaillé de microscopie à force de traction utilisant un gel de polyacrylamide (PAA) incorporé dans des billes fluorescentes11,22,23,24,27,39,41,42,44. Cette méthode détecte et quantifie la force de traction sous le cône de croissance en surveillant les mouvements de billes induits par la force44,45. Un code d’analyse de la force de traction open source est fourni et la méthode de quantification de la force de traction pendant la migration du cône de croissance est expliquée en détail. À l’aide de l’imagerie à faisceau unique et de la microscopie à force de traction, la compréhension de la mécanique moléculaire sous-jacente à la migration et à la navigation des cônes de croissance sera facilitée. Ces techniques sont également applicables pour analyser la mécanique moléculaire sous-jacente à l’élargissement de la colonne vertébrale dendritique, qui est connu pour être important dans l’apprentissage et la mémoire42.
Protocole
Toutes les expériences utilisant des animaux de laboratoire ont été réalisées avec le Comité institutionnel de soins et d’utilisation des animaux de l’Institut des sciences et de la technologie de Nara. Les chercheurs devraient suivre les lignes directrices établies par leurs comités institutionnels et nationaux de réglementation des animaux pour le soin et l’utilisation des animaux de laboratoire.
1. Préparation des solutions et des supports
- Préparer les solutions et les milieux de culture tels que résumés dans le fichier supplémentaire 1
2. Préparation de substrats revêtus de poly-D-lysine (PDL)/laminine
- Enduire les plats à fond de verre de 14 mm de diamètre avec 100 μg/mL de PDL, dissous dans une solution saline tamponnée au phosphate (PBS, pH 7,4), et incuber pendant la nuit dans un incubateur humidifié à 37 °C.
REMARQUE: Ne pas sécher les surfaces vitrées après cette étape - Retirez la solution PDL et pipetez 1 mL de PBS sur le verre pendant 10 s trois fois.
- Enduire les plats de laminine de 5 μg/mL, dissous dans du PBS, et incuber toute la nuit dans un incubateur humidifié à 37 °C.
- Retirer la solution de laminine et pipeter 1 mL de PBS sur le verre pendant 10 s trois fois.
- Retirez le PBS et placez 0,5 mL du milieu neurobasal sur les surfaces vitrées. Conserver la vaisselle dans un incubateur humidifié à 37 °C.
REMARQUE: Les plats enduits de PDL / laminine peuvent être conservés pendant 2-3 jours dans un incubateur humidifié à 37 ° C.
3. Dissection et dissociation de l’hippocampe
- Euthanasier une souris enceinte par luxation cervicale.
REMARQUE : La présente étude utilise des souris disponibles dans le commerce (voir tableau des matériaux). Les chercheurs devraient utiliser des souris élevées et traitées sans cruauté dans un environnement stérilisé. - Disséquez les embryons de souris du jour 16 (E16) et placez-les sur de la glace.
- Dans une hotte à écoulement laminaire, disséquez les cerveaux avec des ciseaux et placez-les sur un plat stérile de 10 cm contenant 10 mL de solution de dissection glacée.
- Avec un stéréomicroscope à dissection, décollez soigneusement les méninges des hémisphères cérébraux, puis disséquez l’hippocampe à l’aide d’une pince.
- Transférer l’hippocampe dans 5 mL de solution de digestion glacée dans un tube de 15 mL. Incuber l’hippocampe au bain-marie pendant 20 min à 37 °C.
- Retirer la solution de digestion et ajouter 3 mL de solution de dissection.
- Pipettez doucement l’hippocampe quatre fois avec une pipette Pasteur.
- Incuber l’hippocampe au bain-marie pendant 20 min à 37 °C.
- Transférer la suspension de la cellule dans un tube de centrifugeuse de 50 mL. Ajouter 3 mL de nouvelle solution de dissection au tissu non dissocié.
- Répétez les étapes 3.7-3.9 jusqu’à ce que les hippocampes soient complètement dissociés.
- Retirez les agrégats flottants d’ADN de la suspension cellulaire en tourbillonnant et en aspirant avec une pipette Pasteur. Centrifuger la suspension de la cellule à 180 x g pendant 20 min à 4 °C.
REMARQUE: L’ADN dérivé de cellules endommagées perturbera le processus de centrifugation. Si les neurones restent dans le surnageant, centrifugez à nouveau la suspension cellulaire après avoir soigneusement retiré l’ADN. - Équilibrer le milieu neurobasal contenant 10 % de sérum fœtal bovin (FBS), de pénicilline (100 U/mL) et de streptomycine (100 μg/mL) dans un incubateur humidifié à 37 °C avec 5 % de CO2.
4. Transfection et culture des neurones
- Retirez le surnageant et ajoutez 5 mL de PBS glacé dans le tube de 50 mL. Remettre en suspension la pastille de cellule par pipetage doux.
- Transférer 10 μL de la suspension cellulaire dans un tube de microcentrifugation, ajouter 10 μL de solution de bleu de trypan à 0,5%, puis compter le nombre de cellules avec un hémocytomètre.
- Centrifuger la suspension de la cellule dans le tube de 50 mL à 180 x g pendant 20 min à 4 °C.
- En attendant, aliquote pour la transfection, 1 μg de pFN21A-HaloTag-actine et 3 μg de pmNeonGreen-N1-Lifeact pour 1 x 106 cellules, dans un tube de microcentrifugation.
- Après centrifugation, retirer le surnageant et ajouter 100 μL de milieu d’électroporation (fourni par kit de transfection) dans le tube de 50 mL. Remettre en suspension la pastille de cellule par pipetage.
- Mélanger la suspension cellulaire avec la solution d’ADN aliquoted et transférer le mélange dans une cuvette (fournie par le kit de transfection).
REMARQUE: Puisque les bulles d’air perturbent l’électroporation, retirez-les de la suspension de la cellule. - Insérez la cuvette dans l’appareil d’électroporation (Table des matériaux) et effectuez l’électroporation à l’aide du programme O-005.
- Ajouter immédiatement 1 mL du milieu neurobasal préchauffé et équilibré contenant 10 % de FBS, de pénicilline (100 U/mL) et de streptomycine (100 μg/mL) à la cuvette.
- Transférer la suspension de la cellule dans un tube de centrifugeuse de 15 mL à l’aide d’une pipette en plastique (fournie par le kit de transfection).
- Transférer 10 μL de la suspension cellulaire dans un microtube, ajouter 10 μL de solution de bleu de trypan à 0,5%, puis compter le nombre de cellules avec un hémocytomètre.
- Retirez les plats à fond de verre enduits de PDL / laminine de l’incubateur et retirez le milieu neurobasal.
- Pipette 0,5 mL de la suspension cellulaire contenant 2,0 x 105 cellules par plat et incuber dans un incubateur humidifié à 37 °C avec 5% de CO2 pendant 3 h.
- Remplacer le milieu par 0,5 mL de milieu neurobasal contenant 2 % de supplément de B-27, de glutamine (1 mM), de pénicilline (100 U/mL) et de streptomycine (100 μg/mL). Cultiver les neurones dans un incubateur humidifié pendant 3 jours à 37 °C avec 5% de CO2.
5. Imagerie par mouchetures uniques aux cônes de croissance neuronale
- Le jour in vitro (DIV) 3, traiter les neurones avec du ligand tétraméthyl-rhodamine (TMR) à une dilution de 1:2000 dilution dans un milieu de culture. Maintenir les neurones pendant 1 h à 37 °C avec 5% de CO2.
- Lavez le ligand TMR trois fois avec du PBS préchauffé.
- Retirer le PBS et pipeter 0,5 mL de milieu L-15 de Leibovitz réchauffé contenant 2 % de supplément de B-27, de glutamine (1 mM), de pénicilline (100 U/mL) et de streptomycine (100 μg/mL).
- Maintenir les neurones pendant 1 h à 37 °C.
- Allumez un microscope à épifluorescence et réglez l’incubateur sur scène à 37 °C.
REMARQUE: Le présent protocole utilise un microscope à épifluorescence équipé d’une caméra semi-conductrice à oxyde métallique complémentaire, d’un objectif d’immersion dans l’huile 100x / 1,40 NA, d’une lampe au mercure et d’un logiciel d’acquisition d’images (voir tableau des matériaux). D’autres microscopes à épifluorescence avec des spécifications équivalentes peuvent également être utilisés pour cette analyse. - Placez les neurones traités par ligand TMR dans la boîte à fond de verre de l’incubateur chauffé.
- Définissez les paramètres d’acquisition d’image comme suit : temps d’exposition, 500 ms pour les canaux fluorescents Lifeact et HaloTag-actine ; binning, 1 x 1 (0,065 μm × 0,065 μm par pixel); intervalle de temps, 3 s; durée, 50 images.
- Sélectionnez un cône de croissance qui exprime fortement Lifeact et exprime faiblement HaloTag-actin.
REMARQUE: L’expression de Lifeact doit être assez forte pour visualiser la morphologie du cône de croissance. L’expression de HaloTag-actine doit être faible et insuffisante pour être détectée (Figure 2A). Les niveaux d’expression des protéines fluorescentes peuvent être ajustés en faisant varier la quantité d’ADN pour la transfection. - Fermez le champ du diaphragme pour éclairer une zone minimale comprenant le cône de croissance (Figure 2B).
REMARQUE: L’éclairage d’une zone minimale réduit le signal de fond et augmente le rapport signal de fluorescence sur bruit (S / N), permettant la détection des taches d’actine (Figure 2B). Pour augmenter encore le rapport S / N, il est recommandé d’éclairer le cône de croissance sans réduire la lumière par des filtres à densité neutre. - Acquérir des images time-lapse (fichier supplémentaire 2). Enregistrez en tant qu’image de pile time-lapse multicanal (format tiff).
6. Quantifications de la vitesse d’écoulement et du taux de polymérisation de la F-actine à l’aide d’un logiciel de traitement et d’analyse d’images Fidji
REMARQUE : Reportez-vous au fichier supplémentaire 3 pour obtenir des données pratiques sur la quantification de la vitesse d’écoulement de la F-actine et du taux de polymérisation de l’actine.
- Ouvrez l’image de pile time-lapse multicanal sur Fidji.
- Sélectionnez Analyser > Définir l’échelle, puis définissez la taille en pixels des images.
- Trouvez une tache d’actine qui s’écoule rétrogradement pendant au moins cinq périodes dans un faisceau de F-actine dans les filopodes ou les lamellipodies.
- Sélectionnez Image > Transformer > Faire pivoter, puis ajustez l’angle des images de sorte que le faisceau F-actine pointe vers le haut (Figure 3A).
- Cliquez sur Rectangle dans la barre d’outils et dessinez une boîte qui comprend des taches d’actine et la pointe du faisceau F-actine (Figure 3B, 1 et 2).
REMARQUE: Les signaux des taches HaloTag-actine sont faibles. De plus, les signaux Lifeact aux extrémités distales du cône de croissance sont faibles et faibles (Figure 2B) parce que les extrémités distales du cône de croissance sont minces par rapport à la partie proximale. Les chercheurs devraient améliorer les signaux afin d’optimiser les quantifications de la vitesse d’écoulement et du taux de polymérisation de la F-actine (figure 3B). Bien que le signal Lifeact au niveau du cône de croissance proximal devienne saturé, cela ne perturbera pas la détermination de l’extrémité distale des F-actines (Figure 3E, ligne jaune). - Sélectionnez Image > Dupliquer et entrez les cinq délais (Figure 3B, 3-5).
- Sélectionnez Image > Stacks > Make Montage, puis entrez les paramètres suivants : Colonnes, 5 ; Lignes, 1; Facteur d’échelle, 1.
- Cliquez sur OK. Le montage d’image apparaîtra à l’écran.
- Sélectionnez Image > Couleur > Couches fractionnées. Cela sépare les deux canaux fluorescents.
- Quantification de la vitesse d’écoulement de la F-actine.
- Sélectionnez Analyser > Définir les mesures, puis choisissez Zone et rectangle englobant.
- Cliquez sur Ovale dans la barre d’outils et dessinez un cercle sur une tache d’actine (Figure 3C, 1 et 2).
- Sélectionnez Image > Superposition > Ajouter une sélection. Le cercle est superposé sur le montage de l’image (Figure 3C, 3 et 4).
REMARQUE : La couleur de la superposition peut être modifiée dans Modifier > Options > Couleurs. - Répétez la superposition de cercles pour les taches restantes (Figure 3C, 5).
REMARQUE: Pour déterminer avec précision les centres de moucheture d’actine, les enquêteurs doivent superposer des cercles sur les mouchetures. - Cliquez sur Droite dans la barre d’outils et tracez une ligne qui relie les centres des cercles (Figure 3D, 1 et 2).
- Sélectionnez Analyser > mesure. Le résultat apparaîtra à l’écran (Figure 3D, 3).
REMARQUE : La hauteur affichée dans le résultat indique la distance de translocation de la moucheture d’actine pendant l’observation (Figure 3D, 3). - Calculer la vitesse d’écoulement de l’actine F en divisant la distance de translocation par le temps d’observation.
- Quantification du taux de polymérisation de la F-actine.
- Tracez une ligne qui relie les extrémités du faisceau F-actine (Figure 3E, 1).
- Sélectionnez Analyser > mesure. Le résultat apparaîtra à l’écran (Figure 3E, 2).
REMARQUE : La hauteur dans le résultat indique la longueur d’extension du faisceau de F-actine pendant l’observation (Figure 3E, 2). - Calculez le taux d’extension de la F-actine en divisant la durée de l’extension par le temps d’observation.
- Calculer le taux de polymérisation de la F-actine comme la somme de la vitesse d’écoulement de la F-actine et de la vitesse d’extension (Figure 3F).
7. Préparation d’un gel PAA pour la microscopie à force de traction et les cultures de neurones
- Préparation des gels PAA nécessaires à la microscopie à force de traction (Figure 4)
- Pipette 0,5 mL de NaOH (100 mM), dissous dans du H2O distillé, sur des plats à fond de verre de 27 mm de diamètre, et incuber pendant 15 min à température ambiante (RT).
- Ajouter 50 μL de 3-aminopropyltriméthyoxysilane (APTMS) dans la solution de NaOH sur les plats et mélanger les solutions par pipetage doux, et incuber pendant 15 min à TA.
- Lavez la vaisselle à fond de verre avec du H2O distillé et séchez-les.
- Pipeter 0,5 mL de solution de glutaraldéhyde à 0,5 %, dissoute dans du PBS, sur la cuve à fond de verre traitée APTMS, et incuber pendant 30 min à TA.
- Laver la vaisselle à fond de verre avec du H2O distillé et sécher.
- Préparer une solution contenant du H2O distillé (412 μL), une solution d’acrylamide à 30 % (p/v) (63 μL), une solution de bis-acrylamide à 2,5 % (p/v) (6 μL) et une solution de microsphère modifiée par carboxylate (bille fluorescente) (20 μL).
REMARQUE: Vortex la solution de billes fluorescentes pour dissocier les billes agrégées avant de les ajouter à la solution d’acrylamide. - Dégazez la solution dans une chambre à vide pendant 30 min à RT.
- À cette solution, ajouter 1 μL de persulfate d’ammonium (APS) à 10 % (p/v), dissous dans du H2O distillé, et 1 μL de N,N,N',N'-tétraméthyléthylènediamine (TEMED), et mélanger par pipetage doux.
- Placez un couvercle en verre (18 mm de diamètre) sur le capuchon d’un tube de centrifugeuse de 15 mL.
- Ajouter immédiatement 25 μL de la solution sur le couvercle, puis placer doucement un plat à fond de verre inversé sur le couvercle. Laisser l’acrylamide polymériser pendant 1 h à TA.
- Appliquez du H2O distillé sur le couvercle et retirez-le du gel à l’aide d’une aiguille de seringue avec une pointe pliée.
- Retirer le H2O et ajouter le sulfo-SANPAH (1 mM), dissous dans le PBS, au gel.
- Dans des conditions stériles, exposez les gels à la lumière ultraviolette pendant 5 min. Lavez le gel trois fois avec du PBS pendant 15 minutes par lavage.
- Détermination de la rigidité du gel à l’aide d’une méthode d’indentation de microsphère (figure 5A)44.
- Transférez une microsphère spécifiée pour le diamètre et la densité au gel, puis placez-la sur l’étage d’échantillon d’un microscope confocal à balayage laser.
- Concentrez-vous sur la surface du gel et enregistrez la position z (Figure 5B).
- De même, concentrez-vous sur le fond de la microsphère et enregistrez la position z (Figure 5C).
- Calculez la profondeur d’indentation déterminée par la différence entre les positions z de la surface du gel et du fond de la microsphère.
- Calculer le module E de Young du gel par :
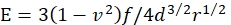
où f est le poids corrigé de la flottabilité de la microsphère, d est la profondeur d’indentation du gel, r est le rayon de la microsphère et v est le rapport de Poisson (dont la valeur est de 0,3, comme déterminé précédemment46).
- Enduire les gels de PDL et de laminine comme décrit à la rubrique 2.
- Comme décrit aux rubriques 3 et 4, disséquer les embryons de souris E16, dissocier l’hippocampe et transfecter avec 5 μg de pEGFP-C1 pour 1,0 x 106 cellules. Semer 2,0 x 105 cellules sur des gels enrobés de PDL/laminine.
REMARQUE: Pour l’analyse de la force de traction, l’imagerie microscopique des cellules marquées par fluorescence est nécessaire pour déterminer la zone du cône de croissance.
8. Microscopie à force de traction au niveau des cônes de croissance neuronale
- Sur DIV3, remplacer le milieu de culture par 0,5 mL de milieu L-15 de Leibovitz réchauffé contenant 2 % de supplément de B-27, de glutamine (1 mM), de pénicilline (100 U/mL) et de streptomycine (100 μg/mL). Maintenir les neurones pendant 1 h à 37 °C.
- Allumez un microscope confocal à balayage laser et réglez l’incubateur sur scène à 37 °C.
REMARQUE : La présente étude utilise un microscope confocal à balayage laser équipé d’une lentille d’objectif à immersion dans l’eau de 63 x 1,2 NA et d’un logiciel d’acquisition et d’analyse d’images (voir tableau des matériaux). - Placez les neurones dans le plat à fond de verre sur l’incubateur préchauffé.
- Définissez les paramètres d’acquisition d’image comme suit : taille de numérisation, 512 × 512 pixels ; zone de numérisation, zoom 1,5-3x; vitesse de numérisation, ~ 1 s par image; longueur d’onde laser, 561 nm (longueur d’onde d’excitation pour les billes fluorescentes) et 488 nm (longueur d’onde d’excitation pour la protéine fluorescente verte améliorée (EGFP)); intervalle de temps, 3 s; durée, 50 images.
- Pour visualiser la morphologie du cône de croissance, sélectionnez un cône de croissance qui exprime fortement l’EGFP.
- Concentrez-vous sur la surface du gel et acquérez des images en accéléré (Figure 6A et Fichier supplémentaire 4).
REMARQUE: Pour l’analyse de traction, utilisez deux canaux fluorescents (pour les billes fluorescentes et EGFP) et un canal de champ lumineux (la capture d’images de contraste d’interférence différentielle (DIC) ou de contraste de phase est recommandée). - Utilisez un logiciel de traitement d’image (par exemple, Fidji) pour produire des piles d’images RVB time-lapse à canal unique et enregistrez ces images sous forme de fichiers tiff.
REMARQUE: Il est essentiel que les enquêteurs enregistrent également les données brutes. - Appliquer 100 μL de dodécylsulfate de sodium (SDS) à 10 % (p/v), dissous dans du H2O distillé, sur la boîte à fond de verre pour détendre le substrat du gel en libérant les neurones du substrat. Incuber le plat pendant 5 min à 37°C pour stabiliser la température.
REMARQUE : L’image des billes dans un substrat non contraint est utilisée pour référencer les positions originales des billes lors de l’analyse de la force de traction (Figure 6C). L’application de la solution SDS modifie les positions xyz en raison du changement thermique dans l’incubateur et de la relaxation de la déformation induite par la cellule. Les enquêteurs doivent donc corriger le plan focal et la position x-y. - Concentrez-vous sur la surface du gel et obtenez une image des billes dans le substrat non contraint (Figure 6B).
- Produisez une image RVB monocanal des perles dans le substrat non contraint et enregistrez cette image sous forme de fichier tiff.
- Correction de la position x-y de l’image de la perle dans le substrat non contraint à l’aide de Fidji
- Aux Fidji, ouvrez l’image de perle dans le substrat non contraint et l’image de pile time-lapse de perles fluorescentes.
- Sélectionnez Piles > d’images > Outils > Concaténer. Une boîte de dialogue s’affiche à l’écran (Figure 7A, 1).
- Sélectionnez l’image de perle dans le substrat non contraint et l’image de pile en accéléré de perles fluorescentes dans Image1 et Image2, respectivement (Figure 7A, 2).
- Cliquez sur OK. L’image de perle dans le substrat non contraint est ajoutée à l’image de pile time-lapse (Figure 7A, 3).
- À l’aide de la barre de défilement (Figure 7B, 1, cadre rouge), affichez la deuxième image de l’image de la pile (flèche rouge).
- Sélectionnez Plugins > StackReg (Figure 7B, 2). Une boîte de dialogue s’affiche à l’écran (Figure 7B, 3).
- Sélectionnez Corps rigide dans la liste déroulante (Figure 7B, 3, cadre rouge) et cliquez sur OK. La correction de la position x-y commence alors.
- Utilisez la barre de défilement pour afficher la première image de l’image de pile corrigée de la position x-y.
- Sélectionnez Image > dupliquer (Figure 7C, 1). Une boîte de dialogue s’affiche à l’écran (Figure 7B, 2).
- Entrez 1 dans la plage, désélectionnez Pile en double (Figure 7C, 2), puis cliquez sur OK. L’image de perle corrigée de la position x-y dans le substrat non contraint apparaîtra à l’écran (Figure 7C, 3). Enregistrez cette image en tant que fichier tiff.
- Quantification de la force de traction
REMARQUE : La méthode décrite ici pour l’analyse de la force de traction utilise MATLAB et deux boîtes à outils MATLAB, « Image Processing Toolbox » et « Parallel Computing Toolbox ». Les enquêteurs devront les installer avant l’analyse. Le code d’analyse de la force de traction a été développé sur la base de la version MATLAB 2018a. Par conséquent, MATLAB version 2018a (ou ultérieure) doit être utilisé pour l’analyse. Les algorithmes utilisés pour l’analyse de la force de traction ont été décrits précédemment22.- Téléchargez le code d’analyse de la force de traction TFM2021 à partir du fichier supplémentaire 5. Ouvrir TFM 2021 dans MATLAB
- Ouvrez main.m dans TFM2021 et exécutez-le. Une interface utilisateur graphique (GUI) apparaîtra à l’écran (Figure 8A).
- Cliquez sur Charger l’image du substrat non tendu et sélectionnez l’image de perle corrigée de la position x-y dans le substrat non contraint.
- Cliquez sur Charger des images de perles fluorescentes et sélectionnez l’image de pile time-lapse des perles.
- Cliquez sur Charger les images en champ lumineux et sélectionnez l’image de pile time-lapse de champ lumineux.
- Cliquez sur Charger les images GFP et sélectionnez l’image de pile time-lapse d’EGFP.
- Sélectionnez GFP dans la liste déroulante de l’interface graphique (Figure 8A, zone rouge).
- Cliquez sur ROI pour spécifier la région rectangulaire d’intérêt (ROI), y compris le cône de croissance, en cliquant sur deux points sur l’image de cellule affichée sur l’interface graphique (Figure 8B).
- Cliquez sur le bouton Enregistrer de l’interface graphique (Figure 8A, flèche rouge). Les images de pile sélectionnées, ainsi que le retour sur investissement, seront enregistrés dans un fichier .mat (fichier au format MATLAB).
- Cliquez sur Spot detect. Une boîte de dialogue apparaîtra à l’écran.
- Entrez une valeur (généralement 50-150) dans la boîte de dialogue pour déterminer un seuil de détection de perles. Cliquez sur OK pour lancer le calcul.
- Une fois le calcul terminé, cliquez sur Tracer la piste pour agrandir la région sélectionnée à l’étape 8.12.8 et afficher les perles détectées sous forme de points blancs (Figure 8C, 1).
REMARQUE: Vérifiez si la détection de perles (points blancs) chevauche les perles fluorescentes. La détection de perles peut également détecter un bruit de fond. Pour réduire ces interférences externes, modifiez la valeur de seuil à Détection ponctuelle. En outre, sélectionnez manuellement les points corrects à l’étape 8.12.13. - Cliquez sur Sélectionner les perles et délimitez une région polygonale qui comprend les points corrects sous le cône de croissance. Appuyez sur Entrée sur le clavier. Les points blancs dans la région polygonale deviennent rouges (Figure 8C, 2).
- Cliquez sur Estimer la force sur l’interface graphique. Ensuite, entrez des valeurs pour les paramètres suivants : taille du pixel, μm/pixel ; Module de Young, la valeur calculée à l’étape 7.2.5; Ratio de Poisson, 0,3.
- Exécutez la force d’estimation pour lancer le calcul. Le logiciel enregistrera automatiquement les résultats de calcul dans un fichier au format feuille de calcul.
REMARQUE : La feuille de calcul montre l’amplitude du composant x, du composant y et de la force à chaque période (Figure 8D). Reportez-vous au dossier supplémentaire 6 pour obtenir des données pratiques sur la quantification de la force de traction.
Résultats
Imagerie à moucheture unique pour quantifier le taux de polymérisation de l’actine et l’efficacité du couplage d’embrayage
Une expression Lifeact élevée permet la visualisation de la F-actine dans le cône de croissance; une faible expression de HaloTag-actine permet de surveiller l’écoulement rétrograde de la F-actine (Figure 3, Fichier supplémentaire 2). Le traçage des taches d’actine permet de mesurer la vitesse d’écoulement de la F-actine (Figure 3C,D). Étant donné que le couplage mécanique de l’écoulement rétrograde de F-actine et du substrat adhésif réduit la vitesse d’écoulement de F-actine, l’efficacité du couplage d’embrayage peut être estimée à partir de la vitesse. De plus, le marquage de la F-actine avec Lifeact facilite la visualisation de l’extension de la F-actine et est utile pour quantifier le taux de polymérisation de l’actine (Figure 3E,F).
Analyse quantitative de la force de traction
Le strict respect de la méthodologie présentée ici révélera les mouvements des billes fluorescentes sous le cône de croissance (Figure 6C, Fichier supplémentaire 4). L’écoulement rétrograde de la F-actine sur le substrat génère une force de traction qui fait que les billes fluorescentes se déplacent vers l’arrière sous le cône de croissance. Le code d’analyse de la force de traction estime la force de traction due au déplacement des billes fluorescentes et exprime la force de traction calculée sous forme de vecteur de force. La direction et l’amplitude de la force de traction sont déterminées à partir des composantes x et y du vecteur force (Figure 8C,D). La case rouge de la figure 8D représente les composantes x et y d’un vecteur de force; La figure 8C représente le cône de croissance correspondant. En termes de coordonnées x-y, le vecteur pointe à -93,8° par rapport à l’axe des x ; cette orientation est dirigée vers l’arrière du cône de croissance (figure 8C). L’amplitude de la force de traction F a été calculée comme suit :
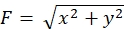
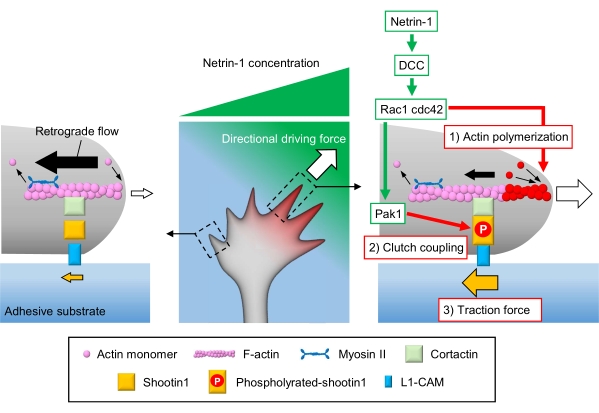
Figure 1 : Une machine à cône de croissance pour la génération de force et la navigation dans les cônes de croissance. Un gradient chimioattractif de nétrine-1 induit une stimulation asymétrique de son récepteur DCC sur un cône de croissance axonal. Cela active Rac1 et Cdc42, ainsi que leur kinase en aval Pak1. Rac1 et Cdc42 favorisent (1) la polymérisation de l’actine, tandis que Pak1 phosphoryle la shootin1, améliorant le couplage d’embrayage médié par la shootine1 (2). L’activation asymétrique de la dynamique de l’actine et le couplage d’embrayage à l’intérieur du cône de croissance augmentent (3) la force de traction du côté de la source de nétrine-1, générant ainsi une force motrice directionnelle pour l’attraction du cône de croissance. Les protocoles présentés ici permettent de quantifier les variables clés (1)-(3) pour la navigation des cônes de croissance. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Images de fluorescence d’un cône de croissance neuronale sous un diaphragme entièrement ouvert et rétréci. Le cône de croissance exprime Lifeact et HaloTag-actin. (A) Une expression Lifeact élevée permet de visualiser la morphologie du cône de croissance. D’autre part, les niveaux d’expression de HaloTag-actine sont très faibles, avec des signaux faibles lorsque le diaphragme est complètement ouvert. (B) Lorsque le diaphragme est correctement rétréci, les signaux de fond diminuent et des taches d’actine simples apparaissent dans le cône de croissance. Barres d’échelle : 5 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3: Étapes de quantification de la vitesse d’écoulement de la F-actine et du taux de polymérisation de l’actine à l’aide d’un logiciel de traitement et d’analyse d’images Fidji. (A) Ajuster l’angle de l’image pour l’analyse. (1) Trouvez et sélectionnez une tache d’actine (pointe de flèche) qui s’écoule dans un faisceau de F-actine. (2) Sélectionnez Image > Transformer > Faire pivoter. (3) Réglez l’angle de manière à ce que le faisceau F-actine soit dirigé vers le haut. (4) L’angle de l’image sera modifié. (B) Délimiter la région, y compris la tache d’actine et le faisceau d’actine F. (1) Cliquez sur Rectangle dans la barre d’outils. (2) Délimitez une région sur l’image. La luminosité et le contraste de l’image sont augmentés pour permettre une visualisation claire de la tache d’actine et de la pointe du faisceau f-actine. (3) Sélectionnez Image > Dupliquer. (4) Entrez les cinq intervalles de temps qui montrent l’écoulement des taches d’actine dans le faisceau de f-actine. (5) L’image de pile sélectionnée apparaîtra à l’écran. (C) Cercles superposés sur la moucheture d’actine. (1) Cliquez sur Ovale dans la barre d’outils. (2) Dessinez un cercle sur une tache d’actine. (3) Sélectionnez Image > Superposition > Ajouter une sélection. (4) Le cercle est superposé. 5) Répétez la superposition des cercles sur les taches d’actine restantes. (D) Mesurer la distance de translocation des mouchetures d’actine au cours des cinq périodes. (1) Cliquez sur Directement dans la barre d’outils. (2) Tracez une ligne qui relie les centres des cercles. (3) Sélectionnez Analyser > mesure. Le résultat, indiqué par le paramètre Height (boîte rouge), relaie la distance de translocation de la moucheture d’actine. (E) Mesurer la variation de la longueur de la saillie de F-actine au cours des cinq périodes. (1) Tracez une ligne qui relie les extrémités de la saillie F-actine. (2) Sélectionnez Analyser > mesure. Le résultat (boîte rouge), Hauteur, indique la longueur d’extension du faisceau F-actine. (F) Le taux de polymérisation de l’actine est calculé à partir de la somme de la vitesse d’écoulement de la F-actine et de la vitesse d’extension. Voir aussi le fichier supplémentaire 2. Le dossier supplémentaire 3 aide les enquêteurs à mettre en pratique la méthodologie décrite ci-dessus. Veuillez cliquer ici pour voir une version agrandie de cette figure.
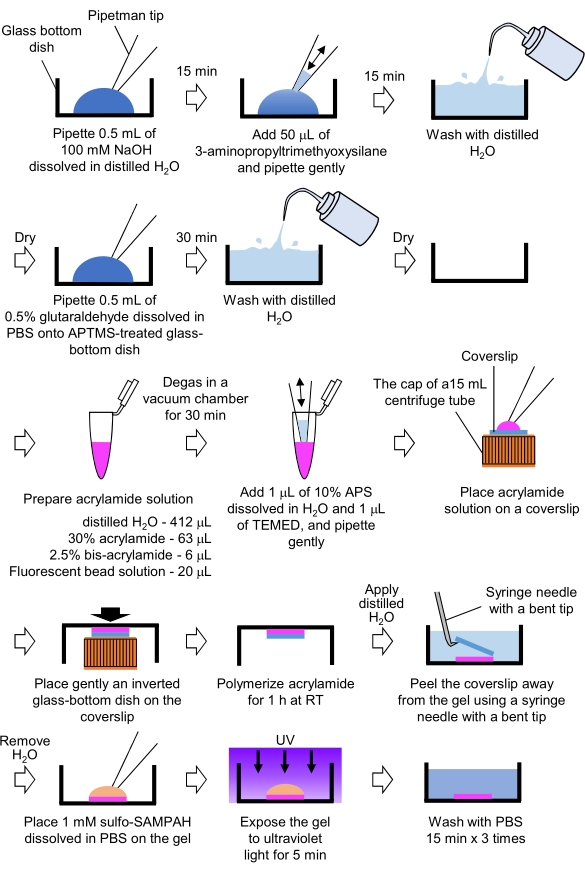
Figure 4 : Étapes de préparation du gel PAA. Veuillez consulter l’étape 7.1 pour une description détaillée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
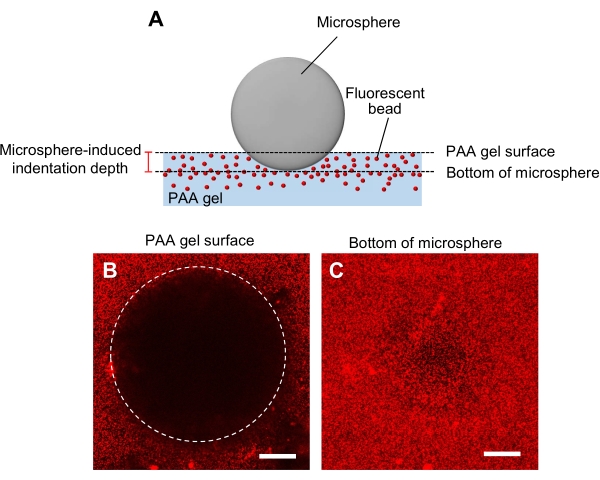
Figure 5 : Détermination de la rigidité du gel PAA. (A) Méthode d’indentation de la microsphère. Lorsqu’une microsphère est placée sur un gel PAA incorporé dans des billes fluorescentes, le poids de la microsphère provoque une indentation dans le gel. La profondeur d’indentation est calculée en soustrayant les positions z de la surface du gel PAA du fond de la microsphère (B,C) Images de fluorescence d’un gel PAA indenté par une microsphère et contenant des billes fluorescentes. Un microscope confocal à balayage laser a été utilisé pour capturer des images de la surface du gel (B) et du fond de la microsphère (C). Les signaux des billes fluorescentes ne sont pas visibles à la surface du gel dans la région en retrait (B, cercle). Ils peuvent cependant être observés au fond de la microsphère. Barres d’échelle : 50 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
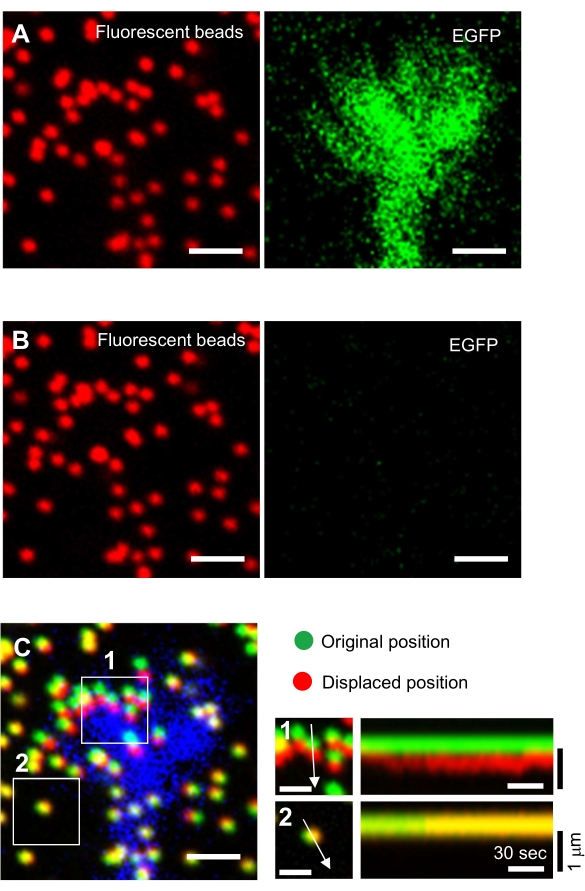
Figure 6 : Cartographie de la force d’un cône de croissance neuronale. (A-C) Images de fluorescence de perles incorporées dans un gel PAA et d’un cône de croissance neurale visualisées avec EGFP. Après l’acquisition d’images time-lapse (A), le neurone a été libéré du substrat de gel en appliquant une solution SDS, et une image des perles dans le substrat non contraint a été capturée (B). (C) L’image des perles dans le substrat non contraint montre les perles dans leurs positions d’origine (vert) et déplacées (rouge). Le signal EGFP du cône de croissance est représenté en bleu. Les kymographes (à droite) montrent les mouvements des perles à des intervalles de 3 s pendant une durée de 147 s, indiqués par les flèches dans les zones encadrées 1 et 2. La perle dans la zone 2 est une perle de référence. Barres d’échelle : 2 μm pour (A), (B) et (C, à gauche), et 1 μm pour (C, au milieu). Voir aussi le fichier supplémentaire 4. Veuillez cliquer ici pour voir une version agrandie de cette figure.
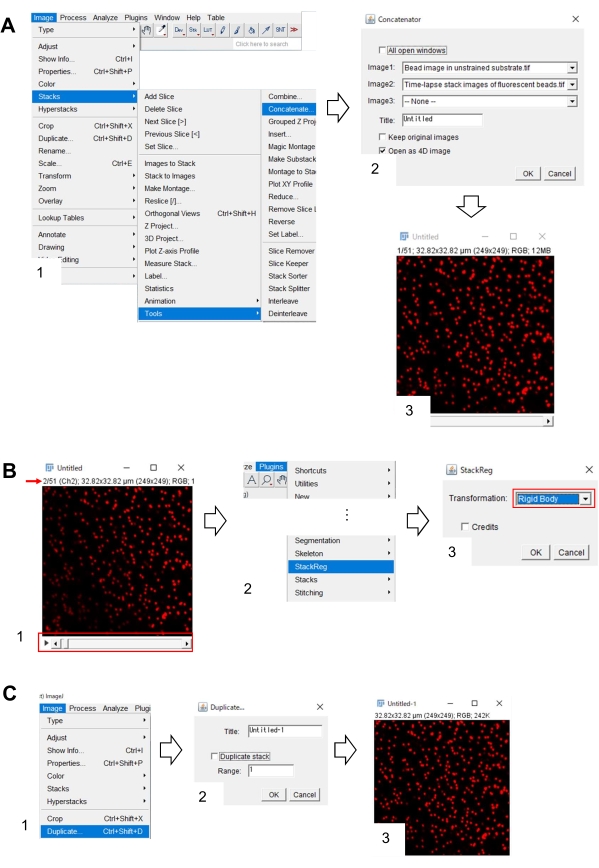
Figure 7: Étapes pour corriger la position x-y de l’image de perle dans un substrat non contraint à l’aide de Fidji. (A) Concaténer l’image de perle dans un substrat non contraint avec l’image de pile time-lapse de perles fluorescentes. (1) Sélectionnez Piles d’images > > Outils > Concaténer. (2) Sélectionnez l’image des perles dans un substrat non contraint et l’image de pile time-lapse des perles fluorescentes dans Image1 et Image2, respectivement. Cliquez sur OK. (3) L’image de perle dans le substrat non contraint est ajoutée à l’image de la pile time-lapse. (B) Corriger la position x-y de l’image de la perle fluorescente. (1) Utilisez la barre de défilement (cadre rouge) pour sélectionner la deuxième image (flèche rouge) dans la pile d’images. (2) Sélectionnez Plugins > StackReg. (3) Sélectionnez Corps rigide dans la liste déroulante (cadre rouge) et cliquez sur OK. La correction de la position x-y va commencer. (C) Enregistrez l’image de perle corrigée de la position x-y. Après avoir sélectionné la première image de l’image de pile corrigée de position x-y, (1) sélectionnez Image > Dupliquer. (2) entrez 1 dans la plage et désélectionnez Pile en double. Cliquez ensuite sur OK. (3) l’image de perle corrigée de la position x-y dans le substrat non contraint apparaîtra à l’écran. Enregistrez cette image en tant que fichier tiff. Le dossier supplémentaire 6 aide les enquêteurs à mettre en pratique la méthodologie décrite ci-dessus. Veuillez cliquer ici pour voir une version agrandie de cette figure.
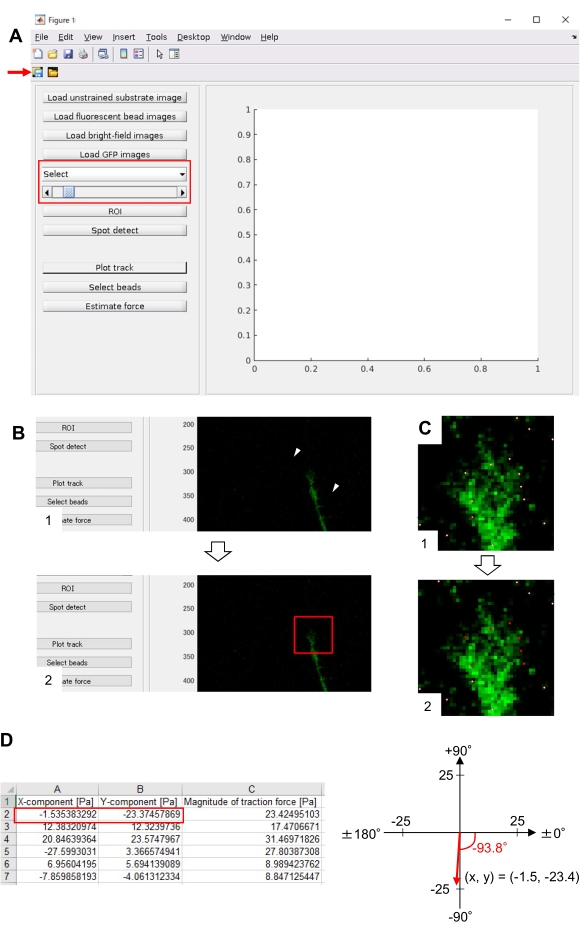
Figure 8 : Analyse de la force de traction sous un cône de croissance neuronal à l’aide d’un code d’analyse de force de traction open source. (A) GUI pour analyser la force de traction. Les images time-lapse sélectionnées sur l’interface graphique peuvent être confirmées dans la liste déroulante et avec le curseur indiqué (boîte rouge). (B) Sélectionnez une région qui inclut le cône de croissance. (1) Cliquez sur ROI sur l’interface graphique. Avec le curseur de la souris, spécifiez deux points (pointes de flèche) sur l’image de la cellule. (2) Une boîte rouge apparaîtra sur l’image de la cellule. Deux clics déterminent l’emplacement de deux coins. (C) Sélectionnez les perles détectées (points blancs) sous le cône de croissance. (1) Sur l’interface graphique, cliquez sur Sélectionner les perles et délimitez une région polygonale qui inclut le cône de croissance en cliquant dessus. Appuyez sur la touche Entrée. (2) Les points blancs dans la région polygonale se transformeront en rouge. (D) Résultats calculés de la direction et de l’amplitude de la force de traction. La case rouge de la feuille de calcul représente les composantes x et y du vecteur de force, estimées par analyse de la force de traction. Sur la coordonnée x-y dans le panneau de droite, le vecteur force généré par le cône de croissance pointe à -93,8° par rapport à l’axe des x ; cette orientation est dirigée vers l’arrière du cône de croissance en (C). Le dossier supplémentaire 6 aide les enquêteurs à mettre en pratique la méthodologie décrite ci-dessus. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Fichier supplémentaire 1 : Recettes de solutions et de supports utilisés dans cette étude. Voir le texte pour une utilisation détaillée. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 2: Imagerie par fluorescence de Lifeact et imagerie par mouchetures fluorescentes de HaloTag-actine dans un cône de croissance nerveuse. Lifeact (vert) et HaloTag-actine (magenta). Les images ont été acquises toutes les 3 s pour une durée totale de 147 s. Barre d’échelle: 2 μm. Voir aussi Figure 3. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 3 : Données pratiques pour quantifier la vitesse d’écoulement de la F-actine et le taux de polymérisation de l’actine. Une image de pile time-lapse multicanal de Lifeact (vert) et HaloTag-actin (magenta). Voir aussi la figure 3. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 4 : Vidéo de cartographie de force détectant la force de traction au niveau d’un cône de croissance neuronale. Les positions d’origine (vert) et déplacées (rouge) des perles. Le signal EGFP dans le cône de croissance est représenté en bleu. Les images ont été acquises toutes les 3 s pendant 147 s. Barre d’échelle: 2 μm. Voir aussi la figure 6. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 5 : Code d’analyse de la force de traction. Veuillez consulter l’étape 8.12 pour une utilisation détaillée. Veuillez cliquer ici pour télécharger ce fichier.
Dossier supplémentaire 6 : Données pratiques pour quantifier la force de traction. Une image RVB monocanal des perles dans un substrat non contraint et un time-lapse monocanal empilent des images RVB de perles fluorescentes sous un cône de croissance, egFP et champ lumineux. Voir également les figures 7 et 8. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Les protocoles décrits dans cette étude utilisent des matériaux et des équipements de microscopie disponibles dans le commerce que l’on trouve couramment dans tous les laboratoires, instituts et universités. Par conséquent, les chercheurs peuvent facilement adopter l’imagerie à faisceau unique et la microscopie à force de traction dans leurs études.
L’imagerie des mouchetures peut analyser la polymérisation de l’actine et le couplage d’embrayage. En outre, l’imagerie des mouchetures peut surveiller le flux rétrograde de molécules d’embrayage telles que la shootin1 et la cortactine, qui interagissent avec le flux rétrograde de F-actine. En utilisant un microscope TIRF, le flux rétrograde de la molécule d’adhésion cellulaire L1-CAM peut également être surveillé23,41; L1-CAM subit des comportements d’adhérence et de glissement qui reflètent l’efficacité de l’accouplement d’embrayage23,41. Bien que la présente étude utilise le système TMR-HaloTag pour l’imagerie des mouchetures, d’autres protéines fluorescentes, telles que l’EGFP et la protéine fluorescente rouge monomère, sont également disponibles dans l’analyse16,18,20,23,24,27,39. Les éléments essentiels pour visualiser les taches d’actine sont un faible niveau d’expression de l’actine fluorescente et l’éclairage d’une zone minimale (Figure 2). Dans ce protocole, les signaux Lifeact et HaloTag-actine sont acquis séquentiellement. Étant donné que l’écoulement rétrograde de l’actine est relativement lent (4,5 ± 0,1 μm/min)24, l’analyse du flux rétrograde de la F-actine et de la polymérisation de l’actine n’est pas affectée par l’acquisition séquentielle d’images de différents canaux fluorescents (intervalle ~1 s). Lifeact est un marqueur F-actine largement utilisé, mais peut rivaliser avec les protéines de liaison à l’actine47. Plus important encore, Lifeact peut modifier la dynamique de l’actine, affectant ainsi les structures de la F-actine et la morphologie cellulaire47,48,49.
La microscopie à force de traction peut détecter des forces pour stimuler l’avancement du cône de croissance. En montant les neurones dans une matrice extracellulaire, les chercheurs peuvent également analyser les forces générées dans un environnement semi-3D11. L’imagerie à fort grossissement est importante pour une quantification précise de la force de traction, car les cônes de croissance génèrent de faibles forces de traction7. Bien que d’autres méthodes avec des nanopiliers ou des biocapteurs sensibles au stress soient également utilisées pour mesurer la force de traction50,51, la méthode à base de gel PAA est très adaptable et permet d’ajuster la rigidité du substrat en faisant varier les concentrations d’acrylamide et de bis-acrylamide41,44,52. Dans ce protocole, le gel PAA est préparé à une concentration finale de 3,75% d’acrylamide et de 0,03% de bis-acrylamide; Le module de Young est d’environ 270 Pa22 et cette rigidité se situe dans la gamme du tissu cérébral (100-10 000 Pa)53,54,55. En raison de l’épaisseur du gel PAA (~100 μm), cette méthode limite l’utilisation de lentilles à fort grossissement pendant la microscopie. Pour obtenir des images à fort grossissement, les chercheurs doivent utiliser la fonction de zoom dans un microscope confocal à balayage laser.
En conclusion, la présente imagerie des mouchetures et la microscopie à force de traction permettent des analyses quantitatives des événements clés des générations de forces. Ces informations seront inestimables pour améliorer la compréhension des mécanismes qui sous-tendent l’avancement et la navigation des cônes de croissance.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Cette recherche a été soutenue en partie par AMED sous le numéro de subvention 21gm0810011h0005 (N.I. et Y.S.), JSPS KAKENHI (JP19H03223, N.I.) et JSPS Grants-in-Aid for Early-Career Scientists (JP19K16258, T.M.), la Osaka Medical Research Foundation for Incurable Diseases (T.M.) et le NAIST Next Generation Interdisciplinary Research Project (Y.S.).
matériels
| Name | Company | Catalog Number | Comments |
| 0.5% trypan blue stain solution | Nacalai | 29853-34 | |
| 3-aminopropyltrimethyoxysilane | Sigma | 281778-100ML | |
| Acrylamide monomer | Nacalai | 00809-85 | |
| Ammonium persulphate | Cytiva | 17-1311-1 | |
| Axio Observer Z1 | Zeiss | 431007-9902-000 | Epi-fluorescence microscope (single speckle imaging) |
| B-27 supplement (50x) | Thermo Fisher Scientific | 17504-044 | |
| Bovine serum albmine | Sigma | A7906-10G | |
| C-Apochromat 63x/1.2 W Corr | Zeiss | 421787-9970-799 | Objective lens (traction force microscopy) |
| Coverslip (diameter 18 mm) | Matsunami | C018001 | |
| D-glucose | Nacalai | 16806-25 | |
| DNaseI | Sigma | DN25-100MG | |
| Fetal bovine serum | Thermo Fisher Scientific | 10270-106 | |
| Fiji | Open source software package | https://imagej.net/software/fiji/ | |
| FluoSpheres carboxylate-modified 0.2 mm, red (580/605), 2% solid | Thermo Fisher Scientific | F8810 | carboxylate-modified microspheres |
| Glass bottom dish (14 mm diameter) | Matsunami | D1130H | |
| Glass bottom dish (27 mm diameter) | Matsunami | D1140H | |
| Glutaraldehyde solution | Sigma | G5882-10X10ML | |
| HaloTag TMR ligand | Promega | G8251 | |
| HBO103 W/2 | Osram | 4050300382128 | Mercury lamp (single speckle imaging) |
| Image Processing Toolbox | MathWork | https://www.mathworks.com/products/image.html | |
| Laminin solution from mouse EHS tumor | Wako | 120-05751 | |
| Leibovitz’s L-15 medium | Thermo Fisher Scientific | 11415064 | |
| L-glutamine | Nacalai | 16919-42 | |
| LSM710 | Zeiss | N/A | Conforcal laser microscope (traction force microscopy) |
| MATLAB2018a | MathWork | https://www.mathworks.com/products/new_products/release2018a.html | |
| Mouse C57BL/6 | Japan SLC | N/A | |
| Mouse neuron nucleofector kit | Lonza | VPG-1001 | |
| N,N,N’,N’-tetramethylethylenediamine (TEMED) | Nacalai | 33401-72 | |
| N,N’-methylenebisacrylamide | Nacalai | 22402-02 | |
| Neurobasal medium | Thermo Fisher Scientific | 21103-049 | |
| Nucleofector I | Amaxa | AAD-1001 | Electroporation apparatus |
| ORCA Flash 4.0 V2 | Hamamatsu | C11440-22CU | CMOS camera (single speckle imaging) |
| Papain | Nacalai | 26036-34 | |
| Parallel Computing Toolbox | MathWork | https://www.mathworks.com/products/parallel-computing.html | |
| pEGFP-C1 | Clontech | 1528177 | |
| Penicillin-streptomycin (100x) | Nacalai Tesque | 26253-84 | |
| pFN21A-HaloTag-actin | (Minegishi et al., 2018) | N/A | |
| Phosphate buffered saline (PBS) pH 7.4 (10x) | Thermo Fisher Scientific | 70011-044 | |
| Plan-Apochromat 100x/1.4 Oil | Zeiss | 420790-9901-000 | Objective lens (single speckle imaging) |
| pmNeonGreen-N1-Lifeact | (Kastian et al., 2021) | N/A | |
| Poly-D-lysine hydrobromide | Sigma | P6407-5MG | |
| Slulfo-SAMPHA | Thermo Fisher Scientific | 22589 | |
| Sodium dodecyl sulfate | Nacalai | 08933-05 | |
| Sodium hydrate (NaOH) | Nacalai | 31511-05 | |
| Steel ball | Sako tekkou | N/A | Microshpere to determin PAA gel rigidity. 0.6 mm diameter, 7.87 g/cm3. |
| ZEN2009 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (traction force microscopy) |
| ZEN2012 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (single speckle imaging) |
Références
- TessierLavigne, M., Goodman, C. S. The molecular biology of axon guidance. Science. 274, 1123-1133 (1996).
- Marin, O., Valiente, M., Ge, X., Tsai, L. H. Guiding neuronal cell migrations. Cold Spring Harbor Perspectives in Biology. 2, 001834 (2010).
- Vitriol, E. A., Zheng, J. Q. Growth cone travel in space and time: the cellular ensemble of cytoskeleton, adhesion, and membrane. Neuron. 73 (6), 1068-1081 (2012).
- Cooper, J. A. Cell biology in neuroscience: mechanisms of cell migration in the nervous system. Journal of Cell Biology. 202, 725-734 (2013).
- Dupraz, S., et al. RhoA controls axon extension independent of specification in the developing brain. Current Biology. 29, 3874-3886 (2019).
- Suter, D. M., Miller, K. E. The emerging role of forces in axonal elongation. Progress in Neurobiology. 94, 91-101 (2011).
- Franze, K. Integrating chemistry and mechanics: the forces driving axon growth. Annual Review of Cell and Developmental Biology. 36, 61-83 (2020).
- Mitchison, T., Kirschner, M. Cytoskeletal dynamics and nerve growth. Neuron. 1, 761-772 (1988).
- Suter, D. M., Forscher, P. Substrate-cytoskeletal coupling as a mechanism for the regulation of growth cone motility and guidance. Journal of Neurobiology. 44, 97-113 (2000).
- Lowery, L. A., Van Vactor, D. The trip of the tip: understanding the growth cone machinery. Nature Reviews Molecular Cell Biology. 10, 332-343 (2009).
- Minegishi, T., et al. Shootin1b mediates a mechanical clutch to produce force for neuronal migration. Cell Reports. 25, 624-639 (2018).
- Minegishi, T., Inagaki, N. Forces to drive neuronal migration steps. Frontiers in Cell and Developmental Biology. 8, 863 (2020).
- Forscher, P., Smith, S. J. Actions of cytochalasins on the organization of actin filaments and microtubules in a neuronal growth cone. Journal of Cell Biology. 107, 1505-1516 (1988).
- Suter, D. M., Forscher, P. An emerging link between cytoskeletal dynamics and cell adhesion molecules in growth cone guidance. Current Opinion in Neurobiology. 8, 106-116 (1998).
- Pollard, T. D., Borisy, G. G. Cellular motility driven by assembly and disassembly of actin filaments. Cell. 112, 453-465 (2003).
- Waterman-Storer, C. M., Desai, A., Bulinski, J. C., Salmon, E. D. Fluorescent speckle microscopy, a method to visualize the dynamics of protein assemblies in living cells. Current Biology. 8, 1227-1230 (1998).
- Katoh, K., Hammar, K., Smith, P. J., Oldenbourg, R. Birefringence imaging directly reveals architectural dynamics of filamentous actin in living growth cones. Molecular Biology of the Cell. 10, 197-210 (1999).
- Watanabe, N., Mitchison, T. J. Single-molecule speckle analysis of actin filament turnover in lamellipodia. Science. 295, 1083-1086 (2002).
- Medeiros, N. A., Burnette, D. T., Forscher, P. Myosin II functions in actin-bundle turnover in neuronal growth cones. Nature Cell Biology. 8, 215-226 (2006).
- Shimada, T., et al. Shootin1 interacts with actin retrograde flow and L1-CAM to promote axon outgrowth. Journal of Cell Biology. 181, 817-829 (2008).
- He, M., Zhang, Z. H., Guan, C. B., Xia, D., Yuan, X. B. Leading tip drives soma translocation via forward F-actin flow during neuronal migration. Jounal of Neuroscience. 30, 10885-10898 (2010).
- Toriyama, M., Kozawa, S., Sakumura, Y., Inagaki, N. Conversion of a signal into forces for axon outgrowth through Pak1-mediated shootin1 phosphorylation. Current Biology. 23, 529-534 (2013).
- Abe, K., et al. Grip and slip of L1-CAM on adhesive substrates direct growth cone haptotaxis. Proceedings of the National Academy of Sciences of the United States of America. 115, 2764-2769 (2018).
- Baba, K., et al. Gradient-reading and mechano-effector machinery for netrin-1-induced axon guidance. Elife. 7, 34593 (2018).
- Nichol, R. I., Hagen, K. M., Lumbard, D. C., Dent, E. W., Gomez, T. M. Guidance of axons by local coupling of retrograde flow to point contact adhesions. Journal of Neuroscience. 36, 2267-2282 (2016).
- Shekarabi, M., et al. Deleted in colorectal cancer binding netrin-1 mediates cell substrate adhesion and recruits Cdc42, Rac1, Pak1, and N-WASP into an intracellular signaling complex that promotes growth cone expansion. Journal of Neuroscience. 25, 3132-3141 (2005).
- Kubo, Y., et al. Shootin1-cortactin interaction mediates signal-force transduction for axon outgrowth. Journal of Cell Biology. 210, 663-676 (2015).
- Huber, A. B., Kolodkin, A. L., Ginty, D. D., Cloutier, J. F. Signaling at the growth cone: ligand-receptor complexes and the control of axon growth and guidance. Annual Review of Neuroscience. 26, 509-563 (2003).
- Kolodkin, A. L., Tessier-Lavigne, M. Mechanisms and molecules of neuronal wiring: a primer. Cold Spring Harbor Perspectives in Biology. 3, 001727 (2011).
- Ayala, R., Shu, T., Tsai, L. H. Trekking across the brain: the journey of neuronal migration. Cell. 128, 29-43 (2007).
- Tatavarty, V., Kim, E. J., Rodionov, V., Yu, J. Investigating sub-spine actin dynamics in rat hippocampal neurons with super-resolution optical imaging. PLoS One. 4, 7724 (2009).
- Frost, N. A., Shroff, H., Kong, H., Betzig, E., Blanpied, T. A. Single-molecule discrimination of discrete perisynaptic and distributed sites of actin filament assembly within dendritic spines. Neuron. 67, 86-99 (2010).
- Chazeau, A., et al. Nanoscale segregation of actin nucleation and elongation factors determines dendritic spine protrusion. EMBO Journal. 33, 2745-2764 (2014).
- Garcia, M., et al. Two-tiered coupling between flowing actin and immobilized N-cadherin/catenin complexes in neuronal growth cones. Proceedings of the National Academy of Sciences of the United States of America. 112, 6997-7002 (2015).
- Swaminathan, V., et al. Actin retrograde flow actively aligns and orients ligand-engaged integrins in focal adhesions. Proceedings of the National Academy of Sciences of the United States of America. 114, 10648-10653 (2017).
- Tsai, T. Y., et al. Efficient front-rear coupling in neutrophil chemotaxis by dynamic myosin II localization. Developmental Cell. 49, 189-205 (2019).
- Zhang, X. F., et al. Regulation of axon growth by myosin II-dependent mechanocatalysis of cofilin activity. Journal of Cell Biology. 218 (7), 2329-2349 (2019).
- Reversat, A., et al. Cellular locomotion using environmental topography. Nature. 582, 582-585 (2020).
- Katsuno, H., et al. Actin migration driven by directional assembly and disassembly of membrane-anchored actin filaments. Cell Reports. 12, 648-660 (2015).
- Urasaki, A., et al. Shootins mediate collective cell migration and organogenesis of the zebrafish posterior lateral line system. Scientific Reports. 9, 12156 (2019).
- Abe, K., et al. Mechanosensitive axon outgrowth mediated by L1-laminin clutch interface. Biophysical Journal. 120, 3566-3576 (2021).
- Kastian, R. F., et al. Shootin1a-mediated actin-adhesion coupling generates force to trigger structural plasticity of dendritic spines. Cell Reports. 35, 109130 (2021).
- Riedl, J., et al. Lifeact: a versatile marker to visualize F-actin. Nature Methods. 5, 605-607 (2008).
- Chan, C. E., Odde, D. J. Traction dynamics of filopodia on compliant substrates. Science. 322, 1687-1691 (2008).
- Wang, Y. L., Pelham, R. J. Preparation of a flexible, porous polyacrylamide substrate for mechanical studies of cultured cells. Methods in Enzymology. 298, 489-496 (1998).
- Li, Y., Hu, Z., Li, C. New method for measuring poisson's ratio in polymer gels. Journal of Applied Polymer Science. 50, 1107-1111 (1993).
- Belyy, A., Merino, F., Sitsel, O., Raunser, S. Structure of the Lifeact-F-actin complex. PLoS Biology. 18, 3000925 (2020).
- Flores, L. R., Keeling, M. C., Zhang, X., Sliogeryte, K., Gavara, N. Lifeact-GFP alters F-actin organization, cellular morphology and biophysical behaviour. Scientific Reports. 9, 3241 (2019).
- Kumari, A., Kesarwani, S., Javoor, M. G., Vinothkumar, K. R., Sirajuddin, M. Structural insights into actin filament recognition by commonly used cellular actin markers. EMBO Journal. 39, 104006 (2020).
- du Roure, O., et al. Force mapping in epithelial cell migration. Proceedings of the National Academy of Sciences of the United States of America. 102, 2390-2395 (2005).
- Grashoff, C., et al. Measuring mechanical tension across vinculin reveals regulation of focal adhesion dynamics. Nature. 466, 263-266 (2010).
- Koch, D., Rosoff, W. J., Jiang, J., Geller, H. M., Urbach, J. S. Strength in the periphery: growth cone biomechanics and substrate rigidity response in peripheral and central nervous system neurons. Biophysical Journal. 102 (3), 452-460 (2012).
- Barnes, J. M., Przybyla, L., Weaver, V. M. Tissue mechanics regulate brain development, homeostasis and disease. Journal of Cell Science. 130, 71-82 (2017).
- Moore, S. W., Roca-Cusachs, P., Sheetz, M. P. Stretchy proteins on stretchy substrates: the important elements of integrin-mediated rigidity sensing. Developmental Cell. 19, 194-206 (2010).
- Tyler, W. J. The mechanobiology of brain function. Nature Reviews Neuroscience. 13, 867-878 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon