Method Article
Análises da Actin Dynamics, Clutch Coupling e Força de Tração para o Avanço do Cone de Crescimento
Neste Artigo
Resumo
Para avançar, os cones de crescimento devem exercer forças de tração contra o ambiente externo. A geração de forças de tração depende da dinâmica actin e do acoplamento da embreagem. O presente estudo descreve métodos para analisar a dinâmica do actin, acoplamento de embreagem e forças de tração para o avanço do cone de crescimento.
Resumo
Para estabelecer redes funcionais, os neurônios devem migrar para seus destinos apropriados e, em seguida, estender axônios em direção às suas células-alvo. Esses processos dependem dos avanços dos cones de crescimento que se localizaram nas pontas dos neurites. Cones de crescimento axonal geram forças motrizes ao sentir seu microambiente local e modular a dinâmica citoesquelletal e o acoplamento de adesão de actina (acoplamento de embreagem). Décadas de pesquisa levaram à identificação de moléculas de orientação, seus receptores e cascatas de sinalização a jusante para regular a migração neuronal e orientação axonal; no entanto, as máquinas moleculares necessárias para gerar forças para impulsionar o avanço do cone de crescimento e a navegação estão apenas começando a ser elucidadas. Na ponta dos cones de crescimento neuronal, os filamentos de actina sofrem fluxo retrógrado, que é alimentado pela polimerização de actina e contração de actomyosina. Um acoplamento de embreagem entre o fluxo retrógrado F-actin e o substrato adesivo gera forças de tração para o avanço do cone de crescimento. O presente estudo descreve um protocolo detalhado para monitorar o fluxo retrógrado F-actin por imagem de uma única mancha. É importante ressaltar que, quando combinada com um marcador F-actin Lifeact, esta técnica pode quantificar 1) a taxa de polimerização F-actin e 2) a eficiência de acoplamento de embreagem entre o fluxo retrógrado F-actin e o substrato adesivo. Ambas são variáveis críticas para a geração de forças para o avanço do cone de crescimento e navegação. Além disso, o presente estudo descreve um protocolo detalhado de microscopia de força de tração, que pode quantificar 3) força de tração gerada por cones de crescimento. Assim, acoplando as análises de imagens de partículas simples e microscopia de força de tração, os pesquisadores podem monitorar a mecânica molecular subjacente ao avanço do cone de crescimento e navegação.
Introdução
No cérebro vertebrado em desenvolvimento, os neurônios passam por migrações elaboradamente organizadas e projetam axônios em direção a parceiros sinápticos apropriados para estabelecer redes neuronais funcionais1,2,3. Cones de crescimento, que são estruturas sensoriais e motil localizadas na ponta dos neurites, determinam a velocidade e a direção da migração neuronal e do crescimento do axônio3,4,5. Uma vez que os neurônios estão cercados por ambientes bem embalados, os cones de crescimento devem exercer forças contra seu ambiente para avançar6,7. Para entender os mecanismos subjacentes à migração neuronal e orientação axonal, as análises da mecânica molecular para o avanço do cone de crescimento são essenciais.
Décadas de análise revelaram que a força de tração para impulsionar o avanço do cone de crescimento é gerada pelo mecanismo de 'embreagem'; acredita-se que esse mecanismo funcione não apenas no cone de crescimento axonal, mas também no cone de crescimento do processo líder de neurônios migratórios8,9,10,11,12. Ou seja, filamentos de actina (F-actins) em cones de crescimento polimerizam na borda principal e despomerizam proximicamente, empurrando para fora a membrana de ponta13,14,15. A força resultante, em conjunto com a contração da actomyosina, induz o movimento para trás das F-actinas chamada fluxo retrógrado7,11,16,17,18,19,20,21. Moléculas de aderência e de aderência celular mediam o acoplamento mecânico entre o fluxo retrógrado F-actin e o substrato adesivo e transmitem a força do fluxo F-actin para o substrato, gerando assim força de tração para o avanço do cone de crescimento7,8,9,11,12,22 . Simultaneamente, o acoplamento actin-substrato reduz a velocidade de fluxo F-actin e converte a polimerização de actina na força para protuberinar a membrana de borda principal9,10.
Os cones de crescimento axonal sentem as pistas químicas locais e as transduzem em uma força motriz direcional para a navegação de cone de crescimento3,23,24,25. Por exemplo, uma molécula de orientação de axônio netrin-1 estimula seu receptor excluído no câncer colorretal (DCC), e ativa o triphosfato de guanosina rho (GTP) -vincula proteínas de divisão celular 42 (Cdc42) e substrato de toxina botulínica C3 1 (Rac1) e sua quinase a jusante p21-ativada quinase 1 (Pak1)26. Cdc42 e Rac1 promovem a polimerização de actina, e Pak1 fosforila uma molécula de embreagem dispara 122,26. O Shootin1 interage com o fluxo retrógrado F-actin através de uma proteína cortactin27 de ligação de actina. O Shootin1 também interage com a molécula de adesão celular L1 (L1-CAM)20,24. A fosforilação shootin1 aumenta as afinidades de ligação para cortactina e L1-CAM, e aumenta o acoplamento de embreagem 24,27 mediado por shootin1. Dentro do cone de crescimento, ativações assimétricas de polimerização de actina e acoplamento de embreagem aumentam 3) força de tração na lateral da fonte netrin-1, gerando assim força motriz direcional para a curva do cone de crescimento (Figura 1)24. Pesquisas intensivas nas últimas décadas em relação à migração neuronal e orientação do axônio aumentaram a compreensão das moléculas de orientação, seus receptores e associaram cascatas de sinalização a jusante2,10,28,29,30. No entanto, as máquinas moleculares para gerar forças para o avanço do cone de crescimento estão apenas começando a ser elucidadas; isso pode ser atribuído ao uso limitado dos protocolos para análises mecanobiológicas.
O presente estudo descreve um protocolo detalhado para o monitoramento do fluxo retrógrado F-actin por imagem de uma única mancha16,18. O monitoramento do fluxo retrógrado F-actin tem sido extensivamente realizado utilizando microscopia de super-resolução, microscopia confocal de disco giratório e fluorescência de reflexo de interferência total (TIRF) microscopia25,31,32,33,34,35,36,37,38 . O protocolo do presente estudo, no entanto, utiliza um microscópio de epifluorescência padrão e, portanto, é prontamente adotável11,16,18,20,22,23,24,27,39,40,41,42. Quando combinada com a rotulagem F-actin por Lifeact43, a imagem de mancha única permite quantificações da taxa de polimerização de actina e da eficiência de acoplamento de embreagem entre o fluxo f-actin retrógrado e o substrato adesivo39,42. O presente estudo descreve ainda um protocolo detalhado de microscopia de força de tração utilizando um gel de poliacrilamida fluorescente (PAA) 11,22,23,24,27,39,41,42,44. Este método detecta e quantifica a força de tração sob o cone de crescimento monitorando movimentos de contas induzidos pela força44,45. Um código de análise de força de tração de código aberto é fornecido, e o método para quantificar a força de tração durante a migração do cone de crescimento é explicado em detalhes. Com o auxílio de imagens de partículas simples e microscopia de força de tração, a compreensão da mecânica molecular subjacente à migração do cone de crescimento e da navegação será facilitada. Essas técnicas também são aplicáveis para analisar a mecânica molecular subjacente ao alargamento da coluna dendrítica, que é conhecido por ser importante na aprendizagem e na memória42.
Protocolo
Todos os experimentos com animais de laboratório foram realizados com o Comitê Institucional de Cuidados e Uso de Animais do Instituto Nara de Ciência e Tecnologia. Os investigadores devem seguir as diretrizes estabelecidas por seus comitês institucionais e nacionais de regulação animal para o cuidado e uso de animais de laboratório.
1. Preparação de soluções e mídia
- Prepare as soluções e os meios de cultura como resumidos no Arquivo Suplementar 1
2. Preparação de substratos revestidos de poli-D-lysine (PDL)/laminin
- Cubra pratos de fundo de vidro de 14 mm de diâmetro com 100 μg/mL PDL, dissolvido em soro fisiológico tamponado de fosfato (PBS, pH 7.4), e incubar durante a noite em uma incubadora umidificada a 37 °C.
NOTA: Não seque as superfícies de vidro após esta etapa - Remova a solução PDL e a pipeta 1 mL de PBS no vidro por 10 s três vezes.
- Cubra os pratos com laminina de 5 μg/mL, dissolvida em PBS e incubada durante a noite em uma incubadora umidificada a 37 °C.
- Remova a solução de laminina e a pipeta 1 mL de PBS no vidro por 10 s três vezes.
- Remova o PBS e coloque 0,5 mL do meio neurobásal nas superfícies de vidro. Guarde os pratos em uma incubadora umidificada a 37 °C.
NOTA: Os pratos revestidos de PDL/laminina podem ser armazenados por 2-3 dias em uma incubadora umidificada a 37 °C.
3. Dissecção e dissociação do hipocampo
- Eutanize um rato grávida por luxação cervical.
NOTA: O presente estudo utiliza camundongos disponíveis comercialmente (ver Tabela de Materiais). Os investigadores devem usar camundongos criados e tratados humanamente em um ambiente esterilizado. - Disseca os embriões de camundongos embrionários do dia 16 (E16) e coloque-os no gelo.
- Em uma capa de fluxo laminar, disseque os cérebros com tesouras e coloque-os em um prato estéril de 10 cm contendo 10 mL de solução de dissecção gelada.
- Com um microscópio estéreo de dissecção, retire cuidadosamente as meninges dos hemisférios cerebrais e, em seguida, disseque o hipocampi usando fórceps.
- Transfira o hipocampi para 5 mL de solução de digestão gelada em um tubo de 15 mL. Incubar o hipocampi em um banho de água por 20 min a 37 °C.
- Remova a solução de digestão e adicione 3 mL de solução de dissecção.
- Cano suavemente o hipocampi quatro vezes com uma pipeta Pasteur.
- Incubar o hipocampi em um banho de água por 20 min a 37 °C.
- Transfira a suspensão da célula para um tubo de centrífuga de 50 mL. Adicione 3 mL de nova solução de dissecção ao tecido não dissociado.
- Repetição passos 3.7-3.9 até que o hipocampi esteja completamente dissociado.
- Remova os agregados flutuantes de DNA da suspensão celular girando e aspirando com uma pipeta Pasteur. Centrifugar a suspensão celular a 180 x g por 20 min a 4 °C.
NOTA: O DNA derivado de células danificadas perturbará o processo de centrifugação. Se os neurônios permanecerem no supernasce, centrifugar a suspensão celular novamente depois de remover cuidadosamente o DNA. - Equilibrate neurobásal médio contendo 10% de soro bovino fetal (FBS), penicilina (100 U/mL) e estreptomicina (100 μg/mL) em uma incubadora umidificada a 37 °C com 5% de CO2.
4. Transfecção e cultivo de neurônios
- Remova o supernatante e adicione 5 mL de PBS gelado no tubo de 50 mL. Resuspend a pelota da célula por pipetação suave.
- Transfira 10 μL da suspensão celular para um tubo de microcentrífuga, adicione 10 μL de solução azul trypan de 0,5% e, em seguida, conte o número de células com um hemótmetro.
- Centrifugar a suspensão celular no tubo de 50 mL a 180 x g por 20 min a 4 °C.
- Enquanto isso, alíquota para transfecção, 1 μg de pFN21A-HaloTag-actin e 3 μg de pmNeonGreen-N1-Lifeact por 1 x 106 células, em um tubo de microcentrifuge.
- Após a centrifugação, remova o supernasce e adicione 100 μL de meio de eletroporação (fornecido pelo kit de transfecção) no tubo de 50 mL. Resuspense a pelota da célula por pipetar.
- Misture a suspensão celular com a solução de DNA aliquoted e transfira a mistura para um cuvette (fornecido pelo kit de transfecção).
NOTA: Como as bolhas de ar perturbam a eletroporação, remova-as da suspensão celular. - Insira a cuvette no aparelho de eletroporação (Tabela de Materiais) e realize a eletroporação utilizando o programa O-005.
- Adicione imediatamente 1 mL do meio neurobásal pré-aquecido e equilibrado contendo 10% de FBS, penicilina (100 U/mL) e estreptomicina (100 μg/mL) à cuvette.
- Transfira a suspensão da célula para um tubo de centrífuga de 15 mL usando uma pipeta plástica (fornecida pelo kit de transfecção).
- Transfira 10 μL da suspensão celular para um microtubo, adicione 10 μL de solução azul trypan de 0,5% e, em seguida, conte o número de células com um hemótmetro.
- Retire os pratos de fundo de vidro revestidos de PDL/laminina da incubadora e remova o meio neurobásal.
- Pipeta 0,5 mL da suspensão celular contendo 2,0 x 105 células por prato e incubar em uma incubadora umidificada a 37 °C com 5% de CO2 por 3h.
- Substitua o médio por 0,5 mL de meio neurobásico contendo suplemento B-27 de 2%, glutamina (1 mM), penicilina (100 U/mL) e estreptomicina (100 μg/mL). Cultura os neurônios em uma incubadora umidificada por 3 dias a 37 °C com 5% de CO2.
5. Imagem de mancha única em cones de crescimento neuronal
- No dia in vitro (DIV) 3, trate os neurônios com ligante de tetrametil-rhodamina (TMR) a uma diluição de 1:2000 em meio de cultura. Mantenha os neurônios por 1h a 37 °C com 5% de CO2.
- Lave o ligante TMR três vezes com PBS pré-aquecido.
- Remova o PBS e a pipeta 0,5 mL do meio L-15 de Leibovitz aquecido contendo 2% de suplemento B-27, glutamina (1 mM), penicilina (100 U/mL) e estreptomicina (100 μg/mL).
- Mantenha os neurônios por 1h a 37 °C.
- Ligue um microscópio de epifluorescência e coloque a incubadora de topo de palco para 37 °C.
NOTA: O presente protocolo utiliza um microscópio de epifluorescência equipado com uma câmera semicondutor de óxido de metal complementar, uma lente objetiva de imersão de óleo 100x/1.40, uma lâmpada de mercúrio e um software de aquisição de imagem (ver Tabela de Materiais). Outros microscópios de epifluorescência com especificações equivalentes também podem ser utilizados para esta análise. - Coloque os neurônios tratados com ligantes TMR no prato de fundo de vidro na incubadora de topo de palco aquecida.
- Defina os parâmetros de aquisição de imagem da seguinte forma: tempo de exposição, 500 ms para canais fluorescentes Lifeact e HaloTag-actin; binning, 1 x 1 (0,065 μm × 0,065 μm por pixel); intervalo de tempo, 3 s; duração, 50 quadros.
- Selecione um cone de crescimento que expresse fortemente o Lifeact e expresse fracamente halotag-actin.
NOTA: A expressão lifeact deve ser forte o suficiente para visualizar a morfologia do cone de crescimento. A expressão halotag-actin deve ser fraca insuficiente para ser detectada (Figura 2A). Os níveis de expressões de proteína fluorescente podem ser ajustados variando a quantidade de DNA para transfecção. - Feche o campo do diafragma para iluminar uma área mínima que inclua o cone de crescimento (Figura 2B).
NOTA: A iluminação de uma área mínima reduz o sinal de fundo e aumenta a relação sinal de fluorescência para ruído (S/N), permitindo a detecção de manchas de actina (Figura 2B). Para aumentar ainda mais a relação S/N, recomenda-se iluminar o cone de crescimento sem reduzir a luz por filtros de densidade neutra. - Adquira imagens de lapso de tempo (Arquivo Suplementar 2). Salvo como uma imagem de pilha de lapso de tempo multicanal (formato de tiff).
6. Quantificações da velocidade de fluxo F-actin e taxa de polimerização usando um software de processamento e análise de imagem Fiji
NOTA: Consulte o Arquivo Suplementar 3 para obter dados de prática para quantificar a velocidade de fluxo F-actin e a taxa de polimerização da actina.
- Abra a imagem da pilha de lapso de tempo multicanal em Fiji.
- Selecione Analisar > escala de conjunto e definir o tamanho do pixel das imagens.
- Encontre uma mancha de actina que flua retrógradamente por um mínimo de cinco períodos de tempo dentro de um pacote F-actin em filopodia ou lamellipodia.
- Selecione Imagem > Transforme > Gire e ajuste o ângulo das imagens para que o pacote F-actin aponte para cima (Figura 3A).
- Clique em Retângulo na barra de ferramentas e desenhe uma caixa que inclua manchas de actin e a ponta do pacote F-actin (Figura 3B, 1 e 2).
NOTA: Os sinais das manchas de halotag-actin são fracos. Além disso, os sinais lifeact nas pontas distais do cone de crescimento são fracos e fracos (Figura 2B) porque as pontas distais do cone de crescimento são finas em comparação com a parte proximal. Os investigadores devem aprimorar os sinais para otimizar as quantificações da velocidade de fluxo F-actin e da taxa de polimerização (Figura 3B). Embora o sinal lifeact no cone de crescimento proximal fique saturado, isso não perturbará a determinação do final distal de F-actins (Figura 3E, linha amarela). - Selecione Imagem > Duplicar e insira os cinco períodos de tempo (Figura 3B, 3-5).
- Selecione > de imagem Stacks > Fazer montagem e insira os parâmetros: Colunas, 5; Linhas, 1; Fator de escala, 1.
- Clique em OK. A montagem da imagem aparecerá na tela.
- Selecione Imagem > Cores > Canais Divididos. Isso separa os dois canais fluorescentes.
- Quantificação da velocidade de fluxo F-actin.
- Selecione Analisar > definir medidas e escolha Área e Retângulo delimitante.
- Clique em Oval na barra de ferramentas e desenhe um círculo sobre uma mancha de actin (Figura 3C, 1 e 2).
- Selecione > sobreposição de imagem > adicionar seleção. O círculo é sobreposto na montagem da imagem (Figura 3C, 3 e 4).
NOTA: A cor da sobreposição pode ser alterada em Editar > Opções > Cores. - Repita a sobreposição de círculos para as manchas restantes (Figura 3C, 5).
NOTA: Para determinar com precisão os centros de mancha de actina, os investigadores devem sobrepor círculos nas manchas. - Clique em Direto na barra de ferramentas e desenhe uma linha que vincule os centros dos círculos (Figura 3D, 1 e 2).
- Selecione Analisar > Medida. O resultado aparecerá na tela (Figura 3D, 3).
NOTA: A altura exibida no resultado indica a distância de translocação da mancha de actin durante a observação (Figura 3D, 3). - Calcule a velocidade de fluxo F-actin dividindo a distância de translocação pelo tempo de observação.
- Quantificação da taxa de polimerização F-actin.
- Desenhe uma linha que vincule as pontas do pacote F-actin (Figura 3E, 1).
- Selecione Analisar > Medida. O resultado aparecerá na tela (Figura 3E, 2).
NOTA: A altura no resultado indica o comprimento de extensão do feixe F-actin durante a observação (Figura 3E, 2). - Calcule a taxa de extensão F-actin dividindo o comprimento de extensão pelo tempo de observação.
- Calcule a taxa de polimerização F-actin como a soma da velocidade de fluxo F-actin e da taxa de extensão (Figura 3F).
7. Preparação de um gel PAA para microscopia de força de tração e culturas de neurônios
- Preparação de géis PAA necessários para microscopia de força de tração (Figura 4)
- Pipeta 0,5 mL de NaOH (100 mM), dissolvida em H2O destilado, em pratos de fundo de vidro de 27 mm de diâmetro, e incubar por 15 min à temperatura ambiente (RT).
- Adicione 50 μL de 3-aminopropiltrimethyoxysilane (APTMS) na solução NaOH nos pratos e misture as soluções por tubulação suave e incubar por 15 minutos na RT.
- Lave os pratos de fundo de vidro com H2O destilado e seque.
- Pipeta 0,5 mL de solução de glutaraldeído de 0,5%, dissolvida em PBS, no prato de fundo de vidro tratado com APTMS, e incubar por 30 min na RT.
- Lave os pratos de fundo de vidro com H2O destilado e seque.
- Prepare uma solução contendo solução de acrilamida H2O destilada (412 μL), solução de acrilamida 30% (w/v) (6 μL) e solução de microesfera modificada por carboxilato (contas fluorescentes) (20 μL).
NOTA: Vortex a solução de contas fluorescentes para dissociar contas agregadas antes de adicionar à solução de acrilamida. - Degas a solução em uma câmara de vácuo por 30 minutos na RT.
- Para esta solução, adicione 1 μL de persulfeto de amônio (APS) de 10% (w/v), dissolvido em H2O destilado e 1 μL de N,N,N',N'-tetrametilenediamina (TEMED), e misture por pipetação suave.
- Coloque uma mancha de vidro (18 mm de diâmetro) na tampa de um tubo centrífuga de 15 mL.
- Adicione imediatamente 25 μL da solução ao deslizamento de tampas e, em seguida, coloque suavemente um prato de fundo de vidro invertido na tampa. Deixe a acrilamida polimerizar por 1h no RT.
- Aplique H2O destilado no deslizamento de cobertura e retire-o do gel usando uma agulha de seringa com uma ponta dobrada.
- Retire o H2O e adicione sulfo-SANPAH (1 mM), dissolvido em PBS, ao gel.
- Em condições estéreis, exponha os géis à luz ultravioleta por 5 minutos. Lave o gel três vezes com PBS por 15 minutos por lavagem.
- Determinação da rigidez do gel com um método de recuo da microesfera (Figura 5A)44.
- Transfira uma microesfera especificada para o diâmetro e densidade, para o gel e, em seguida, coloque-a no estágio de amostra de um microscópio confocal de varredura a laser.
- Concentre-se na superfície do gel e registe a posição z (Figura 5B).
- Da mesma forma, concentre-se na parte inferior da microesfera e regissão a posição z (Figura 5C).
- Calcule a profundidade do recuo conforme determinado pela diferença entre as posições z da superfície do gel e a parte inferior da microesfera.
- Calcule o módulo E do gel de Young por:
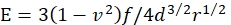
onde f é o peso corrigido pela flutuação da microesfera, d é a profundidade de recuo do gel, r é o raio da microesfera, e v é a razão de Poisson (cujo valor é 0,3, como determinado anteriormente46).
- Cubra os géis com PDL e laminina conforme descrito na seção 2.
- Como descrito nas seções 3 e 4, disseca os embriões de camundongos E16, hipocampi dissociado e transfeto com 5 μg de pEGFP-C1 por células 1,0 x 106 . Sementes 2.0 x 105 células em Géis PDL/laminin.
NOTA: Para a análise da força de tração, é necessária uma imagem microscópica de células fluorescentes com rótulos fluorescentes para determinar a área do cone de crescimento.
8. Microscopia de força de tração em cones de crescimento neuronal
- No DIV3, substitua o meio de cultura por 0,5 mL de meio L-15 aquecido contendo suplemento L-15 de 2% B-27, glutamina (1 mM), penicilina (100 U/mL) e estreptomicina (100 μg/mL). Mantenha os neurônios por 1h a 37 °C.
- Ligue um microscópio confocal de varredura a laser e coloque a incubadora de topo de palco para 37 °C.
NOTA: O presente estudo utiliza um microscópio confocal de varredura a laser equipado com uma lente objetiva de imersão de água 63x/1.2 NA e um software de aquisição e análise de imagens (ver Tabela de Materiais). - Coloque os neurônios no prato de fundo de vidro na incubadora pré-aquecida.
- Defina os parâmetros de aquisição de imagem da seguinte forma: tamanho de digitalização, 512 × 512 pixels; área de varredura, zoom de 1,5-3x; velocidade de varredura, ~1 s por quadro; comprimento de onda laser, 561 nm (o comprimento de onda de excitação para as contas fluorescentes) e 488 nm (o comprimento de onda de excitação para proteína fluorescente verde aprimorada (EGFP)); intervalo de tempo, 3 s; duração, 50 quadros.
- Para visualizar a morfologia do cone de crescimento, selecione um cone de crescimento que expresse fortemente o EGFP.
- Concentre-se na superfície do gel e adquira imagens de lapso de tempo (Figura 6A e Arquivo Suplementar 4).
NOTA: Para análise de tração, utilize dois canais fluorescentes (para contas fluorescentes e EGFP) e um canal de campo brilhante (captura de contraste de interferência diferencial (DIC) ou imagens de contraste de fase. - Use um software de processamento de imagem (por exemplo, Fiji) para produzir pilhas de imagens RGB de lapso de tempo de um único canal e salvar essas imagens como arquivos de tiff.
NOTA: É fundamental que os investigadores também salvem os dados brutos. - Aplique 100 μL de 10% (w/v) sulfato de dodecilo de sódio (SDS), dissolvido em H2O destilado, ao prato de fundo de vidro para relaxar o substrato de gel liberando neurônios do substrato. Incubar o prato por 5 min a 37°C para estabilizar a temperatura.
NOTA: A imagem das contas em um substrato não treinado é usada para referenciar as posições originais da conta durante a análise da força de tração (Figura 6C). A aplicação da solução SDS altera as posições de xiz devido à alteração térmica na incubadora e relaxamento da deformação induzida por células. Os investigadores, portanto, precisam corrigir o plano focal e a posição x-y. - Concentre-se na superfície do gel e adquira uma imagem das contas no substrato não treinado (Figura 6B).
- Produza uma imagem RGB de um único canal das contas no substrato não treinado e salve esta imagem como um arquivo de tiff.
- Correção da posição x-y da imagem de contas no substrato não treinado usando Fiji
- Em Fiji, abra a imagem das contas no substrato não treinado e a imagem da pilha de lapso de tempo de contas fluorescentes.
- Selecione > de imagens > ferramentas > Concatenato. Uma caixa de diálogo aparecerá na tela (Figura 7A, 1).
- Selecione a imagem de contas no substrato não treinado e a imagem da pilha de lapso de tempo de contas fluorescentes em Image1 e Image2, respectivamente (Figura 7A, 2).
- Clique em OK. A imagem de contas no substrato não treinado é adicionada à imagem da pilha de lapso de tempo (Figura 7A, 3).
- Usando a barra de rolagem (Figura 7B, 1, quadro vermelho), exibir o segundo quadro da imagem da pilha (seta vermelha).
- Selecione Plugins > StackReg (Figura 7B, 2). Uma caixa de diálogo aparecerá na tela (Figura 7B, 3).
- Selecione Corpo Rígido na lista de retirada (Figura 7B, 3, quadro vermelho) e clique em OK. Em seguida, começa a correção da posição x-y.
- Use a barra de rolagem para exibir o primeiro quadro da imagem da pilha corrigida pela posição x-y.
- Selecione Imagem > Duplicar (Figura 7C, 1). Uma caixa de diálogo aparecerá na tela (Figura 7B, 2).
- Entrada 1 na Gama, desmarque pilha duplicada (Figura 7C, 2) e clique em OK. A imagem de contas corrigida por posição x-y no substrato não treinado aparecerá na tela (Figura 7C, 3). Salve esta imagem como um arquivo de disputa.
- Quantificação da força de tração
NOTA: O método descrito aqui para análise de força de tração utiliza matlab e duas caixas de ferramentas MATLAB, 'Caixa de ferramentas de processamento de imagem' e 'Caixa de ferramentas de computação paralela'. Os investigadores precisarão instalá-los antes da análise. O código de análise da força de tração foi desenvolvido com base na versão MATLAB 2018a. Portanto, a versão MATLAB 2018a (ou posterior) deve ser utilizada para a análise. Os algoritmos utilizados para análise de força de tração foram descritos anteriormente22.- Baixe o código de análise de força de tração TFM2021 do Arquivo Suplementar 5. Abra o TFM 2021 no MATLAB
- Abra o principal.m em TFM2021 e execute-o. Uma interface gráfica de usuário (GUI) aparecerá na tela (Figura 8A).
- Clique em Carregar imagem de substrato não treinado e selecione a imagem de contas corrigida por posição x-y em substrato não treinado.
- Clique em Carregar imagens de contas fluorescentes e selecione a imagem da pilha de lapso de tempo de contas.
- Clique em Carregar imagens de campo brilhante e selecione a imagem da pilha de lapso de tempo do campo brilhante.
- Clique em Carregar imagens GFP e selecione a imagem da pilha de lapso de tempo do EGFP.
- Selecione GFP na lista de drop-down na GUI (Figura 8A, caixa vermelha).
- Clique no ROI para especificar a região de interesse do retângulo (ROI), incluindo o cone de crescimento, clicando em dois pontos na imagem celular exibida na GUI (Figura 8B).
- Clique no botão Salvar na GUI (Figura 8A, seta vermelha). As imagens de pilha selecionadas, juntamente com o ROI, serão salvas em um arquivo .mat (arquivo em formato MATLAB).
- Clique no Spot detect. Uma caixa de diálogo aparecerá na tela.
- Insira um valor (geralmente 50-150) na caixa de diálogo para determinar um limiar para detecção de contas. Clicar em OK inicia o cálculo.
- Após terminar o cálculo, clique na faixa Plot para ampliar a região selecionada na etapa 8.12.8 e exibir as contas detectadas como pontos brancos (Figura 8C, 1).
NOTA: Confirme se a detecção de contas (pontos brancos) se sobrepõe às contas fluorescentes. A detecção de contas também pode detectar ruído de fundo. Para reduzir essa interferência externa, altere o valor do limiar no Spot detect. Além disso, selecione manualmente os pontos corretos na etapa 8.12.13. - Clique em Selecionar contas e demarcar uma região poligonal que inclua os pontos corretos sob o cone de crescimento. Pressione Enter no teclado. Os pontos brancos dentro da região poligonal se transformarão em uma cor vermelha (Figura 8C, 2).
- Clique em Estimar força na GUI. Em seguida, valores de entrada para os seguintes parâmetros: tamanho do pixel, μm/pixel; Módulo de Young, o valor calculado na Etapa 7.2.5; Razão de Poisson, 0,3.
- Execute a força de estimativa para iniciar o cálculo. O software salvará os resultados de cálculo em um arquivo de formato de planilha automaticamente.
NOTA: A planilha mostra a magnitude do componente x, do componente y e da força em cada período (Figura 8D). Consulte o Arquivo Suplementar 6 para obter dados de prática para quantificar a força de tração.
Resultados
Imagem de uma única mancha para quantificar a taxa de polimerização de actina e eficiência de acoplamento de embreagem
Uma expressão de alta lifeact permite a visualização de F-actin dentro do cone de crescimento; uma expressão de halotag-actina baixa permite o monitoramento do fluxo retrógrado F-actin (Figura 3, Arquivo Suplementar 2). O rastreamento das manchas de actina permite a medição da velocidade de fluxo F-actin (Figura 3C,D). Uma vez que o acoplamento mecânico do fluxo retrógrado F-actin e o substrato adesivo reduz a velocidade de fluxo F-actin, a eficiência do acoplamento da embreagem pode ser estimada a partir da velocidade. Além disso, a rotulagem F-actin com lifeact auxilia a visualização da extensão F-actin e é útil para quantificar a taxa de polimerização de actina (Figura 3E,F).
Análise quantitativa da força de tração
A adesão rigorosa à metodologia aqui apresentada revelará os movimentos das contas fluorescentes sob o cone de crescimento (Figura 6C, Arquivo Suplementar 4). F-actin fluxo retrógrado para o substrato gera força de tração fazendo com que as contas fluorescentes se movam para trás sob o cone de crescimento. O código de análise da força de tração estima a força de tração do deslocamento fluorescente das contas, e expressa a força de tração calculada como vetor de força. A direção e a magnitude da força de tração são determinadas a partir dos componentes x e y do vetor de força (Figura 8C,D). A caixa vermelha na Figura 8D representa os componentes x e y de um vetor de força; A Figura 8C retrata o cone de crescimento correspondente. Em termos de coordenadas x-y, o vetor aponta para -93,8° contra o eixo x; esta orientação é direcionada para a parte traseira do cone de crescimento (Figura 8C). A magnitude da força de tração F foi calculada da seguinte forma:
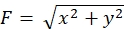
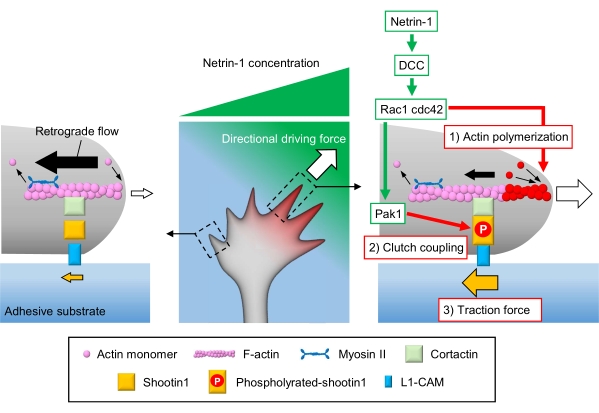
Figura 1: Uma máquina de cone de crescimento para geração de força e navegação de cone de crescimento. Um gradiente de quimioattractant netrin-1 induz a estimulação assimétrica de seu receptor DCC em um cone de crescimento axonal. Isso ativa rac1 e Cdc42, e seu Pak1 de quinase a jusante. Rac1 e Cdc42 promovem (1) polimerização de actina, enquanto pak1 fosforilatos disparam1, aumentando o acoplamento de embreagem mediado por 1 a tiros (2). A ativação assimétrica da dinâmica do actin e o acoplamento da embreagem dentro do cone de crescimento aumenta (3) a força de tração na lateral da fonte netrin-1, gerando assim uma força motriz direcional para a atração do cone de crescimento. Os protocolos aqui apresentados permitem a quantificação das variáveis-chave (1)-(3) para a navegação de cone de crescimento. Clique aqui para ver uma versão maior desta figura.

Figura 2: Imagens de fluorescência de um cone de crescimento neuronal sob um diafragma totalmente aberto e estreito. O cone de crescimento expressa Lifeact e HaloTag-actin. (A) Uma expressão de alta lifeact permite a visualização da morfologia do cone de crescimento. Por outro lado, os níveis de expressão halotag-actin são muito baixos, com sinais fracos quando o diafragma está totalmente aberto. (B) Quando o diafragma é apropriadamente reduzido, os sinais de fundo diminuem e as manchas de actina única aparecem no cone de crescimento. Barras de escala: 5 μm. Clique aqui para ver uma versão maior desta figura.

Figura 3: Etapas para quantificar a velocidade de fluxo F-actin e a taxa de polimerização de actina usando um software de processamento e análise de imagem Fiji. (A) Ajuste o ângulo da imagem para análise. (1) Encontre e selecione uma mancha de actin (ponta de flecha) que flui em um feixe F-actin. (2) Selecione > de imagem Transformar > rota. (3) Defina o ângulo para que o feixe F-actin seja direcionado para cima. (4) O ângulo da imagem será alterado. (B) Demarcar a região, incluindo a mancha de actina e o feixe F-actin. (1) Clique em Retângulo na barra de ferramentas. (2) Delineie uma região na imagem. O brilho e o contraste da imagem são aumentados para permitir uma visualização clara da mancha de actina e da ponta do feixe F-actin. (3) Selecione a > duplicata da imagem. (4) Insira os cinco períodos de tempo que mostram o fluxo de manchas de actina no feixe F-actin. (5) A imagem da pilha selecionada aparecerá na tela. (C) Sobreposição circula sobre a mancha de actina. (1) Clique em Oval na barra de ferramentas. (2) Desenhe um círculo sobre uma mancha de actina. (3) Selecione A sobreposição de > de imagem > adicionar seleção. (4) O círculo está sobreposto. 5) Repita sobreposição dos círculos sobre as manchas restantes de actin. (D) Meça a distância de translocação das manchas de actina durante os cinco períodos de tempo. (1) Clique em Reta na barra de ferramentas. (2) Desenhe uma linha que ligue os centros dos círculos. (3) Selecione Analisar > Medida. O resultado, indicado pelo parâmetro Altura (caixa vermelha), retransmitindo a distância de translocação de manchas de actina. (E) Meça a alteração no comprimento da saliência F-actin durante os cinco períodos de tempo. (1) Desenhe uma linha que vincule as pontas da saliência F-actin. (2) Selecione Analisar > Medida. O resultado (caixa vermelha), Altura, indica o comprimento de extensão do feixe F-actin. (F) A taxa de polimerização de actina é calculada a partir da soma da velocidade de fluxo F-actin e da taxa de extensão. Veja também arquivo suplementar 2. O Arquivo Suplementar 3 auxilia os investigadores a praticar a metodologia descrita acima. Clique aqui para ver uma versão maior desta figura.
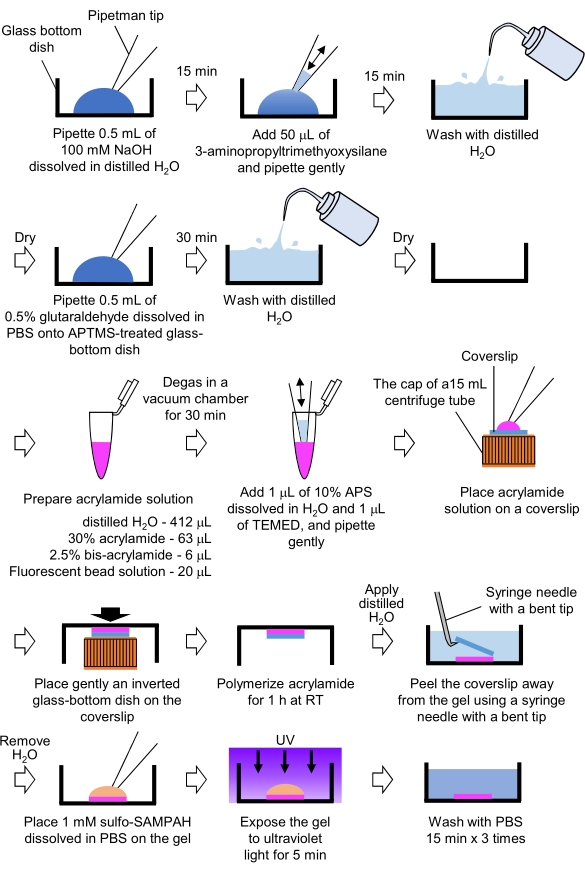
Figura 4: Passos para a preparação do gel PAA. Consulte o passo 7.1 para obter uma descrição detalhada. Clique aqui para ver uma versão maior desta figura.
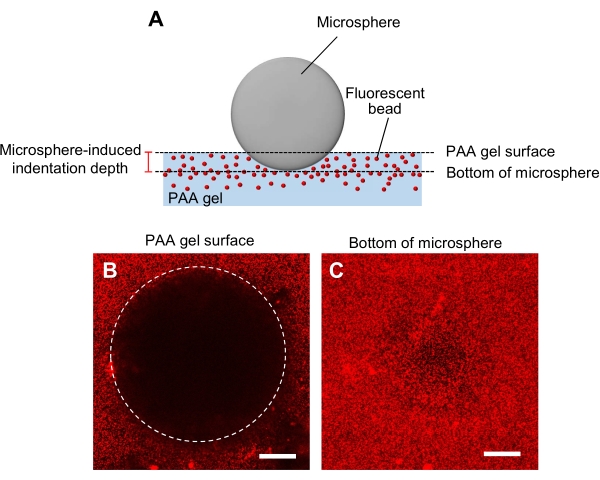
Figura 5: Determinação da rigidez do gel paa. (A) Um método de recuo da microesfera. Quando uma microesfera é colocada em um gel PAA fluorescente embutido em contas, o peso da microesfera causa um recuo no gel. A profundidade do recuo é calculada subtraindo as posições z da superfície do gel PAA da parte inferior da microesfera (B,C) Imagens de fluorescência de um gel paa recuado por uma microesfera e contendo contas fluorescentes. Um microscópio confocal de varredura a laser foi usado para capturar imagens da superfície do gel (B) e da parte inferior da microsfera (C). Os sinais das contas fluorescentes não são visíveis na superfície do gel na região recuada (B, círculo). Eles podem, no entanto, ser observados na parte inferior da microesfera. Barras de escala: 50 μm. Clique aqui para ver uma versão maior desta figura.
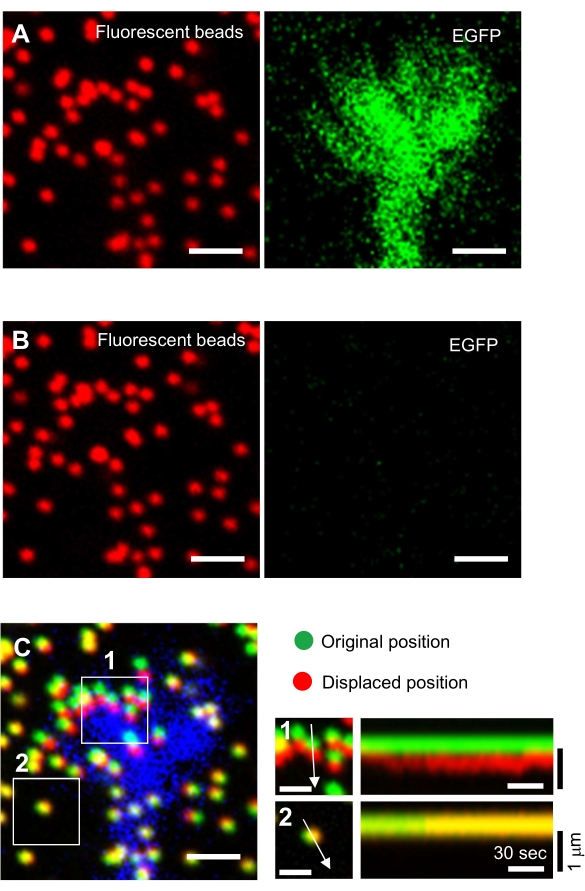
Figura 6: Mapeamento de força de um cone de crescimento neuronal. (A-C) Imagens de fluorescência de contas embutidas em um gel PAA e um cone de crescimento neural visualizado com EGFP. Após a aquisição de imagens de lapso de tempo (A), o neurônio foi liberado do substrato de gel aplicando a solução SDS, e uma imagem das contas no substrato não treinado foi capturada (B). (C) A imagem das contas no substrato não treinado mostra as contas em suas posições originais (verdes) e deslocadas (vermelhas). O sinal EGFP do cone de crescimento é mostrado em azul. Os kymógrafos (à direita) mostram os movimentos das contas em intervalos de 3 s durante uma duração de 147 s, indicadas pelas setas nas áreas encaixotadas 1 e 2. A conta na área 2 é uma conta de referência. Barras de escala: 2 μm para (A), (B) e (C, esquerda) e 1 μm para (C, médio). Veja também arquivo suplementar 4. Clique aqui para ver uma versão maior desta figura.
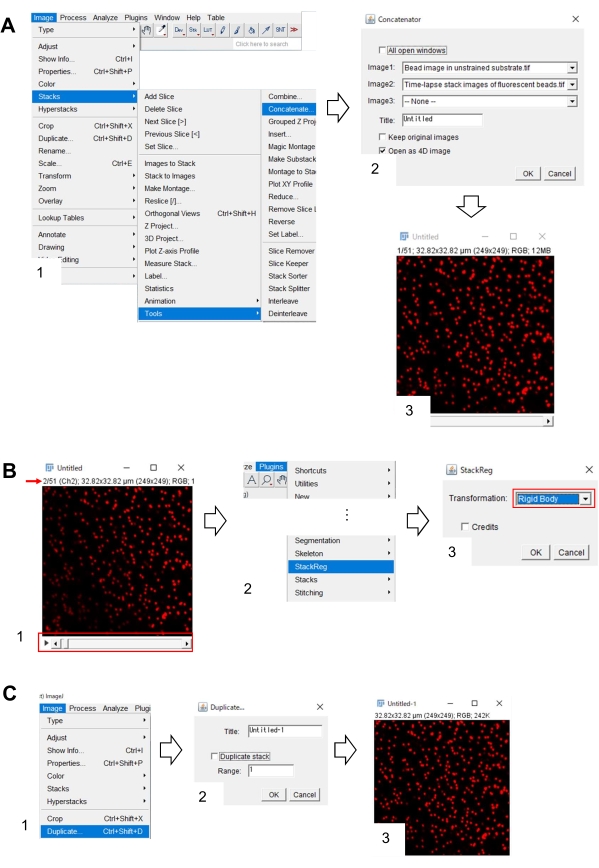
Figura 7: Passos para a posição x-y de correção da imagem de contas em substrato não treinado usando Fiji. (A) Concatenar a imagem da conta em substrato não treinado com a imagem de pilha de lapso de tempo de contas fluorescentes. (1) Selecione > de imagens > Ferramentas > Concatenato. (2) Selecione a imagem das contas em substrato não treinado e a imagem da pilha de lapso de tempo de contas fluorescentes na Imagem1 e na Imagem2, respectivamente. Clique em OK. (3) A imagem da conta no substrato não treinado é adicionada à imagem da pilha de lapso de tempo. (B) Corrija a posição x-y da imagem fluorescente da conta. (1) Use a barra de rolagem (quadro vermelho) para selecionar o segundo quadro (seta vermelha) na pilha de imagens. (2) Selecione Plugins > StackReg. (3) Selecione corpo rígido na lista de gotas (quadro vermelho) e clique em OK. A correção da posição x-y começará. (C) Salve a imagem de contas corrigida por posição x-y. Depois de selecionar o primeiro quadro da imagem de pilha corrigida por x-y, (1) selecione Imagem > Duplicar. (2) entrada 1 na pilha de intervalo e desmarcação duplicada. Em seguida, clique em OK. (3) a imagem de contas corrigida por posição x-y no substrato não treinado aparecerá na tela. Salve esta imagem como um arquivo de disputa. O Arquivo Suplementar 6 auxilia os investigadores a praticar a metodologia descrita acima. Clique aqui para ver uma versão maior desta figura.
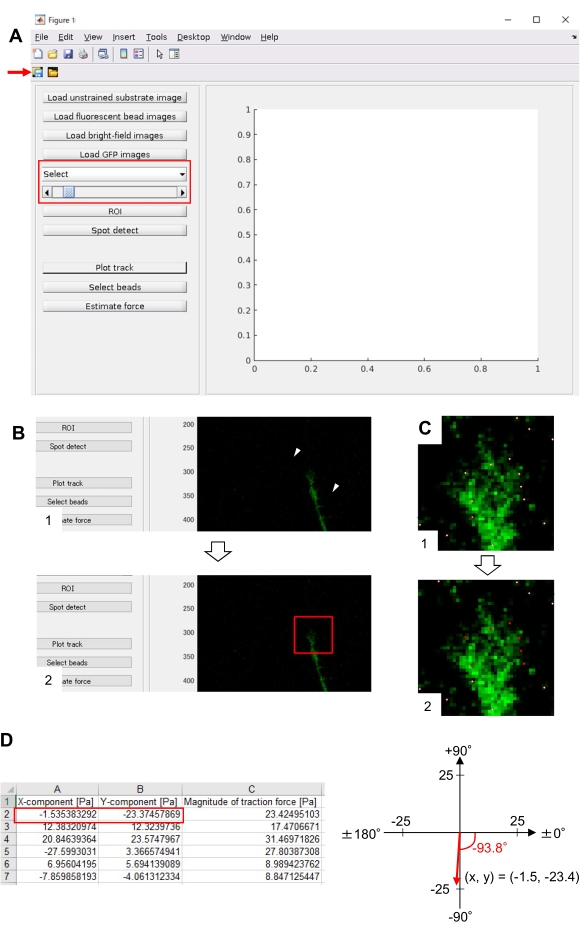
Figura 8: Análise da força de tração sob um cone de crescimento neuronal utilizando um código de análise de força de tração de código aberto. (A) GUI para análise da força de tração. As imagens de lapso de tempo selecionadas na GUI podem ser confirmadas na lista suspensa e com o controle deslizante indicado (caixa vermelha). (B) Selecione uma região que inclua o cone de crescimento. (1) Clique no ROI na GUI. Com o cursor do mouse, especifique dois pontos (pontas de seta) na imagem do celular. (2) Uma caixa vermelha aparecerá na imagem do celular. Dois cliques determinam a localização de dois cantos. (C) Selecione as contas detectadas (pontos brancos) sob o cone de crescimento. (1) Na GUI, clique em Selecionar contas e demarca uma região poligonal que inclua o cone de crescimento clicando. Pressione a tecla Enter . (2) Os pontos brancos dentro da região poligonal se transformarão em uma cor vermelha. (D) Resultados calculados da direção e magnitude da força de tração. A caixa vermelha na planilha representa os componentes x e y do vetor de força, estimados pela análise da força de tração. Na coordenada x-y no painel direito, o vetor de força gerado pelo cone de crescimento aponta para -93,8° contra o eixo x; esta orientação é direcionada para a parte traseira do cone de crescimento em (C). O Arquivo Suplementar 6 auxilia os investigadores a praticar a metodologia descrita acima. Clique aqui para ver uma versão maior desta figura.
Arquivo Suplementar 1: Receitas de soluções e mídias utilizadas neste estudo. Consulte o texto para uso detalhado. Clique aqui para baixar este Arquivo.
Arquivo suplementar 2: Imagem de fluorescência de Lifeact e imagem de manchas fluorescentes de HaloTag-actin em um cone de crescimento nervoso. Lifeact (verde) e HaloTag-actin (magenta). As imagens foram adquiridas a cada 3 s por uma duração total de 147 s. Barra de escala: 2 μm. Veja também Figura 3. Clique aqui para baixar este Arquivo.
Arquivo Suplementar 3: Pratique dados para quantificar a velocidade de fluxo F-actin e a taxa de polimerização de actina. Uma imagem multicanal de pilha de lapso de tempo de Lifeact (verde) e HaloTag-actin (magenta). Veja também a Figura 3. Clique aqui para baixar este Arquivo.
Arquivo Suplementar 4: Um vídeo de mapeamento de força detectando força de tração em um cone de crescimento neuronal. As posições originais (verdes) e deslocadas (vermelhas) das contas. O sinal EGFP no cone de crescimento é mostrado em azul. As imagens foram adquiridas a cada 3 s por 147 s. Barra de escala: 2 μm. Veja também Figura 6. Clique aqui para baixar este Arquivo.
Arquivo Suplementar 5: Código de análise da força de tração. Consulte o Passo 8.12 para uso detalhado. Clique aqui para baixar este Arquivo.
Arquivo Suplementar 6: Pratique dados para quantificar a força de tração. Uma imagem RGB de um único canal das contas em substrato não treinado e imagens RGB de pilha de tempo de um único canal de contas fluorescentes sob um cone de crescimento, EGFP e campo brilhante. Veja também as Figuras 7 e 8. Clique aqui para baixar este Arquivo.
Discussão
Os protocolos descritos neste estudo utilizam materiais e equipamentos de microscopia disponíveis comercialmente encontrados rotineiramente em todos os laboratórios, institutos e universidades. Portanto, os pesquisadores podem facilmente adotar a atual microscopia de imagem de mancha única e força de tração em seus estudos.
A imagem de manchas pode analisar a polimerização de actin e o acoplamento da embreagem. Além disso, a imagem de manchas pode monitorar o fluxo retrógrado de moléculas de embreagem como shootin1 e cortactina, que interagem com f-actin fluxo retrógrado. Usando um microscópio TIRF, o fluxo retrógrado da molécula de adesão celular L1-CAM também pode ser monitorado23,41; O L1-CAM sofre comportamentos de aderência e deslizamento que refletem a eficiência do acoplamento de embreagem23,41. Embora o presente estudo empregue o sistema TMR-HaloTag para imagens de manchas, outras proteínas fluorescentes, como eGFP e proteína fluorescente vermelha monomérica, também estão disponíveis nas análises16,18,20,23,24,27,39. O essencial para visualizar as manchas de actina são um baixo nível de expressão de actina fluorescente e a iluminação de uma área mínima (Figura 2). Neste protocolo, os sinais lifeact e halotag-actin são adquiridos sequencialmente. Como o fluxo retrógrado da actina é relativamente lento (4,5 ± 0,1 μm/min)24, a análise do fluxo f-actin retrógrado e da polimerização de actina não são afetadas pela aquisição de imagem sequencial de diferentes canais fluorescentes (~1 s intervalo). Lifeact é um marcador F-actin amplamente utilizado, mas pode competir com proteínas de ligação actina47. De maior importância, o Lifeact pode alterar a dinâmica da actina, afetando assim as estruturas F-actin e a morfologia celular47,48,49.
Microscopia de força de tração pode detectar forças para impulsionar o avanço do cone de crescimento. Ao montar os neurônios dentro de uma matriz extracelular, os pesquisadores também podem analisar forças geradas em um ambiente semi-3D11. A imagem de alta ampliação é importante para uma quantificação precisa da força de tração porque os cones de crescimento geram forças de tração fracas7. Embora outros métodos com nanopillars ou biosensores sensíveis ao estresse também sejam usados para medir a força de tração50,51, o método baseado em gel paa é altamente adaptável e permite o ajuste da rigidez do substrato variando as concentrações de acrilamida e bis-acrilamida41,44,52. Neste protocolo, o gel PAA é preparado em uma concentração final de 3,75% de acrilamida e 0,03% de bis-acrilamida; O módulo de Young é ~270 Pa22 e essa rigidez está dentro da faixa de tecido cerebral (100-10.000 Pa)53,54,55. Devido à espessura do gel PAA (~100 μm), este método limita o uso de lentes de alta ampliação durante a microscopia. Para obter imagens de alta ampliação, os pesquisadores devem usar a função zoom em um microscópio confocal de varredura a laser.
Em conclusão, a atual microscopia de imagem e força de tração permite análises quantitativas dos principais eventos em vigor gerações. Essas informações serão inestimáveis para melhorar a compreensão dos mecanismos que fundamentam o avanço do cone de crescimento e a navegação.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Esta pesquisa foi apoiada em parte pela AMED sob o número de subvenção 21gm0810011h0005 (N.I. e Y.S.), JSPS KAKENHI (JP19H03223, N.I.) e JSPS Grants-in-Aid for Early-Career Scientists (JP19K16258, T.M.), a Fundação de Pesquisa Médica de Osaka para Doenças Incuráveis (T.M.), e o Naist Next Generation Interdisciplinar Research Project (Y.S.).
Materiais
| Name | Company | Catalog Number | Comments |
| 0.5% trypan blue stain solution | Nacalai | 29853-34 | |
| 3-aminopropyltrimethyoxysilane | Sigma | 281778-100ML | |
| Acrylamide monomer | Nacalai | 00809-85 | |
| Ammonium persulphate | Cytiva | 17-1311-1 | |
| Axio Observer Z1 | Zeiss | 431007-9902-000 | Epi-fluorescence microscope (single speckle imaging) |
| B-27 supplement (50x) | Thermo Fisher Scientific | 17504-044 | |
| Bovine serum albmine | Sigma | A7906-10G | |
| C-Apochromat 63x/1.2 W Corr | Zeiss | 421787-9970-799 | Objective lens (traction force microscopy) |
| Coverslip (diameter 18 mm) | Matsunami | C018001 | |
| D-glucose | Nacalai | 16806-25 | |
| DNaseI | Sigma | DN25-100MG | |
| Fetal bovine serum | Thermo Fisher Scientific | 10270-106 | |
| Fiji | Open source software package | https://imagej.net/software/fiji/ | |
| FluoSpheres carboxylate-modified 0.2 mm, red (580/605), 2% solid | Thermo Fisher Scientific | F8810 | carboxylate-modified microspheres |
| Glass bottom dish (14 mm diameter) | Matsunami | D1130H | |
| Glass bottom dish (27 mm diameter) | Matsunami | D1140H | |
| Glutaraldehyde solution | Sigma | G5882-10X10ML | |
| HaloTag TMR ligand | Promega | G8251 | |
| HBO103 W/2 | Osram | 4050300382128 | Mercury lamp (single speckle imaging) |
| Image Processing Toolbox | MathWork | https://www.mathworks.com/products/image.html | |
| Laminin solution from mouse EHS tumor | Wako | 120-05751 | |
| Leibovitz’s L-15 medium | Thermo Fisher Scientific | 11415064 | |
| L-glutamine | Nacalai | 16919-42 | |
| LSM710 | Zeiss | N/A | Conforcal laser microscope (traction force microscopy) |
| MATLAB2018a | MathWork | https://www.mathworks.com/products/new_products/release2018a.html | |
| Mouse C57BL/6 | Japan SLC | N/A | |
| Mouse neuron nucleofector kit | Lonza | VPG-1001 | |
| N,N,N’,N’-tetramethylethylenediamine (TEMED) | Nacalai | 33401-72 | |
| N,N’-methylenebisacrylamide | Nacalai | 22402-02 | |
| Neurobasal medium | Thermo Fisher Scientific | 21103-049 | |
| Nucleofector I | Amaxa | AAD-1001 | Electroporation apparatus |
| ORCA Flash 4.0 V2 | Hamamatsu | C11440-22CU | CMOS camera (single speckle imaging) |
| Papain | Nacalai | 26036-34 | |
| Parallel Computing Toolbox | MathWork | https://www.mathworks.com/products/parallel-computing.html | |
| pEGFP-C1 | Clontech | 1528177 | |
| Penicillin-streptomycin (100x) | Nacalai Tesque | 26253-84 | |
| pFN21A-HaloTag-actin | (Minegishi et al., 2018) | N/A | |
| Phosphate buffered saline (PBS) pH 7.4 (10x) | Thermo Fisher Scientific | 70011-044 | |
| Plan-Apochromat 100x/1.4 Oil | Zeiss | 420790-9901-000 | Objective lens (single speckle imaging) |
| pmNeonGreen-N1-Lifeact | (Kastian et al., 2021) | N/A | |
| Poly-D-lysine hydrobromide | Sigma | P6407-5MG | |
| Slulfo-SAMPHA | Thermo Fisher Scientific | 22589 | |
| Sodium dodecyl sulfate | Nacalai | 08933-05 | |
| Sodium hydrate (NaOH) | Nacalai | 31511-05 | |
| Steel ball | Sako tekkou | N/A | Microshpere to determin PAA gel rigidity. 0.6 mm diameter, 7.87 g/cm3. |
| ZEN2009 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (traction force microscopy) |
| ZEN2012 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (single speckle imaging) |
Referências
- TessierLavigne, M., Goodman, C. S. The molecular biology of axon guidance. Science. 274, 1123-1133 (1996).
- Marin, O., Valiente, M., Ge, X., Tsai, L. H. Guiding neuronal cell migrations. Cold Spring Harbor Perspectives in Biology. 2, 001834 (2010).
- Vitriol, E. A., Zheng, J. Q. Growth cone travel in space and time: the cellular ensemble of cytoskeleton, adhesion, and membrane. Neuron. 73 (6), 1068-1081 (2012).
- Cooper, J. A. Cell biology in neuroscience: mechanisms of cell migration in the nervous system. Journal of Cell Biology. 202, 725-734 (2013).
- Dupraz, S., et al. RhoA controls axon extension independent of specification in the developing brain. Current Biology. 29, 3874-3886 (2019).
- Suter, D. M., Miller, K. E. The emerging role of forces in axonal elongation. Progress in Neurobiology. 94, 91-101 (2011).
- Franze, K. Integrating chemistry and mechanics: the forces driving axon growth. Annual Review of Cell and Developmental Biology. 36, 61-83 (2020).
- Mitchison, T., Kirschner, M. Cytoskeletal dynamics and nerve growth. Neuron. 1, 761-772 (1988).
- Suter, D. M., Forscher, P. Substrate-cytoskeletal coupling as a mechanism for the regulation of growth cone motility and guidance. Journal of Neurobiology. 44, 97-113 (2000).
- Lowery, L. A., Van Vactor, D. The trip of the tip: understanding the growth cone machinery. Nature Reviews Molecular Cell Biology. 10, 332-343 (2009).
- Minegishi, T., et al. Shootin1b mediates a mechanical clutch to produce force for neuronal migration. Cell Reports. 25, 624-639 (2018).
- Minegishi, T., Inagaki, N. Forces to drive neuronal migration steps. Frontiers in Cell and Developmental Biology. 8, 863 (2020).
- Forscher, P., Smith, S. J. Actions of cytochalasins on the organization of actin filaments and microtubules in a neuronal growth cone. Journal of Cell Biology. 107, 1505-1516 (1988).
- Suter, D. M., Forscher, P. An emerging link between cytoskeletal dynamics and cell adhesion molecules in growth cone guidance. Current Opinion in Neurobiology. 8, 106-116 (1998).
- Pollard, T. D., Borisy, G. G. Cellular motility driven by assembly and disassembly of actin filaments. Cell. 112, 453-465 (2003).
- Waterman-Storer, C. M., Desai, A., Bulinski, J. C., Salmon, E. D. Fluorescent speckle microscopy, a method to visualize the dynamics of protein assemblies in living cells. Current Biology. 8, 1227-1230 (1998).
- Katoh, K., Hammar, K., Smith, P. J., Oldenbourg, R. Birefringence imaging directly reveals architectural dynamics of filamentous actin in living growth cones. Molecular Biology of the Cell. 10, 197-210 (1999).
- Watanabe, N., Mitchison, T. J. Single-molecule speckle analysis of actin filament turnover in lamellipodia. Science. 295, 1083-1086 (2002).
- Medeiros, N. A., Burnette, D. T., Forscher, P. Myosin II functions in actin-bundle turnover in neuronal growth cones. Nature Cell Biology. 8, 215-226 (2006).
- Shimada, T., et al. Shootin1 interacts with actin retrograde flow and L1-CAM to promote axon outgrowth. Journal of Cell Biology. 181, 817-829 (2008).
- He, M., Zhang, Z. H., Guan, C. B., Xia, D., Yuan, X. B. Leading tip drives soma translocation via forward F-actin flow during neuronal migration. Jounal of Neuroscience. 30, 10885-10898 (2010).
- Toriyama, M., Kozawa, S., Sakumura, Y., Inagaki, N. Conversion of a signal into forces for axon outgrowth through Pak1-mediated shootin1 phosphorylation. Current Biology. 23, 529-534 (2013).
- Abe, K., et al. Grip and slip of L1-CAM on adhesive substrates direct growth cone haptotaxis. Proceedings of the National Academy of Sciences of the United States of America. 115, 2764-2769 (2018).
- Baba, K., et al. Gradient-reading and mechano-effector machinery for netrin-1-induced axon guidance. Elife. 7, 34593 (2018).
- Nichol, R. I., Hagen, K. M., Lumbard, D. C., Dent, E. W., Gomez, T. M. Guidance of axons by local coupling of retrograde flow to point contact adhesions. Journal of Neuroscience. 36, 2267-2282 (2016).
- Shekarabi, M., et al. Deleted in colorectal cancer binding netrin-1 mediates cell substrate adhesion and recruits Cdc42, Rac1, Pak1, and N-WASP into an intracellular signaling complex that promotes growth cone expansion. Journal of Neuroscience. 25, 3132-3141 (2005).
- Kubo, Y., et al. Shootin1-cortactin interaction mediates signal-force transduction for axon outgrowth. Journal of Cell Biology. 210, 663-676 (2015).
- Huber, A. B., Kolodkin, A. L., Ginty, D. D., Cloutier, J. F. Signaling at the growth cone: ligand-receptor complexes and the control of axon growth and guidance. Annual Review of Neuroscience. 26, 509-563 (2003).
- Kolodkin, A. L., Tessier-Lavigne, M. Mechanisms and molecules of neuronal wiring: a primer. Cold Spring Harbor Perspectives in Biology. 3, 001727 (2011).
- Ayala, R., Shu, T., Tsai, L. H. Trekking across the brain: the journey of neuronal migration. Cell. 128, 29-43 (2007).
- Tatavarty, V., Kim, E. J., Rodionov, V., Yu, J. Investigating sub-spine actin dynamics in rat hippocampal neurons with super-resolution optical imaging. PLoS One. 4, 7724 (2009).
- Frost, N. A., Shroff, H., Kong, H., Betzig, E., Blanpied, T. A. Single-molecule discrimination of discrete perisynaptic and distributed sites of actin filament assembly within dendritic spines. Neuron. 67, 86-99 (2010).
- Chazeau, A., et al. Nanoscale segregation of actin nucleation and elongation factors determines dendritic spine protrusion. EMBO Journal. 33, 2745-2764 (2014).
- Garcia, M., et al. Two-tiered coupling between flowing actin and immobilized N-cadherin/catenin complexes in neuronal growth cones. Proceedings of the National Academy of Sciences of the United States of America. 112, 6997-7002 (2015).
- Swaminathan, V., et al. Actin retrograde flow actively aligns and orients ligand-engaged integrins in focal adhesions. Proceedings of the National Academy of Sciences of the United States of America. 114, 10648-10653 (2017).
- Tsai, T. Y., et al. Efficient front-rear coupling in neutrophil chemotaxis by dynamic myosin II localization. Developmental Cell. 49, 189-205 (2019).
- Zhang, X. F., et al. Regulation of axon growth by myosin II-dependent mechanocatalysis of cofilin activity. Journal of Cell Biology. 218 (7), 2329-2349 (2019).
- Reversat, A., et al. Cellular locomotion using environmental topography. Nature. 582, 582-585 (2020).
- Katsuno, H., et al. Actin migration driven by directional assembly and disassembly of membrane-anchored actin filaments. Cell Reports. 12, 648-660 (2015).
- Urasaki, A., et al. Shootins mediate collective cell migration and organogenesis of the zebrafish posterior lateral line system. Scientific Reports. 9, 12156 (2019).
- Abe, K., et al. Mechanosensitive axon outgrowth mediated by L1-laminin clutch interface. Biophysical Journal. 120, 3566-3576 (2021).
- Kastian, R. F., et al. Shootin1a-mediated actin-adhesion coupling generates force to trigger structural plasticity of dendritic spines. Cell Reports. 35, 109130 (2021).
- Riedl, J., et al. Lifeact: a versatile marker to visualize F-actin. Nature Methods. 5, 605-607 (2008).
- Chan, C. E., Odde, D. J. Traction dynamics of filopodia on compliant substrates. Science. 322, 1687-1691 (2008).
- Wang, Y. L., Pelham, R. J. Preparation of a flexible, porous polyacrylamide substrate for mechanical studies of cultured cells. Methods in Enzymology. 298, 489-496 (1998).
- Li, Y., Hu, Z., Li, C. New method for measuring poisson's ratio in polymer gels. Journal of Applied Polymer Science. 50, 1107-1111 (1993).
- Belyy, A., Merino, F., Sitsel, O., Raunser, S. Structure of the Lifeact-F-actin complex. PLoS Biology. 18, 3000925 (2020).
- Flores, L. R., Keeling, M. C., Zhang, X., Sliogeryte, K., Gavara, N. Lifeact-GFP alters F-actin organization, cellular morphology and biophysical behaviour. Scientific Reports. 9, 3241 (2019).
- Kumari, A., Kesarwani, S., Javoor, M. G., Vinothkumar, K. R., Sirajuddin, M. Structural insights into actin filament recognition by commonly used cellular actin markers. EMBO Journal. 39, 104006 (2020).
- du Roure, O., et al. Force mapping in epithelial cell migration. Proceedings of the National Academy of Sciences of the United States of America. 102, 2390-2395 (2005).
- Grashoff, C., et al. Measuring mechanical tension across vinculin reveals regulation of focal adhesion dynamics. Nature. 466, 263-266 (2010).
- Koch, D., Rosoff, W. J., Jiang, J., Geller, H. M., Urbach, J. S. Strength in the periphery: growth cone biomechanics and substrate rigidity response in peripheral and central nervous system neurons. Biophysical Journal. 102 (3), 452-460 (2012).
- Barnes, J. M., Przybyla, L., Weaver, V. M. Tissue mechanics regulate brain development, homeostasis and disease. Journal of Cell Science. 130, 71-82 (2017).
- Moore, S. W., Roca-Cusachs, P., Sheetz, M. P. Stretchy proteins on stretchy substrates: the important elements of integrin-mediated rigidity sensing. Developmental Cell. 19, 194-206 (2010).
- Tyler, W. J. The mechanobiology of brain function. Nature Reviews Neuroscience. 13, 867-878 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados