Method Article
Analisi della dinamica dell'actina, dell'accoppiamento della frizione e della forza di trazione per l'avanzamento del cono di crescita
In questo articolo
Riepilogo
Per avanzare, i coni di crescita devono esercitare forze di trazione contro l'ambiente esterno. La generazione delle forze di trazione dipende dalla dinamica dell'actina e dall'accoppiamento della frizione. Il presente studio descrive i metodi per analizzare la dinamica dell'actina, l'accoppiamento della frizione e le forze di trazione per l'avanzamento del cono di crescita.
Abstract
Per stabilire reti funzionali, i neuroni devono migrare verso le loro destinazioni appropriate e quindi estendere gli assoni verso le loro cellule bersaglio. Questi processi dipendono dai progressi dei coni di crescita che si trovano sulle punte dei neuriti. I coni di crescita assonale generano forze motrici rilevando il loro microambiente locale e modulando la dinamica citoscheletrica e l'accoppiamento actina-adesione (accoppiamento frizione). Decenni di ricerca hanno portato all'identificazione di molecole guida, dei loro recettori e di cascate di segnalazione a valle per la regolazione della migrazione neuronale e della guida assonale; tuttavia, i macchinari molecolari necessari per generare forze per guidare l'avanzamento del cono di crescita e la navigazione stanno appena iniziando a essere chiariti. All'avanguardia dei coni di crescita neuronale, i filamenti di actina subiscono un flusso retrogrado, che è alimentato dalla polimerizzazione dell'actina e dalla contrazione dell'actomiosina. Un accoppiamento della frizione tra il flusso retrogrado di F-actina e il substrato adesivo genera forze di trazione per l'avanzamento del cono di crescita. Il presente studio descrive un protocollo dettagliato per il monitoraggio del flusso retrogrado di F-actina mediante imaging a singola macchia. È importante sottolineare che, se combinata con un marcatore F-actina Lifeact, questa tecnica può quantificare 1) il tasso di polimerizzazione F-actina e 2) l'efficienza di accoppiamento della frizione tra il flusso retrogrado F-actina e il substrato adesivo. Entrambe sono variabili critiche per generare forze per l'avanzamento e la navigazione del cono di crescita. Inoltre, il presente studio descrive un protocollo dettagliato di microscopia della forza di trazione, che può quantificare 3) la forza di trazione generata dai coni di crescita. Pertanto, accoppiando le analisi dell'imaging a singola macchia e della microscopia della forza di trazione, i ricercatori possono monitorare la meccanica molecolare alla base dell'avanzamento e della navigazione del cono di crescita.
Introduzione
Nel cervello vertebrato in via di sviluppo, i neuroni subiscono migrazioni organizzate in modo elaborato e proiettano assoni verso partner sinaptici appropriati per stabilire reti neuronali funzionali1,2,3. I coni di crescita, che sono strutture sensoriali e mobili situate sulla punta dei neuriti, determinano la velocità e la direzione della migrazione neuronale e della crescita degli assoni3,4,5. Poiché i neuroni sono circondati da ambienti strettamente imballati, i coni di crescita devono esercitare forze contro il loro ambiente per andare avanti6,7. Per comprendere i meccanismi alla base della migrazione neuronale e della guida assonale, sono essenziali analisi della meccanica molecolare per l'avanzamento del cono di crescita.
Decenni di analisi hanno rivelato che la forza di trazione per guidare l'avanzamento del cono di crescita è generata dal meccanismo della "frizione"; si pensa che questo meccanismo funzioni non solo nel cono di crescita assonale, ma anche nel principale cono di crescita del processo dei neuroni migratori8,9,10,11,12. Vale a dire, i filamenti di actina (F-actine) nei coni di crescita polimerizzano sul bordo d'attacco e depolimerizzano prossimalmente, spingendo fuori la membrana all'avanguardia13,14,15. La forza risultante, in combinazione con la contrazione dell'actomiosina, induce il movimento all'indietro delle F-actine chiamate flusso retrogrado7,11,16,17,18,19,20,21. Le molecole di adesione della frizione e della cellula mediano l'accoppiamento meccanico tra il flusso retrogrado di F-actina e il substrato adesivo e trasmettono la forza del flusso di F-actina sul substrato, generando così forza di trazione per l'avanzamento del cono di crescita7,8,9,11,12,22 . Allo stesso tempo, l'accoppiamento actina-substrato riduce la velocità del flusso F-actina e converte la polimerizzazione dell'actina nella forza di sporgere la membrana all'avanguardia9,10.
I coni di crescita assonale rilevano i segnali chimici locali e li trasducono in una forza motrice direzionale per la navigazione del cono di crescita3,23,24,25. Ad esempio, una molecola guida assonale netrin-1 stimola il suo recettore eliminato nel cancro del colon-retto (DCC) e attiva la proteina di controllo della divisione cellulare 42 (Cdc42) e il substrato 1 (Rac1) della tossina botulinica C3 correlata a Rho guanosina trifosfato (GTP) e la loro chinasi 1 attivata dalla chinasi p21 a valle (Pak1)26. Cdc42 e Rac1 promuovono 1) polimerizzazione dell'actina e Pak1 fosforila una molecola di frizione shootin122,26. Shootin1 interagisce con il flusso retrogrado di F-actina attraverso una proteina legante l'actina cortactin27. Shootin1 interagisce anche con la molecola di adesione cellulare L1 (L1-CAM)20,24. La fosforilazione Shootin1 aumenta le affinità di legame per cortactina e L1-CAM e migliora l'accoppiamento frizione 2) mediato shootin124,27. All'interno del cono di crescita, le attivazioni asimmetriche della polimerizzazione dell'actina e dell'accoppiamento della frizione aumentano 3) la forza di trazione sul lato della sorgente netrin-1, generando così forza motrice direzionale per la rotazione del cono di crescita (Figura 1)24. Un'intensa ricerca negli ultimi decenni per quanto riguarda la migrazione neuronale e la guida degli assoni ha migliorato la comprensione delle molecole guida, dei loro recettori e delle cascate di segnalazione a valle associate2,10,28,29,30. Tuttavia, i macchinari molecolari per generare forze per l'avanzamento del cono di crescita stanno appena iniziando a essere chiariti; ciò può essere attribuito all'uso limitato dei protocolli per le analisi meccanobiologiche.
Il presente studio descrive un protocollo dettagliato per il monitoraggio del flusso retrogrado di F-actina mediante imaging a singola macchia16,18. Il monitoraggio del flusso retrogrado di F-actina è stato ampiamente eseguito utilizzando microscopia a super-risoluzione, microscopia confocale a disco rotante e microscopia a fluorescenza a riflessione totale ad interferenza totale (TIRF)25,31,32,33,34,35,36,37,38 . Il protocollo nel presente studio, tuttavia, utilizza un microscopio a epifluorescenza standard ed è quindi facilmente adottabile11,16,18,20,22,23,24,27,39,40,41,42. In combinazione con l'etichettatura F-actina di Lifeact43, l'imaging a singola macchia consente di quantificare la velocità di polimerizzazione dell'actina e l'efficienza di accoppiamento della frizione tra il flusso retrogrado di F-actina e il substrato adesivo39,42. Il presente studio descrive inoltre un protocollo dettagliato di microscopia della forza di trazione utilizzando un gel di poliacrilammide (PAA) incorporato in perline fluorescenti11,22,23,24,27,39,41,42,44. Questo metodo rileva e quantifica la forza di trazione sotto il cono di crescita monitorando i movimenti delle perline indotti dalla forza44,45. Viene fornito un codice di analisi della forza di trazione open source e viene spiegato in dettaglio il metodo per quantificare la forza di trazione durante la migrazione del cono di crescita. Con l'aiuto dell'imaging a singola macchia e della microscopia della forza di trazione, sarà facilitata la comprensione della meccanica molecolare alla base della migrazione e della navigazione del cono di crescita. Queste tecniche sono applicabili anche per analizzare la meccanica molecolare alla base dell'allargamento della colonna vertebrale dendritica, che è noto per essere importante nell'apprendimento e nella memoria42.
Protocollo
Tutti gli esperimenti che utilizzano animali da laboratorio sono stati eseguiti con il Comitato istituzionale per la cura e l'uso degli animali del Nara Institute of Science and Technology. Gli sperimentatori dovrebbero seguire le linee guida stabilite dai loro comitati istituzionali e nazionali di regolamentazione degli animali per la cura e l'uso degli animali da laboratorio.
1. Preparazione di soluzioni e media
- Preparare le soluzioni e i supporti di coltura come riepilogato nel file supplementare 1
2. Preparazione di substrati rivestiti di poli-D-lisina (PDL)/laminina
- Rivestire piatti con fondo di vetro da 14 mm di diametro con 100 μg/mL PDL, disciolti in soluzione salina tamponata con fosfato (PBS, pH 7,4), e incubare per la notte in un incubatore umidificato a 37 °C.
NOTA: non asciugare le superfici vetrate dopo questo passaggio - Rimuovere la soluzione PDL e la pipetta 1 mL di PBS sul vetro per 10 s tre volte.
- Rivestire le piastre con laminina da 5 μg/mL, sciolta in PBS, e incubare per la notte in un incubatore umidificato a 37 °C.
- Rimuovere la soluzione di laminina e la pipetta 1 mL di PBS sul vetro per 10 s tre volte.
- Rimuovere il PBS e posizionare 0,5 ml del mezzo neurobasale sulle superfici vetrate. Conservare i piatti in un incubatore umidificato a 37 °C.
NOTA: i piatti rivestiti di PDL/laminina possono essere conservati per 2-3 giorni in un incubatore umidificato a 37 °C.
3. Dissezione e dissociazione dell'ippocampo
- Eutanasia di un topo in gravidanza per lussazione cervicale.
NOTA: Il presente studio utilizza topi disponibili in commercio (vedi Tabella dei materiali). I ricercatori dovrebbero usare topi allevati e trattati umanamente in un ambiente sterilizzato. - Seziona gli embrioni embrionali di topo del giorno 16 (E16) e mettili sul ghiaccio.
- In una cappa a flusso laminare, sezionare il cervello con le forbici e metterli su un piatto sterile di 10 cm contenente 10 ml di soluzione di dissezione ghiacciata.
- Con uno stereomicroscopio a dissezione, staccare con cura le meningi degli emisferi cerebrali e quindi sezionare l'ippocampo usando una pinza.
- Trasferire l'ippocampo in 5 mL di soluzione di digestione ghiacciata in un tubo da 15 ml. Incubare l'ippocampo a bagnomaria per 20 minuti a 37 °C.
- Rimuovere la soluzione di digestione e aggiungere 3 ml di soluzione di dissezione.
- Pipettare delicatamente l'ippocampo quattro volte con una pipetta Pasteur.
- Incubare l'ippocampo a bagnomaria per 20 minuti a 37 °C.
- Trasferire la sospensione cellulare in un tubo centrifugo da 50 ml. Aggiungere 3 ml di nuova soluzione di dissezione al tessuto non dissociato.
- Ripeti i passaggi 3.7-3.9 fino a quando l'ippocampo non è completamente dissociato.
- Rimuovere gli aggregati galleggianti di DNA dalla sospensione cellulare ruotando e aspirando con una pipetta Pasteur. Centrifugare la sospensione cellulare a 180 x g per 20 min a 4 °C.
NOTA: il DNA derivato da cellule danneggiate disturberà il processo di centrifugazione. Se i neuroni rimangono nel surnatante, centrifugare nuovamente la sospensione cellulare dopo aver accuratamente rimosso il DNA. - Equilibrare il mezzo neurobasale contenente il 10% di siero bovino fetale (FBS), penicillina (100 U/mL) e streptomicina (100 μg/mL) in un incubatore umidificato a 37 °C con il 5% di CO2.
4. Trasfezione e coltura dei neuroni
- Rimuovere il surnatante e aggiungere 5 ml di PBS ghiacciato nel tubo da 50 ml. Risospesso il pellet cellulare mediante un delicato pipettaggio.
- Trasferire 10 μL della sospensione cellulare in un tubo microcentrifuga, aggiungere 10 μL di soluzione blu di tripano allo 0,5% e quindi contare il numero di cellule con un emocitometro.
- Centrifugare la sospensione cellulare nel tubo da 50 mL a 180 x g per 20 min a 4 °C.
- Nel frattempo, aliquota per la trasfezione, 1 μg di pFN21A-HaloTag-actina e 3 μg di pmNeonGreen-N1-Lifeact per 1 x 106 cellule, in un tubo microcentrifuga.
- Dopo la centrifugazione, rimuovere il surnatante e aggiungere 100 μL di mezzo di elettroporazione (fornito dal kit di trasfezione) nel tubo da 50 ml. Risospesso il pellet cellulare mediante pipettaggio.
- Mescolare la sospensione cellulare con la soluzione di DNA aliquotato e trasferire la miscela in una cuvetta (fornita dal kit di trasfezione).
NOTA: poiché le bolle d'aria disturbano l'elettroporazione, rimuoverle dalla sospensione cellulare. - Inserire la cuvetta nell'apparato di elettroporazione (Tabella dei materiali) ed eseguire l'elettroporazione utilizzando il programma O-005.
- Aggiungere immediatamente 1 mL del mezzo neurobasale preriscaldato ed equilibrato contenente il 10% di FBS, penicillina (100 U/mL) e streptomicina (100 μg/mL) alla cuvetta.
- Trasferire la sospensione cellulare in un tubo centrifugo da 15 ml utilizzando una pipetta di plastica (fornita dal kit di trasfezione).
- Trasferire 10 μL della sospensione cellulare in un microtubo, aggiungere 10 μL di soluzione blu di tripano allo 0,5% e quindi contare il numero di cellule con un emocitometro.
- Estrarre i piatti con fondo di vetro rivestiti di PDL / laminina dall'incubatrice e rimuovere il mezzo neurobasale.
- Pipettare 0,5 mL della sospensione cellulare contenente 2,0 x 105 celle per piatto e incubare in un incubatore umidificato a 37 °C con 5% di CO2 per 3 ore.
- Sostituire il mezzo con 0,5 mL di mezzo neurobasale contenente il 2% di integratore B-27, glutammina (1 mM), penicillina (100 U / mL) e streptomicina (100 μg / mL). Coltivare i neuroni in un incubatore umidificato per 3 giorni a 37 °C con il 5% di CO2.
5. Imaging a singola macchia ai coni di crescita neuronale
- Il giorno in vitro (DIV) 3, trattare i neuroni con ligando tetrametil-rodamina (TMR) ad una diluizione di 1:2000 diluizione in terreno di coltura. Mantenere i neuroni per 1 ora a 37 °C con il 5% di CO2.
- Lavare il ligando TMR tre volte con PBS preriscaldato.
- Rimuovere il PBS e la pipetta 0,5 mL di mezzo L-15 di Leibovitz riscaldato contenente il supplemento B-27 al 2%, glutammina (1 mM), penicillina (100 U / mL) e streptomicina (100 μg / mL).
- Mantenere i neuroni per 1 ora a 37 °C.
- Accendere un microscopio a epifluorescenza e impostare l'incubatore da palcoscenico a 37 °C.
NOTA: Il presente protocollo utilizza un microscopio a epifluorescenza dotato di una fotocamera a semiconduttore complementare di ossido di metallo, una lente obiettivo ad immersione in olio 100x/1,40 NA, una lampada al mercurio e un software di acquisizione di immagini (vedi Tabella dei materiali). Per questa analisi possono essere utilizzati anche altri microscopi a epifluorescenza con specifiche equivalenti. - Posizionare i neuroni trattati con ligando TMR nel piatto con fondo di vetro sull'incubatrice riscaldata superiore dello stadio.
- Impostare i parametri di acquisizione dell'immagine come segue: tempo di esposizione, 500 ms per i canali fluorescenti Lifeact e HaloTag-actina; binning, 1 x 1 (0,065 μm × 0,065 μm per pixel); intervallo di tempo, 3 s; durata, 50 fotogrammi.
- Selezionare un cono di crescita che esprima fortemente Lifeact ed esprima debolmente HaloTag-actina.
NOTA: l'espressione di Lifeact deve essere abbastanza forte da visualizzare la morfologia del cono di crescita. L'espressione di HaloTag-actina deve essere debole e insufficiente per essere rilevata (Figura 2A). I livelli di espressioni proteiche fluorescenti possono essere regolati variando la quantità di DNA per la trasfezione. - Chiudere il campo del diaframma per illuminare un'area minima che include il cono di crescita (Figura 2B).
NOTA: L'illuminazione di un'area minima riduce il segnale di fondo e aumenta il rapporto segnale di fluorescenza / rumore (S/ N), consentendo il rilevamento di macchie di actina (Figura 2B). Per aumentare ulteriormente il rapporto S/N, si consiglia di illuminare il cono di crescita senza ridurre la luce con filtri a densità neutra. - Acquisire immagini time-lapse (File supplementare 2). Salva come immagine stack time-lapse multicanale (formato tiff).
6. Quantificazioni della velocità del flusso di F-actina e della velocità di polimerizzazione utilizzando un software di elaborazione e analisi delle immagini Fiji
NOTA: fare riferimento al file supplementare 3 per i dati di pratica per quantificare la velocità del flusso di F-actina e il tasso di polimerizzazione dell'actina.
- Apri l'immagine dello stack time-lapse multicanale su Fiji.
- Selezionare Analizza > Imposta scala e impostare la dimensione in pixel delle immagini.
- Trova una macchia di actina che scorre retrogradamente per un minimo di cinque intervalli di tempo all'interno di un fascio di F-actina in filopodia o lamellipodia.
- Selezionare Immagine > Trasforma > Ruota e regolare l'angolo delle immagini in modo che il fascio di F-actina punti verso l'alto (Figura 3A).
- Fare clic su Rettangolo sulla barra degli strumenti e disegnare una casella che include macchie di actina e la punta del fascio F-actina (Figura 3B, 1 e 2).
NOTA: I segnali delle macchie di HaloTag-actina sono deboli. Inoltre, i segnali Lifeact sulle punte distali del cono di crescita sono deboli e deboli (Figura 2B) perché le punte distali del cono di crescita sono sottili rispetto alla parte prossimale. I ricercatori dovrebbero migliorare i segnali per ottimizzare le quantificazioni della velocità del flusso di F-actina e della velocità di polimerizzazione (Figura 3B). Sebbene il segnale Lifeact al cono di crescita prossimale diventi saturo, ciò non disturberà la determinazione dell'estremità distale delle F-actine (Figura 3E, linea gialla). - Selezionare Immagine > Duplica e immettere i cinque intervalli di tempo (Figura 3B, 3-5).
- Selezionare Image > Stacks > Make Montage e immettere i parametri: Colonne, 5; File, 1; Fattore di scala, 1.
- Fare clic su OK. Il montaggio dell'immagine apparirà sullo schermo.
- Selezionare Immagine > Colore > Canali divisi. Questo separa i due canali fluorescenti.
- Quantificazione della velocità del flusso di F-actina.
- Selezionate Analizza > Imposta misure (Set Measurements) e Scegliete Area (Area ) e Rettangolo di delimitazione (Bounding Rectangle).
- Fare clic su Oval sulla barra degli strumenti e disegnare un cerchio su una macchia di actina (Figura 3C, 1 e 2).
- Selezionare Immagine > Sovrapposizione > Aggiungi selezione. Il cerchio è sovrapposto al montaggio dell'immagine (Figura 3C, 3 e 4).
NOTA: il colore della sovrapposizione può essere modificato in Modifica opzioni > > colori. - Ripetere la sovrapposizione dei cerchi per le macchie rimanenti (Figura 3C, 5).
NOTA: per determinare con precisione i centri di macchie di actina, gli investigatori dovrebbero sovrapporre cerchi sulle macchie. - Fare clic su Dritto sulla barra degli strumenti e disegnare una linea che collega i centri dei cerchi (Figura 3D, 1 e 2).
- Selezionare Analizza > misura. Il risultato apparirà sullo schermo (Figura 3D, 3).
NOTA: l'altezza visualizzata nel risultato indica la distanza di traslocazione della macchia di actina durante l'osservazione (Figura 3D, 3). - Calcola la velocità del flusso di F-actina dividendo la distanza di traslocazione per il tempo di osservazione.
- Quantificazione del tasso di polimerizzazione della F-actina.
- Disegnare una linea che colleghi le punte del fascio di F-actina (Figura 3E, 1).
- Selezionare Analizza > misura. Il risultato apparirà sullo schermo (Figura 3E, 2).
NOTA: l'altezza nel risultato indica la lunghezza di estensione del fascio di F-actina durante l'osservazione (Figura 3E, 2). - Calcola il tasso di estensione della F-actina dividendo la lunghezza dell'estensione per il tempo di osservazione.
- Calcolare il tasso di polimerizzazione della F-actina come somma della velocità del flusso di F-actina e della velocità di estensione (Figura 3F).
7. Preparazione di un gel PAA per microscopia a forza di trazione e colture neuronali
- Preparazione dei gel PAA necessari per la microscopia con forza di trazione (Figura 4)
- Pipetta 0,5 mL di NaOH (100 mM), disciolta in H2O distillato, su piatti con fondo di vetro da 27 mm di diametro, e incubare per 15 minuti a temperatura ambiente (RT).
- Aggiungere 50 μL di 3-aminopropiltrimetossisilano (APTMS) nella soluzione di NaOH sui piatti e mescolare le soluzioni mediante pipettaggio delicato e incubare per 15 minuti a RT.
- Lavare i piatti con fondo di vetro con H2O distillato e asciugare.
- Pipettare 0,5 mL di soluzione di glutaraldeide allo 0,5%, disciolta in PBS, sul piatto di fondo di vetro trattato con APTMS, e incubare per 30 minuti a RT.
- Lavare i piatti con fondo di vetro con H2O distillato e asciugare.
- Preparare una soluzione contenente soluzione distillata di H2O (412 μL), soluzione di acrilammide al 30% (p/v) (63 μL), soluzione di bis-acrilammide al 2,5% (p/v) (6 μL) e soluzione di microsfera modificata con carbossilato (perla fluorescente) (20 μL).
NOTA: Vortice la soluzione di perline fluorescenti per dissociare le perle aggregate prima di aggiungerle alla soluzione di acrilammide. - Degasare la soluzione in una camera a vuoto per 30 minuti a RT.
- A questa soluzione, aggiungere 1 μL di persolfato di ammonio al 10% (p/v) (APS), disciolto in H2O distillato, e 1 μL di N,N,N',N'-tetrametiletilendiammina (TEMED), e mescolare mediante pipettaggio delicato.
- Posizionare un coperchio di vetro (diametro 18 mm) sul cappuccio di un tubo di centrifuga da 15 ml.
- Aggiungere immediatamente 25 μL della soluzione al coperchio, quindi posizionare delicatamente un piatto con fondo di vetro invertito sul coperchio. Lasciare polimerizzare l'acrilammide per 1 ora a RT.
- Applicare H2O distillato sul coperchio e staccarlo dal gel usando un ago per siringa con una punta piegata.
- Rimuovere l'H2O e aggiungere sulfo-SANPAH (1 mM), sciolto in PBS, al gel.
- In condizioni sterili, esporre i gel alla luce ultravioletta per 5 minuti. Lavare il gel tre volte con PBS per 15 minuti per lavaggio.
- Determinazione della rigidità del gel con un metodo di indentazione della microsfera (Figura 5A)44.
- Trasferire una microsfera specificata per il diametro e la densità al gel e quindi posizionarla sullo stadio campione di un microscopio confocale a scansione laser.
- Concentrarsi sulla superficie del gel e registrare la posizione z (Figura 5B).
- Allo stesso modo, concentrati sul fondo della microsfera e registra la posizione z (Figura 5C).
- Calcolare la profondità di rientro determinata dalla differenza tra le posizioni z della superficie del gel e il fondo della microsfera.
- Calcola il modulo E di Young del gel tramite:
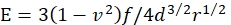
dove f è il peso corretto per galleggiamento della microsfera, d è la profondità di rientranza del gel, r è il raggio della microsfera e v è il rapporto di Poisson (il cui valore è 0,3, come determinato in precedenza46).
- Rivestire i gel con PDL e laminina come descritto nel paragrafo 2.
- Come descritto nei paragrafi 3 e 4, sezionare gli embrioni di topo E16, dissociare l'ippocampo e trasfettare con 5 μg di pEGFP-C1 per 1,0 x 106 cellule. Seme 2,0 x 105 cellule su gel rivestiti di PDL/laminina.
NOTA: per l'analisi della forza di trazione, l'imaging microscopico di cellule marcate fluorescenti è necessario per determinare l'area del cono di crescita.
8. Microscopia della forza di trazione ai coni di crescita neuronale
- Su DIV3, sostituire il terreno di coltura con 0,5 mL di terreno L-15 di Leibovitz riscaldato contenente il 2% di integratore B-27, glutammina (1 mM), penicillina (100 U / mL) e streptomicina (100 μg / mL). Mantenere i neuroni per 1 ora a 37 °C.
- Accendere un microscopio confocale a scansione laser e impostare l'incubatore stage-top a 37 °C.
NOTA: Il presente studio utilizza un microscopio confocale a scansione laser dotato di una lente ad immersione in acqua 63x/1,2 NA e di un software di acquisizione e analisi delle immagini (vedi Tabella dei materiali). - Posiziona i neuroni nel piatto con fondo di vetro sull'incubatrice preriscaldata.
- Impostare i parametri di acquisizione dell'immagine come segue: dimensione della scansione, 512 × 512 pixel; area di scansione, zoom 1,5-3x; velocità di scansione, ~ 1 s per fotogramma; lunghezza d'onda laser, 561 nm (la lunghezza d'onda di eccitazione per le perle fluorescenti) e 488 nm (la lunghezza d'onda di eccitazione per la proteina fluorescente verde potenziata (EGFP)); intervallo di tempo, 3 s; durata, 50 fotogrammi.
- Per visualizzare la morfologia del cono di crescita, selezionare un cono di crescita che esprima fortemente EGFP.
- Concentrati sulla superficie del gel e acquisisci immagini time-lapse (Figura 6A e File supplementare 4).
NOTA: per l'analisi della trazione, utilizzare due canali fluorescenti (per perline fluorescenti ed EGFP) e un canale a campo luminoso (si consiglia di acquisire immagini a contrasto di interferenza differenziale (DIC) o a contrasto di fase). - Utilizzare un software di elaborazione delle immagini (ad esempio, Fiji) per produrre stack di immagini RGB time-lapse a canale singolo e salvare queste immagini come file tiff.
NOTA: è fondamentale che gli investigatori salvino anche i dati grezzi. - Applicare 100 μL di 10% (p/v) di sodio dodecil solfato (SDS), disciolto in H2O distillato, sul piatto di fondo di vetro per rilassare il substrato di gel rilasciando neuroni dal substrato. Incubare il piatto per 5 minuti a 37°C per stabilizzare la temperatura.
NOTA: l'immagine delle perline in un substrato non teso viene utilizzata per fare riferimento alle posizioni originali delle perline durante l'analisi della forza di trazione (Figura 6C). L'applicazione della soluzione SDS altera le posizioni xyz a causa del cambiamento termico nell'incubatore e del rilassamento della deformazione indotta dalla cellula. Gli investigatori, quindi, devono correggere il piano focale e la posizione x-y. - Concentrati sulla superficie del gel e acquisisci un'immagine delle perline nel substrato non teso (Figura 6B).
- Produrre un'immagine RGB a canale singolo delle perline nel substrato non filtrato e salvare questa immagine come file tiff.
- Correzione della posizione x-y dell'immagine del tallone nel substrato non teso utilizzando Fiji
- Nelle Figi, apri l'immagine del tallone nel substrato non teso e l'immagine della pila time-lapse delle perline fluorescenti.
- Selezionate Immagine > Pile > Strumenti (> Concatena). Sullo schermo verrà visualizzata una finestra di dialogo (Figura 7A, 1).
- Selezionare l'immagine del tallone nel substrato non teso e l'immagine dello stack time-lapse delle perline fluorescenti rispettivamente in Image1 e Image2 (Figura 7A, 2).
- Fare clic su OK. L'immagine del tallone nel substrato non teso viene aggiunta all'immagine dello stack time-lapse (Figura 7A, 3).
- Utilizzando la barra di scorrimento (Figura 7B, 1, cornice rossa), visualizzare il secondo fotogramma dell'immagine dello stack (freccia rossa).
- Selezionare Plugin > StackReg (Figura 7B, 2). Sullo schermo apparirà una finestra di dialogo (Figura 7B, 3).
- Selezionate Corpo rigido (Rigid Body ) dall'elenco a discesa (Figura 7B, 3, cornice rossa) e fate clic su OK. Inizia quindi la correzione della posizione x-y.
- Utilizzate la barra di scorrimento per visualizzare il primo fotogramma dell'immagine dello stack con correzione posizione x-y.
- Selezionare Immagine > Duplica (Figura 7C, 1). Sullo schermo apparirà una finestra di dialogo (Figura 7B, 2).
- Immettere 1 nell'intervallo, deselezionare Stack duplicato (Figura 7C, 2) e fare clic su OK. L'immagine del tallone corretto per la posizione x-y nel substrato non teso apparirà sullo schermo (Figura 7C, 3). Salva questa immagine come file tiff.
- Quantificazione della forza di trazione
NOTA: Il metodo qui descritto per l'analisi della forza di trazione utilizza MATLAB e due toolbox MATLAB, 'Image Processing Toolbox' e 'Parallel Computing Toolbox'. Gli investigatori dovranno installarli prima dell'analisi. Il codice di analisi della forza di trazione è stato sviluppato sulla base della versione MATLAB 2018a. Pertanto, MATLAB versione 2018a (o successiva) deve essere utilizzato per l'analisi. Gli algoritmi utilizzati per l'analisi della forza di trazione sono stati descritti in precedenza22.- Scarica il codice di analisi della forza di trazione TFM2021 dal file supplementare 5. Apri TFM 2021 in MATLAB
- Apri main.m in TFM2021 ed eseguilo. Sullo schermo verrà visualizzata un'interfaccia utente grafica (GUI) (Figura 8A).
- Fare clic su Carica immagine del substrato non teso e selezionare l'immagine del tallone corretto per la posizione x-y nel substrato non teso.
- Fare clic su Carica immagini di perline fluorescenti e selezionare l'immagine dello stack time-lapse delle perline.
- Fare clic su Carica immagini a campo luminoso e selezionare l'immagine dello stack time-lapse di bright-field.
- Fare clic su Carica immagini GFP e selezionare l'immagine dello stack time-lapse di EGFP.
- Selezionare GFP dall'elenco a discesa nella GUI (Figura 8A, casella rossa).
- Fare clic su ROI per specificare la regione di interesse del rettangolo (ROI), incluso il cono di crescita, facendo clic su due punti sull'immagine della cella visualizzata nella GUI (Figura 8B).
- Fare clic sul pulsante Salva sulla GUI (Figura 8A, freccia rossa). Le immagini dello stack selezionate, insieme al ROI, verranno salvate in un file .mat (file in formato MATLAB).
- Fare clic su Rilevamento spot. Sullo schermo apparirà una finestra di dialogo.
- Immettere un valore (in genere 50-150) nella finestra di dialogo per determinare una soglia per il rilevamento di perline. Facendo clic su OK viene avviato il calcolo.
- Dopo aver terminato il calcolo, fare clic su Traccia traccia per ingrandire la regione selezionata al passaggio 8.12.8 e visualizzare le perline rilevate come punti bianchi (Figura 8C, 1).
NOTA: verificare se il rilevamento delle perline (punti bianchi) si sovrappone alle perline fluorescenti. Il rilevamento di perline può anche rilevare il rumore di fondo. Per ridurre questa interferenza esterna, modificare il valore di soglia in Rilevamento spot. Inoltre, selezionare manualmente i punti corretti al passaggio 8.12.13. - Fare clic su Seleziona perline e delimitare una regione poligonale che includa i punti corretti sotto il cono di crescita. Premere Invio sulla tastiera. I punti bianchi all'interno della regione poligonale cambieranno in un colore rosso (Figura 8C, 2).
- Fare clic su Stima forza sulla GUI. Quindi, i valori di input per i seguenti parametri: dimensione dei pixel, μm / pixel; modulo di Young, il valore calcolato al passo 7.2.5; Rapporto di Poisson, 0,3.
- Eseguire la forza stima per avviare il calcolo. Il software salverà automaticamente i risultati del calcolo in un file in formato foglio di calcolo.
NOTA: il foglio di calcolo mostra la componente x, la componente y e la magnitudine della forza in ogni intervallo di tempo (Figura 8D). Fare riferimento al file supplementare 6 per i dati di pratica per quantificare la forza di trazione.
Risultati
Imaging a singola macchia per quantificare la velocità di polimerizzazione dell'actina e l'efficienza di accoppiamento della frizione
Un'elevata espressione di Lifeact consente la visualizzazione di F-actina all'interno del cono di crescita; una bassa espressione di HaloTag-actina consente il monitoraggio del flusso retrogrado di F-actina (Figura 3, File supplementare 2). Il tracciamento delle macchie di actina consente di misurare la velocità del flusso di F-actina (Figura 3C,D). Poiché l'accoppiamento meccanico del flusso retrogrado di F-actina e del substrato adesivo riduce la velocità del flusso di F-actina, l'efficienza di accoppiamento della frizione può essere stimata dalla velocità. Inoltre, l'etichettatura di F-actina con Lifeact aiuta la visualizzazione dell'estensione di F-actina ed è utile per quantificare il tasso di polimerizzazione dell'actina (Figura 3E, F).
Analisi quantitativa della forza di trazione
La stretta aderenza alla metodologia qui presentata rivelerà i movimenti delle perle fluorescenti sotto il cono di crescita (Figura 6C, File supplementare 4). Il flusso retrogrado di F-actina sul substrato genera forza di trazione facendo sì che le perle fluorescenti si muovano all'indietro sotto il cono di crescita. Il codice di analisi della forza di trazione stima la forza di trazione dallo spostamento del tallone fluorescente ed esprime la forza di trazione calcolata come vettore di forza. La direzione e l'entità della forza di trazione sono determinate dalle componenti x e y del vettore di forza (Figura 8C,D). La casella rossa nella Figura 8D rappresenta le componenti x e y di un vettore di forza; La Figura 8C raffigura il cono di crescita corrispondente. In termini di coordinate x-y, il vettore punta a -93,8° contro l'asse x; questo orientamento è diretto verso la parte posteriore del cono di crescita (Figura 8C). L'entità della forza di trazione F è stata calcolata come segue:
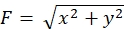
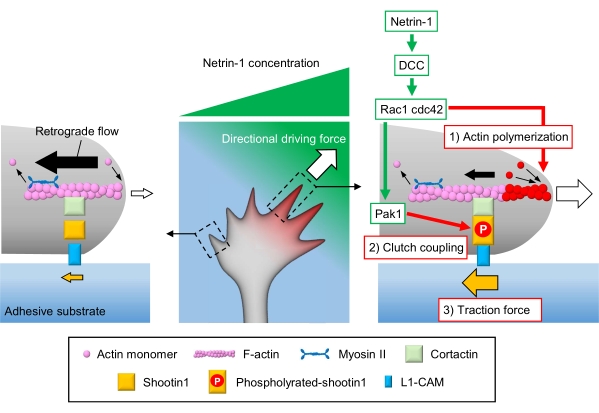
Figura 1: Un meccanismo a cono di crescita per la generazione di forza e la navigazione del cono di crescita. Un gradiente chemioattrattante netrin-1 induce la stimolazione asimmetrica del suo recettore DCC su un cono di crescita assonale. Questo attiva Rac1 e Cdc42 e la loro chinasi a valle Pak1. Rac1 e Cdc42 promuovono (1) la polimerizzazione dell'actina, mentre Pak1 fosforila shootin1, migliorando l'accoppiamento della frizione mediato da shootin1 (2). L'attivazione asimmetrica della dinamica dell'actina e l'accoppiamento della frizione all'interno del cono di crescita aumenta (3) la forza di trazione sul lato della sorgente netrin-1, generando così una forza motrice direzionale per l'attrazione del cono di crescita. I protocolli qui presentati consentono la quantificazione delle variabili chiave (1)-(3) per la navigazione del cono di crescita. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Immagini di fluorescenza di un cono di crescita neuronale sotto un diaframma completamente aperto e ristretto. Il cono di crescita esprime Lifeact e HaloTag-actina. (A) Un'elevata espressione di Lifeact consente la visualizzazione della morfologia del cono di crescita. D'altra parte, i livelli di espressione di HaloTag-actina sono molto bassi, con segnali deboli quando il diaframma è completamente aperto. (B) Quando il diaframma è opportunamente ristretto, i segnali di fondo diminuiscono e singole macchie di actina appaiono nel cono di crescita. Barre della scala: 5 μm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Passaggi per quantificare la velocità del flusso di F-actina e la velocità di polimerizzazione dell'actina utilizzando un software di elaborazione e analisi delle immagini Fiji. (A) Regolare l'angolo dell'immagine per l'analisi. (1) Trova e seleziona una macchia di actina (punta di freccia) che scorre in un fascio di F-actina. (2) Selezionare Immagine > Trasforma > Ruota. (3) Impostare l'angolo in modo che il fascio di F-actina sia diretto verso l'alto. (4) L'angolo dell'immagine verrà modificato. (B) Delimitare la regione, compresi la macchia di actina e il fascio di F-actina. (1) Fare clic su Rettangolo sulla barra degli strumenti. (2) Delineare una regione sull'immagine. La luminosità e il contrasto dell'immagine sono aumentati per consentire una chiara visualizzazione della macchia di actina e della punta del fascio di F-actina. (3) Selezionare Immagine > Duplica. (4) Inserire i cinque intervalli di tempo che mostrano il flusso di macchie di actina nel fascio di F-actina. (5) L'immagine dello stack selezionata apparirà sullo schermo. (C) Sovrapposizione di cerchi sulla macchia di actina. (1) Fare clic su Oval sulla barra degli strumenti. (2) Disegna un cerchio su una macchia di actina. (3) Selezionare Immagine > Sovrapposizione > Aggiungi selezione. (4) Il cerchio è sovrapposto. 5) Ripetere la sovrapposizione dei cerchi sulle macchie di actina rimanenti. (D) Misurare la distanza di traslocazione delle macchie di actina durante i cinque intervalli di tempo. (1) Fare clic su Dritto sulla barra degli strumenti. (2) Disegna una linea che collega i centri dei cerchi. (3) Selezionare Analizza > misura. Il risultato, indicato dal parametro Altezza (riquadro rosso), trasmette la distanza di traslocazione della macchia di actina. (E) Misurare la variazione della lunghezza della protrusione di F-actina durante i cinque intervalli di tempo. (1) Tracciare una linea che collega le punte della protrusione F-actina. (2) Selezionare Analizza > misura. Il risultato (riquadro rosso), Altezza, indica la lunghezza di estensione del fascio di F-actina. (F) Il tasso di polimerizzazione dell'actina è calcolato dalla somma della velocità del flusso di F-actina e della velocità di estensione. Vedere anche File supplementare 2. Il file supplementare 3 aiuta gli investigatori a praticare la metodologia sopra descritta. Fare clic qui per visualizzare una versione più grande di questa figura.
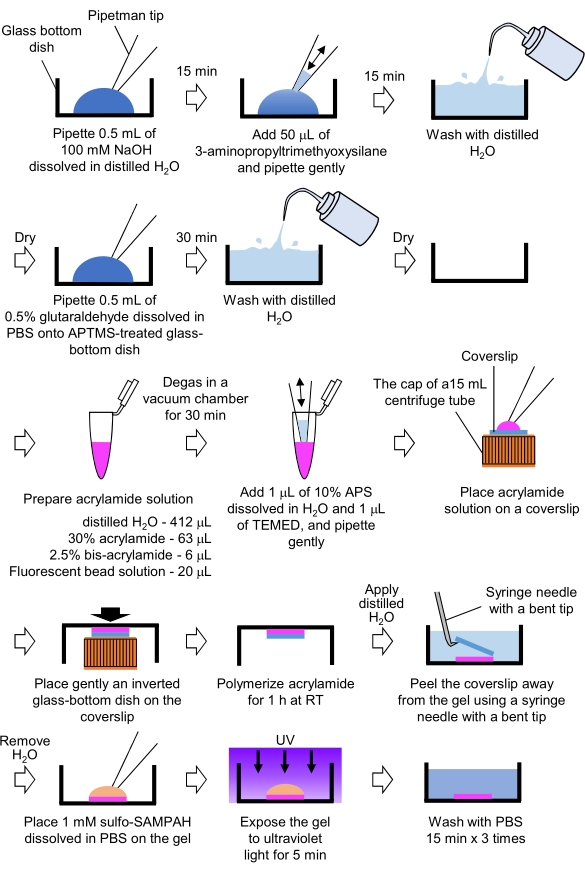
Figura 4: Passaggi per la preparazione del gel PAA. Si prega di consultare il passaggio 7.1 per una descrizione dettagliata. Fare clic qui per visualizzare una versione più grande di questa figura.
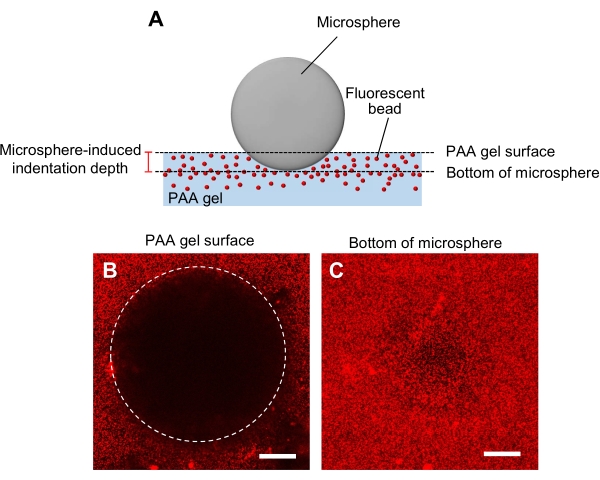
Figura 5: Determinazione della rigidità del gel PAA. (A) Un metodo di indentazione della microsfera. Quando una microsfera viene posizionata su un gel PAA incorporato in perline fluorescenti, il peso della microsfera provoca una rientranza nel gel. La profondità di indentazione viene calcolata sottraendo le posizioni z della superficie del gel PAA dal fondo della microsfera (B,C) Immagini di fluorescenza di un gel PAA rientrato da una microsfera e contenente perline fluorescenti. Un microscopio confocale a scansione laser è stato utilizzato per catturare immagini della superficie del gel (B) e del fondo della microsfera (C). I segnali provenienti dalle perle fluorescenti non sono visibili sulla superficie del gel nella regione rientrata (B, cerchio). Possono, tuttavia, essere osservati nella parte inferiore della microsfera. Barre di scala: 50 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
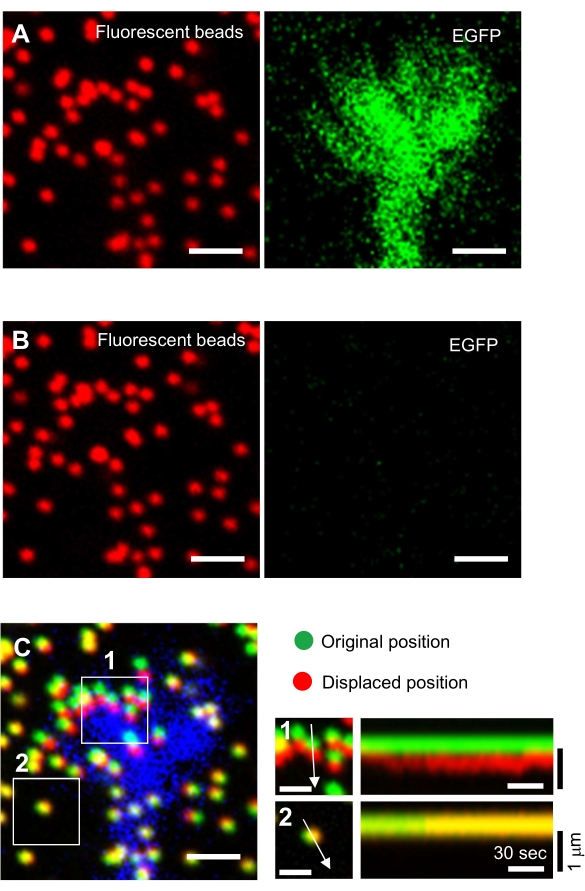
Figura 6: Mappatura della forza di un cono di crescita neuronale. (A-C) Immagini a fluorescenza di perline incorporate in un gel PAA e un cono di crescita neurale visualizzate con EGFP. A seguito dell'acquisizione di immagini time-lapse (A), il neurone è stato rilasciato dal substrato del gel applicando la soluzione SDS e un'immagine delle perline nel substrato non teso è stata catturata (B). (C) L'immagine delle perline nel substrato non teso mostra le perline nelle loro posizioni originali (verde) e spostate (rosso). Il segnale EGFP del cono di crescita è mostrato in blu. I kymografi (a destra) mostrano i movimenti delle perline a intervalli di 3 s per una durata di 147 s, indicati dalle frecce nelle aree in scatola 1 e 2. Il tallone nell'area 2 è un tallone di riferimento. Barre di scala: 2 μm per (A), (B) e (C, a sinistra) e 1 μm per (C, al centro). Vedere anche Il file supplementare 4. Fare clic qui per visualizzare una versione più grande di questa figura.
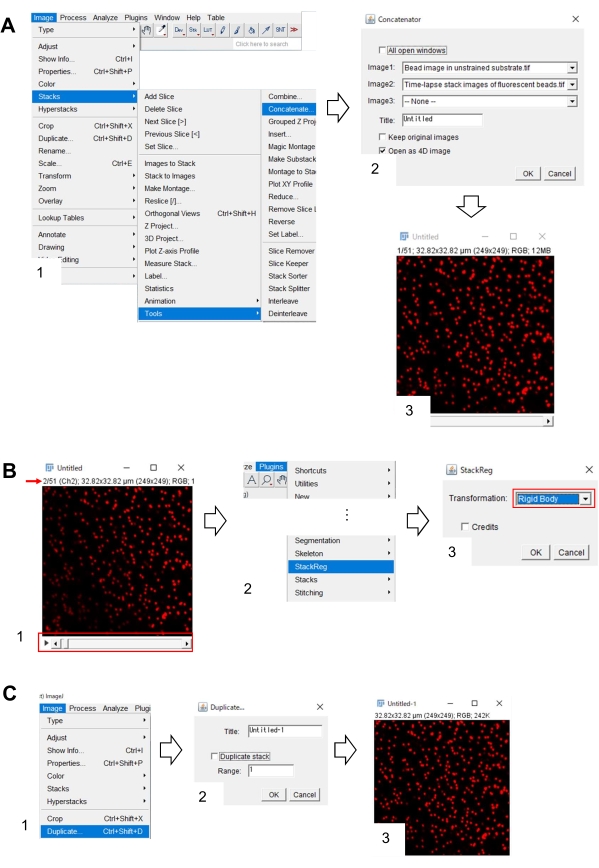
Figura 7: Passaggi per la correzione della posizione x-y dell'immagine del tallone in substrato non teso utilizzando Fiji. (A) Concatenare l'immagine del tallone in un substrato non teso con l'immagine dello stack time-lapse di perline fluorescenti. (1) Selezionare Image > Stacks > Tools > Concatenare. (2) Selezionare l'immagine delle perline in substrato non teso e l'immagine dello stack time-lapse delle perline fluorescenti rispettivamente in Image1 e Image2. Fare clic su OK. (3) L'immagine del tallone nel substrato non teso viene aggiunta all'immagine dello stack time-lapse. (B) Correggere la posizione x-y dell'immagine del tallone fluorescente. (1) Utilizzare la barra di scorrimento (cornice rossa) per selezionare il secondo fotogramma (freccia rossa) nella pila di immagini. (2) Selezionare Plugin > StackReg. (3) Selezionare Corpo rigido dall'elenco a discesa (cornice rossa) e fare clic su OK. Inizierà la correzione della posizione x-y. (C) Salvare l'immagine del tallone corretta per la posizione x-y. Dopo aver selezionato il primo fotogramma dell'immagine dello stack x-y corretta per la posizione, (1) selezionare Immagine > Duplica. (2) inserire 1 nell'intervallo e deselezionare lo stack duplicato. Quindi fare clic su OK. (3) l'immagine del tallone corretto per la posizione x-y nel substrato non teso apparirà sullo schermo. Salva questa immagine come file tiff. Il file supplementare 6 aiuta gli investigatori a praticare la metodologia sopra descritta. Fare clic qui per visualizzare una versione più grande di questa figura.
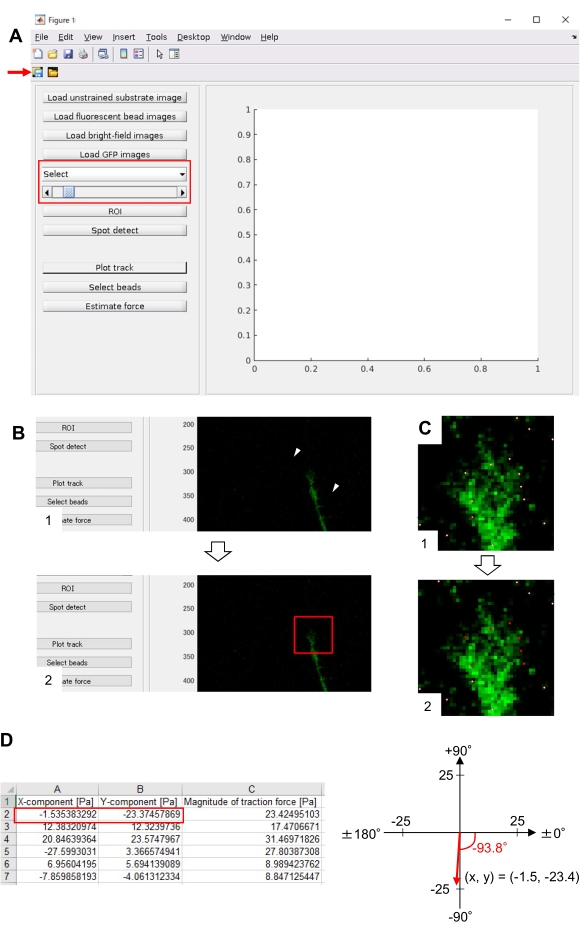
Figura 8: Analisi della forza di trazione sotto un cono di crescita neuronale utilizzando un codice di analisi della forza di trazione open source. (A) GUI per l'analisi della forza di trazione. Le immagini time-lapse selezionate nella GUI possono essere confermate dall'elenco a discesa e con il cursore indicato (casella rossa). (B) Selezionare una regione che includa il cono di crescita. (1) Fare clic su ROI nella GUI. Con il cursore del mouse, specificare due punti (punte di freccia) sull'immagine della cella. (2) Una casella rossa apparirà sull'immagine della cella. Due clic determinano le posizioni di due angoli. (C) Selezionare le perline rilevate (punti bianchi) sotto il cono di crescita. (1) Nella GUI, fare clic su Seleziona perline e delimitare una regione poligonale che include il cono di crescita facendo clic. Premere il tasto Invio . (2) I punti bianchi all'interno della regione poligonale cambieranno in un colore rosso. (D) Risultati calcolati della direzione e dell'entità della forza di trazione. La casella rossa nel foglio di calcolo rappresenta le componenti x e y del vettore di forza, stimate dall'analisi della forza di trazione. Sulla coordinata x-y nel pannello di destra, il vettore di forza generato dal cono di crescita punta a -93,8° contro l'asse x; questo orientamento è diretto verso la parte posteriore del cono di crescita in (C). Il file supplementare 6 aiuta gli investigatori a praticare la metodologia sopra descritta. Fare clic qui per visualizzare una versione più grande di questa figura.
File supplementare 1: Ricette di soluzioni e supporti utilizzati in questo studio. Vedere il testo per un utilizzo dettagliato. Fare clic qui per scaricare questo file.
File supplementare 2: Imaging a fluorescenza di Lifeact e imaging a macchie fluorescenti di HaloTag-actina in un cono di crescita nervosa. Lifeact (verde) e HaloTag-actina (magenta). Le immagini sono state acquisite ogni 3 s per una durata totale di 147 s. Barra di scala: 2 μm. Vedere anche la Figura 3. Fare clic qui per scaricare questo file.
File supplementare 3: Dati pratici per quantificare la velocità del flusso di F-actina e il tasso di polimerizzazione dell'actina. Un'immagine stack time-lapse multicanale di Lifeact (verde) e HaloTag-actina (magenta). Vedere anche la Figura 3. Fare clic qui per scaricare questo file.
File supplementare 4: un video di mappatura della forza che rileva la forza di trazione in un cono di crescita neuronale. Le posizioni originali (verde) e spostate (rosso) delle perline. Il segnale EGFP nel cono di crescita è mostrato in blu. Le immagini sono state acquisite ogni 3 s per 147 s. Barra di scala: 2 μm. Vedere anche la Figura 6. Fare clic qui per scaricare questo file.
File supplementare 5: Codice di analisi della forza di trazione. Vedere il passaggio 8.12 per un utilizzo dettagliato. Fare clic qui per scaricare questo file.
File supplementare 6: Dati pratici per quantificare la forza di trazione. Un'immagine RGB a canale singolo delle perline in substrato non teso e immagini RGB time-lapse a canale singolo di perline fluorescenti sotto un cono di crescita, EGFP e campo luminoso. Cfr . anche figure 7 e 8. Fare clic qui per scaricare questo file.
Discussione
I protocolli descritti in questo studio utilizzano materiali disponibili in commercio e apparecchiature di microscopia che si trovano abitualmente in tutti i laboratori, istituti e università. Pertanto, i ricercatori possono facilmente adottare l'attuale imaging a singola macchia e la microscopia della forza di trazione nei loro studi.
L'imaging speckle può analizzare la polimerizzazione dell'actina e l'accoppiamento della frizione. Inoltre, l'imaging speckle può monitorare il flusso retrogrado di molecole di frizione come shootin1 e cortactin, che interagiscono con il flusso retrogrado di F-actina. Utilizzando un microscopio TIRF, è possibile monitorare anche il flusso retrogrado della molecola di adesione cellulare L1-CAM23,41; L1-CAM subisce comportamenti di presa e slittamento che riflettono l'efficienza di accoppiamento della frizione23,41. Sebbene il presente studio utilizzi il sistema TMR-HaloTag per l'imaging speckle, altre proteine fluorescenti, come EGFP e la proteina fluorescente rossa monomerica, sono disponibili nell'analisi16,18,20,23,24,27,39. Gli elementi essenziali per visualizzare le macchie di actina sono un basso livello di espressione di actina fluorescente e l'illuminazione di un'area minima (Figura 2). In questo protocollo, i segnali Lifeact e HaloTag-actina vengono acquisiti in sequenza. Poiché il flusso retrogrado di actina è relativamente lento (4,5 ± 0,1 μm/min)24, l'analisi del flusso retrogrado di F-actina e la polimerizzazione dell'actina non sono influenzate dall'acquisizione sequenziale di immagini di diversi canali fluorescenti (intervallo ~ 1 s). Lifeact è un marcatore F-actina ampiamente utilizzato, ma può competere con le proteine leganti l'actina47. Di ulteriore importanza, Lifeact può alterare la dinamica dell'actina, influenzando così le strutture F-actina e la morfologia cellulare47,48,49.
La microscopia a forza di trazione può rilevare le forze per guidare l'avanzamento del cono di crescita. Montando i neuroni all'interno di una matrice extracellulare, i ricercatori possono anche analizzare le forze generate in un ambiente semi-3D11. L'imaging ad alto ingrandimento è importante per una quantificazione accurata della forza di trazione perché i coni di crescita generano forze di trazione deboli7. Sebbene per misurare la forza di trazione vengano utilizzati anche altri metodi con nanopilastri o biosensori sensibili allo stress50,51, il metodo a base di gel PAA è altamente adattabile e consente di regolare la rigidità del substrato variando le concentrazioni di acrilammide e bis-acrilammide41,44,52. In questo protocollo, il gel PAA viene preparato ad una concentrazione finale del 3,75% di acrilammide e dello 0,03% di bis-acrilammide; Il modulo di Young è ~270 Pa22 e questa rigidità rientra nell'intervallo del tessuto cerebrale (100-10.000 Pa)53,54,55. A causa dello spessore del gel PAA (~ 100 μm), questo metodo limita l'uso di lenti ad alto ingrandimento durante la microscopia. Per ottenere immagini ad alto ingrandimento, i ricercatori dovrebbero utilizzare la funzione zoom in un microscopio confocale a scansione laser.
In conclusione, l'attuale speckle imaging e la microscopia della forza di trazione consentono analisi quantitative degli eventi chiave nelle generazioni di forze. Queste informazioni saranno preziose per migliorare la comprensione dei meccanismi che sono alla base dell'avanzamento e della navigazione del cono di crescita.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questa ricerca è stata sostenuta in parte da AMED con il numero di sovvenzione 21gm0810011h0005 (N.I. e Y.S.), JSPS KAKENHI (JP19H03223, N.I.) e JSPS Grants-in-Aid for Early-Career Scientists (JP19K16258, T.M.), Osaka Medical Research Foundation for Incurable Diseases (T.M.) e NAIST Next Generation Interdisciplinary Research Project (Y.S.).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.5% trypan blue stain solution | Nacalai | 29853-34 | |
| 3-aminopropyltrimethyoxysilane | Sigma | 281778-100ML | |
| Acrylamide monomer | Nacalai | 00809-85 | |
| Ammonium persulphate | Cytiva | 17-1311-1 | |
| Axio Observer Z1 | Zeiss | 431007-9902-000 | Epi-fluorescence microscope (single speckle imaging) |
| B-27 supplement (50x) | Thermo Fisher Scientific | 17504-044 | |
| Bovine serum albmine | Sigma | A7906-10G | |
| C-Apochromat 63x/1.2 W Corr | Zeiss | 421787-9970-799 | Objective lens (traction force microscopy) |
| Coverslip (diameter 18 mm) | Matsunami | C018001 | |
| D-glucose | Nacalai | 16806-25 | |
| DNaseI | Sigma | DN25-100MG | |
| Fetal bovine serum | Thermo Fisher Scientific | 10270-106 | |
| Fiji | Open source software package | https://imagej.net/software/fiji/ | |
| FluoSpheres carboxylate-modified 0.2 mm, red (580/605), 2% solid | Thermo Fisher Scientific | F8810 | carboxylate-modified microspheres |
| Glass bottom dish (14 mm diameter) | Matsunami | D1130H | |
| Glass bottom dish (27 mm diameter) | Matsunami | D1140H | |
| Glutaraldehyde solution | Sigma | G5882-10X10ML | |
| HaloTag TMR ligand | Promega | G8251 | |
| HBO103 W/2 | Osram | 4050300382128 | Mercury lamp (single speckle imaging) |
| Image Processing Toolbox | MathWork | https://www.mathworks.com/products/image.html | |
| Laminin solution from mouse EHS tumor | Wako | 120-05751 | |
| Leibovitz’s L-15 medium | Thermo Fisher Scientific | 11415064 | |
| L-glutamine | Nacalai | 16919-42 | |
| LSM710 | Zeiss | N/A | Conforcal laser microscope (traction force microscopy) |
| MATLAB2018a | MathWork | https://www.mathworks.com/products/new_products/release2018a.html | |
| Mouse C57BL/6 | Japan SLC | N/A | |
| Mouse neuron nucleofector kit | Lonza | VPG-1001 | |
| N,N,N’,N’-tetramethylethylenediamine (TEMED) | Nacalai | 33401-72 | |
| N,N’-methylenebisacrylamide | Nacalai | 22402-02 | |
| Neurobasal medium | Thermo Fisher Scientific | 21103-049 | |
| Nucleofector I | Amaxa | AAD-1001 | Electroporation apparatus |
| ORCA Flash 4.0 V2 | Hamamatsu | C11440-22CU | CMOS camera (single speckle imaging) |
| Papain | Nacalai | 26036-34 | |
| Parallel Computing Toolbox | MathWork | https://www.mathworks.com/products/parallel-computing.html | |
| pEGFP-C1 | Clontech | 1528177 | |
| Penicillin-streptomycin (100x) | Nacalai Tesque | 26253-84 | |
| pFN21A-HaloTag-actin | (Minegishi et al., 2018) | N/A | |
| Phosphate buffered saline (PBS) pH 7.4 (10x) | Thermo Fisher Scientific | 70011-044 | |
| Plan-Apochromat 100x/1.4 Oil | Zeiss | 420790-9901-000 | Objective lens (single speckle imaging) |
| pmNeonGreen-N1-Lifeact | (Kastian et al., 2021) | N/A | |
| Poly-D-lysine hydrobromide | Sigma | P6407-5MG | |
| Slulfo-SAMPHA | Thermo Fisher Scientific | 22589 | |
| Sodium dodecyl sulfate | Nacalai | 08933-05 | |
| Sodium hydrate (NaOH) | Nacalai | 31511-05 | |
| Steel ball | Sako tekkou | N/A | Microshpere to determin PAA gel rigidity. 0.6 mm diameter, 7.87 g/cm3. |
| ZEN2009 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (traction force microscopy) |
| ZEN2012 | Zeiss | https://www.zeiss.com/microscopy/us/products/microscope-software/zen.html#introduction | Image acquisition software (single speckle imaging) |
Riferimenti
- TessierLavigne, M., Goodman, C. S. The molecular biology of axon guidance. Science. 274, 1123-1133 (1996).
- Marin, O., Valiente, M., Ge, X., Tsai, L. H. Guiding neuronal cell migrations. Cold Spring Harbor Perspectives in Biology. 2, 001834 (2010).
- Vitriol, E. A., Zheng, J. Q. Growth cone travel in space and time: the cellular ensemble of cytoskeleton, adhesion, and membrane. Neuron. 73 (6), 1068-1081 (2012).
- Cooper, J. A. Cell biology in neuroscience: mechanisms of cell migration in the nervous system. Journal of Cell Biology. 202, 725-734 (2013).
- Dupraz, S., et al. RhoA controls axon extension independent of specification in the developing brain. Current Biology. 29, 3874-3886 (2019).
- Suter, D. M., Miller, K. E. The emerging role of forces in axonal elongation. Progress in Neurobiology. 94, 91-101 (2011).
- Franze, K. Integrating chemistry and mechanics: the forces driving axon growth. Annual Review of Cell and Developmental Biology. 36, 61-83 (2020).
- Mitchison, T., Kirschner, M. Cytoskeletal dynamics and nerve growth. Neuron. 1, 761-772 (1988).
- Suter, D. M., Forscher, P. Substrate-cytoskeletal coupling as a mechanism for the regulation of growth cone motility and guidance. Journal of Neurobiology. 44, 97-113 (2000).
- Lowery, L. A., Van Vactor, D. The trip of the tip: understanding the growth cone machinery. Nature Reviews Molecular Cell Biology. 10, 332-343 (2009).
- Minegishi, T., et al. Shootin1b mediates a mechanical clutch to produce force for neuronal migration. Cell Reports. 25, 624-639 (2018).
- Minegishi, T., Inagaki, N. Forces to drive neuronal migration steps. Frontiers in Cell and Developmental Biology. 8, 863 (2020).
- Forscher, P., Smith, S. J. Actions of cytochalasins on the organization of actin filaments and microtubules in a neuronal growth cone. Journal of Cell Biology. 107, 1505-1516 (1988).
- Suter, D. M., Forscher, P. An emerging link between cytoskeletal dynamics and cell adhesion molecules in growth cone guidance. Current Opinion in Neurobiology. 8, 106-116 (1998).
- Pollard, T. D., Borisy, G. G. Cellular motility driven by assembly and disassembly of actin filaments. Cell. 112, 453-465 (2003).
- Waterman-Storer, C. M., Desai, A., Bulinski, J. C., Salmon, E. D. Fluorescent speckle microscopy, a method to visualize the dynamics of protein assemblies in living cells. Current Biology. 8, 1227-1230 (1998).
- Katoh, K., Hammar, K., Smith, P. J., Oldenbourg, R. Birefringence imaging directly reveals architectural dynamics of filamentous actin in living growth cones. Molecular Biology of the Cell. 10, 197-210 (1999).
- Watanabe, N., Mitchison, T. J. Single-molecule speckle analysis of actin filament turnover in lamellipodia. Science. 295, 1083-1086 (2002).
- Medeiros, N. A., Burnette, D. T., Forscher, P. Myosin II functions in actin-bundle turnover in neuronal growth cones. Nature Cell Biology. 8, 215-226 (2006).
- Shimada, T., et al. Shootin1 interacts with actin retrograde flow and L1-CAM to promote axon outgrowth. Journal of Cell Biology. 181, 817-829 (2008).
- He, M., Zhang, Z. H., Guan, C. B., Xia, D., Yuan, X. B. Leading tip drives soma translocation via forward F-actin flow during neuronal migration. Jounal of Neuroscience. 30, 10885-10898 (2010).
- Toriyama, M., Kozawa, S., Sakumura, Y., Inagaki, N. Conversion of a signal into forces for axon outgrowth through Pak1-mediated shootin1 phosphorylation. Current Biology. 23, 529-534 (2013).
- Abe, K., et al. Grip and slip of L1-CAM on adhesive substrates direct growth cone haptotaxis. Proceedings of the National Academy of Sciences of the United States of America. 115, 2764-2769 (2018).
- Baba, K., et al. Gradient-reading and mechano-effector machinery for netrin-1-induced axon guidance. Elife. 7, 34593 (2018).
- Nichol, R. I., Hagen, K. M., Lumbard, D. C., Dent, E. W., Gomez, T. M. Guidance of axons by local coupling of retrograde flow to point contact adhesions. Journal of Neuroscience. 36, 2267-2282 (2016).
- Shekarabi, M., et al. Deleted in colorectal cancer binding netrin-1 mediates cell substrate adhesion and recruits Cdc42, Rac1, Pak1, and N-WASP into an intracellular signaling complex that promotes growth cone expansion. Journal of Neuroscience. 25, 3132-3141 (2005).
- Kubo, Y., et al. Shootin1-cortactin interaction mediates signal-force transduction for axon outgrowth. Journal of Cell Biology. 210, 663-676 (2015).
- Huber, A. B., Kolodkin, A. L., Ginty, D. D., Cloutier, J. F. Signaling at the growth cone: ligand-receptor complexes and the control of axon growth and guidance. Annual Review of Neuroscience. 26, 509-563 (2003).
- Kolodkin, A. L., Tessier-Lavigne, M. Mechanisms and molecules of neuronal wiring: a primer. Cold Spring Harbor Perspectives in Biology. 3, 001727 (2011).
- Ayala, R., Shu, T., Tsai, L. H. Trekking across the brain: the journey of neuronal migration. Cell. 128, 29-43 (2007).
- Tatavarty, V., Kim, E. J., Rodionov, V., Yu, J. Investigating sub-spine actin dynamics in rat hippocampal neurons with super-resolution optical imaging. PLoS One. 4, 7724 (2009).
- Frost, N. A., Shroff, H., Kong, H., Betzig, E., Blanpied, T. A. Single-molecule discrimination of discrete perisynaptic and distributed sites of actin filament assembly within dendritic spines. Neuron. 67, 86-99 (2010).
- Chazeau, A., et al. Nanoscale segregation of actin nucleation and elongation factors determines dendritic spine protrusion. EMBO Journal. 33, 2745-2764 (2014).
- Garcia, M., et al. Two-tiered coupling between flowing actin and immobilized N-cadherin/catenin complexes in neuronal growth cones. Proceedings of the National Academy of Sciences of the United States of America. 112, 6997-7002 (2015).
- Swaminathan, V., et al. Actin retrograde flow actively aligns and orients ligand-engaged integrins in focal adhesions. Proceedings of the National Academy of Sciences of the United States of America. 114, 10648-10653 (2017).
- Tsai, T. Y., et al. Efficient front-rear coupling in neutrophil chemotaxis by dynamic myosin II localization. Developmental Cell. 49, 189-205 (2019).
- Zhang, X. F., et al. Regulation of axon growth by myosin II-dependent mechanocatalysis of cofilin activity. Journal of Cell Biology. 218 (7), 2329-2349 (2019).
- Reversat, A., et al. Cellular locomotion using environmental topography. Nature. 582, 582-585 (2020).
- Katsuno, H., et al. Actin migration driven by directional assembly and disassembly of membrane-anchored actin filaments. Cell Reports. 12, 648-660 (2015).
- Urasaki, A., et al. Shootins mediate collective cell migration and organogenesis of the zebrafish posterior lateral line system. Scientific Reports. 9, 12156 (2019).
- Abe, K., et al. Mechanosensitive axon outgrowth mediated by L1-laminin clutch interface. Biophysical Journal. 120, 3566-3576 (2021).
- Kastian, R. F., et al. Shootin1a-mediated actin-adhesion coupling generates force to trigger structural plasticity of dendritic spines. Cell Reports. 35, 109130 (2021).
- Riedl, J., et al. Lifeact: a versatile marker to visualize F-actin. Nature Methods. 5, 605-607 (2008).
- Chan, C. E., Odde, D. J. Traction dynamics of filopodia on compliant substrates. Science. 322, 1687-1691 (2008).
- Wang, Y. L., Pelham, R. J. Preparation of a flexible, porous polyacrylamide substrate for mechanical studies of cultured cells. Methods in Enzymology. 298, 489-496 (1998).
- Li, Y., Hu, Z., Li, C. New method for measuring poisson's ratio in polymer gels. Journal of Applied Polymer Science. 50, 1107-1111 (1993).
- Belyy, A., Merino, F., Sitsel, O., Raunser, S. Structure of the Lifeact-F-actin complex. PLoS Biology. 18, 3000925 (2020).
- Flores, L. R., Keeling, M. C., Zhang, X., Sliogeryte, K., Gavara, N. Lifeact-GFP alters F-actin organization, cellular morphology and biophysical behaviour. Scientific Reports. 9, 3241 (2019).
- Kumari, A., Kesarwani, S., Javoor, M. G., Vinothkumar, K. R., Sirajuddin, M. Structural insights into actin filament recognition by commonly used cellular actin markers. EMBO Journal. 39, 104006 (2020).
- du Roure, O., et al. Force mapping in epithelial cell migration. Proceedings of the National Academy of Sciences of the United States of America. 102, 2390-2395 (2005).
- Grashoff, C., et al. Measuring mechanical tension across vinculin reveals regulation of focal adhesion dynamics. Nature. 466, 263-266 (2010).
- Koch, D., Rosoff, W. J., Jiang, J., Geller, H. M., Urbach, J. S. Strength in the periphery: growth cone biomechanics and substrate rigidity response in peripheral and central nervous system neurons. Biophysical Journal. 102 (3), 452-460 (2012).
- Barnes, J. M., Przybyla, L., Weaver, V. M. Tissue mechanics regulate brain development, homeostasis and disease. Journal of Cell Science. 130, 71-82 (2017).
- Moore, S. W., Roca-Cusachs, P., Sheetz, M. P. Stretchy proteins on stretchy substrates: the important elements of integrin-mediated rigidity sensing. Developmental Cell. 19, 194-206 (2010).
- Tyler, W. J. The mechanobiology of brain function. Nature Reviews Neuroscience. 13, 867-878 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon