Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Evaluación de la transcitosis intestinal de aislados de bacteriemia neonatal por Escherichia coli
En este artículo
Resumen
Escherichia coli causa sepsis en neonatos que ingieren la bacteria alrededor del momento del nacimiento. El proceso involucrado en la capacidad de E. coli para viajar desde el tracto entérico al torrente sanguíneo es poco conocido. Este modelo in vitro evalúa la capacidad de las cepas de E. coli para viajar a través de las células epiteliales intestinales.
Resumen
Los recién nacidos ingieren cepas maternas de E. coli que colonizan su tracto intestinal alrededor del momento del parto. Las cepas de E. coli con la capacidad de translocarse a través del intestino invaden el torrente sanguíneo del recién nacido, causando bacteriemia potencialmente mortal. La metodología presentada aquí utiliza células epiteliales intestinales polarizadas cultivadas en insertos semipermeables para evaluar la transcitosis de aislados de bacteriemia neonatal por E. coli in vitro. Este método utiliza la línea celular intestinal T84 establecida que tiene la capacidad de crecer para confluir y formar uniones estrechas y desmosomas. Después de alcanzar la confluencia, las monocapas T84 maduras desarrollan resistencia transepitelial (TEER), que se puede cuantificar utilizando un voltímetro. Los valores de TEER están inversamente correlacionados con la permeabilidad paracelular de los componentes extracelulares, incluidas las bacterias, a través de la monocapa intestinal. El paso transcelular de bacterias (transcitosis), por otro lado, no necesariamente altera las mediciones TEER. En este modelo, el paso bacteriano a través de la monocapa intestinal se cuantifica hasta 6 h después de la infección, y se realizan mediciones repetidas de TEER para controlar la permeabilidad paracelular. Además, este método facilita el uso de técnicas como la inmunotinción para estudiar los cambios estructurales en las uniones estrechas y otras proteínas de adhesión de célula a célula durante la transcitosis bacteriana a través del epitelio polarizado. El uso de este modelo contribuye a la caracterización de los mecanismos por los cuales E. coli neonatal se transcita a través del epitelio intestinal para producir bacteriemia.
Introducción
Escherichia coli es la causa más común de sepsis de inicio temprano en recién nacidos 1,2,3. La tasa de mortalidad de la bacteriemia neonatal por E. coli puede alcanzar el 40%, y la meningitis es una posible complicación asociada a discapacidades graves del desarrollo neurológico2. La ingestión de cepas maternas de E. coli por el recién nacido puede producir bacteriemia neonatal; Este proceso ha sido replicado en modelos animales 2,4. Una vez ingeridas, las bacterias patógenas viajan desde la luz intestinal neonatal a través de la barrera intestinal y entran en el torrente sanguíneo, causando septicemia. Las cepas neonatales invasivas de E. coli que producen bacteriemia varían en su capacidad para invadir las células epitelialesintestinales 1,5. Sin embargo, su capacidad para transcitar el epitelio intestinal después de la invasión no ha sido completamente caracterizada.
Este modelo de transcitosis intestinal es un método in vitro útil para emular el paso bacteriano a través del epitelio intestinal. El objetivo general de los métodos presentados en este manuscrito es comparar la capacidad de los aislados neonatales de E. coli para transcitar el epitelio intestinal. El modelo descrito aquí utiliza células T84, que son células de adenocarcinoma intestinal humano inmortalizadas 6,7. Las células T84 se cultivan para confluir en una membrana semipermeable con dos compartimentos separados. La justificación para utilizar esta técnica es que, como sucede in vivo, estas células intestinales se polarizan y desarrollan uniones estrechas maduras 6,8. El lado en contacto con la membrana se convierte en el lado basal. El lado opuesto de las células se convierte en el lado apical, parecido a la luz intestinal donde los patógenos ingeridos se adhieren e invaden. La membrana transwell es permeable a las bacterias, pero las células intestinales polarizadas forman uniones estrechas, que perjudican el movimiento paracelular bacteriano9. Por lo tanto, este método proporciona la ventaja de un entorno in vitro controlado que utiliza una línea celular humana para estudiar el proceso de transcitosis bacteriana, incluida la ruta transcelular. Mientras que existen otros métodos para investigar la transcitosis de bacterias a través del epitelio intestinal, el método transwell presentado aquí proporciona una mayor facilidad y accesibilidad. Se dispone de técnicas alternativas, como las que utilizan muestras ex vivo instaladas en sistemas de cámaras Ussing. Sin embargo, utilizan muestras de tejido que pueden no ser fácilmente accesibles, particularmente si la investigación tiene la intención de estudiar la fisiología humana10. Los organoides intestinales representan otro ejemplo de una alternativa in vitro para estudiar las interacciones huésped-bacteria11. Mientras que las monocapas organoides también pueden ser utilizadas en el sistema transwell para estudiar la transcitosis bacteriana, requieren el aislamiento y crecimiento de células madre y el uso de factores de crecimiento específicos para inducir la diferenciación12. Por lo tanto, su uso requiere más tiempo y se asocia con mayores costos en comparación con el método transwell descrito en este manuscrito.
La evaluación del paso bacteriano a través del epitelio intestinal utilizando este sistema de transwell in vitro se ha realizado con éxito para varios patógenos. Estos estudios han demostrado la utilidad del sistema transwell utilizando células T84 para caracterizar la transcitosis de bacterias a través del epitelio intestinal polarizado13,14,15. Sin embargo, la aplicación de este método transwell para comparar la capacidad de transcitosis de cepas neonatales de E. coli productoras de bacteriemia no se ha descrito en detalle. Este manuscrito proporciona a otros investigadores un protocolo estándar de transwell que es confiable y fácil de usar y no requiere recursos demasiado caros.
Para comparar la capacidad de las cepas neonatales invasivas de E. coli para transcitar el epitelio intestinal, el lado apical de la monocapa epitelial intestinal puede infectarse con un número conocido de células bacterianas. Después de la incubación, se puede recolectar el medio en el lado basal del epitelio y cuantificar las bacterias para determinar la cantidad de transcitosis bacteriana a lo largo del tiempo. En este manuscrito, los métodos presentados se utilizan para estudiar la capacidad de transcitosis de cepas clínicas neonatales de E. coli recuperadas de recién nacidos hospitalizados con bacteriemia. Los criterios de inclusión para la selección de estos aislados clínicos neonatales para estudios de transcitosis han sido publicados previamente 1,2,16. Cuando este método se realiza utilizando diferentes cepas de E. coli, se pueden comparar sus capacidades de transcitosis. A través de este proceso, el modelo de transcitosis intestinal proporciona datos valiosos para caracterizar los factores de virulencia de E. coli que contribuyen al proceso de múltiples pasos que culmina en el desarrollo de bacteriemia neonatal.
Protocolo
NOTA: Realice todas las manipulaciones de las células, bacterias, placas y reactivos T84 en un gabinete de seguridad de nivel de bioseguridad 2 (BSL-2) para evitar la contaminación. Use áreas e incubadoras separadas para todo el trabajo que involucre células T84 estériles, células T84 infectadas y E. coli. Los aislados clínicos de E. coli probados con los métodos aquí descritos fueron obtenidos siguiendo las directrices del Comité de Revisión Institucional de nuestra institución 1,16.
1. Preparación de insertos de transcitosis con células T84 (aproximadamente 1-2 semanas antes del experimento)
- Grow American Type Culture Collection (ATCC) células T84 en medio de cultivo de tejidos (MTC + antibióticos) que consiste en el medio Águila modificada de Dulbecco: mezcla de nutrientes F-12 de Ham (1: 1, concentración final: 50% cada uno), suero bovino fetal al 5% y mezcla antibiótica dual de penicilina / estreptomicina al 1% (100 U / ml). Incubar las células a 37 °C con 5% deCO2.
NOTA: Se utiliza una variación de esta formulación de medio sin penicilina/estreptomicina (MTC sin antibióticos) para los pasos posteriores (sección 2 y posteriores) del procedimiento. Asegúrese de que se utiliza la formulación correcta para cada paso. - Trabajando dentro de un gabinete de bioseguridad (BSC), siembre las células T84 en insertos de transwell de cultivo celular de membrana de tereftalato de polietileno con poros de 3 μm hechos para placas de 24 pocillos. Incluya réplicas de transwell para cada condición experimental deseada más controles no infectados para monitorear la posible contaminación.
- En una placa de 24 pocillos diseñada para contener insertos de transwell, llene el número deseado de pocillos colectores con 1 ml de MTC + antibióticos.
- En cada pocillo, coloque un inserto transwell.
- Sembrar estos insertos con 1 x 105 células T84 suspendidas en 500 μL de MTC + antibióticos. Cuantificar el número de células utilizando un hemocitómetro con tinción de azul de tripano o un contador celular automatizado17.
- Incubar las placas transwell que contienen insertos con semillas en las mismas condiciones en que se cultivaron las células.
- Verifique con microscopía óptica que las monocapas hayan comenzado a confluente después de sembrar el inserto, aproximadamente 48 h después de la siembra.
- Cada 2 días después de la siembra, mida y registre la resistencia eléctrica transepitelial (TEER) utilizando un medidor epitelial volt/ohm (EVOM) para evaluar la madurez de la monocapa. Una vez que los insertos alcanzan un TEER de al menos 1.000 Ωcm2, se consideran listos para el ensayo18.
NOTA: Los insertos suelen tardar de 7 a 10 días en alcanzar este TEER después de la siembra. Las células T84 mostrarán una confluencia del 100% bajo microscopía óptica una vez que se alcance esta resistencia.- Guarde la sonda EVOM con los electrodos sumergidos en 0,15 M KCl cuando no esté en uso.
- Antes de medir el TEER, descontamine la sonda del electrodo sumergiéndola en 5 ml de etanol al 70% en un tubo cónico de 50 ml durante 10-15 min. Retire la sonda, sacuda el exceso de etanol y deje que se seque al aire dentro del BSC durante 10 minutos. Retener el tubo de etanol.
- Pruebe el EVOM y la sonda colocando la sonda seca descontaminada en un pocillo estéril que contenga 1 ml de MTC + antibióticos con un inserto estéril en el interior que contenga 500 μL de MTC + antibióticos. Asegúrese de que la lectura de EVOM sea de <200 Ω. Registre este valor en blanco para utilizarlo en los cálculos de resistencia posteriores descritos en el paso 1.3.6.
- Retire la sonda del tubo de MTC + antibióticos. Conserve este tubo para el almacenamiento de la sonda durante todo el experimento.
- Baje suavemente la sonda en el primer inserto con el electrodo largo en el pozo colector y el electrodo corto dentro del inserto. Permita que el electrodo largo toque el fondo del pocillo colector, pero no empuje hacia abajo, ya que esto puede interrumpir la monocapa epitelial.
- Repita este proceso para medir y registrar la resistencia en ohmios (Ω) para cada inserto. Cuando termine, descontamine la sonda en el etanol sumergiéndola durante otros 10-15 minutos. Luego, mueva la sonda descontaminada de nuevo a la solución KCl para su almacenamiento. Restar la resistencia en blanco obtenida en el paso 1.3.3 de cada valor obtenido de cada inserto que contenga celdas T84. Multiplique la resistencia resultante (Ω) para cada inserto por el área de la parte inferior de cada inserto (cm 2) para obtener la medida TEER final (Ω·cm2).
- Una vez que el TEER alcanza al menos 1.000 Ω · cm2, la monocapa epitelial está madura y lista para los ensayos de infección.
- A medida que el TEER madura, proporcione a las células un medio fresco cada 1-2 días.
- En una nueva placa de 24 pocillos, agregue 1 ml de MTC + antibióticos a un pocillo por cada inserto sembrado que se esté preparando.
- Usando fórceps estériles, transfiera cuidadosamente los insertos a los pozos recién reabastecidos.
- Reemplace el soporte en las inserciones.
- Retire los medios viejos de los insertos inclinando la placa y utilizando un aspirador de vacío interno para extraer suavemente el medio con una punta de pipeta a lo largo del costado del inserto. El aspirador permite la regulación de la succión de bajo nivel para evitar la interrupción de las células. No permita que la punta de la pipeta toque la parte inferior del inserto, ya que esto interrumpirá la monocapa epitelial en desarrollo.
- Agregue 500 μL de MTC + antibióticos a los insertos. Visualice la monocapa con microscopía óptica para verificar que permanezca intacta.
- Cada 1-2 días, mida el TEER en cada inserto como se describe anteriormente en los pasos 1.3.2-1.3.6.
2. Preparación de las células T84 1 día antes del experimento usando MTC sin antibióticos
- Mida y registre el TEER el día antes del experimento.
- Reemplace la MTC de la misma manera que durante la preparación y el mantenimiento de la celda anterior. Sin embargo, la MTC sin antibióticos se usa en su lugar en preparación para la infección (1 ml en el pocillo de la placa y 500 μL en el inserto).
3. Cultivos de E. coli (iniciados 1 día antes del experimento)
PRECAUCIÓN: Use precauciones de nivel de bioseguridad 2 (BSL-2) cuando trabaje con cepas clínicas patógenas de E. coli.
- Tome un tubo cónico marcado de 15 ml con 5 ml de caldo de lisogenia estéril (LB) y use un asa estéril para inocular el caldo con una colonia de una cepa bacteriana (E. coli). Repita este proceso, creando un tubo de cultivo nocturno para cada cepa a probar.
- Incubar el cultivo nocturno, con las tapas de los tubos aflojadas, en un agitador de incubadora (250 rpm, 37 °C).
4. Preparación del inóculo de E. coli , células epiteliales y materiales (en la mañana del experimento)
NOTA: Use MTC sin antibióticos calentados hasta 37 °C a partir de este momento.
- Añadir 250 μL de cada cultivo de LB durante la noche a 25 ml de MTC sin antibióticos en un tubo cónico de 50 ml (uno por cepa individual). Mantenga las tapas de los tubos aflojadas. Coloque estos nuevos tubos de cultivo en el agitador con la misma configuración (250 rpm, 37 °C) durante exactamente 2 h. Realice los subpasos restantes mientras espera.
- Mida el TEER en cada inserto, como se describe en los pasos 1.3.2-1.3.6. Regístrelos como TEER en el momento (t) = 0 h.
- Mover los insertos a pocillos de una placa nueva y cambiar el medio utilizando la técnica descrita en el paso 1.4.3. Sin embargo, esta vez, llene los nuevos pocillos colectores con 500 μL de MTC sin antibióticos, y llene los insertos con 400 μL de MTC sin antibióticos. Mantenga los insertos dentro de la incubadora de cultivo de tejidos hasta el momento de la infección.
- Coloque un número suficiente de placas cuadradas de agar LB para calentar a temperatura ambiente (RT) para su posterior recubrimiento y cuantificación bacteriana.
5. Inoculación de las células (inicio del experimento)
- Después de exactamente 2 h, retire los cultivos bacterianos matutinos del agitador y centrifugarlos durante 10 min (1,900 x g, 4 °C).
NOTA: Para todos los pasos siguientes, mantenga todas las suspensiones bacterianas en hielo para minimizar el crecimiento. - Resuspender el pellet de bacterias en TCM sin antibióticos. Use un espectrofotómetro para ajustar la densidad óptica (OD) a 0.7-0.9, y diluya aún más con TCM sin antibióticos a una concentración de 1 x 106 unidades formadoras de colonias (UFC) / ml (aproximadamente 1:100 dilución). Use esta suspensión bacteriana para infectar cada inserto con 1 x 105 UFC por volumen de 100 μL.
- Etiquetar la placa transwell e infectar cada inserto con 100 μL del inóculo ajustado por OD (total de 1 x 105 UFC por inserto). El ensayo ya ha comenzado. Anote la hora y regístrela como t = 0 h.
- Colocar en placa la suspensión bacteriana del inóculo para cuantificar la UFC/ml utilizando el método de dilución de la vía, colocando alícuotas de 10 μL en placas cuadradas de agar LB19.
6. Cuantificación de la transcitosis
- Cada 30 minutos después de la inoculación, llene los nuevos pocillos con 500 μL de MTC sin antibióticos. Transfiera los insertos a estos nuevos pocillos utilizando un conjunto diferente de pinzas estériles para cada cepa bacteriana diferente.
- Recoja los medios del pozo recolector usado para cada inserto en tubos etiquetados separados. Coloque estos tubos sobre hielo. Devuelva las placas transwell a la incubadora entre puntos de tiempo.
- Para cada inserto, combine los medios recolectados de t = 0.5 h, t = 1 h, t = 1.5 h y t = 2 h, y vórtice brevemente. Coloque los medios recolectados en placas de agar LB utilizando el método de dilución de pista para cuantificar la cantidad de bacterias transcitadas en las primeras 2 h del experimento.
NOTA: Recuperar las bacterias cada 30 minutos y mantenerlas en hielo asegura que el crecimiento bacteriano en los pozos colectores se minimice y las mediciones se realicen en bacterias predominantemente transcitadas. - Coloque las placas de agar LB de dilución de vía etiquetadas en la incubadora bacteriana a 37 °C sinCO2 suplementario y devuelva la placa de pocillo T84 a la incubadora de cultivo de tejidos.
- En t = 4 h, repita el paso 6.3 combinando los medios recolectados de t = 2.5 h, t = 3 h, t = 3.5 h y t = 4 h.
- En t = 6 h, repita el paso 6.3 combinando los medios recolectados de t = 4.5 h, t = 5 h, t = 5.5 h y t = 6 h. Además, a t = 6 h, placa el medio de los pocillos de control.
7. Fin del experimento
- Mida y registre el TEER al final del experimento, t = 6 h. Utilice el procedimiento descrito en los pasos 1.3.2-1.3.6.
- Descontamine la sonda sumergiéndola en etanol al 70% durante 10-15 min. Deseche los insertos o guárdelos/procese para aplicaciones adicionales, si lo desea. Permita que las placas de agar LB se incuben durante la noche y desinfecte y/o deseche de forma segura todos los demás materiales utilizados.
- Después de la incubación durante la noche, cuente las colonias bacterianas manualmente en las placas LB de dilución de la pista para determinar la cantidad de inóculo y la cantidad de transcitosis de E. coli. Asegúrese de que las placas de control no muestren ningún crecimiento bacteriano.
Resultados
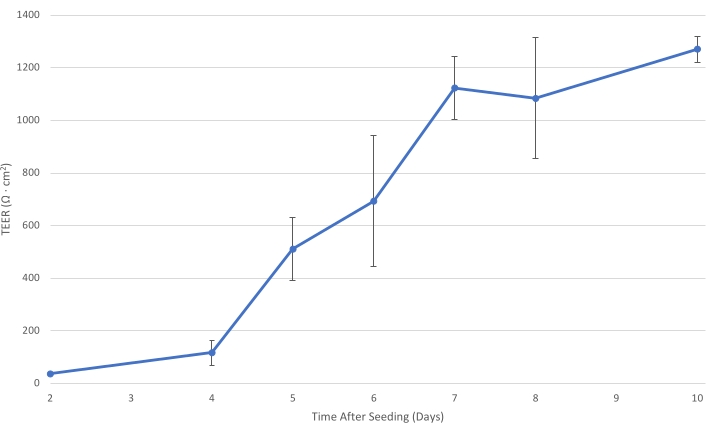
Figura 1: T84 TEER a lo largo del tiempo. A medida que la capa de celda T84 madura en el inserto, la resistencia eléctrica de la monocapa aumenta. A un TEER de al menos 1.000 Ω·cm2, la capa celular está suficientemente desarrollada para disminuir el transporte bacteriano paracelular y permitir la medición del tránsito bacteriano pri...
Discusión
Este método se deriva de técnicas utilizadas en gastroenterología y enfermedades infecciosas20. Se han utilizado modelos in vitro de la barrera epitelial intestinal para dilucidar los mecanismos por los cuales el contenido luminal interactúa con este componente relevante de la inmunidad innata 6,8. Las interacciones huésped-patógeno de E. coli neonatal invasiva también se han caracterizado por separado a través de...
Divulgaciones
Ninguno.
Agradecimientos
Este trabajo fue apoyado por una beca estudiantil Sarah Morrison emitida por la Escuela de Medicina de la Universidad de Missouri-Kansas City a A.I.
Materiales
| Name | Company | Catalog Number | Comments |
| 10,000 U/ mL Penicillin/Streptomycin Mixture | Fisher Scientific | 15-140-122 | |
| 15 mL sterile conical tubes | MidSci | C15B | |
| 2 mL microcentrifuge tubes | Avant | AVSS2000 | |
| 50 mL sterile polypropylene conical tubes | Falcon | 352070 | |
| Aspirator | Corning | 4930 | |
| Biosafety Cabinets | Labconco | 30441010028343 | Three of these are used in the method: one for sterile tissue work, one for infected tissue work, and one for bacterial work. |
| Centrifuge | Sorvall | Legend RT | |
| Disposable inoculation loops | Fisherbrand | 22363605 | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-084 | |
| Epithelial Volt/Ohm Meter | World Precision Instruments | EVOM2 | |
| Fetal Bovine Serum | Fisher Scientific | 10437028 | |
| Ham's F-12 Nutrient Mixture | Gibco | 11765-047 | |
| Hemacytometer | Sigma Aldrich, Bright Line | Z359629 | |
| Incubator shaker | New Brunswick | Innova 4080 | |
| Incubators | Thermo Scientific | 51030284 | Three of these are used in the method: one for sterile tissue culturing, one for infected tissue culturing, and one for bacterial incubation. |
| Lysogeny broth | Difco | 244610 | |
| Lysogeny broth agar | IBI Scientific | IB49101 | |
| Nikon Eclipse TS2R Microscope | Nikon | ||
| Spectrophotometer | Unico | 1100RS | |
| T84 Intestinal Cells | American Tissue Culture Collection | CCL248 | |
| Tissue culture inserts, with polyethylene trephthalate membrane, 3 µm pores, 24 well format | Falcon | 353096 | |
| Tissue culture plate, 24 wells | Falcon | 353504 | |
| Trypan blue stain | Fisher Scientific | T10282 |
Referencias
- Shakir, S. M., Goldbeck, J. M., Robison, D., Eckerd, A. M., Chavez-Bueno, S. Genotypic and phenotypic characterization of invasive neonatal Escherichia coli clinical isolates. American Journal of Perinatology. 31 (11), 975-982 (2014).
- Cole, B. K., et al. Route of infection alters virulence of neonatal septicemia Escherichia coli clinical isolates. PloS One. 12 (12), 0189032 (2017).
- Stoll, B. J., et al. Early-onset neonatal sepsis 2015 to 2017, the rise of Escherichia coli, and the need for novel prevention strategies. Journal of the American Medical Association Pediatrics. 174 (7), 200593 (2020).
- Dalgakiran, F., Witcomb, L. A., McCarthy, A. J., Birchenough, G. M., Taylor, P. W. Non-invasive model of neuropathogenic Escherichia coli infection in the neonatal rat. Journal of Visualized Experiments. (92), e52018 (2014).
- Williams, M., et al. Whole-genome sequencing-based phylogeny, antibiotic resistance, and invasive phenotype of Escherichia coli strains colonizing the cervix of women in preterm labor. BMC Microbiology. 21 (1), 330 (2021).
- Schoultz, I., Keita, &. #. 1. 9. 7. ;. The intestinal barrier and current techniques for the assessment of gut permeability. Cells. 9 (8), 1909 (2020).
- Devriese, S., et al. T84 monolayers are superior to Caco-2 as a model system of colonocytes. Histochemistry and Cell Biology. 148 (1), 85-93 (2017).
- Buckley, A., Turner, J. R. Cell biology of tight junction barrier regulation and mucosal disease. Cold Spring Harbor Perspectives in Biology. 10 (1), 029314 (2018).
- Awad, W. A., Hess, C., Hess, M. Enteric pathogens and their toxin-induced disruption of the intestinal barrier through alteration of tight junctions in chickens. Toxins. 9 (2), 60 (2017).
- Vancamelbeke, M., Vermeire, S. The intestinal barrier: A fundamental role in health and disease. Expert Review of Gastroenterology & Hepatology. 11 (9), 821-834 (2017).
- Aguilar, C., et al. Organoids as host models for infection biology - A review of methods. Experimental and Molecular Medicine. 53 (10), 1471-1482 (2021).
- Nickerson, K. P., et al. A versatile human intestinal organoid-derived epithelial monolayer model for the study of enteric pathogens. Microbiology Spectrum. 9 (1), 0000321 (2021).
- Gavin, H. E., Beubier, N. T., Satchell, K. J. The effector domain region of the Vibrio vulnificus MARTX toxin confers biphasic epithelial barrier disruption and is essential for systemic spread from the intestine. PLoS Pathogens. 13 (1), 1006119 (2017).
- Kobayashi, H., et al. Aeromonas sobria serine protease decreases epithelial barrier function in T84 cells and accelerates bacterial translocation across the T84 monolayer in vitro. PloS One. 14 (8), 0221344 (2019).
- Kalischuk, L. D., Inglis, G. D., Buret, A. G. Campylobacter jejuni induces transcellular translocation of commensal bacteria via lipid rafts. Gut Pathogens. 1 (1), 2 (2009).
- Cole, B. K., Ilikj, M., McCloskey, C. B., Chavez-Bueno, S. Antibiotic resistance and molecular characterization of bacteremia Escherichia coli isolates from newborns in the United States. PloS One. 14 (7), 0219352 (2019).
- Cadena-Herrera, D., et al. Validation of three viable-cell counting methods: Manual, semi-automated, and automated. Biotechnology Reports. 7, 9-16 (2015).
- den Hartog, G., et al. Apurinic/apyrimidinic endonuclease 1 restricts the internalization of bacteria into human intestinal epithelial cells through the inhibition of Rac1. Frontiers in Immunology. 11, 553994 (2020).
- Jett, B. D., Hatter, K. L., Huycke, M. M., Gilmore, M. S. Simplified agar plate method for quantifying viable bacteria. Biotechniques. 23 (4), 648-650 (1997).
- Lievin-Le Moal, V., Servin, A. L. Pathogenesis of human enterovirulent bacteria: Lessons from cultured, fully differentiated human colon cancer cell lines. Microbiology and Molecular Biology Reviews. 77 (3), 380-439 (2013).
- Kaczmarek, A., Budzynska, A., Gospodarek, E. Detection of K1 antigen of Escherichia coli rods isolated from pregnant women and neonates. Folia Microbiologica. 59 (5), 419-422 (2014).
- Kalita, A., Hu, J., Torres, A. G. Recent advances in adherence and invasion of pathogenic Escherichia coli. Current Opinion in Infectious Diseases. 27 (5), 459-464 (2014).
- McCool, D. J., Marcon, M. A., Forstner, J. F., Forstner, G. G. The T84 human colonic adenocarcinoma cell line produces mucin in culture and releases it in response to various secretagogues. Biochemical Journal. 267 (2), 491-500 (1990).
- Resta-Lenert, S., Barrett, K. E. Enteroinvasive bacteria alter barrier and transport properties of human intestinal epithelium: Role of iNOS and COX-2. Gastroenterology. 122 (4), 1070-1087 (2002).
- Elatrech, I., et al. Escherichia coli LF82 differentially regulates ROS production and mucin expression in intestinal epithelial T84 cells: Implication of NOX1. Inflammatory Bowel Diseases. 21 (5), 1018-1026 (2015).
- El-Aouar Filho, R. A., et al. Heterogeneous family of cyclomodulins: Smart weapons that allow bacteria to hijack the eukaryotic cell cycle and promote infections. Frontiers in Cellular and Infection Microbiology. 7, 208 (2017).
- Hopkins, A. M., Walsh, S. V., Verkade, P., Boquet, P., Nusrat, A. Constitutive activation of Rho proteins by CNF-1 influences tight junction structure and epithelial barrier function. Journal of Cell Science. 116, 725-742 (2003).
- Shiou, S. R., et al. Erythropoietin protects intestinal epithelial barrier function and lowers the incidence of experimental neonatal necrotizing enterocolitis. Journal of Biological Chemistry. 286 (14), 12123-12132 (2011).
- Newburg, D. S., Ko, J. S., Leone, S., Nanthakumar, N. N. Human milk oligosaccharides and synthetic galactosyloligosaccharides contain 3’-, 4-, and 6'-galactosyllactose and attenuate inflammation in human T84, NCM-460, and H4 cells and intestinal tissue ex vivo. Journal of Nutrition. 146 (2), 358-367 (2016).
- Burns, J. L., Griffith, A., Barry, J. J., Jonas, M., Chi, E. Y. Transcytosis of gastrointestinal epithelial cells by Escherichia coli K1. Pediatric Research. 49 (1), 30-37 (2001).
- Raut, B., Chen, L. J., Hori, T., Kaji, H. An open-source add-on EVOM((R)) device for real-time transepithelial/endothelial electrical resistance measurements in multiple transwell samples. Micromachines. 12 (3), 282 (2021).
- McCarthy, A. J., Stabler, R. A., Taylor, P. W. Genome-wide identification by transposon insertion sequencing of Escherichia coli K1 genes essential for in vitro growth, gastrointestinal colonizing capacity, and survival in serum. Journal of Bacteriology. 200 (7), 00698 (2018).
- Sayoc-Becerra, A., et al. The JAK-inhibitor tofacitinib rescues human intestinal epithelial cells and colonoids from cytokine-induced barrier dysfunction. Inflammatory Bowel Diseases. 26 (3), 407-422 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados