È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Valutazione della transcitosi intestinale degli isolati neonatali di batteriemia di Escherichia coli
In questo articolo
Riepilogo
Escherichia coli provoca sepsi nei neonati che ingeriscono i batteri intorno al momento della nascita. Il processo coinvolto nella capacità di E. coli di viaggiare dal tratto enterico al flusso sanguigno è poco compreso. Questo modello in vitro valuta la capacità dei ceppi di E. coli di viaggiare attraverso le cellule epiteliali intestinali.
Abstract
I neonati ingeriscono ceppi materni di E. coli che colonizzano il loro tratto intestinale intorno al momento del parto. I ceppi di E. coli con la capacità di traslocare attraverso l'intestino invadono il flusso sanguigno del neonato, causando batteriemia pericolosa per la vita. La metodologia qui presentata utilizza cellule epiteliali intestinali polarizzate cresciute su inserti semipermeabili per valutare la transcitosi degli isolati neonatali di batteriemia di E. coli in vitro. Questo metodo utilizza la linea cellulare intestinale T84 stabilita che ha la capacità di crescere fino alla confluenza e formare giunzioni strette e desmosomi. Dopo aver raggiunto la confluenza, i monostrati maturi T84 sviluppano resistenza transepiteliale (TEER), che può essere quantificata utilizzando un voltmetro. I valori di TEER sono inversamente correlati con la permeabilità paracellulare dei componenti extracellulari, compresi i batteri, attraverso il monostrato intestinale. Il passaggio transcellulare dei batteri (transcitosi), d'altra parte, non altera necessariamente le misurazioni TEER. In questo modello, il passaggio batterico attraverso il monostrato intestinale viene quantificato fino a 6 ore dopo l'infezione e vengono effettuate misurazioni ripetute di TEER per monitorare la permeabilità paracellulare. Inoltre, questo metodo facilita l'uso di tecniche come l'immunocolorazione per studiare i cambiamenti strutturali nelle giunzioni strette e altre proteine di adesione cellula-cellula durante la transcitosi batterica attraverso l'epitelio polarizzato. L'uso di questo modello contribuisce alla caratterizzazione dei meccanismi attraverso i quali E. coli neonatale transcitosi attraverso l'epitelio intestinale per produrre batteriemia.
Introduzione
L'Escherichia coli è la causa più comune di sepsi ad esordio precoce nei neonati 1,2,3. Il tasso di mortalità della batteriemia neonatale di E. coli può raggiungere il 40% e la meningite è una possibile complicanza associata a gravi disabilità dello sviluppo neurologico2. L'ingestione di ceppi materni di E. coli da parte del neonato può produrre batteriemia neonatale; Questo processo è stato replicato in modelli animali 2,4. Una volta ingeriti, i batteri patogeni viaggiano dal lume intestinale neonatale attraverso la barriera intestinale ed entrano nel flusso sanguigno, causando setticemia. I ceppi neonatali invasivi di E. coli che producono batteriemia variano nella loro capacità di invadere le cellule epiteliali intestinali 1,5. Tuttavia, la loro capacità di transcitare l'epitelio intestinale dopo l'invasione non è stata completamente caratterizzata.
Questo modello di transcitosi intestinale è un utile metodo in vitro per emulare il passaggio batterico attraverso l'epitelio intestinale. L'obiettivo generale dei metodi presentati in questo manoscritto è quello di confrontare la capacità degli isolati neonatali di E. coli di transcitare l'epitelio intestinale. Il modello qui descritto utilizza cellule T84, che sono cellule di adenocarcinoma intestinale umano immortalizzate 6,7. Le cellule T84 vengono coltivate fino alla confluenza su una membrana semipermeabile con due compartimenti separati. Il razionale per l'utilizzo di questa tecnica è che, come accade in vivo, queste cellule intestinali si polarizzano e sviluppano giunzioni strette mature 6,8. Il lato a contatto con la membrana diventa il lato basale. Il lato opposto delle cellule diventa il lato apicale, simile al lume intestinale dove i patogeni ingeriti aderiscono e invadono. La membrana transwell è permeabile ai batteri, ma le cellule intestinali polarizzate formano giunzioni strette, che compromettono il movimento paracellulare batterico9. Pertanto, questo metodo offre il vantaggio di un ambiente controllato in vitro che utilizza una linea cellulare umana per studiare il processo di transcitosi batterica, compresa la via transcellulare. Mentre esistono altri metodi per studiare la transcitosi dei batteri attraverso l'epitelio intestinale, il metodo transwell qui presentato offre maggiore facilità e accessibilità. Sono disponibili tecniche alternative, come quelle che utilizzano campioni ex vivo installati nei sistemi di camere di Ussing. Tuttavia, utilizzano campioni di tessuto che potrebbero non essere facilmente accessibili, in particolare se la ricerca intende studiare la fisiologia umana10. Gli organoidi intestinali rappresentano un altro esempio di alternativa in vitro per lo studio delle interazioni ospite-batteri11. Mentre i monostrati organoidi possono anche essere utilizzati nel sistema transwell per studiare la transcitosi batterica, richiedono l'isolamento e la crescita delle cellule staminali e l'uso di specifici fattori di crescita per indurre la differenziazione12. Pertanto, il loro uso è più dispendioso in termini di tempo e associato a costi maggiori rispetto al metodo transwell descritto in questo manoscritto.
La valutazione del passaggio batterico attraverso l'epitelio intestinale utilizzando questo sistema transwell in vitro è stata eseguita con successo per vari agenti patogeni. Questi studi hanno dimostrato l'utilità del sistema transwell utilizzando cellule T84 per caratterizzare la transcitosi dei batteri attraverso l'epitelio intestinale polarizzato13,14,15. Tuttavia, l'applicazione di questo metodo transwell per confrontare la capacità di transcitosi dei ceppi neonatali di E. coli produttori di batteriemia non è stata descritta in dettaglio. Questo manoscritto fornisce ad altri ricercatori un protocollo transwell standard che è affidabile e facile da usare e non richiede risorse troppo costose.
Per confrontare la capacità dei ceppi neonatali invasivi di E. coli di transcitare l'epitelio intestinale, il lato apicale del monostrato epiteliale intestinale può essere infettato da un numero noto di cellule batteriche. Dopo l'incubazione, il mezzo sul lato basale dell'epitelio può essere raccolto e i batteri quantificati per determinare la quantità di transcitosi batterica nel tempo. In questo manoscritto, i metodi presentati sono utilizzati per studiare la capacità di transcitosi di ceppi clinici neonatali di E. coli recuperati da neonati ospedalizzati con batteriemia. I criteri di inclusione per la selezione di questi isolati clinici neonatali per gli studi di transcitosi sono stati pubblicati in precedenza 1,2,16. Quando questo metodo viene eseguito utilizzando diversi ceppi di E. coli, le loro capacità di transcitosi possono essere confrontate. Attraverso questo processo, il modello di transcitosi intestinale fornisce dati preziosi per caratterizzare i fattori di virulenza di E. coli che contribuiscono al processo multifase che culmina nello sviluppo della batteriemia neonatale.
Protocollo
NOTA: Eseguire tutte le manipolazioni delle cellule T84, dei batteri, delle piastre e dei reagenti in un armadio di sicurezza di livello 2 (BSL-2) per evitare la contaminazione. Utilizzare aree separate e incubatori per tutto il lavoro che coinvolge cellule T84 sterili, cellule T84 infette ed E. coli. Gli isolati clinici di E. coli testati con i metodi qui descritti sono stati ottenuti seguendo le linee guida dell'Institutional Review Board presso il nostro istituto 1,16.
1. Preparazione di inserti di transcitosi con cellule T84 (circa 1-2 settimane prima dell'esperimento)
- Coltivare cellule T84 della American Type Culture Collection (ATCC) in terreno di coltura tissutale (MTC + antibiotici) costituito dal terreno di coltura Modified Eagle di Dulbecco: miscela di nutrienti F-12 di prosciutto (1: 1, concentrazione finale: 50% ciascuno), siero bovino fetale al 5% e miscela di antibiotici doppi penicillina / streptomicina all'1% (100 U / ml). Incubare le cellule a 37 °C con il 5% di CO2.
NOTA: Una variante di questa formulazione di mezzo senza penicillina / streptomicina (MTC senza antibiotici) viene utilizzata per le fasi successive (sezione 2 e successive) della procedura. Assicurarsi che venga utilizzata la formulazione corretta per ogni passaggio. - Lavorando all'interno di un armadio di biosicurezza (BSC), seminare le cellule T84 in inserti transwell di coltura cellulare a membrana in polietilene tereftalato con pori da 3 μm realizzati per piastre a 24 pozzetti. Includere repliche di inserti transwell per ogni condizione sperimentale desiderata più controlli non infetti per monitorare la possibile contaminazione.
- In una piastra a 24 pozzetti progettata per contenere inserti transwell, riempire il numero desiderato di pozzetti di raccolta con 1 ml di TCM + antibiotici.
- In ogni pozzetto, posizionare un inserto transwell.
- Semina questi inserti con 1 x 105 cellule T84 sospese in 500 μL di TCM + antibiotici. Quantificare il numero di cellule utilizzando un emocitometro con colorazione blu tripano o un contatore automatico di cellule17.
- Incubare le piastre transwell contenenti inserti seminati nelle stesse condizioni in cui sono state coltivate le cellule.
- Verificare con microscopia ottica che i monostrati abbiano iniziato a diventare confluenti dopo la semina dell'inserto, circa 48 ore dopo la semina.
- Ogni 2 giorni dopo la semina, misurare e registrare la resistenza elettrica transepiteliale (TEER) utilizzando un volt / ohmmetro epiteliale (EVOM) per valutare la maturità del monostrato. Una volta che gli inserti raggiungono un TEER di almeno 1.000 Ω·cm2, sono considerati pronti per il saggio18.
NOTA: Gli inserti impiegano in genere 7-10 giorni per raggiungere questo TEER dopo la semina. Le cellule T84 mostreranno una confluenza del 100% al microscopio ottico una volta raggiunta questa resistenza.- Conservare la sonda EVOM con gli elettrodi immersi in 0,15 M KCl quando non in uso.
- Prima di misurare il TEER, decontaminare la sonda dell'elettrodo immergendola in 5 ml di etanolo al 70% in un tubo conico da 50 ml per 10-15 minuti. Rimuovere la sonda, scrollarsi di dosso l'etanolo in eccesso e lasciarlo asciugare all'aria all'interno del BSC per 10 minuti. Trattenere il tubo di etanolo.
- Testare l'EVOM e la sonda posizionando la sonda decontaminata a secco in un pozzetto sterile contenente 1 ml di TCM + antibiotici con un inserto sterile all'interno contenente 500 μL di TCM + antibiotici. Assicurarsi che la lettura EVOM sia <200 Ω. Registrare questo valore vuoto per utilizzarlo nei successivi calcoli di resistenza descritti al punto 1.3.6.
- Rimuovere la sonda dal tubo di TCM + antibiotici. Conservare questo tubo per la conservazione della sonda durante l'esperimento.
- Abbassare delicatamente la sonda nel primo inserto con l'elettrodo lungo nel pozzetto di raccolta e l'elettrodo corto all'interno dell'inserto. Lasciare che l'elettrodo lungo tocchi il fondo del pozzo di raccolta, ma non spingere verso il basso in quanto ciò potrebbe disturbare il monostrato epiteliale.
- Ripetere questo processo per misurare e registrare la resistenza in Ohm (Ω) per ogni inserto. Al termine, decontaminare la sonda nell'etanolo immergendola per altri 10-15 minuti. Quindi, spostare nuovamente la sonda decontaminata nella soluzione KCl per lo stoccaggio. Sottrarre la resistenza al bianco ottenuta al punto 1.3.3 da ciascun valore ottenuto da ciascun inserto contenente celle T84. Moltiplicare la resistenza risultante (Ω) per ciascun inserto per l'area del fondo di ciascun inserto (cm 2) per ottenere la misura finale TEER (Ω·cm2).
- Una volta che il TEER raggiunge almeno 1.000 Ω·cm2, il monostrato epiteliale è maturo e pronto per i test di infezione.
- Man mano che il TEER matura, fornire alle cellule un terreno fresco ogni 1-2 giorni.
- In una nuova piastra a 24 pozzetti, aggiungere 1 ml di TCM + antibiotici in un pozzetto per ogni inserto seminato in preparazione.
- Usando una pinza sterile, trasferire con attenzione gli inserti nei pozzetti appena riforniti.
- Sostituire il supporto negli inserti.
- Rimuovere i vecchi fluidi dagli inserti inclinando la piastra e utilizzando un aspiratore a vuoto interno per rimuovere delicatamente il fluido con una punta di pipetta lungo il lato dell'inserto. L'aspiratore consente la regolazione dell'aspirazione a basso livello per prevenire la rottura delle celle. Non lasciare che la punta della pipetta tocchi il fondo dell'inserto, poiché ciò interromperebbe il monostrato epiteliale in via di sviluppo.
- Aggiungere 500 μL di MTC + antibiotici agli inserti. Visualizza il monostrato con microscopia ottica per verificare che rimanga intatto.
- Ogni 1-2 giorni, misurare il TEER su ciascun inserto come descritto sopra nei passaggi 1.3.2-1.3.6.
2. Preparazione delle cellule T84 1 giorno prima dell'esperimento con MTC senza antibiotici
- Misurare e registrare il TEER il giorno prima dell'esperimento.
- Sostituire il MTC nello stesso modo in cui si verifica durante la preparazione e la manutenzione della cella precedente. Tuttavia, la MTC senza antibiotici viene invece utilizzata in preparazione all'infezione (1 ml nel pozzetto della piastra e 500 μL nell'inserto).
3. Colture di E. coli (iniziate 1 giorno prima dell'esperimento)
ATTENZIONE: Utilizzare precauzioni di livello 2 (BSL-2) quando si lavora con ceppi clinici patogeni di E. coli.
- Prendi un tubo conico marcato da 15 ml con 5 ml di brodo di lisogenia sterile (LB) e usa un anello sterile per inoculare il brodo con una colonia da un ceppo batterico (E. coli). Ripetere questo processo, creando un tubo di coltura notturno per ogni ceppo da testare.
- Incubare la coltura per la notte, con i tappi dei tubi allentati, in uno shaker incubatore (250 rpm, 37 °C).
4. Preparazione dell'inoculo di E. coli , delle cellule epiteliali e dei materiali (la mattina dell'esperimento)
NOTA: Utilizzare MTC senza antibiotici riscaldati fino a 37 °C da questo punto in poi.
- Aggiungere 250 μL da ogni coltura LB durante la notte a 25 ml di MTC senza antibiotici in un tubo conico da 50 mL (uno per singolo ceppo). Tenere allentati i tappi dei tubi. Posizionare questi nuovi tubi di coltura nello shaker con le stesse impostazioni (250 giri/min, 37 °C) per esattamente 2 ore. Eseguire i passaggi secondari rimanenti durante l'attesa.
- Misurare il TEER su ciascun inserto, come descritto nei passaggi 1.3.2-1.3.6. Registrarli come TEER al tempo (t) = 0 h.
- Spostare gli inserti nei pozzetti in una nuova piastra e cambiare il supporto usando la tecnica descritta al punto 1.4.3. Tuttavia, questa volta, riempire i nuovi pozzetti di raccolta con 500 μL di MTC senza antibiotici e riempire gli inserti con 400 μL di MTC senza antibiotici. Conservare gli inserti all'interno dell'incubatore di coltura tissutale fino al momento dell'infezione.
- Disporre un numero sufficiente di piastre di agar LB quadrate da riscaldare a temperatura ambiente (RT) per una successiva placcatura e quantificazione batterica.
5. Inoculazione delle cellule (inizio dell'esperimento)
- Dopo esattamente 2 ore, rimuovere le colture batteriche mattutine dallo shaker e centrifugarle per 10 minuti (1.900 x g, 4 °C).
NOTA: Per tutti i seguenti passaggi, tenere tutte le sospensioni batteriche sul ghiaccio per ridurre al minimo la crescita. - Risospendere il pellet di batteri in MTC senza antibiotici. Utilizzare uno spettrofotometro per regolare la densità ottica (OD) a 0,7-0,9 e diluire ulteriormente con MTC senza antibiotici a una concentrazione di 1 x 106 unità formanti colonie (CFU)/ml (circa 1:100 diluizione). Utilizzare questa sospensione batterica per infettare ogni inserto con 1 x 105 CFU per 100 μL di volume.
- Etichettare la piastra del pozzetto e infettare ciascun inserto con 100 μL di inoculo OD-aggiustato (totale di 1 x 105 CFU per inserto). Il test è ora iniziato. Annotare l'ora e registrarla come t = 0 h.
- Placcare la sospensione batterica dell'inoculo per quantificare il CFU/mL utilizzando il metodo della diluizione in traccia, placcando 10 μL aliquote su piastre di agar LB quadrate19.
6. Quantificazione della transcitosi
- Ogni 30 minuti dopo l'inoculazione, riempire nuovi pozzetti con 500 μL di MTC senza antibiotici. Trasferire gli inserti in questi nuovi pozzetti utilizzando un diverso set di pinze sterili per ogni diverso ceppo batterico.
- Raccogliere il supporto dal pozzo di raccolta utilizzato per ciascun inserto in tubi etichettati separati. Posizionare questi tubi sul ghiaccio. Riportare le piastre del pozzo all'incubatore tra i punti temporali.
- Per ogni inserto, combinare brevemente i supporti raccolti da t = 0,5 h, t = 1 h, t = 1,5 h e t = 2 h e vortice. Placcare i mezzi raccolti su piastre di agar LB utilizzando il metodo di diluizione della traccia per quantificare la quantità di batteri transcitosi nelle prime 2 ore dell'esperimento.
NOTA: Recuperare i batteri ogni 30 minuti e tenerli sul ghiaccio assicura che la crescita batterica nei pozzetti di raccolta sia ridotta al minimo e che le misurazioni vengano eseguite su batteri prevalentemente transcitosi. - Posizionare le piastre di agar LB marcate per la diluizione del binario nell'incubatore batterico a 37 °C senza CO2 supplementare e riportare la piastra transwell T84 all'incubatore di coltura tissutale.
- A t = 4 h, ripetere il passaggio 6.3 combinando i mezzi raccolti da t = 2,5 h, t = 3 h, t = 3,5 h e t = 4 h.
- A t = 6 h, ripetere il passaggio 6.3 combinando i mezzi raccolti da t = 4,5 h, t = 5 h, t = 5,5 h e t = 6 h. Inoltre, a t = 6 h, placcare il mezzo dai pozzetti di controllo.
7. Fine dell'esperimento
- Misurare e registrare il TEER alla fine dell'esperimento, t = 6 h. Utilizzare la procedura descritta nei passaggi 1.3.2-1.3.6.
- Decontaminare la sonda immergendola in etanolo al 70% per 10-15 minuti. Eliminare gli inserti o salvarli/elaborarli per ulteriori applicazioni, se lo si desidera. Lasciare incubare le piastre di agar LB durante la notte e disinfettare e/o smaltire in modo sicuro tutti gli altri materiali utilizzati.
- Dopo l'incubazione durante la notte, contare manualmente le colonie batteriche sulle piastre LB di diluizione della traccia per determinare la quantità di inoculo e la quantità di transcitosi di E. coli. Assicurarsi che le piastre di controllo non mostrino alcuna crescita batterica.
Risultati
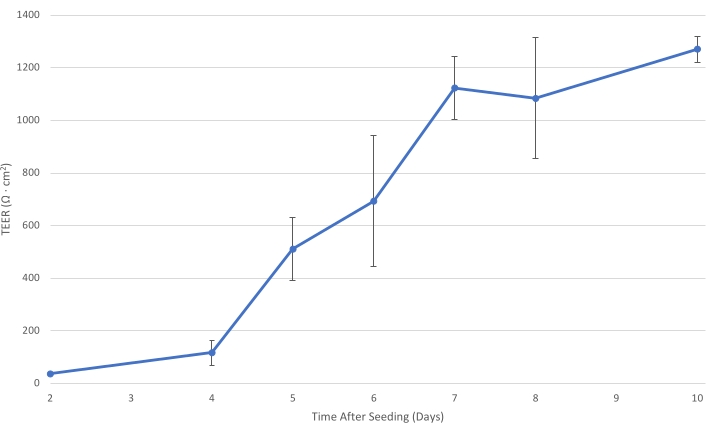
Figura 1: T84 TEER nel tempo. Man mano che lo strato di celle T84 matura sull'inserto, la resistenza elettrica del monostrato aumenta. Con un TEER di almeno 1.000 Ω·cm2, lo strato cellulare è sufficientemente sviluppato per diminuire il trasporto batterico paracellulare e consentire la misurazione del transito batterico principalmente ...
Discussione
Questo metodo deriva da tecniche utilizzate in gastroenterologia e malattie infettive20. Modelli in vitro della barriera epiteliale intestinale sono stati utilizzati per chiarire i meccanismi attraverso i quali i contenuti luminali interagiscono con questa componente rilevante dell'immunità innata 6,8. Le interazioni ospite-patogeno di E. coli neonatale invasivo sono state anche caratterizzate separatamente attraverso an...
Divulgazioni
Nessuno.
Riconoscimenti
Questo lavoro è stato supportato da una borsa di studio Sarah Morrison rilasciata dalla University of Missouri-Kansas City School of Medicine all'IA.
Materiali
| Name | Company | Catalog Number | Comments |
| 10,000 U/ mL Penicillin/Streptomycin Mixture | Fisher Scientific | 15-140-122 | |
| 15 mL sterile conical tubes | MidSci | C15B | |
| 2 mL microcentrifuge tubes | Avant | AVSS2000 | |
| 50 mL sterile polypropylene conical tubes | Falcon | 352070 | |
| Aspirator | Corning | 4930 | |
| Biosafety Cabinets | Labconco | 30441010028343 | Three of these are used in the method: one for sterile tissue work, one for infected tissue work, and one for bacterial work. |
| Centrifuge | Sorvall | Legend RT | |
| Disposable inoculation loops | Fisherbrand | 22363605 | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-084 | |
| Epithelial Volt/Ohm Meter | World Precision Instruments | EVOM2 | |
| Fetal Bovine Serum | Fisher Scientific | 10437028 | |
| Ham's F-12 Nutrient Mixture | Gibco | 11765-047 | |
| Hemacytometer | Sigma Aldrich, Bright Line | Z359629 | |
| Incubator shaker | New Brunswick | Innova 4080 | |
| Incubators | Thermo Scientific | 51030284 | Three of these are used in the method: one for sterile tissue culturing, one for infected tissue culturing, and one for bacterial incubation. |
| Lysogeny broth | Difco | 244610 | |
| Lysogeny broth agar | IBI Scientific | IB49101 | |
| Nikon Eclipse TS2R Microscope | Nikon | ||
| Spectrophotometer | Unico | 1100RS | |
| T84 Intestinal Cells | American Tissue Culture Collection | CCL248 | |
| Tissue culture inserts, with polyethylene trephthalate membrane, 3 µm pores, 24 well format | Falcon | 353096 | |
| Tissue culture plate, 24 wells | Falcon | 353504 | |
| Trypan blue stain | Fisher Scientific | T10282 |
Riferimenti
- Shakir, S. M., Goldbeck, J. M., Robison, D., Eckerd, A. M., Chavez-Bueno, S. Genotypic and phenotypic characterization of invasive neonatal Escherichia coli clinical isolates. American Journal of Perinatology. 31 (11), 975-982 (2014).
- Cole, B. K., et al. Route of infection alters virulence of neonatal septicemia Escherichia coli clinical isolates. PloS One. 12 (12), 0189032 (2017).
- Stoll, B. J., et al. Early-onset neonatal sepsis 2015 to 2017, the rise of Escherichia coli, and the need for novel prevention strategies. Journal of the American Medical Association Pediatrics. 174 (7), 200593 (2020).
- Dalgakiran, F., Witcomb, L. A., McCarthy, A. J., Birchenough, G. M., Taylor, P. W. Non-invasive model of neuropathogenic Escherichia coli infection in the neonatal rat. Journal of Visualized Experiments. (92), e52018 (2014).
- Williams, M., et al. Whole-genome sequencing-based phylogeny, antibiotic resistance, and invasive phenotype of Escherichia coli strains colonizing the cervix of women in preterm labor. BMC Microbiology. 21 (1), 330 (2021).
- Schoultz, I., Keita, &. #. 1. 9. 7. ;. The intestinal barrier and current techniques for the assessment of gut permeability. Cells. 9 (8), 1909 (2020).
- Devriese, S., et al. T84 monolayers are superior to Caco-2 as a model system of colonocytes. Histochemistry and Cell Biology. 148 (1), 85-93 (2017).
- Buckley, A., Turner, J. R. Cell biology of tight junction barrier regulation and mucosal disease. Cold Spring Harbor Perspectives in Biology. 10 (1), 029314 (2018).
- Awad, W. A., Hess, C., Hess, M. Enteric pathogens and their toxin-induced disruption of the intestinal barrier through alteration of tight junctions in chickens. Toxins. 9 (2), 60 (2017).
- Vancamelbeke, M., Vermeire, S. The intestinal barrier: A fundamental role in health and disease. Expert Review of Gastroenterology & Hepatology. 11 (9), 821-834 (2017).
- Aguilar, C., et al. Organoids as host models for infection biology - A review of methods. Experimental and Molecular Medicine. 53 (10), 1471-1482 (2021).
- Nickerson, K. P., et al. A versatile human intestinal organoid-derived epithelial monolayer model for the study of enteric pathogens. Microbiology Spectrum. 9 (1), 0000321 (2021).
- Gavin, H. E., Beubier, N. T., Satchell, K. J. The effector domain region of the Vibrio vulnificus MARTX toxin confers biphasic epithelial barrier disruption and is essential for systemic spread from the intestine. PLoS Pathogens. 13 (1), 1006119 (2017).
- Kobayashi, H., et al. Aeromonas sobria serine protease decreases epithelial barrier function in T84 cells and accelerates bacterial translocation across the T84 monolayer in vitro. PloS One. 14 (8), 0221344 (2019).
- Kalischuk, L. D., Inglis, G. D., Buret, A. G. Campylobacter jejuni induces transcellular translocation of commensal bacteria via lipid rafts. Gut Pathogens. 1 (1), 2 (2009).
- Cole, B. K., Ilikj, M., McCloskey, C. B., Chavez-Bueno, S. Antibiotic resistance and molecular characterization of bacteremia Escherichia coli isolates from newborns in the United States. PloS One. 14 (7), 0219352 (2019).
- Cadena-Herrera, D., et al. Validation of three viable-cell counting methods: Manual, semi-automated, and automated. Biotechnology Reports. 7, 9-16 (2015).
- den Hartog, G., et al. Apurinic/apyrimidinic endonuclease 1 restricts the internalization of bacteria into human intestinal epithelial cells through the inhibition of Rac1. Frontiers in Immunology. 11, 553994 (2020).
- Jett, B. D., Hatter, K. L., Huycke, M. M., Gilmore, M. S. Simplified agar plate method for quantifying viable bacteria. Biotechniques. 23 (4), 648-650 (1997).
- Lievin-Le Moal, V., Servin, A. L. Pathogenesis of human enterovirulent bacteria: Lessons from cultured, fully differentiated human colon cancer cell lines. Microbiology and Molecular Biology Reviews. 77 (3), 380-439 (2013).
- Kaczmarek, A., Budzynska, A., Gospodarek, E. Detection of K1 antigen of Escherichia coli rods isolated from pregnant women and neonates. Folia Microbiologica. 59 (5), 419-422 (2014).
- Kalita, A., Hu, J., Torres, A. G. Recent advances in adherence and invasion of pathogenic Escherichia coli. Current Opinion in Infectious Diseases. 27 (5), 459-464 (2014).
- McCool, D. J., Marcon, M. A., Forstner, J. F., Forstner, G. G. The T84 human colonic adenocarcinoma cell line produces mucin in culture and releases it in response to various secretagogues. Biochemical Journal. 267 (2), 491-500 (1990).
- Resta-Lenert, S., Barrett, K. E. Enteroinvasive bacteria alter barrier and transport properties of human intestinal epithelium: Role of iNOS and COX-2. Gastroenterology. 122 (4), 1070-1087 (2002).
- Elatrech, I., et al. Escherichia coli LF82 differentially regulates ROS production and mucin expression in intestinal epithelial T84 cells: Implication of NOX1. Inflammatory Bowel Diseases. 21 (5), 1018-1026 (2015).
- El-Aouar Filho, R. A., et al. Heterogeneous family of cyclomodulins: Smart weapons that allow bacteria to hijack the eukaryotic cell cycle and promote infections. Frontiers in Cellular and Infection Microbiology. 7, 208 (2017).
- Hopkins, A. M., Walsh, S. V., Verkade, P., Boquet, P., Nusrat, A. Constitutive activation of Rho proteins by CNF-1 influences tight junction structure and epithelial barrier function. Journal of Cell Science. 116, 725-742 (2003).
- Shiou, S. R., et al. Erythropoietin protects intestinal epithelial barrier function and lowers the incidence of experimental neonatal necrotizing enterocolitis. Journal of Biological Chemistry. 286 (14), 12123-12132 (2011).
- Newburg, D. S., Ko, J. S., Leone, S., Nanthakumar, N. N. Human milk oligosaccharides and synthetic galactosyloligosaccharides contain 3’-, 4-, and 6'-galactosyllactose and attenuate inflammation in human T84, NCM-460, and H4 cells and intestinal tissue ex vivo. Journal of Nutrition. 146 (2), 358-367 (2016).
- Burns, J. L., Griffith, A., Barry, J. J., Jonas, M., Chi, E. Y. Transcytosis of gastrointestinal epithelial cells by Escherichia coli K1. Pediatric Research. 49 (1), 30-37 (2001).
- Raut, B., Chen, L. J., Hori, T., Kaji, H. An open-source add-on EVOM((R)) device for real-time transepithelial/endothelial electrical resistance measurements in multiple transwell samples. Micromachines. 12 (3), 282 (2021).
- McCarthy, A. J., Stabler, R. A., Taylor, P. W. Genome-wide identification by transposon insertion sequencing of Escherichia coli K1 genes essential for in vitro growth, gastrointestinal colonizing capacity, and survival in serum. Journal of Bacteriology. 200 (7), 00698 (2018).
- Sayoc-Becerra, A., et al. The JAK-inhibitor tofacitinib rescues human intestinal epithelial cells and colonoids from cytokine-induced barrier dysfunction. Inflammatory Bowel Diseases. 26 (3), 407-422 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon