Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Beurteilung der intestinalen Transzytose von neonatalen Escherichia coli Bacteremia-Isolaten
In diesem Artikel
Zusammenfassung
Escherichia coli verursacht Sepsis bei Neugeborenen, die die Bakterien um den Zeitpunkt der Geburt aufnehmen. Der Prozess, der an der Fähigkeit von E. coli beteiligt ist, vom Darmtrakt in den Blutkreislauf zu gelangen, ist kaum verstanden. Dieses In-vitro-Modell bewertet die Fähigkeit von E. coli-Stämmen, durch die Darmepithelzellen zu wandern.
Zusammenfassung
Neugeborene nehmen mütterliche E. coli-Stämme auf, die ihren Darmtrakt um den Zeitpunkt der Geburt besiedeln. E. coli-Stämme mit der Fähigkeit, über den Darm zu translozieren, dringen in den Blutkreislauf des Neugeborenen ein und verursachen lebensbedrohliche Bakteriämie. Die hier vorgestellte Methodik verwendet polarisierte intestinale Epithelzellen, die auf semipermeablen Einsätzen gezüchtet wurden, um die Transzytose von neonatalen E. coli-Bakteriämie-Isolaten in vitro zu beurteilen. Diese Methode nutzt die etablierte T84-Darmzelllinie, die die Fähigkeit hat, zu Konfluenz zu wachsen und enge Verbindungen und Desmosomen zu bilden. Nach Erreichen der Konfluenz entwickeln reife T84-Monoschichten einen transepithelialen Widerstand (TEER), der mit einem Voltmeter quantifiziert werden kann. Die TEER-Werte korrelieren umgekehrt mit der parazellulären Permeabilität extrazellulärer Komponenten, einschließlich Bakterien, über die intestinale Monoschicht. Die transzelluläre Passage von Bakterien (Transzytose) hingegen verändert die TEER-Messungen nicht notwendigerweise. In diesem Modell wird die bakterielle Passage durch die intestinale Monoschicht bis zu 6 h nach der Infektion quantifiziert, und wiederholte Messungen von TEER werden durchgeführt, um die parazelluläre Permeabilität zu überwachen. Darüber hinaus erleichtert diese Methode den Einsatz von Techniken wie der Immunfärbung, um die strukturellen Veränderungen in Tight Junctions und anderen Zell-zu-Zell-Adhäsionsproteinen während der bakteriellen Transzytose über das polarisierte Epithel zu untersuchen. Die Verwendung dieses Modells trägt zur Charakterisierung der Mechanismen bei, durch die neonatale E. coli durch das Darmepithel transzytieren, um Bakteriämie zu erzeugen.
Einleitung
Escherichia coli ist die häufigste Ursache für eine früh einsetzende Sepsis bei Neugeborenen 1,2,3. Die Sterblichkeitsrate der neonatalen E. coli-Bakteriämie kann 40% erreichen, und Meningitis ist eine mögliche Komplikation, die mit schweren neurologischen Entwicklungsstörungen einhergeht2. Die Aufnahme von mütterlichen E. coli-Stämmen durch das Neugeborene kann zu neonataler Bakteriämie führen; Dieser Prozess wurde in Tiermodellenrepliziert 2,4. Einmal eingenommen, wandern pathogene Bakterien aus dem neonatalen Darmlumen über die Darmbarriere und gelangen in den Blutkreislauf, was zu einer Sepsis führt. Neonatale invasive E. coli-Stämme, die Bakteriämien produzieren, unterscheiden sich in ihrer Fähigkeit, in Darmepithelzellen einzudringen 1,5. Ihre Fähigkeit, das Darmepithel nach der Invasion zu transzytieren, ist jedoch nicht vollständig charakterisiert.
Dieses intestinale Transzytosemodell ist eine nützliche In-vitro-Methode, um die bakterielle Passage durch das Darmepithel zu emulieren. Das übergeordnete Ziel der in diesem Manuskript vorgestellten Methoden ist es, die Fähigkeit von neonatalen E. coli-Isolaten zu vergleichen, das Darmepithel zu transzytieren. Das hier beschriebene Modell verwendet T84-Zellen, die immortalisierte humane intestinale Adenokarzinomzellen 6,7 sind. T84-Zellen werden auf einer semipermeablen Membran mit zwei getrennten Kompartimenten zum Zusammenfluss gezüchtet. Der Grund für die Anwendung dieser Technik ist, dass diese Darmzellen, wie in vivo geschehen, polarisieren und reife Tight Junctions entwickeln 6,8. Die Seite, die mit der Membran in Kontakt kommt, wird zur basalen Seite. Die gegenüberliegende Seite der Zellen wird zur apikalen Seite und ähnelt dem Darmlumen, wo aufgenommene Krankheitserreger haften und eindringen. Die Transwell-Membran ist durchlässig für Bakterien, aber die polarisierten Darmzellen bilden enge Verbindungen, die die bakterielle parazelluläre Bewegung beeinträchtigen9. Somit bietet diese Methode den Vorteil einer kontrollierten In-vitro-Umgebung, in der eine menschliche Zelllinie verwendet wird, um den Prozess der bakteriellen Transzytose, einschließlich des transzellulären Weges, zu untersuchen. Während es andere Methoden gibt, um die Transzytose von Bakterien über das Darmepithel zu untersuchen, bietet die hier vorgestellte Transwell-Methode eine größere Leichtigkeit und Zugänglichkeit. Es stehen alternative Techniken zur Verfügung, z. B. unter Verwendung von Ex-vivo-Proben, die in Ussing-Kammersystemen eingerichtet wurden. Sie verwenden jedoch Gewebeproben, die möglicherweise nicht leicht zugänglich sind, insbesondere wenn die Forschung beabsichtigt, die menschliche Physiologie zu untersuchen10. Intestinale Organoide stellen ein weiteres Beispiel für eine In-vitro-Alternative zur Untersuchung von Wirt-Bakterien-Interaktionen dar11. Während organoide Monoschichten auch im Transwell-System verwendet werden können, um bakterielle Transzytose zu untersuchen, erfordern sie die Isolierung und das Wachstum von Stammzellen und die Verwendung spezifischer Wachstumsfaktoren, um die Differenzierung zu induzieren12. Daher ist ihre Verwendung zeitaufwendiger und mit höheren Kosten verbunden als die in diesem Manuskript beschriebene Transwell-Methode.
Die Beurteilung der bakteriellen Passage durch das Darmepithel mit diesem In-vitro-Transwell-System wurde erfolgreich für verschiedene Krankheitserreger durchgeführt. Diese Studien haben den Nutzen des Transwell-Systems unter Verwendung von T84-Zellen gezeigt, um die Transzytose von Bakterien über das polarisierte Darmepithel zu charakterisieren13,14,15. Die Anwendung dieser Transwell-Methode zum Vergleich der Transzytosefähigkeit von Bakteriämie erzeugenden neonatalen E. coli-Stämmen wurde jedoch nicht detailliert beschrieben. Dieses Manuskript bietet anderen Forschern ein Standard-Transwell-Protokoll, das zuverlässig und einfach zu bedienen ist und keine zu teuren Ressourcen erfordert.
Um die Fähigkeit neonataler invasiver E. coli-Stämme zur Transzytose des Darmepithels zu vergleichen, kann die apikale Seite der intestinalen Epithelmonoschicht mit einer bekannten Anzahl von Bakterienzellen infiziert werden. Nach der Inkubation kann das Medium auf der basalen Seite des Epithels gesammelt und die Bakterien quantifiziert werden, um die Menge der bakteriellen Transzytose im Laufe der Zeit zu bestimmen. In diesem Manuskript werden die vorgestellten Methoden verwendet, um die Transzytosefähigkeit von klinischen E. coli-Stämmen von Neugeborenen zu untersuchen, die von Neugeborenen mit Bakteriämie hospitalisiert wurden. Die Einschlusskriterien für die Auswahl dieser neonatalen klinischen Isolate für Transzytosestudien wurden bereits veröffentlicht 1,2,16. Wenn diese Methode mit verschiedenen E. coli-Stämmen durchgeführt wird, können ihre Transzytosefähigkeiten verglichen werden. Durch diesen Prozess liefert das intestinale Transzytosemodell wertvolle Daten zur Charakterisierung der Virulenzfaktoren von E. coli, die zu dem mehrstufigen Prozess beitragen, der in der Entwicklung einer neonatalen Bakteriämie gipfelt.
Access restricted. Please log in or start a trial to view this content.
Protokoll
HINWEIS: Führen Sie alle Manipulationen der T84-Zellen, Bakterien, Platten und Reagenzien in einer Sicherheitswerkbank der Biosicherheitsstufe 2 (BSL-2) durch, um eine Kontamination zu vermeiden. Verwenden Sie separate Bereiche und Inkubatoren für alle Arbeiten mit sterilen T84-Zellen, infizierten T84-Zellen und E. coli. Die klinischen E. coli-Isolate, die mit den hier beschriebenen Methoden getestet wurden, wurden nach den Richtlinien des Institutional Review Board an unserer Institution 1,16 gewonnen.
1. Herstellung von Transzytose-Inserts mit T84-Zellen (ca. 1-2 Wochen vor dem Experiment)
- Züchten Sie American Type Culture Collection (ATCC) T84-Zellen in Gewebekulturmedium (TCM + Antibiotika), bestehend aus Dulbeccos modifiziertem Adlermedium: Hams F-12-Nährstoffmischung (1:1, Endkonzentration: jeweils 50%), 5% fötalem Rinderserum und 1% (100 U/ml) Penicillin / Streptomycin-Dual-Antibiotika-Mischung. Die Zellen bei 37 °C mit 5%CO2 inkubieren.
HINWEIS: Eine Variation dieser Mediumformulierung ohne Penicillin/Streptomycin (TCM ohne Antibiotika) wird für die späteren Schritte (Abschnitt 2 und weiter) des Verfahrens verwendet. Stellen Sie sicher, dass für jeden Schritt die richtige Formulierung verwendet wird. - In einer Biosicherheitswerkbank (BSC) werden die T84-Zellen in Polyethylenterephthalat-Membran-Zellkultur-Transwell-Einsätze mit 3 μm-Poren für 24-Well-Platten ausgesät. Fügen Sie Transwell-Insert-Replikate für jede gewünschte Versuchsbedingung sowie nicht infizierte Kontrollen hinzu, um mögliche Kontaminationen zu überwachen.
- Füllen Sie in einer 24-Well-Platte zur Aufnahme von Transwell-Einsätzen die gewünschte Anzahl von Sammelmulden mit 1 ml TCM + Antibiotika.
- Legen Sie in jede Vertiefung einen Transwell-Einsatz.
- Säen Sie diese Einsätze mit 1 x 105 T84-Zellen, die in 500 μL TCM + Antibiotika suspendiert sind. Quantifizieren Sie die Anzahl der Zellen mit einem Hämozytometer mit Trypanblaufärbung oder einem automatisierten Zellzähler17.
- Inkubieren Sie die Transwell-Platten mit den gesäten Einsätzen unter den gleichen Bedingungen, unter denen die Zellen gezüchtet wurden.
- Mit Lichtmikroskopie wird überprüft, ob die Monoschichten nach der Aussaat des Einsatzes, etwa 48 Stunden nach der Aussaat, zu konfluieren begonnen haben.
- Messen und notieren Sie alle 2 Tage nach der Aussaat den transepithelialen elektrischen Widerstand (TEER) mit einem epithelialen Volt/Ohm-Messgerät (EVOM), um den Reifegrad der Monoschicht zu beurteilen. Sobald die Einsätze einen TEER von mindestens 1.000 Ω·cm2 erreichen, gelten sie als bereit für den Assay18.
HINWEIS: Die Einsätze benötigen in der Regel 7-10 Tage, um diesen TEER nach der Aussaat zu erreichen. Die T84-Zellen zeigen unter Lichtmikroskopie eine 100%ige Konfluenz, sobald dieser Widerstand erreicht ist.- Lagern Sie die EVOM Sonde mit den Elektroden in 0,15 M KCl getaucht, wenn sie nicht verwendet wird.
- Vor der Messung des TEER dekontaminieren Sie die Elektrodensonde, indem Sie sie 10-15 Minuten lang in ein konisches 50-ml-Röhrchen mit 70 % Ethanol eintauchen. Entfernen Sie die Sonde, schütteln Sie das überschüssige Ethanol ab und lassen Sie es 10 Minuten lang im BSC an der Luft trocknen. Behalten Sie die Röhre mit Ethanol bei.
- Testen Sie das EVOM und die Sonde, indem Sie die trockene dekontaminierte Sonde in eine sterile Vertiefung mit 1 ml TCM + Antibiotika mit einem sterilen Einsatz im Inneren mit 500 μl TCM + Antibiotika legen. Stellen Sie sicher, dass der VOM Messwert <200 Ω beträgt. Notieren Sie diesen leeren Wert, um ihn in den späteren Widerstandsberechnungen zu verwenden, die in Schritt 1.3.6 beschrieben werden.
- Entfernen Sie die Sonde aus dem Röhrchen TCM + Antibiotika. Bewahren Sie dieses Röhrchen für die Aufbewahrung der Sonde während des gesamten Experiments auf.
- Senken Sie die Sonde vorsichtig in den ersten Einsatz mit der langen Elektrode in der Auffangmulde und der kurzen Elektrode im Inneren des Einsatzes. Lassen Sie die lange Elektrode den Boden der Sammelmulde berühren, aber drücken Sie nicht nach unten, da dies die epitheliale Monoschicht stören kann.
- Wiederholen Sie diesen Vorgang, um den Widerstand in Ohm (Ω) für jeden Einsatz zu messen und aufzuzeichnen. Wenn Sie fertig sind, dekontaminieren Sie die Sonde im Ethanol, indem Sie sie für weitere 10-15 Minuten eintauchen. Bringen Sie dann die dekontaminierte Sonde zur Lagerung zurück in die KCl-Lösung. Der in Schritt 1.3.3 ermittelte Blindwiderstand wird von jedem Wert abgezogen, der von jedem Einsatz mit T84-Zellen erhalten wird. Multiplizieren Sie den resultierenden Widerstand (Ω) für jeden Einsatz mit der Fläche der Unterseite jedes Einsatzes (cm 2), um die endgültige TEER-Messung (Ω·cm2) zu erhalten.
- Sobald das TEER mindestens 1.000 Ω·cm2 erreicht, ist die epitheliale Monoschicht ausgereift und bereit für Infektionstests.
- Wenn das TEER reift, versorgen Sie die Zellen alle 1-2 Tage mit frischem Medium.
- In einer neuen 24-Well-Platte 1 ml TCM + Antibiotika zu einer Vertiefung für jede vorbereitete Saateinlage hinzufügen.
- Mit sterilen Pinzetten die Einsätze vorsichtig in die neu aufgefüllten Brunnen geben.
- Ersetzen Sie die Medien in den Einsätzen.
- Entfernen Sie das alte Medium aus den Einsätzen, indem Sie die Platte kippen und mit einem hauseigenen Vakuumsauger das Medium mit einer Pipettenspitze an der Seite des Einsatzes vorsichtig entfernen. Der Absauger ermöglicht die Regulierung der schwachen Saugleistung, um die Störung der Zellen zu verhindern. Lassen Sie die Pipettenspitze nicht die Unterseite des Einsatzes berühren, da dies die sich entwickelnde epitheliale Monoschicht stört.
- Fügen Sie 500 μL TCM + Antibiotika zu den Einsätzen hinzu. Visualisieren Sie die Monoschicht mit Lichtmikroskopie, um zu überprüfen, ob sie intakt bleibt.
- Messen Sie alle 1-2 Tage den TEER an jedem Einsatz, wie oben in den Schritten 1.3.2-1.3.6 beschrieben.
2. Vorbereitung der T84-Zellen 1 Tag vor dem Experiment mit TCM ohne Antibiotika
- Messen und notieren Sie den TEER am Tag vor dem Experiment.
- Ersetzen Sie die TCM auf die gleiche Weise wie bei der vorherigen Zellvorbereitung und -erhaltung. Zur Vorbereitung auf die Infektion wird jedoch TCM ohne Antibiotika eingesetzt (1 ml in der Plattenmulde und 500 μl im Einsatz).
3. E. coli-Kulturen (begonnen 1 Tag vor dem Experiment)
VORSICHT: Verwenden Sie Vorsichtsmaßnahmen der Biosicherheitsstufe 2 (BSL-2), wenn Sie mit pathogenen klinischen E. coli-Stämmen arbeiten.
- Nehmen Sie ein markiertes konisches 15-ml-Röhrchen mit 5 ml steriler Lysogenie-Bouillon (LB) und verwenden Sie eine sterile Schleife, um die Brühe mit einer Kolonie eines Bakterienstamms (E. coli) zu impfen. Wiederholen Sie diesen Vorgang und erstellen Sie ein Übernacht-Kulturröhrchen für jede zu testende Sorte.
- Die Übernachtkultur bei gelösten Deckeln der Röhrchen in einem Inkubatorschüttler (250 U/min, 37 °C) inkubieren.
4. Vorbereitung des E. coli-Inokulums, der Epithelzellen und -materialien (am Morgen des Experiments)
HINWEIS: Verwenden Sie ab diesem Zeitpunkt TCM ohne auf 37 °C erwärmte Antibiotika.
- Fügen Sie 250 μL aus jeder LB-Kultur über Nacht zu 25 ml TCM ohne Antibiotika in einem konischen 50-ml-Röhrchen hinzu (eines pro einzelnen Stamm). Halten Sie die Kappen der Röhrchen gelockert. Legen Sie diese neuen Kulturröhrchen mit den gleichen Einstellungen (250 U/min, 37 °C) für genau 2 h in den Shaker. Führen Sie die verbleibenden Teilschritte aus, während Sie warten.
- Messen Sie den TEER an jedem Einsatz, wie in den Schritten 1.3.2 bis 1.3.6 beschrieben. Notieren Sie diese als TEERs zum Zeitpunkt (t) = 0 h.
- Bewegen Sie die Einsätze in Vertiefungen in einer neuen Platte und wechseln Sie das Medium mit der in Schritt 1.4.3 beschriebenen Technik. Füllen Sie dieses Mal die neuen Auffangbrunnen mit 500 μL TCM ohne Antibiotika und füllen Sie die Einsätze mit 400 μL TCM ohne Antibiotika. Bewahren Sie die Einsätze bis zum Zeitpunkt der Infektion im Gewebekulturinkubator auf.
- Legen Sie eine ausreichende Anzahl von quadratischen LB-Agarplatten vor, um sich für eine spätere Beschichtung und bakterielle Quantifizierung auf Raumtemperatur (RT) zu erwärmen.
5. Impfung der Zellen (Beginn des Experiments)
- Nach genau 2 h die morgendlichen Bakterienkulturen aus dem Shaker nehmen und 10 min (1.900 x g, 4 °C) zentrifugieren.
HINWEIS: Halten Sie für alle folgenden Schritte alle Bakteriensuspensionen auf Eis, um das Wachstum zu minimieren. - Resuspendieren Sie das Bakterienpellet in TCM ohne Antibiotika. Verwenden Sie ein Spektralphotometer, um die optische Dichte (OD) auf 0,7-0,9 einzustellen, und verdünnen Sie sie weiter mit TCM ohne Antibiotika auf eine Konzentration von 1 x 106 koloniebildenden Einheiten (KBE)/ml (ca. 1:100 Verdünnung). Verwenden Sie diese Bakteriensuspension, um jeden Einsatz mit 1 x 105 KBE pro 100 μL Volumen zu infizieren.
- Beschriften Sie die Transwell-Platte und infizieren Sie jeden Einsatz mit 100 μL des OD-adjustierten Inokulums (insgesamt 1 x 105 KBE pro Einsatz). Der Test hat nun begonnen. Notieren Sie sich die Uhrzeit und notieren Sie sie als t = 0 h.
- Zur Quantifizierung der KBE/ml unter Verwendung der Spurverdünnungsmethode wird die Inokulumbakteriensuspension plattiert, wobei 10 μL-Aliquots auf quadratische LB-Agarplattenplattiert werden 19.
6. Quantifizierung der Transzytose
- Füllen Sie alle 30 Minuten nach der Impfung neue Vertiefungen mit 500 μL TCM ohne Antibiotika. Übertragen Sie die Einsätze in diese neuen Vertiefungen mit einer anderen sterilen Pinzette für jeden Bakterienstamm.
- Sammeln Sie das Medium aus der gebrauchten Auffangmulde für jeden Einsatz in separat beschrifteten Röhrchen. Legen Sie diese Röhrchen auf Eis. Geben Sie die Transwell-Platten zwischen den Zeitpunkten in den Inkubator zurück.
- Kombinieren Sie für jede Einlage die gesammelten Medien aus t = 0,5 h, t = 1 h, t = 1,5 h und t = 2 h und Wirbel kurz. Die gesammelten Medien werden auf LB-Agarplatten unter Verwendung der Track-Verdünnungsmethode beschichtet, um die Menge der in den ersten 2 h des Experiments transzytosierten Bakterien zu quantifizieren.
HINWEIS: Wenn die Bakterien alle 30 Minuten entnommen und auf Eis gehalten werden, wird sichergestellt, dass das Bakterienwachstum in den Auffangbrunnen minimiert wird und Messungen an überwiegend transzytosierten Bakterien durchgeführt werden. - Legen Sie die markierten LB-Agarplatten zur Spurverdünnung in den Bakterieninkubator bei 37 °C ohne zusätzlichesCO2 und bringen Sie die T84-Transwell-Platte in den Gewebekulturinkubator zurück.
- Bei t = 4 h wiederholen Sie Schritt 6.3, indem Sie die gesammelten Medien aus t = 2,5 h, t = 3 h, t = 3,5 h und t = 4 h kombinieren.
- Bei t = 6 h wiederholen Sie Schritt 6.3, indem Sie die gesammelten Medien aus t = 4,5 h, t = 5 h, t = 5,5 h und t = 6 h kombinieren. Zusätzlich werden bei t = 6 h die Medien aus den Kontrolltöpfen plattiert.
7. Ende des Experiments
- Messung und Aufzeichnung des TEER am Ende des Experiments, t = 6 h. Verwenden Sie das in den Schritten 1.3.2-1.3.6 beschriebene Verfahren.
- Dekontamination der Sonde, indem Sie sie für 10-15 min in 70% Ethanol tauchen. Entsorgen Sie die Einsätze oder speichern Sie sie für weitere Anwendungen, falls gewünscht. Lassen Sie die LB-Agarplatten über Nacht inkubieren und desinfizieren und/oder entsorgen Sie alle anderen verwendeten Materialien.
- Nach der Inkubation über Nacht werden die Bakterienkolonien manuell auf den LB-Platten der Spurverdünnung gezählt, um die Inokulummenge und die Menge an E. coli-Transzytose zu bestimmen. Stellen Sie sicher, dass die Kontrollplatten kein Bakterienwachstum aufweisen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
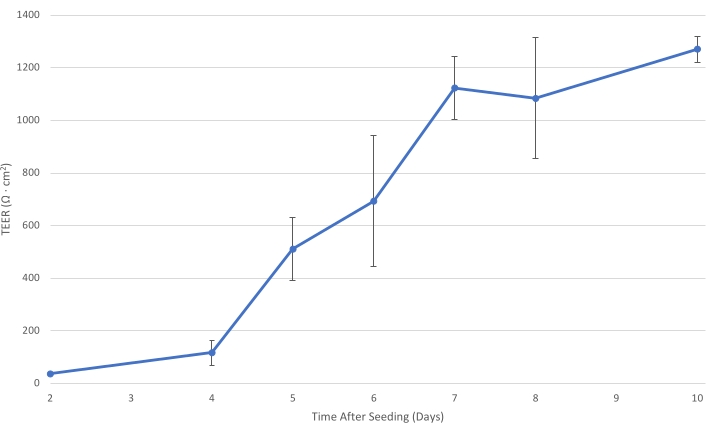
Abbildung 1: T84 TEER im Zeitverlauf. Wenn die T84-Zellschicht auf dem Einsatz reift, steigt der elektrische Widerstand der Monoschicht. Bei einem TEER von mindestens 1.000 Ω·cm2 ist die Zellschicht ausreichend entwickelt, um den parazellulären Bakterientransport zu verringern und die Messung des primär transzellulären Bakterientrans...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Diese Methode leitet sich von Techniken ab, die in der Gastroenterologie und bei Infektionskrankheitenverwendet werden 20. In-vitro-Modelle der intestinalen Epithelbarriere wurden verwendet, um die Mechanismen aufzuklären, durch die die luminalen Gehalte mit dieser relevanten Komponente der angeborenen Immunität interagieren 6,8. Die Wirt-Pathogen-Interaktionen von invasiven neonatalen E. coli wurden auch separat durch ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Nichts.
Danksagungen
Diese Arbeit wurde durch ein Sarah Morrison Studentenstipendium unterstützt, das von der University of Missouri-Kansas City School of Medicine an A.I.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 10,000 U/ mL Penicillin/Streptomycin Mixture | Fisher Scientific | 15-140-122 | |
| 15 mL sterile conical tubes | MidSci | C15B | |
| 2 mL microcentrifuge tubes | Avant | AVSS2000 | |
| 50 mL sterile polypropylene conical tubes | Falcon | 352070 | |
| Aspirator | Corning | 4930 | |
| Biosafety Cabinets | Labconco | 30441010028343 | Three of these are used in the method: one for sterile tissue work, one for infected tissue work, and one for bacterial work. |
| Centrifuge | Sorvall | Legend RT | |
| Disposable inoculation loops | Fisherbrand | 22363605 | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-084 | |
| Epithelial Volt/Ohm Meter | World Precision Instruments | EVOM2 | |
| Fetal Bovine Serum | Fisher Scientific | 10437028 | |
| Ham's F-12 Nutrient Mixture | Gibco | 11765-047 | |
| Hemacytometer | Sigma Aldrich, Bright Line | Z359629 | |
| Incubator shaker | New Brunswick | Innova 4080 | |
| Incubators | Thermo Scientific | 51030284 | Three of these are used in the method: one for sterile tissue culturing, one for infected tissue culturing, and one for bacterial incubation. |
| Lysogeny broth | Difco | 244610 | |
| Lysogeny broth agar | IBI Scientific | IB49101 | |
| Nikon Eclipse TS2R Microscope | Nikon | ||
| Spectrophotometer | Unico | 1100RS | |
| T84 Intestinal Cells | American Tissue Culture Collection | CCL248 | |
| Tissue culture inserts, with polyethylene trephthalate membrane, 3 µm pores, 24 well format | Falcon | 353096 | |
| Tissue culture plate, 24 wells | Falcon | 353504 | |
| Trypan blue stain | Fisher Scientific | T10282 |
Referenzen
- Shakir, S. M., Goldbeck, J. M., Robison, D., Eckerd, A. M., Chavez-Bueno, S. Genotypic and phenotypic characterization of invasive neonatal Escherichia coli clinical isolates. American Journal of Perinatology. 31 (11), 975-982 (2014).
- Cole, B. K., et al. Route of infection alters virulence of neonatal septicemia Escherichia coli clinical isolates. PloS One. 12 (12), 0189032(2017).
- Stoll, B. J., et al. Early-onset neonatal sepsis 2015 to 2017, the rise of Escherichia coli, and the need for novel prevention strategies. Journal of the American Medical Association Pediatrics. 174 (7), 200593(2020).
- Dalgakiran, F., Witcomb, L. A., McCarthy, A. J., Birchenough, G. M., Taylor, P. W. Non-invasive model of neuropathogenic Escherichia coli infection in the neonatal rat. Journal of Visualized Experiments. (92), e52018(2014).
- Williams, M., et al. Whole-genome sequencing-based phylogeny, antibiotic resistance, and invasive phenotype of Escherichia coli strains colonizing the cervix of women in preterm labor. BMC Microbiology. 21 (1), 330(2021).
- Schoultz, I., Keita, Å The intestinal barrier and current techniques for the assessment of gut permeability. Cells. 9 (8), 1909(2020).
- Devriese, S., et al. T84 monolayers are superior to Caco-2 as a model system of colonocytes. Histochemistry and Cell Biology. 148 (1), 85-93 (2017).
- Buckley, A., Turner, J. R. Cell biology of tight junction barrier regulation and mucosal disease. Cold Spring Harbor Perspectives in Biology. 10 (1), 029314(2018).
- Awad, W. A., Hess, C., Hess, M. Enteric pathogens and their toxin-induced disruption of the intestinal barrier through alteration of tight junctions in chickens. Toxins. 9 (2), 60(2017).
- Vancamelbeke, M., Vermeire, S. The intestinal barrier: A fundamental role in health and disease. Expert Review of Gastroenterology & Hepatology. 11 (9), 821-834 (2017).
- Aguilar, C., et al. Organoids as host models for infection biology - A review of methods. Experimental and Molecular Medicine. 53 (10), 1471-1482 (2021).
- Nickerson, K. P., et al. A versatile human intestinal organoid-derived epithelial monolayer model for the study of enteric pathogens. Microbiology Spectrum. 9 (1), 0000321(2021).
- Gavin, H. E., Beubier, N. T., Satchell, K. J. The effector domain region of the Vibrio vulnificus MARTX toxin confers biphasic epithelial barrier disruption and is essential for systemic spread from the intestine. PLoS Pathogens. 13 (1), 1006119(2017).
- Kobayashi, H., et al. Aeromonas sobria serine protease decreases epithelial barrier function in T84 cells and accelerates bacterial translocation across the T84 monolayer in vitro. PloS One. 14 (8), 0221344(2019).
- Kalischuk, L. D., Inglis, G. D., Buret, A. G. Campylobacter jejuni induces transcellular translocation of commensal bacteria via lipid rafts. Gut Pathogens. 1 (1), 2(2009).
- Cole, B. K., Ilikj, M., McCloskey, C. B., Chavez-Bueno, S. Antibiotic resistance and molecular characterization of bacteremia Escherichia coli isolates from newborns in the United States. PloS One. 14 (7), 0219352(2019).
- Cadena-Herrera, D., et al. Validation of three viable-cell counting methods: Manual, semi-automated, and automated. Biotechnology Reports. 7, 9-16 (2015).
- den Hartog, G., et al. Apurinic/apyrimidinic endonuclease 1 restricts the internalization of bacteria into human intestinal epithelial cells through the inhibition of Rac1. Frontiers in Immunology. 11, 553994(2020).
- Jett, B. D., Hatter, K. L., Huycke, M. M., Gilmore, M. S. Simplified agar plate method for quantifying viable bacteria. Biotechniques. 23 (4), 648-650 (1997).
- Lievin-Le Moal, V., Servin, A. L. Pathogenesis of human enterovirulent bacteria: Lessons from cultured, fully differentiated human colon cancer cell lines. Microbiology and Molecular Biology Reviews. 77 (3), 380-439 (2013).
- Kaczmarek, A., Budzynska, A., Gospodarek, E. Detection of K1 antigen of Escherichia coli rods isolated from pregnant women and neonates. Folia Microbiologica. 59 (5), 419-422 (2014).
- Kalita, A., Hu, J., Torres, A. G. Recent advances in adherence and invasion of pathogenic Escherichia coli. Current Opinion in Infectious Diseases. 27 (5), 459-464 (2014).
- McCool, D. J., Marcon, M. A., Forstner, J. F., Forstner, G. G. The T84 human colonic adenocarcinoma cell line produces mucin in culture and releases it in response to various secretagogues. Biochemical Journal. 267 (2), 491-500 (1990).
- Resta-Lenert, S., Barrett, K. E. Enteroinvasive bacteria alter barrier and transport properties of human intestinal epithelium: Role of iNOS and COX-2. Gastroenterology. 122 (4), 1070-1087 (2002).
- Elatrech, I., et al. Escherichia coli LF82 differentially regulates ROS production and mucin expression in intestinal epithelial T84 cells: Implication of NOX1. Inflammatory Bowel Diseases. 21 (5), 1018-1026 (2015).
- El-Aouar Filho, R. A., et al. Heterogeneous family of cyclomodulins: Smart weapons that allow bacteria to hijack the eukaryotic cell cycle and promote infections. Frontiers in Cellular and Infection Microbiology. 7, 208(2017).
- Hopkins, A. M., Walsh, S. V., Verkade, P., Boquet, P., Nusrat, A. Constitutive activation of Rho proteins by CNF-1 influences tight junction structure and epithelial barrier function. Journal of Cell Science. 116, 725-742 (2003).
- Shiou, S. R., et al. Erythropoietin protects intestinal epithelial barrier function and lowers the incidence of experimental neonatal necrotizing enterocolitis. Journal of Biological Chemistry. 286 (14), 12123-12132 (2011).
- Newburg, D. S., Ko, J. S., Leone, S., Nanthakumar, N. N. Human milk oligosaccharides and synthetic galactosyloligosaccharides contain 3’-, 4-, and 6'-galactosyllactose and attenuate inflammation in human T84, NCM-460, and H4 cells and intestinal tissue ex vivo. Journal of Nutrition. 146 (2), 358-367 (2016).
- Burns, J. L., Griffith, A., Barry, J. J., Jonas, M., Chi, E. Y. Transcytosis of gastrointestinal epithelial cells by Escherichia coli K1. Pediatric Research. 49 (1), 30-37 (2001).
- Raut, B., Chen, L. J., Hori, T., Kaji, H. An open-source add-on EVOM((R)) device for real-time transepithelial/endothelial electrical resistance measurements in multiple transwell samples. Micromachines. 12 (3), 282(2021).
- McCarthy, A. J., Stabler, R. A., Taylor, P. W. Genome-wide identification by transposon insertion sequencing of Escherichia coli K1 genes essential for in vitro growth, gastrointestinal colonizing capacity, and survival in serum. Journal of Bacteriology. 200 (7), 00698(2018).
- Sayoc-Becerra, A., et al. The JAK-inhibitor tofacitinib rescues human intestinal epithelial cells and colonoids from cytokine-induced barrier dysfunction. Inflammatory Bowel Diseases. 26 (3), 407-422 (2020).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten