Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Évaluation de la transcytose intestinale d’isolats néonatals de bactériémie à Escherichia coli
Dans cet article
Résumé
Escherichia coli provoque une septicémie chez les nouveau-nés qui ingèrent la bactérie au moment de la naissance. Le processus impliqué dans la capacité d’E. coli à se déplacer des voies entériques à la circulation sanguine est mal compris. Ce modèle in vitro évalue la capacité des souches d’E. coli à traverser les cellules épithéliales intestinales.
Résumé
Les nouveau-nés ingèrent des souches maternelles d’E. coli qui colonisent leur tractus intestinal au moment de l’accouchement. Les souches d’E. coli capables de se déplacer dans l’intestin envahissent la circulation sanguine du nouveau-né, provoquant une bactériémie potentiellement mortelle. La méthodologie présentée ici utilise des cellules épithéliales intestinales polarisées cultivées sur des inserts semi-perméables pour évaluer la transcytose d’isolats néonatals de bactériémie à E. coli in vitro. Cette méthode utilise la lignée cellulaire intestinale T84 établie qui a la capacité de se développer jusqu’à la confluence et de former des jonctions serrées et des desmosomes. Après avoir atteint la confluence, les monocouches T84 matures développent une résistance transépithéliale (TEER), qui peut être quantifiée à l’aide d’un voltmètre. Les valeurs TEER sont inversement corrélées avec la perméabilité paracellulaire des composants extracellulaires, y compris les bactéries, à travers la monocouche intestinale. Le passage transcellulaire des bactéries (transcytose), en revanche, ne modifie pas nécessairement les mesures TEER. Dans ce modèle, le passage bactérien à travers la monocouche intestinale est quantifié jusqu’à 6 heures après l’infection, et des mesures répétées de TEER sont effectuées pour surveiller la perméabilité paracellulaire. En outre, cette méthode facilite l’utilisation de techniques telles que l’immunomarquage pour étudier les changements structurels dans les jonctions serrées et d’autres protéines d’adhésion de cellule à cellule au cours de la transcytose bactérienne à travers l’épithélium polarisé. L’utilisation de ce modèle contribue à la caractérisation des mécanismes par lesquels E. coli néonatal transcytose à travers l’épithélium intestinal pour produire une bactériémie.
Introduction
Escherichia coli est la cause la plus fréquente de septicémie précoce chez les nouveau-nés 1,2,3. Le taux de mortalité de la bactériémie néonatale à E. coli peut atteindre 40 %, et la méningite est une complication possible associée à de graves troubles neurodéveloppementaux2. L’ingestion de souches maternelles d’E. coli par le nouveau-né peut produire une bactériémie néonatale; Ce processus a été reproduit dans les modèles animaux 2,4. Une fois ingérées, les bactéries pathogènes voyagent de la lumière intestinale néonatale à travers la barrière intestinale et pénètrent dans la circulation sanguine, provoquant une septicémie. Les souches néonatales invasives d’E. coli qui produisent une bactériémie varient dans leur capacité à envahir les cellules épithéliales intestinales 1,5. Cependant, leur capacité à transcyser l’épithélium intestinal après l’invasion n’a pas été complètement caractérisée.
Ce modèle de transcytose intestinale est une méthode in vitro utile pour imiter le passage bactérien à travers l’épithélium intestinal. L’objectif global des méthodes présentées dans ce manuscrit est de comparer la capacité des isolats néonatals d’E. coli à transcyser l’épithélium intestinal. Le modèle décrit ici utilise des cellules T84, qui sont des cellules d’adénocarcinome intestinal humainimmortalisées 6,7. Les cellules T84 sont cultivées jusqu’à confluence sur une membrane semi-perméable avec deux compartiments séparés. La raison d’être de cette technique est que, comme cela se produit in vivo, ces cellules intestinales se polarisent et développent des jonctions serrées matures 6,8. Le côté en contact avec la membrane devient le côté basal. Le côté opposé des cellules devient le côté apical, ressemblant à la lumière intestinale où les agents pathogènes ingérés adhèrent et envahissent. La membrane transwell est perméable aux bactéries, mais les cellules intestinales polarisées forment des jonctions serrées, qui nuisent au mouvement paracellulaire bactérien9. Ainsi, cette méthode offre l’avantage d’un environnement in vitro contrôlé utilisant une lignée cellulaire humaine pour étudier le processus de transcytose bactérienne, y compris la voie transcellulaire. Alors que d’autres méthodes existent pour étudier la transcytose des bactéries à travers l’épithélium intestinal, la méthode transwell présentée ici offre une plus grande facilité et accessibilité. Des techniques alternatives, telles que celles utilisant des échantillons ex vivo installés dans des systèmes de chambres d’Ussing, sont disponibles. Cependant, ils utilisent des échantillons de tissus qui peuvent ne pas être facilement accessibles, en particulier si la recherche vise à étudier la physiologie humaine10. Les organoïdes intestinaux représentent un autre exemple d’alternative in vitro pour étudier les interactions hôte-bactérie11. Bien que les monocouches organoïdes puissent également être utilisées dans le système transwell pour étudier la transcytose bactérienne, elles nécessitent l’isolement et la croissance de cellules souches et l’utilisation de facteurs de croissance spécifiques pour induire la différenciation12. Ainsi, leur utilisation prend plus de temps et est associée à des coûts plus élevés par rapport à la méthode transwell décrite dans ce manuscrit.
L’évaluation du passage bactérien à travers l’épithélium intestinal à l’aide de ce système transwell in vitro a été réalisée avec succès pour divers agents pathogènes. Ces études ont montré l’utilité du système transwell utilisant des cellules T84 pour caractériser la transcytose des bactéries à travers l’épithélium intestinal polarisé13,14,15. Cependant, l’application de cette méthode transwell pour comparer la capacité de transcytose des souches néonatales d’E. coli productrices de bactériémie n’a pas été décrite en détail. Ce manuscrit fournit aux autres chercheurs un protocole transwell standard fiable et facile à utiliser et ne nécessitant pas de ressources trop coûteuses.
Pour comparer la capacité des souches néonatales invasives d’E. coli à transcyser l’épithélium intestinal, la face apicale de la monocouche épithéliale intestinale peut être infectée par un nombre connu de cellules bactériennes. Après l’incubation, le milieu sur la face basale de l’épithélium peut être recueilli et les bactéries quantifiées pour déterminer la quantité de transcytose bactérienne au fil du temps. Dans ce manuscrit, les méthodes présentées sont utilisées pour étudier la capacité de transcytose des souches cliniques néonatales d’E. coli récupérées chez des nouveau-nés hospitalisés pour bactériémie. Les critères d’inclusion pour la sélection de ces isolats cliniques néonatals pour les études de transcytose ont été publiés précédemment 1,2,16. Lorsque cette méthode est réalisée en utilisant différentes souches d’E. coli, leurs capacités de transcytose peuvent être comparées. Grâce à ce processus, le modèle de transcytose intestinale fournit des données précieuses pour caractériser les facteurs de virulence d’E. coli qui contribuent au processus en plusieurs étapes qui aboutit au développement de la bactériémie néonatale.
Protocole
REMARQUE : Effectuer toutes les manipulations des cellules, bactéries, plaques et réactifs T84 dans une enceinte de sécurité de niveau de biosécurité 2 (BSL-2) pour éviter toute contamination. Utilisez des zones et des incubateurs séparés pour tous les travaux impliquant des cellules T84 stériles, des cellules T84 infectées et E. coli. Les isolats cliniques d’E. coli testés selon les méthodes décrites ici ont été obtenus conformément aux lignes directrices du comité d’examen institutionnel de notre établissement 1,16.
1. Préparation des inserts de transcytose avec des cellules T84 (environ 1-2 semaines avant l’expérience)
- Cultiver des cellules T84 de l’American Type Culture Collection (ATCC) dans un milieu de culture tissulaire (MTC + antibiotiques) composé du mélange nutritif F-12 F-12 de Dulbecco (1:1, concentration finale : 50 % chacun), de 5 % de sérum bovin fœtal et de 1 % (100 U/mL) de double mélange antibiotique pénicilline/streptomycine. Incuber les cellules à 37 °C avec 5% de CO2.
NOTE: Une variante de cette formulation de milieu sans pénicilline / streptomycine (MTC sans antibiotiques) est utilisée pour les étapes ultérieures (section 2 et suivantes) de la procédure. Assurez-vous que la formulation correcte est utilisée pour chaque étape. - En travaillant à l’intérieur d’une enceinte de biosécurité (ESB), ensemencer les cellules T84 dans des inserts transwell de culture cellulaire à membrane de polyéthylène téréphtalate avec des pores de 3 μm conçus pour des plaques à 24 puits. Inclure des répliques d’insert transwell pour chaque condition expérimentale souhaitée ainsi que des témoins non infectés pour surveiller la contamination possible.
- Dans une plaque de 24 puits conçue pour contenir des inserts transwell, remplissez le nombre désiré de puits collecteurs avec 1 mL de TCM + antibiotiques.
- Dans chaque puits, placez un insert transwell.
- Ensemencer ces inserts avec 1 x 105 cellules T84 en suspension dans 500 μL d’antibiotiques MTC+. Quantifier le nombre de cellules à l’aide d’un hémocytomètre avec coloration bleu trypan ou d’un compteur de cellules automatisé17.
- Incuber les plaques transwell contenant des inserts ensemencés dans les mêmes conditions que celles dans lesquelles les cellules ont été cultivées.
- Vérifier par microscopie optique que les monocouches ont commencé à devenir confluentes après l’ensemencement de l’insert, environ 48 heures après l’ensemencement.
- Tous les 2 jours après l’ensemencement, mesurer et enregistrer la résistance électrique transépithéliale (TEER) à l’aide d’un volt/ohmmètre épithélial (EVOM) pour évaluer la maturité de la monocouche. Une fois que les inserts atteignent un TEER d’au moins 1 000 Ω·cm2, ils sont considérés comme prêts pour le test18.
REMARQUE: Les inserts prennent généralement 7 à 10 jours pour atteindre ce TEER après l’ensemencement. Les cellules T84 montreront une confluence de 100% sous microscopie optique une fois cette résistance atteinte.- Rangez la sonde EVOM avec les électrodes immergées dans 0,15 M KCl lorsqu’elles ne sont pas utilisées.
- Avant de mesurer le TEER, décontaminez la sonde d’électrode en la plongeant dans 5 mL d’éthanol à 70 % dans un tube conique de 50 mL pendant 10 à 15 minutes. Retirez la sonde, secouez l’excès d’éthanol et laissez-le sécher à l’air libre à l’intérieur de l’ESB pendant 10 minutes. Retenir le tube d’éthanol.
- Testez la MVE et la sonde en plaçant la sonde sèche décontaminée dans un puits stérile contenant 1 mL de MTC + antibiotiques avec un insert stérile à l’intérieur contenant 500 μL de MTC + antibiotiques. Assurez-vous que la lecture EVOM est de <200 Ω. Notez cette valeur blanche pour l’utiliser dans les calculs de résistance ultérieurs décrits à l’étape 1.3.6.
- Retirez la sonde du tube de MTC + antibiotiques. Conservez ce tube pour le stockage de la sonde tout au long de l’expérience.
- Abaissez doucement la sonde dans le premier insert avec l’électrode longue dans le puits collecteur et l’électrode courte à l’intérieur de l’insert. Laissez la longue électrode toucher le fond du puits collecteur, mais ne poussez pas vers le bas car cela pourrait perturber la monocouche épithéliale.
- Répétez ce processus pour mesurer et enregistrer la résistance en Ohms (Ω) pour chaque insert. Lorsque vous avez terminé, décontaminez la sonde dans l’éthanol en l’immergeant pendant encore 10 à 15 minutes. Ensuite, replacez la sonde décontaminée dans la solution KCl pour le stockage. Soustraire la résistance à blanc obtenue à l’étape 1.3.3 de chaque valeur obtenue à partir de chaque insert contenant des cellules T84. Multipliez la résistance résultante (Ω) pour chaque insert par l’aire du fond de chaque insert (cm 2) pour obtenir la mesure TEER finale (Ω·cm2).
- Une fois que le TEER atteint au moins 1 000 Ω·cm2, la monocouche épithéliale est mature et prête pour les tests d’infection.
- Au fur et à mesure que le TEER mûrit, fournissez aux cellules un milieu frais tous les 1-2 jours.
- Dans une nouvelle plaque de 24 puits, ajouter 1 mL de MTC + antibiotiques à un puits pour chaque insert ensemencé en préparation.
- À l’aide de pinces stériles, transférez soigneusement les inserts dans les puits nouvellement réapprovisionnés.
- Remplacez le support dans les inserts.
- Retirez l’ancien média des inserts en inclinant la plaque et en utilisant un aspirateur à vide interne pour retirer délicatement le média avec une pointe de pipette sur le côté de l’insert. L’aspirateur permet la régulation de l’aspiration de faible niveau pour éviter la perturbation des cellules. Ne laissez pas l’embout de la pipette toucher le bas de l’insert, car cela perturberait la monocouche épithéliale en développement.
- Ajouter 500 μL de MTC + antibiotiques aux inserts. Visualisez la monocouche avec la microscopie optique pour vérifier qu’elle reste intacte.
- Tous les 1-2 jours, mesurer le TEER sur chaque insert comme décrit ci-dessus aux étapes 1.3.2-1.3.6.
2. Préparation des cellules T84 1 jour avant l’expérience en utilisant la MTC sans antibiotiques
- Mesurer et enregistrer le TEER la veille de l’expérience.
- Remplacer le TCM de la même manière que lors de la préparation et de l’entretien de la cellule précédente. Cependant, la MTC sans antibiotiques est plutôt utilisée en préparation à l’infection (1 mL dans le puits de la plaque et 500 μL dans l’insert).
3. Cultures d’E. coli (commencées 1 jour avant l’expérience)
MISE EN GARDE : Utilisez des précautions de niveau de biosécurité 2 (BSL-2) lorsque vous travaillez avec des souches cliniques pathogènes d’E. coli .
- Prenez un tube conique étiqueté de 15 mL avec 5 mL de bouillon lysogène stérile (LB) et utilisez une boucle stérile pour inoculer le bouillon avec une colonie d’une souche bactérienne (E. coli). Répétez ce processus, en créant un tube de culture de nuit pour chaque souche à tester.
- Incuber la culture de nuit, avec les bouchons des tubes desserrés, dans un agitateur incubateur (250 tr/min, 37 °C).
4. Préparation de l’inoculum E. coli , des cellules épithéliales et du matériel (le matin de l’expérience)
REMARQUE: Utilisez la MTC sans antibiotiques réchauffés à 37 ° C à partir de ce moment.
- Ajouter 250 μL de chaque culture LB pendant la nuit à 25 mL de MTC sans antibiotiques dans un tube conique de 50 ml (un par souche individuelle). Gardez les bouchons des tubes desserrés. Placer ces nouveaux tubes de culture dans l’agitateur aux mêmes réglages (250 tr/min, 37 °C) pendant exactement 2 h. Effectuez les sous-étapes restantes en attendant.
- Mesurer le TEER sur chaque insert, comme décrit aux étapes 1.3.2 à 1.3.6. Enregistrez-les en tant que TEER au temps (t) = 0 h.
- Déplacer les inserts vers des puits dans une nouvelle plaque et changer le support en utilisant la technique décrite à l’étape 1.4.3. Cependant, cette fois, remplissez les nouveaux puits collecteurs avec 500 μL de MTC sans antibiotiques et remplissez les inserts avec 400 μL de MTC sans antibiotiques. Conservez les inserts à l’intérieur de l’incubateur de culture tissulaire jusqu’au moment de l’infection.
- Disposer un nombre suffisant de plaques de gélose LB carrées pour chauffer à température ambiante (RT) pour un placage ultérieur et une quantification bactérienne.
5. Inoculation des cellules (début de l’expérience)
- Après exactement 2 h, retirer les cultures bactériennes du matin de l’agitateur et les centrifuger pendant 10 min (1 900 x g, 4 °C).
REMARQUE: Pour toutes les étapes suivantes, gardez toutes les suspensions bactériennes sur la glace pour minimiser la croissance. - Remettez en suspension la pastille de bactéries dans la MTC sans antibiotiques. Utiliser un spectrophotomètre pour ajuster la densité optique (DO) à 0,7-0,9, et diluer davantage avec la MTC sans antibiotiques à une concentration de 1 x 106 unités formant colonies (UFC)/mL (dilution d’environ 1:100). Utilisez cette suspension bactérienne pour infecter chaque insert avec 1 x 105 UFC par volume de 100 μL.
- Étiqueter la plaque transwell et infecter chaque insert avec 100 μL d’inoculum ajusté en DO (total de 1 x 105 UFC par insert). Le test a maintenant commencé. Notez l’heure et notez-la comme t = 0 h.
- Plaquer la suspension bactérienne d’inoculum pour quantifier l’UFC/mL en utilisant la méthode de dilution de la piste, en plaquant 10 μL aliquotes sur des plaques de gélose LB carrées19.
6. Quantifier la transcytose
- Toutes les 30 minutes suivant l’inoculation, remplir les nouveaux puits avec 500 μL de MTC sans antibiotiques. Transférer les inserts dans ces nouveaux puits à l’aide d’un jeu différent de pinces stériles pour chaque souche bactérienne différente.
- Recueillir le média du puits collecteur utilisé pour chaque insert dans des tubes étiquetés séparés. Placez ces tubes sur de la glace. Remettez les plaques transwell dans l’incubateur entre les points temporels.
- Pour chaque insert, combinez les milieux collectés à partir de t = 0,5 h, t = 1 h, t = 1,5 h et t = 2 h, et vortex brièvement. Plaquer le milieu recueilli sur des plaques de gélose LB en utilisant la méthode de dilution de piste pour quantifier la quantité de bactéries transcytosées au cours des 2 premières heures de l’expérience.
REMARQUE: Le fait de récupérer les bactéries toutes les 30 minutes et de les garder sur la glace permet de minimiser la croissance bactérienne dans les puits collecteurs et de mesurer les bactéries principalement transcytosées. - Placer les plaques de gélose LB à dilution de piste marquées dans l’incubateur bactérien à 37 °C sans supplément de CO2 et remettre la plaque transpuits T84 dans l’incubateur de culture tissulaire.
- À t = 4 h, répéter l’étape 6.3 en combinant les milieux collectés à partir de t = 2,5 h, t = 3 h, t = 3,5 h et t = 4 h.
- À t = 6 h, répéter l’étape 6.3 en combinant les milieux collectés à partir de t = 4,5 h, t = 5 h, t = 5,5 h et t = 6 h. De plus, à t = 6 h, plaquer le fluide des puits de contrôle.
7. Fin de l’expérience
- Mesurer et enregistrer le TEER à la fin de l’expérience, t = 6 h. Utilisez la procédure décrite aux étapes 1.3.2 à 1.3.6.
- Décontaminez la sonde en la plongeant dans de l’éthanol à 70% pendant 10-15 min. Jetez les inserts ou enregistrez-les pour des applications supplémentaires, si vous le souhaitez. Laisser les plaques de gélose LB incuber pendant la nuit et désinfecter et/ou éliminer en toute sécurité tous les autres matériaux utilisés.
- Après une nuit d’incubation, compter manuellement les colonies bactériennes sur les plaques LB de dilution de piste pour déterminer la quantité d’inoculum et la quantité de transcytose E. coli. Assurez-vous que les plaques de contrôle ne montrent aucune croissance bactérienne.
Résultats
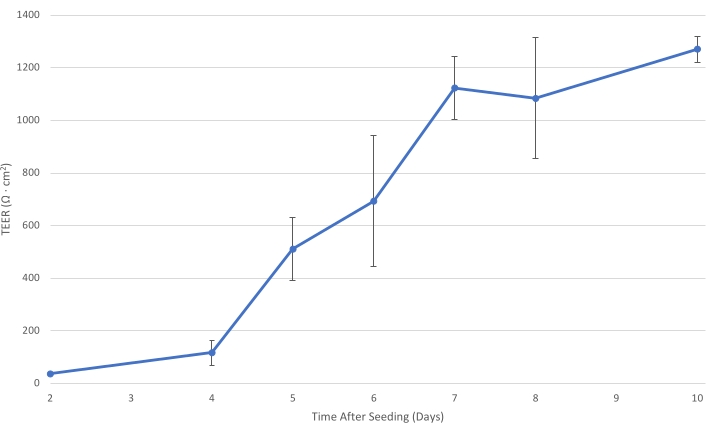
Figure 1 : T84 TEER au fil du temps. Au fur et à mesure que la couche cellulaire T84 mûrit sur l’insert, la résistance électrique de la monocouche augmente. À un TEER d’au moins 1 000 Ω·cm2, la couche cellulaire est suffisamment développée pour diminuer le transport bactérien paracellulaire et permettre la mesure du transit ...
Discussion
Cette méthode est dérivée des techniques utilisées en gastroentérologie et en maladies infectieuses20. Des modèles in vitro de la barrière épithéliale intestinale ont été utilisés pour élucider les mécanismes par lesquels le contenu luminal interagit avec cette composante pertinente de l’immunité innée 6,8. Les interactions hôte-pathogène de la bactérie invasive à E. coli néonatale ont également é...
Déclarations de divulgation
Aucun.
Remerciements
Ce travail a été soutenu par une bourse d’étudiant Sarah Morrison accordée par l’École de médecine de l’Université du Missouri-Kansas City à A.I.
matériels
| Name | Company | Catalog Number | Comments |
| 10,000 U/ mL Penicillin/Streptomycin Mixture | Fisher Scientific | 15-140-122 | |
| 15 mL sterile conical tubes | MidSci | C15B | |
| 2 mL microcentrifuge tubes | Avant | AVSS2000 | |
| 50 mL sterile polypropylene conical tubes | Falcon | 352070 | |
| Aspirator | Corning | 4930 | |
| Biosafety Cabinets | Labconco | 30441010028343 | Three of these are used in the method: one for sterile tissue work, one for infected tissue work, and one for bacterial work. |
| Centrifuge | Sorvall | Legend RT | |
| Disposable inoculation loops | Fisherbrand | 22363605 | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-084 | |
| Epithelial Volt/Ohm Meter | World Precision Instruments | EVOM2 | |
| Fetal Bovine Serum | Fisher Scientific | 10437028 | |
| Ham's F-12 Nutrient Mixture | Gibco | 11765-047 | |
| Hemacytometer | Sigma Aldrich, Bright Line | Z359629 | |
| Incubator shaker | New Brunswick | Innova 4080 | |
| Incubators | Thermo Scientific | 51030284 | Three of these are used in the method: one for sterile tissue culturing, one for infected tissue culturing, and one for bacterial incubation. |
| Lysogeny broth | Difco | 244610 | |
| Lysogeny broth agar | IBI Scientific | IB49101 | |
| Nikon Eclipse TS2R Microscope | Nikon | ||
| Spectrophotometer | Unico | 1100RS | |
| T84 Intestinal Cells | American Tissue Culture Collection | CCL248 | |
| Tissue culture inserts, with polyethylene trephthalate membrane, 3 µm pores, 24 well format | Falcon | 353096 | |
| Tissue culture plate, 24 wells | Falcon | 353504 | |
| Trypan blue stain | Fisher Scientific | T10282 |
Références
- Shakir, S. M., Goldbeck, J. M., Robison, D., Eckerd, A. M., Chavez-Bueno, S. Genotypic and phenotypic characterization of invasive neonatal Escherichia coli clinical isolates. American Journal of Perinatology. 31 (11), 975-982 (2014).
- Cole, B. K., et al. Route of infection alters virulence of neonatal septicemia Escherichia coli clinical isolates. PloS One. 12 (12), 0189032 (2017).
- Stoll, B. J., et al. Early-onset neonatal sepsis 2015 to 2017, the rise of Escherichia coli, and the need for novel prevention strategies. Journal of the American Medical Association Pediatrics. 174 (7), 200593 (2020).
- Dalgakiran, F., Witcomb, L. A., McCarthy, A. J., Birchenough, G. M., Taylor, P. W. Non-invasive model of neuropathogenic Escherichia coli infection in the neonatal rat. Journal of Visualized Experiments. (92), e52018 (2014).
- Williams, M., et al. Whole-genome sequencing-based phylogeny, antibiotic resistance, and invasive phenotype of Escherichia coli strains colonizing the cervix of women in preterm labor. BMC Microbiology. 21 (1), 330 (2021).
- Schoultz, I., Keita, &. #. 1. 9. 7. ;. The intestinal barrier and current techniques for the assessment of gut permeability. Cells. 9 (8), 1909 (2020).
- Devriese, S., et al. T84 monolayers are superior to Caco-2 as a model system of colonocytes. Histochemistry and Cell Biology. 148 (1), 85-93 (2017).
- Buckley, A., Turner, J. R. Cell biology of tight junction barrier regulation and mucosal disease. Cold Spring Harbor Perspectives in Biology. 10 (1), 029314 (2018).
- Awad, W. A., Hess, C., Hess, M. Enteric pathogens and their toxin-induced disruption of the intestinal barrier through alteration of tight junctions in chickens. Toxins. 9 (2), 60 (2017).
- Vancamelbeke, M., Vermeire, S. The intestinal barrier: A fundamental role in health and disease. Expert Review of Gastroenterology & Hepatology. 11 (9), 821-834 (2017).
- Aguilar, C., et al. Organoids as host models for infection biology - A review of methods. Experimental and Molecular Medicine. 53 (10), 1471-1482 (2021).
- Nickerson, K. P., et al. A versatile human intestinal organoid-derived epithelial monolayer model for the study of enteric pathogens. Microbiology Spectrum. 9 (1), 0000321 (2021).
- Gavin, H. E., Beubier, N. T., Satchell, K. J. The effector domain region of the Vibrio vulnificus MARTX toxin confers biphasic epithelial barrier disruption and is essential for systemic spread from the intestine. PLoS Pathogens. 13 (1), 1006119 (2017).
- Kobayashi, H., et al. Aeromonas sobria serine protease decreases epithelial barrier function in T84 cells and accelerates bacterial translocation across the T84 monolayer in vitro. PloS One. 14 (8), 0221344 (2019).
- Kalischuk, L. D., Inglis, G. D., Buret, A. G. Campylobacter jejuni induces transcellular translocation of commensal bacteria via lipid rafts. Gut Pathogens. 1 (1), 2 (2009).
- Cole, B. K., Ilikj, M., McCloskey, C. B., Chavez-Bueno, S. Antibiotic resistance and molecular characterization of bacteremia Escherichia coli isolates from newborns in the United States. PloS One. 14 (7), 0219352 (2019).
- Cadena-Herrera, D., et al. Validation of three viable-cell counting methods: Manual, semi-automated, and automated. Biotechnology Reports. 7, 9-16 (2015).
- den Hartog, G., et al. Apurinic/apyrimidinic endonuclease 1 restricts the internalization of bacteria into human intestinal epithelial cells through the inhibition of Rac1. Frontiers in Immunology. 11, 553994 (2020).
- Jett, B. D., Hatter, K. L., Huycke, M. M., Gilmore, M. S. Simplified agar plate method for quantifying viable bacteria. Biotechniques. 23 (4), 648-650 (1997).
- Lievin-Le Moal, V., Servin, A. L. Pathogenesis of human enterovirulent bacteria: Lessons from cultured, fully differentiated human colon cancer cell lines. Microbiology and Molecular Biology Reviews. 77 (3), 380-439 (2013).
- Kaczmarek, A., Budzynska, A., Gospodarek, E. Detection of K1 antigen of Escherichia coli rods isolated from pregnant women and neonates. Folia Microbiologica. 59 (5), 419-422 (2014).
- Kalita, A., Hu, J., Torres, A. G. Recent advances in adherence and invasion of pathogenic Escherichia coli. Current Opinion in Infectious Diseases. 27 (5), 459-464 (2014).
- McCool, D. J., Marcon, M. A., Forstner, J. F., Forstner, G. G. The T84 human colonic adenocarcinoma cell line produces mucin in culture and releases it in response to various secretagogues. Biochemical Journal. 267 (2), 491-500 (1990).
- Resta-Lenert, S., Barrett, K. E. Enteroinvasive bacteria alter barrier and transport properties of human intestinal epithelium: Role of iNOS and COX-2. Gastroenterology. 122 (4), 1070-1087 (2002).
- Elatrech, I., et al. Escherichia coli LF82 differentially regulates ROS production and mucin expression in intestinal epithelial T84 cells: Implication of NOX1. Inflammatory Bowel Diseases. 21 (5), 1018-1026 (2015).
- El-Aouar Filho, R. A., et al. Heterogeneous family of cyclomodulins: Smart weapons that allow bacteria to hijack the eukaryotic cell cycle and promote infections. Frontiers in Cellular and Infection Microbiology. 7, 208 (2017).
- Hopkins, A. M., Walsh, S. V., Verkade, P., Boquet, P., Nusrat, A. Constitutive activation of Rho proteins by CNF-1 influences tight junction structure and epithelial barrier function. Journal of Cell Science. 116, 725-742 (2003).
- Shiou, S. R., et al. Erythropoietin protects intestinal epithelial barrier function and lowers the incidence of experimental neonatal necrotizing enterocolitis. Journal of Biological Chemistry. 286 (14), 12123-12132 (2011).
- Newburg, D. S., Ko, J. S., Leone, S., Nanthakumar, N. N. Human milk oligosaccharides and synthetic galactosyloligosaccharides contain 3’-, 4-, and 6'-galactosyllactose and attenuate inflammation in human T84, NCM-460, and H4 cells and intestinal tissue ex vivo. Journal of Nutrition. 146 (2), 358-367 (2016).
- Burns, J. L., Griffith, A., Barry, J. J., Jonas, M., Chi, E. Y. Transcytosis of gastrointestinal epithelial cells by Escherichia coli K1. Pediatric Research. 49 (1), 30-37 (2001).
- Raut, B., Chen, L. J., Hori, T., Kaji, H. An open-source add-on EVOM((R)) device for real-time transepithelial/endothelial electrical resistance measurements in multiple transwell samples. Micromachines. 12 (3), 282 (2021).
- McCarthy, A. J., Stabler, R. A., Taylor, P. W. Genome-wide identification by transposon insertion sequencing of Escherichia coli K1 genes essential for in vitro growth, gastrointestinal colonizing capacity, and survival in serum. Journal of Bacteriology. 200 (7), 00698 (2018).
- Sayoc-Becerra, A., et al. The JAK-inhibitor tofacitinib rescues human intestinal epithelial cells and colonoids from cytokine-induced barrier dysfunction. Inflammatory Bowel Diseases. 26 (3), 407-422 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon