Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Prueba de inmunoterapia contra el cáncer en un modelo de ratón humanizado con tumores humanos
* Estos autores han contribuido por igual
En este artículo
Erratum Notice
Resumen
Este protocolo describe la generación de ratones del sistema inmune humano (HIS) para estudios de inmunooncología. Las instrucciones y consideraciones en el uso de este modelo para probar inmunoterapias humanas en tumores humanos implantados en este modelo se presentan con énfasis en la caracterización de la respuesta del sistema inmune humano al tumor.
Resumen
Revertir la naturaleza inmunosupresora del microambiente tumoral es fundamental para el tratamiento exitoso de los cánceres con medicamentos de inmunoterapia. Los modelos de cáncer murino son extremadamente limitados en su diversidad y sufren de una mala traducción a la clínica. Para servir como un modelo preclínico más fisiológico para estudios de inmunoterapia, este protocolo se ha desarrollado para evaluar el tratamiento de tumores humanos en un ratón reconstituido con un sistema inmune humano. Este protocolo único demuestra el desarrollo de ratones del sistema inmune humano (HIS, "humanizados"), seguido de la implantación de un tumor humano, ya sea un xenoinjerto derivado de la línea celular (CDX) o un xenoinjerto derivado del paciente (PDX). Los ratones HIS se generan inyectando células madre hematopoyéticas humanas CD34 + aisladas de la sangre del cordón umbilical en ratones BRGS neonatales (BALB / c Rag2-/- IL2RγC-/- NODSIRPα) altamente inmunodeficientes que también son capaces de aceptar un tumor xenogénico. Se enfatiza la importancia de la cinética y las características del desarrollo del sistema inmune humano y la implantación del tumor. Finalmente, se describe una evaluación en profundidad del microambiente tumoral mediante citometría de flujo. En numerosos estudios que utilizaron este protocolo, se encontró que el microambiente tumoral de tumores individuales se recapitula en ratones HIS-PDX; Los tumores "calientes" exhiben una gran infiltración inmune, mientras que los tumores "fríos" no lo hacen. Este modelo sirve como campo de pruebas para inmunoterapias combinadas para una amplia gama de tumores humanos y representa una herramienta importante en la búsqueda de la medicina personalizada.
Introducción
Los modelos de cáncer de ratón son importantes para establecer mecanismos básicos de crecimiento tumoral y escape inmune. Sin embargo, los estudios de tratamiento del cáncer en modelos de ratón han producido una traducción finita a la clínica debido a los modelos singénicos limitados y las diferencias específicas de la especie 1,2. La aparición de inmunoterapias como un enfoque dominante para controlar los tumores ha reiterado la necesidad de un modelo in vivo con un sistema inmune humano funcional. Los avances en ratones del sistema inmune humano (ratones HIS) durante la última década han hecho posible estudiar inmunooncología in vivo en una amplia variedad de tipos de cáncer y agentes inmunoterapéuticos 3,4,5,6. Los modelos tumorales humanos, incluidos los xenoinjertos derivados de líneas celulares y derivados de pacientes (CDX y PDX, respectivamente), crecen bien en ratones HIS y en la mayoría de los casos son casi idénticos a su crecimiento en el huésped inmunodeficiente que carece de injerto hematopoyético humano 7,8. Con base en este hallazgo clave, los investigadores han estado utilizando el modelo de ratón HIS para estudiar inmunoterapias humanas, incluidas las terapias combinadas diseñadas para alterar el microambiente tumoral (TME) para disminuir la inmunosupresión y, por lo tanto, mejorar la destrucción tumoral dirigida por el sistema inmunitario. Estos modelos preclínicos ayudan a abordar los problemas de heterogeneidad de los cánceres humanos, y también pueden predecir el éxito del tratamiento, así como monitorear las toxicidades de los medicamentos relacionados con el sistema inmunitario 9,10.
La producción de un modelo de ratón con un sistema inmune humano mediante la introducción de células madre hematopoyéticas humanas requiere un ratón inmunodeficiente receptor que no rechace el xenoinjerto. Los modelos actuales de ratón HIS se derivan de cepas de ratón inmunodeficientes que se informaron hace más de 30 años. La primera cepa de ratón inmunodeficiente descrita fueron ratones SCID que carecían de células T y B11, seguidos de un híbrido NOD-SCID con un polimorfismo SIRPα responsable de la tolerancia de los macrófagos de ratón a las células humanas, debido al aumento de la unión del alelo NOD SIRPα a la molécula CD47 humana12,13. A principios de la década de 2000, la deleción de la cadena gamma común del receptor de IL-2 (IL-2Rγc) en cepas inmunodeficientes BALB/c y NOD fue un cambio de juego para el injerto humano mejorado, debido a las deleciones genéticas que prohíben el desarrollo de células NK huésped14,15,16,17. Modelos alternativos, como ratones BRG y NRG, logran deficiencia de células T y B a través de la deleción del gen Rag1 o Rag2, requerido para los reordenamientos de los genes receptores de células T y B y, por lo tanto, la maduración y supervivencia de los linfocitos18,19. El ratón BRGS (BALB/c -Rag2 nullIl2RγCnullSirpα NOD) utilizado aquí combina la deficiencia de la cadena IL-2Rγ y el aleloNOD SIRPα en el fondo Rag2-/-, lo que resulta en un ratón altamente inmunodeficiente sin células T, B o NK, pero con suficiente vigor y salud para permitir un injerto a largo plazo de más de 30 semanas13.
Los ratones HIS se pueden generar de múltiples maneras, siendo la inyección humana de PBMC el método más directo15,18,20. Sin embargo, estos ratones tienen una expansión pronunciada de células T humanas activadas que resulta en la enfermedad de injerto contra huésped (EICH) a las 12 semanas de edad, lo que impide estudios a largo plazo. Alternativamente, las células madre hematopoyéticas humanas de la sangre del cordón umbilical (CB), la médula ósea y el hígado fetal también se pueden usar para el injerto y la producción del sistema inmunológico humano de novo. En este sistema, las células madre hematopoyéticas producen un sistema inmune humano de múltiples linajes con la generación de células T, B e inmunitarias innatas que son significativamente tolerantes al huésped del ratón, en comparación con los ratones PBMC que desarrollan principalmente células T. Por lo tanto, la EICH está ausente o muy retrasada, y los estudios pueden extenderse a ratones de hasta 10 meses de edad. CB proporciona una fuente fácil, accesible y no invasiva de células madre hematopoyéticas humanas CD34+ que facilita el injerto de múltiples ratones HIS con sistemas inmunes genéticamente idénticos 17,18,20,21. En los últimos años, los modelos de ratón HIS se han utilizado ampliamente para estudiar la inmunoterapia y el TME 3,4,5,6. A pesar del desarrollo de sistemas inmunes derivados humanos en estos ratones, los tumores de xenoinjerto humano crecen a tasas similares en comparación con los ratones inmunodeficientes de control y permiten la compleja interacción entre las células cancerosas y las células inmunes, lo cual es importante para mantener el microambiente del PDX injertado 3,7,8 . Este protocolo se ha utilizado para realizar más de 50 estudios que prueban tratamientos en ratones HIS-BRGS con PDX y CDX. Una conclusión importante es que los tumores humanos en los ratones HIS mantienen su TME único definido por la evaluación molecular del tumor en relación con la muestra inicial del paciente y las características del infiltrado inmune 3,22,23. Nuestro grupo se centra en la evaluación en profundidad del HIS tanto en los órganos inmunes como en el tumor mediante citometría de flujo multiparamétrica. Aquí, describimos un protocolo para la humanización de ratones BRGS, evaluación de quimerismo, implantación de tumores humanos, mediciones de crecimiento tumoral, administración de tratamiento del cáncer y análisis de las células HIS por citometría de flujo.
Protocolo
Todo el trabajo con animales se realizó bajo protocolos con animales aprobados por el Comité Institucional de Cuidado y Uso de Animales de la Universidad de Colorado Denver (Protocolos IACUC # 00593 y # 00021). Todo el trabajo con animales se realizó de acuerdo con la Oficina de Recursos para Animales de Laboratorio (OLAR), una instalación acreditada por la Asociación Americana para el Cuidado de Animales de Laboratorio, en el Campus Médico Anschutz de la Universidad de Colorado Denver. Todas las muestras de sangre del cordón umbilical humano se obtuvieron como donaciones de donantes no identificados y, por lo tanto, no están sujetas a la aprobación del comité de ética de investigación humana.
NOTA: Las composiciones de todos los medios y soluciones mencionados en el protocolo se incluyen en el archivo complementario 1. La Figura 1 ilustra el protocolo general para la generación y el análisis de las respuestas inmunes a los tumores en ratones HIS-BRGS.
1. Generación de ratones HIS
- Cría de ratones BALB/c -Rag2 null Il2RγCnull SirpαNOD (BRGS)
NOTA: Esta cepa es extremadamente inmunodeficiente, sin células T, B o NK. Por lo tanto, se deben utilizar medidas rigurosas para prevenir las infecciones oportunistas. Mantenga la colonia en una dieta que contenga trimetoprima y sulfadiazina en un horario alterno de 2 semanas con una dieta normal. Mantenga en el nivel más alto de precaución la habitación de la vivienda posible (por ejemplo, una instalación de ducha de barrera con acceso limitado).- Mantener colonias de ratones homocigotos BALB/c -Rag2 null Il2RγC null Sirpα NOD (BRGS) y BALB/c Rag2 null Il2RγC null SirpaBalb/c (BRG) como reproductores.
- Criar BRGS N/N×BRG B/B para generar crías BRGSB/N, para ser utilizadas como receptoras de células madre humanas. En esta colonia, los BRGSB/N son más saludables que los BRGS N/N, y se injertan a niveles equivalentes (más que BRG).
- Aislamiento de células madre humanas CD34+ a partir de CB umbilical
NOTA: No se utilizan antibióticos para este procedimiento. Por lo tanto, una buena técnica estéril es imperativa.- Coloque un bastidor de tubos cónicos de 50 ml, así como tubos cónicos de 3 x 15 ml y ~ 10 x 50 ml en un gabinete de bioseguridad esterilizado (BSC). Rocíe una bolsa de recolección de sangre con etanol al 70% y déjela secar en el BSC.
- Calcule el número de tubos cónicos de 50 ml necesarios para el aislamiento del gradiente de densidad CB = volumen CB/15, redondeado hacia arriba y a un número de tubo par. Calcular el volumen sanguíneo por tubo = volumen CB/número de tubos. Vierta sangre cuidadosamente de la bolsa CB en cada tubo cónico; esto es un máximo de 15 ml por tubo. Utilice una pipeta automática y una pipeta serológica de 25 ml para mezclar la sangre 1:1 con PBS estéril pipeteando hacia arriba y hacia abajo.
- Utilice una pipeta automática a baja velocidad y una pipeta serológica de 10 ml para cubrir lentamente la sangre con una solución de gradiente de densidad de 1,077 g/ml a temperatura ambiente (RT) (ver Tabla de materiales) sin alterar la interfaz. Evite que la punta de la pipeta toque el fondo del tubo. Repita para todos los tubos. Luego centrifugar durante 30 min a 850 x g, sin frenado, a RT para garantizar el mantenimiento del gradiente de densidad.
- Visualiza la capa leucocitaria celular encima del gradiente de densidad de 1,077 g/ml como una capa blanca turbia. Retire y deseche la capa de plasma hasta unos 10 ml por encima de la capa leucocitaria con una pipeta serológica de 25 ml y una pipeta automática.
- Recoger la capa leucocitaria con una transferencia estéril o pipeta serológica. Use la pipeta como una espátula para raspar las células del lado del tubo cónico mientras suelta el bulbo (o pipetea lentamente) para extraer las células. Combine las capas leucocitarias de dos tubos cónicos de 50 ml en un nuevo tubo cónico de 50 ml.
- Lave las células vertiendo 45 ml de HBSS estéril que contiene 2% de FBS en cada tubo cónico. Centrífuga durante 11 min a 360 x g en RT.
- Aspire el medio de lavado hasta el pellet en todos los tubos. Utilice una pipeta serológica de 10 ml y una pipeta automática para resuspender el primer pellet en 10 ml de HBSS que contenga un 2% de FBS. Resuspenda cada pellet en los mismos 10 ml de HBSS y enjuague cada tubo con 10 ml adicionales de HBSS para recoger todas las células en un solo tubo.
- Vierta 45 ml de HBSS estéril que contenga 2% de FBS en el tubo cónico. Centrifugar durante 10 min a 360 x g, a 4 °C.
- Aspire el tampón de lavado hasta el pellet de celda y vuelva a suspender el pellet en 20 ml de tampón separador magnético de celda (consulte la Tabla de materiales). Retire una pequeña alícuota para contar las células con un hemacitómetro en una dilución 1:20 en azul de metileno. Agregue el número de celdas azules y blancas. Centrifugadora a 360 x g, durante 10 min a 4 °C.
NOTA: Este protocolo utiliza tecnología de perlas magnéticas (consulte la Tabla de materiales). El protocolo se puede modificar para su uso con cualquier tecnología de separación celular, con suficiente pureza y rendimiento de las células madre CD34 +. - Aspirar el sobrenadante y resuspender el pellet de células CD34+ aislado de la sangre del cordón umbilical en 300 μL de tampón separador magnético de células por 1 x 108 células. Agregue primero 100 μL de reactivo bloqueador FcR y luego 100 μL de perlas magnéticas CD34+ por 1 x 108 células. Incubar a 4 °C durante 30 min (sin hielo).
- Añadir 5 ml de tampón separador magnético de células por 1 x 108 células y girar a 360 x g durante 10 min a 4 °C. Repita el paso de lavado y vuelva a suspender el pellet en 500 μL de tampón separador magnético de células por 1 x 108 celdas en un tubo cónico de 15 ml etiquetado como "no fraccionado".
- Etiquete dos tubos cónicos más de 15 ml "CD34-" y "CD34+". Coloque los tres tubos cónicos de 15 ml (no fraccionados, CD34- y CD34+) en las ranuras A1, B1 y C1, respectivamente, en una rejilla de enfriamiento (consulte la Tabla de materiales). Separe las celdas utilizando el programa de selección positiva de dos columnas en un separador automático de celdas magnéticas (consulte la Tabla de materiales) en un BSC, de acuerdo con las instrucciones del instrumento del fabricante.
- Expansión y congelación de células madre humanas CD34+
- Alícuota 10 μL de la suspensión de células CD34+ recuperada (2 ml) en un portaobjetos del hemocitómetro y contar las células con un aumento de 10x. Calcule el número total de células CD34+ multiplicando el recuento de células por 2 x 104. Divida el número total de células CD34+ entre 250.000 para calcular el número de viales a congelar (50.000 células por cachorro de ratón antes de la expansión in vitro ).
- Prepare el FCS al 10% de CB medium Iscove (más 1 ml extra para la pérdida de filtrado), suplementado con 40 ng / ml de factor de células madre, 20 ng / ml Flt3L y 10 ng / ml IL-6, y pase a través de un filtro de 0.22 μm. Resuspender las células CD34+ a 100.000 por ml de medio CB e incubar a 37 °C. El día 3, añadir un volumen equivalente de medio CB sin citocinas al matraz que contiene las células y el medio CB con citocinas.
NOTA: La adición de estas citoquinas al medio CB promueve la supervivencia y la expansión de las células CD34+ al tiempo que previene la diferenciación. - Cosecha las células CD34+ expandidas el día 5. Pipetear la suspensión celular hacia arriba y hacia abajo y recoger en un tubo cónico de 50 ml. Añadir suficiente CB medio para cubrir el fondo del matraz. Con un raspador de celdas, raspe todo el fondo del matraz. Recoja todos los medios en el mismo tubo de 50 ml y centrifugar a 360 x g durante 11 min.
- Resuspender las células en 2 mL de medio CB. Guarde la gota final de la pipeta en una placa de 96 pocillos para contar. Diluir las células 1:1 en azul de tripano y añadir 10 μL al hemacitómetro, luego contar y promediar las células de cuatro cuadrantes. Calcule el número total de células CD34+ multiplicando el recuento de células por 4 x 104 y registre la viabilidad.
- Hacer n+1 ml de medio de congelación, donde n es el número de viales de congelación calculado en el paso 1.3.1. Prepare el medio de congelación agregando un 10% (v / v) de DMSO a FBS y manténgalo en hielo. Etiquete los crioviales con CB#, CD34+ d5 y fecha.
- Girar las células CD34+ a 360 x g durante 10 min a 4 °C. Aspirar el medio hasta el pellet y resuspender el pellet celular en medio de congelación. Alícuota 1 ml de suspensión celular en cada vial y dividir cualquier resto uniformemente entre los viales. Agregue los viales a un congelador de celdas de isopropanol enfriado a 4 ° C, colóquelos a -80 ° C y transfiéralos a nitrógeno líquido para almacenar durante >90 días.
- Irradiación de crías de ratón
- Recoja los cachorrosBRGS B / N , 1-3 días después del nacimiento, en una caja de plástico esterilizada en autoclave con relleno. Agregue una pequeña cantidad de ropa de cama con los cachorros. Etiquete la caja con el número de jaula y el número de cachorros.
- Configure el irradiador (ver Tabla de materiales) para una dosis de 300 rad. Coloque la caja de cachorros en el irradiador y expóngalos a 300 rad. Lleve a los cachorros de vuelta a su jaula, colóquelos en una pila y cúbralos con ropa de cama.
- Inyecciones de cachorros y preparación de células CD34+
- Comience la preparación de células CD34 + ~ 3 h después de la irradiación. Calentar 10 ml de medios CB en un tubo cónico de 50 ml. Realice todos los pasos en un BSC estéril.
- Recupere un vial de células CD34+ expandidas y congeladas in vitro por cada cuatro a seis cachorros para inyectar. Descongelar rápidamente a 55 °C, hasta que solo se vea una pequeña cantidad de hielo, y añadir las células al medio CB calentado (el vial aún debe estar frío al tacto). Use 1 ml de medio para enjuagar cada vial y haga girar las células a 360 x g durante 12 minutos a 4 °C.
NOTA: Se encontró que la descongelación rápida a 55 °C produce una mejor viabilidad celular (90%-95%) que la descongelación a 37 °C. - Aspire el medio con cuidado. Resuspenda el pellet (pequeño) en 2 ml de medios CB, mezcle suavemente y agregue ~ 30 μL de la suspensión celular a un solo pocillo en una placa de conteo. Diluir 1:1 en azul de tripano, luego agregar 10 μL a un hemocitómetro y contar y promediar las células de cuatro cuadrantes.
- Calcule el número total de células CD34+ multiplicando el recuento de células por 4 x 104 y luego registre la viabilidad. Girar a 360 x g durante 12 min a 4 °C.
- Aspirar el medio con cuidado y resuspender el pellet celular en 100 μL de PBS estéril por n + 1 cachorros para inyectar, lo que resulta en 250,000-450,000 células CD34 + por ratón. Coloque el tubo cónico sobre hielo en un recipiente de transporte y viaje al vivero para la inyección del cachorro.
- Lleve una lámpara de calor, pañales, jeringa de 1 ml, aguja de 18 G, aguja de 30 g y la preparación de células CD34 + en recipientes estériles al vivarium BSC. Coloque un pañal estéril ~ 2 pies debajo de la lámpara de calor. Recuperar la jaula con la cama a inyectar y colocar en el BSC.
- Ensamble la jeringa con una aguja biselada de 18 G. Haciendo coincidir el ángulo del tubo cónico con el bisel de la aguja, mezcle suavemente y extraiga la suspensión celular. Coloque a los cachorros en el pañal para que se calienten (esté atento al sobrecalentamiento). Retire el aire de la jeringa y reemplace la aguja de 18 G con la aguja de 30 G, y luego empuje cuidadosamente la jeringa hasta que la suspensión celular esté justo en la punta de la aguja.
NOTA: Alternativamente, se puede usar una jeringa de insulina. - Lleve un cachorro a la vez al borde del pañal lejos de la lámpara de calor. Inmovilice al cachorro de lado debajo del pulgar y el índice, lo que permite una visión clara de la cara. Observe la vena a través de la mejilla debajo de donde estará la oreja. Inserte la aguja superficialmente en la vena (IV) más cercana al ojo e inyecte lentamente 50 μL de células.
- Compruebe si se está formando una burbuja a partir de una inyección subcutánea. Si es así, inserte la aguja más profundamente y proceda a inyectar células. Una pequeña gota de sangre/hematoma será visible cuando tenga éxito.
NOTA: Consulte el procedimiento de Gombash Lampe et al.24 con respecto a la inyección de cachorros. - Con el cachorro aún inmovilizado, realizar una inyección intrahepática (HI) con otros 50 μL de células (100 μL en total por cachorro de IV+IH). El hígado se puede visualizar como una mancha oscura entre la banda de leche blanca y la caja torácica. Coloque al cachorro inyectado en un pañal más alejado de los cachorros no inyectados y caliente.
NOTA: La inyección IV produce un mejor quimerismo a largo plazo que la inyección de IH sola25, pero las inyecciones intravenosas no siempre son exitosas. Por lo tanto, dividir la inyección entre IV e IH asegura el injerto en un mayor porcentaje de ratones. - Repita las inyecciones de venas faciales e IH para todos los cachorros. Limpie la sangre, devuelva a los cachorros al nido en su jaula y cúbralos con ropa de cama.
2. Probar el quimerismo humano en sangre
- Prueba quimerismo en la sangre de ratones HIS a las 10 y 14 semanas de edad. Recolectar 50 μL de sangre a través de la vena retroorbitaria o utilizando un método alternativo aprobado por IACUC.
- Para hemorragias retroorbitales, anestesia a los ratones con un vaporizador de isoflurano ajustado a 5 durante 1-2 min y luego baja el ajuste del vaporizador a 4. Baje el vaporizador según sea necesario para permitir que los ratones permanezcan suficientemente oxigenados mientras están bajo anestesia. No mantenga los ratones bajo isoflurano durante más de 5 minutos.
- Coloque la nariz del ratón anestesiado en el accesorio del cono nasal del vaporizador de isoflurano y coloque una gota del analgésico (solución oftálmica de HCl proparacaína al 0,5%) en el ojo.
- Después de 1 min, retire la proparacaína con una gasa estéril, proptose el ojo e inserte un tubo de hematocrito heparinizado de 75 mm retroorbitalmente para recolectar 50 μL de sangre. Expulse la sangre en un tubo de microfuga de 1,5 ml que contenga 50 μL de heparina y mezcle suavemente.
- Pellizque el ojo cerrado con una gasa estéril para detener el sangrado y aplique una gota de proparacaína. Retire el ratón del isoflurano y recupéralo en una jaula limpia.
- Aislamiento de PBMC de sangre de ratón
- Mezclar la sangre/heparina pipeteando suavemente hacia arriba y hacia abajo y superponer lentamente sobre 500 μL de gradiente de densidad de 1,077 g/ml, teniendo cuidado de no perturbar la interfaz. Centrifugar los tubos a 1.220 x g durante 20 min en RT sin frenos.
- Visualice la capa leucocitaria celular como una capa turbia sobre el gradiente de densidad de 1,077 g/ml, debajo del plasma. Extraer tantas células como sea posible de la capa leucocitaria con una pipeta de 200 μL y añadir a los nuevos tubos de 1,5 ml que contengan 750 μL de medio de cosecha. Centrífuga a 360 x g durante 11 min en RT.
- Aspirar el medio hasta 50 μL y resuspender el pellet en 750 μL de medio de cosecha. Centrifugar a 360 x g durante 10 min a 4 °C y aspirar de nuevo a 50 μL. Las células están listas para ser resuspendidas en la mancha superficial.
- Tinción superficial y análisis citométrico de flujo
- Complete la hoja de trabajo del panel de tinción (Tabla 1; "spectral flow bleed panel" worksheet) con números de ratón y preparar el cóctel de tinción de anticuerpos añadiendo todos los anticuerpos fluorescentes al tampón de tinción (Archivo suplementario 1). Haga un diseño de placa para agregar las muestras a una placa de fondo en U de 96 pocillos. Marque los fondos de los pozos con un marcador permanente. Valorar los anticuerpos utilizando un procedimiento estándar antes de la tinción para determinar la concentración apropiada26.
- Agregue 62 μL del cóctel de manchas superficiales a cada pocillo de la placa de fondo en U de 96 pocillos. Resuspender las células en los 50 μL restantes en el tubo y añadir cada muestra a su pocillo correspondiente. Incluya un pocillo para un control positivo de tinción (PBMC humanas + esplenocitos de ratón, 1 x 106 células cada uno) para teñir junto con las muestras. Mezclar por pipeteo e incubar la mezcla durante 15 min a 4 °C.
- Centrifugar a 680 x g durante 3 min a 4 °C. Mueva la placa en el fregadero para eliminar el sobrenadante y lave las células resuspendiendo en 150 μL de tampón de tinción pipeteando suavemente ~ 10x. Girar y resuspender cada pocillo en 150 μL de tampón de tinción.
- Adquiera los datos de 100 μL de cada muestra en un citómetro de flujo (consulte la Tabla de materiales) y exporte los archivos .fcs. Importe los archivos .fcs al software de edición de datos dinámicos (consulte Tabla de materiales). Aplique una puerta de polígono a una gráfica FSC-A x SSC-A que rodea las celdas, excluyendo cualquier residuo. Seleccione las celdas dentro de la puerta (ver Tabla 1; Hoja de trabajo "Spectral Flow Bleed Gating").
- Cambie los ejes a (FSC-A x FSC-H) y compuerta las celdas contenidas en la diagonal lineal excluyendo los dobletes que sobresalen de la línea. Seleccione estas celdas y cambie los ejes a (hCD45 x mCD45).
- Aplique una puerta de polígono a la población hCD45+ y aplique el nombre "humano". Aplique una puerta de polígono a la población mCD45+ y aplique el nombre "mouse". Cree una estadística de recuento para las poblaciones humanas y de ratón.
- Seleccione la población humana y cambie los ejes a (CD19 x CD3). Aplique una puerta de polígono a las celdas CD19+ y asígnele el nombre "células B". Aplique una puerta de polígono a la población CD3+ y asígnele el nombre "células T". Aplique una puerta de polígono a la población doble negativa y asígnele el nombre "NonTB".
- Seleccione la población de células T y cambie los ejes a (CD8 x CD4). Aplique una puerta de polígono a las poblaciones CD4 y CD8 positivas y llámelas "CD4+" y "CD8+", respectivamente.
- Seleccione la población no TB y cambie los ejes a (CD56 x mieloide). Aplique una puerta de polígono a la población CD56 positiva total, incluidos los eventos positivos dobles, y llámela "células NK". Aplique una puerta de polígono a la población CD56 negativa y mieloide positiva y llámela "mieloide".
- Cree una tabla para las estadísticas de porcentaje y recuento de todas las poblaciones y expórtela a un software de hoja de cálculo (consulte Tabla de materiales). Calcule %hCD45 quimerismo = %hCD45/(%hCD45 + mCD45).
- Excluir los ratones HIS que son <20% hCD45+ (de mCD45 + hCD45) para experimentos adicionales.
NOTA: En este estudio, las PBMC se prepararon utilizando una separación de gradiente de densidad de 1.077 g / ml para una depleción de glóbulos rojos más limpia. Este procedimiento excluyó granulocitos humanos y de ratón de la capa PBMC. Alternativamente, se puede utilizar la lisis de glóbulos rojos.
3. Inyección de tumores en ratones
- Antes de la inyección del tumor, confirme los números de células T a partir de los datos de sangrado de 14 semanas. Los tumores se inyectan para estar listos para la cosecha entre 20-26 semanas si las células T son >20%, y entre 24-28 semanas si las células T son <20% de la población total de células inmunes. Este tiempo de inyección asegura que los ratones tengan entre 20 y 28 semanas de edad y tengan un número suficiente de células T al final del estudio.
- Inyección de xenoinjertos derivados de líneas celulares (CDX)
NOTA: El procedimiento se describe utilizando células de adenocarcinoma de mama MDA-MB-231 (consulte la Tabla de materiales) como ejemplo.- Descongele una alícuota de células congeladas, lave 1x resuspendiendo en 10 ml de DMEM suplementado con 10% de FBS, 1% de PenStrep y 1% de aminoácidos no esenciales, y centrifugar a 360 x g durante 10 min a 4 °C. Aspirar el medio y resuspender en 10 ml de DMEM suplementado con 10% de FBS, 1% de PenStrep y 1% de aminoácidos no esenciales en un matraz T25 e incubar a 37 °C con 5% deCO2.
NOTA: Las líneas celulares se autentican mediante PCR para garantizar el tipo de célula correcto. Los sobrenadantes celulares se prueban para detectar micoplasma a través de un ensayo bioquímico antes de la inyección. - Paso y expansión de las células durante la fase de crecimiento exponencial a aproximadamente el 80% de confluencia.
- Aspirar el medio, enjuagar las células con 5-10 ml de PBS estéril (pH 7,2) e incubar con 1 ml de tripsina-EDTA al 0,25% durante 1-5 min hasta que las células se desprendan del matraz.
- Agregue 5 ml del mismo DMEM y mezcle pipeteando hacia arriba y hacia abajo. Pasar los 6 ml completos de suspensión celular a un nuevo matraz T75 tratado con cultivo tisular (dilución 1:3) y añadir 10 ml adicionales de DMEM.
- Pasar las células cada 2-3 días al 80% de confluencia a 1:3 de placa dilucional en DMEM para expandir las células.
- Cosechar las células con 0,25% de tripsina-EDTA durante la fase de crecimiento exponencial dentro de seis pasajes y lavar con PBS. Mezclar PBS y extracto de membrana basal (ver Tabla de materiales) en una proporción de 1:1 y añadir a las células a una concentración final de 5 x 107 células/ml.
- Anestesiar a los ratones con isoflurano como se describe en el paso 2.1.1. Inyectar por vía subcutánea 100 μL (5 x 106 células) de suspensión de células tumorales en cada flanco con una aguja de 23 G.
- Descongele una alícuota de células congeladas, lave 1x resuspendiendo en 10 ml de DMEM suplementado con 10% de FBS, 1% de PenStrep y 1% de aminoácidos no esenciales, y centrifugar a 360 x g durante 10 min a 4 °C. Aspirar el medio y resuspender en 10 ml de DMEM suplementado con 10% de FBS, 1% de PenStrep y 1% de aminoácidos no esenciales en un matraz T25 e incubar a 37 °C con 5% deCO2.
- Realizar la inyección de xenoinjertos derivados del paciente (PDX) utilizando trócares. El procedimiento de inyección y otras instrucciones de desarrollo y mantenimiento para el modelo PDX están disponibles en la literatura27.
4. Medición del crecimiento tumoral
- Verifique el progreso del tumor una vez a la semana después de la implantación palpando a lo largo del flanco para el crecimiento del tumor. Una vez que los tumores son palpables, anestesiar a los ratones con isoflurano (como se describe en el paso 2.1.1) y afeitarse en cada flanco con un recortador eléctrico, teniendo cuidado alrededor del tumor para prevenir ulceraciones a medida que el tumor crece.
NOTA: Los ratones pueden ser afeitados antes de la inyección del tumor para prevenir lesiones en la piel alrededor de los tumores; Sin embargo, la tasa de recrecimiento del cabello podría obstruir las mediciones del tumor. - Mida la longitud y el ancho de los tumores dos veces por semana usando calibradores y registre las mediciones en mm. Informar las mediciones tumorales como volumen tumoral (mm3) utilizando la fórmula
 . Tenga cuidado de no permitir que la carga tumoral exceda los 2.000 mm3 por tumor individual o un volumen combinado de 3.000mm3.
. Tenga cuidado de no permitir que la carga tumoral exceda los 2.000 mm3 por tumor individual o un volumen combinado de 3.000mm3.
NOTA: Las pautas para el uso de ratones en la investigación del cáncer varían según la ubicación. Consulte las políticas de cuidado y uso de animales en la institución del investigador.
5. Tratamientos farmacológicos
- Asigne los ratones HIS en grupos de tratamiento equivalentes basados en el quimerismo hCD45, el quimerismo hCD3 y el quimerismo hCD8. Una vez que los tumores alcanzan los 100mm3 en promedio, comience los tratamientos farmacológicos.
NOTA: El número de ratones por grupo se basa en el número de ratones HIS generados a partir del CB. Se recomienda un mínimo de cuatro ratones por grupo. La coincidencia multigrupo no bipartita (por ejemplo, en el software R-vivo Manila) es útil para esta agrupación28. - Ruta y frecuencia de las drogas
- Inyecte inhibidores anti-PD-1 (nivolumab, pembrolizumab) intraperitonialmente (i.p.) a 30 mg/kg 1x por semana o 20 mg/kg 2x por semana para tratamientos únicos, o 10-15 mg/kg 2x por semana para tratamientos combinados.
- Dosificar las terapias combinadas, como terapias dirigidas, quimioterapias e irradiación, de acuerdo con el diseño experimental. Las terapias dirigidas y las quimioterapias se pueden administrar a través de sonda nasogástrica oral, inyección i.p. o alimentos.
NOTA: Las dosis pueden variarse y optimizarse de acuerdo con los datos publicados y el modelo tumoral. Los estudios de dosis a menudo se prueban en receptores inmunodeficientes antes de los estudios con ratones HIS.
- Controle a los ratones al menos 3 veces por semana para detectar cambios de salud como pérdida de peso, heces sueltas, postura encorvada, movilidad reducida y pérdida de pelaje. Algunos síntomas pueden ser signos de toxicidad farmacológica o EICH, y la dosificación del fármaco puede necesitar reducción o detención. Eutanasia a los ratones según sea necesario de acuerdo con las políticas de cuidado y uso de animales.
6. Recolección de tejidos y tumores de ratón al final del estudio
- Eutanasia de los ratones individualmente de acuerdo con las directrices institucionales y veterinarias utilizando gasCO2 comprimido con un caudal de 2,75 L / min. Monitorear a los ratones retenidos enCO2 durante 1 minuto después de la muerte y luego realizar la dislocación cervical como una forma secundaria de eutanasia.
- Extracción de sangre
- Recolectar sangre mediante punción intracardíaca. Sostenga el ratón en posición vertical e inserte una jeringa de 1 ml con una aguja de 25 G directamente en el corazón desde una línea justo a la izquierda de la línea media y debajo de las costillas. Recoja la sangre en una jeringa idealmente a través de una extracción larga y transfiérala a un tubo marcado de 1.5 ml.
NOTA: Se pueden realizar inserciones de agujas adicionales para recolectar sangre adicional de las esquinas de la cavidad pulmonar con cuidado. - Dejar la sangre a 4 °C durante 1 h, luego centrifugar la sangre en una microcentrífuga durante 6 min a 7.800 x g. Recoja el líquido transparente (sueros) por encima de la interfaz sanguínea en un segundo tubo limpio y etiquetado de 1,5 ml. Almacene el suero a -20 °C para análisis posteriores, como títulos de citoquinas inflamatorias o inmunoglobulinas humanas y de ratón.
- Recolectar sangre mediante punción intracardíaca. Sostenga el ratón en posición vertical e inserte una jeringa de 1 ml con una aguja de 25 G directamente en el corazón desde una línea justo a la izquierda de la línea media y debajo de las costillas. Recoja la sangre en una jeringa idealmente a través de una extracción larga y transfiérala a un tubo marcado de 1.5 ml.
- Disección de tejidos
- Después de la inyección y el crecimiento de células tumorales en los ratones y la administración de tratamientos farmacológicos, cosechar los tejidos. Coloque los ratones en una tabla de disección de espuma, con alfileres para mantenerlos en su lugar, y los brazos y las piernas extendidos en ángulos de 45 °. Haga una incisión hasta la mitad del torso, comenzando cerca de la pelvis y extendiéndose hasta la barbilla, tratando de evitar cortar el peritoneo (aunque esto no es esencial). Tire de la piel hacia el borde y manténgala en su lugar con alfileres.
- Extraiga los ganglios linfáticos (LN) utilizando fórceps finos en el siguiente orden: inguinal, axilar, cervical, mesentérico, hiatal.
NOTA: En los ratones HIS, los LN son a menudo muy pequeños, se asemejan a anlage, o "inflamados" con una apariencia distinta a la de un ratón de tipo salvaje. Por lo tanto, se toma tejido parecido a LN de cada sitio. Los LN periféricos a menudo aparecen como "bolas" llenas de líquido, mientras que los LN mesentéricos son más densos. Los LN mesentéricos son los más obvios y consistentes y se observan como uno o dos nodos más densos distintos, en oposición a una cadena. - Coloque los LN en un lado de los portaobjetos de vidrio esmerilado en 8 ml de medio de cosecha en una placa de Petri. Sosteniendo los portaobjetos en ángulos perpendiculares con los bordes esmerilados hacia adentro, presione suavemente los tejidos hasta que se libere el contenido celular.
- Enjuague los portaobjetos varias veces separándolos y juntándolos para liberar la cantidad máxima de células. Recoja las células con una pipeta de vidrio de 5 pulgadas y filtre a través de una pipeta de algodón de 9 pulgadas en un tubo cónico de 15 ml etiquetado.
- Extraiga el bazo de la parte superior izquierda del abdomen usando dos pares de fórceps o fórceps y tijeras. Tenga en cuenta el tamaño del bazo como una estimación del volumen de medios de resuspensión. Recolecte y filtre el bazo mediante digestión mecánica utilizando portaobjetos de vidrio esmerilado, como con los LN.
NOTA: Se puede utilizar cualquier técnica para la preparación de tejidos para suspensiones unicelulares. - Realice el conteo y la resuspensión de las células de las muestras de tejido como se describe a continuación.
- Centrifugar las células linfáticas a 360 x g durante 10 min a 4 °C. Aspirar el líquido y resuspender el pellet celular en 1 mL de medio de cosecha con DNasa.
- Eliminar los eritrocitos de las células del bazo incubando con 3 ml de tampón de lisis ACK en RT durante 3 min, seguido de la adición de 10 ml de medio de cosecha/DNasa. Centrifugar las células del bazo nuevamente, aspirar el sobrenadante y resuspender las células en 1-10 ml de medios de cosecha / DNasa, según el tamaño del bazo (por ejemplo, 1 ml para bazos muy pequeños y hasta 10 ml para los bazos más grandes).
- Añadir una alícuota de 10 μL de suspensión celular a 90 μL de medio y contar con un hemocitómetro. Centrifugar los LN y las células del bazo, aspirar los sobrenadantes y resuspender las células en medio de cosecha a una concentración de 1 x 108 células/ml, o un mínimo de 80 μL.
- Extraer los tumores.
- Extirpar el tumor del flanco abierto sosteniendo el tumor con fórceps mientras corta lentamente los márgenes del tumor con tijeras de disección.
- Una vez extirpado el tumor, péselo y extraiga 1/4 para el procesamiento de ARN e inmunohistoquímica (IHC). Dividir el 1/4 del tumor por la mitad; colocar la mitad (1/8 de todo el tumor) en un criovial, congelar rápidamente en líquido N2 y almacenar a -80 °C para estudios genómicos posteriores.
- Coloque el otro 1/8 del tumor en un tubo de muestra marcado que contenga 10% de formalina. Al día siguiente, enjuague y resuspenda el tejido en etanol al 70% hasta su uso futuro. Coloque los 3/4 restantes del tumor en un plato de 6 cm y pique en pedazos de ~ 1 mm con una hoja de bisturí.
- Transportar las piezas tumorales a un tubo de disociación (consulte la Tabla de materiales), enjuague el plato con 5 ml de medio leucocitario infiltrante (TIL) incompleto del tumor y agréguelo al tubo de disociación.
NOTA: Para tumores que pesen >0,4 g, enjuague el plato con 10 ml de medios incompletos TIL y agréguelos al tubo de disociación. - Agregue la preparación de colagenasa (consulte la Tabla de materiales) a una concentración final de 50 mg / ml al tejido en el tubo de disociación. Disociar el tejido mediante disociación mecánica a 37 °C durante 30 min a 1 h, dependiendo de la firmeza del tumor.
- Después de la disociación, pase la suspensión sobre un filtro de 100 μm en un tubo cónico de 50 ml y enjuague el filtro con 10 ml de medio completo TIL. El suero en este medio detendrá la reacción de la colagenasa y protegerá las células de una mayor degradación.
- Centrifugar la suspensión unicelular a 360 x g durante 10 min a 4 °C. Vuelva a suspender el pellet en el medio de cosecha suficiente con DNasa para que la suspensión celular pueda pasar fácilmente a través de una punta de pipeta P1000 y registre el volumen para el análisis posterior.
NOTA: Un volumen más pequeño aumenta la acumulación de células inmunes tumorales, pero es más susceptible a las obstrucciones del citómetro de flujo.
7. Tinción celular y análisis citométricos de flujo
- Preparación de manchas y recubrimiento celular
- Preparar cócteles de tinción: El día de la cosecha, agregue el número de muestras a la hoja de trabajo de tinción (Tabla 2; "Panel de flujo convencional A", "Panel de flujo espectral B" y "Panel de flujo C convencional") para todas las manchas e impresiones. Prepare cócteles de manchas superficiales para las manchas A y B en el tampón de tinción (SB) agregando cada anticuerpo individualmente con una nueva punta, marcando cada reactivo sobre la marcha y guárdelo a 4 °C hasta que sea necesario. Preparar los colorantes de viabilidad adecuados en PBS libre de azida y almacenar a 4 °C hasta que sea necesario (calentar a RT antes de su uso). Preparar el cóctel de tinción superficial C el día 2 en SB junto con tinciones intracelulares B y C en sus respectivos tampones de permeabilización.
- Cree un diseño de placa para todas las muestras en la hoja de trabajo de tinción y alícuota 100 μL de PBS libre de azida a los pocillos. Incluir controles no teñidos para cada tejido y tinción (para citometría espectral).
- Agregue las células a placas de 96 pocillos que contengan 50 μL de PBS. Para la tinción A, resuspender cada grupo de tejido en el volumen apropiado, como se indica en la hoja de trabajo de tinción, y almacenar a 4 °C hasta la adquisición en el citómetro de flujo el mismo día. Para las tinciones B y C, agregue 25 μL de linfa y 60 μL de suspensiones de células de tejido no linfático a los pocillos con PBS.
- Realizar estimulación in vitro de citoquinas para su detección por tinción intracelular mediante tinción C.
- Centrifugar la placa de tinción C de 96 pocillos del paso 7.1.3 a 680 x g durante 3 min a 4 °C. Agregue 200 μL de medios completos TIL (RPMI 1640, 10 mM HEPES [pH 7], 10% FBS) a cada pocillo. Conservar la placa con la suspensión celular a 4 °C durante la noche.
- Temprano a la mañana siguiente, diluya el cóctel de estimulación celular (ver Tabla de materiales) 1:500 en medios TIL completos. Centrifugar la placa que contiene la suspensión celular a 680 x g para granular las células y mover para extraer el medio. Resuspender las células en 200 μL del cóctel de estimulación celular preparado e incubar a 37 °C durante 1 h.
- Añadir 25 μL de solución inhibidora del transporte de proteínas que contenga monensina en una dilución 1:1.000 en medios TIL completos, mezclar las células e incubar a 37 °C durante 4 h adicionales para permitir la acumulación intracelular de citoquinas.
- Realizar tinción celular.
- Para las tinciones A y B (día 1), y para la tinción C (día 2), centrifugar a 680 x g durante 3 min a 4 °C. Mueva las placas y agregue los tintes de viabilidad apropiados a los pocillos. Mezclar bien pipeteando suavemente hacia arriba y hacia abajo con una pipeta multicanal e incubar durante 15 min a RT.
- Centrifugar las placas (como en el paso 7.1.5.1), añadir manchas superficiales adecuadas a las placas y mezclar suavemente pipeteando con una pipeta multicanal. Incubar durante 15 min a 4 °C y centrifugar de nuevo. Mueva las placas, lave las células pipeteando suavemente hacia arriba y hacia abajo con 150 μL de SB y centrifugara.
- Agitar las placas, repetir el lavado pipeteando suavemente hacia arriba y hacia abajo con 150 μL de SB y centrifugar a 680 x g durante 3 min a 4 °C. Para la tinción A, resuspender cada grupo de tejido en el volumen apropiado, como se indica en la hoja de trabajo de tinción, y almacenar a 4 °C. Para la tinción B, fijar con el kit de factor de transcripción FoxP3 fijador (ver Tabla de materiales) durante 30 min en RT. Para la tinción C, fijar en paraformaldehído al 1% (v/v) en SB durante 30 min a RT.
- Centrifugar las placas fijas a 480 x g durante 3 min a 4 °C. Lavar 1x en SB. Para la tinción B, las células se pueden dejar durante la noche.
- Permeabilizar las células: resuspender los pocillos en 150 μL de tampón de permeabilización del kit de factor de transcripción FoxP3 para la tinción B y en saponina al 0,5% (p/v) en SB para la tinción C. Incubar durante 15 min a RT. Centrifugar las placas fijas a 480 x g durante 3 min a 4 °C.
- Mueva las placas y agregue los respectivos cócteles de tinción intracelular para la tinción B y la tinción C. Incubar durante 30 minutos en RT.
- Centrifugar las placas fijas a 480 x g durante 3 min a 4 °C y mover las placas. Lavar las células con 150 μL de los tampones de permeabilización correspondientes (tinción B, tampón de permeabilización del kit del factor de transcripción FoxP3; tinción C, saponina).
- Agitar las placas y lavar pipeteando hacia arriba y hacia abajo en 150 μL de SB. Centrifugar a 480 x g durante 3 min a 4 °C. Mueva las placas y vuelva a suspender las células en SB por grupo de tejidos en los volúmenes apropiados que se indican en la hoja de trabajo de tinción.
NOTA: Las muestras ya están listas para su adquisición por citometría de flujo espectral. - Configure el citómetro de flujo (consulte la Tabla de materiales) con controles teñidos individuales apropiados para cada tinción y muestras no teñidas para cada grupo de tejidos para tener en cuenta las diferencias en la autofluorescencia durante la mezcla. Adquiera los volúmenes apropiados por grupo de tejidos como se define en la hoja de trabajo de tinción (Tabla 2) y exporte los archivos .fcs.
- Análisis de datos de citometría de flujo
- Con el software de análisis por citometría de flujo (consulte Tabla de materiales), cree un nuevo espacio de trabajo. Crear nuevos grupos para cada órgano (LN, bazo y TIL). Importe los archivos .fcs de cada órgano en el grupo.
- Cree un diagrama de puntos bivariado, establezca los ejes en (FSC-A x SSC-A) y aplique una puerta poligonal a los eventos celulares, evitando eventos en los bordes (puerta de todas las celdas). Seleccione los eventos celulares, cambie los ejes a (FSC-A x FSC-H) y aplique una puerta poligonal a los eventos en la diagonal lineal, excluyendo los eventos que se desvían de la diagonal para generar la puerta "singlets". Seleccione la puerta "singlets" y cambie los ejes a (hCD45 x mCD45). Aplique puertas de polígono a los eventos positivos hCD45 y mCD45, y asígneles el nombre "humano" y "ratón", respectivamente.
- Seleccione la población humana y cambie el eje Y a Live/Dead Aqua. Aplique una puerta de polígono a la población positiva hCD45 negativa / muerta y asígnele el nombre "humano vivo". Seleccione la población del ratón y cambie el eje X a Live/Dead Aqua. Aplique una puerta de polígono a la población Live/Dead negative, mCD45 positiva y asígnele el nombre "ratón vivo".
- De manera similar, seleccione la puerta principal y los ejes X e Y como se describe en las hojas de trabajo "Activación de flujo A convencional", "Activación de flujo espectral B" y "Activación de flujo C convencional" (Tabla 2) para aislar las poblaciones indicadas (por ejemplo, células B humanas, células T activadas).
NOTA: La activación representativa para cada mancha (Sangrado, A, B, C) se incluye en el Archivo Suplementario 2. - Cree tablas de exportación de recuento y frecuencia en el software de análisis de citometría de flujo para todas las poblaciones y exporte a un software de hoja de cálculo. La población parental para las frecuencias se indica en la Tabla 2.
- Utilice los datos para generar gráficos basados en grupos de tratamiento experimental.
NOTA: Los datos también se pueden analizar utilizando paquetes R en el software de análisis. Se puede crear un único archivo .fcs de todas las muestras que se van a analizar utilizando la función de concatenación. Estos datos se pueden reducir dimensionalmente con el algoritmo T-SNE mientras se agregan parámetros de palabras clave para el tipo de tejido y el grupo de tratamiento. El algoritmo FlowSOM se puede usar para agrupar poblaciones y la herramienta ClusterExplorer se puede usar para identificar las poblaciones. Las nuevas poblaciones celulares pueden identificarse de esta manera, compararse visualmente y cuantificarse entre grupos de tratamiento o dentro de varios tejidos. - Correlacione los parámetros inmunitarios para los tumores dentro del mismo grupo de tratamiento, con el crecimiento de ese tumor para definir los inmunotipos que se correlacionan con la inhibición del crecimiento tumoral. Cuantificar el crecimiento del tumor por la tasa de crecimiento específico (SGR) para ese tumor, una medida que tiene en cuenta la diferencia en los volúmenes del tumor durante un tiempo específico. Esta medida normaliza los tumores cosechados en diferentes días debido a la salud del ratón y las fechas de inicio del tratamiento.
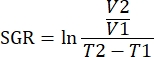
Resultados
Siguiendo el protocolo del tumor del flanco y la línea de tiempo experimental (Figura 1), se estudió el crecimiento tumoral y la respuesta inmunitaria a una terapia dirigida con inhibidores de la tirosina cinasa (TKI) y al tratamiento combinado con nivolumab en dos PDX distintas de cáncer colorrectal humano (CCR). Los fármacos TKI han sido estudiados en hospedadores inmunodeficientes para evaluar el crecimiento tumoral sólo29. Este modelo permitió el estudio de ...
Discusión
En los últimos 6 años, utilizando nuestra experiencia tanto en inmunología como en ratones humanizados, nuestro equipo de investigación ha desarrollado un modelo preclínico muy necesario para probar inmunoterapias en una variedad de tumores humanos 3,7,30,31. Este protocolo enfatiza la consideración de la variabilidad del modelo, con especial atención a las poblaciones de células T huma...
Divulgaciones
Ninguno.
Agradecimientos
Nos gustaría agradecer tanto al Centro de Investigación Animal (OLAR) por su cuidado de nuestros ratones, como al Recurso Compartido de Citometría de Flujo apoyado por la Subvención de Apoyo al Centro de Cáncer (P30CA046934) en nuestro instituto por su inmensa ayuda en todo nuestro trabajo. También agradecemos tanto a Gail Eckhardt como a Anna Capasso por nuestras colaboraciones inaugurales en el estudio de inmunoterapias para PDX humanos en nuestro modelo HIS-BRGS. Este estudio fue apoyado en parte por la Subvención de Apoyo al Centro de Cáncer P30CA06934 de los Institutos Nacionales de Salud con el uso del recurso compartido PHISM (Pre-clinical Human Immune System Mouse Models), RRID: SCR_021990 y Flow Cytometry Shared Resource, RRID: SCR_022035. Esta investigación fue apoyada en parte por el NIAID de los Institutos Nacionales de Salud bajo el número de contrato 75N93020C00058.
Materiales
| Name | Company | Catalog Number | Comments |
| 1 mL syringe w/needles | McKesson | 1031815 | |
| 15 mL tubes | Grenier Bio-One | 188271 | |
| 2-mercaptoethanol | Sigma | M6250 | |
| 50 mL tubes | Grenier Bio-One | 227261 | |
| AutoMACS Pro Separator | Miltenyi | 130-092-545 | |
| BD Golgi Stop Protein Transport Inhibitor with monensin | BD Bioscience | BDB563792 | |
| BSA | Fisher Scientific | BP1600100 | |
| Cell Stim Cocktail | Life Technologies | 509305 | |
| Chill 15 Rack | Miltenyi | 130-092-952 | |
| Cotton-plugged glass pipettes | Fisher Scientific | 13-678-8B | |
| Cultrex Basement membrane extract | R&D Systems | 363200502 | |
| Cytek Aurora | Cytek | ||
| DNase | Sigma | 9003-98-9 | |
| eBioscience FoxP3/Transcription Factor Staining Buffer Set | Invitrogen | 00-5523-00 | |
| Embryonic Stemcell FCS | Gibco | 10439001 | |
| Eppendorf Tubes; 1.5 mL volume | Grenier Bio-One | 616201 | |
| Excel | Microsoft | ||
| FBS | Benchmark | 100-106 500mL | |
| Ficoll Hypaque | GE Healthcare | 45001752 | |
| FlowJo Software | BD Biosciences | ||
| Forceps - fine | Roboz Surgical | RS5045 | |
| Forceps normal | Dumont | RS4919 | |
| Formaldehyde | Fisher | F75P1GAL | |
| Frosted Glass Slides | Corning | 1255310 | |
| Gentlemacs C-Tubes | Miltenyi | 130-096-334 | |
| GentleMACS Dissociator | Miltenyi | 130-093-235 | |
| glass pipettes | DWK Life Sciences | 63A53 | |
| Glutamax | Gibco | 11140050 | |
| HBSS w/ Ca & Mg | Sigma | 55037C | |
| HEPES | Corning | MT25060CI | |
| IgG standard | Sigma | I2511 | |
| IgM standard | Sigma | 401108 | |
| IMDM | Gibco | 12440053 | |
| Liberase DL | Roche | 5466202001 | |
| LIVE/DEAD Fixable Blue | Thermo | L23105 | |
| MDA-MB-231 | ATCC | HTB-26 | |
| MEM | Gibco | 1140050 | |
| mouse anti-human IgG-AP | Southern Biotech | JDC-10 | |
| mouse anti-human IgG-unabeled | Southern Biotech | H2 | |
| mouse anti-human IgM-AP | Southern Biotech | UHB | |
| mouse anti-human IgM-unlabeled | Southern Biotech | SA-DA4 | |
| MultiRad 350 | Precision X-Ray | ||
| PBS | Corning | 45000-446 | |
| Pen Strep | Gibco | 15140122 | |
| Petri Dishes | Fisher Scientific | FB0875713A | |
| p-nitrophenyl substrate | Thermo | 34045 | |
| PRISM | Graphpad | ||
| Rec Hu FLT3L | R&D systems | 308-FK-005/CF | |
| Rec Hu IL6 | R&D systems | 206-IL-010/CF | |
| Rec Hu SCF | R&D systems | 255SC010 | |
| RPMI 1640 | Corning | 45000-39 | |
| Saponin | Sigma | 8047-15-2 | |
| Scissors | McKesson | 862945 | |
| Serological pipettes 25 mL | Fisher Scientific | 1367811 | |
| Sterile filter | Nalgene | 567-0020 | |
| Sterile molecular water | Sigma | 7732-18-5 | |
| Yeti Cell Analyzer | Bio-Rad | 12004279 | |
| Zombie Green | biolegend | 423112 |
Referencias
- Chulpanova, D. S., Kitaeva, K. V., Rutland, C. S., Rizvanov, A. A., Solovyeva, V. V. Mouse tumor models for advanced cancer immunotherapy. International Journal of Molecular Sciences. 21 (11), 4118 (2020).
- Olson, B., Li, Y., Lin, Y., Liu, E. T., Patnaik, A. Mouse models for cancer immunotherapy research. Cancer Discovery. 8 (11), 1358-1365 (2018).
- Marin-Jimenez, J. A., et al. Testing cancer immunotherapy in a human immune system mouse model: correlating treatment responses to human chimerism, therapeutic variables and immune cell phenotypes. Frontiers in Immunology. 12, 607282 (2021).
- Yin, L., Wang, X. J., Chen, D. X., Liu, X. N., Wang, X. J. Humanized mouse model: a review on preclinical applications for cancer immunotherapy. American Journal of Cancer Research. 10 (12), 4568-4584 (2020).
- Cogels, M. M., et al. Humanized mice as a valuable pre-clinical model for cancer immunotherapy research. Frontiers in Oncology. 11, 784947 (2021).
- Jin, K. T., et al. Development of humanized mouse with patient-derived xenografts for cancer immunotherapy studies: A comprehensive review. Cancer Science. 112 (7), 2592-2606 (2021).
- Capasso, A., et al. Characterization of immune responses to anti-PD-1 mono and combination immunotherapy in hematopoietic humanized mice implanted with tumor xenografts. Journal for Immunotherapy of Cancer. 7 (1), 37 (2019).
- Wang, M., et al. Humanized mice in studying efficacy and mechanisms of PD-1-targeted cancer immunotherapy. The FASEB Journal. 32 (3), 1537-1549 (2018).
- Yong, K. S. M., et al. Humanized mouse as a tool to predict immunotoxicity of human biologics. Frontiers in Immunology. 11, 553362 (2020).
- Shen, H. W., Jiang, X. L., Gonzalez, F. J., Yu, A. M. Humanized transgenic mouse models for drug metabolism and pharmacokinetic research. Current Drug Metabolism. 12 (10), 997-1006 (2011).
- Bosma, G. C., Custer, R. P., Bosma, M. J. A severe combined immunodeficiency mutation in the mouse. Nature. 301 (5900), 527-530 (1983).
- Shultz, L. D., et al. Multiple defects in innate and adaptive immunologic function in NOD/LtSz-scid mice. The Journal of Immunology. 154 (1), 180-191 (1995).
- Legrand, N., et al. Functional CD47/signal regulatory protein alpha (SIRP(alpha)) interaction is required for optimal human T- and natural killer- (NK) cell homeostasis in vivo. Proceedings of the National Academy of Sciences. 108 (32), 13224-13229 (2011).
- Ishikawa, F., et al. Development of functional human blood and immune systems in NOD/SCID/IL2 receptor {gamma} chain(null) mice. Blood. 106 (5), 1565-1573 (2005).
- Ito, M., et al. NOD/SCID/gamma(c)(null) mouse: an excellent recipient mouse model for engraftment of human cells. Blood. 100 (9), 3175-3182 (2002).
- Shultz, L. D., et al. Human lymphoid and myeloid cell development in NOD/LtSz-scid IL2R gamma null mice engrafted with mobilized human hemopoietic stem cells. The Journal of Immunology. 174 (10), 6477-6489 (2005).
- Traggiai, E., et al. Development of a human adaptive immune system in cord blood cell-transplanted mice. Science. 304 (5667), 104-107 (2004).
- Theocharides, A. P., Rongvaux, A., Fritsch, K., Flavell, R. A., Manz, M. G. Humanized hemato-lymphoid system mice. Haematologica. 101 (1), 5-19 (2016).
- Goldman, J. P., et al. Enhanced human cell engraftment in mice deficient in RAG2 and the common cytokine receptor gamma chain. British Journal of Haematology. 103 (2), 335-342 (1998).
- Stripecke, R., et al. Innovations, challenges, and minimal information for standardization of humanized mice. EMBO Molecular Medicine. 12 (7), (2020).
- Allen, T. M., et al. Humanized immune system mouse models: progress, challenges and opportunities. Nature Immunology. 20 (7), 770-774 (2019).
- Gammelgaard, O. L., Terp, M. G., Preiss, B., Ditzel, H. J. Human cancer evolution in the context of a human immune system in mice. Molecular Oncology. 12 (10), 1797-1810 (2018).
- Rios-Doria, J., Stevens, C., Maddage, C., Lasky, K., Koblish, H. K. Characterization of human cancer xenografts in humanized mice. Journal for Immunotherapy of Cancer. 8 (1), 000416 (2020).
- Gombash Lampe, S. E., Kaspar, B. K., Foust, K. D. Intravenous injections in neonatal mice. Journal of Visualized Experiments. (93), e52037 (2014).
- Lang, J., Weiss, N., Freed, B. M., Torres, R. M., Pelanda, R. Generation of hematopoietic humanized mice in the newborn BALB/c-Rag2null Il2rγnull mouse model: a multivariable optimization approach. Clinical Immunology. 140 (1), 102-116 (2011).
- Laskowski, T. J., Hazen, A. L., Collazo, R. S., Haviland, D. Rigor and reproducibility of cytometry practices for immuno-oncology: a multifaceted challenge. Cytometry Part A. 97 (2), 116-125 (2020).
- Bagby, S., et al. Development and maintenance of a preclinical patient derived tumor xenograft model for the investigation of novel anti-cancer therapies. Journal of Visualized Experiments. (115), e54393 (2016).
- Laajala, T. D., et al. Optimized design and analysis of preclinical intervention studies in vivo. Scientific Reports. 6, 30723 (2016).
- Na, Y. S., et al. Establishment of patient-derived xenografts from patients with gastrointestinal stromal tumors: analysis of clinicopathological characteristics related to engraftment success. Scientific Reports. 10 (1), 7996 (2020).
- Tentler, J. J., et al. RX-5902, a novel beta-catenin modulator, potentiates the efficacy of immune checkpoint inhibitors in preclinical models of triple-negative breast cancer. BMC Cancer. 20 (1), 1063 (2020).
- Lang, J., et al. Development of an adrenocortical cancer humanized mouse model to characterize anti-PD1 effects on tumor microenvironment. The Journal of Clinical Endocrinology & Metabolism. 105 (1), 26-42 (2020).
- Lang, J., et al. Studies of lymphocyte reconstitution in a humanized mouse model reveal a requirement of T cells for human B cell maturation. The Journal of Immunology. 190 (5), 2090-2101 (2013).
- Katano, I., et al. NOD-Rag2null IL-2Rγnull mice: an alternative to NOG mice for generation of humanized mice. Experimental Animalas. 63 (3), 321-330 (2014).
- Brehm, M. A., et al. Parameters for establishing humanized mouse models to study human immunity: analysis of human hematopoietic stem cell engraftment in three immunodeficient strains of mice bearing the IL2rγ(null) mutation. Clinical Immunology. 135 (1), 84-98 (2010).
- Hasgur, S., Aryee, K. E., Shultz, L. D., Greiner, D. L., Brehm, M. A. Generation of immunodeficient mice bearing human immune systems by the engraftment of hematopoietic stem cells. Methods in Molecular Biology. 1438, 67-78 (2016).
- Andre, M. C., et al. Long-term human CD34+ stem cell-engrafted nonobese diabetic/SCID/IL-2Rγnull mice show impaired CD8+ T cell maintenance and a functional arrest of immature NK cells. The Journal of Immunology. 185 (5), 2710-2720 (2010).
- Wunderlich, M., et al. Improved multilineage human hematopoietic reconstitution and function in NSGS mice. PLoS One. 13 (12), 0209034 (2018).
- Lee, J., Brehm, M. A., Greiner, D., Shultz, L. D., Kornfeld, H. Engrafted human cells generate adaptive immune responses to Mycobacterium bovis BCG infection in humanized mice. BMC Immunology. 14, 53 (2013).
- Masse-Ranson, G., et al. Accelerated thymopoiesis and improved T-cell responses in HLA-A2/-DR2 transgenic BRGS-based human immune system mice. European Journal of Immunology. 49 (6), 954-965 (2019).
- Oswald, E., et al. Immune cell infiltration pattern in non-small cell lung cancer PDX models is a model immanent feature and correlates with a distinct molecular and phenotypic make-up. Journal for Immunotherapy of Cancer. 10 (4), 004412 (2022).
Erratum
Formal Correction: Erratum: Testing Cancer Immunotherapeutics in a Humanized Mouse Model Bearing Human Tumors
Posted by JoVE Editors on 5/25/2023. Citeable Link.
An erratum was issued for: Testing Cancer Immunotherapeutics in a Humanized Mouse Model Bearing Human Tumors. The Authors section was updated from:
Jordi M. Lanis1
Matthew S. Lewis1
Hannah Strassburger1
Stacey M. Bagby2
Adrian T. A. Dominguez2
Juan A. Marín-Jiménez3
Roberta Pelanda1
Todd M. Pitts2
Julie Lang1
1Department of Immunology and Microbiology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
2Division of Oncology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
3Department of Medical Oncology, Catalan Institute of Oncology (ICO-L’Hospitalet)
to:
Jordi M. Lanis1
Matthew S. Lewis1
Hannah Strassburger1
Kristina Larsen1
Stacey M. Bagby2
Adrian T. A. Dominguez2
Juan A. Marín-Jiménez3
Roberta Pelanda1
Todd M. Pitts2
Julie Lang1
1Department of Immunology and Microbiology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
2Division of Oncology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
3Department of Medical Oncology, Catalan Institute of Oncology (ICO-L’Hospitalet)
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados