È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Test di immunoterapie del cancro in un modello murino umanizzato che porta tumori umani
* Questi autori hanno contribuito in egual misura
In questo articolo
Erratum Notice
Riepilogo
Questo protocollo delinea la generazione di topi del sistema immunitario umano (HIS) per studi immuno-oncologici. Le istruzioni e le considerazioni nell'uso di questo modello per testare immunoterapici umani su tumori umani impiantati in questo modello sono presentate con un'enfasi sulla caratterizzazione della risposta del sistema immunitario umano al tumore.
Abstract
Invertire la natura immunosoppressiva del microambiente tumorale è fondamentale per il successo del trattamento dei tumori con farmaci immunoterapici. I modelli di cancro murino sono estremamente limitati nella loro diversità e soffrono di scarsa traduzione in clinica. Per servire come modello preclinico più fisiologico per gli studi di immunoterapia, questo protocollo è stato sviluppato per valutare il trattamento dei tumori umani in un topo ricostituito con un sistema immunitario umano. Questo protocollo unico dimostra lo sviluppo di topi del sistema immunitario umano (HIS, "umanizzati"), seguito dall'impianto di un tumore umano, sia uno xenotrapianto derivato da linea cellulare (CDX) o uno xenotrapianto derivato dal paziente (PDX). I topi HIS sono generati iniettando cellule staminali ematopoietiche umane CD34 + isolate dal sangue del cordone ombelicale in topi neonatali BRGS (BALB / c Rag2-/- IL2RγC-/- NODSIRPα) altamente immunodeficienti che sono anche in grado di accettare un tumore xenogenico. Viene sottolineata l'importanza della cinetica e delle caratteristiche dello sviluppo del sistema immunitario umano e dell'impianto del tumore. Infine, viene descritta una valutazione approfondita del microambiente tumorale mediante citometria a flusso. In numerosi studi che utilizzano questo protocollo, è stato riscontrato che il microambiente tumorale dei singoli tumori è ricapitolato nei topi HIS-PDX; I tumori "caldi" mostrano una grande infiltrazione immunitaria mentre i tumori "freddi" no. Questo modello funge da banco di prova per le immunoterapie combinate per una vasta gamma di tumori umani e rappresenta uno strumento importante nella ricerca di una medicina personalizzata.
Introduzione
I modelli di cancro del topo sono importanti per stabilire i meccanismi di base della crescita tumorale e della fuga immunitaria. Tuttavia, gli studi sul trattamento del cancro in modelli murini hanno prodotto una traduzione finita alla clinica a causa dei limitati modelli singeneici e delle differenze specie-specifiche 1,2. L'emergere di terapie immunitarie come approccio dominante per controllare i tumori ha ribadito la necessità di un modello in vivo con un sistema immunitario umano funzionale. I progressi nei topi del sistema immunitario umano (topi HIS) negli ultimi dieci anni hanno reso possibile studiare l'immuno-oncologia in vivo in un'ampia varietà di tipi di cancro e agenti immunoterapeutici 3,4,5,6. I modelli tumorali umani, compresi gli xenotrapianti derivati da linee cellulari e derivati dal paziente (CDX e PDX, rispettivamente), crescono bene nei topi HIS e nella maggior parte dei casi sono quasi identici alla loro crescita nell'ospite immunodeficiente privo di attecchimento ematopoietico umano 7,8. Sulla base di questa scoperta chiave, i ricercatori hanno utilizzato il modello murino HIS per studiare le immunoterapie umane, comprese le terapie combinate progettate per alterare il microambiente tumorale (TME) per ridurre l'immunosoppressione e quindi migliorare l'uccisione del tumore diretta dal sistema immunitario. Questi modelli preclinici aiutano ad affrontare i problemi di eterogeneità dei tumori umani e possono anche prevedere il successo del trattamento e monitorare la tossicità dei farmaci correlati al sistema immunitario 9,10.
La produzione di un modello murino con un sistema immunitario umano attraverso l'introduzione di cellule staminali ematopoietiche umane richiede un topo immunodeficiente ricevente che non respinga lo xenotrapianto. Gli attuali modelli murini HIS derivano da ceppi murini immunodeficienti che sono stati segnalati oltre 30 anni fa. Il primo ceppo di topo immunodeficiente descritto erano topi SCID privi di cellule T e B11, seguiti da un ibrido NOD-SCID con un polimorfismo SIRPα responsabile della tolleranza dei macrofagi di topo alle cellule umane, a causa dell'aumento del legame per l'allele NOD SIRPα alla molecola umana CD4712,13. Nei primi anni 2000, la delezione della catena gamma comune del recettore IL-2 (IL-2Rγc) su entrambi i ceppi immunodeficienti BALB / c e NOD è stato un punto di svolta per l'attecchimento umano potenziato, a causa delle delezioni genetiche che vietano lo sviluppo delle cellule NK dell'ospite14,15,16,17. Modelli alternativi, come i topi BRG e NRG, raggiungono il deficit delle cellule T e B attraverso la delezione del gene Rag1 o Rag2, richiesto per i riarrangiamenti genici dei recettori delle cellule T e B e quindi la maturazione e la sopravvivenza dei linfociti18,19. Il topo BRGS (BALB/c -Rag2 nullIl2RγCnullSirpα NOD) qui utilizzato combina il deficit della catena IL-2Rγ e l'alleleNOD SIRPα sullo sfondo Rag2-/-, risultando in un topo altamente immunodeficiente senza cellule T, B o NK, ma con sufficiente vigore e salute per consentire un attecchimento a lungo termine di oltre 30 settimane13.
I topi HIS possono essere generati in diversi modi, con l'iniezione umana di PBMC che è il metodo più diretto15,18,20. Tuttavia, questi topi hanno una pronunciata espansione delle cellule T umane attivate che si traduce in malattia del trapianto contro l'ospite (GVHD) entro le 12 settimane di età, impedendo studi a lungo termine. In alternativa, le cellule staminali ematopoietiche umane dal sangue del cordone ombelicale (CB), dal midollo osseo e dal fegato fetale possono anche essere utilizzate per l'attecchimento e la produzione del sistema immunitario umano de novo. In questo sistema, le cellule staminali ematopoietiche producono un sistema immunitario umano multi-lignaggio con la generazione di T, B e cellule immunitarie innate che sono significativamente tolleranti dell'ospite del topo, rispetto ai topi PBMC che sviluppano principalmente cellule T. Pertanto, la GVHD è assente o molto ritardata e gli studi possono essere estesi a topi fino a 10 mesi di età. CB fornisce una fonte facile, accessibile e non invasiva di cellule staminali ematopoietiche umane CD34+ che facilita l'attecchimento di più topi HIS con sistemi immunitari geneticamente identici 17,18,20,21. Negli ultimi anni, i modelli murini HIS sono stati ampiamente utilizzati per studiare l'immunoterapia e la TME 3,4,5,6. Nonostante lo sviluppo di sistemi immunitari di derivazione umana in questi topi, i tumori dello xenotrapianto umano crescono a tassi simili rispetto ai topi immunodeficienti di controllo e consentono la complessa interazione tra le cellule tumorali e le cellule immunitarie, che è importante per mantenere il microambiente delPDX 3,7,8 innestato . Questo protocollo è stato utilizzato per eseguire oltre 50 studi testando trattamenti in topi HIS-BRGS con PDX e CDX. Una conclusione importante è che i tumori umani nei topi HIS mantengono la loro unica TME come definito dalla valutazione molecolare del tumore rispetto al campione iniziale del paziente e alle caratteristiche dell'infiltrato immunitario 3,22,23. Il nostro gruppo si concentra sulla valutazione approfondita dell'HIS sia negli organi immunitari che nel tumore utilizzando la citometria a flusso multiparametrica. Qui, descriviamo un protocollo per l'umanizzazione dei topi BRGS, la valutazione del chimerismo, l'impianto di tumori umani, le misurazioni della crescita tumorale, la somministrazione del trattamento del cancro e l'analisi delle cellule HIS mediante citometria a flusso.
Protocollo
Tutto il lavoro sugli animali è stato eseguito secondo protocolli animali approvati dal Comitato istituzionale per la cura e l'uso degli animali dell'Università del Colorado Denver (protocolli IACUC # 00593 e # 00021). Tutto il lavoro sugli animali è stato eseguito in conformità con l'Office of Laboratory Animal Resources (OLAR), una struttura accreditata dall'American Association for Laboratory Animal Care, presso l'Università del Colorado Denver Anschutz Medical Campus. Tutti i campioni di sangue del cordone ombelicale umano sono stati ottenuti come donazioni da donatori anonimizzati e non sono quindi soggetti all'approvazione del comitato etico per la ricerca umana.
NOTA: le composizioni di tutti i supporti e le soluzioni menzionate nel protocollo sono incluse nel file supplementare 1. La Figura 1 illustra il protocollo generale per la generazione e l'analisi delle risposte immunitarie ai tumori nei topi HIS-BRGS.
1. Generazione di topi HIS
- Allevamento di topi BALB/c -Rag2 null Il2RγCnull Sirpα NOD (BRGS)
NOTA: Questo ceppo è estremamente immunodeficiente, senza cellule T, B o NK. Pertanto, devono essere utilizzate misure rigorose per prevenire infezioni opportunistiche. Mantenere la colonia su una dieta contenente trimetoprim e sulfadiazina su un programma di 2 settimane alternate con una dieta normale. Mantenere il massimo livello di precauzione possibile per l'alloggiamento (ad esempio, una struttura di doccia a barriera con accesso limitato).- Mantenere colonie di topi omozigoti BALB/c -Rag2 null Il2RγC null Sirpα NOD (BRGS) e BALB/c Rag2 null Il2RγC null SirpaBalb/c (BRG) come riproduttori.
- Razza BRGS N/N×BRG B/B per generare cuccioli BRGSB/N, da utilizzare come riceventi di cellule staminali umane. In questa colonia, i BRGSB/N sono più sani dei BRGS N/N e si attecchiscono a livelli equivalenti (più del BRG).
- Isolamento di cellule staminali umane CD34+ da CB ombelicale
NOTA: Non vengono utilizzati antibiotici per questa procedura. Pertanto, una buona tecnica sterile è imperativa.- Posizionare un rack per tubi conici da 50 mL, nonché 3 tubi conici da 15 ml e ~ 10 x 50 ml in un armadio di biosicurezza sterilizzato (BSC). Spruzzare una sacca per la raccolta del sangue con etanolo al 70% e lasciarla asciugare nella BSC.
- Calcolare il numero di tubi conici da 50 mL necessari per l'isolamento del gradiente di densità CB = volume CB/15, arrotondato per eccesso e ad un numero pari. Calcolare il volume del sangue per provetta = volume CB / numero di provette. Versare con cura il sangue dalla sacca CB in ciascun tubo conico; questo è un massimo di 15 ml per tubo. Utilizzare un pipettor automatico e una pipetta sierologica da 25 ml per mescolare il sangue 1:1 con PBS sterile mediante pipettaggio su e giù.
- Utilizzare un pipettor automatico a bassa velocità e una pipetta sierologica da 10 mL per coprire lentamente il sangue con una soluzione a gradiente di densità di 1,077 g/mL a temperatura ambiente (RT) (vedere Tabella dei materiali) senza disturbare l'interfaccia. Evitare che la punta della pipetta tocchi il fondo del tubo. Ripetere l'operazione per tutti i tubi. Quindi centrifugare per 30 minuti a 850 x g, senza frenare, a RT per garantire il mantenimento del gradiente di densità.
- Visualizza il buffy coat cellulare sopra il gradiente di densità di 1,077 g / mL come uno strato bianco nuvoloso. Rimuovere ed eliminare lo strato di plasma fino a circa 10 ml sopra il buffy coat utilizzando una pipetta sierologica da 25 ml e un pipettatore automatico.
- Raccogliere il buffy coat con un trasferimento sterile o una pipetta sierologica. Utilizzare la pipetta come una spatola per raschiare le cellule dal lato del tubo conico mentre si rilascia il bulbo (o pipettaggio lentamente) per aspirare le cellule. Unire i buffy coat di due tubi conici da 50 mL in un nuovo tubo conico da 50 ml.
- Lavare le cellule versando 45 ml di HBSS sterile contenente il 2% di FBS in ciascun tubo conico. Centrifugare per 11 minuti a 360 x g a RT.
- Aspirare il mezzo di lavaggio fino al pellet in tutti i tubi. Utilizzare una pipetta sierologica da 10 mL e un pipettor automatico per risospendere il primo pellet in 10 mL di HBSS contenente il 2% di FBS. Risospendere ogni pellet negli stessi 10 ml di HBSS e sciacquare ogni tubo con altri 10 ml di HBSS per raccogliere tutte le cellule in un singolo tubo.
- Versare 45 mL di HBSS sterile contenente il 2% di FBS nel tubo conico. Centrifugare per 10 min a 360 x g, a 4 °C.
- Aspirare il tampone di lavaggio fino al pellet cellulare e risospendere il pellet in 20 ml di tampone separatore magnetico (vedere Tabella dei materiali). Rimuovere una piccola aliquota per contare le cellule con un emocitometro ad una diluizione 1:20 in blu di metilene. Aggiungere il numero di celle blu e bianche. Centrifugare a 360 x g, per 10 minuti a 4 °C.
NOTA: questo protocollo utilizza la tecnologia delle perline magnetiche (vedere la tabella dei materiali). Il protocollo può essere modificato per l'uso con qualsiasi tecnologia di separazione cellulare, con sufficiente purezza e resa delle cellule staminali CD34+. - Aspirare il surnatante e risospendere il pellet cellulare CD34+ isolato dal sangue del cordone ombelicale in 300 μL di tampone separatore magnetico per 1 x 108 cellule. Aggiungere prima 100 μL di reagente bloccante FcR e poi 100 μL di sfere magnetiche CD34+ per 1 x 108 celle. Incubare a 4 °C per 30 minuti (senza ghiaccio).
- Aggiungere 5 ml di tampone separatore magnetico per 1 x 108 celle e ruotare a 360 x g per 10 minuti a 4 °C. Ripetere la fase di lavaggio e risospendere il pellet in 500 μL di tampone separatore magnetico per 1 x 108 celle in un tubo conico da 15 mL etichettato come "non frazionato".
- Etichettare altri due tubi conici da 15 mL "CD34-" e "CD34+". Posizionare i tre tubi conici da 15 mL (non frazionati, CD34- e CD34+) negli slot A1, B1 e C1, rispettivamente, su un rack di raffreddamento (vedere la tabella dei materiali). Separare le celle utilizzando il programma di selezione positiva a due colonne su un separatore di celle magnetico automatico (vedere la tabella dei materiali) in un BSC, secondo le istruzioni dello strumento del produttore.
- Espansione e congelamento delle cellule staminali umane CD34+
- Aliquot 10 μL della sospensione cellulare CD34+ recuperata (2 ml) su un vetrino emocitometrico e contare le cellule sotto ingrandimento 10x. Calcolare il numero totale di cellule CD34+ moltiplicando il conteggio delle cellule per 2 x 104. Dividere il numero totale di cellule CD34+ per 250.000 per calcolare il numero di fiale da congelare (50.000 cellule per cucciolo di topo prima dell'espansione in vitro ).
- Preparare il mezzo CB FCS al 10% di Iscove (più 1 mL di perdita extraper filtrante), integrato con 40 ng/mL di fattore di cellule staminali, 20 ng/mL Flt3L e 10 ng/mL IL-6, e passare attraverso un filtro da 0,22 μm. Risospendere le cellule CD34+ a 100.000 per ml di terreno CB e incubare a 37 °C. Il giorno 3, aggiungere un volume equivalente di mezzo CB senza citochine al matraccio contenente le cellule e al mezzo CB con citochine.
NOTA: L'aggiunta di queste citochine al mezzo CB promuove la sopravvivenza e l'espansione delle cellule CD34+ prevenendo la differenziazione. - Raccogliere le cellule CD34+ espanse il giorno 5. Pipettare la sospensione cellulare su e giù e raccogliere in un tubo conico da 50 ml. Aggiungere una quantità di CB media sufficiente a coprire il fondo del pallone. Usando un raschietto cellulare, raschiare l'intero fondo del pallone. Raccogliere tutti i fluidi nello stesso tubo da 50 ml e centrifugare a 360 x g per 11 minuti.
- Risospendere le cellule in 2 mL di terreno CB. Salvare la goccia finale dalla pipetta in una piastra da 96 pozzetti per il conteggio. Diluire le cellule 1:1 in blu tripano e aggiungere 10 μL all'ematocitometro, quindi contare e calcolare le cellule medie da quattro quadranti. Calcola il numero totale di cellule CD34+ moltiplicando il conteggio delle cellule per 4 x 104 e registra la vitalità.
- Fabbricare n+1 mL di mezzo di congelamento, dove n è il numero di flaconcini di congelamento calcolato al punto 1.3.1. Preparare il mezzo di congelamento aggiungendo il 10% (v/v) di DMSO a FBS e conservare in ghiaccio. Etichettare i crioviali con CB#, CD34+ d5 e data.
- Far ruotare le cellule CD34+ a 360 x g per 10 minuti a 4 °C. Aspirare il mezzo verso il pellet e risospendere il pellet cellulare nel mezzo di congelamento. Aliquot 1 mL di sospensione cellulare su ciascun flaconcino e dividere uniformemente il resto tra i flaconcini. Aggiungere i flaconcini in un congelatore a celle isopropanolo raffreddato a 4 °C, porre a -80 °C e trasferire in azoto liquido per conservare per >90 giorni.
- Irradiazione di cuccioli di topo
- Raccogliere i cuccioli BRGSB/N , 1-3 giorni dopo la nascita, in una scatola di plastica autoclavata con imbottitura. Aggiungi una piccola quantità di biancheria da letto con i cuccioli. Etichetta la scatola con il numero della gabbia e il numero di cuccioli.
- Impostare l'irradiatore (vedere Tabella dei materiali) per una dose di 300 rad. Metti la scatola dei cuccioli nell'irradiatore ed esponi loro a 300 rad. Riporta i cuccioli nella loro gabbia, mettili in una pila e coprili con la biancheria da letto.
- Iniezioni di cuccioli e preparazione di cellule CD34+
- Iniziare la preparazione delle cellule CD34+ ~3 ore dopo l'irradiazione. Riscaldare 10 mL di fluido CB in un tubo conico da 50 mL. Eseguire tutti i passaggi in un BSC sterile.
- Recuperare una fiala di cellule CD34+ espanse e congelate in vitro ogni quattro-sei cuccioli da iniettare. Scongelare rapidamente a 55 °C, fino a quando non è visibile solo una piccola quantità di ghiaccio, e aggiungere le cellule al mezzo CB riscaldato (il flaconcino dovrebbe essere ancora fresco al tatto). Utilizzare 1 mL di terreno per risciacquare ogni flaconcino e centrifugare le cellule a 360 x g per 12 minuti a 4 °C.
NOTA: È stato riscontrato che il rapido scongelamento a 55 °C produce una migliore vitalità cellulare (90% -95%) rispetto allo scongelamento a 37 °C. - Aspirare il mezzo con attenzione. Risospendere il (piccolo) pellet in 2 ml di mezzo CB, mescolare delicatamente e aggiungere ~ 30 μL della sospensione cellulare in un singolo pozzetto in una piastra di conteggio. Diluire 1:1 in tripano blu, quindi aggiungere 10 μL a un emocitometro e contare e calcolare la media delle cellule da quattro quadranti.
- Calcolare il numero totale di cellule CD34+ moltiplicando il conteggio delle cellule per 4 x 104 e quindi registrare la vitalità. Centrifugare a 360 x g per 12 minuti a 4 °C.
- Aspirare il terreno con cautela e risospendere il pellet cellulare in 100 μL di PBS sterile per n+1 cuccioli da iniettare, ottenendo 250.000-450.000 cellule CD34+ per topo. Posizionare il tubo conico sul ghiaccio in un contenitore di trasporto e recarsi al vivaio per l'iniezione del cucciolo.
- Portare una lampada termica, pannolini, siringa da 1 ml, ago da 18 G, ago da 30 G e la preparazione di cellule CD34+ in contenitori sterili al vivaio BSC. Posizionare un pannolino sterile ~ 2 piedi sotto la lampada di calore. Recuperare la gabbia con la lettiera da iniettare e metterla nella BSC.
- Assemblare la siringa con un ago smussato da 18 G. Abbinando l'angolo del tubo conico con la smussatura dell'ago, mescolare delicatamente e disegnare la sospensione cellulare. Metti i cuccioli sul pannolino per riscaldare (fai attenzione al surriscaldamento). Rimuovere l'aria dalla siringa e sostituire l'ago da 18 G con l'ago da 30 G, quindi spingere con cautela la siringa fino a quando la sospensione cellulare si trova proprio sulla punta dell'ago.
NOTA: In alternativa, è possibile utilizzare una siringa da insulina. - Porta un cucciolo alla volta sul bordo del pannolino lontano dalla lampada di calore. Immobilizza il cucciolo su un lato sotto il pollice e l'indice, consentendo una visione chiara del viso. Nota la vena attraverso la guancia sotto dove sarà l'orecchio. Inserire l'ago superficialmente nella vena (IV) più vicina all'occhio e iniettare lentamente 50 μL di cellule.
- Controlla se si sta formando una bolla da un'iniezione sottocutanea. In tal caso, inserisca l'ago più in profondità e proceda all'iniezione delle cellule. Una piccola goccia di sangue / ematoma sarà visibile in caso di successo.
NOTA: Fare riferimento alla procedura di Gombash Lampe et al.24 per quanto riguarda l'iniezione di cuccioli. - Con il cucciolo ancora immobilizzato, eseguire un'iniezione intraepatica (IH) con altri 50 μL di cellule (100 μL totali per cucciolo di IV+IH). Il fegato può essere visualizzato come una macchia scura tra la fascia bianca del latte e la gabbia toracica. Posizionare il cucciolo iniettato su un pannolino più lontano dai cuccioli non iniettati e riscaldare.
NOTA: L'iniezione endovenosa provoca un migliore chimerismo a lungo termine rispetto alla sola iniezione IH25, ma le iniezioni IV non hanno sempre successo. Pertanto, la suddivisione dell'iniezione tra IV e IH assicura l'attecchimento in una percentuale maggiore di topi. - Ripetere entrambe le iniezioni di vena facciale e IH per tutti i cuccioli. Pulisci il sangue, riporta i cuccioli al nido nella loro gabbia e copri con la biancheria da letto.
2. Test del chimerismo umano nel sangue
- Test chimerismo nel sangue dei topi HIS sia a 10 che a 14 settimane di età. Raccogliere 50 μL di sangue attraverso la vena retroorbitaria o utilizzando un metodo alternativo approvato dalla IACUC.
- Per le spurghe retroorbitali, anestetizzare i topi con un vaporizzatore isoflurano impostato su 5 per 1-2 minuti e quindi abbassare l'impostazione del vaporizzatore a 4. Abbassa il vaporizzatore secondo necessità per consentire ai topi di rimanere sufficientemente ossigenati mentre sono sotto anestesia. Non tenere i topi sotto isoflurano per più di 5 minuti.
- Posizionare il naso del topo anestetizzato nell'attacco del cono nasale del vaporizzatore isoflurano e posizionare una goccia dell'analgesico (soluzione oftalmica HCl allo 0,5% di proparacaina USP) sull'occhio.
- Dopo 1 minuto, rimuovere la proparacaina usando una garza sterile, proptose l'occhio e inserire un tubo di ematocrito eparinizzato da 75 mm retro-orbitalmente per raccogliere 50 μL di sangue. Espellere il sangue in una provetta microfuge da 1,5 mL contenente 50 μL di eparina e mescolare delicatamente.
- Pizzicare l'occhio chiuso con una garza sterile per fermare l'emorragia e applicare una goccia di proparacaina. Rimuovere il mouse dall'isoflurano e recuperarlo in una gabbia pulita.
- Isolamento PBMC dal sangue di topo
- Mescolare il sangue/eparina pipettando delicatamente su e giù e sovrapporre lentamente sopra 500 μL di gradiente di densità di 1,077 g/mL, facendo attenzione a non disturbare l'interfaccia. Centrifugare i tubi a 1.220 x g per 20 minuti a RT senza freni.
- Visualizza il buffy coat cellulare come uno strato torbido sopra il gradiente di densità di 1,077 g / ml, sotto il plasma. Rimuovere il maggior numero possibile di cellule dal buffy coat con una pipetta da 200 μL e aggiungere a nuove provette da 1,5 ml contenenti 750 μL di terreno di raccolta. Centrifugare a 360 x g per 11 minuti a RT.
- Aspirare il terreno fino a 50 μL e risospendere il pellet in 750 μL di terreno di raccolta. Centrifugare a 360 x g per 10 minuti a 4 °C e aspirare nuovamente fino a 50 μL. Le celle sono pronte per essere risospese nella macchia superficiale.
- Colorazione superficiale e analisi citometrica a flusso
- Compilare il foglio di lavoro del pannello di colorazione (Tabella 1; "spectral flow bleed panel") con i numeri del mouse e preparare il cocktail di colorazione degli anticorpi aggiungendo tutti gli anticorpi fluorescenti al tampone di colorazione (file supplementare 1). Crea un layout di piastra per aggiungere i campioni a una piastra inferiore a U a 96 pozzetti. Segna il fondo dei pozzetti usando un pennarello permanente. Titolare gli anticorpi utilizzando una procedura standard prima della colorazione per determinare la concentrazione appropriata26.
- Aggiungere 62 μL del cocktail di macchie superficiali a ciascun pozzetto della piastra inferiore a U a 96 pozzetti. Risospendere le cellule nei 50 μL rimanenti nel tubo e aggiungere ogni campione al suo pozzetto corrispondente. Includere un pozzetto per un controllo positivo della colorazione (PBMC umane + splenociti di topo, 1 x 106 cellule ciascuno) da colorare accanto ai campioni. Miscelare mediante pipettaggio e incubare la miscela per 15 minuti a 4 °C.
- Centrifugare a 680 x g per 3 minuti a 4 °C. Aprire la piastra nel lavandino per rimuovere il surnatante e lavare le celle risospendendo 150 μL di tampone colorante mediante pipettaggio delicato ~10x. Ruotare e risospendere ciascun pozzetto in 150 μL di tampone colorante.
- Acquisire i dati per 100 μL di ciascun campione su un citometro a flusso (vedere Tabella dei materiali) ed esportare i file .fcs. Importare i file .fcs nel software di modifica dei dati di flusso (vedere Tabella dei materiali). Applicare una porta poligonale a un grafico FSC-A x SSC-A che circonda le celle, escludendo eventuali detriti. Selezionare le celle all'interno del cancello (vedere Tabella 1; Foglio di lavoro "Spectral Flow Bleed Gating").
- Modificare gli assi in (FSC-A x FSC-H) e porte le celle contenute nella diagonale lineare escludendo i doppietti che sporgono dalla linea. Selezionare queste celle e modificare gli assi in (hCD45 x mCD45).
- Applicare una porta poligonale alla popolazione hCD45+ e applicare il nome "umano". Applicare un gate poligonale alla popolazione mCD45+ e applicare il nome "mouse". Crea una statistica di conteggio sia per la popolazione umana che per quella di topo.
- Selezionare la popolazione umana e modificare gli assi in (CD19 x CD3). Applicare una porta poligonale alle celle CD19+ e denominarla "cellule B". Applicare una porta poligonale alla popolazione CD3+ e denominarla "cellule T". Applicare una porta poligonale alla popolazione doppia negativa e denominarla "NonTB".
- Selezionare la popolazione di cellule T e modificare gli assi in (CD8 x CD4). Applicare una porta poligonale alle popolazioni CD4 e CD8 positive e denominarle rispettivamente "CD4+" e "CD8+".
- Selezionare la popolazione NonTB e modificare gli assi in (CD56 x mieloide). Applicare una porta poligonale alla popolazione totale CD56 positiva compresi i doppi eventi positivi e denominarla "cellule NK". Applicare un gate poligonale alla popolazione CD56 negativa e mieloide positiva e denominarla "mieloide".
- Creare una tabella per le statistiche percentuali e di conteggio di tutte le popolazioni ed esportarle in un software per fogli di calcolo (vedi Tabella dei materiali). Calcola %hCD45 chimerismo = %hCD45/(%hCD45 + mCD45).
- Escludere i topi HIS che sono <20% hCD45+ (di mCD45 + hCD45) per ulteriori esperimenti.
NOTA: In questo studio, le PBMC sono state preparate utilizzando una separazione del gradiente di densità di 1,077 g / mL per una deplezione dei globuli rossi più pulita. Questa procedura ha escluso i granulociti umani e murini dallo strato PBMC. In alternativa, è possibile utilizzare la lisi dei globuli rossi.
3. Iniezione di tumori nei topi
- Prima dell'iniezione del tumore, confermare il numero di cellule T dai dati di sanguinamento a 14 settimane. I tumori vengono iniettati per essere pronti per il raccolto tra 20-26 settimane se le cellule T sono >20% e tra 24-28 settimane se le cellule T sono <20% della popolazione totale di cellule immunitarie. Questa tempistica di iniezione assicura che i topi abbiano un'età compresa tra 20 e 28 settimane e abbiano un numero sufficiente di cellule T alla fine dello studio.
- Iniezione di xenotrapianti derivati da linee cellulari (CDX)
NOTA: La procedura è descritta utilizzando cellule di adenocarcinoma mammario MDA-MB-231 (vedere la tabella dei materiali) come esempio.- Scongelare un'aliquota di cellule congelate, lavare 1 volta riassumendo in 10 mL di DMEM integrato con 10% FBS, 1% PenStrep e 1% aminoacidi non essenziali e centrifugare a 360 x g per 10 minuti a 4 °C. Aspirare il terreno e risospendere in 10 mL di DMEM integrato con 10% FBS, 1% PenStrep e 1% di aminoacidi non essenziali in un matraccio T25 e incubare a 37 °C con 5% di CO2.
NOTA: Le linee cellulari sono autenticate mediante PCR per garantire il corretto tipo di cellula. I surnatanti cellulari sono testati per il micoplasma tramite un test biochimico prima dell'iniezione. - Passaggio ed espansione delle cellule durante la fase di crescita esponenziale a circa l'80% di confluenza.
- Aspirare il substrato, sciacquare le cellule con 5-10 ml di PBS sterile (pH 7,2) e incubare con 1 ml di tripsina-EDTA allo 0,25% per 1-5 minuti fino a quando le cellule si staccano dal pallone.
- Aggiungere 5 mL dello stesso DMEM e mescolare mediante pipettaggio su e giù. Far passare l'intero 6 mL di sospensione cellulare in un nuovo matraccio T75 trattato con coltura tissutale (diluizione 1:3) e aggiungere altri 10 mL di DMEM.
- Far passare le cellule ogni 2-3 giorni alla confluenza dell'80% a 1:3 di placcatura diluitiva in DMEM per espandere le cellule.
- Raccogliere le cellule con tripsina-EDTA allo 0,25% durante la fase di crescita esponenziale entro sei passaggi e lavare con PBS. Mescolare PBS ed estratto di membrana basale (vedi Tabella dei materiali) in un rapporto 1: 1 e aggiungere alle cellule ad una concentrazione finale di 5 x 107 cellule / ml.
- Anestetizzare i topi con isoflurano come descritto al punto 2.1.1. Iniettare per via sottocutanea 100 μL (5 x 106 cellule) di sospensione di cellule tumorali in ciascun fianco utilizzando un ago da 23 G.
- Scongelare un'aliquota di cellule congelate, lavare 1 volta riassumendo in 10 mL di DMEM integrato con 10% FBS, 1% PenStrep e 1% aminoacidi non essenziali e centrifugare a 360 x g per 10 minuti a 4 °C. Aspirare il terreno e risospendere in 10 mL di DMEM integrato con 10% FBS, 1% PenStrep e 1% di aminoacidi non essenziali in un matraccio T25 e incubare a 37 °C con 5% di CO2.
- Eseguire l'iniezione di xenotrapianti derivati dal paziente (PDX) utilizzando trocar. La procedura di iniezione e altre istruzioni di sviluppo e manutenzione per il modello PDX sono disponibili nella letteratura27.
4. Misurazione della crescita tumorale
- Controllare i progressi del tumore una volta alla settimana dopo l'impianto sentendo lungo il fianco per la crescita del tumore. Una volta che i tumori sono palpabili, anestetizzare i topi con isoflurano (come descritto al punto 2.1.1) e radersi su ciascun fianco con un trimmer elettrico, avendo cura del tumore per prevenire le ulcerazioni man mano che il tumore cresce.
NOTA: I topi possono essere rasati prima dell'iniezione del tumore per prevenire lesioni alla pelle intorno ai tumori; Tuttavia, il tasso di ricrescita dei capelli potrebbe ostacolare le misurazioni del tumore. - Misurare la lunghezza e la larghezza dei tumori due volte alla settimana utilizzando i calibri e registrare le misurazioni in mm. Riportare le misurazioni del tumore come volume tumorale (mm3) utilizzando la formula
 . Fare attenzione a non permettere che il carico tumorale superi i 2.000 mm 3 per singolo tumore o un volume combinato di 3.000 mm3.
. Fare attenzione a non permettere che il carico tumorale superi i 2.000 mm 3 per singolo tumore o un volume combinato di 3.000 mm3.
NOTA: Le linee guida per l'uso dei topi nella ricerca sul cancro variano in base alla località. Si prega di fare riferimento alle politiche di cura e utilizzo degli animali presso l'istituto del ricercatore.
5. Trattamenti farmacologici
- Allocare i topi HIS in gruppi di trattamento equivalenti basati sul chimerismo hCD45, sul chimerismo hCD3 e sul chimerismo hCD8. Una volta che i tumori raggiungono 100 mm3 in media, iniziare i trattamenti farmacologici.
NOTA: Il numero di topi per gruppo si basa sul numero di topi HIS generati dal CB. Si raccomanda un minimo di quattro topi per gruppo. La corrispondenza multigruppo non bipartita (ad esempio, nel software R-vivo Manila) è utile per questo raggruppamento28. - Via e frequenza dei farmaci
- Iniettare inibitori anti-PD-1 (nivolumab, pembrolizumab) per via intraperitoniale (i.p.) a 30 mg/kg 1x a settimana o 20 mg/kg 2x a settimana per trattamenti singoli, o 10-15 mg/kg 2x a settimana per trattamenti combinati.
- Dosare le terapie combinate, come terapie mirate, chemioterapie e irradiazione, in conformità con il disegno sperimentale. Terapie mirate e chemioterapie possono essere somministrate attraverso sonda gastrica orale, iniezione endovenosa o cibo.
NOTA: Le dosi possono essere variate e ottimizzate in base ai dati pubblicati e al modello tumorale. Gli studi sulla dose sono spesso testati in riceventi immunodeficienti prima degli studi sui topi HIS.
- Monitorare i topi almeno 3 volte a settimana per cambiamenti di salute come perdita di peso, feci sciolte, postura curva, mobilità ridotta e perdita di pelo. Alcuni sintomi potrebbero essere segni di tossicità da farmaci o GVHD e potrebbe essere necessario ridurre o interrompere il dosaggio del farmaco. Eutanasia dei topi secondo necessità secondo le politiche di cura e utilizzo degli animali.
6. Raccolta di tessuti e tumori di topo alla fine dello studio
- Eutanasia i topi singolarmente secondo le linee guida istituzionali e veterinarie utilizzando gas CO2 compresso con una portata di 2,75 L / min. Monitorare i topi detenuti in CO2 per 1 minuto dopo la morte e quindi eseguire la dislocazione cervicale come forma secondaria di eutanasia.
- Raccolta del sangue
- Raccogliere il sangue tramite puntura intracardiaca. Tenere il mouse in posizione verticale e inserire una siringa da 1 mL con un ago da 25 G direttamente nel cuore da una linea appena a sinistra della linea mediana e sotto le costole. Raccogliere il sangue in una siringa idealmente attraverso un lungo prelievo e trasferirlo in un tubo marcato da 1,5 ml.
NOTA: Ulteriori inserimenti di aghi possono essere eseguiti per raccogliere ulteriore sangue dagli angoli della cavità polmonare con cura. - Lasciare il sangue a 4 °C per 1 ora, quindi centrifugare il sangue in una microcentrifuga per 6 minuti a 7.800 x g. Raccogliere il liquido trasparente (sieri) sopra l'interfaccia del sangue in un secondo tubo pulito ed etichettato da 1,5 ml. Conservare il siero a -20 °C per l'analisi a valle, come citochine infiammatorie umane e di topo o titoli di immunoglobuline.
- Raccogliere il sangue tramite puntura intracardiaca. Tenere il mouse in posizione verticale e inserire una siringa da 1 mL con un ago da 25 G direttamente nel cuore da una linea appena a sinistra della linea mediana e sotto le costole. Raccogliere il sangue in una siringa idealmente attraverso un lungo prelievo e trasferirlo in un tubo marcato da 1,5 ml.
- Dissezione dei tessuti
- Dopo l'iniezione e la crescita delle cellule tumorali nei topi e la somministrazione di trattamenti farmacologici, raccogliere i tessuti. Posizionare i topi su una tavola di dissezione di schiuma, con perni per tenerli in posizione, e le braccia e le gambe estese ad angoli di 45 °. Fai un'incisione al centro del busto, iniziando vicino al bacino e estendendosi fino al mento, cercando di evitare di tagliare il peritoneo (anche se questo non è essenziale). Tirare la pelle fino al bordo e tenerla in posizione con gli spilli.
- Estrarre i linfonodi (LN) utilizzando una pinza fine nel seguente ordine: inguinale, ascellare, cervicale, mesenterico, iatale.
NOTA: Nei topi HIS, i LN sono spesso molto piccoli, simili ad anlage, o "infiammati" con un aspetto distinto diverso da quello di un topo wild-type. Pertanto, il tessuto simile ai LN viene prelevato da ciascun sito. I LN periferici appaiono spesso come "palline" piene di liquido, mentre i LN mesenterici sono più densi. I LN mesenterici sono i più evidenti e consistenti e sono osservati come uno o due distinti nodi più densi, al contrario di una stringa. - Posizionare i LN su un lato di vetrini smerigliati in 8 ml di terreno di raccolta in una capsula di Petri. Tenendo i vetrini ad angoli perpendicolari con i bordi smerigliati verso l'interno, premere delicatamente i tessuti fino a quando il contenuto cellulare non viene rilasciato.
- Risciacquare i vetrini più volte separandoli e unendoli per rilasciare la quantità massima di cellule. Raccogliere le celle con una pipetta di vetro da 5 pollici e filtrarle attraverso una pipetta con tappo di cotone da 9 pollici in un tubo conico etichettato da 15 ml.
- Estrarre la milza dal lato superiore sinistro dell'addome usando due paia di pinze o pinze e forbici. Si noti la dimensione della milza come stima del volume dei mezzi di risospensione. Raccogliere e filtrare la milza mediante digestione meccanica utilizzando vetrini smerigliati, come con i LN.
NOTA: È possibile utilizzare qualsiasi tecnica per la preparazione di tessuti per sospensioni monocellulari. - Eseguire il conteggio e la risospensione delle cellule dai campioni di tessuto come descritto di seguito.
- Centrifugare le cellule linfatiche a 360 x g per 10 minuti a 4 °C. Aspirare il liquido e risospendere il pellet cellulare in 1 mL di terreno di raccolta con DNasi.
- Rimuovere gli eritrociti dalle cellule della milza incubando con 3 ml di tampone di lisi ACK a RT per 3 minuti, seguito dall'aggiunta di 10 ml di terreno di raccolta / DNasi. Centrifugare nuovamente le cellule della milza, aspirare il surnatante e risospendere le cellule in 1-10 ml di terreno di raccolta / DNasi, in base alla dimensione della milza (ad esempio, 1 ml per milza molto piccola e fino a 10 ml per la milza più grande).
- Aggiungere un'aliquota di 10 μL di sospensione cellulare a 90 μL di terreno e contare su un emocitometro. Centrifugare i LN e le cellule della milza, aspirare i surnatanti e risospendere le cellule nel mezzo di raccolta ad una concentrazione di 1 x 108 cellule / ml o un minimo di 80 μL.
- Estrarre i tumori.
- Rimuovere il tumore dal fianco aperto tenendo il tumore con una pinza mentre si tagliano lentamente i margini del tumore con le forbici di dissezione.
- Una volta rimosso il tumore, pesarlo e rimuovere 1/4 per l'elaborazione dell'RNA e dell'immunoistochimica (IHC). Dividere il tumore 1/4 a metà; porre una metà (1/8 dell'intero tumore) in un crioviale, congelare il liquido N2 e conservare a -80 °C per gli studi genomici a valle.
- Posizionare l'altro 1/8 del tumore in una provetta marcata contenente il 10% di formalina. Il giorno successivo, sciacquare e risospendere il tessuto in etanolo al 70% fino a un uso futuro. Posizionare i restanti 3/4 del tumore in un piatto di 6 cm e tritare in pezzi ~ 1 mm usando una lama di bisturi.
- Trasportare i pezzi di tumore in un tubo di dissociazione (vedere Tabella dei materiali), sciacquare il piatto con 5 ml di terreno incompleto per leucociti infiltranti il tumore (TIL) e aggiungere al tubo di dissociazione.
NOTA: Per i tumori di peso >0,4 g, sciacquare il piatto con 10 ml di terreno incompleto TIL e aggiungere al tubo di dissociazione. - Aggiungere la preparazione di collagenasi (vedere Tabella dei materiali) ad una concentrazione finale di 50 mg/ml nel tessuto nel tubo di dissociazione. Dissociare il tessuto mediante dissociazione meccanica a 37 °C per 30 minuti a 1 ora, a seconda della fermezza del tumore.
- Dopo la dissociazione, passare la sospensione su un filtro da 100 μm in un tubo conico da 50 mL e sciacquare il filtro con 10 mL di mezzo completo TIL. Il siero in questo mezzo fermerà la reazione della collagenasi e proteggerà le cellule da un'ulteriore degradazione.
- Centrifugare la sospensione monocellulare a 360 x g per 10 minuti a 4 °C. Risospendere il pellet in un mezzo di raccolta sufficiente con DNase in modo che la sospensione cellulare possa facilmente passare attraverso una punta di pipetta P1000 e registrare il volume per l'analisi a valle.
NOTA: Un volume più piccolo aumenta la raccolta di cellule immunitarie tumorali ma è più suscettibile agli intasamenti del citometro a flusso.
7. Colorazione cellulare e analisi citometriche a flusso
- Preparazione delle macchie e placcatura cellulare
- Preparare cocktail di colorazione: il giorno della raccolta, aggiungere il numero di campioni al foglio di lavoro di colorazione (Tabella 2; Fogli di lavoro "Pannello Flusso convenzionale A", "Pannello Flusso spettrale B" e "Pannello Flusso convenzionale C") per tutte le macchie e la stampa. Preparare cocktail di macchie superficiali per le macchie A e B nel tampone colorante (SB) aggiungendo ciascun anticorpo singolarmente con una nuova punta, contrassegnando ciascun reagente in movimento e conservare a 4 °C fino al momento del bisogno. Preparare i coloranti vitali appropriati in PBS privo di azoturi e conservare a 4 °C fino al momento del bisogno (caldo a RT prima dell'uso). Preparare il cocktail di colorazione superficiale C il giorno 2 in SB insieme alle macchie intracellulari B e C nei rispettivi tamponi di permeabilizzazione.
- Creare un layout di piastra per tutti i campioni nel foglio di lavoro di colorazione e aliquote 100 μL di PBS privo di azoturi ai pozzetti. Includere controlli non colorati per ciascun tessuto e colorazione (per citometria spettrale).
- Aggiungere le celle a piastre da 96 pozzetti contenenti 50 μL di PBS. Per la colorazione A, risospendere ciascun gruppo tissutale nel volume appropriato, come indicato sul foglio di lavoro di colorazione, e conservare a 4 °C fino all'acquisizione sul citometro a flusso lo stesso giorno. Per le macchie B e C, aggiungere 25 μL di linfa e 60 μL di sospensioni di cellule tissutali non linfatiche ai pozzetti con PBS.
- Eseguire la stimolazione in vitro delle citochine per il rilevamento mediante colorazione intracellulare utilizzando la colorazione C.
- Centrifugare la piastra del pozzetto C 96 del punto 7.1.3 a 680 x g per 3 minuti a 4 °C. Aggiungere 200 μL di terreno TIL completo (RPMI 1640, 10 mM HEPES [pH 7], 10% FBS) a ciascun pozzetto. Conservare la piastra con la sospensione cellulare a 4 °C durante la notte.
- La mattina seguente, diluire il cocktail di stimolazione cellulare (vedi Tabella dei materiali) 1:500 in mezzo TIL completo. Centrifugare la piastra contenente la sospensione cellulare a 680 x g per pellettare le celle e scorrere per rimuovere il supporto. Risospendere le cellule in 200 μL del cocktail di stimolazione cellulare preparato e incubare a 37 °C per 1 ora.
- Aggiungere 25 μL di soluzione inibitrice del trasporto proteico contenente monensin ad una diluizione 1:1.000 in mezzo TIL completo, mescolare le cellule e incubare a 37 °C per altre 4 ore per consentire l'accumulo intracellulare di citochine.
- Eseguire la colorazione cellulare.
- Per le macchie A e B (giorno 1) e per la macchia C (giorno 2), centrifugare a 680 x g per 3 minuti a 4 °C. Sfiorare le piastre e aggiungere i coloranti di vitalità appropriati ai pozzetti. Mescolare bene pipettando delicatamente su e giù con una pipetta multicanale e incubare per 15 minuti a RT.
- Centrifugare le piastre (come al punto 7.1.5.1), quindi aggiungere le opportune macchie superficiali alle piastre e mescolare delicatamente mediante pipettaggio con una pipetta multicanale. Incubare per 15 minuti a 4 °C e centrifugare nuovamente. Sfiorare le piastre, lavare le celle con pipettaggio delicato su e giù con 150 μL di SB e centrifugare.
- Scorrere le piastre, ripetere il lavaggio con pipettaggio delicato su e giù con 150 μL di SB e centrifugare a 680 x g per 3 minuti a 4 °C. Per la colorazione A, risospendere ciascun gruppo di tessuti nel volume appropriato, come indicato sul foglio di lavoro di colorazione, e conservare a 4 °C. Per la macchia B, correggere con il fissativo del kit del fattore di trascrizione FoxP3 (vedi Tabella dei materiali) per 30 minuti su RT. Per la macchia C, fissare la paraformaldeide all'1% (v / v) in SB per 30 minuti a RT.
- Centrifugare le piastre fisse a 480 x g per 3 minuti a 4 °C. Lavare 1x in SB. Per la macchia B, le cellule possono essere lasciate durante la notte.
- Permeabilizzare le cellule: risospendere i pozzetti in 150 μL di tampone di permeabilizzazione del kit del fattore di trascrizione FoxP3 per la colorazione B e in saponina allo 0,5% (p/v) in SB per la statina C. Incubare per 15 minuti a RT. Centrifugare le piastre fisse a 480 x g per 3 minuti a 4 °C.
- Sfiora i piatti e aggiungi i rispettivi cocktail di colorazione intracellulare per la macchia B e la colorazione C. Incubare per 30 minuti a RT.
- Centrifugare le piastre fisse a 480 x g per 3 minuti a 4 °C e far scorrere le piastre. Lavare le cellule con 150 μL dei corrispondenti tamponi di permeabilizzazione (colorazione B, tampone di permeabilizzazione del kit del fattore di trascrizione FoxP3; colorante C, saponina).
- Scorrere le piastre e lavare mediante pipettaggio su e giù in 150 μL di SB. Centrifugare a 480 x g per 3 minuti a 4 °C. Scorrere le piastre e risospendere le cellule in SB per gruppo tissutale nei volumi appropriati annotati sul foglio di lavoro di colorazione.
NOTA: I campioni sono ora pronti per l'acquisizione mediante citometria a flusso spettrale. - Impostare il citometro a flusso (vedere la tabella dei materiali) con appropriati controlli a singola colorazione per ogni macchia e campioni non colorati per ciascun gruppo di tessuto per tenere conto delle differenze nell'autofluorescenza durante la dismiscelazione. Acquisire i volumi appropriati per gruppo di tessuti come definito nel foglio di lavoro di colorazione (Tabella 2) ed esportare i file .fcs.
- Analisi dei dati di citometria a flusso
- Utilizzando il software di analisi della citometria a flusso (vedere Tabella dei materiali), creare una nuova area di lavoro. Creare nuovi gruppi per ogni organo (LN, milza e TIL). Importare i file .fcs per ogni organo nel gruppo.
- Create un dot plot bivariato, impostate gli assi su (FSC-A x SSC-A) e applicate un gate poligonale agli eventi cellulari, evitando eventi sui bordi (gate di tutte le celle). Selezionare gli eventi cellulari, modificare gli assi in (FSC-A x FSC-H) e applicare una porta poligonale agli eventi sulla diagonale lineare, escludendo gli eventi che si discostano dalla diagonale per generare la porta "singoletto". Selezionare il gate "singoletto" e modificare gli assi in (hCD45 x mCD45). Applicare le porte poligonali agli eventi positivi hCD45 e mCD45 e denominarli rispettivamente "umano" e "topo".
- Selezionare la popolazione umana e modificare l'asse Y in Live/Dead Aqua. Applicare un cancello poligonale alla popolazione Live/Dead negativa, hCD45 positiva e chiamarla "vivo umano". Selezionate la popolazione di mouse e impostate l'asse X su Live/Dead Aqua. Applicare una porta poligonale alla popolazione positiva di mCD45 negativa e chiamarla "topo vivo".
- In modo simile, selezionare il gate genitore e gli assi X e Y come indicato nei fogli di lavoro "Gating A Flusso convenzionale", "Gating B a flusso spettrale" e "Gating C di flusso convenzionale" (Tabella 2) per isolare le popolazioni indicate (ad esempio, cellule B umane, cellule T attivate).
NOTA: la gating rappresentativa per ogni macchia (Bleed, A, B, C) è inclusa nel file supplementare 2. - Crea tabelle di conteggio e frequenza di esportazione nel software di analisi della citometria a flusso per tutte le popolazioni ed esporta in un software di foglio di calcolo. La popolazione parentale per le frequenze è indicata nella Tabella 2.
- Utilizzare i dati per generare grafici basati su gruppi di trattamento sperimentali.
NOTA: I dati possono anche essere analizzati utilizzando pacchetti R nel software di analisi. È possibile creare un singolo file .fcs da tutti i campioni da analizzare utilizzando la funzione di concatenazione. Questi dati possono essere ridotti dimensionalmente con l'algoritmo T-SNE aggiungendo parametri di parole chiave per tipo di tessuto e gruppo di trattamento. L'algoritmo FlowSOM può quindi essere utilizzato per raggruppare le popolazioni e lo strumento ClusterExplorer può essere utilizzato per identificare le popolazioni. Nuove popolazioni cellulari possono essere identificate in questo modo, confrontate visivamente e quantificate tra gruppi di trattamento o all'interno di vari tessuti. - Correlare i parametri immunitari per i tumori all'interno dello stesso gruppo di trattamento, con la crescita per quel tumore per definire gli immunotipi correlati con l'inibizione della crescita tumorale. Quantificare la crescita tumorale in base al tasso di crescita specifico (SGR) per quel tumore, una misura che tiene conto della differenza nei volumi tumorali in un tempo specificato. Questa misurazione normalizza i tumori raccolti in giorni diversi a causa della salute del topo e delle date di inizio del trattamento.
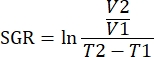
Risultati
Seguendo il protocollo tumorale del fianco e la tempistica sperimentale (Figura 1), la crescita tumorale e la risposta immunitaria a una terapia mirata con inibitori della tirosin-chinasi (TKI) e al trattamento combinato con nivolumab sono state studiate in due distinti PDX per il cancro del colon-retto umano (CRC). I farmaci TKI sono stati studiati in ospiti immunodeficienti per valutare la crescita tumorale solo29. Questo modello ha permesso lo studio dei cambiament...
Discussione
Negli ultimi 6 anni, utilizzando la nostra esperienza sia in immunologia che nei topi umanizzati, il nostro team di ricerca ha sviluppato un modello preclinico molto necessario per testare le immunoterapie su una varietà di tumori umani 3,7,30,31. Questo protocollo enfatizza la considerazione della variabilità del modello, con particolare attenzione alle popolazioni di cellule T umane incentr...
Divulgazioni
Nessuno.
Riconoscimenti
Vorremmo ringraziare sia l'Animal Research Facility (OLAR) per la loro cura dei nostri topi, sia la risorsa condivisa di citometria a flusso supportata dal Cancer Center Support Grant (P30CA046934) presso il nostro istituto per il loro immenso aiuto in tutto il nostro lavoro. Riconosciamo anche Gail Eckhardt e Anna Capasso per le nostre collaborazioni inaugurali che studiano le immunoterapie per PDX umani nel nostro modello HIS-BRGS. Questo studio è stato supportato in parte dal National Institutes of Health P30CA06934 Cancer Center Support Grant con l'uso della risorsa condivisa PHISM (Pre-clinical Human Immune System Mouse Models), RRID: SCR_021990 and Flow Cytometry Shared Resource, RRID: SCR_022035. Questa ricerca è stata sostenuta in parte dal NIAID del National Institutes of Health con il numero di contratto 75N93020C00058.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 mL syringe w/needles | McKesson | 1031815 | |
| 15 mL tubes | Grenier Bio-One | 188271 | |
| 2-mercaptoethanol | Sigma | M6250 | |
| 50 mL tubes | Grenier Bio-One | 227261 | |
| AutoMACS Pro Separator | Miltenyi | 130-092-545 | |
| BD Golgi Stop Protein Transport Inhibitor with monensin | BD Bioscience | BDB563792 | |
| BSA | Fisher Scientific | BP1600100 | |
| Cell Stim Cocktail | Life Technologies | 509305 | |
| Chill 15 Rack | Miltenyi | 130-092-952 | |
| Cotton-plugged glass pipettes | Fisher Scientific | 13-678-8B | |
| Cultrex Basement membrane extract | R&D Systems | 363200502 | |
| Cytek Aurora | Cytek | ||
| DNase | Sigma | 9003-98-9 | |
| eBioscience FoxP3/Transcription Factor Staining Buffer Set | Invitrogen | 00-5523-00 | |
| Embryonic Stemcell FCS | Gibco | 10439001 | |
| Eppendorf Tubes; 1.5 mL volume | Grenier Bio-One | 616201 | |
| Excel | Microsoft | ||
| FBS | Benchmark | 100-106 500mL | |
| Ficoll Hypaque | GE Healthcare | 45001752 | |
| FlowJo Software | BD Biosciences | ||
| Forceps - fine | Roboz Surgical | RS5045 | |
| Forceps normal | Dumont | RS4919 | |
| Formaldehyde | Fisher | F75P1GAL | |
| Frosted Glass Slides | Corning | 1255310 | |
| Gentlemacs C-Tubes | Miltenyi | 130-096-334 | |
| GentleMACS Dissociator | Miltenyi | 130-093-235 | |
| glass pipettes | DWK Life Sciences | 63A53 | |
| Glutamax | Gibco | 11140050 | |
| HBSS w/ Ca & Mg | Sigma | 55037C | |
| HEPES | Corning | MT25060CI | |
| IgG standard | Sigma | I2511 | |
| IgM standard | Sigma | 401108 | |
| IMDM | Gibco | 12440053 | |
| Liberase DL | Roche | 5466202001 | |
| LIVE/DEAD Fixable Blue | Thermo | L23105 | |
| MDA-MB-231 | ATCC | HTB-26 | |
| MEM | Gibco | 1140050 | |
| mouse anti-human IgG-AP | Southern Biotech | JDC-10 | |
| mouse anti-human IgG-unabeled | Southern Biotech | H2 | |
| mouse anti-human IgM-AP | Southern Biotech | UHB | |
| mouse anti-human IgM-unlabeled | Southern Biotech | SA-DA4 | |
| MultiRad 350 | Precision X-Ray | ||
| PBS | Corning | 45000-446 | |
| Pen Strep | Gibco | 15140122 | |
| Petri Dishes | Fisher Scientific | FB0875713A | |
| p-nitrophenyl substrate | Thermo | 34045 | |
| PRISM | Graphpad | ||
| Rec Hu FLT3L | R&D systems | 308-FK-005/CF | |
| Rec Hu IL6 | R&D systems | 206-IL-010/CF | |
| Rec Hu SCF | R&D systems | 255SC010 | |
| RPMI 1640 | Corning | 45000-39 | |
| Saponin | Sigma | 8047-15-2 | |
| Scissors | McKesson | 862945 | |
| Serological pipettes 25 mL | Fisher Scientific | 1367811 | |
| Sterile filter | Nalgene | 567-0020 | |
| Sterile molecular water | Sigma | 7732-18-5 | |
| Yeti Cell Analyzer | Bio-Rad | 12004279 | |
| Zombie Green | biolegend | 423112 |
Riferimenti
- Chulpanova, D. S., Kitaeva, K. V., Rutland, C. S., Rizvanov, A. A., Solovyeva, V. V. Mouse tumor models for advanced cancer immunotherapy. International Journal of Molecular Sciences. 21 (11), 4118 (2020).
- Olson, B., Li, Y., Lin, Y., Liu, E. T., Patnaik, A. Mouse models for cancer immunotherapy research. Cancer Discovery. 8 (11), 1358-1365 (2018).
- Marin-Jimenez, J. A., et al. Testing cancer immunotherapy in a human immune system mouse model: correlating treatment responses to human chimerism, therapeutic variables and immune cell phenotypes. Frontiers in Immunology. 12, 607282 (2021).
- Yin, L., Wang, X. J., Chen, D. X., Liu, X. N., Wang, X. J. Humanized mouse model: a review on preclinical applications for cancer immunotherapy. American Journal of Cancer Research. 10 (12), 4568-4584 (2020).
- Cogels, M. M., et al. Humanized mice as a valuable pre-clinical model for cancer immunotherapy research. Frontiers in Oncology. 11, 784947 (2021).
- Jin, K. T., et al. Development of humanized mouse with patient-derived xenografts for cancer immunotherapy studies: A comprehensive review. Cancer Science. 112 (7), 2592-2606 (2021).
- Capasso, A., et al. Characterization of immune responses to anti-PD-1 mono and combination immunotherapy in hematopoietic humanized mice implanted with tumor xenografts. Journal for Immunotherapy of Cancer. 7 (1), 37 (2019).
- Wang, M., et al. Humanized mice in studying efficacy and mechanisms of PD-1-targeted cancer immunotherapy. The FASEB Journal. 32 (3), 1537-1549 (2018).
- Yong, K. S. M., et al. Humanized mouse as a tool to predict immunotoxicity of human biologics. Frontiers in Immunology. 11, 553362 (2020).
- Shen, H. W., Jiang, X. L., Gonzalez, F. J., Yu, A. M. Humanized transgenic mouse models for drug metabolism and pharmacokinetic research. Current Drug Metabolism. 12 (10), 997-1006 (2011).
- Bosma, G. C., Custer, R. P., Bosma, M. J. A severe combined immunodeficiency mutation in the mouse. Nature. 301 (5900), 527-530 (1983).
- Shultz, L. D., et al. Multiple defects in innate and adaptive immunologic function in NOD/LtSz-scid mice. The Journal of Immunology. 154 (1), 180-191 (1995).
- Legrand, N., et al. Functional CD47/signal regulatory protein alpha (SIRP(alpha)) interaction is required for optimal human T- and natural killer- (NK) cell homeostasis in vivo. Proceedings of the National Academy of Sciences. 108 (32), 13224-13229 (2011).
- Ishikawa, F., et al. Development of functional human blood and immune systems in NOD/SCID/IL2 receptor {gamma} chain(null) mice. Blood. 106 (5), 1565-1573 (2005).
- Ito, M., et al. NOD/SCID/gamma(c)(null) mouse: an excellent recipient mouse model for engraftment of human cells. Blood. 100 (9), 3175-3182 (2002).
- Shultz, L. D., et al. Human lymphoid and myeloid cell development in NOD/LtSz-scid IL2R gamma null mice engrafted with mobilized human hemopoietic stem cells. The Journal of Immunology. 174 (10), 6477-6489 (2005).
- Traggiai, E., et al. Development of a human adaptive immune system in cord blood cell-transplanted mice. Science. 304 (5667), 104-107 (2004).
- Theocharides, A. P., Rongvaux, A., Fritsch, K., Flavell, R. A., Manz, M. G. Humanized hemato-lymphoid system mice. Haematologica. 101 (1), 5-19 (2016).
- Goldman, J. P., et al. Enhanced human cell engraftment in mice deficient in RAG2 and the common cytokine receptor gamma chain. British Journal of Haematology. 103 (2), 335-342 (1998).
- Stripecke, R., et al. Innovations, challenges, and minimal information for standardization of humanized mice. EMBO Molecular Medicine. 12 (7), (2020).
- Allen, T. M., et al. Humanized immune system mouse models: progress, challenges and opportunities. Nature Immunology. 20 (7), 770-774 (2019).
- Gammelgaard, O. L., Terp, M. G., Preiss, B., Ditzel, H. J. Human cancer evolution in the context of a human immune system in mice. Molecular Oncology. 12 (10), 1797-1810 (2018).
- Rios-Doria, J., Stevens, C., Maddage, C., Lasky, K., Koblish, H. K. Characterization of human cancer xenografts in humanized mice. Journal for Immunotherapy of Cancer. 8 (1), 000416 (2020).
- Gombash Lampe, S. E., Kaspar, B. K., Foust, K. D. Intravenous injections in neonatal mice. Journal of Visualized Experiments. (93), e52037 (2014).
- Lang, J., Weiss, N., Freed, B. M., Torres, R. M., Pelanda, R. Generation of hematopoietic humanized mice in the newborn BALB/c-Rag2null Il2rγnull mouse model: a multivariable optimization approach. Clinical Immunology. 140 (1), 102-116 (2011).
- Laskowski, T. J., Hazen, A. L., Collazo, R. S., Haviland, D. Rigor and reproducibility of cytometry practices for immuno-oncology: a multifaceted challenge. Cytometry Part A. 97 (2), 116-125 (2020).
- Bagby, S., et al. Development and maintenance of a preclinical patient derived tumor xenograft model for the investigation of novel anti-cancer therapies. Journal of Visualized Experiments. (115), e54393 (2016).
- Laajala, T. D., et al. Optimized design and analysis of preclinical intervention studies in vivo. Scientific Reports. 6, 30723 (2016).
- Na, Y. S., et al. Establishment of patient-derived xenografts from patients with gastrointestinal stromal tumors: analysis of clinicopathological characteristics related to engraftment success. Scientific Reports. 10 (1), 7996 (2020).
- Tentler, J. J., et al. RX-5902, a novel beta-catenin modulator, potentiates the efficacy of immune checkpoint inhibitors in preclinical models of triple-negative breast cancer. BMC Cancer. 20 (1), 1063 (2020).
- Lang, J., et al. Development of an adrenocortical cancer humanized mouse model to characterize anti-PD1 effects on tumor microenvironment. The Journal of Clinical Endocrinology & Metabolism. 105 (1), 26-42 (2020).
- Lang, J., et al. Studies of lymphocyte reconstitution in a humanized mouse model reveal a requirement of T cells for human B cell maturation. The Journal of Immunology. 190 (5), 2090-2101 (2013).
- Katano, I., et al. NOD-Rag2null IL-2Rγnull mice: an alternative to NOG mice for generation of humanized mice. Experimental Animalas. 63 (3), 321-330 (2014).
- Brehm, M. A., et al. Parameters for establishing humanized mouse models to study human immunity: analysis of human hematopoietic stem cell engraftment in three immunodeficient strains of mice bearing the IL2rγ(null) mutation. Clinical Immunology. 135 (1), 84-98 (2010).
- Hasgur, S., Aryee, K. E., Shultz, L. D., Greiner, D. L., Brehm, M. A. Generation of immunodeficient mice bearing human immune systems by the engraftment of hematopoietic stem cells. Methods in Molecular Biology. 1438, 67-78 (2016).
- Andre, M. C., et al. Long-term human CD34+ stem cell-engrafted nonobese diabetic/SCID/IL-2Rγnull mice show impaired CD8+ T cell maintenance and a functional arrest of immature NK cells. The Journal of Immunology. 185 (5), 2710-2720 (2010).
- Wunderlich, M., et al. Improved multilineage human hematopoietic reconstitution and function in NSGS mice. PLoS One. 13 (12), 0209034 (2018).
- Lee, J., Brehm, M. A., Greiner, D., Shultz, L. D., Kornfeld, H. Engrafted human cells generate adaptive immune responses to Mycobacterium bovis BCG infection in humanized mice. BMC Immunology. 14, 53 (2013).
- Masse-Ranson, G., et al. Accelerated thymopoiesis and improved T-cell responses in HLA-A2/-DR2 transgenic BRGS-based human immune system mice. European Journal of Immunology. 49 (6), 954-965 (2019).
- Oswald, E., et al. Immune cell infiltration pattern in non-small cell lung cancer PDX models is a model immanent feature and correlates with a distinct molecular and phenotypic make-up. Journal for Immunotherapy of Cancer. 10 (4), 004412 (2022).
Erratum
Formal Correction: Erratum: Testing Cancer Immunotherapeutics in a Humanized Mouse Model Bearing Human Tumors
Posted by JoVE Editors on 5/25/2023. Citeable Link.
An erratum was issued for: Testing Cancer Immunotherapeutics in a Humanized Mouse Model Bearing Human Tumors. The Authors section was updated from:
Jordi M. Lanis1
Matthew S. Lewis1
Hannah Strassburger1
Stacey M. Bagby2
Adrian T. A. Dominguez2
Juan A. Marín-Jiménez3
Roberta Pelanda1
Todd M. Pitts2
Julie Lang1
1Department of Immunology and Microbiology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
2Division of Oncology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
3Department of Medical Oncology, Catalan Institute of Oncology (ICO-L’Hospitalet)
to:
Jordi M. Lanis1
Matthew S. Lewis1
Hannah Strassburger1
Kristina Larsen1
Stacey M. Bagby2
Adrian T. A. Dominguez2
Juan A. Marín-Jiménez3
Roberta Pelanda1
Todd M. Pitts2
Julie Lang1
1Department of Immunology and Microbiology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
2Division of Oncology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
3Department of Medical Oncology, Catalan Institute of Oncology (ICO-L’Hospitalet)
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon