Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Test d’immunothérapies anticancéreuses dans un modèle murin humanisé portant des tumeurs humaines
* Ces auteurs ont contribué à parts égales
Dans cet article
Erratum Notice
Résumé
Ce protocole décrit la génération de souris du système immunitaire humain (SIH) pour les études d’immuno-oncologie. Les instructions et les considérations relatives à l’utilisation de ce modèle pour tester les immunothérapies humaines sur les tumeurs humaines implantées dans ce modèle sont présentées en mettant l’accent sur la caractérisation de la réponse du système immunitaire humain à la tumeur.
Résumé
L’inversion de la nature immunosuppressive du microenvironnement tumoral est essentielle au succès du traitement des cancers avec des médicaments d’immunothérapie. Les modèles de cancer murin sont extrêmement limités dans leur diversité et souffrent d’une mauvaise traduction à la clinique. Pour servir de modèle préclinique plus physiologique pour les études d’immunothérapie, ce protocole a été développé pour évaluer le traitement des tumeurs humaines chez une souris reconstituée avec un système immunitaire humain. Ce protocole unique démontre le développement de souris du système immunitaire humain (HIS, « humanisé »), suivi de l’implantation d’une tumeur humaine, soit une xénogreffe dérivée d’une lignée cellulaire (CDX), soit une xénogreffe dérivée du patient (PDX). Les souris HIS sont générées par l’injection de cellules souches hématopoïétiques humaines CD34+ isolées du sang de cordon ombilical dans des souris BRGS néonatales (BALB/c Rag2-/- IL2RγC-/- NODSIRPα) hautement immunodéficientes qui sont également capables d’accepter une tumeur xénogénique. L’importance de la cinétique et des caractéristiques du développement du système immunitaire humain et de l’implantation tumorale est soulignée. Enfin, une évaluation approfondie du microenvironnement tumoral à l’aide de la cytométrie en flux est décrite. Dans de nombreuses études utilisant ce protocole, il a été constaté que le microenvironnement tumoral des tumeurs individuelles est récapitulé chez les souris HIS-PDX; Les tumeurs « chaudes » présentent une infiltration immunitaire importante alors que les tumeurs « froides » ne le font pas. Ce modèle sert de terrain d’essai pour les immunothérapies combinées pour un large éventail de tumeurs humaines et représente un outil important dans la quête de la médecine personnalisée.
Introduction
Les modèles de cancer de la souris sont importants pour établir les mécanismes de base de la croissance tumorale et de l’évasion immunitaire. Cependant, les études sur le traitement du cancer sur des modèles murins ont donné une traduction finie à la clinique en raison des modèles syngéniques limités et des différences spécifiques à l’espèce 1,2. L’émergence des thérapies immunitaires en tant qu’approche dominante pour contrôler les tumeurs a réitéré la nécessité d’un modèle in vivo avec un système immunitaire humain fonctionnel. Les progrès réalisés chez les souris du système immunitaire humain (souris HIS) au cours de la dernière décennie ont permis d’étudier l’immuno-oncologie in vivo dans une grande variété de types de cancer et d’agents immunothérapeutiques 3,4,5,6. Les modèles tumoraux humains, y compris les xénogreffes dérivées de lignées cellulaires et de patients (CDX et PDX, respectivement), se développent bien chez les souris HIS et, dans la plupart des cas, sont presque identiques à leur croissance chez l’hôte immunodéficient dépourvu de greffe hématopoïétique humaine 7,8. Sur la base de cette découverte clé, les chercheurs ont utilisé le modèle murin HIS pour étudier les immunothérapies humaines, y compris les thérapies combinées conçues pour modifier le microenvironnement tumoral (TME) afin de diminuer l’immunosuppression et ainsi améliorer la destruction tumorale dirigée par le système immunitaire. Ces modèles précliniques aident à résoudre les problèmes d’hétérogénéité des cancers humains et peuvent également prédire le succès du traitement ainsi que surveiller les toxicités médicamenteuses liées au système immunitaire 9,10.
La production d’un modèle murin avec un système immunitaire humain par l’introduction de cellules souches hématopoïétiques humaines nécessite une souris immunodéficiente receveuse qui ne rejettera pas la xénogreffe. Les modèles murins HIS actuels sont dérivés de souches de souris immunodéficientes qui ont été signalées il y a plus de 30 ans. La première souche de souris immunodéficiente décrite était des souris SCID dépourvues de cellules T et B11, suivies d’un hybride NOD-SCID avec un polymorphisme SIRPα responsable de la tolérance des macrophages de souris aux cellules humaines, en raison de la liaison accrue de l’allèle NOD SIRPα à la molécule CD47humaine 12,13. Au début des années 2000, la suppression de la chaîne gamma commune du récepteur IL-2 (IL-2Rγc) sur les souches immunodéficientes BALB/c et NOD a changé la donne pour une greffe humaine améliorée, en raison de délétions génétiques interdisant le développement des cellules NK de l’hôte14,15,16,17. Des modèles alternatifs, tels que les souris BRG et NRG, permettent d’obtenir un déficit en lymphocytes T et B par délétion du gène Rag1 ou Rag2, nécessaire aux réarrangements des gènes des récepteurs des cellules T et B et donc à la maturation et à la survie des lymphocytes18,19. La souris BRGS (BALB/c -Rag2 nullIl2RγCnullSirpαNOD) utilisée ici combine le déficit en chaîne IL-2Rγ et l’allèle NOD SIRPα sur le fond Rag2-/-, résultant en une souris hautement immunodéficiente sans cellules T, B ou NK, mais avec une vigueur et une santé suffisantes pour permettre une greffe à long terme de plus de 30 semaines13.
Les souris HIS peuvent être générées de multiples façons, l’injection humaine de PBMC étant la méthode la plus directe15,18,20. Cependant, ces souris ont une expansion prononcée des cellules T humaines activées qui entraîne la maladie du greffon contre l’hôte (GVH) à l’âge de 12 semaines, empêchant les études à long terme. Alternativement, les cellules souches hématopoïétiques humaines du sang de cordon ombilical (CB), de la moelle osseuse et du foie fœtal peuvent également être utilisées pour la greffe et la production du système immunitaire humain de novo. Dans ce système, les cellules souches hématopoïétiques produisent un système immunitaire humain multi-lignée avec la génération de cellules immunitaires T, B et innées qui sont particulièrement tolérantes envers l’hôte souris, par rapport aux souris PBMC qui développent principalement des cellules T. Par conséquent, la GVH est absente ou considérablement retardée, et les études peuvent être étendues aux souris jusqu’à l’âge de 10 mois. Le CB fournit une source facile, accessible et non invasive de cellules souches hématopoïétiques humaines CD34+ qui facilite la greffe de plusieurs souris HIS ayant un système immunitaire génétiquement identique 17,18,20,21. Au cours des dernières années, les modèles murins HIS ont été largement utilisés pour étudier l’immunothérapie et le TME 3,4,5,6. Malgré le développement de systèmes immunitaires dérivés de l’homme chez ces souris, les tumeurs de xénogreffe humaine se développent à des taux similaires à ceux des souris immunodéficientes témoins et permettent l’interaction complexe entre les cellules cancéreuses et les cellules immunitaires, ce qui est important pour maintenir le microenvironnement du PDX 3,7,8 greffé . Ce protocole a été utilisé pour réaliser plus de 50 études testant des traitements chez des souris HIS-BRGS avec PDX et CDX. Une conclusion importante est que les tumeurs humaines chez les souris HIS maintiennent leur TME unique tel que défini par l’évaluation moléculaire de la tumeur par rapport à l’échantillon initial du patient et aux caractéristiques de l’infiltrat immunitaire 3,22,23. Notre groupe se concentre sur l’évaluation approfondie du HIS dans les organes immunitaires et la tumeur à l’aide de la cytométrie de flux multiparamétrique. Nous décrivons ici un protocole pour l’humanisation des souris BRGS, l’évaluation du chimérisme, l’implantation de tumeurs humaines, les mesures de croissance tumorale, l’administration de traitements contre le cancer et l’analyse des cellules HIS par cytométrie en flux.
Protocole
Tous les travaux sur les animaux ont été effectués conformément aux protocoles sur les animaux approuvés par le comité institutionnel de soin et d’utilisation des animaux de l’Université du Colorado à Denver (protocoles IACUC #00593 et #00021). Tous les travaux sur les animaux ont été effectués conformément à l’Office of Laboratory Animal Resources (OLAR), un établissement accrédité par l’American Association for Laboratory Animal Care, au campus médical Anschutz de l’Université du Colorado à Denver. Tous les échantillons de sang de cordon humain ont été prélevés sous forme de dons de donneurs anonymisés et ne sont donc pas assujettis à l’approbation du comité d’éthique de la recherche sur des sujets humains.
REMARQUE : Les compositions de tous les supports et solutions mentionnés dans le protocole sont incluses dans le dossier supplémentaire 1. La figure 1 illustre le protocole global de génération et d’analyse des réponses immunitaires aux tumeurs chez les souris HIS-BRGS.
1. Génération de souris HIS
- Élevage de souris BALB/c -Rag2 null Il2RγCnull SirpαNOD (BRGS)
REMARQUE: Cette souche est extrêmement immunodéficiente, sans cellules T, B ou NK. Par conséquent, des mesures rigoureuses doivent être utilisées pour prévenir les infections opportunistes. Maintenir la colonie sur un régime contenant du triméthoprime et de la sulfadiazine sur un calendrier alterné de 2 semaines avec un régime normal. Maintenir le plus haut niveau possible de salle de logement par précaution (p. ex., une installation de douche à barrière avec un accès limité).- Maintenir les colonies de souris homozygotes BALB/c -Rag2 null Il2RγC null Sirpα NOD (BRGS) et BALB/c Rag2 null Il2RγC null SirpaBalb/c (BRG) homozygotes comme reproducteurs.
- Breed BRGS N/N×BRG B/Bpourgénérer des petits BRGSB/N, à utiliser comme receveurs de cellules souches humaines. Dans cette colonie, les BRGSB/N sont en meilleure santé que les BRGS N/N, et se greffent à des niveaux équivalents (plus que BRG).
- Isolement de cellules souches humaines CD34+ à partir de CB ombilical
REMARQUE: Aucun antibiotique n’est utilisé pour cette procédure. Par conséquent, une bonne technique stérile est impérative.- Placez une grille à tubes coniques de 50 mL, ainsi que 3 tubes coniques de 15 ml et ~10 x 50 ml dans une enceinte de biosécurité stérilisée (ESB). Vaporisez un sac de prélèvement sanguin avec de l’éthanol à 70 % et laissez-le sécher dans l’ESB.
- Calculer le nombre de tubes coniques de 50 mL requis pour l’isolement du gradient de densité CB = volume CB/15, arrondi vers le haut et à un nombre de tubes pairs. Calculer le volume sanguin par tube = volume CB/nombre de tubes. Versez soigneusement le sang du sac CB dans chaque tube conique; il s’agit d’un maximum de 15 mL par tube. Utilisez une pipette automatique et une pipette sérologique de 25 ml pour mélanger le sang 1:1 avec du PBS stérile en pipetant de haut en bas.
- Utilisez une pipette automatique à basse vitesse et une pipette sérologique de 10 mL pour recouvrir lentement le sang d’une solution à gradient de densité de 1,077 g/mL à température ambiante (RT) (voir le tableau des matières) sans perturber l’interface. Empêchez l’embout de la pipette de toucher le fond du tube. Répétez l’opération pour tous les tubes. Puis centrifuger pendant 30 min à 850 x g, sans freinage, à RT pour assurer le maintien du gradient de densité.
- Visualisez la couche leucocytaire cellulaire au-dessus du gradient de densité de 1,077 g/mL sous la forme d’une couche blanche trouble. Retirer et jeter la couche de plasma jusqu’à environ 10 mL au-dessus de la couche leucocytaire à l’aide d’une pipette sérologique de 25 mL et d’un pipetor automatique.
- Recueillir la couche leucocytaire avec un transfert stérile ou une pipette sérologique. Utilisez la pipette comme une spatule pour gratter les cellules sur le côté du tube conique tout en relâchant le bulbe (ou en pipetant lentement) pour aspirer les cellules vers le haut. Combiner les couches leucocytaires de deux tubes coniques de 50 ml en un nouveau tube conique de 50 ml.
- Lavez les cellules en versant 45 mL de HBSS stérile contenant 2% de FBS dans chaque tube conique. Centrifuger pendant 11 min à 360 x g à TA.
- Aspirer le fluide de lavage jusqu’au granulé dans tous les tubes. Utilisez une pipette sérologique de 10 mL et une pipette automatique pour remettre en suspension la première pastille dans 10 mL de HBSS contenant 2 % de FBS. Remettez chaque pastille en suspension dans les mêmes 10 mL de HBSS et rincez chaque tube avec 10 mL supplémentaires de HBSS pour recueillir toutes les cellules dans un seul tube.
- Verser 45 mL d’HBSS stérile contenant 2% de FBS dans le tube conique. Centrifuger pendant 10 min à 360 x g, à 4 °C.
- Aspirer le tampon de lavage jusqu’à la pastille de la cellule et remettre en suspension la pastille dans 20 ml de tampon séparateur de cellules magnétiques (voir le tableau des matériaux). Prélever une petite partie aliquote pour compter les cellules à l’aide d’un hémocytomètre à une dilution de 1:20 dans du bleu de méthylène. Ajoutez le nombre de cellules bleues et blanches. Centrifuger à 360 x g, pendant 10 min à 4 °C.
REMARQUE : Ce protocole utilise la technologie des billes magnétiques (voir le tableau des matériaux). Le protocole peut être modifié pour être utilisé avec n’importe quelle technologie de séparation cellulaire, avec une pureté et un rendement suffisants des cellules souches CD34+. - Aspirer le surnageant et remettre en suspension la pastille de cellules CD34+ isolée du sang de cordon ombilical dans 300 μL de tampon séparateur de cellules magnétiques par 1 x 108 cellules. Ajouter d’abord 100 μL de réactif bloquant le FcR, puis 100 μL de billes magnétiques CD34+ par 1 x 108 cellules. Incuber à 4 °C pendant 30 min (sans glace).
- Ajouter 5 mL de tampon séparateur de cellules magnétiques par 1 x 108 cellules et faire tourner à 360 x g pendant 10 min à 4 °C. Répéter l’étape de lavage et remettre en suspension la pastille dans 500 μL de tampon séparateur de cellules magnétiques par 1 x 108 cellules dans un tube conique de 15 mL étiqueté « non fractionné ».
- Étiquetez deux autres tubes coniques de 15 mL « CD34- » et « CD34+ ». Placez les trois tubes coniques de 15 ml (non fractionnés, CD34 et CD34+) dans les fentes A1, B1 et C1, respectivement, sur une grille de refroidissement (voir le tableau des matériaux). Séparer les cellules à l’aide du programme de sélection positive à deux colonnes d’un séparateur automatique de cellules magnétiques (voir le tableau des matériaux) dans une ESB, conformément aux instructions de l’instrument du fabricant.
- Expansion et congélation de cellules souches humaines CD34+
- Aliquote 10 μL de la suspension cellulaire CD34+ récupérée (2 mL) sur une lame d’hémocytomètre et compter les cellules sous grossissement 10x. Calculez le nombre total de cellules CD34+ en multipliant le nombre de cellules par 2 x 104. Divisez le nombre total de cellules CD34+ par 250 000 pour calculer le nombre de flacons à congeler (50 000 cellules par bébé de souris avant l’expansion in vitro ).
- Préparer le milieu CB à 10 % de FCS d’Iscove (plus 1 mL supplémentaire pour la perte de filtrage), complété par 40 ng/mL de facteur de cellules souches, 20 ng/mL de Flt3L et 10 ng/mL d’IL-6, et passer à travers un filtre de 0,22 μm. Resuspendre les cellules CD34+ à 100 000 par mL de milieu CB et incuber à 37 °C. Le jour 3, ajouter un volume équivalent de milieu CB sans cytokines dans la fiole contenant les cellules et le milieu CB avec cytokines.
REMARQUE : L’ajout de ces cytokines au milieu CB favorise la survie et l’expansion des cellules CD34+ tout en empêchant la différenciation. - Récoltez les cellules CD34+ expansées au jour 5. Pipeter la suspension cellulaire de haut en bas et recueillir dans un tube conique de 50 mL. Ajouter suffisamment de milieu CB pour couvrir le fond de la fiole. À l’aide d’un grattoir à cellules, gratter tout le fond de la fiole. Recueillir tous les milieux dans le même tube de 50 ml et centrifuger à 360 x g pendant 11 min.
- Remettez les cellules en suspension dans 2 mL de milieu CB. Conservez la goutte finale de la pipette dans une plaque de 96 puits pour le comptage. Diluer les cellules 1:1 dans du bleu de trypan et ajouter 10 μL à l’hémocytomètre, puis compter et faire la moyenne des cellules à partir de quatre quadrants. Calculez le nombre total de cellules CD34+ en multipliant le nombre de cellules par 4 x 104 et notez la viabilité.
- Faire n + 1 mL de milieu de congélation, où n est le nombre de flacons congelés calculé à l’étape 1.3.1. Préparer le milieu de congélation en ajoutant 10 % (v/v) de DMSO au FBS et garder sur la glace. Étiquetez les cryoflacons avec CB#, CD34+ d5 et date.
- Faire tourner les cellules CD34+ à 360 x g pendant 10 min à 4 °C. Aspirer le milieu jusqu’à la pastille et remettre en suspension le granulé de cellule dans un milieu de congélation. Aliquote 1 mL de suspension cellulaire dans chaque flacon et répartir le reste uniformément entre les flacons. Ajouter les flacons dans un congélateur à cellules isopropanol refroidis à 4 °C, placer à -80 °C et transférer dans de l’azote liquide pour conserver pendant >90 jours.
- Irradiation des bébés souris
- Rassemblez les chiots BRGSB/N , 1 à 3 jours après la naissance, dans une boîte en plastique autoclavée avec rembourrage. Ajoutez une petite quantité de litière avec les chiots. Étiquetez la boîte avec le numéro de la cage et le nombre de chiots.
- Installer l’irradiateur (voir le tableau des matières) pour une dose de 300 rad. Placez la boîte de chiots dans l’irradiateur et exposez-les à 300 rad. Ramenez les chiots dans leur cage, placez-les en tas et couvrez-les de litière.
- Injections de chiots et préparation de cellules CD34+
- Commencer la préparation des cellules CD34+ ~3 h après l’irradiation. Réchauffer 10 mL de média CB dans un tube conique de 50 mL. Effectuer toutes les étapes d’une ESB stérile.
- Récupérer un flacon de cellules CD34+ expansées et congelées in vitro pour quatre à six petits à injecter. Décongeler rapidement à 55 °C, jusqu’à ce qu’une petite quantité de glace soit visible, et ajouter les cellules au milieu CB réchauffé (le flacon doit encore être froid au toucher.) Utiliser 1 mL de milieu pour rincer chaque flacon et faire tourner les cellules à 360 x g pendant 12 min à 4 °C.
REMARQUE : La décongélation rapide à 55 °C s’est avérée améliorer la viabilité cellulaire (90 % à 95 %) que la décongélation à 37 °C. - Aspirer le milieu avec précaution. Remettez en suspension la (petite) pastille dans 2 mL de milieu CB, mélangez doucement et ajoutez ~30 μL de la suspension cellulaire à un seul puits dans une plaque de comptage. Diluer 1:1 dans du bleu de trypan, puis ajouter 10 μL à un hémocytomètre et compter et faire la moyenne des cellules de quatre quadrants.
- Calculez le nombre total de cellules CD34+ en multipliant le nombre de cellules par 4 x 104 , puis notez la viabilité. Essorage à 360 x g pendant 12 min à 4 °C.
- Aspirer soigneusement le milieu et remettre en suspension la pastille cellulaire dans 100 μL de PBS stérile par bébé n + 1 à injecter, ce qui donne 250 000 à 450 000 cellules CD34+ par souris. Placez le tube conique sur de la glace dans un conteneur de transport et dirigez-vous vers le vivarium pour l’injection de chiots.
- Apportez une lampe chauffante, des couches, une seringue de 1 mL, une aiguille de 18 G, une aiguille de 30 G et la préparation de cellules CD34+ dans des contenants stériles au vivarium BSC. Placez une couche stérile ~ 2 pieds sous la lampe chauffante. Récupérez la cage avec la litière à injecter et placez-la dans l’ESB.
- Assemblez la seringue à l’aide d’une aiguille biseautée de 18 G. En faisant correspondre l’angle du tube conique avec le biseau de l’aiguille, mélanger délicatement et aspirer la suspension cellulaire. Placez les chiots sur la couche pour les réchauffer (surveillez la surchauffe). Retirez l’air de la seringue et remplacez l’aiguille de 18 G par l’aiguille de 30 G, puis poussez soigneusement la seringue jusqu’à ce que la suspension cellulaire soit juste à l’extrémité de l’aiguille.
REMARQUE: Alternativement, une seringue à insuline peut être utilisée. - Emmenez un chiot à la fois au bord de la couche, loin de la lampe chauffante. Immobilisez le chiot sur le côté sous le pouce et l’index, permettant une vue claire du visage. Remarquez la veine à travers la joue sous l’endroit où l’oreille sera. Insérez l’aiguille peu profondément dans la veine (IV) la plus proche de l’œil et injectez lentement 50 μL de cellules.
- Vérifiez si une bulle se forme à partir d’une injection sous-cutanée. Si c’est le cas, insérez l’aiguille plus profondément et procédez à l’injection de cellules. Une petite goutte de sang / hématome sera visible en cas de succès.
NOTE: Se référer à la procédure de Gombash Lampe et al.24 concernant l’injection de chiots. - Avec le chiot toujours immobilisé, effectuer une injection intrahépatique (IH) avec 50 μL supplémentaires de cellules (100 μL au total par chiot de IV + IH). Le foie peut être visualisé comme une tache sombre entre la bande de lait blanc et la cage thoracique. Placez le chiot injecté sur une couche plus éloignée des chiots non injectés et chauffez.
REMARQUE: L’injection IV entraîne un meilleur chimérisme à long terme que l’injection d’HI seule25, mais les injections IV ne sont pas toujours réussies. Par conséquent, la division de l’injection entre IV et IH assure la greffe dans un plus grand pourcentage de souris. - Répétez les injections de veines faciales et d’IH pour tous les chiots. Nettoyez tout sang, remettez les chiots au nid dans leur cage et couvrez-les de litière.
2. Tester le chimérisme humain dans le sang
- Testez le chimérisme dans le sang de souris HIS à l’âge de 10 et 14 semaines. Prélever 50 μL de sang par la veine rétro-orbitaire ou en utilisant une autre méthode approuvée par l’IACUC.
- Pour les saignements rétro-orbitaux, anesthésiez les souris avec un vaporisateur d’isoflurane réglé sur 5 pendant 1-2 minutes, puis réglez le réglage du vaporisateur sur 4. Baissez le vaporisateur au besoin pour permettre aux souris de rester suffisamment oxygénées sous anesthésie. Ne pas garder les souris sous isoflurane pendant plus de 5 minutes.
- Placez le nez de la souris anesthésiée dans le cône nasal du vaporisateur d’isoflurane et placez une goutte de l’analgésique (solution ophtalmique de HCl à 0,5% de proparacaïne USP) sur l’œil.
- Après 1 min, retirer la proparacaïne à l’aide d’une gaze stérile, proptose l’œil et insérer un tube hématocrite hépariné de 75 mm rétro-orbitalement pour recueillir 50 μL de sang. Éjecter le sang dans un tube à microfuge de 1,5 mL contenant 50 μL d’héparine et mélanger doucement.
- Pincez l’œil fermé avec de la gaze stérile pour arrêter le saignement et appliquez une goutte de proparacaïne. Retirez la souris de l’isoflurane et récupérez dans une cage propre.
- Isolement PBMC du sang de souris
- Mélanger le sang/héparine en pipitant doucement de haut en bas et en superposant lentement sur 500 μL de gradient de densité de 1,077 g/mL, en prenant soin de ne pas perturber l’interface. Centrifuger les tubes à 1 220 x g pendant 20 min à TA sans freins.
- Visualisez la couche leucocytaire cellulaire comme une couche trouble au-dessus du gradient de densité de 1,077 g/mL, sous le plasma. Retirer le plus grand nombre possible de cellules de la couche leucocytaire à l’aide d’une pipette de 200 μL et les ajouter à de nouveaux tubes de 1,5 mL contenant 750 μL de milieu de récolte. Centrifuger à 360 x g pendant 11 min à RT.
- Aspirer le milieu jusqu’à 50 μL et remettre en suspension le granulé dans 750 μL de milieu de récolte. Centrifuger à 360 x g pendant 10 min à 4 °C, puis aspirer à nouveau jusqu’à 50 μL. Les cellules sont prêtes à être remises en suspension dans la tache de surface.
- Coloration de surface et analyse cytométrique en flux
- Remplissez la feuille de travail sur les panneaux de coloration (tableau 1; « panneau de purge spectrale » avec les numéros de souris et préparer le cocktail de coloration des anticorps en ajoutant tous les anticorps fluorescents au tampon de coloration (dossier supplémentaire 1). Faites une disposition de plaque pour ajouter les échantillons à une plaque de fond en U de 96 puits. Marquez le fond des puits à l’aide d’un marqueur permanent. Titrer les anticorps en utilisant une procédure standard avant la coloration afin de déterminer la concentration appropriée26.
- Ajouter 62 μL du cocktail de taches de surface à chaque puits de la plaque de fond en U de 96 puits. Remettez en suspension les cellules dans les 50 μL restant dans le tube et ajoutez chaque échantillon à son puits correspondant. Inclure un puits pour un contrôle positif de la coloration (PBMC humains + splénocytes de souris, 1 x 106 cellules chacun) pour colorer à côté des échantillons. Mélanger par pipetage et incuber le mélange pendant 15 min à 4 °C.
- Centrifuger à 680 x g pendant 3 min à 4 °C. Sortez la plaque dans l’évier pour retirer le surnageant et lavez les cellules en remettant en suspension 150 μL de tampon de coloration en pipetant doucement ~10x. Faire tourner et remettre en suspension chaque puits dans 150 μL de tampon de coloration.
- Acquérir les données pour 100 μL de chaque échantillon sur un cytomètre en flux (voir le tableau des matériaux) et exporter les fichiers .fcs. Importez les fichiers .fcs dans le logiciel d’édition des données de flux (voir Table des matières). Appliquez une porte polygonale sur une parcelle FSC-A x SSC-A entourant les cellules, à l’exclusion de tout débris. Sélectionnez les cellules à l’intérieur de la porte (voir le tableau 1; feuille de travail « Spectral Flow Bed Gating »).
- Changez les axes en (FSC-A x FSC-H) et grillez les cellules contenues sur la diagonale linéaire à l’exclusion des doublets qui dépassent de la ligne. Sélectionnez ces cellules et remplacez les axes par (hCD45 x mCD45).
- Appliquez une porte polygonale à la population hCD45+ et appliquez le nom « humain ». Appliquez une porte polygonale à la population mCD45+ et appliquez le nom « souris ». Créez une statistique de comptage pour les populations humaines et de souris.
- Sélectionnez la population humaine et remplacez les axes par (CD19 x CD3). Appliquez une porte polygonale aux cellules CD19+ et nommez-la « cellules B ». Appliquez une porte polygonale à la population de CD3+ et nommez-la « lymphocytes T ». Appliquez une porte polygonale à la population doublement négative et nommez-la « NonTB ».
- Sélectionnez la population de lymphocytes T et remplacez les axes par (CD8 x CD4). Appliquez une porte polygonale aux populations CD4 et CD8 positives et nommez-les respectivement « CD4+ » et « CD8+ ».
- Sélectionnez la population non tuberculeuse et remplacez les axes par (CD56 x myéloïde). Appliquez une porte polygonale à la population totale de CD56 positifs, y compris les événements doublement positifs, et nommez-la « cellules NK ». Appliquez une porte polygonale à la population CD56 négative et myéloïde positive et nommez-la « myéloïde ».
- Créez un tableau pour les statistiques de pourcentage et de dénombrement de toutes les populations et exportez-les vers un tableur (voir Tableau des matières). Calculer %hCD45 chimérisme = %hCD45/(%hCD45 + mCD45).
- Exclure les souris HIS qui sont <20% hCD45+ (de mCD45 + hCD45) pour d’autres expériences.
REMARQUE : Dans cette étude, les PBMC ont été préparés en utilisant une séparation par gradient de densité de 1,077 g/mL pour un appauvrissement plus propre des globules rouges. Cette procédure excluait les granulocytes humains et de souris de la couche PBMC. Alternativement, la lyse de RBC peut être utilisée.
3. Injection de tumeurs chez la souris
- Avant l’injection de la tumeur, confirmez le nombre de lymphocytes T à partir des données de saignement de 14 semaines. Les tumeurs sont injectées pour être prêtes à être récoltées entre 20 et 26 semaines si les cellules T représentent >20% et entre 24 et 28 semaines si les cellules T représentent <20% de la population totale de cellules immunitaires. Ce moment d’injection garantit que les souris sont âgées de 20 à 28 semaines et ont un nombre suffisant de cellules T à la fin de l’étude.
- Injection de xénogreffes dérivées de lignées cellulaires (CDX)
REMARQUE : La procédure est décrite à l’aide des cellules d’adénocarcinome du sein MDA-MB-231 (voir le tableau des matières) à titre d’exemple.- Décongeler une partie aliquote des cellules congelées, laver 1x en remettant en suspension dans 10 mL de DMEM additionné de 10 % de FBS, 1 % de PenStrep et 1 % d’acides aminés non essentiels, et centrifuger à 360 x g pendant 10 min à 4 °C. Aspirer le milieu et remettre en suspension dans 10 mL de DMEM supplémenté de 10% de FBS, 1% PenStrep et 1% d’acides aminés non essentiels dans une fiole T25 et incuber à 37 °C avec 5% de CO2.
REMARQUE: Les lignées cellulaires sont authentifiées par PCR pour assurer le bon type de cellule. Les surnageants cellulaires sont testés pour les mycoplasmes via un test biochimique avant l’injection. - Passage et expansion des cellules pendant la phase de croissance exponentielle à environ 80% de confluence.
- Aspirer le milieu, rincer les cellules avec 5 à 10 mL de PBS stérile (pH 7,2) et incuber avec 1 mL de trypsine-EDTA à 0,25 % pendant 1 à 5 minutes jusqu’à ce que les cellules se détachent de la fiole.
- Ajouter 5 mL du même DMEM et mélanger en pipetant de haut en bas. Passer la totalité des 6 mL de suspension cellulaire dans une nouvelle fiole T75 traitée par culture tissulaire (dilution 1:3) et ajouter 10 mL supplémentaires de DMEM.
- Passage des cellules tous les 2-3 jours à 80% de confluence à 1:3 placage dilutionnel dans DMEM pour dilater les cellules.
- Récoltez les cellules avec 0,25% de trypsine-EDTA pendant la phase de croissance exponentielle en six passages et lavez avec du PBS. Mélanger le PBS et l’extrait de membrane basale (voir le tableau des matériaux) dans un rapport de 1:1 et ajouter aux cellules à une concentration finale de 5 x 107 cellules/mL.
- Anesthésier les souris avec de l’isoflurane comme décrit à l’étape 2.1.1. Injecter par voie sous-cutanée 100 μL (5 x 106 cellules) de suspension de cellules tumorales dans chaque flanc à l’aide d’une aiguille de 23 G.
- Décongeler une partie aliquote des cellules congelées, laver 1x en remettant en suspension dans 10 mL de DMEM additionné de 10 % de FBS, 1 % de PenStrep et 1 % d’acides aminés non essentiels, et centrifuger à 360 x g pendant 10 min à 4 °C. Aspirer le milieu et remettre en suspension dans 10 mL de DMEM supplémenté de 10% de FBS, 1% PenStrep et 1% d’acides aminés non essentiels dans une fiole T25 et incuber à 37 °C avec 5% de CO2.
- Effectuer l’injection de xénogreffes dérivées du patient (PDX) à l’aide de trocarts. La procédure d’injection et d’autres instructions de développement et de maintenance pour le modèle PDX sont disponibles dans la littérature27.
4. Mesure de la croissance tumorale
- Vérifiez la progression tumorale une fois par semaine après l’implantation en palpant le long du flanc pour la croissance tumorale. Une fois que les tumeurs sont palpables, anesthésiez les souris avec de l’isoflurane (comme décrit à l’étape 2.1.1) et rasez-les sur chaque flanc avec une tondeuse électrique, en prenant soin autour de la tumeur pour prévenir les ulcérations à mesure que la tumeur se développe.
REMARQUE: Les souris peuvent être rasées avant l’injection de la tumeur pour prévenir les blessures à la peau autour des tumeurs; Cependant, le taux de repousse des cheveux pourrait obstruer les mesures tumorales. - Mesurez la longueur et la largeur des tumeurs deux fois par semaine à l’aide d’étriers et enregistrez les mesures en mm. Rapportez les mesures tumorales en volume tumoral (mm3) en utilisant la formule
 . Veillez à ne pas laisser la charge tumorale dépasser 2 000mm3 par tumeur unique ou un volume combiné de 3 000 mm3.
. Veillez à ne pas laisser la charge tumorale dépasser 2 000mm3 par tumeur unique ou un volume combiné de 3 000 mm3.
REMARQUE : Les lignes directrices pour l’utilisation des souris dans la recherche sur le cancer varient selon l’emplacement. Veuillez consulter les politiques de soin et d’utilisation des animaux de l’établissement du chercheur.
5. Traitements médicamenteux
- Répartir les souris HIS dans des groupes de traitement équivalents en fonction du chimérisme hCD45, du chimérisme hCD3 et du chimérisme hCD8. Une fois que les tumeurs atteignent 100 mm3 en moyenne, commencez les traitements médicamenteux.
NOTE: Le nombre de souris par groupe est basé sur le nombre de souris HIS générées à partir du CB. Un minimum de quatre souris par groupe est recommandé. L’appariement multigroupe non bipartite (par exemple, dans le logiciel R-vivo de Manille) est utile pour ce groupe28. - Voie et fréquence des médicaments
- Injecter des inhibiteurs anti--1 (nivolumab, pembrolizumab) par voie intrapéritonique (i.p.) à raison de 30 mg/kg 1x par semaine ou 20 mg/kg 2x par semaine pour les traitements uniques, ou 10-15 mg/kg 2x par semaine pour les traitements combinés.
- Doser les thérapies combinées, telles que les thérapies ciblées, les chimiothérapies et l’irradiation, conformément à la conception expérimentale. Les thérapies ciblées et les chimiothérapies peuvent être administrées par gavage oral, injection intraveineuse ou nourriture.
REMARQUE: Les doses peuvent être modifiées et optimisées en fonction des données publiées et du modèle tumoral. Les études posologiques sont souvent testées chez des receveurs immunodéficients avant les études sur les souris SIH.
- Surveillez les souris au moins 3 fois par semaine pour détecter les changements de santé tels que la perte de poids, les excréments lâches, la posture voûtée, la mobilité réduite et la perte de fourrure. Certains symptômes peuvent être des signes de toxicité médicamenteuse ou de GVH, et la posologie du médicament peut devoir être réduite ou arrêtée. Euthanasier les souris au besoin conformément aux politiques de soin et d’utilisation des animaux.
6. Prélèvement de tissus et de tumeurs de souris à la fin de l’étude
- Euthanasier les souris individuellement selon les directives institutionnelles et vétérinaires en utilisant du gaz CO 2 comprimé avec un débit de 2,75 L/min. Surveillez les souris maintenues dans le CO2 pendant 1 minute après la mort, puis effectuez une luxation cervicale comme forme secondaire d’euthanasie.
- Collecte de sang
- Prélever du sang par ponction intracardiaque. Tenez la souris en position verticale et insérez une seringue de 1 mL avec une aiguille de 25 G directement dans le cœur à partir d’une ligne juste à gauche de la ligne médiane et au-dessous des côtes. Recueillir le sang dans une seringue idéalement en un long tirage et transférer dans un tube marqué de 1,5 mL.
REMARQUE: Des insertions d’aiguilles supplémentaires peuvent être effectuées pour prélever du sang supplémentaire dans les coins de la cavité pulmonaire avec soin. - Laisser le sang à 4 °C pendant 1 h, puis centrifuger le sang dans une microcentrifugeuse pendant 6 min à 7 800 x g. Recueillir le liquide clair (sérum) au-dessus de l’interface sanguine dans un deuxième tube propre et étiqueté de 1,5 mL. Conservez le sérum à -20 °C pour l’analyse en aval, comme les titres de cytokines inflammatoires ou d’immunoglobulines humaines et souris.
- Prélever du sang par ponction intracardiaque. Tenez la souris en position verticale et insérez une seringue de 1 mL avec une aiguille de 25 G directement dans le cœur à partir d’une ligne juste à gauche de la ligne médiane et au-dessous des côtes. Recueillir le sang dans une seringue idéalement en un long tirage et transférer dans un tube marqué de 1,5 mL.
- Dissection tissulaire
- Après l’injection et la croissance de cellules tumorales chez les souris et l’administration de traitements médicamenteux, récoltez les tissus. Placez les souris sur une planche de dissection en mousse, avec des épingles pour les maintenir en place, et les bras et les jambes étendus à des angles de 45°. Faites une incision au milieu du torse, en commençant près du bassin et en s’étendant jusqu’au menton, en essayant d’éviter de couper le péritoine (bien que ce ne soit pas essentiel). Tirez la peau vers le bord et maintenez-la en place avec des épingles.
- Extraire les ganglions lymphatiques (LN) à l’aide de pinces fines dans l’ordre suivant : inguinale, axillaire, cervicale, mésentérique, hiatale.
REMARQUE: Chez les souris SEH, les LN sont souvent très petits, ressemblant à un anlage, ou « enflammés » avec une apparence distincte contrairement à celle d’une souris de type sauvage. Par conséquent, des tissus ressemblant à des LN sont prélevés sur chaque site. Les LN périphériques apparaissent souvent comme des « boules » remplies de liquide, tandis que les LN mésentériques sont plus denses. Les LN mésentériques sont les plus évidents et les plus cohérents et sont observés comme un ou deux nœuds plus denses distincts, par opposition à une chaîne. - Placer les LN sur un côté des lames de verre dépoli dans 8 ml de milieu de récolte dans une boîte de Pétri. En tenant les lames à des angles perpendiculaires avec les bords givrés vers l’intérieur, appuyez doucement sur les tissus jusqu’à ce que le contenu cellulaire soit libéré.
- Rincez les lames plusieurs fois en les séparant et en les rassemblant pour libérer le maximum de cellules. Rassemblez les cellules à l’aide d’une pipette en verre de 5 po et filtrez-les à travers une pipette de 9 po en coton dans un tube conique étiqueté de 15 mL.
- Extraire la rate du côté supérieur gauche de l’abdomen à l’aide de deux paires de forceps ou de forceps et de ciseaux. Notez la taille de la rate comme estimation du volume de milieu de remise en suspension. Recueillir et filtrer la rate par digestion mécanique à l’aide de lames de verre dépoli, comme avec les LN.
NOTE: Toute technique de préparation de tissus pour suspensions unicellulaires peut être utilisée. - Effectuer le comptage et la remise en suspension des cellules à partir d’échantillons de tissus comme décrit ci-dessous.
- Centrifuger les cellules lymphatiques à 360 x g pendant 10 min à 4 °C. Aspirer le liquide et remettre en suspension la pastille cellulaire dans 1 mL de milieu de récolte avec de la DNase.
- Retirer les érythrocytes des cellules de la rate en incubant avec 3 mL de tampon de lyse ACK à TA pendant 3 min, puis en ajoutant 10 mL de milieu de récolte/DNase. Centrifuger à nouveau les cellules de la rate, aspirer le surnageant et remettre les cellules en suspension dans 1 à 10 mL de milieu de récolte/DNase, en fonction de la taille de la rate (p. ex. 1 mL pour les très petites rates et jusqu’à 10 mL pour les plus grosses rates).
- Ajouter une partie aliquote de 10 μL de suspension cellulaire à 90 μL de milieu et compter sur un hémocytomètre. Centrifuger les LN et les cellules de la rate, aspirer les surnageants et remettre les cellules en suspension dans le milieu de récolte à une concentration de 1 x 108 cellules/mL, soit un minimum de 80 μL.
- Extraire les tumeurs.
- Retirez la tumeur du flanc ouvert en tenant la tumeur avec une pince tout en coupant lentement les bords de la tumeur avec des ciseaux de dissection.
- Une fois la tumeur enlevée, pesez-la et retirez 1/4 pour le traitement de l’ARN et de l’immunohistochimie (IHC). Diviser la tumeur 1/4 en deux; placer la moitié (1/8 de la tumeur entière) dans un cryoflacon, congeler dans le liquide N2, et conserver à -80 °C pour les études génomiques en aval.
- Placez l’autre 1/8 de la tumeur dans un tube d’échantillon marqué contenant 10% de formol. Le lendemain, rincer et remettre en suspension le tissu dans de l’éthanol à 70% jusqu’à une utilisation ultérieure. Placer les 3/4 restants de la tumeur dans un plat de 6 cm et hacher en morceaux de ~1 mm à l’aide d’une lame de scalpel.
- Transporter les morceaux tumoraux dans un tube de dissociation (voir le tableau des matériaux), rincer le plat avec 5 mL de milieu leucocytaire infiltrant la tumeur incomplète (TIL) et ajouter au tube de dissociation.
REMARQUE: Pour les tumeurs pesant >0,4 g, rincer le plat avec 10 ml de milieu incomplet TIL et ajouter au tube de dissociation. - Ajouter la préparation de collagénase (voir le tableau des matières) à une concentration finale de 50 mg/mL dans le tissu dans le tube de dissociation. Dissocier le tissu par dissociation mécanique à 37 °C pendant 30 min à 1 h, selon la fermeté de la tumeur.
- Après dissociation, passer la suspension sur un filtre de 100 μm dans un tube conique de 50 mL et rincer le filtre avec 10 mL de milieu complet TIL. Le sérum dans ce milieu arrêtera la réaction de collagénase et protégera les cellules contre une dégradation ultérieure.
- Centrifuger la suspension unicellulaire à 360 x g pendant 10 min à 4 °C. Remettez le granulé en suspension dans juste assez de milieu de récolte avec de la DNase pour que la suspension cellulaire puisse facilement passer à travers une pointe de pipette P1000 et enregistrer le volume pour l’analyse en aval.
REMARQUE: Un volume plus petit augmente la collecte de cellules immunitaires tumorales mais est plus sensible aux obstructions du cytomètre en flux.
7. Coloration cellulaire et analyses cytométriques en flux
- Préparation des taches et placage cellulaire
- Préparez des cocktails de coloration : Le jour de la récolte, ajoutez le nombre d’échantillons à la feuille de travail de coloration (tableau 2; Feuilles de travail « Panneau Flux A conventionnel », « Panneau B à flux spectral » et « Panneau Flux C conventionnel ») pour toutes les taches et impressions. Préparer des cocktails de taches de surface pour les taches A et B dans un tampon de coloration (SB) en ajoutant chaque anticorps individuellement avec un nouvel embout, en marquant chaque réactif en déplacement, et conserver à 4 °C jusqu’à ce que vous en ayez besoin. Préparer les colorants de viabilité appropriés dans du PBS sans azoture et conserver à 4 °C jusqu’à ce qu’il soit nécessaire (chaud à TA avant utilisation). Préparer le cocktail de la tache de surface C le jour 2 en SB avec les taches intracellulaires B et C dans leurs tampons de perméabilisation respectifs.
- Créer une disposition de plaque pour tous les échantillons de la feuille de coloration et allouer 100 μL de PBS sans azoture aux puits. Inclure des témoins non colorés pour chaque tissu et coloration (pour la cytométrie spectrale).
- Ajouter les cellules à des plaques de 96 puits contenant 50 μL de PBS. Pour la coloration A, remettre en suspension chaque groupe de tissu dans le volume approprié, comme indiqué sur la feuille de coloration, et conserver à 4 °C jusqu’à l’acquisition sur le cytomètre en flux le même jour. Pour les taches B et C, ajouter 25 μL de lymphe et 60 μL de suspensions de cellules tissulaires non lymphatiques aux puits avec PBS.
- Effectuer une stimulation in vitro des cytokines pour la détection par coloration intracellulaire à l’aide de la coloration C.
- Centrifuger la plaque de coloration C 96 puits de l’étape 7.1.3 à 680 x g pendant 3 min à 4 °C. Ajouter 200 μL de milieu complet TIL (RPMI 1640, HEPES 10 mM [pH 7], 10% FBS) à chaque puits. Conserver la plaque avec la suspension cellulaire à 4 °C pendant la nuit.
- Tôt le lendemain matin, diluer le cocktail de stimulation cellulaire (voir le tableau des matériaux) 1:500 dans un milieu TIL complet. Centrifuger la plaque contenant la suspension cellulaire à 680 x g pour granuler les cellules et effleurer pour retirer le média. Resuspendre les cellules dans 200 μL du cocktail de stimulation cellulaire préparé et incuber à 37 °C pendant 1 h.
- Ajouter 25 μL de solution inhibitrice du transport des protéines contenant du monensin à une dilution de 1:1 000 dans un milieu TIL complet, mélanger les cellules et incuber à 37 °C pendant 4 heures supplémentaires pour permettre l’accumulation intracellulaire de cytokines.
- Effectuer la coloration cellulaire.
- Pour les colorations A et B (Jour 1), et pour la coloration C (Jour 2), centrifuger à 680 x g pendant 3 min à 4 °C. Agitez les plaques et ajoutez les colorants de viabilité appropriés aux puits. Bien mélanger en pipotant doucement de haut en bas avec une pipette multicanal et d’incuber pendant 15 min à TA.
- Centrifuger les plaques (comme à l’étape 7.1.5.1), puis ajouter les taches de surface appropriées sur les plaques et mélanger délicatement en pipetant avec une pipette multicanaux. Incuber pendant 15 min à 4 °C et centrifuger à nouveau. Agiter les plaques, laver les cellules en pipitant doucement de haut en bas avec 150 μL de SB et centrifuger.
- Agiter les plaques, répéter le lavage en pipitant doucement de haut en bas avec 150 μL de SB et centrifuger à 680 x g pendant 3 min à 4 °C. Pour la coloration A, remettre en suspension chaque groupe de tissu dans le volume approprié, comme indiqué sur la feuille de coloration, et conserver à 4 °C. Pour la tache B, corriger avec le fixateur du kit de facteur de transcription FoxP3 (voir le tableau des matériaux) pendant 30 minutes à TA. Pour la coloration C, fixer dans 1% (v/v) de paraformaldéhyde dans SB pendant 30 min à TA.
- Centrifuger les plaques fixes à 480 x g pendant 3 min à 4 °C. Lavez 1x dans SB. Pour la tache B, les cellules peuvent être laissées pendant la nuit.
- Perméabiliser les cellules : remettre les puits en suspension dans 150 μL de tampon de perméabilisation du kit de facteur de transcription FoxP3 pour la coloration B et dans 0,5 % (p/v) de saponine dans SB pour la coloration C. Incuber pendant 15 min à TA. Centrifuger les plaques fixes à 480 x g pendant 3 min à 4 °C.
- Agitez les assiettes et ajoutez les cocktails de coloration intracellulaire respectifs pour la tache B et la tache C. Incuber pendant 30 min à TA.
- Centrifuger les plaques fixes à 480 x g pendant 3 min à 4 °C et agiter les plaques. Laver les cellules avec 150 μL des tampons de perméabilisation correspondants (tache B, tampon de perméabilisation du kit de facteur de transcription FoxP3; coloration C, saponine).
- Agiter les plaques et laver en pipetant de haut en bas dans 150 μL de SB. Centrifuger à 480 x g pendant 3 min à 4 °C. Agiter les plaques et remettre en suspension les cellules de SB par groupe de tissus dans les volumes appropriés indiqués sur la feuille de travail de coloration.
REMARQUE: Les échantillons sont maintenant prêts pour l’acquisition par cytométrie spectrale. - Installez le cytomètre en flux (voir le tableau des matériaux) avec des témoins à coloration unique appropriés pour chaque coloration et des échantillons non colorés pour chaque groupe de tissus afin de tenir compte des différences d’autofluorescence pendant le démélange. Procurez-vous les volumes appropriés par groupe de tissus, tel que défini dans la feuille de calcul de coloration (Tableau 2) et exportez les fichiers .fcs.
- Analyse des données de cytométrie en flux
- À l’aide du logiciel d’analyse de cytométrie en flux (voir Tableau des matériaux), créez un nouvel espace de travail. Créez de nouveaux groupes pour chaque organe (LN, rate et TIL). Importez les fichiers .fcs de chaque organe dans le groupe.
- Créez un diagramme à points bivarié, définissez les axes sur (FSC-A x SSC-A) et appliquez une porte polygonale aux événements cellulaires, en évitant les événements sur les bords (porte toutes les cellules). Sélectionnez les événements cellulaires, remplacez les axes par (FSC-A x FSC-H) et appliquez une porte polygonale aux événements de la diagonale linéaire, en excluant les événements qui s’écartent de la diagonale pour générer la porte « singlets ». Sélectionnez la porte « singlets » et changez les axes en (hCD45 x mCD45). Appliquez des portes polygonales aux événements positifs hCD45 et mCD45 et nommez-les respectivement « humain » et « souris ».
- Sélectionnez la population humaine et changez l’axe des Y en Aqua vivante/morte. Appliquez une porte polygonale à la population positive hCD45 négative vivante/morte et nommez-la « humain vivant ». Sélectionnez la population de souris et changez l’axe X en Live/Dead Aqua. Appliquez une porte polygonale à la population positive mCD45 négative Live/Dead et nommez-la « souris vivante ».
- De la même manière, sélectionnez la porte parente et les axes X et Y, comme indiqué dans les feuilles de travail « Point de contrôle du flux A conventionnel », « Grille du flux spectral B » et « Points de contrôle du flux C conventionnel » (tableau 2) pour isoler les populations indiquées (p. ex. cellules B humaines, cellules T activées).
REMARQUE : Un point de contrôle représentatif pour chaque tache (Bleed, A, B, C) est inclus dans le dossier supplémentaire 2. - Créer des tableaux d’exportation de numération et de fréquence dans le logiciel d’analyse de cytométrie en flux pour toutes les populations et exporter vers un tableur. La population mère pour les fréquences est indiquée dans le tableau 2.
- Utilisez les données pour générer des graphiques basés sur des groupes de traitement expérimentaux.
REMARQUE: Les données peuvent également être analysées à l’aide de packages R dans le logiciel d’analyse. Un seul fichier .fcs de tous les échantillons à analyser peut être créé à l’aide de la fonction de concaténation. Ces données peuvent être réduites dimensionnellement avec l’algorithme T-SNE tout en ajoutant des paramètres de mots-clés pour le type de tissu et le groupe de traitement. L’algorithme FlowSOM peut ensuite être utilisé pour regrouper les populations et l’outil ClusterExplorer peut être utilisé pour identifier les populations. Les nouvelles populations cellulaires peuvent être identifiées de cette manière, comparées visuellement et quantifiées entre les groupes de traitement ou au sein de divers tissus. - Corréler les paramètres immunitaires pour les tumeurs au sein du même groupe de traitement, avec la croissance de cette tumeur pour définir les immunotypes qui sont en corrélation avec l’inhibition de la croissance tumorale. Quantifier la croissance tumorale par le taux de croissance spécifique (SGR) de cette tumeur, une mesure qui prend en compte la différence de volume tumoral sur une période donnée. Cette mesure normalise les tumeurs récoltées à des jours différents en raison de la santé de la souris et des dates de début du traitement.
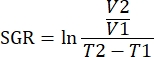
Résultats
Conformément au protocole de tumeur du flanc et à la chronologie expérimentale (Figure 1), la croissance tumorale et la réponse immunitaire à un traitement ciblé par inhibiteur de la tyrosine kinase (ITK) et à un traitement combiné par nivolumab ont été étudiées dans deux PDX distincts de cancer colorectal humain (CCR). Les médicaments TKI ont été étudiés chez des hôtes immunodéficients pour évaluer la croissance tumorale seulement29. Ce modèle a ...
Discussion
Au cours des 6 dernières années, en utilisant notre expertise en immunologie et en souris humanisées, notre équipe de recherche a développé un modèle préclinique indispensable pour tester des immunothérapies sur une variété de tumeurs humaines 3,7,30,31. Ce protocole met l’accent sur la prise en compte de la variabilité du modèle, avec une attention particulière aux populations ...
Déclarations de divulgation
Aucun.
Remerciements
Nous tenons à remercier à la fois l’Animal Research Facility (OLAR) pour les soins qu’ils ont prodigués à nos souris et la ressource partagée en cytométrie en flux soutenue par la subvention de soutien au Centre de cancérologie (P30CA046934) de notre institut pour leur immense aide dans tout notre travail. Nous remercions également Gail Eckhardt et Anna Capasso pour nos collaborations inaugurales sur l’étude des immunothérapies aux PDX humains dans notre modèle HIS-BRGS. Cette étude a été financée en partie par la subvention de soutien du centre de cancérologie P30CA06934 des National Institutes of Health avec l’utilisation de la ressource partagée PHISM (Pre-clinical Human Immune System Mouse Models), RRID: ressource partagée de cytométrie SCR_021990 et de flux, RRID: SCR_022035. Cette recherche a été financée en partie par le NIAID des National Institutes of Health sous le numéro de contrat 75N93020C00058.
matériels
| Name | Company | Catalog Number | Comments |
| 1 mL syringe w/needles | McKesson | 1031815 | |
| 15 mL tubes | Grenier Bio-One | 188271 | |
| 2-mercaptoethanol | Sigma | M6250 | |
| 50 mL tubes | Grenier Bio-One | 227261 | |
| AutoMACS Pro Separator | Miltenyi | 130-092-545 | |
| BD Golgi Stop Protein Transport Inhibitor with monensin | BD Bioscience | BDB563792 | |
| BSA | Fisher Scientific | BP1600100 | |
| Cell Stim Cocktail | Life Technologies | 509305 | |
| Chill 15 Rack | Miltenyi | 130-092-952 | |
| Cotton-plugged glass pipettes | Fisher Scientific | 13-678-8B | |
| Cultrex Basement membrane extract | R&D Systems | 363200502 | |
| Cytek Aurora | Cytek | ||
| DNase | Sigma | 9003-98-9 | |
| eBioscience FoxP3/Transcription Factor Staining Buffer Set | Invitrogen | 00-5523-00 | |
| Embryonic Stemcell FCS | Gibco | 10439001 | |
| Eppendorf Tubes; 1.5 mL volume | Grenier Bio-One | 616201 | |
| Excel | Microsoft | ||
| FBS | Benchmark | 100-106 500mL | |
| Ficoll Hypaque | GE Healthcare | 45001752 | |
| FlowJo Software | BD Biosciences | ||
| Forceps - fine | Roboz Surgical | RS5045 | |
| Forceps normal | Dumont | RS4919 | |
| Formaldehyde | Fisher | F75P1GAL | |
| Frosted Glass Slides | Corning | 1255310 | |
| Gentlemacs C-Tubes | Miltenyi | 130-096-334 | |
| GentleMACS Dissociator | Miltenyi | 130-093-235 | |
| glass pipettes | DWK Life Sciences | 63A53 | |
| Glutamax | Gibco | 11140050 | |
| HBSS w/ Ca & Mg | Sigma | 55037C | |
| HEPES | Corning | MT25060CI | |
| IgG standard | Sigma | I2511 | |
| IgM standard | Sigma | 401108 | |
| IMDM | Gibco | 12440053 | |
| Liberase DL | Roche | 5466202001 | |
| LIVE/DEAD Fixable Blue | Thermo | L23105 | |
| MDA-MB-231 | ATCC | HTB-26 | |
| MEM | Gibco | 1140050 | |
| mouse anti-human IgG-AP | Southern Biotech | JDC-10 | |
| mouse anti-human IgG-unabeled | Southern Biotech | H2 | |
| mouse anti-human IgM-AP | Southern Biotech | UHB | |
| mouse anti-human IgM-unlabeled | Southern Biotech | SA-DA4 | |
| MultiRad 350 | Precision X-Ray | ||
| PBS | Corning | 45000-446 | |
| Pen Strep | Gibco | 15140122 | |
| Petri Dishes | Fisher Scientific | FB0875713A | |
| p-nitrophenyl substrate | Thermo | 34045 | |
| PRISM | Graphpad | ||
| Rec Hu FLT3L | R&D systems | 308-FK-005/CF | |
| Rec Hu IL6 | R&D systems | 206-IL-010/CF | |
| Rec Hu SCF | R&D systems | 255SC010 | |
| RPMI 1640 | Corning | 45000-39 | |
| Saponin | Sigma | 8047-15-2 | |
| Scissors | McKesson | 862945 | |
| Serological pipettes 25 mL | Fisher Scientific | 1367811 | |
| Sterile filter | Nalgene | 567-0020 | |
| Sterile molecular water | Sigma | 7732-18-5 | |
| Yeti Cell Analyzer | Bio-Rad | 12004279 | |
| Zombie Green | biolegend | 423112 |
Références
- Chulpanova, D. S., Kitaeva, K. V., Rutland, C. S., Rizvanov, A. A., Solovyeva, V. V. Mouse tumor models for advanced cancer immunotherapy. International Journal of Molecular Sciences. 21 (11), 4118 (2020).
- Olson, B., Li, Y., Lin, Y., Liu, E. T., Patnaik, A. Mouse models for cancer immunotherapy research. Cancer Discovery. 8 (11), 1358-1365 (2018).
- Marin-Jimenez, J. A., et al. Testing cancer immunotherapy in a human immune system mouse model: correlating treatment responses to human chimerism, therapeutic variables and immune cell phenotypes. Frontiers in Immunology. 12, 607282 (2021).
- Yin, L., Wang, X. J., Chen, D. X., Liu, X. N., Wang, X. J. Humanized mouse model: a review on preclinical applications for cancer immunotherapy. American Journal of Cancer Research. 10 (12), 4568-4584 (2020).
- Cogels, M. M., et al. Humanized mice as a valuable pre-clinical model for cancer immunotherapy research. Frontiers in Oncology. 11, 784947 (2021).
- Jin, K. T., et al. Development of humanized mouse with patient-derived xenografts for cancer immunotherapy studies: A comprehensive review. Cancer Science. 112 (7), 2592-2606 (2021).
- Capasso, A., et al. Characterization of immune responses to anti-PD-1 mono and combination immunotherapy in hematopoietic humanized mice implanted with tumor xenografts. Journal for Immunotherapy of Cancer. 7 (1), 37 (2019).
- Wang, M., et al. Humanized mice in studying efficacy and mechanisms of PD-1-targeted cancer immunotherapy. The FASEB Journal. 32 (3), 1537-1549 (2018).
- Yong, K. S. M., et al. Humanized mouse as a tool to predict immunotoxicity of human biologics. Frontiers in Immunology. 11, 553362 (2020).
- Shen, H. W., Jiang, X. L., Gonzalez, F. J., Yu, A. M. Humanized transgenic mouse models for drug metabolism and pharmacokinetic research. Current Drug Metabolism. 12 (10), 997-1006 (2011).
- Bosma, G. C., Custer, R. P., Bosma, M. J. A severe combined immunodeficiency mutation in the mouse. Nature. 301 (5900), 527-530 (1983).
- Shultz, L. D., et al. Multiple defects in innate and adaptive immunologic function in NOD/LtSz-scid mice. The Journal of Immunology. 154 (1), 180-191 (1995).
- Legrand, N., et al. Functional CD47/signal regulatory protein alpha (SIRP(alpha)) interaction is required for optimal human T- and natural killer- (NK) cell homeostasis in vivo. Proceedings of the National Academy of Sciences. 108 (32), 13224-13229 (2011).
- Ishikawa, F., et al. Development of functional human blood and immune systems in NOD/SCID/IL2 receptor {gamma} chain(null) mice. Blood. 106 (5), 1565-1573 (2005).
- Ito, M., et al. NOD/SCID/gamma(c)(null) mouse: an excellent recipient mouse model for engraftment of human cells. Blood. 100 (9), 3175-3182 (2002).
- Shultz, L. D., et al. Human lymphoid and myeloid cell development in NOD/LtSz-scid IL2R gamma null mice engrafted with mobilized human hemopoietic stem cells. The Journal of Immunology. 174 (10), 6477-6489 (2005).
- Traggiai, E., et al. Development of a human adaptive immune system in cord blood cell-transplanted mice. Science. 304 (5667), 104-107 (2004).
- Theocharides, A. P., Rongvaux, A., Fritsch, K., Flavell, R. A., Manz, M. G. Humanized hemato-lymphoid system mice. Haematologica. 101 (1), 5-19 (2016).
- Goldman, J. P., et al. Enhanced human cell engraftment in mice deficient in RAG2 and the common cytokine receptor gamma chain. British Journal of Haematology. 103 (2), 335-342 (1998).
- Stripecke, R., et al. Innovations, challenges, and minimal information for standardization of humanized mice. EMBO Molecular Medicine. 12 (7), (2020).
- Allen, T. M., et al. Humanized immune system mouse models: progress, challenges and opportunities. Nature Immunology. 20 (7), 770-774 (2019).
- Gammelgaard, O. L., Terp, M. G., Preiss, B., Ditzel, H. J. Human cancer evolution in the context of a human immune system in mice. Molecular Oncology. 12 (10), 1797-1810 (2018).
- Rios-Doria, J., Stevens, C., Maddage, C., Lasky, K., Koblish, H. K. Characterization of human cancer xenografts in humanized mice. Journal for Immunotherapy of Cancer. 8 (1), 000416 (2020).
- Gombash Lampe, S. E., Kaspar, B. K., Foust, K. D. Intravenous injections in neonatal mice. Journal of Visualized Experiments. (93), e52037 (2014).
- Lang, J., Weiss, N., Freed, B. M., Torres, R. M., Pelanda, R. Generation of hematopoietic humanized mice in the newborn BALB/c-Rag2null Il2rγnull mouse model: a multivariable optimization approach. Clinical Immunology. 140 (1), 102-116 (2011).
- Laskowski, T. J., Hazen, A. L., Collazo, R. S., Haviland, D. Rigor and reproducibility of cytometry practices for immuno-oncology: a multifaceted challenge. Cytometry Part A. 97 (2), 116-125 (2020).
- Bagby, S., et al. Development and maintenance of a preclinical patient derived tumor xenograft model for the investigation of novel anti-cancer therapies. Journal of Visualized Experiments. (115), e54393 (2016).
- Laajala, T. D., et al. Optimized design and analysis of preclinical intervention studies in vivo. Scientific Reports. 6, 30723 (2016).
- Na, Y. S., et al. Establishment of patient-derived xenografts from patients with gastrointestinal stromal tumors: analysis of clinicopathological characteristics related to engraftment success. Scientific Reports. 10 (1), 7996 (2020).
- Tentler, J. J., et al. RX-5902, a novel beta-catenin modulator, potentiates the efficacy of immune checkpoint inhibitors in preclinical models of triple-negative breast cancer. BMC Cancer. 20 (1), 1063 (2020).
- Lang, J., et al. Development of an adrenocortical cancer humanized mouse model to characterize anti-PD1 effects on tumor microenvironment. The Journal of Clinical Endocrinology & Metabolism. 105 (1), 26-42 (2020).
- Lang, J., et al. Studies of lymphocyte reconstitution in a humanized mouse model reveal a requirement of T cells for human B cell maturation. The Journal of Immunology. 190 (5), 2090-2101 (2013).
- Katano, I., et al. NOD-Rag2null IL-2Rγnull mice: an alternative to NOG mice for generation of humanized mice. Experimental Animalas. 63 (3), 321-330 (2014).
- Brehm, M. A., et al. Parameters for establishing humanized mouse models to study human immunity: analysis of human hematopoietic stem cell engraftment in three immunodeficient strains of mice bearing the IL2rγ(null) mutation. Clinical Immunology. 135 (1), 84-98 (2010).
- Hasgur, S., Aryee, K. E., Shultz, L. D., Greiner, D. L., Brehm, M. A. Generation of immunodeficient mice bearing human immune systems by the engraftment of hematopoietic stem cells. Methods in Molecular Biology. 1438, 67-78 (2016).
- Andre, M. C., et al. Long-term human CD34+ stem cell-engrafted nonobese diabetic/SCID/IL-2Rγnull mice show impaired CD8+ T cell maintenance and a functional arrest of immature NK cells. The Journal of Immunology. 185 (5), 2710-2720 (2010).
- Wunderlich, M., et al. Improved multilineage human hematopoietic reconstitution and function in NSGS mice. PLoS One. 13 (12), 0209034 (2018).
- Lee, J., Brehm, M. A., Greiner, D., Shultz, L. D., Kornfeld, H. Engrafted human cells generate adaptive immune responses to Mycobacterium bovis BCG infection in humanized mice. BMC Immunology. 14, 53 (2013).
- Masse-Ranson, G., et al. Accelerated thymopoiesis and improved T-cell responses in HLA-A2/-DR2 transgenic BRGS-based human immune system mice. European Journal of Immunology. 49 (6), 954-965 (2019).
- Oswald, E., et al. Immune cell infiltration pattern in non-small cell lung cancer PDX models is a model immanent feature and correlates with a distinct molecular and phenotypic make-up. Journal for Immunotherapy of Cancer. 10 (4), 004412 (2022).
Erratum
Formal Correction: Erratum: Testing Cancer Immunotherapeutics in a Humanized Mouse Model Bearing Human Tumors
Posted by JoVE Editors on 5/25/2023. Citeable Link.
An erratum was issued for: Testing Cancer Immunotherapeutics in a Humanized Mouse Model Bearing Human Tumors. The Authors section was updated from:
Jordi M. Lanis1
Matthew S. Lewis1
Hannah Strassburger1
Stacey M. Bagby2
Adrian T. A. Dominguez2
Juan A. Marín-Jiménez3
Roberta Pelanda1
Todd M. Pitts2
Julie Lang1
1Department of Immunology and Microbiology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
2Division of Oncology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
3Department of Medical Oncology, Catalan Institute of Oncology (ICO-L’Hospitalet)
to:
Jordi M. Lanis1
Matthew S. Lewis1
Hannah Strassburger1
Kristina Larsen1
Stacey M. Bagby2
Adrian T. A. Dominguez2
Juan A. Marín-Jiménez3
Roberta Pelanda1
Todd M. Pitts2
Julie Lang1
1Department of Immunology and Microbiology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
2Division of Oncology, School of Medicine, University of Colorado Denver Anschutz Medical Campus
3Department of Medical Oncology, Catalan Institute of Oncology (ICO-L’Hospitalet)
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon