Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Detección de poro de nodo mecánico: una plataforma rápida y sin etiquetas para mediciones viscoelásticas unicelulares multiparamétricas
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí se presenta un método para fenotipar mecánicamente células individuales utilizando una plataforma microfluídica basada en electrónica llamada detección mecano-nodo-poro (mecano-NPS). Esta plataforma mantiene un rendimiento moderado de 1-10 células/s mientras mide las propiedades biofísicas elásticas y viscosas de las células.
Resumen
Las propiedades mecánicas celulares están involucradas en una amplia variedad de procesos biológicos y enfermedades, que van desde la diferenciación de células madre hasta la metástasis del cáncer. Los métodos convencionales para medir estas propiedades, como la microscopía de fuerza atómica (AFM) y la aspiración de micropipetas (MA), capturan información rica, reflejando la respuesta viscoelástica completa de una célula; Sin embargo, estos métodos están limitados por un rendimiento muy bajo. Los enfoques de alto rendimiento, como la citometría de deformabilidad en tiempo real (RT-DC), solo pueden medir información mecánica limitada, ya que a menudo están restringidos a lecturas de un solo parámetro que solo reflejan las propiedades elásticas de una célula. En contraste con estos métodos, la detección de poro mecano-nodo (mecano-NPS) es una plataforma microfluídica flexible y sin etiquetas que cierra la brecha en el logro de mediciones viscoelásticas multiparamétricas de una célula con un rendimiento moderado. Una medición de corriente continua (CC) se utiliza para monitorear las células a medida que transitan por un canal microfluídico, rastreando su tamaño y velocidad antes, durante y después de que sean forzadas a través de una constricción estrecha. Esta información (es decir, tamaño y velocidad) se utiliza para cuantificar la deformación transversal de cada célula, la resistencia a la deformación y la recuperación de la deformación. En general, esta plataforma microfluídica basada en electrónica proporciona múltiples propiedades de células viscoelásticas y, por lo tanto, una imagen más completa del estado mecánico de una célula. Debido a que requiere una preparación mínima de la muestra, utiliza una medición electrónica directa (en contraste con una cámara de alta velocidad) y aprovecha la fabricación de litografía blanda estándar, la implementación de esta plataforma es simple, accesible y adaptable al análisis posterior. La flexibilidad, utilidad y sensibilidad de esta plataforma han proporcionado información mecánica única sobre una amplia gama de células, con el potencial de muchas más aplicaciones en ciencia básica y diagnóstico clínico.
Introducción
Las células individuales son materiales dinámicos y viscoelásticos1. Una multitud de procesos internos y externos, (por ejemplo, el inicio de la mitosis o la remodelación de la matriz extracelular [MEC]), influyen en su estructura y composición 2,3,4, a menudo resultando en distintas propiedades biofísicas que complementan su estado actual. En particular, las propiedades mecánicas han demostrado ser biomarcadores importantes del desarrollo celular, la fisiología y la patología, produciendo información cuantitativa valiosa que puede complementar los enfoques moleculares y genéticos canónicos 5,6,7. Por ejemplo, Li et al. describieron recientemente las diferencias mecánicas entre las células de leucemia promielocítica aguda resistentes a los medicamentos y las que responden a los medicamentos, mientras que también utilizaron RNA-seq para descubrir genes asociados al citoesqueleto expresados diferencialmente8. Al comprender la compleja interacción entre la mecánica unicelular y la función celular, el mecanofenotipado tiene aplicaciones más amplias en la transformación de la ciencia básica y el diagnóstico clínico9.
La herramienta más ampliamente adoptada para medir la mecánica unicelular es la microscopía de fuerza atómica (AFM). Si bien AFM permite una medición localizada de alta resolución de las propiedades mecánicas celulares, permanece limitado a un rendimiento de <0.01 células / s10. Alternativamente, las camillas ópticas, que utilizan dos rayos láser divergentes para atrapar y deformar celdas individuales suspendidas11, están limitadas a rendimientos marginalmente más altos de <1 celda/s12. Los avances recientes en las tecnologías microfluídicas han permitido una nueva generación de dispositivos para la evaluación mecánica rápida de una sola célula12,13. Estas técnicas emplean canales de constricción estrechos 14,15, flujo de cizallamiento16 o estiramiento hidrodinámico 17 para deformar las células rápidamente a rendimientos de 10-1.000 células/s 18. Si bien la tasa de medición de estos enfoques es considerablemente más rápida que las técnicas convencionales, a menudo intercambian capacidades de alto rendimiento por lecturas mecánicas limitadas (Tabla suplementaria 1). Todos los métodos microfluídicos rápidos mencionados anteriormente se centran en métricas básicas de un solo parámetro, como el tiempo de tránsito o las relaciones de deformabilidad, que solo reflejan las propiedades elásticas de una célula. Sin embargo, dada la naturaleza viscoelástica intrínseca de las células individuales, una caracterización mecánica robusta y exhaustiva de las células requiere considerar no solo los componentes elásticos sino también las respuestas viscosas.
La detección mecano-nodo-poro (mecano-NPS)2,8 (Figura 1A) es una plataforma microfluídica que aborda las limitaciones existentes con el mecanofenotipado unicelular. Este método permite la medición de múltiples parámetros biofísicos simultáneamente, incluido el diámetro celular, la deformabilidad relativa y el tiempo de recuperación de la deformación, con un rendimiento moderado de 1-10 células / s. Esta técnica se basa en la detección nodo-poro (NPS)19,20,21,22,23,24, que consiste en utilizar una sonda de medición de cuatro puntos para medir el pulso de corriente modulada producido por una célula que transita un canal microfluídico que ha sido segmentado por regiones más amplias, denominadas "nodos". El pulso de corriente modulada es el resultado de que la célula bloquea parcialmente el flujo de corriente en los segmentos (es decir, "poros") y nodos, con más corriente bloqueada en los primeros que en los segundos. En mecano-NPS, un segmento, el "canal de contracción", es más estrecho que el diámetro de una célula; en consecuencia, una célula debe deformarse para transitar todo el canal (Figura 1B). El diámetro celular puede determinarse por la magnitud del subpulso producido cuando la célula transita por los poros del nodo antes del canal de contracción (Figuras 1B, C). Aquí, |ΔInp|, la caída de corriente cuando la celda está en el poro, es proporcional a la relación volumen de la celda al poro, V célula /V poro 2,8,19. La rigidez celular puede determinarse mediante ΔTc, la duración del subpulso dramáticamente mayor producido cuando la célula transita por el canal de contracción (Figuras 1B, C). Una célula más rígida tardará más en transitar por el canal que una más blanda 2,8. Finalmente, la "recuperación" celular, la capacidad de la célula para volver a su tamaño y forma originales después de la deformación, puede determinarse por la serie de subpulsos producidos a medida que la célula transita por los poros del nodo después del canal de contracción (Figuras 1B, C). El tiempo de recuperación, ΔTr, es el tiempo que tardan los subpulsos actuales en volver a la magnitud de los subpulsos anteriores, antes de que la célula sea comprimida. En general, los pulsos de corriente modulada producidos cuando una célula transita por el canal microfluídico se registran y analizan para extraer los parámetros mecánicos unicelulares relevantes (Figura 1D)2,8.
La reproducibilidad y facilidad de uso de esta plataforma microfluídica basada en electrónica han sido demostradas previamente25. Además, la plataforma presenta una baja barrera de entrada para el mecanofenotipado unicelular. La litografía blanda estándar se emplea para fabricar dispositivos microfluídicos. El hardware de medición consta de componentes económicos, que incluyen una placa de circuito impreso simple (PCB), fuente de alimentación, preamplificador, placa de adquisición de datos (DAQ) y computadora. Finalmente, el código fácil de usar está disponible para la adquisición y el análisis de datos, lo que permite una implementación sencilla. Esta técnica de mecanofenotipado puede distinguir poblaciones de líneas celulares epiteliales de mama y pulmón no malignas y malignas, discriminar entre sublinajes en células epiteliales mamarias humanas primarias y caracterizar los efectos de las perturbaciones citoesqueléticas y otros agentes farmacológicos 2,8. En general, esta plataforma es un enfoque efectivo para el mecanofenotipado de células individuales.
Protocolo
1. Diseño de la geometría del dispositivo
- Elija el ancho de los segmentos de tamaño y recuperación para que sea más ancho que el diámetro de las celdas más grandes a medir, pero también mantenga una relación señal-ruido (SNR) suficiente. Consulte la Tabla complementaria 2 para obtener ejemplos de diferentes anchos de segmentos de tamaño y recuperación para varias líneas celulares.
- Elija el ancho del segmento de contracción para aplicar una cepa del 30% al 40% al tamaño promedio de las células que se someterán al mecanofenotipado. La deformación se define como
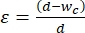 , donde d es el diámetro celular y wc es el ancho del canal de contracción 2,8. Véase la Tabla suplementaria 2 para conocer los diferentes anchos de segmento de contracción para varias líneas celulares.
, donde d es el diámetro celular y wc es el ancho del canal de contracción 2,8. Véase la Tabla suplementaria 2 para conocer los diferentes anchos de segmento de contracción para varias líneas celulares.
NOTA: Si se desea comparar tipos de células o condiciones con diámetros sustancialmente diferentes, se deben usar diseños de dispositivos separados con anchos de segmento de contracción específicos para cada tipo / condición de célula. - Diseñe un dispositivo de referencia para cada geometría de dispositivo única. Esto es necesario para determinar De, el diámetro efectivo del segmento de poro de tamaño del canal microfluídico.
NOTA: El dispositivo de referencia utiliza la misma geometría que el dispositivo principal. La única modificación es que el segmento de contracción debe ser igual en anchura al segmento de poro de tamaño para permitir la calibración con perlas de poliestireno de un tamaño conocido. La ampliación de la contracción evita que las perlas de poliestireno obstruyan el canal de contracción durante la calibración. El proceso de calibración se describe con más detalle en los pasos 4.1 y 5.3.1. La calibración también se puede lograr utilizando un contador de celdas disponible comercialmente, en cuyo caso, no se necesita ningún dispositivo de referencia. Este proceso se describe en el paso 4.2. - Elija la altura del canal de tal manera que las celdas de interés más grandes puedan alargarse completamente sin restricciones dentro del segmento de contracción2. Asegúrese de que la altura del canal sea mayor que hmin
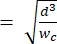 (esto supone que la celda es una predeformación esférica y que la deformación isométrica ocurre a lo largo de la longitud y altura del canal durante la deformación).
(esto supone que la celda es una predeformación esférica y que la deformación isométrica ocurre a lo largo de la longitud y altura del canal durante la deformación).
NOTA: Dada la magnitud de un subpulso de corriente, , cuanto mayor sea el hmin,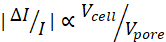 menor será la SNR general.
menor será la SNR general. - Diseñe y cree una fotomáscara utilizando un software de diseño asistido por computadora con los anchos de canal elegidos. Se proporciona un archivo de ejemplo en el archivo complementario 1. Escale el diseño de la máscara microfluídica en un 1,5% para tener en cuenta la contracción del polidimetilsiloxano (PDMS) después de pelar el maestro negativo.
NOTA: Se puede incluir una matriz de dispositivos en una sola máscara siempre que la matriz general no exceda el tamaño de la oblea (Figura suplementaria 1A). - Diseñar y crear una fotomáscara con electrodos que se utilizará para realizar una medición de sonda de cuatro puntos de la corriente del dispositivo microfluídico (Figura 1D). Se proporciona un archivo de ejemplo en el archivo complementario 1.
NOTA: Se puede incluir una matriz de electrodos en una sola máscara siempre que la matriz no exceda el tamaño del portaobjetos de vidrio (Figura suplementaria 1B).
2. Fabricar dispositivos (Figura 2)
- Prepare patrones de electrodos en un sustrato de vidrio.
- Gire el recubrimiento, el patrón y procese una fotorresistencia positiva en un portaobjetos de vidrio liso de acuerdo con la hoja de datos del producto. Un ejemplo de este procedimiento se describe en el Archivo complementario 2.
- Realice la deposición de metal, el despegue y el grabado en oro.
- Realice una deposición de película delgada de 75 Å Ti, 250 Å Pt y 250 Å Au en la diapositiva. Un ejemplo de este procedimiento que utiliza la evaporación del cañón de electrones se describe en el Archivo Suplementario 3.
- Sumerja el portaobjetos en acetona durante 15 minutos para realizar un despegue del exceso de metal.
- En una campana extractora, utilice una pipeta desechable para colocar grabado de oro fundido en la región de los electrodos que estarán expuestos al canal microfluídico, como se muestra en la Figura complementaria 2. Tenga cuidado de evitar dejar caer el grabador en otra parte del tobogán.
PRECAUCIÓN: El grabador de oro puede causar irritación de la piel y los ojos. No respirar vapores, y no ingerir. Manipule con cuidado, use el equipo de protección personal (EPP) adecuado y deseche los desechos de acuerdo con las regulaciones locales de eliminación. - Enjuague el portaobjetos con agua desionizada (DI) y séquelo con nitrógeno seco (N2).
- Si se imprimen varios electrodos en el mismo portaobjetos de vidrio, corte la diapositiva en fichas individuales.
- Use una herramienta de corte de vidrio para marcar la diapositiva a lo largo de los límites del electrodo estampado.
- Rompe el vaso a lo largo de la partitura para dividir la diapositiva en fichas individuales.
- Inspeccione visualmente los electrodos bajo un microscopio. Asegúrese de que los electrodos individuales no estén abiertos eléctricamente o que los electrodos no estén cortocircuitados juntos.
- Fabrica un molde maestro negativo para canales.
- Escurra la capa, el patrón y procese una resistencia epoxi SU-8 sobre una oblea de silicio pulido de acuerdo con la hoja de datos del producto. Un ejemplo de este procedimiento se describe en el Archivo complementario 2.
- Mida las alturas de las características con un perfilómetro e inspeccione visualmente las características bajo un microscopio (Figura complementaria 3). Asegúrese de que las geometrías deseadas estén bien definidas.
- Moldear canales PDMS con litografía blanda.
- Prepare el PDMS pesando un elastómero y un reticulante con una relación de masa de 10:1 en un vaso desechable.
NOTA: Para una oblea con un diámetro de 3, 30 g de PDMS es suficiente. - Mezclar el PDMS vigorosamente durante 30 s con un tenedor desechable, hasta que el PDMS quede opaco con burbujas.
- Desgasifique el PDMS en una cámara de vacío durante aproximadamente 30-90 min, o hasta que el PDMS sea transparente sin burbujas visibles.
- Coloque la oblea con el molde maestro SU-8 en una placa de Petri desechable y vierta PDMS sobre el centro de la oblea.
- Coloque la placa de Petri que contiene el PDMS y la oblea en una cámara de vacío y desgasifieste durante aproximadamente 30 minutos, o hasta que no queden burbujas en el PDMS.
- Hornear el PDMS a 80 °C durante 2 h en un horno o en una placa caliente.
- Con una cuchilla afilada, corte y retire el PDMS del maestro negativo SU-8.
- Corte la losa PDMS moldeada en moldes individuales usando una cuchilla afilada
- Coloque el núcleo de los orificios de acceso de entrada y salida con un punzón de biopsia desechable. Para obtener los mejores resultados, utilice un nuevo punzón para cada losa PDMS. Un punzón más afilado produce agujeros de bordes lisos, minimizando las partículas que podrían obstruir el canal de contracción.
NOTA: El diámetro de los orificios de acceso debe ser ligeramente menor que el diámetro exterior del tubo. Por ejemplo, si se utiliza un tubo de politetrafluoroetileno (PTFE) con un diámetro exterior de 1/32 pulgada, se debe perforar un orificio de 1,5 mm.
- Prepare el PDMS pesando un elastómero y un reticulante con una relación de masa de 10:1 en un vaso desechable.
- Unir un sustrato de vidrio/electrodo a los canales PDMS.
- Limpie los portaobjetos de vidrio del electrodo con metanol (≥99,8%). Secar con N2 seco.
- Limpie el dispositivo PDMS con cinta adhesiva, seguido de un enjuague con alcohol isopropílico (IPA) y agua desionizada (DI; 18 MΩ/cm2). Secar con N2 seco. Luego, limpie con cinta adhesiva una vez más.
- Coloque el sustrato de vidrio con electrodos prefabricados y el molde PDMS preparado (con el lado de la característica hacia arriba) en un limpiador de plasma.
- Exponer ambos al plasma de oxígeno durante 2 min (100-300 mTorr, 30 W).
- Alinee y coloque el molde PDMS con el lado de la característica boca abajo sobre el sustrato de vidrio con electrodos prefabricados.
NOTA: La unión es instantánea una vez que el PDMS tratado con plasma y el vidrio entran en contacto; En consecuencia, no serán posibles más modificaciones de alineación. Para facilitar la alineación, se pueden pipetear 20 μL de una dilución 2:1 de metanol en agua DI sobre la superficie de vidrio tratada con plasma. La solución de metanol actúa como una barrera física entre el vidrio tratado y el PDMS, lo que permite ajustes de alineación. Si utiliza metanol, hornee el dispositivo alineado y acoplado a 50 °C durante 2 h para evaporar la solución y completar el proceso de unión. - Inspeccione visualmente el dispositivo unido bajo un microscopio. Asegúrese de que los electrodos y las geometrías del canal estén correctamente alineados.
3. Medir celdas (Figura 1D)
- Prepare la fuente de presión, la PCB, el hardware de sobremesa y el software de adquisición de datos.
- Conecte el dispositivo microfluídico a la PCB usando la abrazadera. Un ejemplo de la PCB se proporciona en el Archivo complementario 4 (archivos GERBER) y el Archivo complementario 5 (archivos de lista de piezas de PCB, esquemáticos, de placa y de PCB).
- Alinee los pines accionados por resorte de la abrazadera con las almohadillas de contacto del electrodo en el dispositivo microfluídico y alinee los pines del cabezal de la abrazadera con los orificios de la PCB.
- Inserte firmemente los pines del cabezal de la abrazadera en los orificios de PCB, asegurándose de que los pines accionados por resorte permanezcan alineados con las almohadillas de contacto del electrodo.
- Configure y conecte el hardware electrónico.
- Conecte dos de los puertos de salida de la fuente de alimentación al puerto de voltaje de alimentación de la PCB con un adaptador hembra doble banana plug-to-Bayonet Neill-Concelman (BNC) y un cable BNC.
- Encienda la fuente de alimentación. Ajuste la salida conectada al conductor interno del BNC a +15 V y ajuste la otra salida a -15 V. Habilite ambas salidas para alimentar el circuito.
- Conecte el tercero de los puertos de salida de la fuente de alimentación al puerto de voltaje de entrada de la PCB con un cable BNC. Establezca la salida en el voltaje aplicado deseado, pero no lo habilite hasta que comience el experimento.
- Conecte el puerto de corriente de salida de la PCB a la entrada del preamplificador de corriente con un cable BNC.
- Conecte la salida del preamplificador de corriente a una entrada analógica en el bloque de terminales BNC del sistema de adquisición de datos con un cable BNC. Opcionalmente, conecte un filtro analógico de paso bajo en línea con el cable BNC para filtrar las interferencias de alta frecuencia.
NOTA: Para mejorar la SNR, la PCB y el dispositivo pueden estar alojados dentro de una carcasa metálica gruesa. Todos los cables BNC y tubos fluídicos se pueden enrutar a través de orificios perforados en el gabinete.
- Instalar y configurar el software necesario en el ordenador personal (PC)
- Encienda y conecte el controlador de presión al PC. Instale cualquier software de control de presión requerido según las instrucciones del fabricante.
- Instale MATLAB y Data Acquisition Toolbox en el PC. Asegúrese de que los controladores necesarios para el sistema de adquisición de datos estén instalados para que la interfaz de MATLAB Data Acquisition Toolbox pueda detectarlos.
- Descargue el script de adquisición de datos incluido, "NPS.m", desde https://github.com/sohnlab/node-pore-sensing-public.
- Abra y configure el script de adquisición de datos.
- Establezca los valores correctos para inicializar la sesión de adquisición de datos, que incluye el ID de proveedor, el ID de dispositivo del DAQ y el número de canal de entrada analógica (líneas 34-36 en el script incluido).
NOTA: El ID del dispositivo se puede encontrar utilizando la función "daq.getDevices" o "daqlist". - Establezca la frecuencia de muestreo deseada para la adquisición (línea 23 en el script incluido). Para obtener resultados óptimos, debe ajustarse a al menos 10 kHz.
- Establezca los valores correctos para inicializar la sesión de adquisición de datos, que incluye el ID de proveedor, el ID de dispositivo del DAQ y el número de canal de entrada analógica (líneas 34-36 en el script incluido).
- Conecte el dispositivo microfluídico a la PCB usando la abrazadera. Un ejemplo de la PCB se proporciona en el Archivo complementario 4 (archivos GERBER) y el Archivo complementario 5 (archivos de lista de piezas de PCB, esquemáticos, de placa y de PCB).
- Prepare la suspensión celular.
- Preparar una solución de suero bovino fetal al 2% (FBS) en solución salina tamponada con fosfato 1x (PBS) y filtrar con un filtro de 0,22 μm.
- Cultivar y preparar las células de acuerdo con el protocolo de cultivo celular apropiado de la línea celular de elección. Suspender las células en la solución preparada de FBS al 2% en 1x PBS a una concentración de 1-5 x 105 células/ml. Mantenga las células en hielo durante la duración de los experimentos.
- Medir las propiedades físicas de las células.
- Cargue la muestra de celda en el tubo y conéctela a la entrada del dispositivo.
- Corte 30 cm de tubo de PTFE con una cuchilla de afeitar o un cuchillo afilado.
- Conecte un extremo del tubo a una jeringa luer lock. Use la jeringa para extraer la muestra celular en el otro extremo del tubo.
- Inserte cuidadosamente el tubo en la entrada del dispositivo.
- Conecte el extremo opuesto del tubo al controlador de presión microfluídica.
NOTA: Se puede agregar un filtro entre el controlador de presión microfluídica y el tubo para evitar el reflujo de líquido en el controlador de presión.
- Ejecute el experimento.
- Ajuste la presión de conducción constante deseada en el software del controlador de presión y permita que la muestra llene el dispositivo.
NOTA: La presión es típicamente de 2-21 kPa. La velocidad de flujo debe ser lo suficientemente lenta como para permitir pulsos claramente definidos, pero lo suficientemente rápida como para permitir un rendimiento adecuado.- Si se forman burbujas en los canales microfluídicos, use un llenado sin salida: enchufe la salida del dispositivo y aplique una presión baja a la entrada para forzar la salida de aire a través del PDMS permeable al gas. Dejar burbujas en el canal conducirá a una línea de base actual inestable e impedirá mediciones precisas.
- Si los residuos obstruyen el canal microfluídico, desalojarlo presionando ligeramente la parte superior del dispositivo PDMS mientras aplica la presión de conducción, "pulsando" una presión más alta activando y apagando la presión, o retirando el tubo y volviéndolo a insertar. Si los residuos permanecen, puede ser necesario cambiar a un nuevo dispositivo.
- Ajuste el voltaje deseado girando la perilla de voltaje en la fuente de alimentación y habilite el voltaje presionando el botón On .
NOTA: El voltaje suele ser de 1-5 V. Elija el voltaje más bajo necesario para una SNR adecuada. Se debe usar el mismo voltaje en todas las condiciones a comparar. - Encienda el preamplificador de corriente y ajuste la sensibilidad (A/V) lo más baja posible; alternativamente, ajuste la ganancia (V / A) lo más alta posible sin sobrecargar el preamplificador o exceder el voltaje de entrada analógico máximo del DAQ. En este estudio, la sensibilidad se estableció en 10-7 A/V.
NOTA: El valor adecuado de sensibilidad/ganancia dependerá tanto del voltaje aplicado como de la resistencia de referencia del canal microfluídico. - Pulse el botón verde Ejecutar en el menú de la cinta de opciones de MATLAB para iniciar el script de adquisición de datos NPS.m y comenzar a muestrear y guardar los datos.
- Para finalizar el experimento, presione el botón Detener en la esquina inferior izquierda de la ventana de la figura para detener el script de adquisición de datos. Desactive la salida de la fuente de alimentación pulsando el botón On . Ajuste la fuente de presión a presión cero en el software del controlador de presión.
- En este punto, el experimento se puede pausar para realizar una o varias de las siguientes acciones:
- Reemplace el dispositivo actual por uno nuevo.
- Vuelva a cargar el tubo con más muestras de células.
NOTA: Para evitar la contaminación cruzada de muestras, utilice nuevos dispositivos para medir células de diferentes tipos o condiciones. - Desenganche el dispositivo de la PCB y examine la condición del canal bajo un microscopio. Para reiniciar el experimento utilizando el mismo dispositivo, se debe tener cuidado de no introducir burbujas de aire. Puede ser necesario aplicar una presión suave al émbolo de la jeringa para mantener la muestra de la célula en el extremo del tubo mientras se inserta en la entrada del dispositivo.
- Ajuste la presión de conducción constante deseada en el software del controlador de presión y permita que la muestra llene el dispositivo.
- Cargue la muestra de celda en el tubo y conéctela a la entrada del dispositivo.
4. Calibre el dispositivo microfluídico
- Opción 1: Medir las perlas de poliestireno en dispositivos de referencia.
- Elija un tamaño de perla de poliestireno que sea más pequeño que el canal de tamaño.
- Agregue 1.5% de perlas de Tween y poliestireno a la solución filtrada de PBS y FBS utilizada durante los experimentos celulares, a una concentración de 1-3 x 105 perlas/ml.
- Continuar con el experimento como se describe en la sección 3, utilizando el dispositivo de referencia descrito en el paso 1.3, y aplicar el mismo voltaje utilizado durante la experimentación. Utilice la magnitud media de la caída de corriente producida a medida que las perlas transitan por los poros de tamaño y el diámetro conocido de las perlas para calcular De, como se describe en la sección 5.
- Opción 2: Mida de forma independiente el tamaño de la celda con un dispositivo de medición separado.
- En lugar de seguir el protocolo del paso 4.1, utilice un instrumento de medición del tamaño celular disponible comercialmente para medir el tamaño promedio de las células en la muestra. En este caso, no se necesita ningún dispositivo de referencia. Utilice la caída media de corriente producida a medida que las células transitan por el poro de tamaño y el diámetro medio de la célula medido para calcular De como se describe en la sección 5.
5. Analizar datos para extraer fenotipos celulares
NOTA: El procesamiento de datos se puede realizar utilizando el archivo de programa de interfaz de línea de comandos de MATLAB mNPS_procJOVE.m en https://github.com/sohnlab/NPS-analysis-JOVE. Consulte el Archivo complementario 6 para obtener más instrucciones.
- Preprocesar los datos (Figura 3A).
- Calcule la corriente eléctrica medida aplicando el valor de ganancia utilizado en el preamplificador de corriente a voltaje a los datos sin procesar adquiridos por el DAQ.
- Elimine el ruido de alta frecuencia aplicando una función de suavizado rectangular y/o un filtro de paso bajo a la medición de corriente bruta. A continuación, vuelva a muestrear los datos filtrados a una frecuencia de muestreo más baja. Además, calcule los datos de marca de tiempo correspondientes a esta frecuencia de muestreo más baja.
- Calcule una señal de corriente de referencia ajustada aplicando un método como el suavizado asimétrico de mínimos cuadrados26.
- Calcule la primera derivada aproximada (señal de diferencia) de los datos de corriente preprocesados tomando la diferencia entre los puntos de datos posteriores.
- Identificar eventos celulares y extraer datos de subpulso (Figura 3B).
- Busque eventos de celdas candidatas examinando los datos preprocesados. Rechazar eventos celulares que se superponen con otros eventos celulares (es decir, eventos de coincidencia) (Figura complementaria 4), exhiben un ajuste basal deficiente o tienen una forma de pulso inesperada o errónea (por ejemplo, donde una obstrucción puede haber estado presente en el canal).
- Extraiga los datos de subpulso para cada evento celular.
- Cada segmento nodo-poro aparecerá como un subpulso correspondiente dentro del pulso de señal general (Figuras 1B, C). Identifique el inicio de cada subpulso calculando el punto de tiempo cuando la señal de diferencia alcanza un valor mínimo local. Identifique el final de cada subpulso calculando el punto de tiempo cuando la señal de diferencia alcanza un valor máximo local.
- Determine el ancho de cada subpulso como el tiempo transcurrido entre los puntos de tiempo de inicio y finalización. Determine la amplitud de cada subpulso calculando la media de la diferencia entre la corriente medida y la corriente de referencia para todos los puntos de datos entre los puntos de tiempo inicial y final.
- Determine el mecanofenotipo celular para cada evento celular basándose en los datos de subpulso.
- Determine el diámetro de celda d basado en la ecuación definida por Deblois y Bean24:
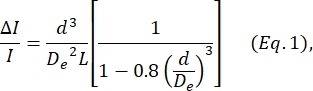
donde ΔI/I es la relación media entre la amplitud del subpulso y la corriente de referencia en los subpulsos de tamaño, De es el diámetro efectivo del canal (medido en el paso 4) y L es la longitud total del canal nodo-poro.- D e se determina calculando el promedio de ΔI/I producido por un conjunto de partículas de un diámetro conocido (ya sean células o cuentas, ver paso 4), usando ese diámetro conocido como d, y resolviendo Eq. 1 para De.
- Cuantificar la resistencia de la célula a la deformación.
- Determine el flujo de la velocidad del fluido U calculando la velocidad media de la célula en los subpulsos de tamaño, utilizando las longitudes de segmento conocidas y la duración medida de cada subpulso.
- Determinar el índice de deformabilidad de toda la célula (wCDI), definido por Kim et al.2 como:
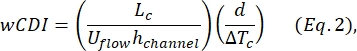
donde L c es la longitud del segmento de contracción, el canal h es la altura del canal y ΔT c es la duración del subpulso de contracción.
- Identificar el tiempo de recuperación de la célula a partir de la deformación, definido como el primer subpulso de recuperación con una amplitud dentro del 8% de la amplitud media del subpulso de tamaño2.
- Calcule la deformación transversal de la célula dentro del segmento de contracción.
- Calcule el diámetro efectivo del segmento de contracción (De,c) según lo definido por Kim et al.2:
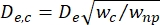 , donde w c es el ancho del segmento de contracción y wnp es el ancho de todos los demás segmentos.
, donde w c es el ancho del segmento de contracción y wnp es el ancho de todos los demás segmentos. - Calcule el diámetro esférico equivalente dc de la celda dentro de la contracción usando nuevamente la ecuación definida por Deblois y Bean24:
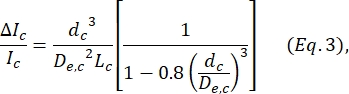
donde ΔI c/I c es la relación entre la amplitud del subpulso y la corriente basal en el subpulso de contracción y Lc es la longitud del segmento de contracción. - Calcule la longitud de elongación de la célula Ldeformarse como se describe en Kim et al.2:
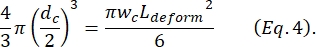
- Finalmente, calcule la deformación transversal δdeformación de la célula, que Kim et al.2 define como
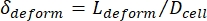 .
.
- Calcule el diámetro efectivo del segmento de contracción (De,c) según lo definido por Kim et al.2:
- Determine el diámetro de celda d basado en la ecuación definida por Deblois y Bean24:
Resultados
La plataforma de mecanofenotipado presentada aquí es un enfoque simple y versátil para medir las propiedades biofísicas de células individuales con un rendimiento moderado. Las células fluyen a través del canal microfluídico (Figura 1A) utilizando un flujo constante impulsado por presión. A medida que las células transitan, la longitud del canal microfluídico y los pulsos de corriente producidos se registran utilizando el hardware de adquisición de datos. La señal adquirida (
Discusión
La medición de las propiedades mecánicas de células individuales utilizando esta técnica de mecanofenotipado consta de tres etapas: fabricación de dispositivos, adquisición de datos y análisis de datos. Dentro de cada etapa, hay aspectos notables que pueden afectar significativamente los resultados experimentales. Durante la fabricación del dispositivo, las geometrías de canal consistentes y la uniformidad de dispositivo a dispositivo son esenciales para obtener resultados precisos y repetibles. Específicamente...
Divulgaciones
L. L. S posee la patente estadounidense No. 11,383,241: "Detección de poro de nodo mecánico", J. Kim, S. Han y L. L. Sohn, emitida el 12 de julio de 2022.
Agradecimientos
Esta investigación fue apoyada por subvenciones de NIBIB 1R01EB024989-01 y NCI 1R01CA190843-01. A. L. y R. R. fueron apoyados por una beca de investigación de posgrado de la Asociación H2H8. K. L. C. fue apoyado por una beca de investigación de posgrado de la Fundación Nacional de Ciencias y una beca académica Siebel.
Materiales
| Name | Company | Catalog Number | Comments |
| Acetone | J.T. Baker | 5356-05 | Purity (GC) ≥ 99.5% (https://us.vwr.com/store/product/6057739/acetone-99-5-vlsi-j-t-baker) |
| Aluminum Foil | n/a | n/a | |
| Analog Low-Pass Filter | ThorLabs | EF504 | ≤240 kHz Passband, Coaxial BNC Feedthrough (https://www.thorlabs.com/thorproduct.cfm?partnumber=EF504#ad-image-0) |
| Biopsy Punch | Integra Miltex | 33-31AA-P/25 | 1mm, Disposable, with Plunger (https://mms.mckesson.com/product/573313/Miltex-33-31AA-P25) |
| Blade | n/a | n/a | |
| BNC Cable | Pomona Electronics | 2249-C-12 | https://www.digikey.com/en/products/detail/pomona-electronics/2249-C-12/603323?utm_adgroup=Coaxial%20Cables%20%28RF%29&utm_source=google&utm_ medium=cpc&utm_campaign= Shopping_Product_Cable%20Assemblies_NEW&utm_term= &utm_content=Coaxial%20Cables%20%28RF%29&gclid=Cj0KCQjwlK-WBhDjARIsAO2sErQqnVJ pj5OXVObuTI8ZUf1ZeIn7zvzGnx mCWdePrG6SdEJMF3X6ubUaAs w-EALw_wcB |
| Cleanroom Polyester Swab | Thermo Fisher Scientific | 18383 | https://www.fishersci.com/shop/products/texwipe-cleantip-alpha-polyester-series-swabs-6/18383 |
| Current Preamplifier | DL Instruments | 1211 | https://www.brltest.com/index.php?main_page=product_info&products_ id=1419 |
| Custom PCB (w/ components) | n/a | n/a | see Supplemental files 4 and 5 |
| DAQ Terminal Block | National Instruments | BNC-2120 | https://www.ni.com/en-in/support/model.bnc-2120.html |
| DAQ to BNC-2110 cable | National Instruments | SHC68-68-EPM | https://www.ni.com/en-in/support/model.shc68-68-epm.html |
| Data Acquisition Board (DAQ) | National Instruments | PCI-6251 | https://www.ni.com/docs/en-US/bundle/pci-6251-feature/page/overview.html |
| Dessicator | Thermo Fisher Scientific | 5311-0250 | https://www.thermofisher.com/order/catalog/product/5311-0250 |
| Female BNC To Banana Plug Adapter | Pomona Electronics | 72909 | https://www.digikey.com/en/products/detail/pomona-electronics/72909/1196318 |
| Fetal Bovine Serum (FBS) | VWR | 89510-186 | https://us.vwr.com/store/product/18706419/avantor-seradigm-select-grade-usda-approved-origin-fetal-bovine-serum-fbs |
| Glass Cutter | Chemglass | CG-1179-21 | https://chemglass.com/plate-glass-cutters-diamond-tips |
| Gold Etchant TFA | Transene | NC0977944 | https://www.fishersci.com/shop/products/NC0977944/NC0977944 |
| Hot Plate | Thermo Fisher Scientific | SP131825 | |
| Isopropyl Alcohol | Spectrum Chemical | I1056-4LTPL | Purity (GC) ≥99.5% (https://www.spectrumchemical.com/isopropyl-alcohol-99-percent-fcc-i1056) |
| Metal Hardware Enclosure | Hammond Manufacturing | EJ12126 | https://www.digikey.com/en/products/detail/hammond-manufacturing/EJ12126/2423415 |
| Methanol | Sigma-Aldrich | 34860 | Purity (GC) ≥99.8% (https://www.sigmaaldrich.com/IN/en/substance/methanol320467561) |
| MF-321 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/mf-321/ |
| MICROPOSIT S1813 Positive Photoresist | DuPont | n/a | https://kayakuam.com/products/microposit-s1800-g2-series-photoresists/ |
| Phosphate Buffered Saline (PBS) | Thermo Fisher Scientific | 10010049 | https://www.thermofisher.com/order/catalog/product/10010049?SID=srch-hj-10010049 |
| Photomask | Fineline Imaging | n/a | Photomask are custom ordered from our CAD designs (https://www.fineline-imaging.com/) |
| Plain Glass Microscope Slide | Fisher Scientific | 12-553-5B | Material: Soda Lime, L75 x W50 mm, Thickness: 0.90–1.10 mm |
| Plasma Cleaner | Harrick Plasma | PDC-001 | https://harrickplasma.com/plasma-cleaners/expanded-plasma-cleaner/ |
| Plastic Petri Dish | Thermo Fisher Scientific | FB0875712 | 100 mm (https://www.fishersci.com/shop/products/fisherbrand-petri-dishes-clear-lid-raised-ridge-100-x-15mm/FB0875712) |
| Pressure Controller | Fluigent | MFCS-EZ | https://www.fluigent.com/research/instruments/pressure-flow-controllers/mfcs-series/ |
| Pressure Controller Software | Fluigent | MAESFLO | |
| Programming & Computation Software | MATLAB | R2021b | for data acquisition and analysis (https://www.mathworks.com/products/matlab.html) |
| PTFE Tubing | Cole Parmer | 06417-31 | 0.032" ID x 0.056" (https://www.coleparmer.com/i/masterflex-transfer-tubing-microbore-ptfe-0-032-id-x-0-056-od-100-ft-roll/0641731) |
| Scepter 2.0 Handheld Automatic Cell Counter | Millapore Sigma | PHCC20060 | https://www.sigmaaldrich.com/IN/en/product/mm/phcc20060 |
| Silicon Wafer | Wafer World | 2885 | 76.2 mm, Single Side Polished (https://www.waferworld.com/product/2885) |
| Spin Coater | n/a | n/a | |
| SU-8 3025 Negative Photoresist | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-2000/ |
| SU8 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-developer/ |
| Sygard 184 Polydimethlysiloxane | Dow Chemical | 4019862 | https://www.ellsworth.com/products/by-market/consumer-products/encapsulants/silicone/dow-sylgard-184-silicone-encapsulant-clear-0.5-kg-kit/ |
| Tape | Scotch | 810-341296 | https://www.staples.com/Scotch-Magic-Tape-810-3-4-x-36-yds-1-Core/product_130567?cid=PS:GS:SBD:PLA:OS&gclid= Cj0KCQjwlK-WBhDjARIsAO 2sErRwzrrgjU0NjFkDkne1xm vT7ekS3tdzvAgiMDwPoxocgH VTQZi7vJgaAvQZEALw_wcB |
| Titanium, Platinum, Gold | n/a | n/a | |
| Triple Output Power Supply | Keysight | E36311A | https://www.newark.com/keysight-technologies/e36311a/dc-power-supply-3o-p-6v-5a-prog/dp/15AC9653 |
| UV Mask Aligner | Karl Suss America | MJB3 Mask Aligner |
Referencias
- Pegoraro, A. F., Janmey, P., Weitz, D. A. Mechanical properties of the cytoskeleton and cells. Cold Spring Harbor Perspectives in Biology. 9 (11), 022038 (2017).
- Kim, J., et al. Characterizing cellular mechanical phenotypes with mechano-node-pore sensing. Microsystems & Nanoengineering. 4, 17091 (2018).
- Mierke, C. T. Bidirectional mechanical response between cells and their microenvironment. Frontiers in Physics. 9, 619 (2021).
- Kumar, S., Weaver, V. M. Mechanics, malignancy, and metastasis: The force journey of a tumor cell. Cancer and Metastasis Reviews. 28 (1), 113-127 (2009).
- Nia, H. T., Munn, L. L., Jain, R. K. Physical traits of cancer. Science. 370 (6516), (2020).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463 (7280), 485-492 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: The role of physical interactions and mechanical forces in metastasis. Nature Reviews Cancer. 11 (7), 512-522 (2011).
- Li, B., et al. Mechanical phenotyping reveals unique biomechanical responses in retinoic acid-resistant acute promyelocytic leukemia. iScience. 25 (2), 103772 (2022).
- Kozminsky, M., Sohn, L. L. The promise of single-cell mechanophenotyping for clinical applications. Biomicrofluidics. 14 (3), 031301 (2020).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Wottawah, F., et al. Optical rheology of biological cells. Physical Review Letters. 94 (9), 1-4 (2005).
- Darling, E. M., Di Carlo, D. High-throughput assessment of cellular mechanical properties. Annual Review of Biomedical Engineering. 17 (1), 35-62 (2015).
- Carey, T. R., Cotner, K. L., Li, B., Sohn, L. L. Developments in label-free microfluidic methods for single-cell analysis and sorting. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 11 (1), 1529 (2019).
- Bagnall, J. S., et al. Deformability of tumor cells versus blood cells. Scientific Reports. 5, 18542 (2015).
- Byun, S., et al. Characterizing deformability and surface friction of cancer cells. Proceedings of the National Academy of Sciences. 110 (19), 7580-7585 (2013).
- Otto, O., et al. Real-time deformability cytometry: On-the-fly cell mechanical phenotyping. Nature Methods. 12 (3), 199-202 (2015).
- Gossett, D. R., et al. Hydrodynamic stretching of single cells for large population mechanical phenotyping. Proceedings of the National Academy of Sciences. 109 (20), 7630-7635 (2012).
- Guck, J., Chilvers, E. R. Mechanics meets medicine. Science Translational Medicine. 5 (212), 3-6 (2013).
- Balakrishnan, K. R., et al. Node-pore sensing: A robust, high-dynamic range method for detecting biological species. Lab on a Chip. 13 (7), 1302-1307 (2013).
- Carbonaro, A., Sohn, L. L. A resistive-pulse sensor chip for multianalyte immunoassays. Lab on a Chip. 5 (10), 1155-1160 (2005).
- Saleh, O. A., Sohn, L. L. Direct detection of antibody-antigen binding using an on-chip artificial pore. Proceedings of the National Academy of Sciences. 100 (3), 820-824 (2003).
- Saleh, O. A., Sohn, L. L. An artificial nanopore for molecular sensing. Nano Letters. 3 (1), 37-38 (2003).
- Saleh, O. A., Sohn, L. L. Quantitative sensing of nanoscale colloids using a microchip Coulter counter. Review of Scientific Instruments. 72 (12), 4449-4451 (2001).
- DeBlois, R. W., Bean, C. P. Counting and sizing of submicron particles by the resistive pulse technique. Review of Scientific Instruments. 41 (7), 909-916 (1970).
- Li, B., et al. Evaluating sources of technical variability in the mechano-node-pore sensing pipeline and their effect on the reproducibility of single-cell mechanical phenotyping. PLoS ONE. 16 (10), 0258982 (2021).
- Zhang, Z. M., Chen, S., Liang, Y. Z. Baseline correction using adaptive iteratively reweighted penalized least squares. Analyst. 135 (5), 1138-1146 (2010).
- Alibert, C., Goud, B., Manneville, J. B. Are cancer cells really softer than normal cells. Biology of the Cell. 109 (5), 167-189 (2017).
- Fujiwara, I., Zweifel, M. E., Courtemanche, N., Pollard, T. D. Latrunculin A accelerates actin filament depolymerization in addition to sequestering actin monomers. Current Biology. 28 (19), 3183-3192 (2018).
- Saleh, O. A. . A novel resistive pulse sensor for biological measurements. , (2003).
- Dokukin, M. E., Guz, N. V., Sokolov, I. Quantitative study of the elastic modulus of loosely attached cells in AFM indentation experiments. Biophysical Journal. 104 (10), 2123-2131 (2013).
- Li, Q., Lim, C. T., Goh, J. C. H., et al. Probing the elasticity of breast cancer cells using AFM. 13th International Conference on Biomedical Engineering. IFMBE Proceedings. 23, 2122-2125 (2009).
- Rother, J., et al. Atomic force microscopy-based microrheology reveals significant differences in the viscoelastic response between malign and benign cell lines. Open Biology. 4 (5), 140046 (2014).
- Li, Q., et al. AFM indentation study of breast cancer cells. Biochemical and Biophysical Research Communications. 374 (4), 609-613 (2008).
- Xu, C., et al. Elasticity measurement of breast cancer cells by atomic force microscopy. Proc. SPIE 9230. Twelfth International Conference on Photonics and Imaging in Biology and Medicine. (PIBM 2014). 92300, (2014).
- Alcaraz, J., et al. Microrheology of human lung epithelial cells measured by atomic force microscopy. Biophysical Journal. 84 (3), 2071-2079 (2003).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Urbanska, M., et al. A comparison of microfluidic methods for high-throughput cell deformability measurements. Nature Methods. 17, 587-593 (2020).
- Hill, R. T., Chilkoti, A. Surface Patterning. Biomaterials Science: An Introduction to Materials: Third Edition. , 276-301 (2013).
- Wang, Z., Volinsky, A. A., Gallant, N. D. Crosslinking effect on polydimethylsiloxane elastic modulus measured by custom-built compression instrument. Journal of Applied Polymer Science. 131 (22), 41050 (2014).
- Gibson, L. J. The hierarchical structure and mechanics of plant materials. Journal of the Royal Society Interface. 9 (76), 2749-2766 (2012).
- Stephens, A. D., Banigan, E. J., Adam, S. A., Goldman, R. D., Marko, J. F. Chromatin and lamin a determine two different mechanical response regimes of the cell nucleus. Molecular Biology of the Cell. 28 (14), 1984-1996 (2017).
- Rosenbluth, M. J., Lam, W. A., Fletcher, D. A. Force microscopy of nonadherent cells: A comparison of leukemia cell deformability. Biophysical Journal. 90 (8), 2994-3003 (2006).
- Evers, T. M. J., Holt, L. J., Alberti, S., Mashaghi, A. Reciprocal regulation of cellular mechanics and metabolism. Nature Metabolism. 3 (4), 456-468 (2021).
- Balakrishnan, K. R., et al. Node-pore sensing enables label-free surface-marker profiling of single cells. Analytical Chemistry. 87 (5), 2988-2995 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados