È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Rilevamento meccano-nodo-pore: una piattaforma rapida e senza etichette per misure viscoelastiche monocellulari multiparametriche
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Presentato qui è un metodo per fenotipizzare meccanicamente singole cellule utilizzando una piattaforma microfluidica basata sull'elettronica chiamata mechano-node-pore sensing (mechano-NPS). Questa piattaforma mantiene un throughput moderato di 1-10 cellule / s misurando sia le proprietà biofisiche elastiche che viscose delle cellule.
Abstract
Le proprietà meccaniche cellulari sono coinvolte in un'ampia varietà di processi biologici e malattie, che vanno dalla differenziazione delle cellule staminali alle metastasi del cancro. I metodi convenzionali per misurare queste proprietà, come la microscopia a forza atomica (AFM) e l'aspirazione con micropipette (MA), acquisiscono informazioni ricche, riflettendo la risposta viscoelastica completa di una cellula; Tuttavia, questi metodi sono limitati da una velocità effettiva molto bassa. Gli approcci ad alto rendimento, come la citometria a deformabilità in tempo reale (RT-DC), possono misurare solo informazioni meccaniche limitate, poiché sono spesso limitati a letture a singolo parametro che riflettono solo le proprietà elastiche di una cellula. In contrasto con questi metodi, il rilevamento meccano-nodo-poro (mechano-NPS) è una piattaforma microfluidica flessibile e senza etichetta che colma il divario nel raggiungimento di misurazioni viscoelastiche multiparametriche di una cella con throughput moderato. Una misurazione di corrente continua (DC) viene utilizzata per monitorare le cellule mentre transitano su un canale microfluidico, monitorandone le dimensioni e la velocità prima, durante e dopo che sono forzate attraverso una stretta costrizione. Queste informazioni (cioè dimensioni e velocità) vengono utilizzate per quantificare la deformazione trasversale di ogni cellula, la resistenza alla deformazione e il recupero dalla deformazione. In generale, questa piattaforma microfluidica basata sull'elettronica fornisce molteplici proprietà viscoelastiche della cella e quindi un quadro più completo dello stato meccanico di una cella. Poiché richiede una preparazione minima del campione, utilizza una misurazione elettronica semplice (a differenza di una telecamera ad alta velocità) e sfrutta la fabbricazione standard della litografia morbida, l'implementazione di questa piattaforma è semplice, accessibile e adattabile all'analisi a valle. La flessibilità, l'utilità e la sensibilità di questa piattaforma hanno fornito informazioni meccaniche uniche su una vasta gamma di cellule, con il potenziale per molte altre applicazioni nella scienza di base e nella diagnostica clinica.
Introduzione
Le singole celle sono materiali viscoelastici dinamici1. Una moltitudine di processi interni ed esterni, (ad esempio, insorgenza di mitosi o rimodellamento della matrice extracellulare [ECM]), influenzano la loro struttura e composizione 2,3,4, spesso risultando in proprietà biofisiche distinte che completano il loro stato attuale. In particolare, le proprietà meccaniche hanno dimostrato di essere importanti biomarcatori dello sviluppo cellulare, della fisiologia e della patologia, fornendo preziose informazioni quantitative che possono integrare gli approcci molecolari e genetici canonici 5,6,7. Ad esempio, Li et al. hanno recentemente descritto le differenze meccaniche tra le cellule di leucemia promielocitica acuta resistenti ai farmaci e quelle responsive, utilizzando anche RNA-seq per scoprire geni associati al citoscheletro differenzialmente espressi8. Comprendendo la complessa interazione tra la meccanica delle singole cellule e la funzione cellulare, la meccanofenotipizzazione ha applicazioni più ampie nella trasformazione della scienza di base e della diagnostica clinica9.
Lo strumento più ampiamente adottato per misurare la meccanica a singola cellula è la microscopia a forza atomica (AFM). Mentre l'AFM consente una misurazione localizzata ad alta risoluzione delle proprietà meccaniche cellulari, rimane limitata a un throughput di <0,01 cellule / s10. In alternativa, le barelle ottiche, che utilizzano due raggi laser divergenti per intrappolare e deformare le singole celle sospese11, sono limitate a portate marginalmente più elevate di <1 cella/s12. I recenti progressi nelle tecnologie microfluidiche hanno permesso una nuova generazione di dispositivi per la valutazione rapida, a cella singola, meccanica12,13. Queste tecniche impiegano stretti canali di costrizione 14,15, flusso di taglio16 o allungamento idrodinamico 17 per deformare rapidamente le cellule a portate di 10-1.000 cellule / s 18. Mentre la velocità di misurazione di questi approcci è considerevolmente più veloce rispetto alle tecniche convenzionali, spesso scambiano capacità ad alto rendimento per letture meccaniche limitate (Tabella supplementare 1). Tutti i suddetti metodi microfluidici rapidi si concentrano su metriche di base a singolo parametro, come il tempo di transito o i rapporti di deformabilità, che riflettono solo le proprietà elastiche di una cella. Tuttavia, data la natura viscoelastica intrinseca delle singole cellule, una caratterizzazione meccanica robusta e completa delle cellule richiede la considerazione non solo dei componenti elastici ma anche delle risposte viscose.
Il rilevamento meccano-nodo-poro (mechano-NPS)2,8 (Figura 1A) è una piattaforma microfluidica che affronta le limitazioni esistenti con la meccanofenotipizzazione a singola cellula. Questo metodo consente la misurazione simultanea di più parametri biofisici, tra cui il diametro della cella, la deformabilità relativa e il tempo di recupero dalla deformazione, con una produttività moderata di 1-10 celle/s. Questa tecnica si basa sul rilevamento dei pori dei nodi (NPS)19,20,21,22,23,24, che prevede l'utilizzo di una misura di sonda a quattro punti per misurare l'impulso di corrente modulata prodotto da una cellula che transita in un canale microfluidico che è stato segmentato da regioni più ampie, denominate "nodi". L'impulso di corrente modulato è il risultato della cella che blocca parzialmente il flusso di corrente nei segmenti (cioè "pori") e nei nodi, con più corrente bloccata nel primo che nel secondo. In meccano-NPS, un segmento, il "canale di contrazione", è più stretto del diametro di una cellula; di conseguenza, una cella deve deformarsi per transitare sull'intero canale (Figura 1B). Il diametro della cellula può essere determinato dall'entità del sottoimpulso prodotto quando la cellula transita nei pori del nodo prima del canale di contrazione (Figure 1B,C). Qui, |ΔInp|, la caduta di corrente quando la cellula è nel poro, è proporzionale al rapporto volumetrico della cellula al poro, cellula V / poro V 2,8,19. La rigidità cellulare può essere determinata da ΔTc, la durata del sottoimpulso drammaticamente più grande prodotto quando la cellula transita nel canale di contrazione (Figure 1B,C). Una cella più rigida impiegherà più tempo a transitare nel canale rispetto a una più morbida 2,8. Infine, il "recupero" cellulare, la capacità della cellula di tornare alla sua dimensione e forma originale dopo la deformazione, può essere determinato dalla serie di sottoimpulsi prodotti mentre la cellula transita nei pori del nodo dopo il canale di contrazione (Figure 1B,C). Il tempo di recupero, ΔTr, è il tempo necessario affinché i sottoimpulsi di corrente ritornino alla grandezza dei sottoimpulsi precedenti, prima che la cella venga schiacciata. Nel complesso, gli impulsi di corrente modulata prodotti al transito di una cellula nel canale microfluidico vengono registrati e analizzati per estrarre i parametri meccanici rilevanti della singola cellula (Figura 1D)2,8.
La riproducibilità e la facilità d'uso di questa piattaforma microfluidica basata sull'elettronica sono state precedentemente dimostrate25. Inoltre, la piattaforma presenta una bassa barriera all'ingresso per la meccanofenotipizzazione unicellulare. La litografia morbida standard viene impiegata per fabbricare dispositivi microfluidici. L'hardware di misura è costituito da componenti economici, tra cui un semplice circuito stampato (PCB), alimentatore, preamplificatore, scheda di acquisizione dati (DAQ) e computer. Infine, è disponibile un codice intuitivo per l'acquisizione e l'analisi dei dati, consentendo un'implementazione semplice. Questa tecnica di meccanofenotipizzazione può distinguere popolazioni di linee cellulari epiteliali mammarie e polmonari non maligne e maligne, discriminare tra i sottolignaggi nelle cellule epiteliali mammarie umane primarie e caratterizzare gli effetti delle perturbazioni citoscheletriche e di altri agenti farmacologici 2,8. Nel complesso, questa piattaforma è un approccio efficace per la meccanofenotipizzazione di singole cellule.
Protocollo
1. Progetta la geometria del dispositivo
- Scegliere la larghezza dei segmenti di dimensionamento e recupero in modo che sia più ampia del diametro delle celle più grandi da misurare, ma mantenga anche un rapporto segnale-rumore (SNR) sufficiente. Vedere la Tabella supplementare 2 per esempi di diverse larghezze dei segmenti di dimensionamento e recupero per varie linee di celle.
- Scegliere la larghezza del segmento di contrazione per applicare un ceppo del 30% -40% alla dimensione media delle cellule che devono essere sottoposte a meccanofenotipizzazione. La deformazione è definita come
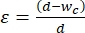 , dove d è il diametro della cella e wc è la larghezza del canale di contrazione 2,8. Vedere la Tabella supplementare 2 per le diverse larghezze dei segmenti di contrazione per varie linee cellulari.
, dove d è il diametro della cella e wc è la larghezza del canale di contrazione 2,8. Vedere la Tabella supplementare 2 per le diverse larghezze dei segmenti di contrazione per varie linee cellulari.
NOTA: Se si desidera confrontare tipi di celle o condizioni con diametri sostanzialmente diversi, è necessario utilizzare progetti di dispositivi separati con larghezze di segmento di contrazione specifiche per ciascun tipo / condizione di cella. - Progettare un dispositivo di riferimento per ogni geometria univoca del dispositivo. Questo è necessario per determinare De, il diametro effettivo del segmento dei pori di dimensionamento del canale microfluidico.
NOTA: la periferica di riferimento utilizza la stessa geometria della periferica principale. L'unica modifica è che il segmento di contrazione dovrebbe essere uguale in larghezza al segmento dei pori di dimensionamento per consentire la calibrazione con perle di polistirene di dimensioni note. L'allargamento della contrazione impedisce alle perle di polistirene di intasare il canale di contrazione durante la calibrazione. Il processo di taratura è ulteriormente descritto nei punti 4.1 e 5.3.1. La calibrazione può anche essere ottenuta utilizzando un contatore di celle disponibile in commercio, nel qual caso non è necessario alcun dispositivo di riferimento. Questo processo è descritto nel passaggio 4.2. - Scegli l'altezza del canale in modo tale che le celle di interesse più grandi possano allungarsi completamente senza restrizioni all'interno del segmento di contrazione2. Assicurarsi che l'altezza del canale sia maggiore di hmin
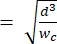 (questo presuppone che la cella sia una pre-deformazione sferica e che la deformazione isometrica si verifichi lungo la lunghezza e l'altezza del canale durante la deformazione).
(questo presuppone che la cella sia una pre-deformazione sferica e che la deformazione isometrica si verifichi lungo la lunghezza e l'altezza del canale durante la deformazione).
NOTA: Data la grandezza di un impulso di corrente, , maggiore è l'hmin,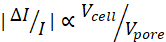 minore sarà l'SNR complessivo.
minore sarà l'SNR complessivo. - Progetta e crea una fotomaschera utilizzando un software di progettazione assistita da computer con le larghezze dei canali scelte. Un file di esempio è fornito nel file supplementare 1. Ridimensionare il design della maschera microfluidica dell'1,5% per tenere conto del restringimento del polidimetilsilossano (PDMS) dopo il peeling dal master negativo.
NOTA: è possibile includere un array di dispositivi su una singola maschera purché l'array complessivo non superi le dimensioni del wafer (Figura supplementare 1A). - Progettare e creare una fotomaschera con elettrodi che verrà utilizzata per eseguire una misura a quattro punti della misura della corrente del dispositivo microfluidico (Figura 1D). Un file di esempio è fornito nel file supplementare 1.
NOTA: Una matrice di elettrodi può essere inclusa su una singola maschera purché l'array non superi le dimensioni del vetrino (Figura supplementare 1B).
2. Fabbricare dispositivi (Figura 2)
- Preparare modelli di elettrodi su un substrato di vetro.
- Gira il rivestimento, il modello ed elabora un fotoresist positivo su un vetrino semplice secondo la scheda tecnica del prodotto. Un esempio di questa procedura è descritto nel fascicolo supplementare 2.
- Eseguire la deposizione di metalli, il decollo e l'incisione dell'oro.
- Eseguire la deposizione di film sottile di 75 Å Ti, 250 Å Pt e 250 Å Au sul vetrino. Un esempio di questa procedura che utilizza l'evaporazione elettrone-pistola è descritto nel file supplementare 3.
- Immergere il vetrino in acetone per 15 minuti per eseguire un sollevamento del metallo in eccesso.
- In una cappa aspirante, utilizzare una pipetta usa e getta per far cadere l'etchant d'oro sulla regione degli elettrodi che saranno esposti al canale microfluidico, come mostrato nella figura supplementare 2. Fai attenzione per evitare di far cadere etchant altrove sulla diapositiva.
ATTENZIONE: L'incantesimo d'oro può causare irritazione della pelle e degli occhi. Non respirare vapori e non ingerire. Maneggiare con cura, indossare dispositivi di protezione individuale (DPI) appropriati e gettare i rifiuti secondo le normative locali sullo smaltimento. - Risciacquare il vetrino con acqua deionizzata (DI) e asciugarlo con azoto secco (N2).
- Se più elettrodi sono stampati sullo stesso vetrino, tagliare il vetrino in singoli chip.
- Utilizzare uno strumento di taglio del vetro per segnare la diapositiva lungo i bordi dell'elettrodo modellati.
- Rompere il vetro lungo la partitura per suddividere la diapositiva in singoli chip.
- Ispezionare visivamente gli elettrodi al microscopio. Assicurarsi che i singoli elettrodi non siano elettricamente aperti o che gli elettrodi non siano collegati in cortocircuito.
- Fabbricare uno stampo master negativo per i canali.
- Spin coat, pattern e processa una resistenza epossidica SU-8 su un wafer di silicio lucidato secondo la scheda tecnica del prodotto. Un esempio di questa procedura è descritto nel fascicolo supplementare 2.
- Misurare le altezze delle caratteristiche utilizzando un profilometro e ispezionare visivamente le caratteristiche al microscopio (Figura supplementare 3). Assicurarsi che le geometrie desiderate siano ben definite.
- Stampare canali PDMS con litografia morbida.
- Preparare il PDMS pesando un elastomero e un reticolante con un rapporto di massa di 10:1 in un bicchiere usa e getta.
NOTA: Per un wafer con un diametro di 3, sono sufficienti 30 g di PDMS. - Mescolare energicamente il PDMS per 30 s con una forchetta usa e getta, fino a quando il PDMS è opaco con le bolle.
- Degassare il PDMS in una camera a vuoto per circa 30-90 minuti, o fino a quando il PDMS è trasparente senza bolle visibili.
- Posizionare il wafer con lo stampo principale SU-8 in una capsula di Petri usa e getta e versare PDMS al centro del wafer.
- Posizionare la capsula di Petri contenente il PDMS e il wafer in una camera a vuoto e degassare per circa 30 minuti, o fino a quando non rimangono bolle nel PDMS.
- Cuocere il PDMS a 80 °C per 2 ore in forno o su una piastra calda.
- Con una lama affilata, tagliare e rimuovere il PDMS dal master negativo SU-8.
- Tagliare la lastra PDMS stampata in singoli stampi usando una lama affilata
- Nucleo dei fori di accesso in ingresso e in uscita utilizzando un punzone monouso per biopsia. Per risultati ottimali, utilizzare un nuovo punzone per ogni lastra PDMS. Un punzone più affilato produce fori dai bordi lisci, riducendo al minimo il particolato che potrebbe ostruire il canale di contrazione.
NOTA: il diametro dei fori di accesso deve essere leggermente inferiore al diametro esterno del tubo. Ad esempio, se si utilizzano tubi in politetrafluoroetilene (PTFE) con un diametro esterno di 1/32 di pollice, è necessario perforare un foro di 1,5 mm.
- Preparare il PDMS pesando un elastomero e un reticolante con un rapporto di massa di 10:1 in un bicchiere usa e getta.
- Incollare un substrato di vetro/elettrodo ai canali PDMS.
- Pulire i vetrini dell'elettrodo con metanolo (≥99,8%). Asciugare con secco N2.
- Pulire il dispositivo PDMS con nastro adesivo, seguito da un risciacquo con alcool isopropilico (IPA) e acqua deionizzata (DI; 18 MΩ/cm2). Asciugare con secco N2. Quindi, pulire ancora una volta con lo scotch.
- Posizionare il substrato di vetro con elettrodi prefabbricati e lo stampo PDMS preparato (lato verso l'alto) in un pulitore al plasma.
- Esporre entrambi al plasma di ossigeno per 2 minuti (100-300 mTorr, 30 W).
- Allineare e posizionare lo stampo PDMS con il lato della caratteristica rivolto verso il basso sul substrato di vetro con elettrodi prefabbricati.
NOTA: L'incollaggio è istantaneo una volta che il PDMS trattato al plasma e il vetro entrano in contatto; Di conseguenza, non saranno possibili ulteriori modifiche all'allineamento. Per facilitare l'allineamento, 20 μL di una diluizione 2:1 di metanolo in acqua DI possono essere pipettati sulla superficie del vetro trattato al plasma. La soluzione di metanolo funge da barriera fisica tra il vetro trattato e il PDMS, consentendo regolazioni di allineamento. Se si utilizza metanolo, cuocere il dispositivo allineato e accoppiato a 50 °C per 2 ore per far evaporare la soluzione e completare il processo di incollaggio. - Ispezionare visivamente il dispositivo incollato al microscopio. Assicurarsi che gli elettrodi e le geometrie dei canali siano allineati correttamente.
3. Misurare le celle (Figura 1D)
- Preparare la sorgente di pressione, il PCB, l'hardware da banco e il software di acquisizione dati.
- Collegare il dispositivo microfluidico al PCB utilizzando il morsetto. Un esempio del PCB è fornito nel file supplementare 4 (file GERBER) e nel file supplementare 5 (file di schemi, schede e file di elenco delle parti PCB).
- Allineare i pin a molla del morsetto con i cuscinetti di contatto dell'elettrodo sul dispositivo microfluidico e allineare i pin della testata del morsetto con i fori sul PCB.
- Inserire saldamente i pin di intestazione del morsetto nei fori del PCB, assicurandosi che i pin caricati a molla rimangano allineati con i pad di contatto dell'elettrodo.
- Configurare e collegare l'hardware elettronico.
- Collegare due delle porte di uscita dell'alimentatore alla porta di tensione di alimentazione del PCB con un adattatore femmina Neill-Concelman (BNC) a doppia banana plug-to-Bayonet e un cavo BNC.
- Accendere l'alimentatore. Impostare l'uscita collegata al conduttore interno del BNC su +15 V e impostare l'altra uscita su -15 V. Abilitare entrambe le uscite per alimentare il circuito.
- Collegare la terza delle porte di uscita dell'alimentatore alla porta di tensione di ingresso del PCB con un cavo BNC. Impostare l'uscita sulla tensione applicata desiderata, ma non attivarla fino all'avvio dell'esperimento.
- Collegare la porta di corrente di uscita del PCB all'ingresso del preamplificatore di corrente con un cavo BNC.
- Collegare l'uscita del preamplificatore di corrente a un ingresso analogico sulla morsettiera BNC del sistema di acquisizione dati con un cavo BNC. Opzionalmente, collegare un filtro passa-basso analogico in linea con il cavo BNC per filtrare le interferenze ad alta frequenza.
NOTA: per migliorare l'SNR, il PCB e il dispositivo possono essere alloggiati all'interno di uno spesso involucro metallico. Tutti i cavi BNC e i tubi fluidici possono essere instradati attraverso fori praticati nella custodia.
- Installare e configurare il software richiesto sul personal computer (PC)
- Accendere e collegare il regolatore di pressione al PC. Installare qualsiasi software di controllo della pressione richiesto secondo le istruzioni del produttore.
- Installa MATLAB e Data Acquisition Toolbox sul PC. Assicurarsi che i driver necessari per il sistema di acquisizione dati siano installati in modo che l'interfaccia di MATLAB Data Acquisition Toolbox possa rilevarlo.
- Scaricare lo script di acquisizione dati incluso, "NPS.m", da https://github.com/sohnlab/node-pore-sensing-public.
- Aprire e configurare lo script di acquisizione dati.
- Impostare i valori corretti per inizializzare la sessione di acquisizione dati, che include l'ID fornitore, l'ID dispositivo del DAQ e il numero del canale di ingresso analogico (righe 34-36 nello script incluso).
NOTA: l'ID dispositivo può essere trovato utilizzando la funzione "daq.getDevices" o "daqlist". - Impostare la frequenza di campionamento desiderata per l'acquisizione (riga 23 nello script incluso). Per risultati ottimali, dovrebbe essere impostato su almeno 10 kHz.
- Impostare i valori corretti per inizializzare la sessione di acquisizione dati, che include l'ID fornitore, l'ID dispositivo del DAQ e il numero del canale di ingresso analogico (righe 34-36 nello script incluso).
- Collegare il dispositivo microfluidico al PCB utilizzando il morsetto. Un esempio del PCB è fornito nel file supplementare 4 (file GERBER) e nel file supplementare 5 (file di schemi, schede e file di elenco delle parti PCB).
- Preparare la sospensione cellulare.
- Preparare una soluzione di siero bovino fetale al 2% (FBS) in soluzione salina tamponata fosfato 1x (PBS) e filtrare con un filtro da 0,22 μm.
- Coltura e preparazione delle cellule secondo il protocollo di coltura cellulare appropriato della linea cellulare scelta. Sospendere le cellule nella soluzione preparata di FBS al 2% in 1x PBS ad una concentrazione di 1-5 x 105 cellule/ml. Mantenere le cellule sul ghiaccio per tutta la durata degli esperimenti.
- Misurare le proprietà fisiche delle cellule.
- Caricare il campione di cella nel tubo e collegarlo all'ingresso del dispositivo.
- Tagliare 30 cm di tubo in PTFE con una lama di rasoio o un coltello affilato.
- Collegare un'estremità del tubo a una siringa luer lock. Utilizzare la siringa per aspirare il campione di cellula nell'altra estremità del tubo.
- Inserire con attenzione il tubo nell'ingresso del dispositivo.
- Collegare l'estremità opposta del tubo al regolatore di pressione microfluidica.
NOTA: È possibile aggiungere un filtro tra il regolatore di pressione microfluidica e il tubo per evitare il riflusso del liquido nel regolatore di pressione.
- Esegui l'esperimento.
- Impostare la pressione di guida costante desiderata sul software del regolatore di pressione e consentire al campione di riempire il dispositivo.
NOTA: La pressione è tipicamente 2-21 kPa. La velocità del flusso deve essere sufficientemente lenta da consentire impulsi chiaramente definiti, ma abbastanza veloce da consentire una portata adeguata.- Se si formano bolle nei canali microfluidici, utilizzare il riempimento senza uscita: collegare l'uscita del dispositivo e applicare una bassa pressione all'ingresso per forzare l'aria attraverso il PDMS permeabile al gas. Lasciare le bolle nel canale porterà a una linea di base corrente instabile e impedirà misurazioni accurate.
- Se i detriti ostruiscono il canale microfluidico, spostarlo premendo leggermente sulla parte superiore del dispositivo PDMS mentre si applica la pressione di guida, "pulsando" una pressione più alta attivando e disattivando la pressione o rimuovendo il tubo e reinserendolo. Se i detriti rimangono, potrebbe essere necessario passare a un nuovo dispositivo.
- Impostare la tensione desiderata ruotando la manopola Voltage sull'alimentatore e abilitare la tensione premendo il pulsante On .
NOTA: la tensione è in genere 1-5 V. Scegliere la tensione più bassa necessaria per un SNR adeguato. La stessa tensione dovrebbe essere utilizzata in tutte le condizioni da confrontare. - Accendere il preamplificatore di corrente e impostare la sensibilità (A/V) il più bassa possibile; in alternativa, impostare il guadagno (V/A) il più alto possibile senza sovraccaricare il preamplificatore o superare la tensione di ingresso analogica massima del DAQ. In questo studio, la sensibilità è stata impostata su 10-7 A / V.
NOTA: il valore corretto di sensibilità/guadagno dipenderà sia dalla tensione applicata che dalla resistenza di base del canale microfluidico. - Premere il pulsante verde Esegui nel menu della barra multifunzione di MATLAB per avviare lo script di acquisizione dati NPS.m e avviare il campionamento e il salvataggio dei dati.
- Per terminare l'esperimento, premere il pulsante Stop nell'angolo inferiore sinistro della finestra della figura per interrompere lo script di acquisizione dati. Disattivare l'uscita dell'alimentatore premendo il pulsante On . Impostare la sorgente di pressione su pressione zero nel software del regolatore di pressione.
- A questo punto, l'esperimento può essere sospeso per eseguire una o più delle operazioni seguenti:
- Sostituire il dispositivo corrente con uno nuovo.
- Ricaricare il tubo con più campioni di celle.
NOTA: Per evitare la contaminazione incrociata del campione, utilizzare nuovi dispositivi per misurare cellule di diversi tipi o condizioni. - Sblocca il dispositivo dal PCB ed esamina le condizioni del canale al microscopio. Per riavviare l'esperimento utilizzando lo stesso dispositivo, è necessario prestare attenzione a non introdurre bolle d'aria. Potrebbe essere necessario applicare una leggera pressione allo stantuffo della siringa per mantenere il campione di cella all'estremità del tubo mentre lo si inserisce nell'ingresso del dispositivo.
- Impostare la pressione di guida costante desiderata sul software del regolatore di pressione e consentire al campione di riempire il dispositivo.
- Caricare il campione di cella nel tubo e collegarlo all'ingresso del dispositivo.
4. Calibrare il dispositivo microfluidico
- Opzione 1: misurare le perle di polistirolo nei dispositivi di riferimento.
- Scegli una dimensione del tallone di polistirolo più piccola del canale di dimensionamento.
- Aggiungere l'1,5% di Tween e perle di polistirene alla soluzione filtrata PBS e FBS utilizzata durante gli esperimenti cellulari, ad una concentrazione di 1-3 x 105 perle / ml.
- Procedere con l'esperimento come descritto nella sezione 3, utilizzando il dispositivo di riferimento descritto al punto 1.3, e applicare la stessa tensione utilizzata durante la sperimentazione. Utilizzare l'entità media della goccia di corrente prodotta mentre le perle transitano nei pori di dimensionamento e il diametro noto delle perle per calcolare De, come descritto nella sezione 5.
- Opzione 2: misurare in modo indipendente la dimensione della cella con un dispositivo di misurazione separato.
- Invece di seguire il protocollo nel passaggio 4.1, utilizzare uno strumento di misurazione delle dimensioni delle celle disponibile in commercio per misurare la dimensione media delle cellule nel campione. In questo caso, non è necessario alcun dispositivo di riferimento. Utilizzare la caduta di corrente media prodotta mentre le celle transitano nel poro di dimensionamento e il diametro medio della cella misurato per calcolare De come descritto nella sezione 5.
5. Analizzare i dati per estrarre fenotipi cellulari
NOTA: L'elaborazione dei dati può essere eseguita utilizzando il file di programma dell'interfaccia a riga di comando MATLAB mNPS_procJOVE.m at https://github.com/sohnlab/NPS-analysis-JOVE. Per ulteriori istruzioni, vedere il file supplementare 6 .
- Pre-elaborare i dati (Figura 3A).
- Calcolare la corrente elettrica misurata applicando il valore di guadagno utilizzato nel preamplificatore corrente-tensione ai dati grezzi acquisiti dal DAQ.
- Rimuovere il rumore ad alta frequenza applicando una funzione di livellamento rettangolare e/o un filtro passa-basso alla misurazione della corrente grezza. Quindi, ricampionare i dati filtrati a una frequenza di campionamento inferiore. Inoltre, calcola i dati del timestamp corrispondenti a questa frequenza di campionamento inferiore.
- Calcolare un segnale di corrente di base adattato applicando un metodo come lo smoothing asimmetrico dei minimi quadrati26.
- Calcolare la derivata prima approssimativa (segnale di differenza) dei dati correnti pre-elaborati prendendo la differenza tra i punti dati successivi.
- Identificare gli eventi cellulari ed estrarre i dati del subimpulso (Figura 3B).
- Cercare gli eventi della cella candidata esaminando i dati pre-elaborati. Rifiutare gli eventi cellulari che si sovrappongono ad altri eventi cellulari (cioè eventi di coincidenza) (Figura supplementare 4), mostrano uno scarso adattamento basale o hanno una forma di impulso inaspettata o errata (ad esempio, dove potrebbe essere stato presente un intasamento nel canale).
- Estrai i dati del subimpulso per ogni evento cellulare.
- Ogni segmento del poro del nodo apparirà come un sottoimpulso corrispondente all'interno dell'impulso del segnale complessivo (Figure 1B, C). Identifica l'inizio di ogni impulso calcolando il punto temporale quando il segnale di differenza raggiunge un valore minimo locale. Identifica la fine di ogni sottoimpulso calcolando il timepoint quando il segnale di differenza raggiunge un valore massimo locale.
- Determinare la larghezza di ciascun impulso secondario come il tempo trascorso tra i punti di inizio e fine. Determinare l'ampiezza di ciascun sottoimpulso calcolando la media della differenza tra la corrente misurata e la corrente di base per tutti i punti dati tra i punti di inizio e di fine.
- Determinare il meccanofenotipo cellulare per ciascun evento cellulare in base ai dati del subimpulso.
- Determinare il diametro della cella d in base all'equazione definita da Deblois e Bean24:
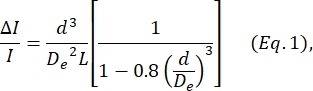
dove ΔI/I è il rapporto medio tra l'ampiezza del sottoimpulso e la corrente di base nei sottoimpulsi di dimensionamento, D e è il diametro effettivo del canale (misurato nella fase 4) e L è la lunghezza totale del canale nodo-pore.- D e è determinato calcolando il ΔI/I medio prodotto da un insieme di particelle di diametro noto (celle o perline, vedi punto 4), usando quel diametro noto come d e risolvendo Eq. 1 per De.
- Quantificare la resistenza della cellula alla deformazione.
- Determinare la velocità del fluido Uflusso calcolando la velocità media della cella nei sottoimpulsi di dimensionamento, utilizzando le lunghezze di segmento note e la durata misurata di ciascun sottoimpulso.
- Determinare l'indice di deformabilità dell'intera cellula (wCDI), definito da Kim et al.2 come:
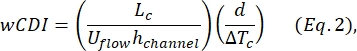
dove L c è la lunghezza del segmento di contrazione, il canale h è l'altezza del canale e ΔTc è la durata del sottoimpulso di contrazione.
- Identificare il tempo di recupero della cella dalla deformazione, definito come il primo sottoimpulso di recupero con un'ampiezza compresa nell'8% dell'ampiezza media dal sottoimpulso di dimensionamento2.
- Calcola la deformazione trasversale della cellula all'interno del segmento di contrazione.
- Calcola il diametro effettivo del segmento di contrazione (D e,c) come definito da Kim et al.2:
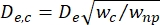 , dove w c è la larghezza del segmento di contrazione e wnp è la larghezza di tutti gli altri segmenti.
, dove w c è la larghezza del segmento di contrazione e wnp è la larghezza di tutti gli altri segmenti. - Calcolare il diametro sferico equivalente dc della cella all'interno della contrazione usando nuovamente l'equazione definita da Deblois e Bean24:
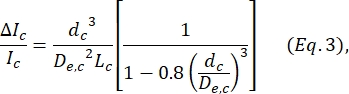
dove ΔI c/I c è il rapporto tra l'ampiezza del sottoimpulso e la corrente basale nel sottoimpulso di contrazione e Lc è la lunghezza del segmento di contrazione. - Calcola la lunghezza di allungamento della cella Ldeforma come descritto da Kim et al.2:
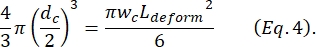
- Infine, calcolare la deformazione trasversale δdeformazione della cellula, che è definita da Kim et al.2 come
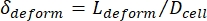 .
.
- Calcola il diametro effettivo del segmento di contrazione (D e,c) come definito da Kim et al.2:
- Determinare il diametro della cella d in base all'equazione definita da Deblois e Bean24:
Risultati
La piattaforma di meccanofenotipizzazione qui presentata è un approccio semplice e versatile per misurare le proprietà biofisiche di singole cellule con un rendimento moderato. Le cellule vengono fatte fluire attraverso il canale microfluidico (Figura 1A) utilizzando un flusso guidato da pressione costante. Durante il transito delle cellule, la lunghezza del canale microfluidico e gli impulsi di corrente prodotti vengono registrati utilizzando l'hardware di acquisizione dati. Il segnale ac...
Discussione
La misurazione delle proprietà meccaniche delle singole cellule utilizzando questa tecnica di meccanofenotipizzazione consiste in tre fasi: fabbricazione del dispositivo, acquisizione dei dati e analisi dei dati. All'interno di ogni fase, ci sono aspetti notevoli che possono avere un impatto significativo sui risultati sperimentali. Durante la fabbricazione del dispositivo, geometrie di canale coerenti e uniformità da dispositivo a dispositivo sono essenziali per risultati accurati e ripetibili. In particolare, le pare...
Divulgazioni
L. L. S detiene il brevetto statunitense n. 11.383.241: "Mechano-node-pore sensing", J. Kim, S. Han e L. L. Sohn, rilasciato il 12 luglio 2022.
Riconoscimenti
Questa ricerca è stata sostenuta da sovvenzioni NIBIB 1R01EB024989-01 e NCI 1R01CA190843-01. A. L. e R. R. sono stati supportati da una borsa di ricerca universitaria dell'Associazione H2H8. K. L. C. è stato supportato da una National Science Foundation Graduate Research Fellowship e da una Siebel Scholar Fellowship.
Materiali
| Name | Company | Catalog Number | Comments |
| Acetone | J.T. Baker | 5356-05 | Purity (GC) ≥ 99.5% (https://us.vwr.com/store/product/6057739/acetone-99-5-vlsi-j-t-baker) |
| Aluminum Foil | n/a | n/a | |
| Analog Low-Pass Filter | ThorLabs | EF504 | ≤240 kHz Passband, Coaxial BNC Feedthrough (https://www.thorlabs.com/thorproduct.cfm?partnumber=EF504#ad-image-0) |
| Biopsy Punch | Integra Miltex | 33-31AA-P/25 | 1mm, Disposable, with Plunger (https://mms.mckesson.com/product/573313/Miltex-33-31AA-P25) |
| Blade | n/a | n/a | |
| BNC Cable | Pomona Electronics | 2249-C-12 | https://www.digikey.com/en/products/detail/pomona-electronics/2249-C-12/603323?utm_adgroup=Coaxial%20Cables%20%28RF%29&utm_source=google&utm_ medium=cpc&utm_campaign= Shopping_Product_Cable%20Assemblies_NEW&utm_term= &utm_content=Coaxial%20Cables%20%28RF%29&gclid=Cj0KCQjwlK-WBhDjARIsAO2sErQqnVJ pj5OXVObuTI8ZUf1ZeIn7zvzGnx mCWdePrG6SdEJMF3X6ubUaAs w-EALw_wcB |
| Cleanroom Polyester Swab | Thermo Fisher Scientific | 18383 | https://www.fishersci.com/shop/products/texwipe-cleantip-alpha-polyester-series-swabs-6/18383 |
| Current Preamplifier | DL Instruments | 1211 | https://www.brltest.com/index.php?main_page=product_info&products_ id=1419 |
| Custom PCB (w/ components) | n/a | n/a | see Supplemental files 4 and 5 |
| DAQ Terminal Block | National Instruments | BNC-2120 | https://www.ni.com/en-in/support/model.bnc-2120.html |
| DAQ to BNC-2110 cable | National Instruments | SHC68-68-EPM | https://www.ni.com/en-in/support/model.shc68-68-epm.html |
| Data Acquisition Board (DAQ) | National Instruments | PCI-6251 | https://www.ni.com/docs/en-US/bundle/pci-6251-feature/page/overview.html |
| Dessicator | Thermo Fisher Scientific | 5311-0250 | https://www.thermofisher.com/order/catalog/product/5311-0250 |
| Female BNC To Banana Plug Adapter | Pomona Electronics | 72909 | https://www.digikey.com/en/products/detail/pomona-electronics/72909/1196318 |
| Fetal Bovine Serum (FBS) | VWR | 89510-186 | https://us.vwr.com/store/product/18706419/avantor-seradigm-select-grade-usda-approved-origin-fetal-bovine-serum-fbs |
| Glass Cutter | Chemglass | CG-1179-21 | https://chemglass.com/plate-glass-cutters-diamond-tips |
| Gold Etchant TFA | Transene | NC0977944 | https://www.fishersci.com/shop/products/NC0977944/NC0977944 |
| Hot Plate | Thermo Fisher Scientific | SP131825 | |
| Isopropyl Alcohol | Spectrum Chemical | I1056-4LTPL | Purity (GC) ≥99.5% (https://www.spectrumchemical.com/isopropyl-alcohol-99-percent-fcc-i1056) |
| Metal Hardware Enclosure | Hammond Manufacturing | EJ12126 | https://www.digikey.com/en/products/detail/hammond-manufacturing/EJ12126/2423415 |
| Methanol | Sigma-Aldrich | 34860 | Purity (GC) ≥99.8% (https://www.sigmaaldrich.com/IN/en/substance/methanol320467561) |
| MF-321 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/mf-321/ |
| MICROPOSIT S1813 Positive Photoresist | DuPont | n/a | https://kayakuam.com/products/microposit-s1800-g2-series-photoresists/ |
| Phosphate Buffered Saline (PBS) | Thermo Fisher Scientific | 10010049 | https://www.thermofisher.com/order/catalog/product/10010049?SID=srch-hj-10010049 |
| Photomask | Fineline Imaging | n/a | Photomask are custom ordered from our CAD designs (https://www.fineline-imaging.com/) |
| Plain Glass Microscope Slide | Fisher Scientific | 12-553-5B | Material: Soda Lime, L75 x W50 mm, Thickness: 0.90–1.10 mm |
| Plasma Cleaner | Harrick Plasma | PDC-001 | https://harrickplasma.com/plasma-cleaners/expanded-plasma-cleaner/ |
| Plastic Petri Dish | Thermo Fisher Scientific | FB0875712 | 100 mm (https://www.fishersci.com/shop/products/fisherbrand-petri-dishes-clear-lid-raised-ridge-100-x-15mm/FB0875712) |
| Pressure Controller | Fluigent | MFCS-EZ | https://www.fluigent.com/research/instruments/pressure-flow-controllers/mfcs-series/ |
| Pressure Controller Software | Fluigent | MAESFLO | |
| Programming & Computation Software | MATLAB | R2021b | for data acquisition and analysis (https://www.mathworks.com/products/matlab.html) |
| PTFE Tubing | Cole Parmer | 06417-31 | 0.032" ID x 0.056" (https://www.coleparmer.com/i/masterflex-transfer-tubing-microbore-ptfe-0-032-id-x-0-056-od-100-ft-roll/0641731) |
| Scepter 2.0 Handheld Automatic Cell Counter | Millapore Sigma | PHCC20060 | https://www.sigmaaldrich.com/IN/en/product/mm/phcc20060 |
| Silicon Wafer | Wafer World | 2885 | 76.2 mm, Single Side Polished (https://www.waferworld.com/product/2885) |
| Spin Coater | n/a | n/a | |
| SU-8 3025 Negative Photoresist | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-2000/ |
| SU8 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-developer/ |
| Sygard 184 Polydimethlysiloxane | Dow Chemical | 4019862 | https://www.ellsworth.com/products/by-market/consumer-products/encapsulants/silicone/dow-sylgard-184-silicone-encapsulant-clear-0.5-kg-kit/ |
| Tape | Scotch | 810-341296 | https://www.staples.com/Scotch-Magic-Tape-810-3-4-x-36-yds-1-Core/product_130567?cid=PS:GS:SBD:PLA:OS&gclid= Cj0KCQjwlK-WBhDjARIsAO 2sErRwzrrgjU0NjFkDkne1xm vT7ekS3tdzvAgiMDwPoxocgH VTQZi7vJgaAvQZEALw_wcB |
| Titanium, Platinum, Gold | n/a | n/a | |
| Triple Output Power Supply | Keysight | E36311A | https://www.newark.com/keysight-technologies/e36311a/dc-power-supply-3o-p-6v-5a-prog/dp/15AC9653 |
| UV Mask Aligner | Karl Suss America | MJB3 Mask Aligner |
Riferimenti
- Pegoraro, A. F., Janmey, P., Weitz, D. A. Mechanical properties of the cytoskeleton and cells. Cold Spring Harbor Perspectives in Biology. 9 (11), 022038 (2017).
- Kim, J., et al. Characterizing cellular mechanical phenotypes with mechano-node-pore sensing. Microsystems & Nanoengineering. 4, 17091 (2018).
- Mierke, C. T. Bidirectional mechanical response between cells and their microenvironment. Frontiers in Physics. 9, 619 (2021).
- Kumar, S., Weaver, V. M. Mechanics, malignancy, and metastasis: The force journey of a tumor cell. Cancer and Metastasis Reviews. 28 (1), 113-127 (2009).
- Nia, H. T., Munn, L. L., Jain, R. K. Physical traits of cancer. Science. 370 (6516), (2020).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463 (7280), 485-492 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: The role of physical interactions and mechanical forces in metastasis. Nature Reviews Cancer. 11 (7), 512-522 (2011).
- Li, B., et al. Mechanical phenotyping reveals unique biomechanical responses in retinoic acid-resistant acute promyelocytic leukemia. iScience. 25 (2), 103772 (2022).
- Kozminsky, M., Sohn, L. L. The promise of single-cell mechanophenotyping for clinical applications. Biomicrofluidics. 14 (3), 031301 (2020).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Wottawah, F., et al. Optical rheology of biological cells. Physical Review Letters. 94 (9), 1-4 (2005).
- Darling, E. M., Di Carlo, D. High-throughput assessment of cellular mechanical properties. Annual Review of Biomedical Engineering. 17 (1), 35-62 (2015).
- Carey, T. R., Cotner, K. L., Li, B., Sohn, L. L. Developments in label-free microfluidic methods for single-cell analysis and sorting. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 11 (1), 1529 (2019).
- Bagnall, J. S., et al. Deformability of tumor cells versus blood cells. Scientific Reports. 5, 18542 (2015).
- Byun, S., et al. Characterizing deformability and surface friction of cancer cells. Proceedings of the National Academy of Sciences. 110 (19), 7580-7585 (2013).
- Otto, O., et al. Real-time deformability cytometry: On-the-fly cell mechanical phenotyping. Nature Methods. 12 (3), 199-202 (2015).
- Gossett, D. R., et al. Hydrodynamic stretching of single cells for large population mechanical phenotyping. Proceedings of the National Academy of Sciences. 109 (20), 7630-7635 (2012).
- Guck, J., Chilvers, E. R. Mechanics meets medicine. Science Translational Medicine. 5 (212), 3-6 (2013).
- Balakrishnan, K. R., et al. Node-pore sensing: A robust, high-dynamic range method for detecting biological species. Lab on a Chip. 13 (7), 1302-1307 (2013).
- Carbonaro, A., Sohn, L. L. A resistive-pulse sensor chip for multianalyte immunoassays. Lab on a Chip. 5 (10), 1155-1160 (2005).
- Saleh, O. A., Sohn, L. L. Direct detection of antibody-antigen binding using an on-chip artificial pore. Proceedings of the National Academy of Sciences. 100 (3), 820-824 (2003).
- Saleh, O. A., Sohn, L. L. An artificial nanopore for molecular sensing. Nano Letters. 3 (1), 37-38 (2003).
- Saleh, O. A., Sohn, L. L. Quantitative sensing of nanoscale colloids using a microchip Coulter counter. Review of Scientific Instruments. 72 (12), 4449-4451 (2001).
- DeBlois, R. W., Bean, C. P. Counting and sizing of submicron particles by the resistive pulse technique. Review of Scientific Instruments. 41 (7), 909-916 (1970).
- Li, B., et al. Evaluating sources of technical variability in the mechano-node-pore sensing pipeline and their effect on the reproducibility of single-cell mechanical phenotyping. PLoS ONE. 16 (10), 0258982 (2021).
- Zhang, Z. M., Chen, S., Liang, Y. Z. Baseline correction using adaptive iteratively reweighted penalized least squares. Analyst. 135 (5), 1138-1146 (2010).
- Alibert, C., Goud, B., Manneville, J. B. Are cancer cells really softer than normal cells. Biology of the Cell. 109 (5), 167-189 (2017).
- Fujiwara, I., Zweifel, M. E., Courtemanche, N., Pollard, T. D. Latrunculin A accelerates actin filament depolymerization in addition to sequestering actin monomers. Current Biology. 28 (19), 3183-3192 (2018).
- Saleh, O. A. . A novel resistive pulse sensor for biological measurements. , (2003).
- Dokukin, M. E., Guz, N. V., Sokolov, I. Quantitative study of the elastic modulus of loosely attached cells in AFM indentation experiments. Biophysical Journal. 104 (10), 2123-2131 (2013).
- Li, Q., Lim, C. T., Goh, J. C. H., et al. Probing the elasticity of breast cancer cells using AFM. 13th International Conference on Biomedical Engineering. IFMBE Proceedings. 23, 2122-2125 (2009).
- Rother, J., et al. Atomic force microscopy-based microrheology reveals significant differences in the viscoelastic response between malign and benign cell lines. Open Biology. 4 (5), 140046 (2014).
- Li, Q., et al. AFM indentation study of breast cancer cells. Biochemical and Biophysical Research Communications. 374 (4), 609-613 (2008).
- Xu, C., et al. Elasticity measurement of breast cancer cells by atomic force microscopy. Proc. SPIE 9230. Twelfth International Conference on Photonics and Imaging in Biology and Medicine. (PIBM 2014). 92300, (2014).
- Alcaraz, J., et al. Microrheology of human lung epithelial cells measured by atomic force microscopy. Biophysical Journal. 84 (3), 2071-2079 (2003).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Urbanska, M., et al. A comparison of microfluidic methods for high-throughput cell deformability measurements. Nature Methods. 17, 587-593 (2020).
- Hill, R. T., Chilkoti, A. Surface Patterning. Biomaterials Science: An Introduction to Materials: Third Edition. , 276-301 (2013).
- Wang, Z., Volinsky, A. A., Gallant, N. D. Crosslinking effect on polydimethylsiloxane elastic modulus measured by custom-built compression instrument. Journal of Applied Polymer Science. 131 (22), 41050 (2014).
- Gibson, L. J. The hierarchical structure and mechanics of plant materials. Journal of the Royal Society Interface. 9 (76), 2749-2766 (2012).
- Stephens, A. D., Banigan, E. J., Adam, S. A., Goldman, R. D., Marko, J. F. Chromatin and lamin a determine two different mechanical response regimes of the cell nucleus. Molecular Biology of the Cell. 28 (14), 1984-1996 (2017).
- Rosenbluth, M. J., Lam, W. A., Fletcher, D. A. Force microscopy of nonadherent cells: A comparison of leukemia cell deformability. Biophysical Journal. 90 (8), 2994-3003 (2006).
- Evers, T. M. J., Holt, L. J., Alberti, S., Mashaghi, A. Reciprocal regulation of cellular mechanics and metabolism. Nature Metabolism. 3 (4), 456-468 (2021).
- Balakrishnan, K. R., et al. Node-pore sensing enables label-free surface-marker profiling of single cells. Analytical Chemistry. 87 (5), 2988-2995 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon