Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mechano-node-pore Sensing: une plate-forme rapide et sans marquage pour les mesures viscoélastiques multi-paramètres à cellule unique
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Présenté ici est une méthode pour phénotyper mécaniquement des cellules individuelles en utilisant une plate-forme microfluidique basée sur l’électronique appelée détection mécano-nœud-pore (mécano-NPS). Cette plateforme maintient un débit modéré de 1 à 10 cellules/s tout en mesurant les propriétés biophysiques élastiques et visqueuses des cellules.
Résumé
Les propriétés mécaniques cellulaires sont impliquées dans une grande variété de processus biologiques et de maladies, allant de la différenciation des cellules souches aux métastases cancéreuses. Les méthodes conventionnelles de mesure de ces propriétés, telles que la microscopie à force atomique (AFM) et l’aspiration par micropipette (MA), capturent des informations riches, reflétant la réponse viscoélastique complète d’une cellule; Cependant, ces méthodes sont limitées par un très faible débit. Les approches à haut débit, telles que la cytométrie de déformabilité en temps réel (RT-DC), ne peuvent mesurer que des informations mécaniques limitées, car elles sont souvent limitées à des lectures à paramètre unique qui ne reflètent que les propriétés élastiques d’une cellule. Contrairement à ces méthodes, la détection mécano-nœud-pores (mécano-NPS) est une plate-forme microfluidique flexible et sans marquage qui comble l’écart dans la réalisation de mesures viscoélastiques multiparamètres d’une cellule avec un débit modéré. Une mesure en courant continu (DC) est utilisée pour surveiller les cellules lorsqu’elles transitent dans un canal microfluidique, en suivant leur taille et leur vitesse avant, pendant et après qu’elles soient forcées à travers une constriction étroite. Cette information (c.-à-d. la taille et la vitesse) est utilisée pour quantifier la déformation transversale, la résistance à la déformation et la récupération de chaque cellule. En général, cette plate-forme microfluidique basée sur l’électronique fournit de multiples propriétés de cellule viscoélastique, et donc une image plus complète de l’état mécanique d’une cellule. Parce qu’elle nécessite une préparation minimale des échantillons, utilise une mesure électronique simple (contrairement à une caméra haute vitesse) et tire parti de la fabrication standard par lithographie douce, la mise en œuvre de cette plate-forme est simple, accessible et adaptable à l’analyse en aval. La flexibilité, l’utilité et la sensibilité de cette plate-forme ont fourni des informations mécaniques uniques sur une gamme variée de cellules, avec le potentiel de nombreuses autres applications en science fondamentale et en diagnostic clinique.
Introduction
Les cellules individuelles sont des matériaux dynamiques et viscoélastiques1. Une multitude de processus internes et externes (par exemple, l’apparition de la mitose ou le remodelage de la matrice extracellulaire [ECM]), influencent leur structure et leur composition 2,3,4, ce qui entraîne souvent des propriétés biophysiques distinctes qui complètent leur état actuel. En particulier, les propriétés mécaniques se sont révélées être des biomarqueurs importants du développement cellulaire, de la physiologie et de la pathologie, fournissant des informations quantitatives précieuses qui peuvent compléter les approches moléculaires et génétiques canoniques 5,6,7. Par exemple, Li et al. ont récemment décrit les différences mécaniques entre les cellules de leucémie promyélocytaire aiguë résistantes aux médicaments et les cellules de leucémie promyélocytaire sensibles aux médicaments, tout en utilisant le séquençage de l’ARN pour découvrir des gènes associés au cytosquelette exprimés différentiellement8. En comprenant l’interaction complexe entre la mécanique unicellulaire et la fonction cellulaire, le mécanophénotypage a des applications plus larges dans la transformation de la science fondamentale et du diagnostic clinique9.
L’outil le plus largement adopté pour mesurer la mécanique unicellulaire est la microscopie à force atomique (AFM). Bien que l’AFM permette une mesure localisée à haute résolution des propriétés mécaniques cellulaires, il reste limité à un débit de <0,01 cellules/s10. Alternativement, les civières optiques, qui utilisent deux faisceaux laser divergents pour piéger et déformer les cellules individuelles suspendues11, sont limitées à des débits légèrement plus élevés de <1 cellule/s12. Les progrès récents des technologies microfluidiques ont permis une nouvelle génération de dispositifs pour l’évaluation mécanique rapide à cellule unique12,13. Ces techniques utilisent des canaux de constriction étroits 14,15, un écoulement de cisaillement 16 ou un étirement hydrodynamique17 pour déformer rapidement les cellules à des débits de 10 à 1 000 cellules/s 18. Bien que le taux de mesure de ces approches soit considérablement plus rapide que les techniques conventionnelles, elles échangent souvent des capacités à haut débit contre des lectures mécaniques limitées (tableau supplémentaire 1). Toutes les méthodes microfluidiques rapides susmentionnées se concentrent sur des mesures de base à paramètre unique, telles que le temps de transit ou les rapports de déformabilité, qui ne reflètent que les propriétés élastiques d’une cellule. Cependant, étant donné la nature viscoélastique intrinsèque des cellules individuelles, une caractérisation mécanique robuste et approfondie des cellules nécessite de prendre en compte non seulement les composants élastiques, mais aussi les réponses visqueuses.
Mechano-node-pore sensing (mechano-NPS)2,8 (Figure 1A) est une plateforme microfluidique qui répond aux limitations existantes du mécano-phénophénotypage unicellulaire. Cette méthode permet de mesurer simultanément plusieurs paramètres biophysiques, y compris le diamètre de la cellule, la déformabilité relative et le temps de récupération de la déformation, avec un débit modéré de 1 à 10 cellules/s. Cette technique est basée sur la détection des pores des nœuds (NPS)19,20,21,22,23,24, qui consiste à utiliser une mesure de sonde à quatre points pour mesurer l’impulsion de courant modulée produite par une cellule transitant par un canal microfluidique qui a été segmenté par des régions plus larges, appelées « nœuds ». L’impulsion de courant modulée est le résultat du blocage partiel par la cellule du flux de courant dans les segments (c’est-à-dire les « pores ») et les nœuds, avec plus de courant bloqué dans le premier que dans le second. Dans le mécano-NPS, un segment, le « canal de contraction », est plus étroit qu’un diamètre de cellule ; par conséquent, une cellule doit se déformer pour transiter par tout le canal (Figure 1B). Le diamètre de la cellule peut être déterminé par l’amplitude de la sous-impulsion produite lorsque la cellule transite par les pores des nœuds avant le canal de contraction (figures 1B, C). Ici, |ΔInp|, la baisse de courant lorsque la cellule est dans le pore, est proportionnelle au rapport volumique de la cellule au pore, Vcellule/V pore2,8,19. La rigidité cellulaire peut être déterminée par ΔTc, la durée de la sous-impulsion considérablement plus grande produite lorsque la cellule transite dans le canal de contraction (figures 1B, C). Une cellule plus rigide mettra plus de temps à transiter dans le canal qu’une cellule plus molle 2,8. Enfin, la « récupération » cellulaire, c’est-à-dire la capacité de la cellule à retrouver sa taille et sa forme d’origine après la déformation, peut être déterminée par la série de sous-impulsions produites lorsque la cellule transite par les pores des nœuds après le canal de contraction (figures 1B, C). Le temps de récupération, ΔTr, est le temps qu’il faut aux sous-impulsions actuelles pour revenir à l’amplitude des sous-impulsions précédentes, avant que la cellule ne soit comprimée. Dans l’ensemble, les impulsions de courant modulées produites lorsqu’une cellule transite dans le canal microfluidique sont enregistrées et analysées pour extraire les paramètres mécaniques pertinents de cellule unique (Figure 1D)2,8.
La reproductibilité et la facilité d’utilisation de cette plateforme microfluidique basée sur l’électronique ont déjà été démontrées25. De plus, la plateforme présente une faible barrière à l’entrée pour le mécanophénotypage unicellulaire. La lithographie douce standard est utilisée pour fabriquer des dispositifs microfluidiques. Le matériel de mesure se compose de composants peu coûteux, y compris une simple carte de circuit imprimé (PCB), une alimentation, un préamplificateur, une carte d’acquisition de données (DAQ) et un ordinateur. Enfin, un code convivial est disponible pour l’acquisition et l’analyse des données, permettant une mise en œuvre simple. Cette technique de mécanophénotypage permet de distinguer des populations de lignées épithéliales mammaires et pulmonaires malignes et malignes, de discriminer entre les sous-lignées dans les cellules épithéliales mammaires humaines primaires et de caractériser les effets des perturbations cytosquelettiques et d’autres agents pharmacologiques 2,8. Dans l’ensemble, cette plateforme est une approche efficace pour le mécanoophénotypage de cellules individuelles.
Protocole
1. Concevoir la géométrie de l’appareil
- Choisissez la largeur des segments de dimensionnement et de récupération afin qu’elle soit plus large que le diamètre des plus grandes cellules à mesurer, tout en maintenant un rapport signal sur bruit (SNR) suffisant. Voir le tableau supplémentaire 2 pour des exemples de différentes largeurs de dimensionnement et de segment de récupération pour diverses lignées cellulaires.
- Choisissez la largeur du segment de contraction pour appliquer une souche de 30% à 40% à la taille moyenne des cellules qui doivent subir un mécanophénotypage. La déformation est définie par
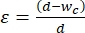 , où d est le diamètre de la cellule et wc est la largeur du canal de contraction 2,8. Voir le tableau supplémentaire 2 pour différentes largeurs de segments de contraction pour diverses lignées cellulaires.
, où d est le diamètre de la cellule et wc est la largeur du canal de contraction 2,8. Voir le tableau supplémentaire 2 pour différentes largeurs de segments de contraction pour diverses lignées cellulaires.
REMARQUE : Si l’on souhaite comparer des types de cellules ou des conditions ayant des diamètres sensiblement différents, des conceptions de dispositifs distinctes doivent être utilisées avec des largeurs de segment de contraction spécifiques à chaque type ou condition de cellule. - Concevez un dispositif de référence pour chaque géométrie de périphérique unique. Ceci est nécessaire pour déterminer De, le diamètre effectif du segment de pores de dimensionnement du canal microfluidique.
Remarque : Le périphérique de référence utilise la même géométrie que le périphérique principal. La seule modification est que le segment de contraction doit avoir une largeur égale à celle du segment de pores de dimensionnement pour permettre l’étalonnage avec des billes de polystyrène d’une taille connue. L’élargissement de la contraction empêche les billes de polystyrène de boucher le canal de contraction pendant l’étalonnage. Le processus d’étalonnage est décrit plus en détail aux étapes 4.1 et 5.3.1. L’étalonnage peut également être réalisé à l’aide d’un compteur de cellules disponible dans le commerce, auquel cas aucun dispositif de référence n’est nécessaire. Ce processus est décrit à l’étape 4.2. - Choisissez la hauteur du canal de telle sorte que les plus grandes cellules d’intérêt puissent s’allonger complètement sans restriction dans le segmentde contraction 2. Assurez-vous que la hauteur du canal est supérieure à hmin
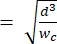 (cela suppose que la cellule est une prédéformation sphérique et que la déformation isométrique se produit le long de la longueur et de la hauteur du canal pendant la déformation).
(cela suppose que la cellule est une prédéformation sphérique et que la déformation isométrique se produit le long de la longueur et de la hauteur du canal pendant la déformation).
REMARQUE: Compte tenu de l’ampleur d’une sous-impulsion de courant, , plus le hmin est grand,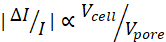 plus le SNR global sera faible.
plus le SNR global sera faible. - Concevez et créez un photomasque à l’aide d’un logiciel de conception assistée par ordinateur avec les largeurs de canal choisies. Un exemple de fichier est fourni dans le dossier supplémentaire 1. Dimensionner la conception du masque microfluidique de 1,5 % pour tenir compte du retrait du polydiméthylsiloxane (PDMS) après le décollement du masque négatif.
REMARQUE : Un tableau de périphériques peut être inclus sur un seul masque tant que le tableau global ne dépasse pas la taille de la plaquette (Figure supplémentaire 1A). - Concevoir et créer un photomasque avec des électrodes qui seront utilisées pour effectuer une mesure de sonde à quatre points du courant du dispositif microfluidique (Figure 1D). Un exemple de fichier est fourni dans le dossier supplémentaire 1.
REMARQUE : Un réseau d’électrodes peut être inclus sur un seul masque tant que le réseau ne dépasse pas la taille de la lame de verre (Figure supplémentaire 1B).
2. Fabriquer des appareils (Figure 2)
- Préparer des motifs d’électrodes sur un substrat en verre.
- Essorez, modelez et traitez une résine photosensible positive sur une lame de verre ordinaire conformément à la fiche technique du produit. Un exemple de cette procédure est présenté dans le dossier supplémentaire 2.
- Effectuer le dépôt de métal, le décollage et la gravure à l’or.
- Effectuer un dépôt de film mince de 75 Å Ti, 250 Å Pt et 250 Å Au sur la lame. Un exemple de cette procédure utilisant l’évaporation par canon à électrons est présenté dans le dossier supplémentaire 3.
- Plongez la lame dans l’acétone pendant 15 min pour effectuer un décollage de l’excès de métal.
- Dans une hotte, utiliser une pipette jetable pour déposer une gravure en or coulé sur la région des électrodes qui seront exposées au canal microfluidique, comme illustré à la figure supplémentaire 2. Soyez prudent pour éviter de laisser tomber des gravures ailleurs sur la diapositive.
ATTENTION : La gravure de l’or peut provoquer une irritation de la peau et des yeux. Ne respirez pas de vapeurs et n’ingérez pas. Manipulez avec soin, portez un équipement de protection individuelle (EPI) approprié et jetez les déchets conformément aux réglementations locales en matière d’élimination. - Rincer la lame à l’eau désionisée (DI) et la sécher à l’azote sec (N2).
- Si plusieurs électrodes sont imprimées sur la même lame de verre, coupez la lame en copeaux individuels.
- Utilisez un outil de coupe de verre pour marquer la lame le long des limites de l’électrode à motifs.
- Cassez le verre le long de la partition pour diviser la diapositive en puces individuelles.
- Inspectez visuellement les électrodes au microscope. Assurez-vous que les électrodes individuelles ne sont pas ouvertes électriquement ou que les électrodes ne sont pas court-circuitées ensemble.
- Fabriquez un moule maître négatif pour les canaux.
- Enduire de rotation, modeler et traiter une résine époxy SU-8 sur une plaquette de silicium poli conformément à la fiche technique du produit. Un exemple de cette procédure est présenté dans le dossier supplémentaire 2.
- Mesurez la hauteur des caractéristiques à l’aide d’un profilomètre et inspectez visuellement les caractéristiques au microscope (figure supplémentaire 3). Assurez-vous que les géométries souhaitées sont bien définies.
- Mouler les canaux PDMS avec lithographie douce.
- Préparez le PDMS en pesant un élastomère et un agent de réticulation à un rapport massique de 10:1 dans un gobelet jetable.
REMARQUE: Pour une plaquette de 3 pouces de diamètre, 30 g de PDMS suffisent. - Mélanger vigoureusement le PDMS pendant 30 s avec une fourchette jetable, jusqu’à ce que le PDMS soit opaque avec des bulles.
- Dégazez le PDMS dans une chambre à vide pendant environ 30 à 90 minutes, ou jusqu’à ce que le PDMS soit transparent sans bulles visibles.
- Placez la plaquette avec le moule maître SU-8 dans une boîte de Petri jetable et versez PDMS au centre de la plaquette.
- Placer la boîte de Petri contenant le PDMS et la plaquette dans une chambre à vide et dégazer pendant environ 30 minutes, ou jusqu’à ce qu’il ne reste plus de bulles dans le PDMS.
- Cuire le PDMS à 80 °C pendant 2 h dans un four ou sur une plaque chauffante.
- À l’aide d’une lame tranchante, coupez et retirez le PDMS du maître négatif SU-8.
- Découper la dalle PDMS moulée en moules individuels à l’aide d’une lame tranchante
- Creusez les trous d’accès à l’entrée et à la sortie à l’aide d’un poinçon de biopsie jetable. Pour de meilleurs résultats, utilisez un nouveau poinçon pour chaque dalle PDMS. Un poinçon plus net produit des trous lisses minimisant les particules qui pourraient obstruer le canal de contraction.
REMARQUE : Le diamètre des trous d’accès doit être légèrement inférieur au diamètre extérieur du tube. Par exemple, si vous utilisez des tubes en polytétrafluoroéthylène (PTFE) d’un diamètre extérieur de 1/32 po, un trou de 1,5 mm doit être percé.
- Préparez le PDMS en pesant un élastomère et un agent de réticulation à un rapport massique de 10:1 dans un gobelet jetable.
- Collez un substrat de verre/électrode aux canaux PDMS.
- Nettoyez les lames de verre d’électrode avec du méthanol (≥99,8%). Sécher avec le N2 sec.
- Nettoyez le PDMS avec du scotch, puis rincez à l’alcool isopropylique (IPA) et à l’eau désionisée (DI; 18 MΩ/cm2). Sécher avec le N2 sec. Ensuite, nettoyez à nouveau avec du scotch.
- Placez le substrat en verre avec des électrodes préfabriquées et le moule PDMS préparé (côté caractéristique vers le haut) dans un nettoyant plasma.
- Exposer les deux au plasma d’oxygène pendant 2 min (100-300 mTorr, 30 W).
- Alignez et placez le moule PDMS avec le côté caractéristique face vers le bas sur le substrat de verre avec des électrodes préfabriquées.
REMARQUE: Le collage est instantané une fois que le PDMS traité au plasma et le verre entrent en contact; Par conséquent, d’autres modifications de l’alignement ne seront pas possibles. Pour faciliter l’alignement, 20 μL d’une dilution 2:1 de méthanol dans de l’eau DI peuvent être pipetés sur la surface du verre traité au plasma. La solution de méthanol agit comme une barrière physique entre le verre traité et le PDMS, permettant des ajustements d’alignement. Si vous utilisez du méthanol, cuire le dispositif aligné et accouplé à 50 °C pendant 2 h pour évaporer la solution et terminer le processus de liaison. - Inspectez visuellement l’appareil collé au microscope. Assurez-vous que les électrodes et les géométries des canaux sont correctement alignées.
3. Mesurer les cellules (Figure 1D)
- Préparez la source de pression, le circuit imprimé, le matériel de paillasse et le logiciel d’acquisition de données.
- Connectez le dispositif microfluidique au PCB à l’aide de la pince. Un exemple de PCB est fourni dans le fichier supplémentaire 4 (fichiers GERBER) et le fichier supplémentaire 5 (fichiers schématiques, cartes et listes de pièces PCB).
- Alignez les broches à ressort de la pince avec les plaquettes de contact d’électrode sur le dispositif microfluidique et alignez les goupilles d’en-tête de la pince avec les trous sur le circuit imprimé.
- Insérez fermement les goupilles d’en-tête de la pince dans les trous du circuit imprimé, en vous assurant que les broches à ressort restent alignées avec les plaquettes de contact des électrodes.
- Configurez et connectez le matériel électronique.
- Connectez deux des ports de sortie de l’alimentation au port de tension d’alimentation du circuit imprimé à l’aide d’un adaptateur femelle BNC (BNC) double fiche banane à baïonnette Neill-Concelman (BNC) et d’un câble BNC.
- Mettez le bloc d’alimentation sous tension. Réglez la sortie connectée au conducteur interne du BNC sur +15 V et réglez l’autre sortie sur -15 V. Activez les deux sorties pour alimenter le circuit.
- Connectez le troisième des ports de sortie de l’alimentation au port de tension d’entrée du circuit imprimé à l’aide d’un câble BNC. Réglez la sortie sur la tension appliquée souhaitée, mais ne l’activez pas avant le début de l’expérience.
- Connectez le port de courant de sortie du PCB à l’entrée du préamplificateur de courant à l’aide d’un câble BNC.
- Connectez la sortie du préamplificateur de courant à une entrée analogique sur le bornier BNC du système d’acquisition de données à l’aide d’un câble BNC. En option, connectez un filtre passe-bas analogique aligné avec le câble BNC pour filtrer les interférences haute fréquence.
REMARQUE: Pour améliorer le SNR, le PCB et le dispositif peuvent être logés dans un boîtier métallique épais. Tous les câbles BNC et les tubes fluidiques peuvent être acheminés à travers des trous percés dans le boîtier.
- Installer et configurer le logiciel requis sur l’ordinateur personnel (PC)
- Mettez sous tension et connectez le régulateur de pression au PC. Installez tout logiciel de régulateur de pression requis conformément aux instructions du fabricant.
- Installez MATLAB et Data Acquisition Toolbox sur le PC. Assurez-vous que les pilotes requis pour le système d’acquisition de données sont installés afin que l’interface de MATLAB Data Acquisition Toolbox puisse le détecter.
- Téléchargez le script d’acquisition de données inclus, « NPS.m », à partir de https://github.com/sohnlab/node-pore-sensing-public.
- Ouvrez et configurez le script d’acquisition de données.
- Définissez les valeurs correctes pour initialiser la session d’acquisition de données, qui inclut l’ID fournisseur, l’ID de périphérique du DAQ et le numéro de canal d’entrée analogique (lignes 34 à 36 dans le script inclus).
REMARQUE: L’ID de périphérique peut être trouvé à l’aide de la fonction « daq.getDevices » ou « daqlist ». - Définissez la fréquence d’échantillonnage souhaitée pour l’acquisition (ligne 23 dans le script inclus). Pour des résultats optimaux, il doit être réglé sur au moins 10 kHz.
- Définissez les valeurs correctes pour initialiser la session d’acquisition de données, qui inclut l’ID fournisseur, l’ID de périphérique du DAQ et le numéro de canal d’entrée analogique (lignes 34 à 36 dans le script inclus).
- Connectez le dispositif microfluidique au PCB à l’aide de la pince. Un exemple de PCB est fourni dans le fichier supplémentaire 4 (fichiers GERBER) et le fichier supplémentaire 5 (fichiers schématiques, cartes et listes de pièces PCB).
- Préparez la suspension cellulaire.
- Préparer une solution de sérum fœtal bovin à 2% (FBS) dans 1x solution saline tamponnée au phosphate (PBS) et filtrer avec un filtre de 0,22 μm.
- Culture et préparation des cellules selon le protocole de culture cellulaire approprié de la lignée cellulaire de choix. Suspendre les cellules dans la solution préparée de FBS à 2% dans 1x PBS à une concentration de 1-5 x 105 cellules / mL. Gardez les cellules sur la glace pendant toute la durée des expériences.
- Mesurer les propriétés physiques des cellules.
- Chargez l’échantillon de cellule dans le tube et connectez-le à l’entrée de l’appareil.
- Coupez 30 cm de tube en PTFE avec une lame de rasoir ou un couteau tranchant.
- Fixez une extrémité du tube à une seringue Luer Lock. Utilisez la seringue pour aspirer l’échantillon de cellule à l’autre extrémité du tube.
- Insérez délicatement le tube dans l’entrée de l’appareil.
- Connectez l’extrémité opposée du tube au régulateur de pression microfluidique.
REMARQUE: Un filtre peut être ajouté entre le régulateur de pression microfluidique et le tube pour empêcher le refoulement de liquide dans le régulateur de pression.
- Exécutez l’expérience.
- Réglez la pression d’entraînement constante souhaitée sur le logiciel du régulateur de pression et laissez l’échantillon remplir l’appareil.
REMARQUE: La pression est généralement de 2-21 kPa. La vitesse d’écoulement doit être suffisamment lente pour permettre des impulsions clairement définies, mais suffisamment rapide pour permettre un débit adéquat.- Si des bulles se forment dans les canaux microfluidiques, utilisez un remplissage en cul-de-sac : branchez la prise de l’appareil et appliquez une faible pression à l’entrée pour forcer l’air à sortir à travers le PDMS perméable aux gaz. Laisser des bulles dans le canal conduira à une ligne de base de courant instable et empêchera des mesures précises.
- Si des débris obstruent le canal microfluidique, délogez-le en appuyant légèrement sur le dessus du dispositif PDMS tout en appliquant la pression d’entraînement, en « pulsant » une pression plus élevée en activant et en désactivant la pression, ou en retirant le tube et en le réinsérant. Si les débris restent, il peut être nécessaire de passer à un nouvel appareil.
- Réglez la tension souhaitée en tournant le bouton Tension sur l’alimentation et activez la tension en appuyant sur le bouton On .
REMARQUE: La tension est généralement de 1 à 5 V. Choisissez la tension la plus basse nécessaire pour un SNR adéquat. La même tension doit être utilisée dans toutes les conditions à comparer. - Allumez le préamplificateur de courant et réglez la sensibilité (A/V) aussi faible que possible ; Vous pouvez également régler le gain (V/A) aussi haut que possible sans surcharger le préamplificateur ou dépasser la tension d’entrée analogique maximale du DAQ. Dans cette étude, la sensibilité a été fixée à 10-7 A / V.
REMARQUE: La valeur de sensibilité/gain appropriée dépendra à la fois de la tension appliquée et de la résistance de base du canal microfluidique. - Appuyez sur le bouton vert Exécuter dans le menu du ruban MATLAB pour lancer le script d’acquisition de données NPS.m et commencer à échantillonner et à enregistrer les données.
- Pour terminer l’expérience, appuyez sur le bouton Arrêter dans le coin inférieur gauche de la fenêtre de la figure pour arrêter le script d’acquisition de données. Désactivez la sortie du bloc d’alimentation en appuyant sur le bouton On . Réglez la source de pression sur zéro pression dans le logiciel du régulateur de pression.
- À ce stade, l’expérience peut être interrompue pour effectuer une ou plusieurs des opérations suivantes :
- Remplacez l’appareil actuel par un nouveau.
- Rechargez le tube avec plus d’échantillons de cellules.
REMARQUE : Pour éviter la contamination croisée des échantillons, utilisez de nouveaux appareils pour mesurer des cellules de différents types ou conditions. - Détachez le dispositif du circuit imprimé et examinez l’état du canal au microscope. Pour recommencer l’expérience en utilisant le même appareil, il faut veiller à ne pas introduire de bulles d’air. Il peut être nécessaire d’appliquer une légère pression sur le piston de la seringue pour maintenir l’échantillon de cellule à la toute fin du tube tout en l’insérant dans l’entrée du dispositif.
- Réglez la pression d’entraînement constante souhaitée sur le logiciel du régulateur de pression et laissez l’échantillon remplir l’appareil.
- Chargez l’échantillon de cellule dans le tube et connectez-le à l’entrée de l’appareil.
4. Calibrer le dispositif microfluidique
- Option 1 : Mesurer les billes de polystyrène dans les dispositifs de référence.
- Choisissez une taille de bille de polystyrène plus petite que le canal de dimensionnement.
- Ajouter 1,5 % de billes d’interpolation et de polystyrène à la solution filtrée de PBS et de FBS utilisée pendant les expériences cellulaires, à une concentration de 1-3 x 105 billes / mL.
- Poursuivre l’expérience comme indiqué à la section 3, en utilisant le dispositif de référence décrit à l’étape 1.3, et appliquer la même tension que celle utilisée lors de l’expérimentation. Utiliser l’amplitude moyenne de la goutte de courant produite lorsque les billes transitent par les pores de calibrage et le diamètre connu des billes pour calculer De, tel que décrit à la section 5.
- Option 2 : Mesurer indépendamment la taille de la cellule à l’aide d’un appareil de mesure distinct.
- Au lieu de suivre le protocole de l’étape 4.1, utilisez un instrument de mesure de la taille des cellules disponible dans le commerce pour mesurer la taille moyenne des cellules de l’échantillon. Dans ce cas, aucun dispositif de référence n’est nécessaire. Utiliser la chute de courant moyenne produite lors du transit des cellules dans le pore de dimensionnement et le diamètre moyen mesuré de la cellule pour calculer De comme décrit à la section 5.
5. Analyser les données pour extraire les phénotypes cellulaires
REMARQUE : Le traitement des données peut être effectué à l’aide du fichier programme de l’interface de ligne de commande MATLAB mNPS_procJOVE.m à https://github.com/sohnlab/NPS-analysis-JOVE. Voir le dossier supplémentaire 6 pour plus d’instructions.
- Prétraiter les données (Figure 3A).
- Calculez le courant électrique mesuré en appliquant la valeur de gain utilisée dans le préamplificateur courant-tension aux données brutes acquises par le DAQ.
- Éliminez le bruit haute fréquence en appliquant une fonction de lissage rectangulaire et/ou un filtre passe-bas à la mesure du courant brut. Ensuite, rééchantillonnez les données filtrées à une fréquence d’échantillonnage inférieure. Calculez également les données d’horodatage correspondantes à cette fréquence d’échantillonnage inférieure.
- Calculer un signal de courant de base ajusté en appliquant une méthode telle que le lissage asymétrique des moindres carrés26.
- Calculez la première dérivée approximative (signal de différence) des données actuelles prétraitées en prenant la différence entre les points de données suivants.
- Identifiez les événements cellulaires et extrayez les données des sous-impulsions (Figure 3B).
- Recherchez les événements de cellules candidates en examinant les données prétraitées. Rejeter les événements cellulaires qui se chevauchent avec d’autres événements cellulaires (c.-à-d. événements de coïncidence) (figure supplémentaire 4), présentent un mauvais ajustement de base ou ont une forme d’impulsion inattendue ou erronée (p. ex. lorsqu’un bouchon peut avoir été présent dans le canal).
- Extraire les données de sous-impulsion pour chaque événement cellulaire.
- Chaque segment de pores de nœud apparaîtra comme une sous-impulsion correspondante dans l’impulsion globale du signal (figures 1B, C). Identifiez le début de chaque sous-impulsion en calculant le point temporel où le signal de différence atteint une valeur minimale locale. Identifiez la fin de chaque sous-impulsion en calculant le point temporel où le signal de différence atteint une valeur maximale locale.
- Déterminez la largeur de chaque sous-impulsion en tant que temps écoulé entre les points de début et de fin. Déterminer l’amplitude de chaque sous-impulsion en calculant la moyenne de la différence entre le courant mesuré et le courant de base pour tous les points de données entre les points de début et de fin.
- Déterminer le mécanophénotype cellulaire pour chaque événement cellulaire en fonction des données des impulsions.
- Déterminer le diamètre de la cellule d en fonction de l’équation définie par Deblois et Bean24 :
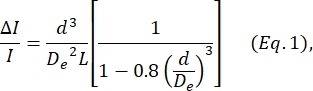
où ΔI/I est le rapport moyen entre l’amplitude de la sous-impulsion et le courant de base dans les sous-impulsions dimensionnelles, De est le diamètre effectif du canal (mesuré à l’étape 4) et L est la longueur totale du canal nœud-pores.- D e est déterminé en calculant la moyenne ΔI/I produite par un ensemble de particules d’un diamètre connu (cellules ou billes, voir étape 4), en utilisant ce diamètre connu en tant que d, et en résolvant Eq. 1 pour De.
- Quantifier la résistance de la cellule à la déformation.
- Déterminer l’écoulement U de la vitesse du fluide en calculant la vitesse cellulaire moyenne dans les sous-impulsions de dimensionnement, en utilisant les longueurs de segment connues et la durée mesurée de chaque sous-impulsion.
- Déterminer l’indice de déformabilité des cellules entières (iDCw), défini par Kim et al.2 comme suit :
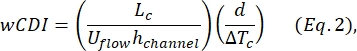
où L c est la longueur du segment de contraction, le canal h est la hauteur du canal et ΔT c est la durée de la sous-impulsion de contraction.
- Identifier le temps de récupération de la cellule à partir de la déformation, défini comme la première sous-impulsion de récupération avec une amplitude inférieure à 8% de l’amplitude moyenne de la sous-impulsion de dimensionnement2.
- Calculer la déformation transversale de la cellule dans le segment de contraction.
- Calculer le diamètre effectif du segment de contraction (De,c) tel que défini par Kim et al.2 :
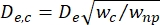 , où w c est la largeur du segment de contraction et wnp est la largeur de tous les autres segments.
, où w c est la largeur du segment de contraction et wnp est la largeur de tous les autres segments. - Calculer le diamètre sphérique équivalent dc de la cellule dans la contraction en utilisant à nouveau l’équation définie par Deblois et Bean24:
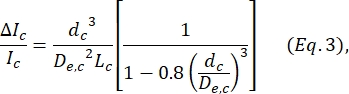
où ΔI c/I c est le rapport entre l’amplitude de la sous-impulsion et le courant de base dans la sous-impulsion de contraction et Lc est la longueur du segment de contraction. - Calculer la longueur d’allongement de la cellule Lse déforme comme décrit par Kim et al.2:
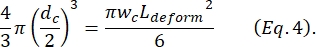
- Enfin, calculez la déformation transversale δla déformation de la cellule, qui est définie par Kim et al.2 comme étant
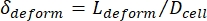 .
.
- Calculer le diamètre effectif du segment de contraction (De,c) tel que défini par Kim et al.2 :
- Déterminer le diamètre de la cellule d en fonction de l’équation définie par Deblois et Bean24 :
Résultats
La plateforme de mécano-phénotypage présentée ici est une approche simple et polyvalente pour mesurer les propriétés biophysiques de cellules individuelles à débit modéré. Les cellules circulent dans le canal microfluidique (Figure 1A) en utilisant un flux entraîné par pression constante. Au fur et à mesure que les cellules transitent, la longueur du canal microfluidique et les impulsions de courant produites sont enregistrées à l’aide du matériel d’acquisition de donnée...
Discussion
La mesure des propriétés mécaniques de cellules individuelles à l’aide de cette technique de mécanophénotypage comprend trois étapes: la fabrication du dispositif, l’acquisition de données et l’analyse des données. À chaque étape, il y a des aspects notables qui peuvent avoir un impact significatif sur les résultats expérimentaux. Lors de la fabrication des appareils, des géométries de canaux cohérentes et une uniformité d’un appareil à l’autre sont essentielles pour obtenir des résultats pr?...
Déclarations de divulgation
L. L. S détient le brevet américain n ° 11,383,241: « Mechano-node-pore sensing », J. Kim, S. Han et L. L. Sohn, délivré le 12 juillet 2022.
Remerciements
Cette recherche a été financée par des subventions du NIBIB 1R01EB024989-01 et du NCI 1R01CA190843-01. A. L. et R. R. ont bénéficié d’une bourse de recherche d’études supérieures de l’Association H2H8. K. L. C. a été soutenu par une bourse de recherche d’études supérieures de la National Science Foundation et une bourse Siebel Scholar.
matériels
| Name | Company | Catalog Number | Comments |
| Acetone | J.T. Baker | 5356-05 | Purity (GC) ≥ 99.5% (https://us.vwr.com/store/product/6057739/acetone-99-5-vlsi-j-t-baker) |
| Aluminum Foil | n/a | n/a | |
| Analog Low-Pass Filter | ThorLabs | EF504 | ≤240 kHz Passband, Coaxial BNC Feedthrough (https://www.thorlabs.com/thorproduct.cfm?partnumber=EF504#ad-image-0) |
| Biopsy Punch | Integra Miltex | 33-31AA-P/25 | 1mm, Disposable, with Plunger (https://mms.mckesson.com/product/573313/Miltex-33-31AA-P25) |
| Blade | n/a | n/a | |
| BNC Cable | Pomona Electronics | 2249-C-12 | https://www.digikey.com/en/products/detail/pomona-electronics/2249-C-12/603323?utm_adgroup=Coaxial%20Cables%20%28RF%29&utm_source=google&utm_ medium=cpc&utm_campaign= Shopping_Product_Cable%20Assemblies_NEW&utm_term= &utm_content=Coaxial%20Cables%20%28RF%29&gclid=Cj0KCQjwlK-WBhDjARIsAO2sErQqnVJ pj5OXVObuTI8ZUf1ZeIn7zvzGnx mCWdePrG6SdEJMF3X6ubUaAs w-EALw_wcB |
| Cleanroom Polyester Swab | Thermo Fisher Scientific | 18383 | https://www.fishersci.com/shop/products/texwipe-cleantip-alpha-polyester-series-swabs-6/18383 |
| Current Preamplifier | DL Instruments | 1211 | https://www.brltest.com/index.php?main_page=product_info&products_ id=1419 |
| Custom PCB (w/ components) | n/a | n/a | see Supplemental files 4 and 5 |
| DAQ Terminal Block | National Instruments | BNC-2120 | https://www.ni.com/en-in/support/model.bnc-2120.html |
| DAQ to BNC-2110 cable | National Instruments | SHC68-68-EPM | https://www.ni.com/en-in/support/model.shc68-68-epm.html |
| Data Acquisition Board (DAQ) | National Instruments | PCI-6251 | https://www.ni.com/docs/en-US/bundle/pci-6251-feature/page/overview.html |
| Dessicator | Thermo Fisher Scientific | 5311-0250 | https://www.thermofisher.com/order/catalog/product/5311-0250 |
| Female BNC To Banana Plug Adapter | Pomona Electronics | 72909 | https://www.digikey.com/en/products/detail/pomona-electronics/72909/1196318 |
| Fetal Bovine Serum (FBS) | VWR | 89510-186 | https://us.vwr.com/store/product/18706419/avantor-seradigm-select-grade-usda-approved-origin-fetal-bovine-serum-fbs |
| Glass Cutter | Chemglass | CG-1179-21 | https://chemglass.com/plate-glass-cutters-diamond-tips |
| Gold Etchant TFA | Transene | NC0977944 | https://www.fishersci.com/shop/products/NC0977944/NC0977944 |
| Hot Plate | Thermo Fisher Scientific | SP131825 | |
| Isopropyl Alcohol | Spectrum Chemical | I1056-4LTPL | Purity (GC) ≥99.5% (https://www.spectrumchemical.com/isopropyl-alcohol-99-percent-fcc-i1056) |
| Metal Hardware Enclosure | Hammond Manufacturing | EJ12126 | https://www.digikey.com/en/products/detail/hammond-manufacturing/EJ12126/2423415 |
| Methanol | Sigma-Aldrich | 34860 | Purity (GC) ≥99.8% (https://www.sigmaaldrich.com/IN/en/substance/methanol320467561) |
| MF-321 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/mf-321/ |
| MICROPOSIT S1813 Positive Photoresist | DuPont | n/a | https://kayakuam.com/products/microposit-s1800-g2-series-photoresists/ |
| Phosphate Buffered Saline (PBS) | Thermo Fisher Scientific | 10010049 | https://www.thermofisher.com/order/catalog/product/10010049?SID=srch-hj-10010049 |
| Photomask | Fineline Imaging | n/a | Photomask are custom ordered from our CAD designs (https://www.fineline-imaging.com/) |
| Plain Glass Microscope Slide | Fisher Scientific | 12-553-5B | Material: Soda Lime, L75 x W50 mm, Thickness: 0.90–1.10 mm |
| Plasma Cleaner | Harrick Plasma | PDC-001 | https://harrickplasma.com/plasma-cleaners/expanded-plasma-cleaner/ |
| Plastic Petri Dish | Thermo Fisher Scientific | FB0875712 | 100 mm (https://www.fishersci.com/shop/products/fisherbrand-petri-dishes-clear-lid-raised-ridge-100-x-15mm/FB0875712) |
| Pressure Controller | Fluigent | MFCS-EZ | https://www.fluigent.com/research/instruments/pressure-flow-controllers/mfcs-series/ |
| Pressure Controller Software | Fluigent | MAESFLO | |
| Programming & Computation Software | MATLAB | R2021b | for data acquisition and analysis (https://www.mathworks.com/products/matlab.html) |
| PTFE Tubing | Cole Parmer | 06417-31 | 0.032" ID x 0.056" (https://www.coleparmer.com/i/masterflex-transfer-tubing-microbore-ptfe-0-032-id-x-0-056-od-100-ft-roll/0641731) |
| Scepter 2.0 Handheld Automatic Cell Counter | Millapore Sigma | PHCC20060 | https://www.sigmaaldrich.com/IN/en/product/mm/phcc20060 |
| Silicon Wafer | Wafer World | 2885 | 76.2 mm, Single Side Polished (https://www.waferworld.com/product/2885) |
| Spin Coater | n/a | n/a | |
| SU-8 3025 Negative Photoresist | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-2000/ |
| SU8 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-developer/ |
| Sygard 184 Polydimethlysiloxane | Dow Chemical | 4019862 | https://www.ellsworth.com/products/by-market/consumer-products/encapsulants/silicone/dow-sylgard-184-silicone-encapsulant-clear-0.5-kg-kit/ |
| Tape | Scotch | 810-341296 | https://www.staples.com/Scotch-Magic-Tape-810-3-4-x-36-yds-1-Core/product_130567?cid=PS:GS:SBD:PLA:OS&gclid= Cj0KCQjwlK-WBhDjARIsAO 2sErRwzrrgjU0NjFkDkne1xm vT7ekS3tdzvAgiMDwPoxocgH VTQZi7vJgaAvQZEALw_wcB |
| Titanium, Platinum, Gold | n/a | n/a | |
| Triple Output Power Supply | Keysight | E36311A | https://www.newark.com/keysight-technologies/e36311a/dc-power-supply-3o-p-6v-5a-prog/dp/15AC9653 |
| UV Mask Aligner | Karl Suss America | MJB3 Mask Aligner |
Références
- Pegoraro, A. F., Janmey, P., Weitz, D. A. Mechanical properties of the cytoskeleton and cells. Cold Spring Harbor Perspectives in Biology. 9 (11), 022038 (2017).
- Kim, J., et al. Characterizing cellular mechanical phenotypes with mechano-node-pore sensing. Microsystems & Nanoengineering. 4, 17091 (2018).
- Mierke, C. T. Bidirectional mechanical response between cells and their microenvironment. Frontiers in Physics. 9, 619 (2021).
- Kumar, S., Weaver, V. M. Mechanics, malignancy, and metastasis: The force journey of a tumor cell. Cancer and Metastasis Reviews. 28 (1), 113-127 (2009).
- Nia, H. T., Munn, L. L., Jain, R. K. Physical traits of cancer. Science. 370 (6516), (2020).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463 (7280), 485-492 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: The role of physical interactions and mechanical forces in metastasis. Nature Reviews Cancer. 11 (7), 512-522 (2011).
- Li, B., et al. Mechanical phenotyping reveals unique biomechanical responses in retinoic acid-resistant acute promyelocytic leukemia. iScience. 25 (2), 103772 (2022).
- Kozminsky, M., Sohn, L. L. The promise of single-cell mechanophenotyping for clinical applications. Biomicrofluidics. 14 (3), 031301 (2020).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Wottawah, F., et al. Optical rheology of biological cells. Physical Review Letters. 94 (9), 1-4 (2005).
- Darling, E. M., Di Carlo, D. High-throughput assessment of cellular mechanical properties. Annual Review of Biomedical Engineering. 17 (1), 35-62 (2015).
- Carey, T. R., Cotner, K. L., Li, B., Sohn, L. L. Developments in label-free microfluidic methods for single-cell analysis and sorting. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 11 (1), 1529 (2019).
- Bagnall, J. S., et al. Deformability of tumor cells versus blood cells. Scientific Reports. 5, 18542 (2015).
- Byun, S., et al. Characterizing deformability and surface friction of cancer cells. Proceedings of the National Academy of Sciences. 110 (19), 7580-7585 (2013).
- Otto, O., et al. Real-time deformability cytometry: On-the-fly cell mechanical phenotyping. Nature Methods. 12 (3), 199-202 (2015).
- Gossett, D. R., et al. Hydrodynamic stretching of single cells for large population mechanical phenotyping. Proceedings of the National Academy of Sciences. 109 (20), 7630-7635 (2012).
- Guck, J., Chilvers, E. R. Mechanics meets medicine. Science Translational Medicine. 5 (212), 3-6 (2013).
- Balakrishnan, K. R., et al. Node-pore sensing: A robust, high-dynamic range method for detecting biological species. Lab on a Chip. 13 (7), 1302-1307 (2013).
- Carbonaro, A., Sohn, L. L. A resistive-pulse sensor chip for multianalyte immunoassays. Lab on a Chip. 5 (10), 1155-1160 (2005).
- Saleh, O. A., Sohn, L. L. Direct detection of antibody-antigen binding using an on-chip artificial pore. Proceedings of the National Academy of Sciences. 100 (3), 820-824 (2003).
- Saleh, O. A., Sohn, L. L. An artificial nanopore for molecular sensing. Nano Letters. 3 (1), 37-38 (2003).
- Saleh, O. A., Sohn, L. L. Quantitative sensing of nanoscale colloids using a microchip Coulter counter. Review of Scientific Instruments. 72 (12), 4449-4451 (2001).
- DeBlois, R. W., Bean, C. P. Counting and sizing of submicron particles by the resistive pulse technique. Review of Scientific Instruments. 41 (7), 909-916 (1970).
- Li, B., et al. Evaluating sources of technical variability in the mechano-node-pore sensing pipeline and their effect on the reproducibility of single-cell mechanical phenotyping. PLoS ONE. 16 (10), 0258982 (2021).
- Zhang, Z. M., Chen, S., Liang, Y. Z. Baseline correction using adaptive iteratively reweighted penalized least squares. Analyst. 135 (5), 1138-1146 (2010).
- Alibert, C., Goud, B., Manneville, J. B. Are cancer cells really softer than normal cells. Biology of the Cell. 109 (5), 167-189 (2017).
- Fujiwara, I., Zweifel, M. E., Courtemanche, N., Pollard, T. D. Latrunculin A accelerates actin filament depolymerization in addition to sequestering actin monomers. Current Biology. 28 (19), 3183-3192 (2018).
- Saleh, O. A. . A novel resistive pulse sensor for biological measurements. , (2003).
- Dokukin, M. E., Guz, N. V., Sokolov, I. Quantitative study of the elastic modulus of loosely attached cells in AFM indentation experiments. Biophysical Journal. 104 (10), 2123-2131 (2013).
- Li, Q., Lim, C. T., Goh, J. C. H., et al. Probing the elasticity of breast cancer cells using AFM. 13th International Conference on Biomedical Engineering. IFMBE Proceedings. 23, 2122-2125 (2009).
- Rother, J., et al. Atomic force microscopy-based microrheology reveals significant differences in the viscoelastic response between malign and benign cell lines. Open Biology. 4 (5), 140046 (2014).
- Li, Q., et al. AFM indentation study of breast cancer cells. Biochemical and Biophysical Research Communications. 374 (4), 609-613 (2008).
- Xu, C., et al. Elasticity measurement of breast cancer cells by atomic force microscopy. Proc. SPIE 9230. Twelfth International Conference on Photonics and Imaging in Biology and Medicine. (PIBM 2014). 92300, (2014).
- Alcaraz, J., et al. Microrheology of human lung epithelial cells measured by atomic force microscopy. Biophysical Journal. 84 (3), 2071-2079 (2003).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Urbanska, M., et al. A comparison of microfluidic methods for high-throughput cell deformability measurements. Nature Methods. 17, 587-593 (2020).
- Hill, R. T., Chilkoti, A. Surface Patterning. Biomaterials Science: An Introduction to Materials: Third Edition. , 276-301 (2013).
- Wang, Z., Volinsky, A. A., Gallant, N. D. Crosslinking effect on polydimethylsiloxane elastic modulus measured by custom-built compression instrument. Journal of Applied Polymer Science. 131 (22), 41050 (2014).
- Gibson, L. J. The hierarchical structure and mechanics of plant materials. Journal of the Royal Society Interface. 9 (76), 2749-2766 (2012).
- Stephens, A. D., Banigan, E. J., Adam, S. A., Goldman, R. D., Marko, J. F. Chromatin and lamin a determine two different mechanical response regimes of the cell nucleus. Molecular Biology of the Cell. 28 (14), 1984-1996 (2017).
- Rosenbluth, M. J., Lam, W. A., Fletcher, D. A. Force microscopy of nonadherent cells: A comparison of leukemia cell deformability. Biophysical Journal. 90 (8), 2994-3003 (2006).
- Evers, T. M. J., Holt, L. J., Alberti, S., Mashaghi, A. Reciprocal regulation of cellular mechanics and metabolism. Nature Metabolism. 3 (4), 456-468 (2021).
- Balakrishnan, K. R., et al. Node-pore sensing enables label-free surface-marker profiling of single cells. Analytical Chemistry. 87 (5), 2988-2995 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon