JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Mechano-Node-Pore 감지: 다중 파라미터 단일 셀 점탄성 측정을 위한 신속한 무표지 플랫폼
* 이 저자들은 동등하게 기여했습니다
요약
여기에 제시된 것은 mechano-node-pore sensing (mechano-NPS)라고하는 전자 기반 미세 유체 플랫폼을 사용하여 단일 세포를 기계적으로 표현형하는 방법입니다. 이 플랫폼은 1-10 cells/s의 적당한 처리량을 유지하면서 세포의 탄성 및 점성 생물물리학적 특성을 모두 측정합니다.
초록
세포의 기계적 특성은 줄기 세포 분화에서 암 전이에 이르기까지 다양한 생물학적 과정과 질병에 관여합니다. 원자력 현미경 (AFM) 및 마이크로 피펫 흡인 (MA)과 같은 이러한 특성을 측정하는 기존의 방법은 세포의 전체 점탄성 반응을 반영하여 풍부한 정보를 캡처합니다. 그러나 이러한 방법은 처리량이 매우 낮기 때문에 제한됩니다. 실시간 변형성 세포분석(RT-DC)과 같은 고처리량 접근 방식은 종종 세포의 탄성 특성만 반영하는 단일 파라미터 판독값으로 제한되기 때문에 제한된 기계적 정보만 측정할 수 있습니다. 이러한 방법과 달리 메카노-노드-기공 감지(mechano-NPS)는 중간 처리량으로 세포의 다중 파라미터 점탄성 측정을 달성하는 데 있어 격차를 해소하는 유연하고 비표지 미세유체 플랫폼입니다. 직류(DC) 측정은 세포가 미세유체 채널을 통과할 때 세포를 모니터링하여 좁은 수축을 통과하기 전, 도중 및 후에 세포의 크기와 속도를 추적하는 데 사용됩니다. 이 정보(즉, 크기 및 속도)는 각 셀의 횡방향 변형, 변형에 대한 저항 및 변형으로부터의 회복을 정량화하는 데 사용됩니다. 일반적으로이 전자 기반 미세 유체 플랫폼은 여러 점탄성 세포 특성을 제공하므로 세포의 기계적 상태를보다 완벽하게 파악할 수 있습니다. 최소한의 샘플 준비가 필요하고 간단한 전자 측정(고속 카메라와 달리)을 활용하며 표준 소프트 리소그래피 제조를 활용하기 때문에 이 플랫폼의 구현은 간단하고 액세스 가능하며 다운스트림 분석에 적용할 수 있습니다. 이 플랫폼의 유연성, 유용성 및 감도는 다양한 범위의 세포에 대한 고유한 기계적 정보를 제공했으며 기초 과학 및 임상 진단 분야에서 더 많은 응용 분야를 지원할 수 있습니다.
서문
단일 세포는 역동적이고 점탄성 재료입니다1. 다수의 내부 및 외부 과정 (예 : 유사 분열의 발병 또는 세포 외 기질 [ECM]의 리모델링)은 구조와 구성 2,3,4에 영향을 미치며 종종 현재 상태를 보완하는 뚜렷한 생물 물리학 적 특성을 초래합니다. 특히, 기계적 특성은 세포 발달, 생리학 및 병리학의 중요한 바이오마커로 나타났으며, 표준 분자 및 유전적 접근 5,6,7을 보완할 수 있는 귀중한 정량적 정보를 산출합니다. 예를 들어, Li 등은 최근 약물 내성 및 약물 반응성 급성 전골수성 백혈병 세포 사이의 기계적 차이를 설명하면서 RNA-seq를 사용하여 차등적으로 발현된 세포골격 관련 유전자8를 밝혀냈습니다. 단일 세포 역학과 세포 기능 간의 복잡한 상호 작용을 이해함으로써 기계 분석은 기초 과학 및 임상 진단을 변화시키는 데 더 광범위하게 적용됩니다9.
단일 세포 역학을 측정하기 위해 가장 널리 채택되는 도구는 원자력 현미경(AFM)입니다. AFM은 세포 기계적 특성의 고분해능, 국부적 인 측정을 가능하게하지만 <0.01 cells/s10의 처리량으로 제한됩니다. 대안적으로, 부유된 단일 세포(11)를 포획하고 변형시키기 위해 2개의 발산 레이저 빔을 사용하는 광학 들것은 <1 cell/s(12)의 약간 더 높은 처리량으로 제한된다. 최근 미세유체 기술의 발전으로 신속한 단일 세포 기계적 평가를 위한 차세대 장치가 가능해졌습니다12,13. 이들 기술은 좁은 수축 채널(14,15), 전단 흐름(16) 또는 유체역학적 스트레칭(17)을 사용하여 10-1,000 cells/s(18)의 처리량으로 세포를 신속하게 변형시킨다. 이러한 접근 방식의 측정 속도는 기존 기술보다 훨씬 빠르지만 제한된 기계적 판독을 위해 높은 처리량 기능을 교환하는 경우가 많습니다(보충 표 1). 앞서 언급한 모든 신속한 미세유체 방법은 세포의 탄성 특성만 반영하는 이동 시간 또는 변형성 비율과 같은 기본 단일 매개변수 메트릭에 중점을 둡니다. 그러나 단일 세포의 고유 점탄성 특성을 고려할 때 세포의 견고하고 철저한 기계적 특성화에는 탄성 성분뿐만 아니라 점성 반응도 고려해야 합니다.
Mechano-node-pore sensing (mechano-NPS) 2,8 (그림 1A)은 단일 세포 Mechanophenotyping의 기존 한계를 해결하는 미세 유체 플랫폼입니다. 이 방법을 사용하면 세포 직경, 상대 변형성 및 변형 후 회복 시간을 포함한 여러 생물물리학적 매개변수를 동시에 측정할 수 있으며 1-10 cells/s의 적당한 처리량으로 측정할 수 있습니다. 이 기술은 노드 기공 감지 (NPS) 19,20,21,22,23,24를 기반으로하며, 여기에는 4 점 프로브 측정을 사용하여 "노드"라고하는 더 넓은 영역으로 분할 된 미세 유체 채널을 통과하는 세포에 의해 생성 된 변조 전류 펄스를 측정하는 것이 포함됩니다. 변조된 전류 펄스는 셀이 세그먼트(즉, "기공") 및 노드에서 전류의 흐름을 부분적으로 차단한 결과이며, 후자보다 전자에서 더 많은 전류가 차단됩니다. mechano-NPS에서 하나의 세그먼트 인 "수축 채널"은 셀 직경보다 좁습니다. 결과적으로 셀은 전체 채널을 통과하기 위해 변형되어야 합니다(그림 1B). 셀 직경은 셀이 수축 채널 이전에 노드 기공을 통과할 때 생성되는 서브펄스의 크기에 의해 결정될 수 있습니다(그림 1B,C). 여기서, |ΔIenp|, 즉 세포가 기공 내에 있을 때의 전류 강하는 기공에 대한 세포의 부피비, V cell/V기공 2,8,19에 비례한다. 세포 강성은 세포가 수축 채널을 통과할 때 생성되는 극적으로 더 큰 서브펄스의 지속시간인 ΔTc에 의해 결정될 수 있다(도 1B,C). 더 단단한 셀은 부드러운 셀보다 채널을 통과하는 데 더 오래 걸립니다 2,8. 마지막으로, 변형 후 원래 크기와 모양으로 돌아가는 세포의 능력인 셀 "회복"은 세포가 수축 채널 후 노드 기공을 통과할 때 생성되는 일련의 서브펄스에 의해 결정될 수 있습니다(그림 1B,C). 회복 시간 ΔTr은 셀이 압착되기 전에 현재 서브 펄스가 이전 서브 펄스의 크기로 돌아가는 데 걸리는 시간입니다. 전반적으로, 세포가 미세유체 채널을 통과할 때 생성된 변조 전류 펄스를 기록하고 분석하여 관련 단일 세포 기계적 파라미터를 추출합니다(그림 1D)2,8.
이 전자 기반 미세 유체 플랫폼의 재현성 및 사용 용이성은 이전에 입증되었습니다25. 또한 이 플랫폼은 단일 셀 기계형 분석을 위한 진입 장벽이 낮습니다. 표준 소프트 리소그래피는 미세 유체 장치를 제조하는 데 사용됩니다. 측정 하드웨어는 간단한 인쇄 회로 기판(PCB), 전원 공급 장치, 프리앰프, 데이터 수집 보드(DAQ) 및 컴퓨터를 포함한 저렴한 구성 요소로 구성됩니다. 마지막으로, 사용자 친화적인 코드를 데이터 수집 및 분석에 사용할 수 있으므로 간단하게 구현할 수 있습니다. 이 기계 형 분석 기술은 비 악성 및 악성 유방 및 폐 상피 세포주의 집단을 구별하고, 일차 인간 유방 상피 세포의 하위 계통을 구별하고, 세포 골격 섭동 및 기타 약리학 적 제제의 효과를 특성화 할 수 있습니다 2,8. 전반적으로이 플랫폼은 단일 세포의 기계 성형을위한 효과적인 접근 방식입니다.
프로토콜
1. 장치 형상 설계
- 측정할 가장 큰 셀의 직경보다 넓지만 충분한 신호 대 잡음비(SNR)를 유지하도록 크기 조정 및 복구 세그먼트의 너비를 선택합니다. 다양한 세포주에 대한 다양한 크기 조정 및 복구 세그먼트 너비의 예는 보충 표 2 를 참조하십시오.
- 수축 세그먼트 너비를 선택하여 기계 성형 수술을 받을 세포의 평균 크기에 30%-40% 변형을 적용합니다. 변형률은 로
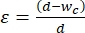 정의되며, 여기서 d는 세포 직경이고 wc는 수축 채널 폭 2,8이다. 다양한 세포주에 대한 상이한 수축 세그먼트 폭에 대해서는 보충 표 2를 참조한다.
정의되며, 여기서 d는 세포 직경이고 wc는 수축 채널 폭 2,8이다. 다양한 세포주에 대한 상이한 수축 세그먼트 폭에 대해서는 보충 표 2를 참조한다.
참고: 직경이 실질적으로 다른 셀 유형 또는 조건을 비교하려면 각 셀 유형/조건에 특정한 수축 세그먼트 너비와 함께 별도의 장치 설계를 사용해야 합니다. - 각각의 고유한 장치 형상에 대한 참조 장치를 설계합니다. 이는 마이크로유체 채널의 사이징 공극 세그먼트의 유효 직경인 Die를 결정하는데 필요하다.
참고: 참조 장치는 기본 장치와 동일한 형상을 사용합니다. 유일한 수정은 수축 세그먼트가 알려진 크기의 폴리스티렌 비드로 교정할 수 있도록 사이징 공극 세그먼트와 너비가 같아야 한다는 것입니다. 수축을 넓히면 폴리스티렌 비드가 교정 중에 수축 채널을 막는 것을 방지할 수 있습니다. 교정 프로세스는 4.1단계 및 5.3.1단계에서 자세히 설명합니다. 교정은 또한 상업적으로 이용 가능한 셀 카운터를 사용하여 달성 될 수 있으며,이 경우 기준 장치가 필요하지 않습니다. 이 프로세스는 4.2단계에서 설명합니다. - 관심있는 가장 큰 세포가 수축 세그먼트2 내에서 제한없이 완전히 신장될 수 있도록 채널 높이를 선택하십시오. 채널 높이가 hmin
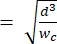 보다 큰지 확인합니다(셀이 구형 사전 변형이고 변형 중에 채널 길이와 높이를 따라 등척성 변형이 발생한다고 가정).
보다 큰지 확인합니다(셀이 구형 사전 변형이고 변형 중에 채널 길이와 높이를 따라 등척성 변형이 발생한다고 가정).
참고 : 전류 서브 펄스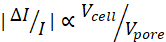 의 크기를 감안할 때 hmin 이 클수록 전체 SNR이 낮아집니다.
의 크기를 감안할 때 hmin 이 클수록 전체 SNR이 낮아집니다. - 선택한 채널 너비로 컴퓨터 지원 설계 소프트웨어를 사용하여 포토마스크를 설계하고 생성합니다. 예제 파일은 보충 파일 1에 제공됩니다. 네거티브 마스터에서 박리한 후 폴리디메틸실록산(PDMS) 수축을 설명하기 위해 마이크로유체 마스크 디자인을 1.5% 스케일링합니다.
참고: 전체 어레이가 웨이퍼의 크기를 초과하지 않는 한 단일 마스크에 장치 어레이를 포함할 수 있습니다(보충 그림 1A). - 미세유체 소자 전류의 4점 프로브 측정을 수행하는 데 사용할 전극이 있는 포토마스크를 설계하고 만듭니다(그림 1D). 예제 파일은 보충 파일 1에 제공됩니다.
알림: 어레이가 유리 슬라이드의 크기를 초과하지 않는 한 단일 마스크에 전극 어레이를 포함할 수 있습니다(보충 그림 1B).
2. 장치 제작(그림 2)
- 유리 기판에 전극 패턴을 준비하십시오.
- 스핀 코팅, 패턴 및 제품 데이터 시트에 따라 일반 유리 슬라이드에 포지티브 포토레지스트를 처리합니다. 이 절차의 예는 보충 파일 2에 요약되어 있습니다.
- 금속 증착, 리프트 오프 및 금 에칭을 수행합니다.
- 슬라이드에 75 Å Ti, 250 Å Pt 및 250 Å Au의 박막 증착을 수행합니다. 전자총 증발을 사용하는 이 절차의 예는 보충 파일 3에 요약되어 있습니다.
- 슬라이드를 아세톤에 15분 동안 담그면 과도한 금속이 들어 올려집니다.
- 흄 후드에서 일회용 피펫을 사용하여 보충 그림 2와 같이 미세 유체 채널에 노출될 전극 영역에 금 에천트를 드롭캐스트합니다. 슬라이드의 다른 곳에 에천트를 떨어뜨리지 않도록 주의하십시오.
주의: 금 에천트는 피부와 눈에 자극을 줄 수 있습니다. 증기를들이 마시지 말고 섭취하지 마십시오. 주의해서 취급하고 적절한 개인 보호 장비(PPE)를 착용하고 현지 폐기 규정에 따라 폐기물을 폐기하십시오. - 슬라이드를 탈이온수(DI)로 헹구고 건조 질소(N2)로 건조시킵니다.
- 동일한 유리 슬라이드에 여러 전극이 인쇄된 경우 슬라이드를 개별 칩에 깍둑썰기합니다.
- 유리 절삭 공구를 사용하여 패턴화된 전극 경계를 따라 슬라이드에 점수를 매깁니다.
- 악보를 따라 유리를 깨서 슬라이드를 개별 칩으로 나눕니다.
- 현미경으로 전극을 육안으로 검사하십시오. 개별 전극이 전기적으로 열려 있지 않거나 전극이 함께 단락되지 않았는지 확인하십시오.
- 채널에 대한 네거티브 마스터 몰드를 제작합니다.
- 제품 데이터 시트에 따라 SU-8 에폭시 레지스트를 연마된 실리콘 웨이퍼에 스핀 코팅, 패턴 및 처리합니다. 이 절차의 예는 보충 파일 2에 요약되어 있습니다.
- 프로파일로미터를 사용하여 피처 높이를 측정하고 현미경으로 피처를 육안으로 검사합니다(보충 그림 3). 원하는 지오메트리가 잘 정의되어 있는지 확인합니다.
- 부드러운 리소그래피로 PDMS 채널을 성형합니다.
- 일회용 컵에서 엘라스토머와 가교결합제를 10:1 질량비로 칭량하여 PDMS를 준비합니다.
참고: 직경이 3인 웨이퍼의 경우 30g의 PDMS로 충분합니다. - PDMS가 거품으로 불투명해질 때까지 일회용 포크로 PDMS를 30초 동안 격렬하게 혼합합니다.
- 진공 챔버에서 약 30-90분 동안 또는 PDMS가 눈에 보이는 기포 없이 투명해질 때까지 PDMS를 탈기합니다.
- SU-8 마스터 몰드가 있는 웨이퍼를 일회용 페트리 접시에 넣고 웨이퍼 중앙에 PDMS를 붓습니다.
- PDMS와 웨이퍼가 들어 있는 페트리 접시를 진공 챔버에 넣고 약 30분 동안 또는 PDMS에 기포가 남지 않을 때까지 가스를 제거합니다.
- PDMS를 80°C에서 오븐 또는 핫플레이트에서 2시간 동안 굽습니다.
- 날카로운 날로 SU-8 네거티브 마스터에서 PDMS를 자르고 제거하십시오.
- 날카로운 칼날을 사용하여 성형된 PDMS 슬래브를 개별 주형으로 깍둑썰기합니다.
- 일회용 생검 펀치를 사용하여 입구 및 출구 접근 구멍을 코어에 넣습니다. 최상의 결과를 얻으려면 각 PDMS 슬래브에 새 펀치를 사용하십시오. 더 날카로운 펀치는 가장자리가 매끄러운 구멍을 생성하여 수축 채널을 방해할 수 있는 미립자를 최소화합니다.
알림: 액세스 구멍의 직경은 튜브의 외부 직경보다 약간 작아야 합니다. 예를 들어, 외경이 1/32인치인 폴리테트라플루오로에틸렌(PTFE) 튜브를 사용하는 경우 1.5mm 구멍을 펀칭해야 합니다.
- 일회용 컵에서 엘라스토머와 가교결합제를 10:1 질량비로 칭량하여 PDMS를 준비합니다.
- 유리/전극 기판을 PDMS 채널에 접합합니다.
- 전극 유리 슬라이드를 메탄올(≥99.8%)로 청소합니다. 건조한 N2로 건조하십시오.
- 스카치 테이프로 PDMS 장치를 청소한 다음 이소프로필 알코올(IPA)과 탈이온수(DI; 18MΩ/cm2)로 헹굽니다. 건조한 N2로 건조하십시오. 그런 다음 스카치 테이프로 한 번 더 청소하십시오.
- 조립식 전극이 있는 유리 기판과 준비된 PDMS 몰드(피처 면이 위로 향함)를 플라즈마 클리너에 넣습니다.
- 둘 다 산소 플라즈마에 2분 동안 노출시킵니다(100-300mTorr, 30W).
- PDMS 몰드를 형상 면이 아래를 향하도록 정렬하고 조립식 전극이 있는 유리 기판에 놓습니다.
참고: 접합은 플라즈마 처리된 PDMS와 유리가 접촉하면 즉각적입니다. 따라서 추가 정렬 수정이 불가능합니다. 정렬을 용이하게 하기 위해, DI 물에서 메탄올의 2:1 희석액 20μL를 플라즈마 처리된 유리 표면 상에 피펫팅할 수 있다. 메탄올 용액은 처리된 유리와 PDMS 사이의 물리적 장벽 역할을 하여 정렬 조정을 허용합니다. 메탄올을 사용하는 경우 정렬되고 결합된 장치를 50°C에서 2시간 동안 베이킹하여 용액을 증발시키고 접합 공정을 완료합니다. - 현미경으로 접합 된 장치를 육안으로 검사하십시오. 전극과 채널 형상이 올바르게 정렬되었는지 확인하십시오.
3. 세포 측정(그림 1D)
- 압력 소스, PCB, 벤치탑 하드웨어 및 데이터 수집 소프트웨어를 준비합니다.
- 클램프를 사용하여 미세 유체 장치를 PCB에 연결합니다. PCB의 예는 보충 파일 4(GERBER 파일) 및 보충 파일 5(회로도, 보드 및 PCB 부품 목록 파일)에 제공됩니다.
- 클램프의 스프링 장착 핀을 미세 유체 장치의 전극 접촉 패드에 맞추고 클램프의 헤더 핀을 PCB의 구멍에 맞춥니다.
- 클램프의 헤더 핀을 PCB 구멍에 단단히 삽입하여 스프링 장착 핀이 전극 접촉 패드와 정렬되어 있는지 확인합니다.
- 전자 하드웨어를 설정하고 연결합니다.
- 이중 바나나 플러그-베이요넷 Neill-Concelman(BNC) 암 어댑터와 BNC 케이블을 사용하여 전원 공급 장치의 출력 포트 2개를 PCB의 공급 전압 포트에 연결합니다.
- 전원 공급 장치를 켭니다. BNC의 내부 컨덕터에 연결된 출력을 +15V로 설정하고 다른 출력을 -15V로 설정합니다. 두 출력을 모두 활성화하여 회로에 전원을 공급합니다.
- 전원 공급 장치의 출력 포트 중 세 번째를 BNC 케이블을 사용하여 PCB의 입력 전압 포트에 연결합니다. 출력을 원하는 인가 전압으로 설정하되 실험을 시작할 때까지 활성화하지 마십시오.
- PCB의 출력 전류 포트를 연결합니다.ampBNC 케이블을 사용하여 전류 전치 증폭기의 입력에.
- BNC 케이블을 사용하여 전류 전치 증폭기의 출력을 데이터 수집 시스템의 BNC 터미널 블록에 있는 하나의 아날로그 입력에 연결합니다. 선택적으로 아날로그 저역 통과 필터를 BNC 케이블과 나란히 연결하여 고주파 간섭을 필터링합니다.
참고: SNR을 개선하기 위해 PCB 및 장치를 두꺼운 금속 인클로저 내에 보관할 수 있습니다. 모든 BNC 케이블과 유체 튜브는 인클로저에 뚫린 구멍을 통해 배선할 수 있습니다.
- 개인용 컴퓨터(PC)에 필요한 소프트웨어 설치 및 설정
- 전원을 켜고 압력 컨트롤러를 PC에 연결합니다. 제조업체의 지침에 따라 필요한 압력 컨트롤러 소프트웨어를 설치합니다.
- PC에 MATLAB과 데이터 수집 툴박스를 설치합니다. MATLAB 데이터 수집 툴박스 인터페이스가 이를 감지할 수 있도록 데이터 수집 시스템에 필요한 드라이버가 설치되어 있는지 확인하십시오.
- https://github.com/sohnlab/node-pore-sensing-public 에서 포함된 데이터 수집 스크립트 "NPS.m"을 다운로드합니다.
- 데이터 수집 스크립트를 열고 구성합니다.
- 벤더 ID, DAQ의 디바이스 ID, 아날로그 입력 채널 번호 (포함된 스크립트의 34-36행 포함)를 포함하는 데이터 수집 세션을 초기화하려면 올바른 값을 설정하십시오.
참고: 장치 ID는 "daq.getDevices" 또는 "daqlist" 함수를 사용하여 찾을 수 있습니다. - 수집에 대해 원하는 샘플 속도를 설정합니다(포함된 스크립트의 23행). 최적의 결과를 얻으려면 최소 10kHz로 설정해야 합니다.
- 벤더 ID, DAQ의 디바이스 ID, 아날로그 입력 채널 번호 (포함된 스크립트의 34-36행 포함)를 포함하는 데이터 수집 세션을 초기화하려면 올바른 값을 설정하십시오.
- 클램프를 사용하여 미세 유체 장치를 PCB에 연결합니다. PCB의 예는 보충 파일 4(GERBER 파일) 및 보충 파일 5(회로도, 보드 및 PCB 부품 목록 파일)에 제공됩니다.
- 세포 현탁액을 준비한다.
- 1x 인산염 완충 식염수(PBS) 중 2% 소 태아 혈청(FBS) 용액을 준비하고, 0.22μm 필터로 여과한다.
- 배양하고 선택한 세포주의 적절한 세포 배양 프로토콜에 따라 세포를 준비한다. 세포를 1-5 x 105 cells/mL의 농도로 1x PBS 중 2% FBS의 준비된 용액에 현탁시킨다. 실험 기간 동안 세포를 얼음 위에 두십시오.
- 세포의 물리적 특성을 측정합니다.
- 셀 샘플을 튜브에 넣고 장치 입구에 연결합니다.
- 면도날이나 날카로운 칼로 PTFE 튜브 30cm를 자릅니다.
- 튜브의 한쪽 끝을 루어 잠금 주사기에 부착합니다. 주사기를 사용하여 세포 샘플을 튜브의 다른 쪽 끝으로 끌어 올립니다.
- 튜브를 장치의 입구에 조심스럽게 삽입하십시오.
- 튜브의 반대쪽 끝을 미세 유체 압력 컨트롤러에 연결합니다.
알림: 미세 유체 압력 컨트롤러와 튜브 사이에 필터를 추가하여 액체가 압력 컨트롤러로 역류하는 것을 방지할 수 있습니다.
- 실험을 실행합니다.
- 압력 컨트롤러 소프트웨어에서 원하는 일정한 구동 압력을 설정하고 샘플이 장치를 채우도록 합니다.
알림: 압력은 일반적으로 2-21kPa입니다. 유속은 명확하게 정의된 펄스를 허용할 만큼 충분히 느리지만 적절한 처리량을 허용할 만큼 충분히 빨라야 합니다.- 미세 유체 채널에 기포가 형성되면 막 다른 골목 충전물을 사용하십시오 : 장치 콘센트를 막고 입구에 저압을 적용하여 가스 투과성 PDMS를 통해 공기를 강제로 배출하십시오. 채널에 기포를 남기면 전류 기준선이 불안정해지고 정확한 측정이 방해됩니다.
- 이물질이 미세유체 채널을 막히게 하는 경우 구동 압력을 가하는 동안 PDMS 장치 상단을 가볍게 누르거나, 압력을 켜고 끄면서 더 높은 압력을 "펄싱"하거나, 튜브를 제거했다가 다시 삽입하여 제거합니다. 이물질이 남아 있으면 새 장치로 전환해야 할 수 있습니다.
- 전원 공급 장치의 전압 노브를 돌려 원하는 전압을 설정하고 ON( ON ) 버튼을 눌러 전압을 활성화합니다.
알림: 전압은 일반적으로 1-5V입니다. 적절한 SNR에 필요한 최저 전압을 선택합니다. 비교할 모든 조건에서 동일한 전압을 사용해야 합니다. - 전류 전치를 켜고 감도(A/V)를 가능한 한 낮게 설정하십시오. 또는 프리앰프에 과부하가 걸리거나 DAQ의 최대 아날로그 입력 전압을 초과하지 않고 게인(V/A)을 가능한 한 높게 설정하십시오. 이 연구에서는 감도를 10-7 A / V로 설정했습니다.
알림: 적절한 감도/이득 값은 적용된 전압과 미세 유체 채널의 기준 저항에 따라 달라집니다. - MATLAB 리본 메뉴에서 녹색의 Run 버튼을 눌러 데이터 수집 스크립트 NPS.m 을 시작하고 데이터 샘플링 및 저장을 시작합니다.
- 실험을 종료하려면 Figure 창의 왼쪽 하단 모서리에 있는 중지 버튼을 눌러 데이터 수집 스크립트를 중지 합니다. On 버튼을 눌러 전원 공급 장치 출력을 비활성화합니다. 압력 컨트롤러 소프트웨어에서 압력 소스를 0 압력으로 설정합니다.
- 이 시점에서 실험을 일시 중지하여 다음 중 하나 이상을 수행할 수 있습니다.
- 현재 장치를 새 장치로 교체하십시오.
- 더 많은 셀 샘플로 튜브를 다시 로드합니다.
알림: 샘플 교차 오염을 방지하려면 새 장치를 사용하여 다양한 유형 또는 조건의 세포를 측정하십시오. - PCB에서 장치의 클램프를 풀고 현미경으로 채널의 상태를 검사합니다. 동일한 장치를 사용하여 실험을 다시 시작하려면 기포가 발생하지 않도록주의해야합니다. 장치 입구에 삽입하는 동안 세포 샘플을 튜브의 맨 끝에 유지하기 위해 주사기 플런저에 부드러운 압력을 가해야 할 수도 있습니다.
- 압력 컨트롤러 소프트웨어에서 원하는 일정한 구동 압력을 설정하고 샘플이 장치를 채우도록 합니다.
- 셀 샘플을 튜브에 넣고 장치 입구에 연결합니다.
4. 미세유체 장치 보정
- 옵션 1: 기준 장치에서 폴리스티렌 비드를 측정합니다.
- 사이징 채널보다 작은 폴리스티렌 비드 크기를 선택하십시오.
- 셀 실험 중에 사용된 여과된 PBS 및 FBS 용액에 1.5% 트윈 및 폴리스티렌 비드를 1-3 x 105 beads/mL의 농도로 추가합니다.
- 3단계에 설명된 참조 장치를 사용하여 섹션 1.3에 설명된 대로 실험을 진행하고 실험 중에 사용된 것과 동일한 전압을 적용합니다. 섹션 5에 설명된 바와 같이, 비드가 사이징 공극을 통과할 때 생성된 전류 방울의 평균 크기 및 비드의 알려진 직경을 사용하여 De를 계산한다.
- 옵션 2: 별도의 측정 장치로 셀 크기를 독립적으로 측정합니다.
- 단계 4.1의 프로토콜을 따르는 대신, 시판되는 세포 크기 측정 기기를 사용하여 샘플 내의 세포의 평균 크기를 측정한다. 이 경우 참조 장치가 필요하지 않습니다. 세포가 사이징 공극을 통과할 때 생성된 평균 전류 강하 및 측정된 평균 세포 직경을 사용하여 섹션 5에 설명된 바와 같이 De를 계산한다.
5. 데이터를 분석하여 세포 표현형 추출
참고: 데이터 처리는 MATLAB 명령줄 인터페이스 프로그램 파일 mNPS_procJOVE.m(https://github.com/sohnlab/NPS-analysis-JOVE )을 사용하여 수행할 수 있습니다. 자세한 내용은 보충 파일 6 을 참조하십시오.
- 데이터를 전처리합니다(그림 3A).
- 전류-전압 프리앰프에 사용된 이득 값을 DAQ에서 수집한 원시 데이터에 적용하여 측정된 전류를 계산합니다.
- 원시 전류 측정에 직사각형 평활화 기능 및/또는 저역 통과 필터를 적용하여 고주파 노이즈를 제거합니다. 그런 다음 필터링된 데이터를 더 낮은 샘플 속도로 리샘플링합니다. 또한 이 낮은 샘플 속도로 해당 타임스탬프 데이터를 계산합니다.
- 비대칭 최소제곱 평활화(26)와 같은 방법을 적용하여 피팅된 기준선 전류 신호를 계산합니다.
- 후속 데이터 포인트 간의 차이를 취하여 전처리된 전류 데이터의 대략적인 1차 도함수(차분 신호)를 계산합니다.
- 세포 이벤트를 식별하고 서브펄스 데이터를 추출합니다(그림 3B).
- 전처리된 데이터를 검사하여 후보 세포 이벤트를 검색합니다. 다른 세포 이벤트와 겹치는 세포 이벤트(즉, 우연의 일치 이벤트)(보충 그림 4)를 거부하거나, 기준선 적합성이 좋지 않거나, 예상치 못한 또는 잘못된 펄스 모양(예: 채널에 막힘이 존재했을 수 있는 경우)을 나타냅니다.
- 각 셀 이벤트에 대한 서브펄스 데이터를 추출합니다.
- 각 노드-기공 세그먼트는 전체 신호 펄스 내에서 해당 서브펄스로 나타납니다(그림 1B, C). 차이 신호가 로컬 최소값에 도달하는 시점을 계산하여 각 서브펄스의 시작을 식별합니다. 차분 신호가 로컬 최대값에 도달하는 시점을 계산하여 각 서브펄스의 끝을 식별합니다.
- 각 서브펄스의 폭을 시작 시점과 종료 시점 사이의 경과 시간으로 결정합니다. 시작 시점과 종료 시점 사이의 모든 데이터 포인트에 대해 측정된 전류와 기준 전류 간의 차이 평균을 계산하여 각 서브펄스의 진폭을 결정합니다.
- 서브펄스 데이터를 기반으로 각 세포 이벤트에 대한 세포 기계형을 결정합니다.
- Deblois and Bean24에 의해 정의된 방정식에 기초하여 세포 직경 d를 결정한다:
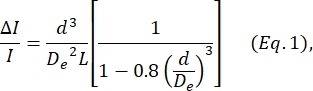
여기서 ΔI/I는 사이징 서브펄스에서 기준선 전류에 대한 서브펄스 진폭의 평균 비율이고, De는 채널의 유효 직경(4단계에서 측정)이고, L은 노드-공극 채널의 전체 길이이다.- De는 알려진 직경(세포 또는 비드, 단계 4 참조)의 입자 세트에 의해 생성된 평균 ΔI/I를 계산하고, 알려진 직경을 d로 사용하고, De에 대해 식 1을 해결함으로써 결정된다.
- 변형에 대한 세포의 저항을 정량화합니다.
- 알려진 세그먼트 길이와 각 서브펄스의 측정된 지속 시간을 사용하여 사이징 서브펄스의 평균 셀 속도를 계산하여 유체 속도 U흐름을 결정합니다.
- Kim et al.2에 의해 정의된 전체 세포 변형성 지수(wCDI)를 다음과 같이 결정합니다.
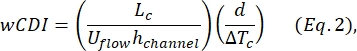
여기서, Lc는 수축 세그먼트의 길이이고, h 채널은 채널 높이이고, ΔTC는 수축 서브펄스의 지속시간이다.
- 사이징 서브펄스2에서 평균 진폭의 8% 이내의 진폭을 갖는 첫 번째 복구 서브펄스로 정의되는 변형으로부터 셀의 회복 시간을 식별합니다.
- 수축 세그먼트 내에서 셀의 횡방향 변형을 계산합니다.
- Kim et al.2에
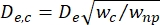 의해 정의된 바와 같이 수축 세그먼트(De,c)의 유효 직경을 계산하고, 여기서 wc는 수축 세그먼트의 너비이고 wnp는 다른 모든 세그먼트의 너비이다.
의해 정의된 바와 같이 수축 세그먼트(De,c)의 유효 직경을 계산하고, 여기서 wc는 수축 세그먼트의 너비이고 wnp는 다른 모든 세그먼트의 너비이다. - Deblois and Bean24에 의해 정의된 방정식을 사용하여 수축 내 세포의 등가 구형 직경 dc를 다시 계산합니다.
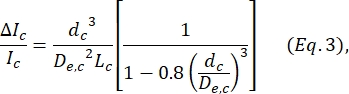
여기서 ΔIC/Ic는 수축 서브펄스에서 기준선 전류에 대한 서브펄스 진폭의 비율이고 Lc는 수축 세그먼트의 길이이다. - Kim et al.2에 설명된 대로 셀의 신장 길이 L변형을 계산합니다.
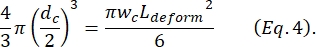
- 마지막으로, 세포의 횡방향 변형 δ변형을 계산하며, 이는 Kim et al.2에 의해 다음과 같이
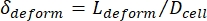 정의됩니다.
정의됩니다.
- Kim et al.2에
- Deblois and Bean24에 의해 정의된 방정식에 기초하여 세포 직경 d를 결정한다:
결과
여기에 제시된 기계 성형 플랫폼은 중간 처리량으로 단일 세포의 생물 물리학 적 특성을 측정하기위한 간단하고 다양한 접근 방식입니다. 세포는 일정한 압력 구동 유동을 사용하여 마이크로유체 채널 (도 1A)을 통해 유동된다. 세포가 통과함에 따라, 생성된 마이크로유체 채널의 길이 및 전류 펄스는 데이터 획득 하드웨어를 사용하여 기록된다. 수집된 신호(...
토론
이 기계 성형 기술을 사용하여 단일 셀의 기계적 특성을 측정하는 것은 장치 제조, 데이터 수집 및 데이터 분석의 세 단계로 구성됩니다. 각 단계에는 실험 결과에 큰 영향을 미칠 수있는 주목할만한 측면이 있습니다. 장치 제작 중에 정확하고 반복 가능한 결과를 얻으려면 일관된 채널 형상과 장치 간 균일성이 필수적입니다. 특히 각 장치의 측벽은 비교적 매끄러워야 하며(그림 4A...
공개
L. L. S는 2022년 7월 12일 발행된 미국 특허 제11,383,241호 "Mechano-node-pore sensing", J. Kim, S. Han 및 L. L. Sohn을 보유하고 있습니다.
감사의 말
이 연구는 NIBIB 1R01EB024989-01 및 NCI 1R01CA190843-01의 보조금으로 지원되었습니다. AL과 R. R.은 H2H8 협회 대학원 연구 펠로우십의 지원을 받았습니다. KLC는 National Science Foundation Graduate Research Fellowship과 Siebel Scholar Fellowship의 지원을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| Acetone | J.T. Baker | 5356-05 | Purity (GC) ≥ 99.5% (https://us.vwr.com/store/product/6057739/acetone-99-5-vlsi-j-t-baker) |
| Aluminum Foil | n/a | n/a | |
| Analog Low-Pass Filter | ThorLabs | EF504 | ≤240 kHz Passband, Coaxial BNC Feedthrough (https://www.thorlabs.com/thorproduct.cfm?partnumber=EF504#ad-image-0) |
| Biopsy Punch | Integra Miltex | 33-31AA-P/25 | 1mm, Disposable, with Plunger (https://mms.mckesson.com/product/573313/Miltex-33-31AA-P25) |
| Blade | n/a | n/a | |
| BNC Cable | Pomona Electronics | 2249-C-12 | https://www.digikey.com/en/products/detail/pomona-electronics/2249-C-12/603323?utm_adgroup=Coaxial%20Cables%20%28RF%29&utm_source=google&utm_ medium=cpc&utm_campaign= Shopping_Product_Cable%20Assemblies_NEW&utm_term= &utm_content=Coaxial%20Cables%20%28RF%29&gclid=Cj0KCQjwlK-WBhDjARIsAO2sErQqnVJ pj5OXVObuTI8ZUf1ZeIn7zvzGnx mCWdePrG6SdEJMF3X6ubUaAs w-EALw_wcB |
| Cleanroom Polyester Swab | Thermo Fisher Scientific | 18383 | https://www.fishersci.com/shop/products/texwipe-cleantip-alpha-polyester-series-swabs-6/18383 |
| Current Preamplifier | DL Instruments | 1211 | https://www.brltest.com/index.php?main_page=product_info&products_ id=1419 |
| Custom PCB (w/ components) | n/a | n/a | see Supplemental files 4 and 5 |
| DAQ Terminal Block | National Instruments | BNC-2120 | https://www.ni.com/en-in/support/model.bnc-2120.html |
| DAQ to BNC-2110 cable | National Instruments | SHC68-68-EPM | https://www.ni.com/en-in/support/model.shc68-68-epm.html |
| Data Acquisition Board (DAQ) | National Instruments | PCI-6251 | https://www.ni.com/docs/en-US/bundle/pci-6251-feature/page/overview.html |
| Dessicator | Thermo Fisher Scientific | 5311-0250 | https://www.thermofisher.com/order/catalog/product/5311-0250 |
| Female BNC To Banana Plug Adapter | Pomona Electronics | 72909 | https://www.digikey.com/en/products/detail/pomona-electronics/72909/1196318 |
| Fetal Bovine Serum (FBS) | VWR | 89510-186 | https://us.vwr.com/store/product/18706419/avantor-seradigm-select-grade-usda-approved-origin-fetal-bovine-serum-fbs |
| Glass Cutter | Chemglass | CG-1179-21 | https://chemglass.com/plate-glass-cutters-diamond-tips |
| Gold Etchant TFA | Transene | NC0977944 | https://www.fishersci.com/shop/products/NC0977944/NC0977944 |
| Hot Plate | Thermo Fisher Scientific | SP131825 | |
| Isopropyl Alcohol | Spectrum Chemical | I1056-4LTPL | Purity (GC) ≥99.5% (https://www.spectrumchemical.com/isopropyl-alcohol-99-percent-fcc-i1056) |
| Metal Hardware Enclosure | Hammond Manufacturing | EJ12126 | https://www.digikey.com/en/products/detail/hammond-manufacturing/EJ12126/2423415 |
| Methanol | Sigma-Aldrich | 34860 | Purity (GC) ≥99.8% (https://www.sigmaaldrich.com/IN/en/substance/methanol320467561) |
| MF-321 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/mf-321/ |
| MICROPOSIT S1813 Positive Photoresist | DuPont | n/a | https://kayakuam.com/products/microposit-s1800-g2-series-photoresists/ |
| Phosphate Buffered Saline (PBS) | Thermo Fisher Scientific | 10010049 | https://www.thermofisher.com/order/catalog/product/10010049?SID=srch-hj-10010049 |
| Photomask | Fineline Imaging | n/a | Photomask are custom ordered from our CAD designs (https://www.fineline-imaging.com/) |
| Plain Glass Microscope Slide | Fisher Scientific | 12-553-5B | Material: Soda Lime, L75 x W50 mm, Thickness: 0.90–1.10 mm |
| Plasma Cleaner | Harrick Plasma | PDC-001 | https://harrickplasma.com/plasma-cleaners/expanded-plasma-cleaner/ |
| Plastic Petri Dish | Thermo Fisher Scientific | FB0875712 | 100 mm (https://www.fishersci.com/shop/products/fisherbrand-petri-dishes-clear-lid-raised-ridge-100-x-15mm/FB0875712) |
| Pressure Controller | Fluigent | MFCS-EZ | https://www.fluigent.com/research/instruments/pressure-flow-controllers/mfcs-series/ |
| Pressure Controller Software | Fluigent | MAESFLO | |
| Programming & Computation Software | MATLAB | R2021b | for data acquisition and analysis (https://www.mathworks.com/products/matlab.html) |
| PTFE Tubing | Cole Parmer | 06417-31 | 0.032" ID x 0.056" (https://www.coleparmer.com/i/masterflex-transfer-tubing-microbore-ptfe-0-032-id-x-0-056-od-100-ft-roll/0641731) |
| Scepter 2.0 Handheld Automatic Cell Counter | Millapore Sigma | PHCC20060 | https://www.sigmaaldrich.com/IN/en/product/mm/phcc20060 |
| Silicon Wafer | Wafer World | 2885 | 76.2 mm, Single Side Polished (https://www.waferworld.com/product/2885) |
| Spin Coater | n/a | n/a | |
| SU-8 3025 Negative Photoresist | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-2000/ |
| SU8 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-developer/ |
| Sygard 184 Polydimethlysiloxane | Dow Chemical | 4019862 | https://www.ellsworth.com/products/by-market/consumer-products/encapsulants/silicone/dow-sylgard-184-silicone-encapsulant-clear-0.5-kg-kit/ |
| Tape | Scotch | 810-341296 | https://www.staples.com/Scotch-Magic-Tape-810-3-4-x-36-yds-1-Core/product_130567?cid=PS:GS:SBD:PLA:OS&gclid= Cj0KCQjwlK-WBhDjARIsAO 2sErRwzrrgjU0NjFkDkne1xm vT7ekS3tdzvAgiMDwPoxocgH VTQZi7vJgaAvQZEALw_wcB |
| Titanium, Platinum, Gold | n/a | n/a | |
| Triple Output Power Supply | Keysight | E36311A | https://www.newark.com/keysight-technologies/e36311a/dc-power-supply-3o-p-6v-5a-prog/dp/15AC9653 |
| UV Mask Aligner | Karl Suss America | MJB3 Mask Aligner |
참고문헌
- Pegoraro, A. F., Janmey, P., Weitz, D. A. Mechanical properties of the cytoskeleton and cells. Cold Spring Harbor Perspectives in Biology. 9 (11), 022038 (2017).
- Kim, J., et al. Characterizing cellular mechanical phenotypes with mechano-node-pore sensing. Microsystems & Nanoengineering. 4, 17091 (2018).
- Mierke, C. T. Bidirectional mechanical response between cells and their microenvironment. Frontiers in Physics. 9, 619 (2021).
- Kumar, S., Weaver, V. M. Mechanics, malignancy, and metastasis: The force journey of a tumor cell. Cancer and Metastasis Reviews. 28 (1), 113-127 (2009).
- Nia, H. T., Munn, L. L., Jain, R. K. Physical traits of cancer. Science. 370 (6516), (2020).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463 (7280), 485-492 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: The role of physical interactions and mechanical forces in metastasis. Nature Reviews Cancer. 11 (7), 512-522 (2011).
- Li, B., et al. Mechanical phenotyping reveals unique biomechanical responses in retinoic acid-resistant acute promyelocytic leukemia. iScience. 25 (2), 103772 (2022).
- Kozminsky, M., Sohn, L. L. The promise of single-cell mechanophenotyping for clinical applications. Biomicrofluidics. 14 (3), 031301 (2020).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Wottawah, F., et al. Optical rheology of biological cells. Physical Review Letters. 94 (9), 1-4 (2005).
- Darling, E. M., Di Carlo, D. High-throughput assessment of cellular mechanical properties. Annual Review of Biomedical Engineering. 17 (1), 35-62 (2015).
- Carey, T. R., Cotner, K. L., Li, B., Sohn, L. L. Developments in label-free microfluidic methods for single-cell analysis and sorting. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 11 (1), 1529 (2019).
- Bagnall, J. S., et al. Deformability of tumor cells versus blood cells. Scientific Reports. 5, 18542 (2015).
- Byun, S., et al. Characterizing deformability and surface friction of cancer cells. Proceedings of the National Academy of Sciences. 110 (19), 7580-7585 (2013).
- Otto, O., et al. Real-time deformability cytometry: On-the-fly cell mechanical phenotyping. Nature Methods. 12 (3), 199-202 (2015).
- Gossett, D. R., et al. Hydrodynamic stretching of single cells for large population mechanical phenotyping. Proceedings of the National Academy of Sciences. 109 (20), 7630-7635 (2012).
- Guck, J., Chilvers, E. R. Mechanics meets medicine. Science Translational Medicine. 5 (212), 3-6 (2013).
- Balakrishnan, K. R., et al. Node-pore sensing: A robust, high-dynamic range method for detecting biological species. Lab on a Chip. 13 (7), 1302-1307 (2013).
- Carbonaro, A., Sohn, L. L. A resistive-pulse sensor chip for multianalyte immunoassays. Lab on a Chip. 5 (10), 1155-1160 (2005).
- Saleh, O. A., Sohn, L. L. Direct detection of antibody-antigen binding using an on-chip artificial pore. Proceedings of the National Academy of Sciences. 100 (3), 820-824 (2003).
- Saleh, O. A., Sohn, L. L. An artificial nanopore for molecular sensing. Nano Letters. 3 (1), 37-38 (2003).
- Saleh, O. A., Sohn, L. L. Quantitative sensing of nanoscale colloids using a microchip Coulter counter. Review of Scientific Instruments. 72 (12), 4449-4451 (2001).
- DeBlois, R. W., Bean, C. P. Counting and sizing of submicron particles by the resistive pulse technique. Review of Scientific Instruments. 41 (7), 909-916 (1970).
- Li, B., et al. Evaluating sources of technical variability in the mechano-node-pore sensing pipeline and their effect on the reproducibility of single-cell mechanical phenotyping. PLoS ONE. 16 (10), 0258982 (2021).
- Zhang, Z. M., Chen, S., Liang, Y. Z. Baseline correction using adaptive iteratively reweighted penalized least squares. Analyst. 135 (5), 1138-1146 (2010).
- Alibert, C., Goud, B., Manneville, J. B. Are cancer cells really softer than normal cells. Biology of the Cell. 109 (5), 167-189 (2017).
- Fujiwara, I., Zweifel, M. E., Courtemanche, N., Pollard, T. D. Latrunculin A accelerates actin filament depolymerization in addition to sequestering actin monomers. Current Biology. 28 (19), 3183-3192 (2018).
- Saleh, O. A. . A novel resistive pulse sensor for biological measurements. , (2003).
- Dokukin, M. E., Guz, N. V., Sokolov, I. Quantitative study of the elastic modulus of loosely attached cells in AFM indentation experiments. Biophysical Journal. 104 (10), 2123-2131 (2013).
- Li, Q., Lim, C. T., Goh, J. C. H., et al. Probing the elasticity of breast cancer cells using AFM. 13th International Conference on Biomedical Engineering. IFMBE Proceedings. 23, 2122-2125 (2009).
- Rother, J., et al. Atomic force microscopy-based microrheology reveals significant differences in the viscoelastic response between malign and benign cell lines. Open Biology. 4 (5), 140046 (2014).
- Li, Q., et al. AFM indentation study of breast cancer cells. Biochemical and Biophysical Research Communications. 374 (4), 609-613 (2008).
- Xu, C., et al. Elasticity measurement of breast cancer cells by atomic force microscopy. Proc. SPIE 9230. Twelfth International Conference on Photonics and Imaging in Biology and Medicine. (PIBM 2014). 92300, (2014).
- Alcaraz, J., et al. Microrheology of human lung epithelial cells measured by atomic force microscopy. Biophysical Journal. 84 (3), 2071-2079 (2003).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Urbanska, M., et al. A comparison of microfluidic methods for high-throughput cell deformability measurements. Nature Methods. 17, 587-593 (2020).
- Hill, R. T., Chilkoti, A. Surface Patterning. Biomaterials Science: An Introduction to Materials: Third Edition. , 276-301 (2013).
- Wang, Z., Volinsky, A. A., Gallant, N. D. Crosslinking effect on polydimethylsiloxane elastic modulus measured by custom-built compression instrument. Journal of Applied Polymer Science. 131 (22), 41050 (2014).
- Gibson, L. J. The hierarchical structure and mechanics of plant materials. Journal of the Royal Society Interface. 9 (76), 2749-2766 (2012).
- Stephens, A. D., Banigan, E. J., Adam, S. A., Goldman, R. D., Marko, J. F. Chromatin and lamin a determine two different mechanical response regimes of the cell nucleus. Molecular Biology of the Cell. 28 (14), 1984-1996 (2017).
- Rosenbluth, M. J., Lam, W. A., Fletcher, D. A. Force microscopy of nonadherent cells: A comparison of leukemia cell deformability. Biophysical Journal. 90 (8), 2994-3003 (2006).
- Evers, T. M. J., Holt, L. J., Alberti, S., Mashaghi, A. Reciprocal regulation of cellular mechanics and metabolism. Nature Metabolism. 3 (4), 456-468 (2021).
- Balakrishnan, K. R., et al. Node-pore sensing enables label-free surface-marker profiling of single cells. Analytical Chemistry. 87 (5), 2988-2995 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유