Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Mechano-Node-Pore Sensing: Eine schnelle, markierungsfreie Plattform für viskoelastische Multiparameter-Einzelzellmessungen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier wird eine Methode zur mechanischen Phänotypisierung einzelner Zellen unter Verwendung einer elektronikbasierten mikrofluidischen Plattform namens Mechano-Node-Pore Sensing (Mechano-NPS) vorgestellt. Diese Plattform hält einen moderaten Durchsatz von 1-10 Zellen / s aufrecht und misst gleichzeitig die elastischen und viskosen biophysikalischen Eigenschaften der Zellen.
Zusammenfassung
Zelluläre mechanische Eigenschaften sind an einer Vielzahl von biologischen Prozessen und Krankheiten beteiligt, die von der Stammzelldifferenzierung bis zur Krebsmetastasierung reichen. Herkömmliche Methoden zur Messung dieser Eigenschaften, wie Rasterkraftmikroskopie (AFM) und Mikropipettenaspiration (MA), erfassen reichhaltige Informationen, die die volle viskoelastische Reaktion einer Zelle widerspiegeln. Diese Methoden sind jedoch durch einen sehr geringen Durchsatz begrenzt. Hochdurchsatzansätze wie die Echtzeit-Deformierbarkeitszytometrie (RT-DC) können nur begrenzte mechanische Informationen messen, da sie oft auf Einzelparameter-Anzeigen beschränkt sind, die nur die elastischen Eigenschaften einer Zelle widerspiegeln. Im Gegensatz zu diesen Methoden ist die Mechano-Node-Poren-Sensorik (Mechano-NPS) eine flexible, markierungsfreie mikrofluidische Plattform, die die Lücke schließt, um multiparameter-viskoelastische Messungen einer Zelle mit moderatem Durchsatz zu erreichen. Eine Gleichstrommessung (DC) wird verwendet, um Zellen zu überwachen, während sie einen mikrofluidischen Kanal durchlaufen, und verfolgt ihre Größe und Geschwindigkeit vor, während und nachdem sie durch eine enge Verengung gezwungen werden. Diese Informationen (d. H. Größe und Geschwindigkeit) werden verwendet, um die transversale Verformung jeder Zelle, den Widerstand gegen Verformung und die Erholung von der Verformung zu quantifizieren. Im Allgemeinen bietet diese elektronikbasierte mikrofluidische Plattform mehrere viskoelastische Zelleigenschaften und damit ein vollständigeres Bild des mechanischen Zustands einer Zelle. Da sie eine minimale Probenvorbereitung erfordert, eine einfache elektronische Messung (im Gegensatz zu einer Hochgeschwindigkeitskamera) verwendet und die Vorteile der Standard-Softlithographie-Fertigung nutzt, ist die Implementierung dieser Plattform einfach, zugänglich und an die nachgelagerte Analyse anpassbar. Die Flexibilität, der Nutzen und die Empfindlichkeit dieser Plattform haben einzigartige mechanische Informationen zu einer Vielzahl von Zellen geliefert, mit dem Potenzial für viele weitere Anwendungen in der Grundlagenforschung und klinischen Diagnostik.
Einleitung
Einzelzellen sind dynamische, viskoelastische Materialien1. Eine Vielzahl von internen und externen Prozessen (z. B. Beginn der Mitose oder Umbau der extrazellulären Matrix [ECM]) beeinflussen ihre Struktur und Zusammensetzung 2,3,4, was oft zu unterschiedlichen biophysikalischen Eigenschaften führt, die ihren aktuellen Zustand ergänzen. Insbesondere mechanische Eigenschaften haben sich als wichtige Biomarker für die Zellentwicklung, Physiologie und Pathologie erwiesen und liefern wertvolle quantitative Informationen, die kanonische molekulare und genetische Ansätze ergänzen können 5,6,7. Zum Beispiel beschrieben Li et al. kürzlich die mechanischen Unterschiede zwischen arzneimittelresistenten und medikamentenresponsiven akuten promyelozytären Leukämiezellen, während sie auch RNA-seq verwendeten, um differentiell exprimierte Zytoskelett-assoziierte Gene aufzudecken8. Durch das Verständnis des komplexen Zusammenspiels zwischen Einzelzellmechanik und Zellfunktion hat die Mechanophänotypisierung breitere Anwendungen bei der Transformation der Grundlagenforschung und der klinischen Diagnostik9.
Das am weitesten verbreitete Werkzeug zur Messung der Einzelzellmechanik ist die Rasterkraftmikroskopie (AFM). Während AFM eine hochauflösende, lokalisierte Messung zellulärer mechanischer Eigenschaften ermöglicht, bleibt es auf einen Durchsatz von <0,01 Zellen/sbegrenzt 10. Alternativ sind optische Stretcher, die zwei divergente Laserstrahlen verwenden, um suspendierte Einzelzellen11 einzufangen und zu verformen, auf geringfügig höhere Durchsätze von <1 Zelle/n12 beschränkt. Jüngste Fortschritte in der mikrofluidischen Technologie haben eine neue Generation von Geräten für die schnelle, einzellige, mechanische Bewertung ermöglicht12,13. Diese Techniken verwenden enge Verengungskanäle14,15, Scherströmung16 oder hydrodynamische Dehnung 17, um Zellen bei Durchsätzen von 10-1.000 Zellen / s 18 schnell zu verformen. Während die Messrate dieser Ansätze wesentlich schneller ist als bei herkömmlichen Techniken, tauschen sie häufig Hochdurchsatzfähigkeiten gegen begrenzte mechanische Auslesungen aus (Zusatztabelle 1). Alle oben genannten schnellen mikrofluidischen Methoden konzentrieren sich auf grundlegende Einzelparametermetriken wie Transitzeit oder Verformbarkeitsverhältnisse, die nur die elastischen Eigenschaften einer Zelle widerspiegeln. Angesichts der intrinsischen viskoelastischen Natur einzelner Zellen erfordert eine robuste und gründliche mechanische Charakterisierung von Zellen jedoch nicht nur die Berücksichtigung elastischer Komponenten, sondern auch viskoser Reaktionen.
Mechano-node-pore sensing (mechano-NPS)2,8 (Abbildung 1A) ist eine mikrofluidische Plattform, die bestehende Einschränkungen bei der Einzelzell-Mechanophänotypisierung adressiert. Diese Methode ermöglicht die gleichzeitige Messung mehrerer biophysikalischer Parameter, einschließlich Zelldurchmesser, relativer Verformbarkeit und Erholungszeit von Verformung, mit einem moderaten Durchsatz von 1-10 Zellen / s. Diese Technik basiert auf Node-Pore Sensing (NPS) 19,20,21,22,23,24, bei der eine Vier-Punkt-Sondenmessung verwendet wird, um den modulierten Stromimpuls zu messen, der von einer Zelle erzeugt wird, die einen mikrofluidischen Kanal durchquert, der durch breitere Regionen, sogenannte "Knoten", segmentiert wurde. Der modulierte Stromimpuls ist das Ergebnis der teilweisen Blockierung des Stromflusses in den Segmenten (d. H. "Poren") und Knoten, wobei in ersteren mehr Strom blockiert wird als in letzteren. Beim Mechano-NPS ist ein Segment, der "Kontraktionskanal", schmaler als ein Zelldurchmesser; Folglich muss sich eine Zelle verformen, um den gesamten Kanal zu durchlaufen (Abbildung 1B). Der Zelldurchmesser kann durch die Größe des Subpulses bestimmt werden, der erzeugt wird, wenn die Zelle die Knotenporen vor dem Kontraktionskanal durchquert (Abbildungen 1B,C). Hier ist |ΔInp|, der Stromabfall, wenn sich die Zelle in der Pore befindet, proportional zum Volumenverhältnis der Zelle zur Pore, V-Zelle/V-Pore 2,8,19. Die Zellsteifigkeit kann durch ΔTc bestimmt werden, die Dauer des dramatisch größeren Subpulses, der entsteht, wenn die Zelle den Kontraktionskanal durchquert (Abbildungen 1B,C). Eine steifere Zelle braucht länger, um den Kanal zu passieren als eine weichere 2,8. Schließlich kann die Zellerholung, die Fähigkeit der Zelle, nach der Verformung zu ihrer ursprünglichen Größe und Form zurückzukehren, durch die Reihe von Subpulsen bestimmt werden, die erzeugt werden, wenn die Zelle die Knotenporen nach dem Kontraktionskanal durchquert (Abbildungen 1B,C). Die Erholungszeit, ΔTr, ist die Zeit, die benötigt wird, bis die aktuellen Subpulse wieder die Größe der vorherigen Subpulse erreichen, bevor die Zelle gequetscht wird. Insgesamt werden die modulierten Stromimpulse, die erzeugt werden, wenn eine Zelle den mikrofluidischen Kanal durchquert, aufgezeichnet und analysiert, um die relevanten mechanischen Einzelzellparameter zu extrahieren (Abbildung 1D)2,8.
Die Reproduzierbarkeit und Benutzerfreundlichkeit dieser elektronikbasierten mikrofluidischen Plattform wurde bereits nachgewiesen25. Darüber hinaus bietet die Plattform eine niedrige Eintrittsbarriere für die Einzelzell-Mechanophänotypisierung. Standard-Softlithographie wird verwendet, um mikrofluidische Geräte herzustellen. Die Messhardware besteht aus kostengünstigen Komponenten, darunter eine einfache Leiterplatte (PCB), ein Netzteil, ein Vorverstärker, eine Datenerfassungsplatine (DAQ) und ein Computer. Schließlich steht benutzerfreundlicher Code für die Datenerfassung und -analyse zur Verfügung, der eine einfache Implementierung ermöglicht. Diese Mechanophänotypisierungstechnik kann Populationen von nicht-malignen und malignen Brust- und Lungenepithelzelllinien unterscheiden, zwischen Unterlinien in primären menschlichen Brustepithelzellen unterscheiden und die Auswirkungen von Zytoskelettstörungen und anderen pharmakologischen Wirkstoffen charakterisieren 2,8. Insgesamt ist diese Plattform ein effektiver Ansatz für die Mechanophänotypisierung einzelner Zellen.
Protokoll
1. Gerätegeometrie entwerfen
- Wählen Sie die Breite der Größen- und Wiederherstellungssegmente so, dass sie breiter als der Durchmesser der größten zu messenden Zellen ist, aber auch ein ausreichendes Signal-Rausch-Verhältnis (SNR) beibehält. In der ergänzenden Tabelle 2 finden Sie Beispiele für unterschiedliche Größen- und Rückgewinnungssegmentbreiten für verschiedene Zelllinien.
- Wählen Sie die Breite des Kontraktionssegments, um eine Dehnung von 30% -40% auf die durchschnittliche Größe der Zellen anzuwenden, die einer Mechanophenotypisierung unterzogen werden sollen. Die Dehnung ist definiert als
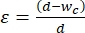 , wobei d der Zelldurchmesser und wc die Kontraktionskanalbreite 2,8 ist. Siehe Zusatztabelle 2 für unterschiedliche Kontraktionssegmentbreiten für verschiedene Zelllinien.
, wobei d der Zelldurchmesser und wc die Kontraktionskanalbreite 2,8 ist. Siehe Zusatztabelle 2 für unterschiedliche Kontraktionssegmentbreiten für verschiedene Zelllinien.
HINWEIS: Wenn man Zelltypen oder -bedingungen mit wesentlich unterschiedlichen Durchmessern vergleichen möchte, sollten separate Gerätedesigns mit Kontraktionssegmentbreiten verwendet werden, die für jeden Zelltyp / jede Zellbedingung spezifisch sind. - Entwerfen Sie ein Referenzgerät für jede eindeutige Gerätegeometrie. Dies ist notwendig, um De, den effektiven Durchmesser des Größenporensegments des mikrofluidischen Kanals, zu bestimmen.
HINWEIS: Das Referenzgerät verwendet dieselbe Geometrie wie das primäre Gerät. Die einzige Änderung besteht darin, dass das Kontraktionssegment gleich breit sein sollte wie das Porengrößensegment, um eine Kalibrierung mit Polystyrolperlen bekannter Größe zu ermöglichen. Die Verbreiterung der Kontraktion verhindert, dass die Polystyrolperlen den Kontraktionskanal während der Kalibrierung verstopfen. Der Kalibrierungsvorgang wird in den Schritten 4.1 und 5.3.1 näher beschrieben. Die Kalibrierung kann auch mit einem handelsüblichen Zellzähler erfolgen, wobei in diesem Fall kein Referenzgerät benötigt wird. Dieser Vorgang wird in Schritt 4.2 beschrieben. - Wählen Sie die Kanalhöhe so, dass sich die größten interessierenden Zellen innerhalb des Kontraktionssegments2 ohne Einschränkung vollständig verlängern können. Stellen Sie sicher, dass die Kanalhöhe größer als hmin
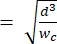 ist (dies setzt voraus, dass die Zelle eine sphärische Vorverformung aufweist und dass während der Verformung eine isometrische Verformung entlang der Kanallänge und -höhe auftritt).
ist (dies setzt voraus, dass die Zelle eine sphärische Vorverformung aufweist und dass während der Verformung eine isometrische Verformung entlang der Kanallänge und -höhe auftritt).
HINWEIS: Angesichts der Größe eines Stromsubpulses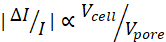 ist das Gesamt-SNR umso niedriger, je größer das hmin ist.
ist das Gesamt-SNR umso niedriger, je größer das hmin ist. - Entwerfen und erstellen Sie eine Fotomaske mit computergestützter Designsoftware mit den gewählten Kanalbreiten. Eine Beispieldatei finden Sie in Ergänzungsdatei 1. Skalieren Sie das mikrofluidische Maskendesign um 1,5 %, um die Schrumpfung von Polydimethylsiloxan (PDMS) nach dem Abziehen vom Negativmaster zu berücksichtigen.
HINWEIS: Ein Array von Geräten kann auf einer einzelnen Maske enthalten sein, solange das gesamte Array die Größe des Wafers nicht überschreitet (ergänzende Abbildung 1A). - Entwerfen und erstellen Sie eine Fotomaske mit Elektroden, die verwendet wird, um eine Vier-Punkt-Sondenmessung des mikrofluidischen Gerätestroms durchzuführen (Abbildung 1D). Eine Beispieldatei finden Sie in Ergänzungsdatei 1.
HINWEIS: Eine Anordnung von Elektroden kann auf einer einzelnen Maske angebracht werden, solange die Anordnung die Größe des Objektträgers nicht überschreitet (ergänzende Abbildung 1B).
2. Geräte herstellen (Abbildung 2)
- Bereiten Sie Elektrodenmuster auf einem Glassubstrat vor.
- Schleudern, mustern und verarbeiten Sie einen positiven Fotolack auf einem einfachen Glasobjektträger gemäß Produktdatenblatt. Ein Beispiel für dieses Verfahren ist in der Zusatzakte 2 beschrieben.
- Führen Sie Metallabscheidung, Abheben und Goldätzen durch.
- Führen Sie eine Dünnschichtabscheidung von 75 Å Ti, 250 Å Pt und 250 Å Au auf dem Objektträger durch. Ein Beispiel für dieses Verfahren mit Elektronenkanonenverdampfung ist in der Zusatzakte 3 beschrieben.
- Tauchen Sie den Objektträger 15 Minuten lang in Aceton, um überschüssiges Metall abzuheben.
- Verwenden Sie in einem Abzug eine Einwegpipette, um Goldätzmittel auf den Bereich der Elektroden zu tropfen, die dem mikrofluidischen Kanal ausgesetzt werden, wie in der ergänzenden Abbildung 2 gezeigt. Seien Sie vorsichtig, um zu vermeiden, dass Ätzmittel an anderer Stelle auf der Folie fallen.
ACHTUNG: Goldätzmittel können Haut- und Augenreizungen verursachen. Atmen Sie keine Dämpfe ein und nehmen Sie sie nicht ein. Behandeln Sie vorsichtig, tragen Sie geeignete persönliche Schutzausrüstung (PSA) und entsorgen Sie Abfälle gemäß den örtlichen Entsorgungsvorschriften. - Spülen Sie den Objektträger mit entionisiertem (DI) Wasser ab und trocknen Sie ihn mit trockenem Stickstoff (N2).
- Wenn mehrere Elektroden auf denselben Objektträger gedruckt sind, würfeln Sie den Objektträger in einzelne Chips.
- Verwenden Sie ein Glasschneidewerkzeug, um den Objektträger entlang der gemusterten Elektrodengrenzen zu ritzen.
- Zerbrechen Sie das Glas entlang der Partitur, um den Objektträger in einzelne Chips aufzuteilen.
- Untersuchen Sie die Elektroden visuell unter einem Mikroskop. Stellen Sie sicher, dass einzelne Elektroden nicht elektrisch geöffnet sind oder dass Elektroden nicht miteinander kurzgeschlossen sind.
- Stellen Sie eine Negativ-Masterform für Kanäle her.
- Schichten, Mustern und Verarbeiten eines SU-8-Epoxidlacks auf einem polierten Siliziumwafer gemäß Produktdatenblatt. Ein Beispiel für dieses Verfahren ist in der Zusatzakte 2 beschrieben.
- Messen Sie die Merkmalshöhen mit einem Profilometer und untersuchen Sie die Merkmale visuell unter einem Mikroskop (ergänzende Abbildung 3). Stellen Sie sicher, dass die gewünschten Geometrien genau definiert sind.
- Form-PDMS-Kanäle mit weicher Lithographie.
- Bereiten Sie PDMS vor, indem Sie ein Elastomer und einen Vernetzer im Massenverhältnis 10:1 in einem Einwegbecher wiegen.
HINWEIS: Für einen Wafer mit einem Durchmesser von 3 Zoll sind 30 g PDMS ausreichend. - Mischen Sie das PDMS kräftig für 30 s mit einer Einweggabel, bis das PDMS undurchsichtig mit Blasen ist.
- Entgasen Sie das PDMS in einer Vakuumkammer für ca. 30-90 min oder bis das PDMS transparent und ohne sichtbare Blasen ist.
- Legen Sie den Wafer mit der SU-8-Masterform in eine Einweg-Petrischale und gießen Sie PDMS über die Mitte des Wafers.
- Die Petrischale mit PDMS und Wafer in eine Vakuumkammer stellen und ca. 30 min entgasen, oder bis keine Blasen mehr im PDMS verbleiben.
- Backen Sie das PDMS bei 80 °C für 2 h im Backofen oder auf einer Kochplatte.
- Schneiden Sie das PDMS mit einer scharfen Klinge ab und entfernen Sie es vom SU-8 Negativmaster.
- Die geformte PDMS-Platte mit einer scharfen Klinge in einzelne Formen schneiden
- Entkernen Sie die Ein- und Auslassöffnungen mit einem Einweg-Biopsiestempel. Die besten Ergebnisse erzielen Sie, wenn Sie für jede PDMS-Platte einen neuen Stempel verwenden. Ein schärferer Stempel erzeugt glattkantige Löcher und minimiert Partikel, die den Kontraktionskanal behindern könnten.
HINWEIS: Der Durchmesser der Zugangslöcher sollte etwas kleiner sein als der Außendurchmesser des Rohres. Wenn beispielsweise Polytetrafluorethylen (PTFE)-Schläuche mit einem Außendurchmesser von 1/32 Zoll verwendet werden, sollte ein 1,5-mm-Loch gestanzt werden.
- Bereiten Sie PDMS vor, indem Sie ein Elastomer und einen Vernetzer im Massenverhältnis 10:1 in einem Einwegbecher wiegen.
- Verkleben Sie ein Glas-/Elektrodensubstrat mit den PDMS-Kanälen.
- Reinigen Sie die Elektrodenglasträger mit Methanol (≥99,8%). Trocken mit trockenem N2.
- Reinigen Sie das PDMS-Gerät mit Klebeband, gefolgt von einer Spülung mit Isopropylalkohol (IPA) und entionisiertem Wasser (DI; 18 MΩ/cm2). Trocken mit trockenem N2. Dann noch einmal mit Klebeband reinigen.
- Legen Sie das Glassubstrat mit vorgefertigten Elektroden und die vorbereitete PDMS-Form (Feature Side Up) in einen Plasmareiniger.
- Beide 2 min (100-300 mTorr, 30 W) Sauerstoffplasma aussetzen.
- Richten Sie die PDMS-Form aus und platzieren Sie sie mit der Feature-Seite nach unten auf dem Glassubstrat mit vorgefertigten Elektroden.
HINWEIS: Die Verklebung erfolgt sofort, sobald das plasmabehandelte PDMS und das Glas in Kontakt kommen. Weitere Trassenänderungen sind daher nicht möglich. Um die Ausrichtung zu erleichtern, können 20 μL einer 2:1-Verdünnung von Methanol in DI-Wasser auf die plasmabehandelte Glasoberfläche pipettiert werden. Die Methanollösung fungiert als physikalische Barriere zwischen dem behandelten Glas und PDMS und ermöglicht Ausrichtungsanpassungen. Wenn Sie Methanol verwenden, backen Sie die ausgerichtete und gesteckte Vorrichtung bei 50 °C für 2 h ein, um die Lösung zu verdampfen und den Klebevorgang abzuschließen. - Untersuchen Sie das verklebte Gerät visuell unter einem Mikroskop. Stellen Sie sicher, dass die Elektroden und Kanalgeometrien richtig ausgerichtet sind.
3. Zellen messen (Abbildung 1D)
- Bereiten Sie Druckquelle, Leiterplatte, Tischhardware und Datenerfassungssoftware vor.
- Verbinden Sie das mikrofluidische Gerät über die Klemme mit der Leiterplatte. Ein Beispiel für die Leiterplatte finden Sie in Supplementary File 4 (GERBER-Dateien) und Supplementary File 5 (Schaltplan-, Platinen- und PCB-Stücklistendateien).
- Richten Sie die federbelasteten Stifte der Klemme mit den Elektrodenkontaktpads auf dem mikrofluidischen Gerät aus und richten Sie die Stifte der Klemme mit den Löchern auf der Leiterplatte aus.
- Führen Sie die Stifte der Klemme fest in die Leiterplattenlöcher ein und stellen Sie sicher, dass die federbelasteten Stifte mit den Elektrodenkontaktpads ausgerichtet bleiben.
- Richten Sie die elektronische Hardware ein und schließen Sie sie an.
- Verbinden Sie zwei der Ausgangsanschlüsse des Netzteils mit einer Doppelbananenstecker-Bajonett-Neill-Concelman-Buchse (BNC) und einem BNC-Kabel mit dem Versorgungsspannungsanschluss der Leiterplatte.
- Schalten Sie das Netzteil ein. Stellen Sie den an den Innenleiter des BNC angeschlossenen Ausgang auf +15 V und den anderen Ausgang auf -15 V ein. Aktivieren Sie beide Ausgänge, um den Stromkreis mit Strom zu versorgen.
- Verbinden Sie den dritten Ausgangsanschluss des Netzteils mit einem BNC-Kabel mit dem Eingangsspannungsanschluss der Leiterplatte. Stellen Sie den Ausgang auf die gewünschte angelegte Spannung ein, aktivieren Sie ihn jedoch erst, wenn Sie das Experiment starten.
- Verbinden Sie den Ausgangsstromanschluss der Leiterplatte mit einem BNC-Kabel mit dem Eingang des Stromvorverstärkers.
- Verbinden Sie den Ausgang des Stromvorverstärkers mit einem BNC-Kabel mit einem analogen Eingang am BNC-Klemmblock des Datenerfassungssystems. Schließen Sie optional einen analogen Tiefpassfilter in Linie mit dem BNC-Kabel an, um hochfrequente Störungen herauszufiltern.
HINWEIS: Um das SNR zu verbessern, können die Leiterplatte und das Gerät in einem dicken Metallgehäuse untergebracht sein. Alle BNC-Kabel und fluidischen Schläuche können durch in das Gehäuse gebohrte Löcher geführt werden.
- Installieren und Einrichten der erforderlichen Software auf dem PC
- Schalten Sie den Druckregler ein und schließen Sie ihn an den PC an. Installieren Sie die erforderliche Druckreglersoftware gemäß den Anweisungen des Herstellers.
- Installieren Sie MATLAB und die Data Acquisition Toolbox auf dem PC. Stellen Sie sicher, dass die erforderlichen Treiber für das Datenerfassungssystem installiert sind, damit die MATLAB Data Acquisition Toolbox-Schnittstelle es erkennen kann.
- Laden Sie das mitgelieferte Datenerfassungsskript "NPS.m" von https://github.com/sohnlab/node-pore-sensing-public herunter.
- Öffnen und konfigurieren Sie das Datenerfassungsskript.
- Legen Sie die richtigen Werte fest, um die Datenerfassungssitzung zu initialisieren, einschließlich der Hersteller-ID, der Geräte-ID des DAQ und der Nummer des analogen Eingangskanals (Zeilen 34-36 im enthaltenen Skript).
HINWEIS: Die Geräte-ID kann über die Funktion "daq.getDevices" oder "daqlist" gefunden werden. - Stellen Sie die gewünschte Abtastrate für die Erfassung ein (Zeile 23 im mitgelieferten Skript). Für optimale Ergebnisse sollte es auf mindestens 10 kHz eingestellt werden.
- Legen Sie die richtigen Werte fest, um die Datenerfassungssitzung zu initialisieren, einschließlich der Hersteller-ID, der Geräte-ID des DAQ und der Nummer des analogen Eingangskanals (Zeilen 34-36 im enthaltenen Skript).
- Verbinden Sie das mikrofluidische Gerät über die Klemme mit der Leiterplatte. Ein Beispiel für die Leiterplatte finden Sie in Supplementary File 4 (GERBER-Dateien) und Supplementary File 5 (Schaltplan-, Platinen- und PCB-Stücklistendateien).
- Bereiten Sie die Zellsuspension vor.
- Eine Lösung von 2% fetalem Rinderserum (FBS) in 1x phosphatgepufferter Kochsalzlösung (PBS) herstellen und mit einem 0,22 μm Filter filtrieren.
- Kultur und Vorbereitung der Zellen nach dem entsprechenden Zellkulturprotokoll der Zelllinie der Wahl. Die Zellen werden in der vorbereiteten Lösung von 2% FBS in 1x PBS in einer Konzentration von 1-5 x 105 Zellen/ml suspendiert. Halten Sie die Zellen für die Dauer der Experimente auf Eis.
- Messen Sie die physikalischen Eigenschaften der Zellen.
- Legen Sie die Zellprobe in den Schlauch und verbinden Sie sie mit dem Geräteeinlass.
- Schneiden Sie 30 cm PTFE-Schläuche mit einer Rasierklinge oder einem scharfen Messer ab.
- Befestigen Sie ein Ende des Schlauchs an einer Luer-Lock-Spritze. Verwenden Sie die Spritze, um die Zellprobe in das andere Ende des Schlauchs zu ziehen.
- Führen Sie den Schlauch vorsichtig in den Einlass des Geräts ein.
- Verbinden Sie das gegenüberliegende Ende des Schlauchs mit dem mikrofluidischen Druckregler.
HINWEIS: Ein Filter kann zwischen dem mikrofluidischen Druckregler und dem Schlauch hinzugefügt werden, um einen Flüssigkeitsrückfluss in den Druckregler zu verhindern.
- Führen Sie das Experiment aus.
- Stellen Sie den gewünschten konstanten Steuerdruck auf der Druckreglersoftware ein und lassen Sie die Probe das Gerät füllen.
HINWEIS: Der Druck beträgt typischerweise 2-21 kPa. Die Strömungsgeschwindigkeit muss langsam genug sein, um klar definierte Impulse zu ermöglichen, aber schnell genug, um einen ausreichenden Durchsatz zu ermöglichen.- Wenn sich in den mikrofluidischen Kanälen Blasen bilden, verwenden Sie eine Sackgassenfüllung: Stecken Sie den Geräteauslass und üben Sie einen niedrigen Druck auf den Einlass aus, um Luft durch das gasdurchlässige PDMS zu drücken. Das Hinterlassen von Blasen im Kanal führt zu einer instabilen Strombasislinie und verhindert genaue Messungen.
- Wenn Schmutz den mikrofluidischen Kanal verstopft, entfernen Sie ihn, indem Sie leicht auf die Oberseite des PDMS-Geräts drücken, während Sie den Antriebsdruck ausüben, einen höheren Druck "pulsieren", indem Sie den Druck ein- und ausschalten oder den Schlauch entfernen und wieder einsetzen. Wenn die Trümmer zurückbleiben, kann es notwendig sein, auf ein neues Gerät umzusteigen.
- Stellen Sie die gewünschte Spannung ein, indem Sie den Spannungsregler am Netzteil drehen, und aktivieren Sie die Spannung durch Drücken der Ein-Taste .
HINWEIS: Die Spannung beträgt typischerweise 1-5 V. Wählen Sie die niedrigste Spannung, die für ein angemessenes SNR erforderlich ist. Für alle zu vergleichenden Bedingungen sollte die gleiche Spannung verwendet werden. - Schalten Sie den Stromvorverstärker ein und stellen Sie die Empfindlichkeit (A/V) so niedrig wie möglich ein; Alternativ können Sie die Verstärkung (V/A) so hoch wie möglich einstellen, ohne den Vorverstärker zu überlasten oder die maximale analoge Eingangsspannung des Datenerfassungsgeräts zu überschreiten. In dieser Studie wurde die Empfindlichkeit auf 10-7 A / V eingestellt.
HINWEIS: Der richtige Empfindlichkeits-/Verstärkungswert hängt sowohl von der angelegten Spannung als auch vom Basiswiderstand des mikrofluidischen Kanals ab. - Klicken Sie auf die grüne Schaltfläche Ausführen im MATLAB-Menübandmenü, um das Datenerfassungsskript NPS.m zu starten und mit der Abtastung und Speicherung der Daten zu beginnen.
- Um das Experiment zu beenden, drücken Sie die Schaltfläche Stopp in der unteren linken Ecke des Abbildungsfensters, um das Datenerfassungsskript zu stoppen. Deaktivieren Sie den Netzteilausgang, indem Sie die Ein-Taste drücken . Stellen Sie die Druckquelle in der Druckreglersoftware auf Nulldruck ein.
- An diesem Punkt kann das Experiment angehalten werden, um eine oder mehrere der folgenden Aktionen auszuführen:
- Ersetzen Sie das aktuelle Gerät durch ein neues.
- Beladen Sie den Schlauch mit weiteren Zellproben.
HINWEIS: Um eine Kreuzkontamination der Probe zu vermeiden, verwenden Sie neue Geräte, um Zellen verschiedener Typen oder Bedingungen zu messen. - Lösen Sie das Gerät von der Leiterplatte und untersuchen Sie den Zustand des Kanals unter einem Mikroskop. Um das Experiment mit demselben Gerät neu zu starten, muss darauf geachtet werden, dass keine Luftblasen entstehen. Es kann notwendig sein, sanften Druck auf den Spritzenkolben auszuüben, um die Zellprobe am Ende des Schlauchs zu halten, während sie in den Geräteeinlass eingeführt wird.
- Stellen Sie den gewünschten konstanten Steuerdruck auf der Druckreglersoftware ein und lassen Sie die Probe das Gerät füllen.
- Legen Sie die Zellprobe in den Schlauch und verbinden Sie sie mit dem Geräteeinlass.
4. Kalibrieren Sie das mikrofluidische Gerät
- Option 1: Messen Sie die Polystyrolperlen in Referenzgeräten.
- Wählen Sie eine Polystyrolperlengröße, die kleiner als der Größenkanal ist.
- Fügen Sie der gefilterten PBS- und FBS-Lösung, die während der Zellexperimente verwendet wurde, 1,5% Tween- und Styroporperlen in einer Konzentration von 1-3 x 105 Perlen / ml hinzu.
- Fahren Sie mit dem in Abschnitt 3 beschriebenen Experiment unter Verwendung des in Schritt 1.3 beschriebenen Referenzgeräts fort und legen Sie dieselbe Spannung an, die während des Experiments verwendet wurde. Verwenden Sie die durchschnittliche Größe des aktuellen Abfalls, der erzeugt wird, wenn Perlen die Größenporen passieren, und den bekannten Durchmesser der Perlen, um De zu berechnen, wie in Abschnitt 5 beschrieben.
- Option 2: Messen Sie die Zellgröße selbstständig mit einem separaten Messgerät.
- Anstatt dem Protokoll in Schritt 4.1 zu folgen, verwenden Sie ein handelsübliches Zellgrößenmessgerät, um die durchschnittliche Größe der Zellen in der Probe zu messen. In diesem Fall wird kein Referenzgerät benötigt. Verwenden Sie den durchschnittlichen Stromabfall, der erzeugt wird, wenn Zellen die Größenpore passieren, und den gemessenen durchschnittlichen Zelldurchmesser, um De wie in Abschnitt 5 beschrieben zu berechnen.
5. Analysieren Sie Daten, um Zellphänotypen zu extrahieren
HINWEIS: Die Datenverarbeitung kann mit der MATLAB-Befehlszeilenschnittstellen-Programmdatei mNPS_procJOVE.m auf https://github.com/sohnlab/NPS-analysis-JOVE durchgeführt werden. Weitere Anweisungen finden Sie in der Zusatzdatei 6 .
- Vorverarbeitung der Daten (Abbildung 3A).
- Berechnen Sie den gemessenen elektrischen Strom, indem Sie den im Strom-Spannungs-Vorverstärker verwendeten Verstärkungswert auf die vom Datenerfassungsgerät erfassten Rohdaten anwenden.
- Entfernen Sie hochfrequentes Rauschen, indem Sie eine rechteckige Glättungsfunktion und/oder einen Tiefpassfilter auf die Rohstrommessung anwenden. Berechnen Sie dann die gefilterten Daten erneut auf eine niedrigere Abtastrate. Berechnen Sie außerdem die entsprechenden Zeitstempeldaten mit dieser niedrigeren Abtastrate.
- Berechnen Sie ein angepasstes Basisstromsignal, indem Sie eine Methode wie asymmetrische Glättung der kleinsten Quadrateanwenden 26.
- Berechnen Sie die ungefähre erste Ableitung (Differenzsignal) der vorverarbeiteten aktuellen Daten, indem Sie die Differenz zwischen nachfolgenden Datenpunkten nehmen.
- Identifizieren Sie Zellereignisse und extrahieren Sie Subpulsdaten (Abbildung 3B).
- Suchen Sie nach Kandidatenzellereignissen, indem Sie die vorverarbeiteten Daten untersuchen. Zellereignisse zurückweisen, die sich mit anderen Zellereignissen überschneiden (d. h. Koinzidenzereignisse) (ergänzende Abbildung 4), eine schlechte Baseline-Anpassung aufweisen oder eine unerwartete oder fehlerhafte Pulsform aufweisen (z. B. wenn eine Verstopfung im Kanal vorhanden gewesen sein könnte).
- Extrahieren Sie Subpulsdaten für jedes Zellereignis.
- Jedes Knotenporensegment erscheint als entsprechender Subimpuls innerhalb des Gesamtsignalimpulses (Abbildungen 1B, C). Identifizieren Sie den Beginn jedes Subimpulses, indem Sie den Zeitpunkt berechnen, an dem das Differenzsignal einen lokalen Mindestwert erreicht. Identifizieren Sie das Ende jedes Subimpulses, indem Sie den Zeitpunkt berechnen, an dem das Differenzsignal einen lokalen Maximalwert erreicht.
- Bestimmen Sie die Breite jedes Subpulses als verstrichene Zeit zwischen Start- und Endzeitpunkt. Bestimmen Sie die Amplitude jedes Subimpulses, indem Sie den Mittelwert der Differenz zwischen dem gemessenen Strom und dem Basisstrom für alle Datenpunkte zwischen den Start- und Endzeitpunkten berechnen.
- Bestimmen Sie den Zellmechanophänotyp für jedes Zellereignis basierend auf Subpulsdaten.
- Bestimmen Sie den Zellendurchmesser d basierend auf der Gleichung von Deblois und Bean24:
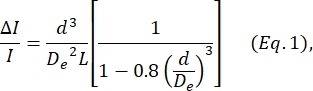
Dabei ist ΔI/I das mittlere Verhältnis von Subpulsamplitude zu Basislinienstrom in den Größensubpulsen, De der effektive Durchmesser des Kanals (gemessen in Schritt 4) und L die Gesamtlänge des Knotenporenkanals.- D e wird bestimmt, indem der Mittelwert ΔI/I berechnet wird, der von einer Gruppe von Partikeln eines bekannten Durchmessers (entweder Zellen oder Perlen, siehe Schritt 4) erzeugt wird, wobei der bekannte Durchmesser als d verwendet wird und Gleichung 1 für De gelöst wird.
- Quantifizieren Sie den Widerstand der Zelle gegen Verformung.
- Bestimmen Sie die Strömung der Fluidgeschwindigkeit U, indem Sie die mittlere Zellgeschwindigkeit in den Größensubpulsen unter Verwendung der bekannten Segmentlängen und der gemessenen Dauer jedes Subpulses berechnen.
- Bestimmen Sie den Ganzzellverformbarkeitsindex (wCDI), definiert von Kim et al.2 als:
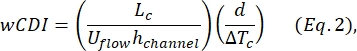
wobei L c die Länge des Kontraktionssegments, derh-Kanal die Kanalhöhe und ΔTc die Dauer des Kontraktionssubpulses ist.
- Identifizieren Sie die Erholungszeit der Zelle von der Verformung, definiert als der erste Erholungssubpuls mit einer Amplitude innerhalb von 8% der mittleren Amplitude aus dem Größensubpuls2.
- Berechnen Sie die Querverformung der Zelle innerhalb des Kontraktionssegments.
- Berechnen Sie den effektiven Durchmesser des Kontraktionssegments (De,c) gemäß der Definition von Kim et al.2:
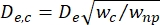 , wobei w c die Breite des Kontraktionssegments und wnp die Breite aller anderen Segmente ist.
, wobei w c die Breite des Kontraktionssegments und wnp die Breite aller anderen Segmente ist. - Berechnen Sie den äquivalenten sphärischen Durchmesser dc der Zelle innerhalb der Kontraktion, indem Sie erneut die Gleichung von Deblois und Bean24 verwenden:
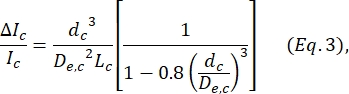
wobei ΔI c/I c das Verhältnis von Subpulsamplitude zu Basisstrom im Kontraktionssubpuls und Lc die Länge des Kontraktionssegments ist. - Berechnen Sie die Dehnungslänge derZelle , wie von Kim et al.2 beschrieben:
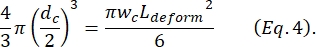
- Schließlich berechnet man die transversale Verformung der Zelle δVerformung, die von Kim et al.2 als definiert wird
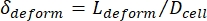 .
.
- Berechnen Sie den effektiven Durchmesser des Kontraktionssegments (De,c) gemäß der Definition von Kim et al.2:
- Bestimmen Sie den Zellendurchmesser d basierend auf der Gleichung von Deblois und Bean24:
Ergebnisse
Die hier vorgestellte Mechanophänotypisierungsplattform ist ein einfacher und vielseitiger Ansatz, um die biophysikalischen Eigenschaften einzelner Zellen mit moderatem Durchsatz zu messen. Die Zellen werden durch den mikrofluidischen Kanal (Abbildung 1A) unter Verwendung eines konstanten druckgetriebenen Flusses geleitet. Während des Passierens der Zellen werden die Länge des mikrofluidischen Kanals und die erzeugten Stromimpulse mit der Datenerfassungshardware aufgezeichnet. Das erfasst...
Diskussion
Die Messung der mechanischen Eigenschaften einzelner Zellen mit dieser Mechanophänotypisierungstechnik besteht aus drei Phasen: Geräteherstellung, Datenerfassung und Datenanalyse. Innerhalb jeder Phase gibt es bemerkenswerte Aspekte, die die experimentellen Ergebnisse erheblich beeinflussen können. Bei der Geräteherstellung sind konsistente Kanalgeometrien und die Einheitlichkeit von Gerät zu Gerät für genaue und wiederholbare Ergebnisse unerlässlich. Insbesondere sollten die Seitenwände jedes Geräts relativ gl...
Offenlegungen
L. L. S hält das US-Patent Nr. 11,383,241: "Mechano-node-pore sensing", J. Kim, S. Han und L. L. Sohn, erteilt am 12. Juli 2022.
Danksagungen
Diese Forschung wurde durch Zuschüsse von NIBIB 1R01EB024989-01 und NCI 1R01CA190843-01 unterstützt. A. L. und R. R. wurden durch ein H2H8 Association Graduate Research Fellowship unterstützt. K. L. C. wurde durch ein National Science Foundation Graduate Research Fellowship und ein Siebel Scholar Fellowship unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Acetone | J.T. Baker | 5356-05 | Purity (GC) ≥ 99.5% (https://us.vwr.com/store/product/6057739/acetone-99-5-vlsi-j-t-baker) |
| Aluminum Foil | n/a | n/a | |
| Analog Low-Pass Filter | ThorLabs | EF504 | ≤240 kHz Passband, Coaxial BNC Feedthrough (https://www.thorlabs.com/thorproduct.cfm?partnumber=EF504#ad-image-0) |
| Biopsy Punch | Integra Miltex | 33-31AA-P/25 | 1mm, Disposable, with Plunger (https://mms.mckesson.com/product/573313/Miltex-33-31AA-P25) |
| Blade | n/a | n/a | |
| BNC Cable | Pomona Electronics | 2249-C-12 | https://www.digikey.com/en/products/detail/pomona-electronics/2249-C-12/603323?utm_adgroup=Coaxial%20Cables%20%28RF%29&utm_source=google&utm_ medium=cpc&utm_campaign= Shopping_Product_Cable%20Assemblies_NEW&utm_term= &utm_content=Coaxial%20Cables%20%28RF%29&gclid=Cj0KCQjwlK-WBhDjARIsAO2sErQqnVJ pj5OXVObuTI8ZUf1ZeIn7zvzGnx mCWdePrG6SdEJMF3X6ubUaAs w-EALw_wcB |
| Cleanroom Polyester Swab | Thermo Fisher Scientific | 18383 | https://www.fishersci.com/shop/products/texwipe-cleantip-alpha-polyester-series-swabs-6/18383 |
| Current Preamplifier | DL Instruments | 1211 | https://www.brltest.com/index.php?main_page=product_info&products_ id=1419 |
| Custom PCB (w/ components) | n/a | n/a | see Supplemental files 4 and 5 |
| DAQ Terminal Block | National Instruments | BNC-2120 | https://www.ni.com/en-in/support/model.bnc-2120.html |
| DAQ to BNC-2110 cable | National Instruments | SHC68-68-EPM | https://www.ni.com/en-in/support/model.shc68-68-epm.html |
| Data Acquisition Board (DAQ) | National Instruments | PCI-6251 | https://www.ni.com/docs/en-US/bundle/pci-6251-feature/page/overview.html |
| Dessicator | Thermo Fisher Scientific | 5311-0250 | https://www.thermofisher.com/order/catalog/product/5311-0250 |
| Female BNC To Banana Plug Adapter | Pomona Electronics | 72909 | https://www.digikey.com/en/products/detail/pomona-electronics/72909/1196318 |
| Fetal Bovine Serum (FBS) | VWR | 89510-186 | https://us.vwr.com/store/product/18706419/avantor-seradigm-select-grade-usda-approved-origin-fetal-bovine-serum-fbs |
| Glass Cutter | Chemglass | CG-1179-21 | https://chemglass.com/plate-glass-cutters-diamond-tips |
| Gold Etchant TFA | Transene | NC0977944 | https://www.fishersci.com/shop/products/NC0977944/NC0977944 |
| Hot Plate | Thermo Fisher Scientific | SP131825 | |
| Isopropyl Alcohol | Spectrum Chemical | I1056-4LTPL | Purity (GC) ≥99.5% (https://www.spectrumchemical.com/isopropyl-alcohol-99-percent-fcc-i1056) |
| Metal Hardware Enclosure | Hammond Manufacturing | EJ12126 | https://www.digikey.com/en/products/detail/hammond-manufacturing/EJ12126/2423415 |
| Methanol | Sigma-Aldrich | 34860 | Purity (GC) ≥99.8% (https://www.sigmaaldrich.com/IN/en/substance/methanol320467561) |
| MF-321 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/mf-321/ |
| MICROPOSIT S1813 Positive Photoresist | DuPont | n/a | https://kayakuam.com/products/microposit-s1800-g2-series-photoresists/ |
| Phosphate Buffered Saline (PBS) | Thermo Fisher Scientific | 10010049 | https://www.thermofisher.com/order/catalog/product/10010049?SID=srch-hj-10010049 |
| Photomask | Fineline Imaging | n/a | Photomask are custom ordered from our CAD designs (https://www.fineline-imaging.com/) |
| Plain Glass Microscope Slide | Fisher Scientific | 12-553-5B | Material: Soda Lime, L75 x W50 mm, Thickness: 0.90–1.10 mm |
| Plasma Cleaner | Harrick Plasma | PDC-001 | https://harrickplasma.com/plasma-cleaners/expanded-plasma-cleaner/ |
| Plastic Petri Dish | Thermo Fisher Scientific | FB0875712 | 100 mm (https://www.fishersci.com/shop/products/fisherbrand-petri-dishes-clear-lid-raised-ridge-100-x-15mm/FB0875712) |
| Pressure Controller | Fluigent | MFCS-EZ | https://www.fluigent.com/research/instruments/pressure-flow-controllers/mfcs-series/ |
| Pressure Controller Software | Fluigent | MAESFLO | |
| Programming & Computation Software | MATLAB | R2021b | for data acquisition and analysis (https://www.mathworks.com/products/matlab.html) |
| PTFE Tubing | Cole Parmer | 06417-31 | 0.032" ID x 0.056" (https://www.coleparmer.com/i/masterflex-transfer-tubing-microbore-ptfe-0-032-id-x-0-056-od-100-ft-roll/0641731) |
| Scepter 2.0 Handheld Automatic Cell Counter | Millapore Sigma | PHCC20060 | https://www.sigmaaldrich.com/IN/en/product/mm/phcc20060 |
| Silicon Wafer | Wafer World | 2885 | 76.2 mm, Single Side Polished (https://www.waferworld.com/product/2885) |
| Spin Coater | n/a | n/a | |
| SU-8 3025 Negative Photoresist | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-2000/ |
| SU8 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-developer/ |
| Sygard 184 Polydimethlysiloxane | Dow Chemical | 4019862 | https://www.ellsworth.com/products/by-market/consumer-products/encapsulants/silicone/dow-sylgard-184-silicone-encapsulant-clear-0.5-kg-kit/ |
| Tape | Scotch | 810-341296 | https://www.staples.com/Scotch-Magic-Tape-810-3-4-x-36-yds-1-Core/product_130567?cid=PS:GS:SBD:PLA:OS&gclid= Cj0KCQjwlK-WBhDjARIsAO 2sErRwzrrgjU0NjFkDkne1xm vT7ekS3tdzvAgiMDwPoxocgH VTQZi7vJgaAvQZEALw_wcB |
| Titanium, Platinum, Gold | n/a | n/a | |
| Triple Output Power Supply | Keysight | E36311A | https://www.newark.com/keysight-technologies/e36311a/dc-power-supply-3o-p-6v-5a-prog/dp/15AC9653 |
| UV Mask Aligner | Karl Suss America | MJB3 Mask Aligner |
Referenzen
- Pegoraro, A. F., Janmey, P., Weitz, D. A. Mechanical properties of the cytoskeleton and cells. Cold Spring Harbor Perspectives in Biology. 9 (11), 022038 (2017).
- Kim, J., et al. Characterizing cellular mechanical phenotypes with mechano-node-pore sensing. Microsystems & Nanoengineering. 4, 17091 (2018).
- Mierke, C. T. Bidirectional mechanical response between cells and their microenvironment. Frontiers in Physics. 9, 619 (2021).
- Kumar, S., Weaver, V. M. Mechanics, malignancy, and metastasis: The force journey of a tumor cell. Cancer and Metastasis Reviews. 28 (1), 113-127 (2009).
- Nia, H. T., Munn, L. L., Jain, R. K. Physical traits of cancer. Science. 370 (6516), (2020).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463 (7280), 485-492 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: The role of physical interactions and mechanical forces in metastasis. Nature Reviews Cancer. 11 (7), 512-522 (2011).
- Li, B., et al. Mechanical phenotyping reveals unique biomechanical responses in retinoic acid-resistant acute promyelocytic leukemia. iScience. 25 (2), 103772 (2022).
- Kozminsky, M., Sohn, L. L. The promise of single-cell mechanophenotyping for clinical applications. Biomicrofluidics. 14 (3), 031301 (2020).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Wottawah, F., et al. Optical rheology of biological cells. Physical Review Letters. 94 (9), 1-4 (2005).
- Darling, E. M., Di Carlo, D. High-throughput assessment of cellular mechanical properties. Annual Review of Biomedical Engineering. 17 (1), 35-62 (2015).
- Carey, T. R., Cotner, K. L., Li, B., Sohn, L. L. Developments in label-free microfluidic methods for single-cell analysis and sorting. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 11 (1), 1529 (2019).
- Bagnall, J. S., et al. Deformability of tumor cells versus blood cells. Scientific Reports. 5, 18542 (2015).
- Byun, S., et al. Characterizing deformability and surface friction of cancer cells. Proceedings of the National Academy of Sciences. 110 (19), 7580-7585 (2013).
- Otto, O., et al. Real-time deformability cytometry: On-the-fly cell mechanical phenotyping. Nature Methods. 12 (3), 199-202 (2015).
- Gossett, D. R., et al. Hydrodynamic stretching of single cells for large population mechanical phenotyping. Proceedings of the National Academy of Sciences. 109 (20), 7630-7635 (2012).
- Guck, J., Chilvers, E. R. Mechanics meets medicine. Science Translational Medicine. 5 (212), 3-6 (2013).
- Balakrishnan, K. R., et al. Node-pore sensing: A robust, high-dynamic range method for detecting biological species. Lab on a Chip. 13 (7), 1302-1307 (2013).
- Carbonaro, A., Sohn, L. L. A resistive-pulse sensor chip for multianalyte immunoassays. Lab on a Chip. 5 (10), 1155-1160 (2005).
- Saleh, O. A., Sohn, L. L. Direct detection of antibody-antigen binding using an on-chip artificial pore. Proceedings of the National Academy of Sciences. 100 (3), 820-824 (2003).
- Saleh, O. A., Sohn, L. L. An artificial nanopore for molecular sensing. Nano Letters. 3 (1), 37-38 (2003).
- Saleh, O. A., Sohn, L. L. Quantitative sensing of nanoscale colloids using a microchip Coulter counter. Review of Scientific Instruments. 72 (12), 4449-4451 (2001).
- DeBlois, R. W., Bean, C. P. Counting and sizing of submicron particles by the resistive pulse technique. Review of Scientific Instruments. 41 (7), 909-916 (1970).
- Li, B., et al. Evaluating sources of technical variability in the mechano-node-pore sensing pipeline and their effect on the reproducibility of single-cell mechanical phenotyping. PLoS ONE. 16 (10), 0258982 (2021).
- Zhang, Z. M., Chen, S., Liang, Y. Z. Baseline correction using adaptive iteratively reweighted penalized least squares. Analyst. 135 (5), 1138-1146 (2010).
- Alibert, C., Goud, B., Manneville, J. B. Are cancer cells really softer than normal cells. Biology of the Cell. 109 (5), 167-189 (2017).
- Fujiwara, I., Zweifel, M. E., Courtemanche, N., Pollard, T. D. Latrunculin A accelerates actin filament depolymerization in addition to sequestering actin monomers. Current Biology. 28 (19), 3183-3192 (2018).
- Saleh, O. A. . A novel resistive pulse sensor for biological measurements. , (2003).
- Dokukin, M. E., Guz, N. V., Sokolov, I. Quantitative study of the elastic modulus of loosely attached cells in AFM indentation experiments. Biophysical Journal. 104 (10), 2123-2131 (2013).
- Li, Q., Lim, C. T., Goh, J. C. H., et al. Probing the elasticity of breast cancer cells using AFM. 13th International Conference on Biomedical Engineering. IFMBE Proceedings. 23, 2122-2125 (2009).
- Rother, J., et al. Atomic force microscopy-based microrheology reveals significant differences in the viscoelastic response between malign and benign cell lines. Open Biology. 4 (5), 140046 (2014).
- Li, Q., et al. AFM indentation study of breast cancer cells. Biochemical and Biophysical Research Communications. 374 (4), 609-613 (2008).
- Xu, C., et al. Elasticity measurement of breast cancer cells by atomic force microscopy. Proc. SPIE 9230. Twelfth International Conference on Photonics and Imaging in Biology and Medicine. (PIBM 2014). 92300, (2014).
- Alcaraz, J., et al. Microrheology of human lung epithelial cells measured by atomic force microscopy. Biophysical Journal. 84 (3), 2071-2079 (2003).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Urbanska, M., et al. A comparison of microfluidic methods for high-throughput cell deformability measurements. Nature Methods. 17, 587-593 (2020).
- Hill, R. T., Chilkoti, A. Surface Patterning. Biomaterials Science: An Introduction to Materials: Third Edition. , 276-301 (2013).
- Wang, Z., Volinsky, A. A., Gallant, N. D. Crosslinking effect on polydimethylsiloxane elastic modulus measured by custom-built compression instrument. Journal of Applied Polymer Science. 131 (22), 41050 (2014).
- Gibson, L. J. The hierarchical structure and mechanics of plant materials. Journal of the Royal Society Interface. 9 (76), 2749-2766 (2012).
- Stephens, A. D., Banigan, E. J., Adam, S. A., Goldman, R. D., Marko, J. F. Chromatin and lamin a determine two different mechanical response regimes of the cell nucleus. Molecular Biology of the Cell. 28 (14), 1984-1996 (2017).
- Rosenbluth, M. J., Lam, W. A., Fletcher, D. A. Force microscopy of nonadherent cells: A comparison of leukemia cell deformability. Biophysical Journal. 90 (8), 2994-3003 (2006).
- Evers, T. M. J., Holt, L. J., Alberti, S., Mashaghi, A. Reciprocal regulation of cellular mechanics and metabolism. Nature Metabolism. 3 (4), 456-468 (2021).
- Balakrishnan, K. R., et al. Node-pore sensing enables label-free surface-marker profiling of single cells. Analytical Chemistry. 87 (5), 2988-2995 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten