このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
メカノノードポアセンシング:マルチパラメータシングルセル粘弾性測定のための迅速でラベルフリーのプラットフォーム
* これらの著者は同等に貢献しました
要約
ここでは、メカノノード細孔センシング(mechano-NPS)と呼ばれるエレクトロニクスベースのマイクロ流体プラットフォームを使用して、単一細胞を機械的に表現型化する方法を示します。このプラットフォームは、細胞の弾性と粘性の両方の生物物理学的特性を測定しながら、1〜10細胞/秒の中程度のスループットを維持します。
要約
細胞の機械的特性は、幹細胞の分化から癌の転移に至るまで、さまざまな生物学的プロセスや疾患に関与しています。原子間力顕微鏡(AFM)やマイクロピペット吸引(MA)など、これらの特性を測定する従来の方法は、細胞の完全な粘弾性応答を反映した豊富な情報をキャプチャします。ただし、これらの方法は非常に低いスループットによって制限されます。リアルタイム変形能サイトメトリー(RT-DC)などのハイスループットアプローチは、細胞の弾性特性のみを反映する単一パラメータ読み出しに制限されることが多いため、限られた機械的情報しか測定できません。これらの方法とは対照的に、メカノノードポアセンシング(mechano-NPS)は、中程度のスループットでセルのマルチパラメータ粘弾性測定を達成するためのギャップを埋める、柔軟でラベルフリーのマイクロ流体プラットフォームです。直流(DC)測定は、マイクロ流体チャネルを通過する細胞を監視し、狭い狭窄を通過する前、最中、および後に細胞のサイズと速度を追跡するために使用されます。この情報(サイズと速度)は、各セルの横方向の変形、変形に対する抵抗、および変形からの回復を定量化するために使用されます。一般に、このエレクトロニクスベースのマイクロ流体プラットフォームは、複数の粘弾性セル特性を提供し、したがって細胞の機械的状態のより完全な全体像を提供します。最小限のサンプル調製で済み、(高速カメラとは対照的に)簡単な電子測定を利用し、標準的なソフトリソグラフィ製造を利用するため、このプラットフォームの実装はシンプルでアクセスしやすく、ダウンストリーム分析に適応できます。このプラットフォームの柔軟性、有用性、感度は、多様な細胞に関する独自の機械的情報を提供し、基礎科学や臨床診断においてさらに多くのアプリケーションの可能性を秘めています。
概要
単一セルは動的な粘弾性材料です1。多数の内部および外部プロセス(例えば、有糸分裂の開始または細胞外マトリックス[ECM]のリモデリング)は、それらの構造および組成2,3,4に影響を与え、しばしばそれらの現在の状態を補完する明確な生物物理学的特性をもたらす。特に、機械的特性は、細胞の発生、生理学、および病理学の重要なバイオマーカーであることが示されており、標準的な分子的および遺伝的アプローチを補完できる貴重な定量的情報を生み出しています5、6、7。例えば、Liらは最近、薬剤耐性と薬剤応答性の急性前骨髄球性白血病細胞の機械的な違いを説明し、RNA-seqを使用して発現差のある細胞骨格関連遺伝子を明らかにしました8。単一細胞力学と細胞機能の間の複雑な相互作用を理解することにより、メカノフェノタイピングは基礎科学と臨床診断の変革においてより広い用途を持っています9。
単一細胞の力学を測定するために最も広く採用されているツールは、原子間力顕微鏡(AFM)です。AFMは、細胞の機械的特性の高分解能の局所的な測定を可能にしますが、スループットは<0.01細胞/秒10に制限されています。あるいは、2つの発散レーザービームを使用して吊り下げられた単一細胞11をトラップおよび変形させる光学ストレッチャーは、<1細胞/秒12のわずかに高いスループットに制限される。マイクロ流体技術の最近の進歩により、迅速な単一セルの機械的評価のための新世代のデバイスが可能になりました12,13。これらの技術は、狭い狭窄チャネル14、15、せん断流16、または流体力学的伸張17を使用して、10〜1,000セル/秒18のスループットで細胞を迅速に変形させる。これらのアプローチの測定速度は従来の技術よりもかなり高速ですが、限られた機械的読み出しのために高スループット能力と引き換えによく見られます(補足表1)。前述の迅速なマイクロ流体法はすべて、通過時間や変形可能性比など、細胞の弾性特性のみを反映する基本的な単一パラメータのメトリックに焦点を当てています。しかし、単一細胞の固有の粘弾性性を考えると、細胞の堅牢で徹底的な機械的特性評価には、弾性成分だけでなく粘性応答も考慮する必要があります。
メカノノードポアセンシング(mechano-NPS)2,8(図1A)は、シングルセルメカノフェノタイピングの既存の制限に対処するマイクロ流体プラットフォームです。この方法では、細胞径、相対変形能、変形からの回復時間など、複数の生物物理学的パラメータを同時に測定でき、1〜10細胞/秒の中程度のスループットが得られます。この手法は、ノード細孔センシング(NPS)19,20,21,22,23,24に基づいており、4点プローブ測定を使用して、「ノード」と呼ばれるより広い領域によってセグメント化されたマイクロ流体チャネルを通過するセルによって生成される変調電流パルスを測定します。変調された電流パルスは、セルがセグメント(すなわち「細孔」)およびノード内の電流の流れを部分的に遮断した結果であり、前者では後者よりも多くの電流が遮断される。メカノNPSでは、1つのセグメントである「収縮チャネル」はセル直径よりも狭くなります。したがって、チャネル全体を通過するには、セルが変形する必要があります(図1B)。細胞直径は、細胞が収縮チャネルの前にノード孔を通過するときに生成されるサブパルスの大きさによって決定できます(図1B、C)。ここで、|ΔYnp|は、セルが細孔内にあるときの電流降下であり、細孔に対するセルの体積比、Vセル/V細孔2,8,19に比例します。細胞の剛性は、細胞が収縮チャネルを通過するときに生成される劇的に大きなサブパルスの持続時間であるΔTcによって決定できます(図1B、C)。硬いセルは、柔らかいセルよりもチャネルを通過するのに時間がかかります2,8。最後に、細胞の「回復」、つまり変形後に元のサイズと形状に戻る細胞の能力は、細胞が収縮チャネルの後に節孔を通過するときに生成される一連のサブパルスによって決定できます(図1B、C)。回復時間ΔTrは、セルが圧迫される前に、現在のサブパルスが前のサブパルスの大きさに戻るのにかかる時間です。全体として、セルがマイクロ流体チャネルを通過するときに生成される変調電流パルスが記録され、分析されて、関連する単一セルの機械的パラメータが抽出されます(図1D)2,8。
このエレクトロニクスベースのマイクロ流体プラットフォームの再現性と使いやすさは、以前に実証されています25。さらに、このプラットフォームは、シングルセルメカノフェノタイピングの参入障壁が低い。標準的なソフトリソグラフィは、マイクロ流体デバイスの作製に採用されています。計測ハードウェアは、シンプルなプリント回路基板(PCB)、電源、プリアンプ、データ集録ボード(DAQ)、コンピュータなどの安価なコンポーネントで構成されています。最後に、データの取得と分析にユーザーフレンドリーなコードが用意されているため、簡単に実装できます。このメカノフェノタイピング技術は、非悪性および悪性の乳腺および肺上皮細胞株の集団を区別し、初代ヒト乳腺上皮細胞におけるサブ系統を区別し、細胞骨格摂動および他の薬理学的物質の影響を特徴付けることができる2,8。全体として、このプラットフォームは、単一細胞のメカノフェノタイピングに効果的なアプローチです。
プロトコル
1. デバイス形状の設計
- サイジングセグメントとリカバリセグメントの幅を選択して、測定する最大のセルの直径よりも広く、十分な信号対雑音比(SNR)を維持します。さまざまな細胞株の異なるサイジングおよび回収セグメント幅の例については、 補足表2 を参照してください。
- 収縮セグメント幅を選択して、メカノフェノタイピングを受ける細胞の平均サイズに30%〜40%のひずみを適用します。ひずみは次のように
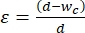 定義されます。ここで、dはセル直径、wcは収縮チャネル幅2,8です。さまざまな細胞株のさまざまな収縮セグメント幅については、補足表2を参照してください。
定義されます。ここで、dはセル直径、wcは収縮チャネル幅2,8です。さまざまな細胞株のさまざまな収縮セグメント幅については、補足表2を参照してください。
注:直径が大きく異なる細胞の種類または条件を比較する場合は、各細胞の種類/条件に固有の収縮セグメント幅で個別のデバイス設計を使用する必要があります。 - 一意のデバイス ジオメトリごとに参照デバイスを設計します。これは、De、マイクロ流体チャネルのサイジング細孔セグメントの有効直径を決定するために必要である。
メモ: 参照デバイスは、プライマリデバイスと同じジオメトリを使用します。唯一の変更は、既知のサイズのポリスチレンビーズによるキャリブレーションを可能にするために、収縮セグメントの幅がサイジング細孔セグメントと等しくなければならないことです。収縮を広げることで、キャリブレーション中にポリスチレンビーズが収縮チャネルを詰まらせるのを防ぎます。キャリブレーションプロセスは、ステップ4.1および5.3.1でさらに説明されています。較正は、市販のセルカウンターを用いても達成することができ、その場合、参照装置は必要ない。このプロセスについては、ステップ 4.2 で説明します。 - 目的の最大のセルが収縮セグメント2内で制限なく完全に伸長できるようにチャネルの高さを選択します。チャネルの高さが hmin
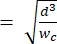 より大きいことを確認します(これは、セルが球面の事前変形であり、変形中にチャネルの長さと高さに沿って等角変形が発生することを前提としています)。
より大きいことを確認します(これは、セルが球面の事前変形であり、変形中にチャネルの長さと高さに沿って等角変形が発生することを前提としています)。
注:電流サブパルスの大きさを考えると、hminが大きいほど、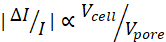 全体的なSNRは低くなります。
全体的なSNRは低くなります。 - 選択したチャネル幅のコンピューター支援設計ソフトウェアを使用して、フォトマスクを設計および作成します。サンプル・ファイルは、 補足ファイル 1 にあります。マイクロ流体マスクの設計を1.5%拡大して、ネガマスターから剥離した後のポリジメチルシロキサン(PDMS)収縮を考慮します。
注:アレイ全体がウェーハのサイズを超えない限り、デバイスのアレイを1つのマスクに含めることができます(補足図1A)。 - マイクロ流体デバイス電流の4点プローブ測定を実行するために使用される電極を備えたフォトマスクを設計および作成します(図1D)。サンプル・ファイルは、 補足ファイル 1 にあります。
注意: 電極のアレイは、アレイがスライドガラスのサイズを超えない限り、単一のマスクに含めることができます(補足図1B)。
2. デバイスの作製(図2)
- ガラス基板上に電極パターンを作製する。
- 製品データシートに従って、ポジ型フォトレジストを無地のスライドガラスにスピンコート、パターン化、および処理します。この手順の例については、 補足ファイル 2 を参照してください。
- 金属蒸着、リフトオフ、および金エッチングを実行します。
- スライド上に75 Å Ti、250 Å Pt、および250 Å Auの薄膜堆積を行います。電子銃蒸発を使用したこの手順の例は、 補足ファイル3に概説されています。
- スライドをアセトンに15分間浸して、余分な金属をリフトオフします。
- ドラフト内で、補足 図2に示すように、使い捨てピペットを使用して、マイクロ流体チャネルにさらされる電極の領域に金エッチング液をドロップキャストします。スライドの他の場所にエッチング液を落とさないように注意してください。
注意: 金のエッチング液は、皮膚や目の炎症を引き起こす可能性があります。蒸気を吸い込んだり、摂取したりしないでください。取り扱いには注意し、適切な個人用保護具(PPE)を着用し、地域の廃棄規制に従って廃棄物を廃棄してください。 - スライドを脱イオン(DI)水ですすぎ、乾燥窒素(N2)で乾燥させます。
- 同じスライドガラスに複数の電極が印刷されている場合は、スライドを個々のチップにダイスします。
- ガラス切削工具を使用して、パターン化された電極境界に沿ってスライドにスコアを付けます。
- スコアに沿ってガラスを割って、スライドを個々のチップに分割します。
- 顕微鏡で電極を目視検査します。個々の電極が電気的に開いていないこと、または電極が互いに短絡していないことを確認してください。
- チャネル用のネガティブマスターモールドを製作します。
- 製品データシートに従って、研磨されたシリコンウェーハ上にSU-8エポキシレジストをスピンコート、パターン化、および処理します。この手順の例については、 補足ファイル 2 を参照してください。
- プロファイラー(表面形状測定機)を使用して特徴の高さを測定し、顕微鏡下で特徴を視覚的に検査します(補足図3)。目的のジオメトリが適切に定義されていることを確認します。
- ソフトリソグラフィでPDMSチャンネルを成形します。
- 使い捨てカップでエラストマーと架橋剤を10:1の質量比で計量してPDMSを調製します。
注:直径3のウェーハの場合、30gのPDMSで十分です。 - PDMSが気泡で不透明になるまで、使い捨てフォークでPDMSを30秒間激しく混合します。
- 真空チャンバー内でPDMSを約30〜90分間、またはPDMSが透明になり、気泡が見えなくなるまで脱ガスします。
- SU-8マスターモールドを備えたウェーハを使い捨てのペトリ皿に入れ、ウェーハの中央にPDMSを注ぎます。
- PDMSとウェーハが入ったペトリ皿を真空チャンバーに入れ、約30分間、またはPDMSに気泡がなくなるまで脱ガスします。
- PDMSを80°Cでオーブンまたはホットプレートで2時間焼きます。
- 鋭利な刃で、PDMSを切り取り、SU-8ネガティブマスターから取り外します。
- 成形されたPDMSスラブを鋭利な刃を使用して個々の金型にダイスします
- 使い捨ての生検パンチを使用して、入口と出口のアクセス穴をコアします。最良の結果を得るには、PDMSスラブごとに新しいパンチを使用します。より鋭いパンチは滑らかなエッジの穴を生成し、収縮チャネルを塞ぐ可能性のある粒子を最小限に抑えます。
注意: アクセス穴の直径は、チューブの外径よりわずかに小さくする必要があります。たとえば、外径が1/32インチのポリテトラフルオロエチレン(PTFE)チューブを使用する場合は、1.5mmの穴を開ける必要があります。
- 使い捨てカップでエラストマーと架橋剤を10:1の質量比で計量してPDMSを調製します。
- ガラス/電極基板をPDMSチャネルに接着します。
- 電極ガラススライドをメタノール(≥99.8%)で洗浄します。乾燥N2で乾燥させる。
- PDMSデバイスをスコッチテープで洗浄し、イソプロピルアルコール(IPA)と脱イオン水(DI; 18 MΩ / cm2)ですすいでください。乾燥N2で乾燥させる。次に、もう一度スコッチテープで拭きます。
- プレハブ電極を備えたガラス基板と準備したPDMSモールド(フィーチャー面を上にして)をプラズマクリーナーに入れます。
- 両方を酸素プラズマに2分間さらします(100〜300 mTorr、30 W)。
- PDMSモールドを、フィーチャー側を下向きにして、プレハブ電極を備えたガラス基板上に位置合わせして配置します。
注:プラズマ処理されたPDMSとガラスが接触すると、接合は瞬時に行われます。したがって、それ以上のアライメントの変更はできません。アライメントを容易にするために、DI水中のメタノールの2:1希釈の20 μLをプラズマ処理ガラス表面にピペットで移すことができます。メタノール溶液は、処理されたガラスとPDMSの間の物理的バリアとして機能し、アライメント調整を可能にします。メタノールを使用する場合は、整列して嵌合したデバイスを50°Cで2時間ベークして溶液を蒸発させ、接着プロセスを完了します。 - 接着したデバイスを顕微鏡で目視検査します。電極とチャネルの形状が正しく位置合わせされていることを確認します。
3. 細胞を測定する(図1D)
- 圧力源、PCB、ベンチトップハードウェア、およびデータ収集ソフトウェアを準備します。
- クランプを使用してマイクロ流体デバイスをPCBに接続します。PCBの例は、補足ファイル4(ガーバーファイル)および補足ファイル5(回路図、ボード、およびPCB部品リストファイル)に記載されています。
- クランプのバネ仕掛けのピンをマイクロ流体デバイスの電極コンタクトパッドに合わせ、クランプのヘッダーピンをPCBの穴に合わせます。
- clをしっかりと挿入しますamp のヘッダーピンをPCBの穴に、バネ仕掛けのピンが電極コンタクトパッドと位置合わせされていることを確認します。
- 電子ハードウェアをセットアップして接続します。
- 電源の2つの出力ポートを、ダブルバナナプラグ-バヨネットニールコンセルマン(BNC)メスアダプタとBNCケーブルを使用してPCBの電源電圧ポートに接続します。
- 電源をオンにします。BNCの内部導体に接続されている出力を+15 Vに設定し、もう一方の出力を-15 Vに設定します。
- 電源の出力ポートの3番目をPCBの入力電圧ポートにBNCケーブルで接続します。出力を希望の印加電圧に設定しますが、実験を開始するまで有効にしないでください。
- PCBの出力電流ポートをBNCケーブルで電流プリアンプの入力に接続します。
- 電流プリアンプの出力を、BNCケーブルでデータ集録システムのBNC端子台の1つのアナログ入力に接続します。オプションで、アナログローパスフィルタをBNCケーブルに沿って接続して、高周波干渉を除去します。
メモ: SNR を改善するために、PCB とデバイスは厚い金属製の筐体に収納できます。すべてのBNCケーブルと流体チューブは、エンクロージャにドリルで開けられた穴に通すことができます。
- 必要なソフトウェアをパーソナルコンピュータ(PC)にインストールしてセットアップする
- 電源を入れ、圧力コントローラーをPCに接続します。製造元の指示に従って、必要な圧力コントローラソフトウェアをインストールします。
- MATLABとデータ収集ツールボックスをPCにインストールします。MATLABデータ収集ツールボックスインターフェイスがそれを検出できるように、データ収集システムに必要なドライバがインストールされていることを確認してください。
- 付属のデータ収集スクリプト「NPS.m」を https://github.com/sohnlab/node-pore-sensing-public からダウンロードします。
- データ集録スクリプトを開いて設定します。
- 正しい値を設定して、ベンダーID、DAQのデバイスID、アナログ入力チャンネル番号(付属のスクリプトの34〜36行目)を含むデータ集録セッションを初期化します。
注: デバイス ID は、関数 "daq.getDevices" または "daqlist" を使用して見つけることができます。 - 集録に必要なサンプルレートを設定します(付属のスクリプトの23行目)。最適な結果を得るには、少なくとも10kHzに設定する必要があります。
- 正しい値を設定して、ベンダーID、DAQのデバイスID、アナログ入力チャンネル番号(付属のスクリプトの34〜36行目)を含むデータ集録セッションを初期化します。
- クランプを使用してマイクロ流体デバイスをPCBに接続します。PCBの例は、補足ファイル4(ガーバーファイル)および補足ファイル5(回路図、ボード、およびPCB部品リストファイル)に記載されています。
- 細胞懸濁液を調製する。
- 1xリン酸緩衝生理食塩水(PBS)中の2%ウシ胎児血清(FBS)の溶液を調製し、0.22 μmフィルターでろ過します。
- 培養し、選択した細胞株の適切な細胞培養プロトコルに従って細胞を調製する。細胞を1x PBS中の2%FBSの調製溶液に1-5 x 105 細胞/ mLの濃度で懸濁します。実験期間中、細胞を氷上に置いておきます。
- 細胞の物性を測定します。
- セルサンプルをチューブにロードし、デバイスのインレットに接続します。
- かみそりの刃または鋭利なナイフで30cmのPTFEチューブを切ります。
- チューブの一方の端をルアーロックシリンジに取り付けます。シリンジを使用して、細胞サンプルをチューブのもう一方の端に引き込みます。
- チューブをデバイスの入口に慎重に挿入します。
- チューブの反対側の端をマイクロ流体圧力コントローラーに接続します。
注意: マイクロ流体圧力コントローラーとチューブの間にフィルターを追加して、圧力コントローラーへの液体の逆流を防ぐことができます。
- 実験を実行します。
- 圧力コントローラソフトウェアで希望の一定の駆動圧力を設定し、サンプルがデバイスを満たすようにします。
注意: 圧力は通常2〜21 kPaです。流速は、明確に定義されたパルスを可能にするのに十分遅く、適切なスループットを可能にするのに十分な速さでなければなりません。- マイクロ流体チャネルに気泡が形成された場合は、行き止まりの充填を使用します:デバイスの出口を塞ぎ、入口に低圧を加えて、気体透過性PDMSから空気を強制的に排出します。チャネルに気泡を残すと、電流ベースラインが不安定になり、正確な測定が妨げられます。
- 破片がマイクロ流体チャネルを詰まらせた場合は、駆動圧力をかけながらPDMSデバイスの上部を軽く押すか、圧力のオンとオフを切り替えてより高い圧力を「パルス」するか、チューブを取り外して再挿入して、マイクロ流体チャネルを取り除きます。破片が残っている場合は、新しいデバイスに切り替える必要がある場合があります。
- 電圧を回して目的の電圧を設定します ボリューム 電源のノブとボリュームを有効にしますtageを押して オン ボタン。
注意: 電圧は通常1〜5 Vです。 適切なSNRに必要な最低電圧を選択してください。比較するすべての条件で同じ電圧を使用する必要があります。 - 電流プリアンプをオンにし、感度(A / V)をできるだけ低く設定します。あるいは、プリアンプに過負荷をかけたり、DAQの最大アナログ入力電圧を超えたりすることなく、ゲイン(V/A)をできるだけ高く設定することもできます。この研究では、感度を10-7 A / Vに設定しました。
注意: 適切な感度/ゲイン値は、印加電圧とマイクロ流体チャネルのベースライン抵抗の両方に依存します。 - MATLABリボンメニューの緑色の [実行 ]ボタンを押して、データ集録スクリプト NPS.m を開始し、データのサンプリングと保存を開始します。
- 実験を終了するには、図ウィンドウの左下隅にある [停止 ]ボタンを押して、データ集録スクリプトを停止します。Onボタンを押して、電源出力を無効に します 。圧力コントローラソフトウェアで圧力源をゼロ圧力に設定します。
- この時点で、実験を一時停止して、次の 1 つ以上の操作を実行できます。
- 現在のデバイスを新しいデバイスと交換します。
- より多くの細胞サンプルでチューブをリロードします。
注:サンプルの相互汚染を避けるために、新しいデバイスを使用してさまざまなタイプまたは条件のセルを測定します。 - PCBからデバイスのクランプを外し、顕微鏡でチャネルの状態を調べます。同じ装置を使用して実験を再開するには、気泡が発生しないように注意する必要があります。シリンジプランジャーに穏やかな圧力を加えて、細胞サンプルをデバイスの入口に挿入しながらチューブの一番端に保つ必要がある場合があります。
- 圧力コントローラソフトウェアで希望の一定の駆動圧力を設定し、サンプルがデバイスを満たすようにします。
- セルサンプルをチューブにロードし、デバイスのインレットに接続します。
4.マイクロ流体デバイスのキャリブレーション
- オプション1:参照デバイスのポリスチレンビーズを測定します。
- サイジングチャネルよりも小さいポリスチレンビーズサイズを選択してください。
- 細胞実験中に使用したろ過したPBSおよびFBS溶液に、1.5%トゥイーンビーズおよびポリスチレンビーズを1〜3 x 105 ビーズ/mLの濃度で添加します。
- ステップ1.3で説明した参照デバイスを使用して、セクション3で概説されているように実験を進め、実験中に使用したのと同じ電圧を印加します。セクション5で説明したように、ビーズがサイジング細孔を通過するときに生成される電流液滴の平均の大きさとビーズの既知の直径を使用して、 Deを計算します。
- オプション2:別の測定装置でセルサイズを個別に測定します。
- ステップ4.1のプロトコルに従う代わりに、市販の細胞サイズ測定装置を使用して、サンプル中の細胞の平均サイズを測定します。この場合、参照デバイスは必要ありません。セルがサイジング孔を通過するときに生成される平均電流降下と測定された平均セル直径を使用して、セクション5で説明したようにDesを計算します。
5.データを分析して細胞表現型を抽出する
注 : データ処理は、https://github.com/sohnlab/NPS-analysis-JOVE の MATLAB コマンド ライン インターフェイス プログラム ファイル mNPS_procJOVE.m を使用して実行できます。詳細については、補足ファイル 6 を参照してください。
- データを前処理します(図3A)。
- 電流/電圧プリアンプで使用されるゲイン値をDAQによって集録された生データに適用することにより、測定電流を計算します。
- 生電流測定に矩形平滑化機能やローパスフィルタを適用して、高周波ノイズを除去します。次に、フィルタリングされたデータをより低いサンプルレートにリサンプリングします。また、この低いサンプルレートで対応するタイムスタンプデータを計算します。
- 非対称最小二乗平滑化26などの方法を適用して、適合ベースライン電流信号を計算する。
- 後続のデータポイント間の差を取ることにより、前処理された現在のデータの近似一次導関数(差分信号)を計算します。
- セルイベントを特定し、サブパルスデータを抽出します(図3B)。
- 前処理されたデータを調べて、候補セル イベントを検索します。他の細胞イベントと重複する細胞イベント(すなわち、一致イベント)をリジェクトする(補足図4)、ベースライン適合度が低い、または予期しないまたは誤ったパルス形状を有する(例えば、チャネルに詰まりが存在していた可能性がある)。
- 各セルイベントのサブパルスデータを抽出します。
- 各ノードポアセグメントは、信号パルス全体に対応するサブパルスとして表示されます(図1B、C)。差分信号が極小値に達する時点を計算することにより、各サブパルスの開始を特定します。差分信号がローカル最大値に達する時点を計算することにより、各サブパルスの終わりを特定します。
- 各サブパルスの幅を、開始時点と終了時点の間の経過時間として決定します。開始時点と終了時点の間のすべてのデータポイントについて、測定電流とベースライン電流の差の平均を計算することにより、各サブパルスの振幅を決定します。
- サブパルスデータに基づいて、各細胞イベントの細胞メカノフェノタイプを決定します。
- DebloisとBean24によって定義された式に基づいてセル直径dを決定します。
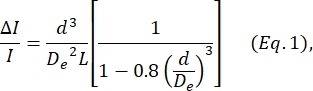
ここで、ΔI/Iはサイジングサブパルスのベースライン電流に対するサブパルス振幅の平均比、Deはチャネルの有効直径(ステップ4で測定)、Lはノードポアチャネルの全長です。- Dleは、既知の直径の粒子のセット(セルまたはビーズのいずれか、ステップ4を参照)によって生成される平均ΔI/Iを計算し、その既知の直径をdとして使用し、Deの式1を解くことによって決定されます。
- 変形に対するセルの抵抗を定量化します。
- 既知のセグメント長と各サブパルスの測定された持続時間を使用して、サイジングサブパルスの平均セル速度を計算することにより、流体速度 Uフロー を決定します。
- Kimら2によって次のように定義された全細胞変形能指数(wCDI)を決定します。
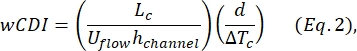
ここで、Lcは収縮セグメントの長さ、hチャネルはチャネルの高さ、ΔTcは収縮サブパルスの持続時間です。
- サイジングサブパルス2からの平均振幅の8%以内の振幅を有する第1の回復サブパルスとして定義される、変形からの細胞の回復時間を特定する。
- 収縮セグメント内のセルの横方向の変形を計算します。
- Kimら2で定義されている収縮セグメントの有効直径(De,c)を計算します:
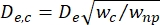 ここで、w cは収縮セグメントの幅、wnpは他のすべてのセグメントの幅です。
ここで、w cは収縮セグメントの幅、wnpは他のすべてのセグメントの幅です。 - DebloisとBean24で定義された式を再度使用して、収縮内のセルの等価球直径dcを計算します。
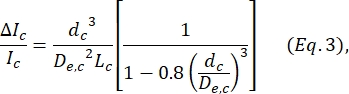
ここで、ΔIc /Icは、収縮サブパルスのベースライン電流に対するサブパルス振幅の比であり、Lcは収縮セグメントの長さです。 - Kimら2で説明されているように、セルの伸長L変形を計算します。
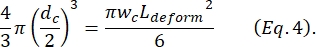
- 最後に、Kim et al.2
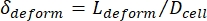 によって定義されている変形δセルの横方向の変形を計算します。
によって定義されている変形δセルの横方向の変形を計算します。
- Kimら2で定義されている収縮セグメントの有効直径(De,c)を計算します:
- DebloisとBean24によって定義された式に基づいてセル直径dを決定します。
結果
ここで紹介するメカノフェノタイピングプラットフォームは、単一細胞の生物物理学的特性を中程度のスループットで測定するためのシンプルで汎用性の高いアプローチです。細胞は、一定の圧力駆動の流れを使用してマイクロ流体チャネル(図1A)を通って流れます。細胞が通過すると、マイクロ流体チャネルの長さと生成される電流パルスがデータ収集ハードウェアを...
ディスカッション
このメカノフェノタイピング技術を使用した単一細胞の機械的特性の測定は、デバイス製造、データ取得、およびデータ分析の3つの段階で構成されています。各段階では、実験結果に大きな影響を与える可能性のある注目すべき側面があります。デバイスの製造時には、正確で再現性のある結果を得るには、一貫したチャネル形状とデバイス間の均一性が不可欠です。具体的には、各デバイ...
開示事項
L. L. Sは、2022年7月12日に発行された米国特許第11,383,241号「メカノノードポアセンシング」、J.キム、S.ハン、L.L.ソンを保有しています。
謝辞
本研究は、NIBIB 1R01EB024989-01およびNCI 1R01CA190843-01からの助成金によって支援されました。A.L.とR.R.は、H2H8協会大学院研究フェローシップの支援を受けました。KLCは、国立科学財団大学院研究フェローシップとシーベル奨学生フェローシップによってサポートされました。
資料
| Name | Company | Catalog Number | Comments |
| Acetone | J.T. Baker | 5356-05 | Purity (GC) ≥ 99.5% (https://us.vwr.com/store/product/6057739/acetone-99-5-vlsi-j-t-baker) |
| Aluminum Foil | n/a | n/a | |
| Analog Low-Pass Filter | ThorLabs | EF504 | ≤240 kHz Passband, Coaxial BNC Feedthrough (https://www.thorlabs.com/thorproduct.cfm?partnumber=EF504#ad-image-0) |
| Biopsy Punch | Integra Miltex | 33-31AA-P/25 | 1mm, Disposable, with Plunger (https://mms.mckesson.com/product/573313/Miltex-33-31AA-P25) |
| Blade | n/a | n/a | |
| BNC Cable | Pomona Electronics | 2249-C-12 | https://www.digikey.com/en/products/detail/pomona-electronics/2249-C-12/603323?utm_adgroup=Coaxial%20Cables%20%28RF%29&utm_source=google&utm_ medium=cpc&utm_campaign= Shopping_Product_Cable%20Assemblies_NEW&utm_term= &utm_content=Coaxial%20Cables%20%28RF%29&gclid=Cj0KCQjwlK-WBhDjARIsAO2sErQqnVJ pj5OXVObuTI8ZUf1ZeIn7zvzGnx mCWdePrG6SdEJMF3X6ubUaAs w-EALw_wcB |
| Cleanroom Polyester Swab | Thermo Fisher Scientific | 18383 | https://www.fishersci.com/shop/products/texwipe-cleantip-alpha-polyester-series-swabs-6/18383 |
| Current Preamplifier | DL Instruments | 1211 | https://www.brltest.com/index.php?main_page=product_info&products_ id=1419 |
| Custom PCB (w/ components) | n/a | n/a | see Supplemental files 4 and 5 |
| DAQ Terminal Block | National Instruments | BNC-2120 | https://www.ni.com/en-in/support/model.bnc-2120.html |
| DAQ to BNC-2110 cable | National Instruments | SHC68-68-EPM | https://www.ni.com/en-in/support/model.shc68-68-epm.html |
| Data Acquisition Board (DAQ) | National Instruments | PCI-6251 | https://www.ni.com/docs/en-US/bundle/pci-6251-feature/page/overview.html |
| Dessicator | Thermo Fisher Scientific | 5311-0250 | https://www.thermofisher.com/order/catalog/product/5311-0250 |
| Female BNC To Banana Plug Adapter | Pomona Electronics | 72909 | https://www.digikey.com/en/products/detail/pomona-electronics/72909/1196318 |
| Fetal Bovine Serum (FBS) | VWR | 89510-186 | https://us.vwr.com/store/product/18706419/avantor-seradigm-select-grade-usda-approved-origin-fetal-bovine-serum-fbs |
| Glass Cutter | Chemglass | CG-1179-21 | https://chemglass.com/plate-glass-cutters-diamond-tips |
| Gold Etchant TFA | Transene | NC0977944 | https://www.fishersci.com/shop/products/NC0977944/NC0977944 |
| Hot Plate | Thermo Fisher Scientific | SP131825 | |
| Isopropyl Alcohol | Spectrum Chemical | I1056-4LTPL | Purity (GC) ≥99.5% (https://www.spectrumchemical.com/isopropyl-alcohol-99-percent-fcc-i1056) |
| Metal Hardware Enclosure | Hammond Manufacturing | EJ12126 | https://www.digikey.com/en/products/detail/hammond-manufacturing/EJ12126/2423415 |
| Methanol | Sigma-Aldrich | 34860 | Purity (GC) ≥99.8% (https://www.sigmaaldrich.com/IN/en/substance/methanol320467561) |
| MF-321 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/mf-321/ |
| MICROPOSIT S1813 Positive Photoresist | DuPont | n/a | https://kayakuam.com/products/microposit-s1800-g2-series-photoresists/ |
| Phosphate Buffered Saline (PBS) | Thermo Fisher Scientific | 10010049 | https://www.thermofisher.com/order/catalog/product/10010049?SID=srch-hj-10010049 |
| Photomask | Fineline Imaging | n/a | Photomask are custom ordered from our CAD designs (https://www.fineline-imaging.com/) |
| Plain Glass Microscope Slide | Fisher Scientific | 12-553-5B | Material: Soda Lime, L75 x W50 mm, Thickness: 0.90–1.10 mm |
| Plasma Cleaner | Harrick Plasma | PDC-001 | https://harrickplasma.com/plasma-cleaners/expanded-plasma-cleaner/ |
| Plastic Petri Dish | Thermo Fisher Scientific | FB0875712 | 100 mm (https://www.fishersci.com/shop/products/fisherbrand-petri-dishes-clear-lid-raised-ridge-100-x-15mm/FB0875712) |
| Pressure Controller | Fluigent | MFCS-EZ | https://www.fluigent.com/research/instruments/pressure-flow-controllers/mfcs-series/ |
| Pressure Controller Software | Fluigent | MAESFLO | |
| Programming & Computation Software | MATLAB | R2021b | for data acquisition and analysis (https://www.mathworks.com/products/matlab.html) |
| PTFE Tubing | Cole Parmer | 06417-31 | 0.032" ID x 0.056" (https://www.coleparmer.com/i/masterflex-transfer-tubing-microbore-ptfe-0-032-id-x-0-056-od-100-ft-roll/0641731) |
| Scepter 2.0 Handheld Automatic Cell Counter | Millapore Sigma | PHCC20060 | https://www.sigmaaldrich.com/IN/en/product/mm/phcc20060 |
| Silicon Wafer | Wafer World | 2885 | 76.2 mm, Single Side Polished (https://www.waferworld.com/product/2885) |
| Spin Coater | n/a | n/a | |
| SU-8 3025 Negative Photoresist | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-2000/ |
| SU8 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-developer/ |
| Sygard 184 Polydimethlysiloxane | Dow Chemical | 4019862 | https://www.ellsworth.com/products/by-market/consumer-products/encapsulants/silicone/dow-sylgard-184-silicone-encapsulant-clear-0.5-kg-kit/ |
| Tape | Scotch | 810-341296 | https://www.staples.com/Scotch-Magic-Tape-810-3-4-x-36-yds-1-Core/product_130567?cid=PS:GS:SBD:PLA:OS&gclid= Cj0KCQjwlK-WBhDjARIsAO 2sErRwzrrgjU0NjFkDkne1xm vT7ekS3tdzvAgiMDwPoxocgH VTQZi7vJgaAvQZEALw_wcB |
| Titanium, Platinum, Gold | n/a | n/a | |
| Triple Output Power Supply | Keysight | E36311A | https://www.newark.com/keysight-technologies/e36311a/dc-power-supply-3o-p-6v-5a-prog/dp/15AC9653 |
| UV Mask Aligner | Karl Suss America | MJB3 Mask Aligner |
参考文献
- Pegoraro, A. F., Janmey, P., Weitz, D. A. Mechanical properties of the cytoskeleton and cells. Cold Spring Harbor Perspectives in Biology. 9 (11), 022038 (2017).
- Kim, J., et al. Characterizing cellular mechanical phenotypes with mechano-node-pore sensing. Microsystems & Nanoengineering. 4, 17091 (2018).
- Mierke, C. T. Bidirectional mechanical response between cells and their microenvironment. Frontiers in Physics. 9, 619 (2021).
- Kumar, S., Weaver, V. M. Mechanics, malignancy, and metastasis: The force journey of a tumor cell. Cancer and Metastasis Reviews. 28 (1), 113-127 (2009).
- Nia, H. T., Munn, L. L., Jain, R. K. Physical traits of cancer. Science. 370 (6516), (2020).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463 (7280), 485-492 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: The role of physical interactions and mechanical forces in metastasis. Nature Reviews Cancer. 11 (7), 512-522 (2011).
- Li, B., et al. Mechanical phenotyping reveals unique biomechanical responses in retinoic acid-resistant acute promyelocytic leukemia. iScience. 25 (2), 103772 (2022).
- Kozminsky, M., Sohn, L. L. The promise of single-cell mechanophenotyping for clinical applications. Biomicrofluidics. 14 (3), 031301 (2020).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Wottawah, F., et al. Optical rheology of biological cells. Physical Review Letters. 94 (9), 1-4 (2005).
- Darling, E. M., Di Carlo, D. High-throughput assessment of cellular mechanical properties. Annual Review of Biomedical Engineering. 17 (1), 35-62 (2015).
- Carey, T. R., Cotner, K. L., Li, B., Sohn, L. L. Developments in label-free microfluidic methods for single-cell analysis and sorting. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 11 (1), 1529 (2019).
- Bagnall, J. S., et al. Deformability of tumor cells versus blood cells. Scientific Reports. 5, 18542 (2015).
- Byun, S., et al. Characterizing deformability and surface friction of cancer cells. Proceedings of the National Academy of Sciences. 110 (19), 7580-7585 (2013).
- Otto, O., et al. Real-time deformability cytometry: On-the-fly cell mechanical phenotyping. Nature Methods. 12 (3), 199-202 (2015).
- Gossett, D. R., et al. Hydrodynamic stretching of single cells for large population mechanical phenotyping. Proceedings of the National Academy of Sciences. 109 (20), 7630-7635 (2012).
- Guck, J., Chilvers, E. R. Mechanics meets medicine. Science Translational Medicine. 5 (212), 3-6 (2013).
- Balakrishnan, K. R., et al. Node-pore sensing: A robust, high-dynamic range method for detecting biological species. Lab on a Chip. 13 (7), 1302-1307 (2013).
- Carbonaro, A., Sohn, L. L. A resistive-pulse sensor chip for multianalyte immunoassays. Lab on a Chip. 5 (10), 1155-1160 (2005).
- Saleh, O. A., Sohn, L. L. Direct detection of antibody-antigen binding using an on-chip artificial pore. Proceedings of the National Academy of Sciences. 100 (3), 820-824 (2003).
- Saleh, O. A., Sohn, L. L. An artificial nanopore for molecular sensing. Nano Letters. 3 (1), 37-38 (2003).
- Saleh, O. A., Sohn, L. L. Quantitative sensing of nanoscale colloids using a microchip Coulter counter. Review of Scientific Instruments. 72 (12), 4449-4451 (2001).
- DeBlois, R. W., Bean, C. P. Counting and sizing of submicron particles by the resistive pulse technique. Review of Scientific Instruments. 41 (7), 909-916 (1970).
- Li, B., et al. Evaluating sources of technical variability in the mechano-node-pore sensing pipeline and their effect on the reproducibility of single-cell mechanical phenotyping. PLoS ONE. 16 (10), 0258982 (2021).
- Zhang, Z. M., Chen, S., Liang, Y. Z. Baseline correction using adaptive iteratively reweighted penalized least squares. Analyst. 135 (5), 1138-1146 (2010).
- Alibert, C., Goud, B., Manneville, J. B. Are cancer cells really softer than normal cells. Biology of the Cell. 109 (5), 167-189 (2017).
- Fujiwara, I., Zweifel, M. E., Courtemanche, N., Pollard, T. D. Latrunculin A accelerates actin filament depolymerization in addition to sequestering actin monomers. Current Biology. 28 (19), 3183-3192 (2018).
- Saleh, O. A. . A novel resistive pulse sensor for biological measurements. , (2003).
- Dokukin, M. E., Guz, N. V., Sokolov, I. Quantitative study of the elastic modulus of loosely attached cells in AFM indentation experiments. Biophysical Journal. 104 (10), 2123-2131 (2013).
- Li, Q., Lim, C. T., Goh, J. C. H., et al. Probing the elasticity of breast cancer cells using AFM. 13th International Conference on Biomedical Engineering. IFMBE Proceedings. 23, 2122-2125 (2009).
- Rother, J., et al. Atomic force microscopy-based microrheology reveals significant differences in the viscoelastic response between malign and benign cell lines. Open Biology. 4 (5), 140046 (2014).
- Li, Q., et al. AFM indentation study of breast cancer cells. Biochemical and Biophysical Research Communications. 374 (4), 609-613 (2008).
- Xu, C., et al. Elasticity measurement of breast cancer cells by atomic force microscopy. Proc. SPIE 9230. Twelfth International Conference on Photonics and Imaging in Biology and Medicine. (PIBM 2014). 92300, (2014).
- Alcaraz, J., et al. Microrheology of human lung epithelial cells measured by atomic force microscopy. Biophysical Journal. 84 (3), 2071-2079 (2003).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Urbanska, M., et al. A comparison of microfluidic methods for high-throughput cell deformability measurements. Nature Methods. 17, 587-593 (2020).
- Hill, R. T., Chilkoti, A. Surface Patterning. Biomaterials Science: An Introduction to Materials: Third Edition. , 276-301 (2013).
- Wang, Z., Volinsky, A. A., Gallant, N. D. Crosslinking effect on polydimethylsiloxane elastic modulus measured by custom-built compression instrument. Journal of Applied Polymer Science. 131 (22), 41050 (2014).
- Gibson, L. J. The hierarchical structure and mechanics of plant materials. Journal of the Royal Society Interface. 9 (76), 2749-2766 (2012).
- Stephens, A. D., Banigan, E. J., Adam, S. A., Goldman, R. D., Marko, J. F. Chromatin and lamin a determine two different mechanical response regimes of the cell nucleus. Molecular Biology of the Cell. 28 (14), 1984-1996 (2017).
- Rosenbluth, M. J., Lam, W. A., Fletcher, D. A. Force microscopy of nonadherent cells: A comparison of leukemia cell deformability. Biophysical Journal. 90 (8), 2994-3003 (2006).
- Evers, T. M. J., Holt, L. J., Alberti, S., Mashaghi, A. Reciprocal regulation of cellular mechanics and metabolism. Nature Metabolism. 3 (4), 456-468 (2021).
- Balakrishnan, K. R., et al. Node-pore sensing enables label-free surface-marker profiling of single cells. Analytical Chemistry. 87 (5), 2988-2995 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved