É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Mechano-Node-Pore Sensing: Uma plataforma rápida e sem rótulos para medições viscoelásticas de célula única multiparâmetro
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Apresentamos aqui um método para fenótipo mecânico de células individuais usando uma plataforma microfluídica baseada em eletrônica chamada detecção de mechano-node-pore (mechano-NPS). Esta plataforma mantém um rendimento moderado de 1-10 células/s enquanto mede as propriedades biofísicas elásticas e viscosas das células.
Resumo
As propriedades mecânicas celulares estão envolvidas em uma ampla variedade de processos biológicos e doenças, que vão desde a diferenciação de células-tronco até a metástase do câncer. Métodos convencionais para medir essas propriedades, como microscopia de força atômica (AFM) e aspiração de micropipeta (MA), capturam informações ricas, refletindo a resposta viscoelástica completa de uma célula; no entanto, esses métodos são limitados por uma taxa de transferência muito baixa. Abordagens de alto rendimento, como a citometria de deformabilidade em tempo real (RT-DC), só podem medir informações mecânicas limitadas, pois geralmente são restritas a leituras de parâmetro único que refletem apenas as propriedades elásticas de uma célula. Em contraste com esses métodos, o sensoriamento de poros de nós mecânicos (mecanano-NPS) é uma plataforma microfluídica flexível e livre de rótulos que preenche a lacuna na obtenção de medições viscoelásticas multiparâmetros de uma célula com rendimento moderado. Uma medição de corrente contínua (CC) é usada para monitorar as células à medida que elas transitam por um canal microfluídico, rastreando seu tamanho e velocidade antes, durante e depois de serem forçadas através de uma constrição estreita. Essas informações (ou seja, tamanho e velocidade) são usadas para quantificar a deformação transversal de cada célula, a resistência à deformação e a recuperação da deformação. Em geral, essa plataforma microfluídica baseada em eletrônica fornece múltiplas propriedades de células viscoelásticas e, portanto, uma imagem mais completa do estado mecânico de uma célula. Como requer preparação mínima da amostra, utiliza uma medição eletrônica direta (em contraste com uma câmera de alta velocidade) e aproveita a fabricação de litografia suave padrão, a implementação dessa plataforma é simples, acessível e adaptável à análise a jusante. A flexibilidade, a utilidade e a sensibilidade dessa plataforma forneceram informações mecânicas exclusivas em uma gama diversificada de células, com o potencial para muitas outras aplicações em ciência básica e diagnóstico clínico.
Introdução
As células individuais são materiais dinâmicos e viscoelásticos1. Uma infinidade de processos internos e externos (por exemplo, início da mitose ou remodelamento da matriz extracelular [MEC]) influenciam sua estrutura e composição 2,3,4, resultando muitas vezes em propriedades biofísicas distintas que complementam seu estado atual. Em particular, as propriedades mecânicas têm se mostrado importantes biomarcadores do desenvolvimento celular, fisiologia e patologia, produzindo informações quantitativas valiosas que podem complementar as abordagens moleculares e genéticas canônicas 5,6,7. Por exemplo, Li et al. descreveram recentemente as diferenças mecânicas entre células de leucemia promielocítica aguda resistentes e responsivas a medicamentos, ao mesmo tempo em que usaram RNA-seq para descobrir genes associados ao citoesqueleto diferencialmente expressos8. Ao compreender a complexa interação entre a mecânica unicelular e a função celular, a mecanofenotipagem tem aplicações mais amplas na transformação da ciência básica e do diagnóstico clínico9.
A ferramenta mais amplamente adotada para medir a mecânica unicelular é a microscopia de força atômica (AFM). Embora o AFM permita uma medição localizada de alta resolução das propriedades mecânicas celulares, ele permanece limitado a uma taxa de transferência de <0,01 células/s10. Alternativamente, as macas ópticas, que usam dois feixes de laser divergentes para prender e deformar células únicas suspensas11, são limitadas a rendimentos marginalmente mais altos de <1 célula/s12. Avanços recentes em tecnologias microfluídicas permitiram uma nova geração de dispositivos para avaliação mecânica rápida, unicelular12,13. Essas técnicas empregam canais de constrição estreitos14,15, fluxo de cisalhamento 16 ou alongamento hidrodinâmico17 para deformar células rapidamente em rendimentos de 10-1.000 células/s 18. Embora a taxa de medição dessas abordagens seja consideravelmente mais rápida do que as técnicas convencionais, elas geralmente trocam recursos de alto rendimento por leituras mecânicas limitadas (Tabela Suplementar 1). Todos os métodos microfluídicos rápidos acima mencionados se concentram em métricas básicas de parâmetro único, como tempo de trânsito ou taxas de deformabilidade, que refletem apenas as propriedades elásticas de uma célula. No entanto, dada a natureza viscoelástica intrínseca de células individuais, uma caracterização mecânica robusta e completa das células requer a consideração não apenas de componentes elásticos, mas também de respostas viscosas.
Mechano-node-pore sensing (mechano-NPS)2,8 (Figura 1A) é uma plataforma microfluídica que aborda as limitações existentes com a mechanofenotipagem de célula única. Este método permite a medição de múltiplos parâmetros biofísicos simultaneamente, incluindo diâmetro celular, deformabilidade relativa e tempo de recuperação da deformação, com um rendimento moderado de 1-10 células/s. Essa técnica é baseada no node-pore sensing (NPS)19,20,21,22,23,24, que envolve o uso de uma medição de sonda de quatro pontos para medir o pulso de corrente modulada produzido por uma célula transitando por um canal microfluídico que foi segmentado por regiões mais amplas, referidas como "nós". O pulso de corrente modulada é resultado de a célula bloquear parcialmente o fluxo de corrente nos segmentos (ou seja, "poros") e nós, com mais corrente bloqueada no primeiro do que no segundo. No mecano-NPS, um segmento, o "canal de contração", é mais estreito do que o diâmetro de uma célula; consequentemente, uma célula deve se deformar para transitar por todo o canal (Figura 1B). O diâmetro da célula pode ser determinado pela magnitude do subpulso produzido quando a célula transita pelos poros do nó antes do canal de contração (Figuras 1B,C). Aqui, |ΔInp|, a queda de corrente quando a célula está no poro, é proporcional à razão de volume da célula para o poro, célula V/poro V 2,8,19. A rigidez celular pode ser determinada por ΔTc, a duração do subpulso dramaticamente maior produzido quando a célula transita pelo canal de contração (Figuras 1B,C). Uma célula mais rígida levará mais tempo para transitar pelo canal do que uma mais macia 2,8. Finalmente, a "recuperação" celular, a capacidade da célula de retornar ao seu tamanho original e forma pós-deformação, pode ser determinada pela série de subpulsos produzidos à medida que a célula transita pelos poros dos nós após o canal de contração (Figuras 1B,C). O tempo de recuperação, ΔTr, é o tempo que leva para que os subpulsos atuais retornem à magnitude dos subpulsos anteriores, antes da célula ser espremida. De modo geral, os pulsos de corrente modulada produzidos à medida que uma célula transita pelo canal microfluídico são registrados e analisados para extrair os parâmetros mecânicos de célula única relevantes (Figura 1D)2,8.
A reprodutibilidade e a facilidade de uso dessa plataforma microfluídica baseada em eletrônica foram previamente demonstradas25. Além disso, a plataforma apresenta uma baixa barreira de entrada para a mecanofenotipagem unicelular. A litografia suave padrão é empregada para fabricar dispositivos microfluídicos. O hardware de medição consiste em componentes baratos, incluindo uma placa de circuito impresso simples (PCB), fonte de alimentação, pré-amplificador, placa de aquisição de dados (DAQ) e computador. Finalmente, o código amigável está disponível para aquisição e análise de dados, permitindo uma implementação direta. Essa técnica de mecanofenotipagem pode distinguir populações de linhagens celulares epiteliais mamárias e pulmonares não malignas e malignas, discriminar sublinhagens em células epiteliais mamárias humanas primárias e caracterizar os efeitos de perturbações citoesqueléticas e outros agentes farmacológicos 2,8. No geral, esta plataforma é uma abordagem eficaz para a mecanofenotipagem de células individuais.
Protocolo
1. Projete a geometria do dispositivo
- Escolha a largura dos segmentos de dimensionamento e recuperação para que seja maior do que o diâmetro das maiores células a serem medidas, mas também mantenha uma relação sinal-ruído (SNR) suficiente. Consulte a Tabela Suplementar 2 para obter exemplos de diferentes larguras de segmento de dimensionamento e recuperação para várias linhas celulares.
- Escolha a largura do segmento de contração para aplicar uma tensão de 30% a 40% ao tamanho médio das células que serão submetidas à mecanofenotipagem. A deformação é definida como
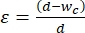 , onde d é o diâmetro da célula e wc é a largura do canal de contração 2,8. Consulte a Tabela Suplementar 2 para diferentes larguras de segmento de contração para várias linhas celulares.
, onde d é o diâmetro da célula e wc é a largura do canal de contração 2,8. Consulte a Tabela Suplementar 2 para diferentes larguras de segmento de contração para várias linhas celulares.
NOTA: Se alguém deseja comparar tipos de células ou condições com diâmetros substancialmente diferentes, projetos de dispositivos separados devem ser usados com larguras de segmento de contração específicas para cada tipo/condição de célula. - Projete um dispositivo de referência para cada geometria de dispositivo exclusiva. Isso é necessário para determinar De, o diâmetro efetivo do segmento de poros de dimensionamento do canal microfluídico.
Observação : O dispositivo de referência usa a mesma geometria que o dispositivo primário. A única modificação é que o segmento de contração deve ser igual em largura ao segmento de poros de dimensionamento para permitir a calibração com esferas de poliestireno de tamanho conhecido. O alargamento da contração impede que as esferas de poliestireno entupam o canal de contração durante a calibração. O processo de calibração é descrito em pormenor nas etapas 4.1 e 5.3.1. A calibração também pode ser obtida usando um contador de células comercialmente disponível, caso em que nenhum dispositivo de referência é necessário. Esse processo é descrito na etapa 4.2. - Escolha a altura do canal de tal forma que as maiores células de interesse possam se alongar totalmente sem restrição dentro do segmento de contração2. Certifique-se de que a altura do canal é maior que hmin
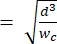 (isso pressupõe que a célula é pré-deformação esférica e que a deformação isométrica ocorre ao longo do comprimento e altura do canal durante a deformação).
(isso pressupõe que a célula é pré-deformação esférica e que a deformação isométrica ocorre ao longo do comprimento e altura do canal durante a deformação).
NOTA: Dada a magnitude de um subpulso de corrente,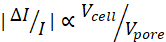 , quanto maior for o hmin , menor será o SNR geral.
, quanto maior for o hmin , menor será o SNR geral. - Projete e crie uma fotomáscara usando um software de design assistido por computador com as larguras de canal escolhidas. Um arquivo de exemplo é fornecido no Arquivo Suplementar 1. Dimensione o design da máscara microfluídica em 1,5% para explicar o encolhimento do polidimetilsiloxano (PDMS) após o peeling do mestre negativo.
NOTA: Uma matriz de dispositivos pode ser incluída em uma única máscara, desde que a matriz geral não exceda o tamanho da bolacha (Figura 1A Suplementar). - Projetar e criar uma fotomáscara com eletrodos que serão usados para realizar uma medição de sonda de quatro pontos da corrente do dispositivo microfluídico (Figura 1D). Um arquivo de exemplo é fornecido no Arquivo Suplementar 1.
NOTA: Uma matriz de eletrodos pode ser incluída em uma única máscara, desde que a matriz não exceda o tamanho da lâmina de vidro (Figura 1B suplementar).
2. Fabricar dispositivos (Figura 2)
- Prepare padrões de eletrodos em um substrato de vidro.
- Gire o revestimento, o padrão e processe uma fotorresistência positiva em uma lâmina de vidro liso de acordo com a folha de dados do produto. Um exemplo desse procedimento é descrito no Arquivo Suplementar 2.
- Realize deposição de metal, decolagem e gravação de ouro.
- Execute a deposição de filme fino de 75 Å Ti, 250 Å Pt e 250 Å Au no slide. Um exemplo desse procedimento usando evaporação por pistola de elétrons é descrito no Arquivo Suplementar 3.
- Mergulhe a lâmina em acetona por 15 minutos para realizar uma decolagem do excesso de metal.
- Em um exaustor, use uma pipeta descartável para gota de ouro fundido etchant na região dos eletrodos que serão expostos ao canal microfluídico, como mostrado na Figura Suplementar 2. Seja cauteloso para evitar deixar cair etchant em outro lugar no slide.
CUIDADO: O etchant dourado pode causar irritação da pele e dos olhos. Não respire vapores e não ingerie. Manuseie com cuidado, use equipamentos de proteção individual (EPI) apropriados e descarte os resíduos de acordo com os regulamentos locais de descarte. - Enxaguar a lâmina com água deionizada (DI) e secar com nitrogênio seco (N2).
- Se vários eletrodos forem impressos na mesma lâmina de vidro, cubra a lâmina em chips individuais.
- Use uma ferramenta de corte de vidro para marcar a deslizamento ao longo dos limites padronizados do eletrodo.
- Quebre o vidro ao longo da pontuação para dividir o slide em chips individuais.
- Inspecione visualmente os eletrodos sob um microscópio. Certifique-se de que os eletrodos individuais não estejam eletricamente abertos ou que os eletrodos não estejam em curto-circuito.
- Fabrice um molde mestre negativo para canais.
- Gire o revestimento, o padrão e o processo de um SU-8 epóxi resistem a uma bolacha de silício polido de acordo com a folha de dados do produto. Um exemplo desse procedimento é descrito no Arquivo Suplementar 2.
- Meça as alturas dos recursos usando um perfilômetro e inspecione visualmente os recursos sob um microscópio (Figura Suplementar 3). Certifique-se de que as geometrias desejadas estejam bem definidas.
- Molde de canais PDMS com litografia suave.
- Prepare o PDMS pesando um elastômero e um reticulador na proporção de massa de 10:1 em um copo descartável.
NOTA: Para uma bolacha com 3 de diâmetro, 30 g de PDMS são suficientes. - Misture o PDMS vigorosamente por 30 s com um garfo descartável, até que o PDMS fique opaco com bolhas.
- Desgaseifique o PDMS em uma câmara de vácuo por aproximadamente 30-90 min, ou até que o PDMS seja transparente sem bolhas visíveis.
- Coloque a bolacha com o molde mestre SU-8 em uma placa de Petri descartável e despeje PDMS sobre o centro da bolacha.
- Coloque a placa de Petri contendo o PDMS e a bolacha em uma câmara de vácuo e desgaseifique por aproximadamente 30 min, ou até que nenhuma bolha permaneça no PDMS.
- Asse o PDMS a 80 °C durante 2 h no forno ou numa placa quente.
- Com uma lâmina afiada, corte e remova o PDMS do mestre negativo SU-8.
- Corte a laje PDMS moldada em moldes individuais usando uma lâmina afiada
- Núcleo dos orifícios de acesso de entrada e saída usando um soco de biópsia descartável. Para obter melhores resultados, use um novo punção para cada laje PDMS. Um soco mais nítido produz orifícios de bordas lisas, minimizando as partículas que poderiam obstruir o canal de contração.
NOTA: O diâmetro dos orifícios de acesso deve ser ligeiramente inferior ao diâmetro exterior da tubagem. Por exemplo, se estiver usando tubos de politetrafluoroetileno (PTFE) com um diâmetro externo de 1/32 pol, um orifício de 1,5 mm deve ser perfurado.
- Prepare o PDMS pesando um elastômero e um reticulador na proporção de massa de 10:1 em um copo descartável.
- Ligue um substrato de vidro/eletrodo aos canais PDMS.
- Limpe as lâminas de vidro do eletrodo com metanol (≥99,8%). Secar com N2 seco.
- Limpar o dispositivo PDMS com fita adesiva, seguido de enxágue com álcool isopropílico (IPA) e água deionizada (DI; 18 MΩ/cm2). Secar com N2 seco. Em seguida, limpe com fita adesiva mais uma vez.
- Coloque o substrato de vidro com eletrodos pré-fabricados e o molde PDMS preparado (lado a lado do recurso) em um limpador a plasma.
- Expor ambos ao plasma de oxigênio por 2 min (100-300 mTorr, 30 W).
- Alinhe e coloque o molde PDMS com o lado do recurso virado para baixo no substrato de vidro com eletrodos pré-fabricados.
NOTA: A ligação é instantânea uma vez que o PDMS tratado com plasma e o vidro entram em contato; consequentemente, não serão possíveis novas modificações de alinhamento. Para facilitar o alinhamento, 20 μL de uma diluição 2:1 de metanol em água DI podem ser pipetados na superfície de vidro tratada com plasma. A solução de metanol atua como uma barreira física entre o vidro tratado e o PDMS, permitindo ajustes de alinhamento. Se utilizar metanol, cozer o dispositivo alinhado e acoplado a 50 °C durante 2 h para evaporar a solução e completar o processo de colagem. - Inspecione visualmente o dispositivo ligado sob um microscópio. Certifique-se de que os eletrodos e as geometrias do canal estejam devidamente alinhados.
3. Meça as células (Figura 1D)
- Prepare a fonte de pressão, a PCB, o hardware de bancada e o software de aquisição de dados.
- Conecte o dispositivo microfluídico à PCB usando a braçadeira. Um exemplo da PCB é fornecido no Arquivo Suplementar 4 (arquivos GERBER) e no Arquivo Suplementar 5 (arquivos esquemáticos, de placa e de lista de peças PCB).
- Alinhe os pinos carregados por mola da braçadeira com as almofadas de contato do eletrodo no dispositivo microfluídico e alinhe os pinos do conector da braçadeira com os orifícios da PCB.
- Insira firmemente os pinos do cabeçote da braçadeira nos orifícios da PCB, certificando-se de que os pinos carregados por mola permaneçam alinhados com as almofadas de contato do eletrodo.
- Configure e conecte o hardware eletrônico.
- Conecte duas das portas de saída da fonte de alimentação à porta de tensão de alimentação da PCB com um adaptador fêmea duplo banana plug-to-Bayonet Neill-Concelman (BNC) e um cabo BNC.
- Ligue a fonte de alimentação. Defina a saída conectada ao condutor interno do BNC para +15 V e defina a outra saída como -15 V. Habilite ambas as saídas para alimentar o circuito.
- Conecte a terceira das portas de saída da fonte de alimentação à porta de tensão de entrada da PCB com um cabo BNC. Defina a saída para a tensão aplicada desejada, mas não a habilite até iniciar o experimento.
- Conecte a porta de corrente de saída da PCB à entrada do pré-amplificador de corrente com um cabo BNC.
- Conecte a saída do pré-amplificador de corrente a uma entrada analógica no bloco de terminais BNC do sistema de aquisição de dados com um cabo BNC. Opcionalmente, conecte um filtro passa-baixas analógico alinhado com o cabo BNC para filtrar a interferência de alta frequência.
NOTA: Para melhorar o SNR, a PCB e o dispositivo podem ser alojados dentro de um gabinete de metal espesso. Todos os cabos BNC e tubulações fluídicas podem ser encaminhados através de furos perfurados no gabinete.
- Instalar e configurar o software necessário no computador pessoal (PC)
- Ligue e conecte o controlador de pressão ao PC. Instale qualquer software controlador de pressão necessário de acordo com as instruções do fabricante.
- Instale o MATLAB e a Caixa de Ferramentas de Aquisição de Dados no PC. Verifique se os drivers necessários para o sistema de aquisição de dados estão instalados para que a interface do MATLAB Data Acquisition Toolbox possa detectá-lo.
- Baixe o script de aquisição de dados incluído, "NPS.m", de https://github.com/sohnlab/node-pore-sensing-public.
- Abra e configure o script de aquisição de dados.
- Defina os valores corretos para inicializar a sessão de aquisição de dados, que inclui a ID do fornecedor, a ID do dispositivo do DAQ e o número do canal de entrada analógico (linhas 34-36 no script incluído).
NOTA: O ID do dispositivo pode ser encontrado usando a função "daq.getDevices" ou "daqlist". - Defina a taxa de amostragem desejada para a aquisição (linha 23 no script incluído). Para obter resultados ótimos, ele deve ser ajustado para pelo menos 10 kHz.
- Defina os valores corretos para inicializar a sessão de aquisição de dados, que inclui a ID do fornecedor, a ID do dispositivo do DAQ e o número do canal de entrada analógico (linhas 34-36 no script incluído).
- Conecte o dispositivo microfluídico à PCB usando a braçadeira. Um exemplo da PCB é fornecido no Arquivo Suplementar 4 (arquivos GERBER) e no Arquivo Suplementar 5 (arquivos esquemáticos, de placa e de lista de peças PCB).
- Prepare a suspensão celular.
- Preparar uma solução de soro fetal bovino a 2% (FBS) em solução salina tamponada com fosfato (PBS) a 1x e filtrar com um filtro de 0,22 μm.
- Cultivar e preparar as células de acordo com o protocolo de cultura celular apropriado da linhagem celular de escolha. Suspender as células na solução preparada de FBS a 2% em 1x PBS na concentração de 1-5 x 105 células/mL. Mantenha as células no gelo durante a duração dos experimentos.
- Meça as propriedades físicas das células.
- Carregue a amostra de célula na tubulação e conecte-a à entrada do dispositivo.
- Corte 30 cm de tubo de PTFE com uma lâmina de barbear ou faca afiada.
- Fixe uma extremidade da tubulação a uma seringa luer lock. Utilize a seringa para extrair a amostra de células para a outra extremidade da tubagem.
- Insira cuidadosamente a tubulação na entrada do dispositivo.
- Conecte a extremidade oposta da tubulação ao controlador de pressão microfluídica.
NOTA: Um filtro pode ser adicionado entre o controlador de pressão microfluídico e a tubulação para evitar o refluxo de líquido para o controlador de pressão.
- Execute o experimento.
- Defina a pressão de acionamento constante desejada no software controlador de pressão e permita que a amostra encha o dispositivo.
NOTA: A pressão é tipicamente de 2-21 kPa. A velocidade do fluxo deve ser lenta o suficiente para permitir pulsos claramente definidos, mas rápida o suficiente para permitir uma taxa de transferência adequada.- Se formarem bolhas nos canais microfluídicos, use o enchimento sem saída: conecte a saída do dispositivo e aplique uma baixa pressão na entrada para forçar a saída do ar através do PDMS permeável ao gás. Deixar bolhas no canal levará a uma linha de base de corrente instável e impedirá medições precisas.
- Se os detritos obstruírem o canal microfluídico, desaloje-o pressionando levemente a parte superior do dispositivo PDMS enquanto aplica a pressão de condução, "pulsando" uma pressão mais alta alternando a pressão para cima e para fora, ou removendo a tubulação e reinserindo-a. Se os detritos permanecerem, pode ser necessário mudar para um novo dispositivo.
- Defina a tensão desejada girando o botão Voltage na fonte de alimentação e ative a tensão pressionando o botão On .
NOTA: A tensão é tipicamente de 1-5 V. Escolha a tensão mais baixa necessária para um SNR adequado. A mesma tensão deve ser usada em todas as condições a serem comparadas. - Ligue o pré-amplificador de corrente e defina a sensibilidade (A/V) o mais baixa possível; alternativamente, defina o ganho (V/A) o mais alto possível sem sobrecarregar o pré-amplificador ou exceder a tensão máxima de entrada analógica do DAQ. Neste estudo, a sensibilidade foi ajustada para 10-7 A/V.
NOTA: O valor adequado de sensibilidade/ganho dependerá tanto da tensão aplicada quanto da resistência basal do canal microfluídico. - Pressione o botão verde Executar no menu da faixa de opções do MATLAB para iniciar o script de aquisição de dados NPS.m e iniciar a amostragem e o salvamento dos dados.
- Para encerrar o experimento, pressione o botão Parar no canto inferior esquerdo da janela de figura para interromper o script de aquisição de dados. Desative a saída da fonte de alimentação pressionando o botão Ligado . Ajuste a fonte de pressão para pressão zero no software do controlador de pressão.
- Neste ponto, o experimento pode ser pausado para executar um ou mais dos seguintes procedimentos:
- Substitua o dispositivo atual por um novo.
- Recarregue a tubulação com mais amostras de células.
NOTA: Para evitar a contaminação cruzada da amostra, use novos dispositivos para medir células de diferentes tipos ou condições. - Desaperte o dispositivo da PCB e examine a condição do canal sob um microscópio. Para reiniciar o experimento usando o mesmo dispositivo, deve-se tomar cuidado para não introduzir bolhas de ar. Pode ser necessário aplicar uma pressão suave no êmbolo da seringa para manter a amostra de célula no final da tubulação enquanto a insere na entrada do dispositivo.
- Defina a pressão de acionamento constante desejada no software controlador de pressão e permita que a amostra encha o dispositivo.
- Carregue a amostra de célula na tubulação e conecte-a à entrada do dispositivo.
4. Calibre o dispositivo microfluídico
- Opção 1: Meça as esferas de poliestireno em dispositivos de referência.
- Escolha um tamanho de grânulo de poliestireno que seja menor que o canal de dimensionamento.
- Adicionar contas de Tween e poliestireno a 1,5% à solução filtrada de PBS e FBS utilizada durante os experimentos celulares, na concentração de 1-3 x 105 contas/mL.
- Prosseguir com a experiência conforme descrito na secção 3, utilizando o dispositivo de referência descrito no passo 1.3, e aplicar a mesma tensão utilizada durante a experimentação. Utilizar a magnitude média da gota de corrente produzida à medida que as esferas transitam pelos poros de dimensionamento e o diâmetro conhecido das esferas para calcular De, conforme descrito na secção 5.
- Opção 2: Meça independentemente o tamanho da célula com um dispositivo de medição separado.
- Em vez de seguir o protocolo na etapa 4.1, use um instrumento de medição de tamanho de célula comercialmente disponível para medir o tamanho médio das células na amostra. Nesse caso, nenhum dispositivo de referência é necessário. Utilizar a gota de corrente média produzida à medida que as células transitam pelo poro de dimensionamento e o diâmetro médio da célula medido para calcular De conforme descrito na secção 5.
5. Analise dados para extrair fenótipos celulares
Observação : processamento de dados pode ser executado usando o arquivo de programa de interface de linha de comando MATLAB mNPS_procJOVE.m em https://github.com/sohnlab/NPS-analysis-JOVE. Consulte Arquivo Suplementar 6 para obter mais instruções.
- Pré-processe os dados (Figura 3A).
- Calcule a corrente elétrica medida aplicando o valor de ganho usado no pré-amplificador de corrente para tensão aos dados brutos adquiridos pelo DAQ.
- Remova o ruído de alta frequência aplicando uma função de suavização retangular e/ou um filtro passa-baixas à medição de corrente bruta. Em seguida, refaça a amostra dos dados filtrados para uma taxa de amostragem mais baixa. Além disso, calcule os dados de carimbo de data/hora correspondentes a essa taxa de amostragem mais baixa.
- Calcule um sinal de corrente de linha de base ajustado aplicando um método como a suavização assimétrica de mínimos quadrados26.
- Calcule a primeira derivada aproximada (sinal de diferença) dos dados atuais pré-processados tomando a diferença entre os pontos de dados subsequentes.
- Identifique eventos celulares e extraia dados de subpulso (Figura 3B).
- Procure eventos de célula candidata examinando os dados pré-processados. Rejeite eventos celulares que se sobreponham a outros eventos celulares (ou seja, eventos de coincidência) (Figura 4 Suplementar), exiba um ajuste basal ruim ou tenha uma forma de pulso inesperada ou errônea (por exemplo, onde um entupimento pode ter estado presente no canal).
- Extraia dados de subpulso para cada evento de célula.
- Cada segmento nó-poro aparecerá como um subpulso correspondente dentro do pulso de sinal global (Figuras 1B, C). Identifique o início de cada subpulso calculando o ponto de tempo em que o sinal de diferença atinge um valor mínimo local. Identifique o final de cada subpulso calculando o ponto de tempo em que o sinal de diferença atinge um valor máximo local.
- Determine a largura de cada subpulso como o tempo decorrido entre os pontos de tempo inicial e final. Determine a amplitude de cada subpulso calculando a média da diferença entre a corrente medida e a corrente de linha de base para todos os pontos de dados entre os pontos de tempo inicial e final.
- Determine o mecanofenótipo celular para cada evento celular com base em dados de subpulso.
- Determine o diâmetro da célula d com base na equação definida por Deblois e Bean24:
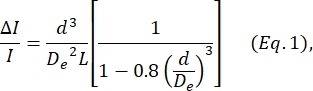
onde ΔI/I é a razão média da amplitude do subpulso para a corrente basal nos subpulsos de dimensionamento, D e é o diâmetro efetivo do canal (medido na etapa 4) e L é o comprimento total do canal nó-poro.- D e é determinado calculando-se a média ΔI/I produzida por um conjunto de partículas de diâmetro conhecido (células ou contas, ver passo 4), utilizando esse diâmetro conhecido como d, e resolvendo Eq. 1 para De.
- Quantificar a resistência da célula à deformação.
- Determine a velocidade do fluido Ufluindo calculando a velocidade média da célula nos subpulsos de dimensionamento, usando os comprimentos de segmento conhecidos e a duração medida de cada subpulso.
- Determinar o índice de deformabilidade de células inteiras (wCDI), definido por Kim et al.2 como:
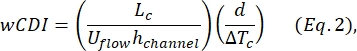
onde L c é o comprimento do segmento de contração, o canal h é a altura do canal e ΔT c é a duração do subpulso de contração.
- Identificar o tempo de recuperação da célula a partir da deformação, definido como o primeiro subpulso de recuperação com uma amplitude dentro de 8% da amplitude média do subpulso de dimensionamento2.
- Calcule a deformação transversal da célula dentro do segmento de contração.
- Calcule o diâmetro efetivo do segmento de contração (D e,c) conforme definido por Kim et al.2:
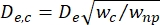 , onde w c é a largura do segmento de contração e wnp é a largura de todos os outros segmentos.
, onde w c é a largura do segmento de contração e wnp é a largura de todos os outros segmentos. - Calcule o diâmetro esférico equivalente dc da célula dentro da contração usando novamente a equação definida por Deblois e Bean24:
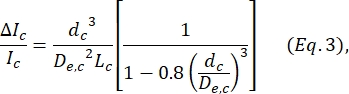
onde ΔI c/I c é a razão entre a amplitude do subpulso e a corrente basal no subpulso de contração e Lc é o comprimento do segmento de contração. - Calcule o comprimento de alongamento da célula Ldeforma conforme descrito por Kim et al.2:
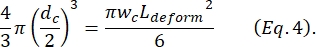
- Finalmente, calcule-se a deformação transversal da célula δdeforma, que é definida por Kim et al.2 como sendo
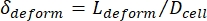 .
.
- Calcule o diâmetro efetivo do segmento de contração (D e,c) conforme definido por Kim et al.2:
- Determine o diâmetro da célula d com base na equação definida por Deblois e Bean24:
Resultados
A plataforma de mecanofenotipagem aqui apresentada é uma abordagem simples e versátil para medir as propriedades biofísicas de células individuais com rendimento moderado. As células são fluídas através do canal microfluídico (Figura 1A) usando fluxo constante impulsionado por pressão. À medida que as células transitam, o comprimento do canal microfluídico e os pulsos de corrente produzidos são registrados usando o hardware de aquisição de dados. O sinal adquirido (
Discussão
A medição das propriedades mecânicas de células individuais usando essa técnica de mecanofenotipagem consiste em três etapas: fabricação de dispositivos, aquisição de dados e análise de dados. Dentro de cada etapa, há aspectos notáveis que podem impactar significativamente os resultados experimentais. Durante a fabricação do dispositivo, geometrias de canal consistentes e uniformidade dispositivo-a-dispositivo são essenciais para resultados precisos e repetíveis. Especificamente, as paredes laterais de c...
Divulgações
L. L. S detém a patente dos EUA nº 11.383.241: "Mechano-node-pore sensing", J. Kim, S. Han e L. L. Sohn, emitida em 12 de julho de 2022.
Agradecimentos
Esta pesquisa foi apoiada por bolsas do NIBIB 1R01EB024989-01 e NCI 1R01CA190843-01. A. L. e R. R. foram apoiados por uma bolsa de pesquisa de pós-graduação da H2H8 Association. K. L. C. foi apoiado por uma National Science Foundation Graduate Research Fellowship e uma Siebel Scholar Fellowship.
Materiais
| Name | Company | Catalog Number | Comments |
| Acetone | J.T. Baker | 5356-05 | Purity (GC) ≥ 99.5% (https://us.vwr.com/store/product/6057739/acetone-99-5-vlsi-j-t-baker) |
| Aluminum Foil | n/a | n/a | |
| Analog Low-Pass Filter | ThorLabs | EF504 | ≤240 kHz Passband, Coaxial BNC Feedthrough (https://www.thorlabs.com/thorproduct.cfm?partnumber=EF504#ad-image-0) |
| Biopsy Punch | Integra Miltex | 33-31AA-P/25 | 1mm, Disposable, with Plunger (https://mms.mckesson.com/product/573313/Miltex-33-31AA-P25) |
| Blade | n/a | n/a | |
| BNC Cable | Pomona Electronics | 2249-C-12 | https://www.digikey.com/en/products/detail/pomona-electronics/2249-C-12/603323?utm_adgroup=Coaxial%20Cables%20%28RF%29&utm_source=google&utm_ medium=cpc&utm_campaign= Shopping_Product_Cable%20Assemblies_NEW&utm_term= &utm_content=Coaxial%20Cables%20%28RF%29&gclid=Cj0KCQjwlK-WBhDjARIsAO2sErQqnVJ pj5OXVObuTI8ZUf1ZeIn7zvzGnx mCWdePrG6SdEJMF3X6ubUaAs w-EALw_wcB |
| Cleanroom Polyester Swab | Thermo Fisher Scientific | 18383 | https://www.fishersci.com/shop/products/texwipe-cleantip-alpha-polyester-series-swabs-6/18383 |
| Current Preamplifier | DL Instruments | 1211 | https://www.brltest.com/index.php?main_page=product_info&products_ id=1419 |
| Custom PCB (w/ components) | n/a | n/a | see Supplemental files 4 and 5 |
| DAQ Terminal Block | National Instruments | BNC-2120 | https://www.ni.com/en-in/support/model.bnc-2120.html |
| DAQ to BNC-2110 cable | National Instruments | SHC68-68-EPM | https://www.ni.com/en-in/support/model.shc68-68-epm.html |
| Data Acquisition Board (DAQ) | National Instruments | PCI-6251 | https://www.ni.com/docs/en-US/bundle/pci-6251-feature/page/overview.html |
| Dessicator | Thermo Fisher Scientific | 5311-0250 | https://www.thermofisher.com/order/catalog/product/5311-0250 |
| Female BNC To Banana Plug Adapter | Pomona Electronics | 72909 | https://www.digikey.com/en/products/detail/pomona-electronics/72909/1196318 |
| Fetal Bovine Serum (FBS) | VWR | 89510-186 | https://us.vwr.com/store/product/18706419/avantor-seradigm-select-grade-usda-approved-origin-fetal-bovine-serum-fbs |
| Glass Cutter | Chemglass | CG-1179-21 | https://chemglass.com/plate-glass-cutters-diamond-tips |
| Gold Etchant TFA | Transene | NC0977944 | https://www.fishersci.com/shop/products/NC0977944/NC0977944 |
| Hot Plate | Thermo Fisher Scientific | SP131825 | |
| Isopropyl Alcohol | Spectrum Chemical | I1056-4LTPL | Purity (GC) ≥99.5% (https://www.spectrumchemical.com/isopropyl-alcohol-99-percent-fcc-i1056) |
| Metal Hardware Enclosure | Hammond Manufacturing | EJ12126 | https://www.digikey.com/en/products/detail/hammond-manufacturing/EJ12126/2423415 |
| Methanol | Sigma-Aldrich | 34860 | Purity (GC) ≥99.8% (https://www.sigmaaldrich.com/IN/en/substance/methanol320467561) |
| MF-321 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/mf-321/ |
| MICROPOSIT S1813 Positive Photoresist | DuPont | n/a | https://kayakuam.com/products/microposit-s1800-g2-series-photoresists/ |
| Phosphate Buffered Saline (PBS) | Thermo Fisher Scientific | 10010049 | https://www.thermofisher.com/order/catalog/product/10010049?SID=srch-hj-10010049 |
| Photomask | Fineline Imaging | n/a | Photomask are custom ordered from our CAD designs (https://www.fineline-imaging.com/) |
| Plain Glass Microscope Slide | Fisher Scientific | 12-553-5B | Material: Soda Lime, L75 x W50 mm, Thickness: 0.90–1.10 mm |
| Plasma Cleaner | Harrick Plasma | PDC-001 | https://harrickplasma.com/plasma-cleaners/expanded-plasma-cleaner/ |
| Plastic Petri Dish | Thermo Fisher Scientific | FB0875712 | 100 mm (https://www.fishersci.com/shop/products/fisherbrand-petri-dishes-clear-lid-raised-ridge-100-x-15mm/FB0875712) |
| Pressure Controller | Fluigent | MFCS-EZ | https://www.fluigent.com/research/instruments/pressure-flow-controllers/mfcs-series/ |
| Pressure Controller Software | Fluigent | MAESFLO | |
| Programming & Computation Software | MATLAB | R2021b | for data acquisition and analysis (https://www.mathworks.com/products/matlab.html) |
| PTFE Tubing | Cole Parmer | 06417-31 | 0.032" ID x 0.056" (https://www.coleparmer.com/i/masterflex-transfer-tubing-microbore-ptfe-0-032-id-x-0-056-od-100-ft-roll/0641731) |
| Scepter 2.0 Handheld Automatic Cell Counter | Millapore Sigma | PHCC20060 | https://www.sigmaaldrich.com/IN/en/product/mm/phcc20060 |
| Silicon Wafer | Wafer World | 2885 | 76.2 mm, Single Side Polished (https://www.waferworld.com/product/2885) |
| Spin Coater | n/a | n/a | |
| SU-8 3025 Negative Photoresist | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-2000/ |
| SU8 Developer | Kayaku Advanced Materials | n/a | https://kayakuam.com/products/su-8-developer/ |
| Sygard 184 Polydimethlysiloxane | Dow Chemical | 4019862 | https://www.ellsworth.com/products/by-market/consumer-products/encapsulants/silicone/dow-sylgard-184-silicone-encapsulant-clear-0.5-kg-kit/ |
| Tape | Scotch | 810-341296 | https://www.staples.com/Scotch-Magic-Tape-810-3-4-x-36-yds-1-Core/product_130567?cid=PS:GS:SBD:PLA:OS&gclid= Cj0KCQjwlK-WBhDjARIsAO 2sErRwzrrgjU0NjFkDkne1xm vT7ekS3tdzvAgiMDwPoxocgH VTQZi7vJgaAvQZEALw_wcB |
| Titanium, Platinum, Gold | n/a | n/a | |
| Triple Output Power Supply | Keysight | E36311A | https://www.newark.com/keysight-technologies/e36311a/dc-power-supply-3o-p-6v-5a-prog/dp/15AC9653 |
| UV Mask Aligner | Karl Suss America | MJB3 Mask Aligner |
Referências
- Pegoraro, A. F., Janmey, P., Weitz, D. A. Mechanical properties of the cytoskeleton and cells. Cold Spring Harbor Perspectives in Biology. 9 (11), 022038 (2017).
- Kim, J., et al. Characterizing cellular mechanical phenotypes with mechano-node-pore sensing. Microsystems & Nanoengineering. 4, 17091 (2018).
- Mierke, C. T. Bidirectional mechanical response between cells and their microenvironment. Frontiers in Physics. 9, 619 (2021).
- Kumar, S., Weaver, V. M. Mechanics, malignancy, and metastasis: The force journey of a tumor cell. Cancer and Metastasis Reviews. 28 (1), 113-127 (2009).
- Nia, H. T., Munn, L. L., Jain, R. K. Physical traits of cancer. Science. 370 (6516), (2020).
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463 (7280), 485-492 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: The role of physical interactions and mechanical forces in metastasis. Nature Reviews Cancer. 11 (7), 512-522 (2011).
- Li, B., et al. Mechanical phenotyping reveals unique biomechanical responses in retinoic acid-resistant acute promyelocytic leukemia. iScience. 25 (2), 103772 (2022).
- Kozminsky, M., Sohn, L. L. The promise of single-cell mechanophenotyping for clinical applications. Biomicrofluidics. 14 (3), 031301 (2020).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Wottawah, F., et al. Optical rheology of biological cells. Physical Review Letters. 94 (9), 1-4 (2005).
- Darling, E. M., Di Carlo, D. High-throughput assessment of cellular mechanical properties. Annual Review of Biomedical Engineering. 17 (1), 35-62 (2015).
- Carey, T. R., Cotner, K. L., Li, B., Sohn, L. L. Developments in label-free microfluidic methods for single-cell analysis and sorting. Wiley Interdisciplinary Reviews: Nanomedicine and Nanobiotechnology. 11 (1), 1529 (2019).
- Bagnall, J. S., et al. Deformability of tumor cells versus blood cells. Scientific Reports. 5, 18542 (2015).
- Byun, S., et al. Characterizing deformability and surface friction of cancer cells. Proceedings of the National Academy of Sciences. 110 (19), 7580-7585 (2013).
- Otto, O., et al. Real-time deformability cytometry: On-the-fly cell mechanical phenotyping. Nature Methods. 12 (3), 199-202 (2015).
- Gossett, D. R., et al. Hydrodynamic stretching of single cells for large population mechanical phenotyping. Proceedings of the National Academy of Sciences. 109 (20), 7630-7635 (2012).
- Guck, J., Chilvers, E. R. Mechanics meets medicine. Science Translational Medicine. 5 (212), 3-6 (2013).
- Balakrishnan, K. R., et al. Node-pore sensing: A robust, high-dynamic range method for detecting biological species. Lab on a Chip. 13 (7), 1302-1307 (2013).
- Carbonaro, A., Sohn, L. L. A resistive-pulse sensor chip for multianalyte immunoassays. Lab on a Chip. 5 (10), 1155-1160 (2005).
- Saleh, O. A., Sohn, L. L. Direct detection of antibody-antigen binding using an on-chip artificial pore. Proceedings of the National Academy of Sciences. 100 (3), 820-824 (2003).
- Saleh, O. A., Sohn, L. L. An artificial nanopore for molecular sensing. Nano Letters. 3 (1), 37-38 (2003).
- Saleh, O. A., Sohn, L. L. Quantitative sensing of nanoscale colloids using a microchip Coulter counter. Review of Scientific Instruments. 72 (12), 4449-4451 (2001).
- DeBlois, R. W., Bean, C. P. Counting and sizing of submicron particles by the resistive pulse technique. Review of Scientific Instruments. 41 (7), 909-916 (1970).
- Li, B., et al. Evaluating sources of technical variability in the mechano-node-pore sensing pipeline and their effect on the reproducibility of single-cell mechanical phenotyping. PLoS ONE. 16 (10), 0258982 (2021).
- Zhang, Z. M., Chen, S., Liang, Y. Z. Baseline correction using adaptive iteratively reweighted penalized least squares. Analyst. 135 (5), 1138-1146 (2010).
- Alibert, C., Goud, B., Manneville, J. B. Are cancer cells really softer than normal cells. Biology of the Cell. 109 (5), 167-189 (2017).
- Fujiwara, I., Zweifel, M. E., Courtemanche, N., Pollard, T. D. Latrunculin A accelerates actin filament depolymerization in addition to sequestering actin monomers. Current Biology. 28 (19), 3183-3192 (2018).
- Saleh, O. A. . A novel resistive pulse sensor for biological measurements. , (2003).
- Dokukin, M. E., Guz, N. V., Sokolov, I. Quantitative study of the elastic modulus of loosely attached cells in AFM indentation experiments. Biophysical Journal. 104 (10), 2123-2131 (2013).
- Li, Q., Lim, C. T., Goh, J. C. H., et al. Probing the elasticity of breast cancer cells using AFM. 13th International Conference on Biomedical Engineering. IFMBE Proceedings. 23, 2122-2125 (2009).
- Rother, J., et al. Atomic force microscopy-based microrheology reveals significant differences in the viscoelastic response between malign and benign cell lines. Open Biology. 4 (5), 140046 (2014).
- Li, Q., et al. AFM indentation study of breast cancer cells. Biochemical and Biophysical Research Communications. 374 (4), 609-613 (2008).
- Xu, C., et al. Elasticity measurement of breast cancer cells by atomic force microscopy. Proc. SPIE 9230. Twelfth International Conference on Photonics and Imaging in Biology and Medicine. (PIBM 2014). 92300, (2014).
- Alcaraz, J., et al. Microrheology of human lung epithelial cells measured by atomic force microscopy. Biophysical Journal. 84 (3), 2071-2079 (2003).
- Li, M., Dang, D., Liu, L., Xi, N., Wang, Y. Atomic force microscopy in characterizing cell mechanics for biomedical applications: A review. IEEE Transactions on Nanobioscience. 16 (6), 523-540 (2017).
- Urbanska, M., et al. A comparison of microfluidic methods for high-throughput cell deformability measurements. Nature Methods. 17, 587-593 (2020).
- Hill, R. T., Chilkoti, A. Surface Patterning. Biomaterials Science: An Introduction to Materials: Third Edition. , 276-301 (2013).
- Wang, Z., Volinsky, A. A., Gallant, N. D. Crosslinking effect on polydimethylsiloxane elastic modulus measured by custom-built compression instrument. Journal of Applied Polymer Science. 131 (22), 41050 (2014).
- Gibson, L. J. The hierarchical structure and mechanics of plant materials. Journal of the Royal Society Interface. 9 (76), 2749-2766 (2012).
- Stephens, A. D., Banigan, E. J., Adam, S. A., Goldman, R. D., Marko, J. F. Chromatin and lamin a determine two different mechanical response regimes of the cell nucleus. Molecular Biology of the Cell. 28 (14), 1984-1996 (2017).
- Rosenbluth, M. J., Lam, W. A., Fletcher, D. A. Force microscopy of nonadherent cells: A comparison of leukemia cell deformability. Biophysical Journal. 90 (8), 2994-3003 (2006).
- Evers, T. M. J., Holt, L. J., Alberti, S., Mashaghi, A. Reciprocal regulation of cellular mechanics and metabolism. Nature Metabolism. 3 (4), 456-468 (2021).
- Balakrishnan, K. R., et al. Node-pore sensing enables label-free surface-marker profiling of single cells. Analytical Chemistry. 87 (5), 2988-2995 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados