Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
In vitro Mapeo químico de estructuras de ADN G-cuádruple por bis-3-cloropiperidinas
En este artículo
Resumen
Las bis-3-cloroperiperidinas (B-CeP) son sondas químicas útiles para identificar y caracterizar estructuras G-cuádruples en plantillas de ADN in vitro. Este protocolo detalla el procedimiento para realizar reacciones de sondeo con B-CePs y para resolver los productos de reacción mediante electroforesis en gel de poliacrilamida de alta resolución.
Resumen
Los G-cuádruples (G4) son estructuras de ADN no canónicas biológicamente relevantes que desempeñan un papel importante en la expresión génica y las enfermedades, representando importantes dianas terapéuticas. Se requieren métodos accesibles para la caracterización in vitro del ADN dentro de posibles secuencias formadoras de G-cuádruples (PQS). Los B-CeP son una clase de agentes alquilantes que han demostrado ser sondas químicas útiles para la investigación de la estructura de orden superior de los ácidos nucleicos. En este trabajo se describe un nuevo ensayo de mapeo químico que explota la reactividad específica de los B-CeP con el N7 de las guaninas, seguido de la escisión directa de la hebra en los Gs alquilados.
Es decir, para distinguir los pliegues G4 de las formas de ADN desplegadas, utilizamos B-CeP 1 para sondear el aptámero de unión a trombina (TBA), un ADN de 15 meros capaz de asumir la disposición G4. La reacción de las guaninas que responden a B-CeP con B-CeP 1 produce productos que pueden resolverse mediante electroforesis en gel de poliacrilamida (PAGE) de alta resolución a nivel de un solo nucleótido mediante la localización de aductos de alquilación individuales y la escisión de la cadena de ADN en las guaninas alquiladas. El mapeo mediante B-CePs es una herramienta sencilla y potente para la caracterización in vitro de secuencias de ADN formadoras de G-cuádruples, lo que permite la localización precisa de guaninas implicadas en la formación de G-tétradas.
Introducción
Además de la típica doble hélice de Watson-Crick, los ácidos nucleicos pueden adoptar varias estructuras secundarias, como la forma alternativa G-quadruplex (G4), debido a sus secuencias ricas en guanina. La estructura de G4 se basa en la formación de tetrámeros planos, llamados G-tétradas, en los que cuatro guaninas interactúan a través de enlaces de hidrógeno de Hoogsteen. Las tétradas G se apilan y se estabilizan aún más mediante cationes monovalentes que se coordinan en el centro del núcleo de guanina (Figura 1)1.
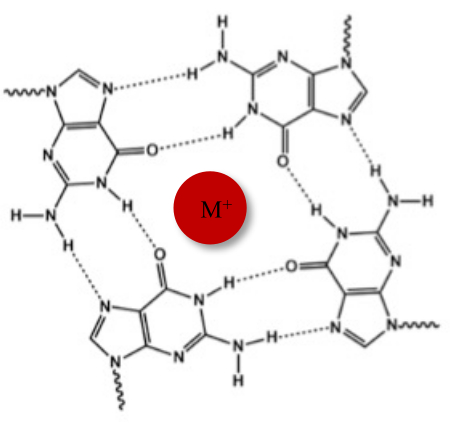
Figura 1: Representación esquemática de una estructura G-cuádruple. (A) Representación esquemática de una G-tétrada. La matriz plana se estabiliza mediante el apareamiento de bases de Hoogsteen y mediante un catión central (M+). Haga clic aquí para ver una versión más grande de esta figura.
Las secuencias con cuatro o más corridas de al menos dos nucleótidos consecutivos de guanina son secuencias potenciales de formación de G-cuádruples (PQS) que pueden plegarse en estructuras G-cuádruples. Las PQS se localizan en muchos contextos celulares diferentes, como los telómeros, los promotores de genes, el ADN ribosómico y los sitios de recombinación, y están involucradas en la regulación de muchos procesos biológicos2. Por lo tanto, la identificación y validación experimental de G4s en el genoma humano, que actualmente se realiza principalmente a través de herramientas computacionales, es un tema biológicamente relevante3. Con el fin de apoyar las predicciones computacionales o detectar estructuras G4 no predichas, aquí se muestra un método accesible basado en el mapeo químico para identificar la formación de G4 en una plantilla de ADN, que permite la identificación precisa de las guaninas que forman la estructura de la tétrada G.
El ensayo de mapeo químico reportado explota la diferente reactividad de las bis-3-cloroperiperidinas (B-CePs) con guaninas después de la formación de estructuras G4. Debido a su alta reactividad con los nucleófilos 4,5,6,7,8,9, los B-CeP son agentes alquilantes de ácidos nucleicos con la capacidad de reaccionar de manera muy eficiente con la posición N7 de los nucleótidos de guanina10. La alquilación es seguida por la desurinación y la escisión de la hebra en las construcciones de ADN monocatenario y bicatenario. Por el contrario, las guaninas involucradas en la formación de las tétradas G en los arreglos G4 son impermeables a la alquilación B-CeP, ya que la posición N7 de las guaninas está implicada en los enlaces de hidrógeno de Hoogsteen. Esta reactividad específica de los B-CeP permite no solo la detección de estructuras G4, sino también la identificación de las guaninas que forman la(s) tétrada(s), ya que se pueden deducir de su relativa protección contra la alquilación en comparación con las guaninas en ADN monocatenario y bicatenario.
El protocolo de mapeo químico se presenta utilizando B-CeP 1 (Figura 2A) como sonda para la caracterización del aptámero de unión a trombina (TBA), un ADN de 15 meros capaz de asumir la disposición G4 en presencia de cationes de potasio11,12. La disposición G4 de TBA (G4-TBA) se compara directamente con dos controles, a saber, TBA en forma monocatenaria (ssTBA) y TBA recocido a su secuencia complementaria para formar la construcción bicatenaria (dsTBA) (Tabla 1). Los productos de las reacciones de sondeo se resuelven mediante electroforesis en gel de poliacrilamida (PAGE) de alta resolución a nivel de un solo nucleótido mediante la localización de aductos de alquilación individuales y la escisión de la cadena de ADN en las guaninas alquiladas. La visualización en el gel es posible mediante la conjugación del oligonucleótido TBA con un fluoróforo en su extremo 3' (Tabla 1). Este protocolo muestra cómo plegar TBA en sus diferentes conformaciones (G4 y controles), y cómo realizar reacciones de sondaje con B-CeP seguidas de PAGE.
Protocolo
1. Preparación de ácido nucleico y sonda química
- Ácidos nucleicos
NOTA: El oligonucleótido llamado "TBA" es la secuencia de ADN de 15 meros 5'-GGT-TGG-TGT-GGT-TGG-3' marcada en el extremo 3' por el fluoróforo 5-carboxifluoresceína (FAM) para permitir la visualización en el gel. El oligonucleótido no marcado "cTBA" es su secuencia complementaria de ADN 5'-CCA-ACC-ACA-CCA-ACC-3'. TBA y cTBA se emplean para obtener las tres estructuras diferentes, como se muestra en la Tabla 1.- Puntas de autoclave y tubos de 0,5 mL para obtener desechables estériles y evitar la contaminación.
- Preparar soluciones madre solubilizando cada oligonucleótido en agua ultrapura hasta una concentración final de 100 μM. Determinar la concentración exacta de oligonucleótidos con un espectrofotómetro ultravioleta-visible (UV-Vis), utilizando el coeficiente de extinción a 260 nm proporcionado por el fabricante.
NOTA: Se utilizaron coeficientes de extinción: 164.300 M-1 cm-1 y 138.600 M-1 cm-1 para TBA y cTBA, respectivamente. - Almacenar las soluciones madre de TBA y cTBA a -20 °C (durante meses en estas condiciones).
- Compuesto B-CeP 1
NOTA: El compuesto B-CeP 1 se sintetiza como se informó anteriormente6.- Prepare la solución madre B-CeP 1 a ~10 mM. Pesar ~1 mg del compuesto liofilizado utilizando una balanza analítica ubicada en una campana extractora y solubilizarlo en dimetilsulfóxido (DMSO) al 100%.
- Calcule la concentración exacta del compuesto en función de la cantidad real de compuesto y DMSO (d = 1,1 g/cm3) utilizados.
NOTA: Manipular el compuesto con guantes en todo momento (tanto liofilizado como disuelto en DMSO)13,14.
Tabla 1: Estructuras de oligonucleótidos utilizadas en este protocolo. Haga clic aquí para descargar esta tabla.
2. Plegamiento de construcciones de ácidos nucleicos
- Preparación de tampones
- Prepare una solución tampón de BPE (ácido bifosfato-etilendiaminotetraacético [EDTA], 5x: 2 mM NaH 2 PO 4, 6 mM Na 2 HPO 4, 1 mMNa2EDTA, pH7,4) y una solución de 500 mM KCl en agua ultrapura. Filtre las soluciones a través de filtros de tamaño de poro de 0,22 μm.
NOTA: Para obtener los mejores resultados, utilice soluciones recién preparadas. El tampón BPE puede almacenarse a 4 °C durante un máximo de 15 días.
- Prepare una solución tampón de BPE (ácido bifosfato-etilendiaminotetraacético [EDTA], 5x: 2 mM NaH 2 PO 4, 6 mM Na 2 HPO 4, 1 mMNa2EDTA, pH7,4) y una solución de 500 mM KCl en agua ultrapura. Filtre las soluciones a través de filtros de tamaño de poro de 0,22 μm.
- Plegamiento de muestras G4-TBA, ssTBA y dsTBA mediante el procedimiento de repliegue térmico
NOTA: Los cationes de potasio son necesarios para plegar la estructura G4 (G4-TBA). No agregue cationes de potasio en la solución de plegado de los controles ssTBA y dsTBA.- Prepare 40 μL de una solución de 4 μM de G4-TBA en 1x BPE y 100 mM de KCl. Desnaturalice la solución de oligonucleótido G4-TBA calentando el tubo a 95 °C durante 5 min y enfríelo lentamente a temperatura ambiente (RT) para permitir que el TBA se pliegue en G-cuádruples.
- Prepare 40 μL de una solución de 4 μM de ssTBA en 1x BPE. Realice el procedimiento de repliegue térmico, como se menciona en el paso 2.2.1, para plegar TBA en su forma monocatenaria.
- Prepare 40 μL de una solución de 4 μM de dsTBA mezclando cantidades equimolares de TBA y cTBA en 1x BPE. Realice el procedimiento de repliegue térmico como se mencionó anteriormente (paso 2.2.1) para que TBA se recociera a su secuencia complementaria cTBA y formara la forma de doble cadena de TBA.
NOTA: El volumen final de cada solución de plegamiento se basa en el número de muestras para las reacciones de sondeo, considerando que se necesitarán 5 μL de 4 μM de solución para cada muestra. Prepare un poco de exceso de volumen de cada solución para evitar errores de pipeteo.
3. Reacciones de sondeo
NOTA: Las reacciones de sondeo deben realizarse inmediatamente después del procedimiento de repliegue por calor.
- Cuando las soluciones de plegado de G4-TBA, ssTBA y dsTBA se hayan enfriado a RT, realice una centrifugación de giro corto (7.000 × g durante 5-8 s a RT) e inicie las reacciones de sondeo.
- Prepare 21 tubos vacíos de 0,5 ml esterilizados en autoclave. Organícelos en tres juegos de siete tubos cada uno en el estante para muestras de laboratorio, como se informa en la Tabla 2.
NOTA: Cada conjunto de columnas corresponde a las tres condiciones de plegado de TBA diferentes G4-TBA, ssTBA y dsTBA. Cada hilera corresponde a tres tiempos de incubación diferentes. Cada celda dentro de la columna corresponde a la concentración final de la sonda B-CeP 1 (Tabla 2). Asegúrate de etiquetar claramente los tubos. - Añadir 3 μL de agua ultrapura en cada tubo.
- Añadir 5 μL de G4-TBA plegado en cada tubo del primer juego (paso 3.2). Añadir 5 μL de ssTBA plegado en cada tubo del segundo juego. Añadir 5 μL de dsTBA plegado en cada tubo del tercer juego.
- Diluir la solución madre B-CeP 1 a 250 μM y 25 μM en agua ultrapura.
NOTA: Las diluciones de la sonda química B-CeP 1 deben prepararse recién y reaccionar inmediatamente con el sustrato de ADN para evitar reacciones competitivas con el agua. - Añadir 2 μL de la dilución adecuada de B-CeP 1 (25 μM y 250 μM) a las muestras. Reemplace el compuesto con agua ultrapura en las tres muestras de control (C) para el análisis de los TBA plegados de manera diferente en ausencia del compuesto. Incubar todas las muestras a 37 °C.
- Después de 1 h, 4 h y 15 h de incubación, detenga la reacción colocando los tubos a -20 °C hasta el siguiente paso.
NOTA: Las muestras se pueden almacenar en estas condiciones durante un par de días. - Secar las muestras en una centrífuga al vacío.
NOTA: Las muestras secas pueden almacenarse a -20 °C durante semanas antes de proceder con el análisis PAGE.
Tabla 2: Muestras para las reacciones de sondeo (estructuras, concentraciones de sonda y tiempo de incubación). Cada conjunto de columnas corresponde a las tres condiciones de plegado de TBA diferentes (G4-TBA, ssTBA y dsTBA). Cada hilera corresponde a tres tiempos de incubación diferentes (1, 4, 15 h). Cada celda dentro de la columna corresponde a la concentración final de la sonda B-CeP 1 (5 o 50 μM). El control (C) para cada serie corresponde a una muestra de los TBAs de diferentes pliegues incubados durante el tiempo más largo (15 h) en ausencia de compuesto. Haga clic aquí para descargar esta tabla.
4. PÁGINA de alta resolución
- Preparación de la solución de poliacrilamida desnaturalizante
NOTA: Prepare con anticipación 500 mL de solución de gel de poliacrilamida desnaturalizante al 20%. Se utilizarán alrededor de 80 ml de esta solución para cada experimento. Use una botella de vidrio ámbar o cubra una botella de vidrio con papel de aluminio para almacenar la solución en RT.
PRECAUCIÓN: La poliacrilamida es neurotóxica. Durante todos los pasos de preparación y vertido del gel, use guantes y una bata de laboratorio. Deseche la acrilamida polimerizada en una caja apropiada para materiales contaminados.- Pesar 210 g de urea en un vaso de precipitados de 1 litro. Añadir 250 ml de solución de acrilamida/bisacrilamida al 40% (19/1) y 50 ml de TBE 10x (890 mM de Tris-HCl, 890 mM de borato, 20 mM de EDTA, pH 8).
- Coloque el vaso de precipitados en una placa de agitación y mezcle la solución con una varilla de agitación. Cubra el vaso de precipitados con papel de aluminio durante la mezcla para evitar salpicaduras y contaminación.
- Mezcle la solución hasta que la urea se disuelva por completo y la solución esté clara.
NOTA: Este paso puede tardar muchas horas. Para favorecer la disolución de la urea, añadir una pequeña alícuota de agua sin ir más allá del volumen final deseado. - Retire la varilla de agitación. Vierta la solución en un cilindro y agregue agua hasta un volumen final exacto de 500 ml.
- Instalación de aparatos de gel
- Limpie dos placas (una con muescas y otra sin muescas) con etanol al 70%, déjelas secar y luego trate las placas con una solución de dimetildiclorosilano.
NOTA: La silanización se puede omitir, aunque ayuda a la liberación del gel de una de las placas cuando se desmonta el sándwich de gel.
PRECAUCIÓN: Manipule la solución de silanización con guantes y realice el tratamiento de la placa con esta solución en una campana extractora. - Coloque los espaciadores de 0,4 mm a lo largo de los bordes largos de la placa más larga, coloque la placa corta encima de la otra y alinee las dos placas en la parte inferior.
- Coloque varias capas de cinta adhesiva a lo largo de todos los bordes, excepto en la parte superior.
- Para evitar fugas durante la fundición, agregue una capa adicional de cinta adhesiva en la parte inferior del gel.
- Sujete los lados del sándwich de vidrio con abrazaderas limpias siguiendo las instrucciones del proveedor (diferentes proveedores usan aparatos, abrazaderas sándwich y juntas ligeramente diferentes).
- Limpie dos placas (una con muescas y otra sin muescas) con etanol al 70%, déjelas secar y luego trate las placas con una solución de dimetildiclorosilano.
- Verter el gel
NOTA: Verter el gel a RT (25 °C), ya que la polimerización por poliacrilamida es sensible a la temperatura.- En un vaso de precipitados, verter 80 ml de la solución de poliacrilamida desnaturalizante previamente preparada (paso 4.1), 450 μl de una solución de persulfato de amonio (APS) al 10 % m/V y 45 μl de tetrametiletilendiamina (TEMED) inmediatamente antes de usar.
- Mezcle la solución y viértala rápidamente entre las placas de vidrio con una jeringa de 50 ml. Introducir el peine con el número deseado de pocillos entre las placas de vidrio, evitando burbujas. Agregue una solución de gel para llenar el sándwich por completo, si es necesario. Coloque cuatro abrazaderas en el peine para presionar hacia abajo y permitir una distribución uniforme de los pocillos.
- Deje que el gel polimerice durante al menos 45 minutos.
- Ejecutar el gel
- Después de la polimerización, retire todas las abrazaderas y las capas de cinta de papel. Retire el peine lentamente y enjuague bien los pozos con agua destilada.
- Siga las instrucciones del proveedor específico para colocar correctamente el sándwich de gel en el aparato de electroforesis de gel vertical.
- Prepare el tampón de funcionamiento TBE (1x: 89 mM de Tris-HCl, 89 mM de borato, 2 mM de EDTA, pH 8) en agua desionizada y llene los depósitos superior e inferior con el tampón.
- Calentar las placas realizando una ejecución previa de la electroforesis en gel durante al menos 30 minutos a 50 W.
- Preparar tampón de carga de gel desnaturalizante (DGLB: 1 M Tris-HCl, 80% formamida, 50% glicerol, 0,05% azul de bromofenol) en agua ultrapura.
NOTA: GLB ayuda a rastrear el movimiento de las muestras de oligonucleótidos en el sistema de gel y permite cargar las muestras en los pocillos del gel. La presencia del agente desnaturalizante formamida permite que las especies de ADN se separen según el tamaño, incluso en una página no desnaturalizante. - Vuelva a suspender las muestras secas (muestras del paso 3.8) en 5 μl de DGLB.
- Antes de cargar las muestras, limpie los pocillos con una jeringa pequeña y el tampón TBE en la cámara tampón superior para eliminar la urea de los pocillos.
NOTA: Repita este paso varias veces para limpiar con precisión los pocillos, evitando así bandas difíciles de interpretar. - Cargue las muestras en los pozos limpios y tome nota del orden de carga.
- Ejecute la electroforesis en gel durante 2 h a 50 W, o al menos hasta que el tinte azul de bromofenol haya corrido 2/3 por el gel.
- Obtención de imágenes del gel
- Después de la electroforesis, apague la fuente de alimentación, retire el sándwich de vidrio y limpie los vasos.
- Detecte la fluorescencia de las bandas de oligonucleótidos marcadas con FAM mediante el escaneo con un generador de imágenes en gel.
Resultados
La Figura 2 muestra un resultado representativo de un ensayo de mapeo químico realizado, como se describe en el protocolo con B-CeP 1 sobre el oligonucleótido TBA plegado en tres estructuras diferentes. La disposición G-cuádruple de TBA (G4-TBA) se obtuvo mediante el plegamiento del oligonucleótido en BPE y en presencia del catión K+, mientras que la forma monocatenaria de la misma secuencia de TBA (ssTBA) se plegó en ausencia de potasio. La construcción bicatenaria (dsTBA...
Discusión
Los G-cuádruples son estructuras secundarias de ácidos nucleicos que normalmente se pliegan dentro de secuencias de ADN ricas en guanina, y son importantes objetivos de investigación debido a su asociación con el control genético y las enfermedades. El mapeo químico por B-CePs es un protocolo útil para la caracterización de los G4 de ADN, que se puede utilizar para identificar las bases de guanina involucradas en la formación de G-tétradas en condiciones fisiológicas de sal.
La sond...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Este trabajo contó con el apoyo del Departamento de Ciencias Farmacéuticas y Farmacológicas de la Universidad de Padua (PRIDJ-BIRD2019).
Materiales
| Name | Company | Catalog Number | Comments |
| Acrylamide/bis-acrylamide solution 40% | Applichem | A3658 | R45-46-20/21-25-36/38-43-48/23/ 24/25-62 |
| Ammonium per-sulfate (APS) | Sigma Aldrich | A7460 | |
| Analytical balance | Mettler Toledo | ||
| Autoclave | pbi international | ||
| Boric acid | Sigma Aldrich | B0252 | |
| Bromophenol blue Brilliant blue R | Sigma Aldrich | B0149 | |
| di-Sodium hydrogen phosphate dodecahydrate | Fluka | 71649 | |
| DMSO | Sigma Aldrich | 276855 | |
| DNA oligonucleotides | Integrated DNA Technologies | synthesis of custom sequences | |
| EDTA disodium | Sigma Aldrich | E5134 | |
| Formamide | Fluka | 40248 | H351-360D-373 |
| Gel imager | GE Healtcare | STORM B40 | |
| Glycerol | Sigma Aldrich | G5516 | |
| Micro tubes 0.5 mL | Sarstedt | 72.704 | |
| Potassium Chloride | Sigma Aldrich | P9541 | |
| Sequencing apparatus | Biometra | Model S2 | |
| Silanization solution I | Fluka | 85126 | H225, 314, 318, 336, 304, 400, 410 |
| Sodium phosphate monobasic | Carlo Erba | 480086 | |
| Speedvac concentrator | Thermo Scientific | Savant DNA 120 | |
| TEMED | Fluka | 87689 | R11-21/22-23-34 |
| Tris-HCl | MERCK | 1.08387.2500 | |
| Urea | Sigma Aldrich | 51456 | |
| UV-Vis spectrophotometer | Thermo Scientific | Nanodrop 1000 |
Referencias
- Davis, J. T. G-quartets 40 years later: from 5'-GMP to molecular biology and supramolecular chemistry. Angewandte Chemie. 43 (6), 668-698 (2004).
- Varshney, D., Spiegel, J., Zyner, K., Tannahill, D., Balasubramanian, S. The regulation and functions of DNA and RNA G-quadruplexes. Nature Reviews Molecular Cell Biology. 21 (8), 459-474 (2020).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nature Biotechnology. 33 (8), 877-881 (2015).
- Zuravka, I., Sosic, A., Gatto, B., Gottlich, R. Synthesis and evaluation of a bis-3-chloropiperidine derivative incorporating an anthraquinone pharmacophore. Bioorganic & Medicinal Chemistry Letters. 25 (20), 4606-4609 (2015).
- Zuravka, I., Roesmann, R., Sosic, A., Gottlich, R., Gatto, B. Bis-3-chloropiperidines containing bridging lysine linkers: Influence of side chain structure on DNA alkylating activity. Bioorganic & Medicinal Chemistry. 23 (6), 1241-1250 (2015).
- Zuravka, I., et al. Synthesis and DNA cleavage activity of bis-3-chloropiperidines as alkylating agents. ChemMedChem. 9 (9), 2178-2185 (2014).
- Sosic, A., Gottlich, R., Fabris, D., Gatto, B. B-CePs as cross-linking probes for the investigation of RNA higher-order structure. Nucleic Acids Research. 49 (12), 6660-6672 (2021).
- Sosic, A., et al. Bis-3-chloropiperidines targeting TAR RNA as a novel strategy to impair the HIV-1 nucleocapsid protein. Molecules. 26 (7), 1874 (2021).
- Sosic, A., et al. In vitro evaluation of bis-3-chloropiperidines as RNA modulators targeting TAR and TAR-protein interaction. International Journal of Molecular Sciences. 23 (2), 582 (2022).
- Sosic, A., et al. Direct and topoisomerase II mediated DNA damage by bis-3-chloropiperidines: The importance of being an earnest G. ChemMedChem. 12 (17), 1471-1479 (2017).
- Bock, L. C., Griffin, L. C., Latham, J. A., Vermaas, E. H., Toole, J. J. Selection of single-stranded DNA molecules that bind and inhibit human thrombin. Nature. 355 (6360), 564-566 (1992).
- Paborsky, L. R., McCurdy, S. N., Griffin, L. C., Toole, J. J., Leung, L. L. The single-stranded DNA aptamer-binding site of human thrombin. The Journal of Biological Chemistry. 268 (28), 20808-20811 (1993).
- Carraro, C., et al. Behind the mirror: chirality tunes the reactivity and cytotoxicity of chloropiperidines as potential anticancer agents. ACS Medicinal Chemistry Letters. 10 (4), 552-557 (2019).
- Carraro, C., et al. Appended aromatic moieties in flexible bis-3-chloropiperidines confer tropism against pancreatic cancer cells. ChemMedChem. 16 (5), 860-868 (2021).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Onel, B., Wu, G., Sun, D., Lin, C., Yang, D. Electrophoretic mobility shift assay and dimethyl sulfate footprinting for characterization of G-quadruplexes and G-quadruplex-protein complexes. Methods in Molecular Biology. 2035, 201-222 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados