È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
In vitro Mappatura chimica delle strutture del DNA G-quadruplex mediante bis-3-cloroperidine
In questo articolo
Riepilogo
Le bis-3-cloropepidine (B-CePs) sono sonde chimiche utili per identificare e caratterizzare le strutture G-quadruplex in modelli di DNA in vitro. Questo protocollo descrive in dettaglio la procedura per eseguire reazioni di sondaggio con B-CeP e per risolvere i prodotti di reazione mediante elettroforesi su gel di poliacrilammide ad alta risoluzione.
Abstract
I G-quadruplex (G4) sono strutture di DNA biologicamente rilevanti e non canoniche che svolgono un ruolo importante nell'espressione genica e nelle malattie, rappresentando bersagli terapeutici significativi. Sono necessari metodi accessibili per la caratterizzazione in vitro del DNA all'interno di potenziali sequenze formanti G-quadruplex (PQS). I B-CeP sono una classe di agenti alchilanti che si sono dimostrati utili sonde chimiche per lo studio della struttura di ordine superiore degli acidi nucleici. Questo articolo descrive un nuovo saggio di mappatura chimica che sfrutta la reattività specifica dei B-CeP con l'N7 delle guanine, seguita da una scissione diretta del filamento ai G alchilati.
Vale a dire, per distinguere le pieghe G4 dalle forme di DNA non ripiegate, usiamo B-CeP 1 per sondare l'aptamero legante la trombina (TBA), un DNA a 15 mer in grado di assumere la disposizione G4. La reazione delle guaine che rispondono a B-CeP con B-CeP 1 produce prodotti che possono essere risolti mediante elettroforesi su gel di poliacrilammide (PAGE) ad alta risoluzione a livello di singolo nucleotide localizzando i singoli addotti di alchilazione e la scissione del filamento di DNA sulle guanine alchilate. La mappatura mediante B-CeP è uno strumento semplice e potente per la caratterizzazione in vitro di sequenze di DNA formanti G-quadruplex, consentendo la localizzazione precisa delle guanine coinvolte nella formazione delle G-tetrade.
Introduzione
Oltre alla tipica doppia elica di Watson-Crick, gli acidi nucleici possono adottare varie strutture secondarie, come la forma alternativa G-quadruplex (G4), a causa delle loro sequenze ricche di guanina. La struttura G4 si basa sulla formazione di tetrameri planari, chiamati G-tetrade, in cui quattro guanine interagiscono attraverso legami idrogeno di Hoogsteen. Le G-tetradi sono impilate e ulteriormente stabilizzate da cationi monovalenti coordinati al centro del nucleo guaninico (Figura 1)1.
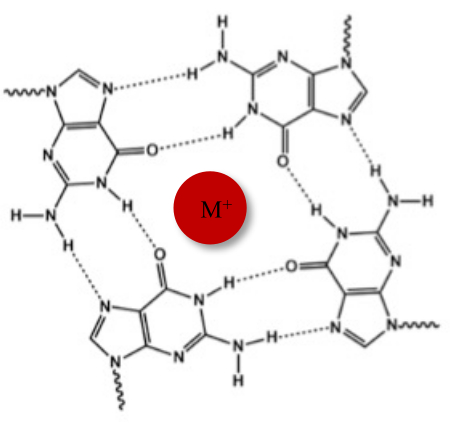
Figura 1: Rappresentazione schematica di una struttura G-quadruplex. (A) Rappresentazione schematica di una G-tetrade. L'array planare è stabilizzato dall'appaiamento di basi di Hoogsteen e da un catione centrale (M+). Fare clic qui per visualizzare una versione più grande di questa figura.
Le sequenze con quattro o più cicli di almeno due nucleotidi guanina consecutivi sono potenziali sequenze formanti G-quadruplex (PQS) che possono ripiegarsi in strutture G-quadruplex. Le PQS sono localizzate in molti contesti cellulari diversi, come i telomeri, i promotori genici, il DNA ribosomiale e i siti di ricombinazione, e sono coinvolte nella regolazione di molti processi biologici2. Quindi, l'identificazione e la validazione sperimentale di G4 nel genoma umano, che attualmente viene eseguita principalmente attraverso strumenti computazionali, è una questione biologicamente rilevante3. Al fine di supportare le previsioni computazionali o rilevare strutture G4 non previste, viene mostrato un metodo accessibile basato sulla mappatura chimica per identificare la formazione di G4 in un modello di DNA, che consente l'identificazione precisa delle guanine che formano la struttura G-tetrade.
Il saggio di mappatura chimica riportato sfrutta la diversa reattività delle bis-3-cloropiperidine (B-CePs) con le guanine in seguito alla formazione di strutture G4. A causa della loro elevata reattività con i nucleofili 4,5,6,7,8,9, i B-CeP sono agenti alchilanti dell'acido nucleico con la capacità di reagire in modo molto efficiente con la posizione N7 dei nucleotidi guanina10. L'alchilazione è seguita dalla depurazione e dalla scissione del filamento nei costrutti di DNA a singolo e doppio filamento. Al contrario, le guaine coinvolte nella formazione delle G-tetradi nelle disposizioni G4 sono impermeabili all'alchilazione B-CeP, poiché la posizione N7 delle guanine è implicata nei legami idrogeno di Hoogsteen. Questa specifica reattività dei B-CeP permette non solo la rilevazione delle strutture G4, ma anche l'identificazione delle guanine che formano la/e tetrade/e, come si può dedurre dalla loro relativa protezione dall'alchilazione rispetto alle guanine nel DNA a singolo e doppio filamento.
Il protocollo di mappatura chimica è riportato qui utilizzando B-CeP 1 (Figura 2A) come sonda per la caratterizzazione dell'aptamero legante la trombina (TBA), un DNA a 15 meri in grado di assumere la disposizione G4 in presenza di cationi di potassio11,12. La disposizione G4 di TBA (G4-TBA) è direttamente confrontata con due controlli, vale a dire TBA nella forma a singolo filamento (ssTBA) e TBA ricotto alla sua sequenza complementare per formare il costrutto a doppio filamento (dsTBA) (Tabella 1). I prodotti delle reazioni di sondaggio vengono risolti mediante elettroforesi su gel di poliacrilammide (PAGE) ad alta risoluzione a livello di singolo nucleotide localizzando i singoli addotti di alchilazione e la scissione del filamento di DNA sulle guanine alchilate. La visualizzazione sul gel è resa possibile dalla coniugazione dell'oligonucleotide TBA con un fluoroforo alla sua estremità 3' (Tabella 1). Questo protocollo mostra come ripiegare TBA nelle sue diverse conformazioni (G4 e controlli) e come eseguire reazioni di sondaggio con B-CeP seguite da PAGE.
Protocollo
1. Preparazione dell'acido nucleico e della sonda chimica
- Acidi nucleici
NOTA: L'oligonucleotide denominato "TBA" è la sequenza di DNA a 15 meri 5'-GGT-TGG-TGT-GGT-TGG-3' marcata all'estremità 3' dal fluoroforo 5-carbossifluoresceina (FAM) per consentire la visualizzazione sul gel. L'oligonucleotide non marcato "cTBA" è la sua sequenza complementare al DNA 5'-CCA-ACC-ACA-CCA-ACC-3'. TBA e cTBA sono impiegati per ottenere le tre diverse strutture, come mostrato nella Tabella 1.- Puntali per autoclave e provette da 0,5 mL per ottenere prodotti monouso sterili ed evitare contaminazioni.
- Preparare soluzioni madre solubilizzando ciascun oligonucleotide in acqua ultrapura fino a una concentrazione finale di 100 μM. Determinare l'esatta concentrazione di oligonucleotidi con uno spettrofotometro UV-visibile (UV-Vis), utilizzando il coefficiente di estinzione a 260 nm fornito dal produttore.
NOTA: I coefficienti di estinzione: 164.300 M-1 cm-1 e 138.600 M-1 cm-1 sono stati utilizzati rispettivamente per TBA e cTBA. - Conservare le soluzioni madre TBA e cTBA a -20 °C (per mesi in queste condizioni).
- Mescola B-CeP 1
NOTA: Il composto B-CeP 1 è sintetizzato come riportato in precedenza6.- Preparare la soluzione madre B-CeP 1 a ~10 mM. Pesare ~1 mg del composto liofilizzato utilizzando una bilancia analitica situata in una cappa aspirante e solubilizzarlo in dimetilsolfossido (DMSO) al 100%.
- Calcolare l'esatta concentrazione del composto in base alla quantità effettiva di composto e DMSO (d = 1,1 g/cm3) utilizzati.
NOTA: Maneggiare sempre il composto con i guanti (sia quando liofilizzato che quando disciolto in DMSO)13,14.
Tabella 1: Strutture oligonucleotidiche utilizzate in questo protocollo. Clicca qui per scaricare questa tabella.
2. Folding dei costrutti degli acidi nucleici
- Preparazione dei tamponi
- Preparare una soluzione tampone BPE (acido bifosfato-etilendiamminotetraacetico [EDTA], 5x: 2 mM NaH 2 PO 4, 6mM Na 2 HPO 4, 1 mM Na2EDTA,pH7,4) e una soluzione di 500 mM KCl in acqua ultrapura. Filtrare le soluzioni attraverso filtri con pori da 0,22 μm.
NOTA: Per ottenere i migliori risultati, utilizzare soluzioni appena preparate. Il tampone BPE può essere conservato a 4 °C per un massimo di 15 giorni.
- Preparare una soluzione tampone BPE (acido bifosfato-etilendiamminotetraacetico [EDTA], 5x: 2 mM NaH 2 PO 4, 6mM Na 2 HPO 4, 1 mM Na2EDTA,pH7,4) e una soluzione di 500 mM KCl in acqua ultrapura. Filtrare le soluzioni attraverso filtri con pori da 0,22 μm.
- Piegatura di campioni G4-TBA, ssTBA e dsTBA mediante la procedura di ripiegamento termico
NOTA: I cationi di potassio sono necessari per ripiegare la struttura G4 (G4-TBA). Non aggiungere cationi di potassio nella soluzione pieghevole dei controlli ssTBA e dsTBA.- Preparare 40 μL di una soluzione da 4 μM di G4-TBA in 1x BPE e 100 mM KCl. Denaturare la soluzione oligonucleotidica G4-TBA riscaldando la provetta a 95 °C per 5 minuti e raffreddarla lentamente a temperatura ambiente (RT) per consentire al TBA di ripiegarsi in G-quadruplex.
- Preparare 40 μL di una soluzione da 4 μM di ssTBA in 1x BPE. Eseguire la procedura di ripiegamento termico, come indicato al punto 2.2.1, per piegare TBA nella sua forma a filo singolo.
- Preparare 40 μL di una soluzione da 4 μM di dsTBA mescolando quantità equimolari di TBA e cTBA in 1x BPE. Eseguire la procedura di ripiegamento termico come indicato sopra (passaggio 2.2.1) affinché TBA si ritempri nella sua sequenza complementare cTBA e formi la forma a doppio filamento di TBA.
NOTA: Il volume finale di ciascuna soluzione di ripiegamento si basa sul numero di campioni per le reazioni di sonda, considerando che saranno necessari 5 μL di soluzione da 4 μM per ciascun campione. Preparare un po' di volume in eccesso di ogni soluzione per evitare errori di pipettaggio.
3. Reazioni di sondaggio
NOTA: Le reazioni di tastatura devono essere eseguite immediatamente dopo la procedura di ripiegamento termico.
- Quando le soluzioni di ripiegamento di G4-TBA, ssTBA e dsTBA si sono raffreddate a RT, eseguire una centrifugazione a rotazione breve (7.000 × g per 5-8 s a RT) e avviare le reazioni di sondaggio.
- Preparare 21 provette vuote da 0,5 mL sterilizzate in autoclave. Organizzarle in tre serie di sette provette ciascuna nel rack per i campioni di laboratorio, come riportato nella Tabella 2.
NOTA: Ogni set di colonne corrisponde alle tre diverse condizioni di riduzione TBA G4-TBA, ssTBA e dsTBA. Ogni fila corrisponde a tre diversi tempi di incubazione. Ogni cella all'interno della colonna corrisponde alla concentrazione finale della sonda B-CeP 1 (Tabella 2). Assicurati di etichettare chiaramente i tubi. - Aggiungere 3 μL di acqua ultrapura in ogni provetta.
- Aggiungere 5 μL di G4-TBA piegato in ciascuna provetta del primo set (passaggio 3.2). Aggiungere 5 μL di ssTBA piegato in ciascuna provetta del secondo set. Aggiungere 5 μL di dsTBA piegato in ciascuna provetta del terzo set.
- Diluire la soluzione madre B-CeP 1 a 250 μM e 25 μM in acqua ultrapura.
NOTA: Le diluizioni della sonda chimica B-CeP 1 devono essere preparate al momento e reagite immediatamente con il substrato di DNA per evitare reazioni concorrenti con l'acqua. - Aggiungere 2 μL della diluizione B-CeP 1 appropriata (25 μM e 250 μM) ai campioni. Sostituire il composto con acqua ultrapura nei tre campioni di controllo (C) per l'analisi dei TBA diversamente ripiegati in assenza del composto. Incubare tutti i campioni a 37 °C.
- Dopo 1 h, 4 h e 15 h di incubazione, arrestare la reazione ponendo le provette a -20 °C fino alla fase successiva.
NOTA: I campioni possono essere conservati in queste condizioni per un paio di giorni. - Essiccare i campioni in una centrifuga sottovuoto.
NOTA: I campioni essiccati possono essere conservati a -20 °C per settimane prima di procedere con l'analisi PAGE.
Tabella 2: Campioni per le reazioni di sondaggio (strutture, concentrazioni della sonda e tempo di incubazione). Ogni set di colonne corrisponde alle tre diverse condizioni di riduzione TBA (G4-TBA, ssTBA e dsTBA). Ogni fila corrisponde a tre diversi tempi di incubazione (1, 4, 15 h). Ogni cella all'interno della colonna corrisponde alla concentrazione finale della sonda B-CeP 1 (5 o 50 μM). Il controllo (C) per ogni set corrisponde a un campione di TBA diversamente ripiegati incubati per il tempo più lungo (15 h) in assenza di composto. Clicca qui per scaricare questa tabella.
4. PAGINA ad alta risoluzione
- Preparazione della soluzione denaturante di poliacrilammide
NOTA: Preparare in anticipo 500 mL di soluzione in gel di poliacrilammide denaturante al 20%. Per ogni esperimento verranno utilizzati circa 80 ml di questa soluzione. Utilizzare un flacone di vetro ambrato o coprire un flacone di vetro con un foglio di alluminio per conservare la soluzione a RT.
ATTENZIONE: La poliacrilammide è neurotossica. Durante tutte le fasi di preparazione e versamento del gel, indossare guanti e camice da laboratorio. Scartare l'acrilammide polimerizzata in una scatola appropriata per i materiali contaminati.- Pesare 210 g di urea in un becher da 1 L. Aggiungere 250 mL di soluzione di acrilammide/bisacrilammide (19/1) al 40% e 50 mL di TBE 10x (890 mM Tris-HCl, 890 mM borato, 20 mM EDTA, pH 8).
- Posizionare il becher su un piatto per mescolare e mescolare la soluzione con una bacchetta per mescolare. Coprire il becher con un foglio di alluminio durante la miscelazione per evitare schizzi e contaminazioni.
- Mescolare la soluzione fino a quando l'urea non è completamente disciolta e la soluzione è limpida.
NOTA: questo passaggio può richiedere molte ore. Per favorire lo scioglimento dell'urea, aggiungere una piccola aliquota d'acqua senza superare il volume finale desiderato. - Rimuovere l'asta di agitazione. Versare la soluzione in un cilindro e aggiungere acqua fino a un volume finale esatto di 500 ml.
- Messa a punto dell'apparecchio gel
- Pulire due piastre (una dentellata e una non dentellata) con etanolo al 70%, lasciarle asciugare, quindi trattare le piastre con una soluzione di dimetildiclorosilano.
NOTA: La silanizzazione può essere saltata, anche se aiuta il rilascio del gel da una delle piastre quando il sandwich di gel viene smontato.
ATTENZIONE: Maneggiare la soluzione di silanizzazione con i guanti ed eseguire il trattamento della piastra con questa soluzione in una cappa aspirante. - Posizionare i distanziatori da 0.4 mm lungo i bordi lunghi della piastra più lunga, posizionare la piastra corta sopra l'altra e allineare le due piastre in basso.
- Posiziona più strati di nastro di carta lungo tutti i bordi tranne la parte superiore.
- Per evitare perdite durante la colata, aggiungere un ulteriore strato di nastro adesivo sul fondo del gel.
- Agganciare i lati del sandwich di vetro con morsetti puliti seguendo le istruzioni del fornitore (diversi fornitori utilizzano apparecchi, morsetti a sandwich e guarnizioni leggermente diversi).
- Pulire due piastre (una dentellata e una non dentellata) con etanolo al 70%, lasciarle asciugare, quindi trattare le piastre con una soluzione di dimetildiclorosilano.
- Versare il gel
NOTA: Versare il gel a RT (25 °C), poiché la polimerizzazione del poliacrilammide è sensibile alla temperatura.- In un becher, versare 80 mL della soluzione denaturante di poliacrilammide precedentemente preparata (fase 4.1), 450 μL di una soluzione di persolfato di ammonio (APS) al 10% m/V e 45 μL di tetrametiletilendiammina (TEMED) immediatamente prima dell'uso.
- Mescolare la soluzione e versarla rapidamente tra le lastre di vetro utilizzando una siringa da 50 ml. Introdurre il pettine con il numero desiderato di pozzetti tra le lastre di vetro, evitando la formazione di bolle. Aggiungere una soluzione di gel per riempire completamente il panino, se necessario. Posizionare quattro morsetti sul pettine per premere verso il basso e consentire una distribuzione uniforme dei pozzetti.
- Lasciare polimerizzare il gel per almeno 45 min.
- Esecuzione del gel
- Dopo la polimerizzazione, rimuovere tutte le pinze e gli strati di nastro di carta. Rimuovere lentamente il pettine e sciacquare accuratamente i pozzetti con acqua distillata.
- Seguire le istruzioni del fornitore specifico per posizionare correttamente il sandwich di gel nell'apparecchio di elettroforesi verticale su gel.
- Preparare il tampone di funzionamento TBE (1x: 89 mM Tris-HCl, 89 mM borato, 2 mM EDTA, pH 8) in acqua deionizzata e riempire entrambi i serbatoi superiore e inferiore con il tampone.
- Riscaldare le piastre eseguendo una pre-corsa dell'elettroforesi su gel per almeno 30 minuti a 50 W.
- Preparare il tampone di caricamento del gel denaturante (DGLB: 1 M Tris-HCl, 80% formammide, 50% glicerolo, 0,05% blu di bromofenolo) in acqua ultrapura.
NOTA: GLB aiuta a tracciare il movimento dei campioni di oligonucleotidi nel sistema del gel e consente di caricare i campioni nei pozzetti del gel. La presenza dell'agente denaturante formammide permette alle specie di DNA di separarsi in base alle dimensioni, anche in una PAGE non denaturante. - Risospendere i campioni essiccati (campioni della fase 3.8) in 5 μL di DGLB.
- Prima di caricare i campioni, pulire i pozzetti utilizzando una piccola siringa e il tampone TBE nella camera tampone superiore per rimuovere l'urea dai pozzetti.
NOTA: Ripetere questo passaggio più volte per pulire accuratamente i pozzetti, evitando così bande difficili da interpretare. - Caricare i campioni nei pozzetti puliti e prendere nota dell'ordine di caricamento.
- Eseguire l'elettroforesi del gel per 2 ore a 50 W, o almeno fino a quando il colorante blu di bromofenolo non è colato per 2/3 lungo il gel.
- Imaging del gel
- Dopo l'elettroforesi, spegnere l'alimentazione, rimuovere il sandwich di vetro e pulire i bicchieri.
- Rilevare la fluorescenza delle bande oligonucleotidiche marcate con FAM mediante scansione utilizzando un gel imager.
Risultati
La Figura 2 mostra un risultato rappresentativo di un saggio di mappatura chimica eseguito, come descritto nel protocollo con B-CeP 1 sull'oligonucleotide TBA ripiegato in tre diverse strutture. La disposizione G-quadruplex di TBA (G4-TBA) è stata ottenuta ripiegando l'oligonucleotide in BPE e in presenza del catione K+, mentre la forma a singolo filamento della stessa sequenza TBA (ssTBA) è stata ripiegata in assenza di potassio. Il costrutto a doppio filamento (dsTBA) è stato...
Discussione
I G-quadruplex sono strutture secondarie di acido nucleico che tipicamente si ripiegano all'interno di sequenze di DNA ricche di guanina e sono obiettivi di ricerca significativi a causa della loro associazione con il controllo genetico e le malattie. La mappatura chimica mediante B-CePs è un protocollo utile per la caratterizzazione del DNA G4s, che può essere utilizzato per identificare le basi guaninica coinvolte nella formazione delle G-tetrade in condizioni fisiologiche di sale.
La sond...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dal Dipartimento di Scienze Farmaceutiche e Farmacologiche dell'Università di Padova (PRIDJ-BIRD2019).
Materiali
| Name | Company | Catalog Number | Comments |
| Acrylamide/bis-acrylamide solution 40% | Applichem | A3658 | R45-46-20/21-25-36/38-43-48/23/ 24/25-62 |
| Ammonium per-sulfate (APS) | Sigma Aldrich | A7460 | |
| Analytical balance | Mettler Toledo | ||
| Autoclave | pbi international | ||
| Boric acid | Sigma Aldrich | B0252 | |
| Bromophenol blue Brilliant blue R | Sigma Aldrich | B0149 | |
| di-Sodium hydrogen phosphate dodecahydrate | Fluka | 71649 | |
| DMSO | Sigma Aldrich | 276855 | |
| DNA oligonucleotides | Integrated DNA Technologies | synthesis of custom sequences | |
| EDTA disodium | Sigma Aldrich | E5134 | |
| Formamide | Fluka | 40248 | H351-360D-373 |
| Gel imager | GE Healtcare | STORM B40 | |
| Glycerol | Sigma Aldrich | G5516 | |
| Micro tubes 0.5 mL | Sarstedt | 72.704 | |
| Potassium Chloride | Sigma Aldrich | P9541 | |
| Sequencing apparatus | Biometra | Model S2 | |
| Silanization solution I | Fluka | 85126 | H225, 314, 318, 336, 304, 400, 410 |
| Sodium phosphate monobasic | Carlo Erba | 480086 | |
| Speedvac concentrator | Thermo Scientific | Savant DNA 120 | |
| TEMED | Fluka | 87689 | R11-21/22-23-34 |
| Tris-HCl | MERCK | 1.08387.2500 | |
| Urea | Sigma Aldrich | 51456 | |
| UV-Vis spectrophotometer | Thermo Scientific | Nanodrop 1000 |
Riferimenti
- Davis, J. T. G-quartets 40 years later: from 5'-GMP to molecular biology and supramolecular chemistry. Angewandte Chemie. 43 (6), 668-698 (2004).
- Varshney, D., Spiegel, J., Zyner, K., Tannahill, D., Balasubramanian, S. The regulation and functions of DNA and RNA G-quadruplexes. Nature Reviews Molecular Cell Biology. 21 (8), 459-474 (2020).
- Chambers, V. S., et al. High-throughput sequencing of DNA G-quadruplex structures in the human genome. Nature Biotechnology. 33 (8), 877-881 (2015).
- Zuravka, I., Sosic, A., Gatto, B., Gottlich, R. Synthesis and evaluation of a bis-3-chloropiperidine derivative incorporating an anthraquinone pharmacophore. Bioorganic & Medicinal Chemistry Letters. 25 (20), 4606-4609 (2015).
- Zuravka, I., Roesmann, R., Sosic, A., Gottlich, R., Gatto, B. Bis-3-chloropiperidines containing bridging lysine linkers: Influence of side chain structure on DNA alkylating activity. Bioorganic & Medicinal Chemistry. 23 (6), 1241-1250 (2015).
- Zuravka, I., et al. Synthesis and DNA cleavage activity of bis-3-chloropiperidines as alkylating agents. ChemMedChem. 9 (9), 2178-2185 (2014).
- Sosic, A., Gottlich, R., Fabris, D., Gatto, B. B-CePs as cross-linking probes for the investigation of RNA higher-order structure. Nucleic Acids Research. 49 (12), 6660-6672 (2021).
- Sosic, A., et al. Bis-3-chloropiperidines targeting TAR RNA as a novel strategy to impair the HIV-1 nucleocapsid protein. Molecules. 26 (7), 1874 (2021).
- Sosic, A., et al. In vitro evaluation of bis-3-chloropiperidines as RNA modulators targeting TAR and TAR-protein interaction. International Journal of Molecular Sciences. 23 (2), 582 (2022).
- Sosic, A., et al. Direct and topoisomerase II mediated DNA damage by bis-3-chloropiperidines: The importance of being an earnest G. ChemMedChem. 12 (17), 1471-1479 (2017).
- Bock, L. C., Griffin, L. C., Latham, J. A., Vermaas, E. H., Toole, J. J. Selection of single-stranded DNA molecules that bind and inhibit human thrombin. Nature. 355 (6360), 564-566 (1992).
- Paborsky, L. R., McCurdy, S. N., Griffin, L. C., Toole, J. J., Leung, L. L. The single-stranded DNA aptamer-binding site of human thrombin. The Journal of Biological Chemistry. 268 (28), 20808-20811 (1993).
- Carraro, C., et al. Behind the mirror: chirality tunes the reactivity and cytotoxicity of chloropiperidines as potential anticancer agents. ACS Medicinal Chemistry Letters. 10 (4), 552-557 (2019).
- Carraro, C., et al. Appended aromatic moieties in flexible bis-3-chloropiperidines confer tropism against pancreatic cancer cells. ChemMedChem. 16 (5), 860-868 (2021).
- Kypr, J., Kejnovska, I., Renciuk, D., Vorlickova, M. Circular dichroism and conformational polymorphism of DNA. Nucleic Acids Research. 37 (6), 1713-1725 (2009).
- Onel, B., Wu, G., Sun, D., Lin, C., Yang, D. Electrophoretic mobility shift assay and dimethyl sulfate footprinting for characterization of G-quadruplexes and G-quadruplex-protein complexes. Methods in Molecular Biology. 2035, 201-222 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon