Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Methods Article
Purificación de cromatina del locus del gen de copia única en Saccharomyces cerevisiae
En este artículo
Resumen
Este protocolo presenta un método de aislamiento de cromatina específico de locus basado en la recombinación específica del sitio para purificar un locus de gen de una sola copia de interés en su contexto de cromatina nativa a partir de la levadura en ciernes, Saccharomyces cerevisiae.
Resumen
La unidad organizativa básica de la cromatina eucariota es la partícula del núcleo del nucleosoma (NCP), que comprende el ADN envuelto ~ 1,7 veces alrededor de un octámero de histonas. La cromatina se define como la entidad de los NCP y muchos otros complejos proteicos, incluidos los factores de transcripción, la remodelación de la cromatina y las enzimas modificadoras. Todavía no está claro cómo se orquestan estas interacciones proteína-ADN a nivel de loci genómicos específicos durante las diferentes etapas del ciclo celular. Esto se debe principalmente a las limitaciones técnicas actuales, que dificultan la obtención de mediciones precisas de dichas interacciones dinámicas. Aquí, describimos un método mejorado que combina la recombinación específica del sitio con un protocolo eficiente de purificación de afinidad de un solo paso para aislar un locus de interés de un gen de copia única en su estado nativo de cromatina. El método permite el enriquecimiento robusto del locus diana sobre la cromatina genómica, lo que convierte a esta técnica en una estrategia eficaz para identificar y cuantificar las interacciones de proteínas de forma imparcial y sistemática, por ejemplo, mediante espectrometría de masas. Además de estos análisis de composición, la cromatina nativa purificada por este método probablemente refleje la situación in vivo con respecto al posicionamiento de los nucleosomas y las modificaciones de las histonas y, por lo tanto, es susceptible de análisis estructurales y bioquímicos adicionales de la cromatina derivada de prácticamente cualquier locus genómico en la levadura.
Introducción
La organización dinámica de los genomas eucariotas en cromatina compacta el ADN para que quepa dentro de los confines del núcleo, al tiempo que garantiza una dinámica suficiente para la expresión génica y la accesibilidad para los factores reguladores. En parte, esta versatilidad está mediada por el nucleosoma, la unidad básica de la cromatina, que comprende una partícula central con 147 pb de ADN envuelto ~ 1,7 veces alrededor del octámerode histonas 1. El nucleosoma es una estructura altamente dinámica con respecto a su composición, con numerosas variantes de histonas y modificaciones postraduccionales (PTM) en las colas de histonas N- y C-terminales. Además, la cromatina eucariota interactúa con una multitud de otros componentes esenciales, como factores de transcripción, maquinaria de procesamiento de ADN y ARN, proteínas arquitectónicas, enzimas involucradas en la remodelación y modificación de la cromatina y moléculas de ARN asociadas con la cromatina. Estas maquinarias cruciales involucradas en la transcripción, replicación y reparación requieren acceso a la cromatina, que sirve como sustrato natural para estos procesos. En consecuencia, la comprensión de los mecanismos moleculares que subyacen a estas transacciones de ADN requiere una definición precisa de las alteraciones colectivas en la estructura de la cromatina en las regiones genómicas específicas donde estas maquinarias convergen y facilitan las reacciones biológicas.
A pesar de la identificación de numerosos factores de cromatina a través de estudios genéticos y de interacción proteína-proteína, la realización de análisis directos, imparciales y exhaustivos de las interacciones de la cromatina en sitios genómicos particulares ha seguido siendo un obstáculo importante 2,3. Inicialmente, solo se pudieron aislar regiones muy abundantes del genoma (es decir, loci repetitivos) o plásmidos de copia múltiple en cantidades y pureza suficientes para la identificación por espectrometría de masas de las proteínas asociadas 4,5,6,7. Una serie de nuevos enfoques basados en la hibridación directa de sondas de captura con ADN cromatinizado, la biotinilación de proximidad mediante el sistema CRISPR-dCas9 o la unión de proteínas adaptadoras específicas de secuencia al locus de interés han comenzado a desentrañar el proteoma de loci de copia única de genomas de levaduras y mamíferos 8,9,10. Sin embargo, todos estos métodos requieren reticulación de formaldehído para estabilizar las interacciones proteína-ADN y sonicación para solubilizar la cromatina para su posterior purificación. Juntas, ambas manipulaciones excluyen la posibilidad de estudios estructurales y funcionales posteriores de la cromatina purificada.
Para superar estas limitaciones, previamente ideamos una metodología que emplea la recombinación específica del sitio para extraer dominios cromosómicos específicos de la levadura11,12. En esencia, la región genómica de interés está rodeada por sitios de reconocimiento (RS) para la R-recombinasa específica del sitio de Zygosaccharomyces rouxi, mientras que simultáneamente incorpora un grupo de tres sitios de unión al ADN para la proteína LexA represora transcripcional procariota (LexA) dentro de la misma región. Las células de levadura contienen un casete de expresión para la expresión simultánea de R-recombinasa y una proteína LexA fusionada a una etiqueta de purificación de afinidad en tándem (TAP). Después de la inducción de la R-recombinasa, la enzima extirpa eficientemente la región objetivo del cromosoma en forma de un dominio circular de cromatina. Este dominio se puede purificar a través de la proteína adaptadora LexA-TAP, que se une a los sitios de unión del ADN LexA, así como a un soporte de afinidad. Este método se ha utilizado recientemente para aislar distintos dominios de cromatina que contienen orígenes de replicación seleccionados del cromosoma III13 de levadura.
Una de las principales ventajas de este enfoque ex vivo es que permite realizar análisis funcionales del material aislado. Por ejemplo, los dominios de origen de replicación purificados con este método pueden someterse a ensayos de replicación in vitro para evaluar la eficiencia de la cocción de origen en un tubo de ensayo a partir de plantillas de cromatina ensambladas in vivo nativas. En última instancia, la caracterización bioquímica y funcional del material aislado puede permitir la reconstitución de procesos nucleares utilizando proteínas purificadas junto con la plantilla de cromatina nativa. En resumen, esta metodología abre una vía emocionante en la investigación de la cromatina, ya que será posible seguir los cambios colectivos en la composición y la estructura de la cromatina de una región genómica específica que sufre una determinada transacción cromosómica.
Protocolo
Consulte la Tabla de materiales para obtener detalles relacionados con todos los materiales e instrumentos utilizados en este protocolo. Consulte la Tabla 1 para obtener una lista de las soluciones, los búferes y los medios utilizados.
1. Construcción de cepas de levadura recombinante
- Para construir una cepa de levadura competente para la recombinación, transforme el plásmido K238 digerido por SbfI en una cepa de levadura con sitios de unión a LexA integrados y sitios de recombinación RS en el locus de interés13,14.
NOTA: El fragmento de restricción de SbfI transformado contiene el casete de expresión requerido para la expresión constitutiva de la proteína de fusión LexA-TAP y la R-recombinasa junto con secuencias homólogas del cromosoma I de levadura para la integración genómica del casete de expresión por recombinación homóloga (Figura 1). - Para construir una cepa de control, se toma una cepa de levadura isogénica que carece de sitios integrados de unión a RS y LexA, y se transforma con plásmido K238 digerido por SbfI en las mismas condiciones que se utilizan para la cepa competente para la recombinación.
- Seleccione las células de levadura competentes basándose en el marcador de selección de LEU2 en placas de agar SCD-LEU14.
2. Acoplamiento de anticuerpos IgG a perlas magnéticas activadas por epoxi
NOTA: Acoplar anticuerpos IgG contra perlas magnéticas activadas por epoxi de acuerdo con el siguiente protocolo publicado11.
- Suspender 300 mg de perlas activadas con epoxi en 10 ml de acetona al 50% (300 mg corresponden a ~5,1 ×10 perlas) en un tubo cónico de 50 ml. Agite vigorosamente en una batidora de vórtice.
- Centrifugar el tubo que contiene las perlas a 820 × g y 4 °C durante 2 min. Retire el sobrenadante.
- Lavar las perlas 3 veces con 20 ml de tampón de fosfato de sodio 0,1 M (pH 7,4). Retire el sobrenadante después de cada paso de lavado por centrifugación como se describe en el paso 2.2.
- Suspender las perlas en 16 mL de tampón fosfato sódico 0,1 M (pH 7,4) y girar suavemente en un horno de hibridación durante 5 min a temperatura ambiente.
- Disolver las IgG de conejo (100 mg) en 7 mL dedH2O (concentración final: 14 mg/mL).
- Centrifugar la suspensión de IgG a 13.000 × g y 4 °C durante 10 min para aclarar la suspensión.
- Transferir 3,5 mL del sobrenadante (correspondiente a 50 mg de IgG de conejo) a un nuevo tubo cónico de 50 mL.
- Diluir la solución de IgG con 9,85 mL de tampón fosfato de sodio 0,1 M (pH 7,4), seguido de la adición gota a gota de 6,65 mL de sulfato de amonio 3 M en fosfato sódico 0,1 M (pH 7,4) bajo una mezcla suave.
NOTA: Evite agregar sulfato de amonio rápidamente a la solución, ya que una alta concentración local de la sal provocará la precipitación de las IgG de conejo. - Centrifugar la solución de IgG a 820 × g y 4 °C durante 3 min, y añadir el sobrenadante resultante a la suspensión de perlas magnéticas.
- Incubar el tubo durante la noche o al menos durante 18 h a 30 °C con una rotación suave en un horno de hibridación.
- Retire el sobrenadante como se describe en el paso 2.2.
- Lavar las perlas con 20 mL de 100 mM de glicina-HCl (pH 2,5). Retire la solución rápidamente como se describe en el paso 2.2 para evitar la desnaturalización de los polipéptidos IgG.
- Lavar las perlas una vez con 20 mL de Tris-HCl 10 mM (pH 8,8). Aspire el sobrenadante como se describe en el paso 2.2.
- Añadir 20 ml de solución de trietilamina 0,1M durante 5-10 min bajo una rotación suave para inactivar los grupos epoxi reactivos residuales. Retire el sobrenadante como se describe en el paso 2.2.
- Lave las perlas 4 veces con 20 ml de PBS (pH 7,4) durante 5 minutos con una rotación suave. Retire el sobrenadante como se describe en el paso 2.2 después de cada paso de lavado.
- Lave las perlas 2 veces con 20 ml de PBS (pH 7,4) con Triton X-100 al 0,5% (p/v) durante 5 minutos y 15 minutos cada una con una rotación suave en un horno de hibridación. Retire el sobrenadante como se describe en el paso 2.2.
- Suspender las perlas en un volumen final de 16 mL de PBS (pH 7,4) con azida sódica al 0,02% (p/v). Conservar en forma de alícuotas de 1 ml a 4 °C hasta su uso.
3. Cultivo y recolección de células de levadura
- Inocular cepas de levadura competentes para el control y la recombinación de placas YPD en 5 mL de medio YPR e incubar durante la noche a 30 °C y 200 rpm.
- Inocular 2 mL de este cultivo en 100 mL de medio YPR e incubar durante la noche a 30 °C y 200 rpm.
- Para cada cepa, dispensar 1.800 ml de medio YP esterilizado en autoclave y 200 ml de solución de rafinosa esterilizada en autoclave (20% p/v) en cada uno de los dos matraces Erlenmeyer de 5 L (4 L de medio de cultivo en total para cada cepa, divididos en dos frascos).
- Inocular las células de levadura en crecimiento a DO600 0,2 en su medio respectivo e incubar como se describe en el paso 3.1 durante aproximadamente 6 h o hasta que las células alcancen el DOdeseado 600 de 1,0. Para garantizar un crecimiento normal de las células, libres de cualquier contaminación, compruebe el OD a intervalos de 2 h.
- Añadir 200 mL de galactosa (20% p/v) para inducir la recombinación específica del sitio.
- Añadir 110 μL (50 ng/mL) de YMFA (al mismo tiempo que la galactosa, paso 3.5) para detener las células en la fase G1, e incubar durante 2 h.
NOTA: La adición de YMFA a las células depende de las condiciones experimentales y de la cuestión biológica que se vaya a abordar. Precisamente, para este experimento, las células se detienen en fase G1 para obtener una población celular homogénea con orígenes de replicación autorizados; en este proceso, el complejo pre-replicativo se une a los orígenes en la fase G1 antes del inicio de la replicación del ADN en la fase S. - Transfiera la suspensión celular a cubos de centrífuga de 1 L y centrifugue a 6.000 × g y 4 °C durante 10 min. Deseche el sobrenadante y vuelva a suspender los gránulos celulares en un volumen total de 10-15 mL de dH2O.
- Selle una jeringa de 25 ml con un tapón Luer y colóquela dentro de un tubo cónico de 50 ml lleno de agua. Transfiera la suspensión de la celda al conjunto de la jeringa y centrifugue a 2.397 × g durante 10 minutos a temperatura ambiente. Deseche el sobrenadante.
- Retire el tapón Luer de la jeringa y extruya las células en nitrógeno líquido para formar "espaguetis" celulares.
NOTA: El uso de 4 L de medio de cultivo proporciona un peso húmedo de los gránulos de celda final de 7-10 g. - Transfiera los espaguetis celulares congelados a un tubo cónico de 50 ml y guárdelos a -80 °C hasta su nuevo uso.
4. Purificación del locus de la cromatina
NOTA: Consulte la Figura 2 para obtener una descripción general esquemática de los pasos involucrados en este protocolo de purificación de cromatina específico del locus.
- Enfríe un molinillo de café comercial moliendo 30-50 g de hielo seco agitando vigorosamente el molinillo de café 2 veces durante 30 s cada vez. Una vez enfriado, deseche el polvo de hielo seco.
- Mezcle 3 g de espaguetis congelados con células de levadura con ~ 30-50 g de hielo seco en el molinillo de café preenfriado.
- Selle la unión entre la tapa y el molinillo con parafilm para evitar la pérdida de polvo de células de levadura durante la molienda.
- Moler la mezcla de hielo seco y celda de levadura 10 veces durante 30 s cada vez, con intervalos de 30 s entre cada ronda (tiempo total estimado: ~10 min). Para evitar que el hielo seco y el polvo celular se adhieran a las paredes del molinillo de café, golpee los lados del molinillo de café continuamente durante la molienda.
- Transfiera el polvo de hielo seco de células de levadura resultante a un vaso de plástico con una espátula limpia y seca.
- Mantenga los vasos de precipitados a temperatura ambiente para evaporar el hielo seco.
- Una vez que el hielo seco se evapore, agregue 2,25 ml de tampón MB con 1x inhibidores de proteasa y fosfatasa (750 μL/g de células de levadura) al polvo de células de levadura.
- Pipetear vigorosamente la mezcla de células y tampones para asegurar la suspensión completa de las células con el tampón y transferirla a un tubo cónico de 15 ml.
- Tomar muestras para análisis de ADN (0,1%) y proteínas (0,05%) de la suspensión celular resultante (extractos celulares crudos [CCE]). Conservar a -20 °C hasta su posterior procesamiento (descrito en la sección 7).
- Transfiera la suspensión celular a tubos de reacción de 2 ml de baja unión y centrifugue a 21,130 × g durante 30 min a 4 °C para separar los restos celulares del lisado celular crudo.
- Agrupe los sobrenadantes de todos los tubos en un tubo cónico de 15 ml.
- Tomar muestras del sobrenadante (entrada [IN]) para análisis de ADN (0,1%) y proteínas (0,05%). Conservar a -20 °C hasta su posterior procesamiento (descrito en la sección 7).
- Equilibre 500 μL de lodo magnético de perlas acoplado a IgG (para cada cepa de levadura) lavando 2 veces con 500 μL de tampón MB frío con 1 inhibidor de proteasa y fosfatasa durante 5 min cada uno a 4 °C en rotación a 20 rpm. Deseche el sobrenadante de las perlas con una rejilla magnética después de cada paso de lavado.
- Incubar las perlas magnéticas acopladas a IgG en 500 μL de tampón MB frío con inhibidores de proteasa y fosfatasa 1x durante 1 h a 4 °C en rotación a 20 rpm. Deseche el sobrenadante de las cuentas con una rejilla magnética.
- Mezclar la suspensión de perlas magnéticas equilibradas con el lisado celular e incubar con rotación a 20 rpm durante 2 h a 4 °C.
- Transfiera la suspensión completa de lisado de células de perlas magnéticas a tubos de reacción de baja unión.
- Separe las perlas magnéticas, que ahora llevan los anillos de cromatina de interés, del lisado celular utilizando una rejilla magnética.
- Transfiera el flujo de paso restante (FT) de cada tubo a un tubo cónico nuevo de 15 ml y tome muestras para el análisis de ADN (0,1 %) y proteínas (0,05 %). Conservar a -20 °C hasta su posterior procesamiento (descrito en la sección 7).
- Suspenda las perlas magnéticas de cada tubo en 300 μL de tampón MB frío y agrupe las perlas en un tubo de reacción. Lavar 5 veces durante 10 minutos cada una con 750 μL de tampón MB frío con 1x inhibidores de la proteasa y la fosfatasa a 4 °C en rotación a 20 rpm. Realizar el lavado final con 750 μL de tampón MB frío sin inhibidores de proteasa y fosfatasa.
- Suspender las perlas en 40 μL de tampón MB frío sin inhibidores de la proteasa y la fosfatasa.
NOTA: El volumen final del eluido se puede ajustar de acuerdo con su aplicación posterior anticipada. La elución de TEV generalmente funciona de manera eficiente dentro del rango de 100-300 μL.
5. Elución mediada por proteasa TEV
- Para liberar los complejos de anillos de cromatina LexA-CBP de las perlas, incube las perlas con 2 μL de proteasa TEV recombinante marcada con 6x His durante la noche a 4 °C y 450 rpm en un termomezclador.
- Separe las perlas del eluido final con una rejilla magnética. Transfiera el eluido, que contiene anillos de cromatina escindidos, a un nuevo tubo de reacción de 1,5 ml.
- Mantenga los tubos en una rejilla magnética nuevamente para separar las perlas residuales del eluido final.
- Resuspender las perlas (TB) en 750 μL de tampón AC frío y tomar muestras para análisis de ADN (0,1%) y proteínas (0,05%). Conservar a -20 °C hasta su posterior procesamiento (descrito en la sección 7).
- A partir del eluido final (TE), tomar muestras para análisis de ADN (0,5%) y proteínas (0,25%). Conservar a -20 °C hasta su posterior procesamiento (descrito en la sección 7).
NOTA: Debido al bajo volumen del eluido final, se incrementa el porcentaje de las muestras recogidas para el análisis de ADN y proteínas para obtener una mejor representación de su contenido en proteínas y ADN durante el análisis.
6. Elución por desnaturalización
- Lave las perlas 2 veces en 750 μL de tampón de CA frío durante 20 minutos cada vez a 4 °C en rotación a 20 rpm.
- Para extraer los complejos de cromatina LexA unidos restantes, se lleva a cabo la elución por desnaturalización añadiendo 500 μL de 0,5M de NH4OH a las perlas y se incuba durante 30 min a temperatura ambiente.
- Separe las perlas de la suspensión con una rejilla magnética y transfiera el eluido a un tubo de reacción de baja unión.
- Vuelva a incubar las perlas como en el paso 6.2 para recuperar los loci máximos de cromatina en el eluido.
- Separe las perlas de la suspensión con una rejilla magnética y agrupe el eluido resultante en el mismo tubo que contiene el eluido del paso 6.3.
- Resuspender las perlas (DB) en 750 μL dedH2O, y tomar muestras para análisis de ADN (0,1%) y proteínas (0,05%).
- A partir del eluido final (DE), tomar muestras para análisis de ADN (0,5%) y proteínas (0,25%).
7. Análisis de ADN y proteínas
- Análisis de ADN
- Preparación de la muestra
- A las muestras de ADN, añadir 100 μL de tampón IRN y aumentar el volumen condH2O hasta un volumen final de 200 μL.
- Agregue 1 ng de ADN plasmídico con pico K071.
- Añadir 1 μL de ARNasa A (10 mg/mL) e incubar a 37 °C durante 1 h.
- Añadir 5 μL de proteinasa K (10 mg/mL) y 10 μL de SDS (20%), e incubar durante 1 h a 56 °C.
- Añadir 200 μL de fenol/cloroformo/alcohol isopropílico (25:24:1) y vórtice a fondo 2 veces durante 10 s cada vez.
- Centrifugar las muestras a 21.130 × g durante 7 min para separar las fases orgánica y acuosa.
- Transfiera el sobrenadante a nuevos tubos de 1,5 ml que contengan 1,5 μl de glucógeno (5 mg/ml) y 2,5 veces etanol al 100 %.
- Incubar los tubos a -20 °C durante un mínimo de 2 h y un máximo de toda la noche para precipitar el ADN.
- Centrifugar a 21.130 × g, 4 °C durante 45 min. Deseche el sobrenadante.
- Añadir 150 μL de etanol al 70% sin resuspender el gránulo de ADN, y centrifugar de nuevo a 21.130 × g a 4 °C durante 10 min.
- Secar los gránulos de ADN a temperatura ambiente durante 10-15 min, y volver a suspender en 40 μL dedH2O.
- Realice la digestión de la enzima de restricción para linealizar el ADN aislado en el paso 7.1.1.11 con endonucleasa de restricción de HpaI. Para una mezcla de reacción de 50 μL, incubar 40 μL de ADN aislado + 2 μL de enzima HpaI + 5 μL de tampón enzimático de restricción + 3 μL de dH2O durante la noche a 37 °C.
- Análisis de qPCR
- Diluir el ADN digerido por restricción en una proporción de 1:5 (10 μL del ADN digerido por restricción obtenido del paso 7.1.1.12 en 40 μL dedH2O).
- Utilice pares de cebadores diseñados para la región ARS316, PDC1 y ADN plasmídico de pico.
- Ejecute qPCR utilizando el programa que se indica en la Tabla 2.
- Analice los resultados de la qPCR mediante cuantificación relativa utilizando la región ARS316 y el ADN plasmídico de espiga como dianas y PDC1 (o cualquier otro gen o región genómica) como locus de referencia.
- Evalúe el porcentaje de recuperación del locus ARS316 sobre PDC1 normalizando los valores relativos de cuantificación de los cebadores ARS316 y PDC1 por el porcentaje del volumen de fracción tomado para cada muestra. Por ejemplo, multiplique el 0,1% para el flujo continuo por un factor de 1.000 y el 0,5% para el eluido TEV por un factor de 200.
- Normalice los porcentajes de recuperación de ARS316 y PDC1 con los valores de cuantificación relativa del ADN plasmídico de pico.
- Evaluar el enriquecimiento del locus ARS316 sobre la cromatina total mediante la evaluación de los valores nominales/referencia de cuantificación relativos obtenidos utilizando la ecuación (1).
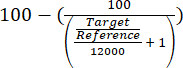 (1)
(1)
- Análisis de Southern blot
- A 20 μL de muestra de ADN digerido por enzima de restricción, agregue 5 μL de colorante de carga de gel.
- Pasar las muestras en un gel de agarosa al 1% a 110 V durante ~3 h.
- Transfiera el gel a una bandeja y sumérjalo en una solución de desurinación agitando suavemente durante 20 minutos.
- Enjuague el gel con dH2O y sumérjalo en una solución de desnaturalización durante 15 minutos agitando. Deseche la solución de desnaturalización.
- Remojar el gel en una solución de desnaturalización durante 15 minutos agitando suavemente.
- Enjuague el gel con dH2O y lávelo 2 veces durante 15 minutos cada vez con el tampón de transferencia agitando suavemente.
- Llene una bandeja con 1-1,5 L de tampón de transferencia y colóquela sobre una plataforma estable.
- Corta un trozo largo de papel Whatman y colócalo en la plataforma de tal manera que los dos extremos del papel queden empapados en el búfer de transferencia.
- Corta una tira de membrana de nailon positivo y cuatro tiras de papel Whatman iguales al tamaño del gel.
- Coloque dos trozos de papel Whatman empapados en el búfer de transferencia en la plataforma.
- Coloca el gel boca abajo sobre los pedazos de papel Whatman.
- Encima del gel, coloque la membrana de nailon positivo, seguida de los dos trozos restantes de papel Whatman empapados en tampón de transferencia. Asegúrese de mantener la pila húmeda con el tampón de transferencia.
- Elimine las burbujas de aire atrapadas rodándolas con una varilla de vidrio.
- Coloque una pila de toallas de papel encima de la pila ensamblada, seguida de un objeto de 0,5 kg (por ejemplo, una botella de vidrio).
- Deje el conjunto para transferirlo durante la noche a temperatura ambiente.
- Después de la transferencia, reticulación UV de la membrana con una producción de energía total de 1.200 J/cm2.
- Para la detección del locus ARS316, realice la hibridación de Southern blot utilizando sondas radiactivas sintetizadas utilizando el sistema de marcaje de ADN referenciado.
NOTA: Realice todos los pasos siguientes en hielo hasta que se indique lo contrario. - En un tubo de reacción de baja unión, diluir 25-40 ng de fragmento de PCR (sonda) en 19 μL dedH2O, e incubar a 95 °C durante 5 min. Deje enfriar inmediatamente sobre hielo.
- Agregue 1 μL de 500 μM de dCTP, 500 μM de dGTP y 500 μM de dTTP al tubo.
- Añadir 20 μL del tampón que contiene hexámeros de nucleótidos aleatorios suministrado con el kit.
- Agregue 5 μL del nucleótido marcado radiactivamente [α-32P] dATP al tubo. Realice todos los pasos que involucren material marcado radiactivamente en un entorno protector aprobado por radiactividad.
- Añadir 1 μL de fragmento de Klenow e incubar a 37 °C en un termomezclador durante 15 min para la síntesis de ADN monocatenario.
- Añadir 5 μL de tampón de parada para detener la reacción.
- Centrifugar una columna de exclusión de tamaño durante 2 minutos a 1.500 × g para eliminar el búfer de almacenamiento. Deseche el flujo y coloque la columna en un tubo nuevo.
- Pase la mezcla de la sonda a través de la columna para eliminar los nucleótidos radiactivos no incorporados. Centrifugar durante 30 s a 1.500 × g para recoger la sonda.
- Añadir 150 μL de ADN de esperma de salmón (1:100) para bloquear la unión inespecífica de la sonda a la membrana.
- Incubar la mezcla de la sonda a 95 °C durante 5 min para desnaturalizar el ADN bicatenario.
- Congelar en hielo durante unos segundos y centrifugar durante unos segundos a 500 × g para reducir las gotas de agua condensadas en la tapa.
- Lave brevemente la membrana de Southern blot con el tampón de hibridación durante 5-10 min con rotación.
- Prehibridar la membrana con el tampón de hibridación durante 1 h a 55 °C con rotación. Deseche el tampón de hibridación.
- Añadir 15 ml de tampón de hibridación fresco (precalentado a 55 °C) a la membrana junto con la sonda preparada. Incubar la membrana durante la noche a 55 °C con rotación.
- Realice lavados posteriores a la hibridación 2 veces durante 15 minutos cada uno con 0,3x SSC, 0,1% SDS a 55 °C con rotación.
- Realice lavados posteriores a la hibridación 2 veces durante 15 minutos cada uno con 0,1x SSC, 0,1% SDS a 55 °C con rotación.
- Realice lavados posteriores a la hibridación 2 veces durante 15 minutos cada uno con 0,1x SSC, 1,5% SDS a 55 °C con rotación.
- Retire la membrana del tubo de hibridación y exponga la membrana a una pantalla de fosforografía durante un máximo de 3 días para obtener las huellas radiactivas en la película.
- Adquiera imágenes de la película utilizando un sistema de escaneo láser de imágenes de fósforo.
- Preparación de la muestra
- Análisis de proteínas
- Preparación de la muestra
- Ajuste todas las muestras de proteínas a sus volúmenes finales (200 μL, 100 μL, 100 μL, 20 μL y 20 μL para el extracto de células crudas, entrada, flujo, perlas y muestras de eluido, respectivamente) agregando 1 μL de β-mercaptoetanol, tampón de muestra PAGE LDS (1x) y dH2O.
- Desnaturalizar las muestras a 95 °C durante 5 min.
- Análisis de Western blot
NOTA: Realice SDS-PAGE y Western blot utilizando los protocolos estándar15,16.- Cargue 15 μL de cada muestra en cada pocillo de un gel SDS-PAGE de 1,5 mm.
- Realizar la inmunotinción del blot utilizando anticuerpos PAP (1:2.000) y LexA (1:1.000) con incubación nocturna a 4 °C. Diluir en una solución de leche en polvo al 5% / PBST.
NOTA: Después del análisis de Papanicolaou, la transferencia se puede eliminar utilizando un protocolo de extracción suave17 antes de la inmunotinción con el segundo anticuerpo LexA.
- Preparación de la muestra
Resultados
La purificación del dominio de cromatina ARS316 de ~1,4 kb fue mediada por la proteína adaptadora LexA-TAP expresada constitutivamente. Para que sirviera como control negativo, realizamos purificaciones utilizando una cepa isogénica que expresa LexA-TAP pero que no contiene sitios integrados de unión a RS y LexA. La Figura 3 ilustra el resultado del análisis de ADN de un experimento de purificación estándar realizado tanto en el control como en una cepa competente para la recombinaci?...
Discusión
La identificación de los factores y el panorama de la cromatina de una región genómica diana específica sigue planteando un reto importante en la investigación de la cromatina18. Este protocolo describe un sistema eficiente para extirpar y purificar específicamente distintos dominios de cromatina de los cromosomas de levadura. Hasta donde sabemos, la pureza y el rendimiento de esta purificación en un solo paso superan muchas de las limitaciones de los métodos de purificación de cromatina ...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
El trabajo en el laboratorio de S.H. contó con el apoyo de la DFG a través de SFB1064 (ID de proyecto 213249687), el Consejo Europeo de Investigación (ERC Starting Grant 852798 ConflictResolution) y la Helmholtz Gesellschaft.
Materiales
| Name | Company | Catalog Number | Comments |
| Yeast strains | |||
| Control Strain: MATa; ura3Δ0; leu2Δ0; his3Δ1; met15Δ0; bar1::kanMX4; Chr I 212kb::LEU2 pTEF2-LEXA-TAP pGAL1-10 RecR | Section 1, see references 13 and 14 | ||
| Recombination Strain: MATa; ura3Δ0; leu2Δ0; his3Δ1; met15Δ0; bar1::kanMX4; RS_LEXA_NS-3_ARS316_NS+3_RS; Chr I 212kb::LEU2 pTEF2-LEXA-TAP pGAL1-10 RecR | Section 1, see reference 13 and 14 | ||
| Plasmid | |||
| K238 plasmid | Section 1, see reference 13 Storage: Store at -20 °C | ||
| K071 Spike-in plasmid DNA | Section 7.1, see reference 13 Storage: Store at -20 °C | ||
| Reagents | |||
| Acetone | Carl Roth | 5025.1 | Section 2 Storage: Store at room temperature |
| Ammonium acetate (NH4Ac) | Sigma Aldrich | A7262 | Section 6 and 7.1 Storage: Store at room temperature |
| Ammonium solution (NH4OH) 25% | Merck Millipore | 533003 | Section 6 Storage: Store at room temperature |
| Ammonium sulfate | Santa Cruz | Sc-29085 | Section 2 Storage: Store at room temperature |
| Bacto agar | BD (VWR) | 90000-760 | Section 3 Storage: Store at room temperature |
| Bacto peptone | BD (VWR) | 211820 | Section 3 Storage: Store at room temperature |
| β-Mercaptoethanol | Sigma Aldrich | 07604 | Section 7.2 Storage: Store at 4 °C |
| Chemiluminescent substrate kit | ThermoFisher | 34580 | Section 7.2 Storage: Store at 4 °C |
| Di-Sodium Hydrogen phosphate dodecahydrate | Merck | 1.06579.1000 | Section 2 and 7.1 Storage: Store at room temperature |
| Dithiothreitol (DTT) | ThermoFisher | 15508013 | Section 4 Storage: Store at 4 °C |
| Ethanol | Merck | 100983 | Section 7.1 Storage: Store at room temperature |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma Aldrich | ED | Section 7.1 Storage: Store at room temperature |
| Galactose (20% (w/v) stock) | Sigma Aldrich | G0625-1KG / 5KG | Section 3 Storage: Store at room temperature |
| Gel loading dye (6x) | BioLabs | B7024A | Section 7.1 Storage: Store at -20 °C |
| Glusose | Sigma-Aldrich | G8270 | Section Storage: Store at room temperature |
| Glycine | Carl Roth | .0079.4 | Section 2 Storage: Store at room temperature |
| Glycogen (5 mg/mL) | Invitrogen | AM9510 | Section 7.1 Storage: Store at -20 °C |
| Hydrochloric acid (HCl) | PanReac AppliChem | 182109.1211 | Section 2, 4 and 7.1 Storage: Store at room temperature |
| Magnesium Acetate (MgAc) | Bernd Kraft | 15274.2600/C035 | Section 4 Storage: Store at room temperature |
| Magnesium chloride (MgCl2) | Sigma Aldrich | M8266 | Section 6 Storage: Store at room temperature |
| Nu PAGE LDS sample buffer (4x) | Invitrogen | 2399549 | Section 7.2 Storage: Store at room temperature |
| Phenol/Chloroform/Isoamyl alcohol (25:24:1 v/v) | Invitrogen | 15593-031 | Section 7.1 Storage: Store at 4 °C |
| Potassium chloride (KCl) | Sigma | P9541 | Section 4 Storage: Store at room temperature |
| Radioactively labeled α-32P dATP (3,000 Ci/mmol, 10 mCi/mL) | Hartmann Analytic | SRP-203 | Section 7.1 Storage: Store at 4 °C |
| RadPrime labeling system | ThermoFisher | 18428-011 | Section 7.1 Storage: Store at -20 °C |
| Raffinose (20% (w/v) stock) | SERVA | 34140.03 | Section 3 Storage: Store at room temperature |
| Sodium chloride (NaCl) | Merck | K53710504142 | Section 7.1 Storage: Store at room temperature |
| Sodium citrate (Na3C6H5O7) | Sigma-Aldrich | 71402 | Section 7.1 Storage: Store at room temperature |
| Sodium hydroxide (NaOH) | Sigma Aldrich | S5881 | Section 7.1 Storage: Store at room temperature |
| Sodium n-dodecyl sulfate (SDS) (5% stock (w/v) ) | Alfa Aesar | A11183 | Section 7.1 Storage: Store at room temperature |
| Sodium phosphate monobasic | Sigma-Aldrich | 71496 | Section 2 and 7.1 Storage: Store at room temperature |
| Sodium azide | Santa Cruz Biotechnology | sc-208393 | Section 2 Storage: Store at -20 °C |
| Triethylamine | Sigma Aldrich | 90340 | Section 2 Storage: Store at room temperature |
| Tris base | Chem Cruz | SC-3715B | Section 2 and 4 Storage: Store at room temperature |
| Triton X-100 | Sigma Aldrich | X100 | Section 2 and 4 Storage: Store at room temperature |
| Tween-20 | Bernd Kraft | 18014332 | Section 4 Storage: Store at room temperature |
| Yeast extract | BD (VWR) | 212720 | Section 3 Storage: Store at room temperature |
| Yeast mating factor alpha (1 µg/mL stock ) | Biomol | Y2016.5 | Section 3 Storage: Store at -20 °C |
| Yeast Synthetic Drop-out medium Supplements without LEUCINE | Sigma Aldrich | Y1376 | Section 1, see reference 14 |
| Enzymes | |||
| HpaI restriction enzyme (5,000 U/mL) | NEB | R0105S | Section 7.1 Storage: Store at -20 °C |
| Protease and Phosphatase Inhibitor Cocktail (100x) | ThermoFisher Scientific | 78446 | Section 4 Storage: Store at4 °C |
| Proteinase K (10 mg/mL) | SERVA | 33756 | Section 7.1 Storage: Store at -20 °C |
| RNase A (10 mg/mL) | ThermoFisher | EN0531 | Section 7.1 Storage: Store at -20 °C |
| TEV protease (10000 U/µL) | NEB | P8112S | Section 5 Storage: Store at -20 °C |
| Materials | |||
| BcMag Epoxy-Activated Magnetic Beads | Bioclone Inc. | FC-102 | Section 2 Storage: Store at 4 °C |
| Dry ice | Section 4 | ||
| Low-binding centrifuge tubes 2.0 mL | Eppendorf | 22431102 | Section 4 |
| Microspin G-25 Columns | Cytiva | 27-5325-01 | Section 7.1 Storage: Store at room temperature |
| Parafilm | Merck | P7793 | Section 4 |
| Positive nylon membrane | Biozol | 11MEMP0001 | Section 7.1 Storage: Store at room temperature |
| PVDF transfer membrane | Immobilon-Merck Millipore | IPVH00010 | Section 7.2 Storage: Store at room temperature |
| SDS-PAGE gel 4-12% bis-tris (15 well, 1.5 mm) | Invitrogen | NP0336BOX | Section 7.2 Storage: Store at 4 °C |
| Syringe (25 mL) with luer fitting | Henke Sass Wolf | 4200-000V0 | Section 3 |
| Whatman paper (Grade 3MM CHR Cellulose Western Blotting Paper Sheet) | Cytiva | 3030-917 | Section 7.1 Storage: Store at room temperature |
| Antibodies | |||
| Anti-LexA, rabbit polyclonal IgG, DNA binding region antibody | Merck Millipore | 06-719 | Section 7.2 Storage: Store at -20 °C |
| Goat Anti-Rabbit IgG (H+L), Horseradish peroxidase conjugate | Invitrogen | G21234 | Section 7.2 Storage: Store at -20 °C |
| Peroxidase Anti-Peroxidase (PAP) antibody produced in rabbit for the detection of TAP-tagged proteins | Sigma Aldrich | P1291-500UL | Section 7.2 Storage: Store at -20 °C |
| Rabbit IgG antibodies | Sigma | I5006-100MG | Section 2 Storage: Store at 4 °C |
| Primers (10 µM) | |||
| ARS316: fwd 5'- CGGCATTATCGTACACAACCT, rev 5'- GTTCTTCGTTGCCTACATTTTCT | Section 7.1 | ||
| K071 Spike-in plasmid DNA: fwd: 5'-TTTTCGCTGCTTGTCCTTTT, rev 5'- CATTTTCGTCCTCCCAACAT | Section 7.1 | ||
| PCR fragment from yeast genomic DNA as a template for ARS316 amplification (for southern blot): fwd 5’- AAATTCTGCCCTTGATTCGT rev 5’- TTTGTTTATCTCATCACTAAT | Section 7.1 | ||
| PDC1: fwd 5'- CATGATCAGATGGGGCTTCA, rev 5'-ACCGGTGGTAGCGACTCTGT | Section 7.1 | ||
| Equipment | |||
| Coffee grinder | Gastroback | 42601 | Section 4 |
| Dewar flask | NAL GENE | 4150-2000 | Section 3 |
| DynaMag TM-2 magnetic rack | Invitrogen | 12321D | Section 4, 5 and 6 |
| Hybridization oven | Hybaid Mini10 | Ri418 | Section 2 |
| Microcentrifuge | Eppendorf | 5424R | Section 4 and 7.1 |
| UV-crosslinker | Analytikjena | 95-0174-02 | Section 7.1 |
Referencias
- Kornberg, R. D., Lorch, Y. Twenty-five years of the nucleosome, fundamental particle of the eukaryote chromosome. Cell. 98 (3), 285-294 (1999).
- Gauchier, M., van Mierlo, G., Vermeulen, M., Déjardin, J. Purification and enrichment of specific chromatin loci. Nature Methods. 17 (4), 380-389 (2020).
- Korthout, T., et al. Decoding the chromatin proteome of a single genomic locus by DNA sequencing. PLoS Biology. 16 (7), e2005542 (2018).
- Antão, J. M., Mason, J. M., Déjardin, J., Kingston, R. E. Protein landscape at Drosophila melanogaster telomere-associated sequence repeats. Molecular and Cellular Biology. 32 (12), 2170-2182 (2012).
- Déjardin, J., Kingston, R. E. Purification of proteins associated with specific genomic loci. Cell. 136 (1), 175-186 (2009).
- Ide, S., Dejardin, J. End-targeting proteomics of isolated chromatin segments of a mammalian ribosomal RNA gene promoter. Nature Communications. 6, 6674 (2015).
- Unnikrishnan, A., Gafken, P. R., Tsukiyama, T. Dynamic changes in histone acetylation regulate origins of DNA replication. Nature Structural & Molecular Biology. 17 (4), 430 (2010).
- Buxton, K. E., et al. Elucidating protein-DNA interactions in human alphoid chromatin via hybridization capture and mass spectrometry. Journal of Proteome Research. 16 (9), 3433-3442 (2017).
- Kennedy-Darling, J., et al. Discovery of chromatin-associated proteins via sequence-specific capture and mass spectrometric protein identification in Saccharomyces cerevisiae. Journal of Proteome Research. 13 (8), 3810-3825 (2014).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355, 7111 (2017).
- Hamperl, S., et al. Purification of specific chromatin domains from single-copy gene loci in Saccharomyces cerevisiae. Functional Analysis of DNA and Chromatin. 1094, 329-341 (2014).
- Hamperl, S., et al. Compositional and structural analysis of selected chromosomal domains from Saccharomyces cerevisiae. Nucleic Acids Research. 42 (1), 2 (2014).
- Weiβ, M. Single-copy locus proteomics of early- and late-firing DNA replication origins identifies a role of Ask1/DASH complex in replication timing control. Cell Reports. 42 (2), 112045 (2023).
- Gietz, R. D., Schiestl, R. H. High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method. Nature Protocols. 2 (1), 31-34 (2007).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. Proceedings of the National Academy of Sciences of the United States of America. 76 (9), 4350-4354 (1979).
- Western blot membrane stripping for restaining protocol. Abcam Available from: https://www.abcam.com/protocols/western-blot-membrane-stripping-for-restaining-protocol (2023)
- Vermeulen, M., Déjardin, J. Locus-specific chromatin isolation. Nature Reviews. Molecular Cell Biology. 21 (5), 249-250 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados