É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Purificação de cromatina de locus gênico de cópia única em Saccharomyces cerevisiae
Neste Artigo
Resumo
Este protocolo apresenta um método de isolamento de cromatina locus-específico baseado em recombinação sítio-específica para purificar um locus gênico de cópia única de interesse em seu contexto de cromatina nativa a partir de levedura brotante, Saccharomyces cerevisiae.
Resumo
A unidade organizacional básica da cromatina eucariótica é a partícula núcleo do nucleossomo (NCP), que compreende DNA envolto ~1,7 vezes em torno de um octâmero de histona. A cromatina é definida como a entidade de NCPs e numerosos outros complexos proteicos, incluindo fatores de transcrição, remodelamento da cromatina e enzimas modificadoras. Ainda não está claro como essas interações proteína-DNA são orquestradas no nível de loci genômicos específicos durante diferentes estágios do ciclo celular. Isso se deve principalmente às limitações técnicas atuais, que tornam difícil obter medidas precisas dessas interações dinâmicas. Aqui, descrevemos um método aprimorado combinando recombinação sítio-específica com um eficiente protocolo de purificação de afinidade em etapa única para isolar um locus gênico de cópia única de interesse em seu estado nativo de cromatina. O método permite o enriquecimento robusto do locus alvo sobre a cromatina genômica, tornando esta técnica uma estratégia eficaz para identificar e quantificar interações proteicas de forma imparcial e sistemática, por exemplo, por espectrometria de massas. Além dessas análises composicionais, a cromatina nativa purificada por este método provavelmente reflete a situação in vivo em relação ao posicionamento do nucleossomo e modificações de histonas e, portanto, é passível de análises estruturais e bioquímicas adicionais da cromatina derivada de praticamente qualquer locus genômico em levedura.
Introdução
A organização dinâmica de genomas eucarióticos em cromatina compacta o DNA para caber dentro dos limites do núcleo, garantindo dinâmica suficiente para a expressão gênica e acessibilidade para fatores regulatórios. Em parte, essa versatilidade é mediada pelo nucleossomo, a unidade básica da cromatina, que compreende uma partícula central com 147 pb de DNA envolto ~1,7 vezes em torno do oitâmero de histona1. O nucleossomo é uma estrutura altamente dinâmica em relação à sua composição, com numerosas variantes de histonas e modificações pós-traducionais (MPTs) nas caudas das histonas N- e C-terminais. Além disso, a cromatina eucariótica interage com uma infinidade de outros componentes essenciais, tais como fatores de transcrição, maquinário de processamento de DNA e RNA, proteínas arquitetônicas, enzimas envolvidas no remodelamento e modificação da cromatina e moléculas de RNA associadas à cromatina. Essas máquinas cruciais envolvidas na transcrição, replicação e reparo requerem acesso à cromatina, que serve como substrato natural para esses processos. Consequentemente, compreender os mecanismos moleculares subjacentes a essas transações de DNA requer uma definição precisa das alterações coletivas na estrutura da cromatina nas regiões genômicas específicas onde essas máquinas convergem e facilitam as reações biológicas.
Apesar da identificação de numerosos fatores da cromatina através de estudos genéticos e de interação proteína-proteína, a realização de análises diretas, imparciais e abrangentes das interações da cromatina em sítios genômicos específicos tem permanecido um obstáculo significativo 2,3. Inicialmente, apenas regiões altamente abundantes do genoma (isto é, loci repetitivos) ou plasmídeos multicópias poderiam ser isolados em quantidade e pureza suficientes para a identificação espectrométrica de massa das proteínas associadas 4,5,6,7. Uma série de novas abordagens baseadas na hibridização direta de sondas de captura para DNA cromatinizado, biotinilação de proximidade usando o sistema CRISPR-dCas9 ou a ligação de proteínas adaptadoras sequenciais específicas ao locus de interesse começaram a desvendar o proteoma de loci de cópia única de genomas de leveduras e mamíferos8,9,10 . No entanto, todos esses métodos requerem reticulação de formaldeído para estabilizar as interações proteína-DNA e sonicação para solubilizar a cromatina para posterior purificação. Juntas, ambas as manipulações excluem a possibilidade de estudos estruturais e funcionais subsequentes da cromatina purificada.
Para superar essas limitações, desenvolvemos previamente uma metodologia que emprega recombinação sítio-específica para extrair domínios cromossômicos direcionados de leveduras11,12. Em essência, a região genômica de interesse é cercada por sítios de reconhecimento (RS) para a R-recombinase sítio-específica de Zygosaccharomyces rouxi, incorporando simultaneamente um grupo de três sítios de ligação ao DNA para a proteína LexA repressora transcricional procariótica (LexA) dentro da mesma região. As células de levedura contêm um de expressão para a expressão simultânea de R-recombinase e uma proteína LexA fundida a uma etiqueta de purificação de afinidade em tandem (TAP). Após a indução da R-recombinase, a enzima excisa eficientemente a região alvo do cromossomo na forma de um domínio circular da cromatina. Este domínio pode ser purificado através da proteína adaptadora LexA-TAP, que se liga aos sítios de ligação do ADN LexA, bem como a um suporte de afinidade. Este método tem sido recentemente utilizado para isolar domínios distintos da cromatina contendo origens de replicação selecionadas do cromossomo III da levedura13.
Uma grande vantagem desta abordagem ex vivo é que ela permite análises funcionais do material isolado. Por exemplo, domínios de origem de replicação purificados com este método podem ser submetidos a ensaios de replicação in vitro para avaliar a eficiência de disparo de origem em um tubo de ensaio a partir de modelos de cromatina montados in vivo nativos. Em última análise, a caracterização bioquímica e funcional do material isolado pode permitir a reconstituição de processos nucleares utilizando proteínas purificadas em conjunto com o molde de cromatina nativa. Em resumo, esta metodologia abre um caminho empolgante na pesquisa da cromatina, pois será possível acompanhar as mudanças composicionais e estruturais coletivas da cromatina de uma região genômica específica submetida a uma determinada transação cromossômica.
Protocolo
Consulte a Tabela de Materiais para obter detalhes relacionados a todos os materiais e instrumentos utilizados neste protocolo. Consulte a Tabela 1 para obter uma lista das soluções, buffers e mídia usados.
1. Construção de cepas de leveduras recombinantes
- Para construir uma linhagem de levedura competente em recombinação, transforme o plasmídeo K238 digerido por SbfI em uma linhagem de levedura com sítios de ligação de LexA integrados e sítios de recombinação de RS no locus de interesse13,14.
NOTA: O fragmento de restrição SbfI transformado contém o de expressão necessário para a expressão constitutiva da proteína de fusão LexA-TAP e da R-recombinase, juntamente com sequências homólogas do cromossoma I da levedura para integração genómica do de expressão por recombinação homóloga (Figura 1). - Para construir uma cepa controle, pegue uma cepa de levedura isogênica sem sítios integrados de ligação RS e LexA e transforme-a com o plasmídeo K238 digerido por SbfI nas mesmas condições usadas para a cepa competente em recombinação.
- Selecionar as células de levedura competentes com base no marcador de seleção LEU2 em placas de ágar SCD-LEU14.
2. Acoplamento de anticorpos IgG a esferas magnéticas epóxi-ativadas
NOTA: Acoplar anticorpos IgG contra esferas magnéticas epóxi-ativadas de acordo com o seguinte protocolo publicado11.
- Suspender 300 mg de esferas epóxi-ativadas em 10 mL de acetona a 50% (300 mg corresponde a ~5,1 × 10 contas10 ) em um tubo cônico de 50 mL. Agite vigorosamente em uma batedeira de vórtices.
- Centrifugar o tubo contendo as esferas a 820 × g e 4 °C durante 2 min. Remova o sobrenadante.
- Lavar as esferas 3x com 20 mL de tampão fosfato de sódio 0,1 M (pH 7,4). Retire o sobrenadante após cada etapa de lavagem por centrifugação, conforme descrito na etapa 2.2.
- Suspender as esferas em 16 mL de tampão fosfato de sódio 0,1 M (pH 7,4) e girar suavemente em estufa de hibridização por 5 min à temperatura ambiente.
- Dissolver as IgGs de coelho (100 mg) em 7 mL de dH2O (concentração final: 14 mg/mL).
- Centrifugar a suspensão de IgG a 13.000 × g e 4 °C por 10 min para esclarecer a suspensão.
- Transferir 3,5 mL do sobrenadante (correspondente a 50 mg de IgGs de coelho) para um novo tubo cônico de 50 mL.
- Diluir a solução de IgG com 9,85 mL de tampão fosfato de sódio 0,1 M (pH 7,4), seguido da adição gota a gota de 6,65 mL de sulfato de amônio 3 M em fosfato de sódio 0,1 M (pH 7,4) sob mistura suave.
NOTA: Evite adicionar sulfato de amônio rapidamente à solução, pois uma alta concentração local do sal causará a precipitação das IgGs de coelho. - Centrifugar a solução de IgG a 820 × g e 4 °C durante 3 minutos e adicionar o sobrenadante resultante à suspensão do cordão magnético.
- Incubar o tubo durante a noite ou, pelo menos, durante 18 h a 30 °C com rotação suave num forno de hibridização.
- Remova o sobrenadante conforme descrito no passo 2.2.
- Lavar as contas com 20 mL de glicina-HCl 100 mM (pH 2,5). Remova a solução rapidamente conforme descrito na etapa 2.2 para evitar a desnaturação dos polipeptídeos IgG.
- Lavar as contas uma vez com 20 mL de Tris-HCl 10 mM (pH 8,8). Aspirar o sobrenadante conforme descrito no passo 2.2.
- Adicionar 20 mL de solução de trietilamina 0,1M por 5-10 min sob rotação suave para inativar os grupos epóxi reativos residuais. Remova o sobrenadante conforme descrito no passo 2.2.
- Lave as contas 4x com 20 mL de PBS (pH 7,4) por 5 min com rotação suave. Retire o sobrenadante conforme descrito no passo 2.2 após cada etapa de lavagem.
- Lavar as contas 2x com 20 mL de PBS (pH 7,4) com Triton X-100 a 0,5% (p/v) por 5 min e 15 min cada em rotação suave em estufa de hibridização. Remova o sobrenadante conforme descrito no passo 2.2.
- Suspender as esferas em volume final de 16 mL de PBS (pH 7,4) com azida sódica a 0,02% (p/v). Conservar como alíquotas de 1 ml a 4 °C até à utilização.
3. Cultura e colheita de células de levedura
- Inocular o controle e a recombinação de cepas de leveduras competentes de placas YPD em 5 mL de meio YPR e incubar durante a noite a 30 °C e 200 rpm.
- Inocular 2 mL dessa cultura em 100 mL de meio YPR e incubar durante a noite a 30 °C e 200 rpm.
- Para cada cepa, dispensar 1.800 mL de meio YP autoclavado e 200 mL de solução de rafinose autoclavada (20% p/v) em cada um dos dois frascos de Erlenmeyer de 5 L (4 L de meio de cultura no total para cada cepa, divididos em dois frascos).
- Inocular as células de levedura em crescimento a OD600 0,2 em seu respectivo meio e incubar conforme descrito na etapa 3.1 por aproximadamente 6 h ou até que as células atinjam o ODdesejado 600 de 1,0. Para garantir o crescimento normal das células, livre de qualquer contaminação, verifique o DO em intervalos de 2 h.
- Adicionar 200 mL de galactose (20% p/v) para induzir a recombinação sítio-específica.
- Adicionar 110 μL (50 ng/mL) de YMFA (ao mesmo tempo que a galactose, passo 3.5) para prender as células na fase G1 e incubar por 2 h.
NOTA: A adição de YMFA às células depende das condições experimentais e da questão biológica a ser abordada. Precisamente, para este experimento, as células são presas na fase G1 para obter uma população celular homogênea com origens de replicação licenciadas; nesse processo, o complexo pré-replicativo liga-se às origens na fase G1 antes do início da replicação do DNA na fase S. - Transfira a suspensão da célula para baldes de centrífuga de 1 L e centrifuja a 6.000 × g e 4 °C por 10 min. Descarte o sobrenadante e ressuspenda os pellets celulares em um volume total de 10-15 mL de dH2O.
- Sele uma seringa de 25 mL com um plugue Luer e coloque-a dentro de um tubo cônico de 50 mL cheio de água. Transfira a suspensão da célula para o conjunto da seringa e centrifugar a 2.397 × g por 10 min à temperatura ambiente. Descarte o sobrenadante.
- Retire o plugue Luer da seringa e extruda as células em nitrogênio líquido para formar o "espaguete" celular.
NOTA: Usando 4 L de meio de cultura fornece um peso úmido de pellets de células finais de 7-10 g. - Transfira o espaguete de células congeladas para um tubo cônico de 50 mL e armazene a -80 °C até uso posterior.
4. Purificação do locus da cromatina
NOTA: Consulte a Figura 2 para obter uma visão geral esquemática das etapas envolvidas neste protocolo de purificação de cromatina locus-específico.
- Resfriar um moedor de café comercial moendo 30-50 g de gelo seco agitando vigorosamente o moedor de café 2x por 30 s de cada vez. Depois de resfriado, descarte o pó de gelo seco.
- Misture 3 g de espaguete de célula de levedura congelado com ~30-50 g de gelo seco no moedor de café pré-resfriado.
- Sele a junção entre a tampa e o moedor com parafilme para evitar a perda de pó de célula de levedura durante a moagem.
- Moer a mistura de gelo seco da célula de levedura 10x por 30 s de cada vez, com intervalos de 30 s entre cada rodada (estimativa de tempo total: ~10 min). Para evitar que o gelo seco e o pó celular grudem nas paredes do moedor de café, bata nas laterais do moedor de café continuamente durante a moagem.
- Transfira o pó de gelo seco da célula de levedura resultante para um copo de plástico usando uma espátula limpa e seca.
- Mantenha os copos à temperatura ambiente para evaporar o gelo seco.
- Uma vez que o gelo seco evaporar, adicione 2,25 mL de tampão MB com 1x inibidores de protease e fosfatase (750 μL/g de células de levedura) ao pó de células de levedura.
- Pipetar a mistura célula-tampão vigorosamente para garantir a suspensão completa das células com o tampão e transferir para um tubo cônico de 15 mL.
- Coletar amostras para análise de DNA (0,1%) e proteína (0,05%) da suspensão celular resultante (extratos de células brutas [CCE]). Conservar a -20 °C até nova transformação (descrito na secção 7).
- Transferir a suspensão celular para tubos de reação de 2 mL de baixa ligação e centrifugar a 21.130 × g por 30 min a 4 °C para separar os restos celulares do lisado bruto de células.
- Junte os sobrenadantes de todos os tubos em um tubo cônico de 15 mL.
- Colher amostras do sobrenadante (entrada [IN]) para análise de DNA (0,1%) e proteína (0,05%). Conservar a -20 °C até nova transformação (descrito na secção 7).
- Equilibrar 500 μL de pasta magnética de esferas acoplada a IgG (para cada cepa de levedura) lavando 2x com 500 μL de tampão MB frio com 1x inibidores de protease e fosfatase por 5 min cada a 4 °C em rotação a 20 rpm. Descarte o sobrenadante das contas usando um rack magnético após cada etapa de lavagem.
- Incubar as esferas magnéticas acopladas a IgG em 500 μL de tampão MB frio com 1x inibidores de protease e fosfatase por 1 h a 4 °C em rotação a 20 rpm. Descarte o sobrenadante das contas usando um rack magnético.
- Misturar a pasta de esferas magnéticas equilibrada com o lisado celular e incubar com rotação a 20 rpm durante 2 h a 4 °C.
- Transfira a suspensão completa de lisado de células de esferas magnéticas para tubos de reação de baixa ligação.
- Separe as esferas magnéticas, agora carregando os anéis de cromatina de interesse, do lisado celular usando um rack magnético.
- Transferir o fluxo restante (FT) de cada tubo para um tubo cônico fresco de 15 mL e coletar amostras para análise de DNA (0,1%) e proteína (0,05%). Conservar a -20 °C até nova transformação (descrito na secção 7).
- Suspenda as esferas magnéticas de cada tubo em 300 μL de tampão MB frio e junte as contas em um tubo de reação. Lavar 5x por 10 min cada com 750 μL de tampão MB frio com 1x inibidores de protease e fosfatase a 4 °C em rotação a 20 rpm. Realizar a lavagem final com 750 μL de tampão MB frio sem inibidores de protease e fosfatase.
- Suspender as esferas em 40 μL de tampão MB frio sem inibidores de protease e fosfatase.
NOTA: O volume final de eluato pode ser ajustado de acordo com a sua aplicação a jusante prevista. A eluição do TEV geralmente funciona eficientemente dentro da faixa de 100-300 μL.
5. Eluição mediada por protease TEV
- Para liberar complexos de anéis de cromatina LexA-CBP das contas, incube as esferas com 2 μL de protease TEV recombinante marcada com 6x His durante a noite a 4 °C e 450 rpm em um termomisturador.
- Separe as contas do eluato final usando um rack magnético. Transferir o eluato, contendo anéis de cromatina clivados, para um novo tubo de reação de 1,5 mL.
- Mantenha os tubos em um rack magnético novamente para separar quaisquer contas residuais do eluato final.
- Ressuspender as esferas (TB) em 750 μL de tampão AC frio e colher amostras para análise de DNA (0,1%) e proteína (0,05%). Conservar a -20 °C até nova transformação (descrito na secção 7).
- A partir do eluato final (TE), colher amostras para análise de DNA (0,5%) e proteína (0,25%). Conservar a -20 °C até nova transformação (descrito na secção 7).
NOTA: Devido ao baixo volume do eluato final, a porcentagem das amostras coletadas para análise de DNA e proteínas é aumentada para obter uma melhor representação de seu conteúdo de proteína e DNA durante a análise.
6. Eluição da desnaturação
- Lavar as esferas 2x em 750 μL de tampão CA frio durante 20 min de cada vez a 4 °C em rotação a 20 rpm.
- Para extrair os complexos LexA-cromatina ligados restantes, realizar a eluição da desnaturação adicionando 500 μL de NH4OH 0,5M às esferas e incubar por 30 min à temperatura ambiente.
- Separe as esferas da suspensão usando um rack magnético e transfira o eluato para um tubo de reação de baixa ligação.
- Incubar novamente as esferas como no passo 6.2 para recuperar os loci máximos de cromatina no eluato.
- Separe as esferas da suspensão usando um rack magnético e junte o eluato resultante no mesmo tubo contendo o eluato da etapa 6.3.
- Ressuspender as esferas (DB) em 750 μL de dH2O e colher amostras para análise de DNA (0,1%) e proteína (0,05%).
- A partir do eluato final (DE), colher amostras para análise de DNA (0,5%) e proteína (0,25%).
7. Análise de DNA e proteínas
- Análise de DNA
- Preparo da amostra
- Às amostras de DNA, adicionar 100 μL de tampão IRN e aumentar o volume com dH2O para um volume final de 200 μL.
- Adicione 1 ng de DNA plasmidial de espícula K071.
- Adicionar 1 μL de RNAse A (10 mg/mL) e incubar a 37 °C por 1 h.
- Adicionar 5 μL de proteinase K (10 mg/mL) e 10 μL de SDS (20%) e incubar por 1 h a 56 °C.
- Adicionar 200 μL de fenol/clorofórmio/álcool isopropílico (25:24:1) e agitar completamente 2x por 10 s de cada vez.
- Centrifugar as amostras a 21.130 × g por 7 min para separar as fases orgânica e aquosa.
- Transferir o sobrenadante para novos tubos de 1,5 mL contendo 1,5 μL de glicogênio (5 mg/mL) e 2,5 x 100% de etanol.
- Incubar os tubos a -20 °C durante um mínimo de 2 h e um máximo de durante a noite para precipitar o ADN.
- Centrifugar a 21.130 × g, 4 °C por 45 min. Descarte o sobrenadante.
- Adicionar 150 μL de etanol a 70% sem ressuspender o pellet de DNA e centrifugar novamente a 21.130 × g a 4 °C por 10 min.
- Secar os pellets de DNA à temperatura ambiente por 10-15 min e ressuspender em 40 μL de dH2O.
- Realizar digestão enzimática de restrição para linearizar o DNA isolado na etapa 7.1.1.11 com endonuclease de restrição HpaI. Para uma mistura de reação de 50 μL, incubar 40 μL de DNA isolado + 2 μL de enzima HpaI + 5 μL de tampão de enzima de restrição + 3 μL de dH2O durante a noite a 37 °C.
- Análise de qPCR
- Diluir o ADN digerido por restrição numa proporção de 1:5 (10 μL do ADN digerido por restrição obtido a partir da etapa 7.1.1.12 em 40 μL de dH2O).
- Use pares de primers projetados para a região ARS316, PDC1 e DNA plasmidial spike-in.
- Execute o qPCR usando o programa fornecido na Tabela 2.
- Analisar os resultados da qPCR por quantificação relativa usando a região ARS316 e o DNA plasmidial spike-in como alvos e PDC1 (ou qualquer outro gene ou região genômica) como locus de referência.
- Avaliar a porcentagem de recuperação do locus ARS316 sobre PDC1 normalizando os valores de quantificação relativa dos primers ARS316 e PDC1 pela porcentagem do volume da fração tomada para cada amostra. Por exemplo, multiplique 0,1% para o flow-through por um fator de 1.000 e 0,5% para o eluato de TEV por um fator de 200.
- Normalize as porcentagens de recuperação ARS316 e PDC1 com os valores de quantificação relativa do DNA plasmidial spike-in.
- Avaliar o enriquecimento do locus ARS316 sobre a cromatina total, avaliando os valores-alvo de quantificação relativa/de referência obtidos usando a equação (1).
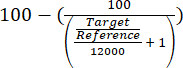 (1º)
(1º)
- Análise de Southern blot
- Para 20 μL de amostra de DNA digerido por enzima de restrição, adicionar 5 μL de corante de carga de gel.
- Executar as amostras em gel de agarose a 1% a 110 V por ~3 h.
- Transfira o gel para uma bandeja e mergulhe-o em solução de depuração com agitação suave por 20 min.
- Enxaguar o gel com dH2O e mergulhá-lo em solução de desnaturação por 15 min com agitação. Descarte a solução de desnaturação.
- Mergulhe o gel em solução de desnaturação por 15 min com agitação suave.
- Enxaguar o gel com dH2O e lavá-lo 2x por 15 min cada vez com o tampão de transferência com agitação suave.
- Encha uma bandeja com 1-1,5 L de buffer de transferência e coloque-a em uma plataforma estável.
- Corte um pedaço longo de papel Whatman e coloque-o na plataforma de tal forma que as duas extremidades do papel fiquem embebidas no buffer de transferência.
- Corte uma tira de membrana de nylon positiva e quatro tiras de papel Whatman igual ao tamanho do gel.
- Coloque dois pedaços de papel Whatman embebidos no buffer de transferência na plataforma.
- Coloque o gel virado para baixo em cima dos pedaços de papel Whatman.
- Por cima do gel, coloque a membrana de nylon positiva, seguida dos dois pedaços restantes de papel Whatman embebidos em tampão de transferência. Certifique-se de manter a pilha molhada com o buffer de transferência.
- Remova quaisquer bolhas de ar presas rolando-as usando uma haste de vidro.
- Coloque uma pilha de toalhas de papel em cima da pilha montada, seguida de um objeto de 0,5 kg (por exemplo, uma garrafa de vidro).
- Deixe o conjunto para transferência durante a noite à temperatura ambiente.
- Após a transferência, reticule UV a membrana com uma saída de energia total de 1.200 J/cm2.
- Para a detecção do locus ARS316, realizar a hibridização southern blot utilizando sondas radioativas sintetizadas usando o sistema de marcação de DNA referenciado.
NOTA: Execute todas as etapas a seguir no gelo até mencionar o contrário. - Em um tubo de reação de baixa ligação, diluir 25-40 ng do fragmento de PCR (sonda) em 19 μL de dH2O e incubar a 95 °C por 5 min. Resfriar imediatamente no gelo.
- Adicionar 1 μL cada de 500 μM dCTP, 500 μM dGTP e 500 μM dTTP ao tubo.
- Adicionar 20 μL do tampão contendo hexâmeros nucleotídicos aleatórios fornecidos com o kit.
- Adicionar 5 μL do nucleotídeo radioativamente marcado [α-32P] dATP ao tubo. Executar todas as etapas que envolvem material marcado radioativamente sob um ambiente de proteção aprovado por radioatividade.
- Adicionar 1 μL de fragmento de Klenow e incubar a 37 °C num termomisturador durante 15 minutos para síntese de ADN de fita simples.
- Adicionar 5 μL de tampão de paragem para parar a reacção.
- Centrifugar uma coluna de exclusão de tamanho por 2 min a 1.500 × g para remover o buffer de armazenamento. Descarte o fluxo e coloque a coluna em um tubo novo.
- Passe a mistura de sonda através da coluna para remover nucleotídeos radioativos não incorporados. Centrifugar por 30 s a 1.500 × g para coletar a sonda.
- Adicionar 150 μL de ADN de esperma de salmão (1:100) para bloquear a ligação inespecífica da sonda à membrana.
- Incubar a mistura de sonda a 95 °C durante 5 min para desnaturar o ADN de fita dupla.
- Congele no gelo por alguns segundos e centrifugue por alguns segundos a 500 × g para derrubar as gotículas de água condensadas na tampa.
- Lavar brevemente a membrana do Southern blot com o tampão de hibridização por 5-10 min com rotação.
- Pré-hibridizar a membrana com o tampão de hibridização por 1 h a 55 °C com rotação. Descarte o buffer de hibridização.
- Adicionar 15 ml de tampão de hibridização fresco (pré-aquecido a 55 °C) à membrana juntamente com a sonda preparada. Incubar a membrana durante a noite a 55 °C com rotação.
- Realizar lavagens pós-hibridização 2x por 15 min cada com 0,3x SSC, 0,1% SDS a 55 °C com rotação.
- Realizar lavagens pós-hibridização 2x por 15 min cada com 0,1x SSC, 0,1% SDS a 55 °C com rotação.
- Realizar lavagens pós-hibridização 2x por 15 min cada com 0,1x SSC, 1,5% SDS a 55 °C com rotação.
- Remova a membrana do tubo de hibridização e exponha a membrana a uma tela de fosforimageador por até 3 dias para obter as impressões radioativas no filme.
- Adquira imagens do filme usando um sistema de varredura a laser de imagem de fósforo.
- Preparo da amostra
- Análise de proteínas
- Preparo da amostra
- Ajustar todas as amostras de proteína aos seus volumes finais (200 μL, 100 μL, 100 μL, 20 μL e 20 μL para as amostras de extrato bruto de células, entrada, fluxo, contas e eluato, respectivamente) adicionando 1 μL de β-mercaptoetanol, tampão de amostra PAGE LDS (1x) e dH2O.
- Desnaturar as amostras a 95 °C durante 5 min.
- Análise de Western blot
NOTA: Executar SDS-PAGE e western blotting usando protocolos padrão15,16.- Carregar 15 μL de cada amostra em cada poço de um gel SDS-PAGE de 1,5 mm.
- Realizar a imunomarcação do blot usando anticorpos PAP (1:2.000) e LexA (1:1.000) com incubação noturna a 4 °C. Diluir em solução de leite a 5% em pó/PBST.
NOTA: Após a análise PAP, o blot pode ser removido usando um protocolo de stripping leve17 antes da imunomarcação com o segundo anticorpo LexA.
- Preparo da amostra
Resultados
A purificação do domínio da cromatina ARS316 de ~1,4 kb foi mediada pela proteína adaptadora LexA-TAP constitutivamente expressa. Para servir como controle negativo, realizamos purificações usando uma cepa isogênica que expressa LexA-TAP, mas não contém sítios integrados de ligação de RS e LexA. A Figura 3 ilustra o resultado da análise de DNA de um experimento de purificação padrão realizado tanto no controle quanto em uma cepa competente em recombinação visando o locus AR...
Discussão
A identificação dos fatores e da paisagem da cromatina de uma região genômica alvo específica continua a representar um grande desafio na pesquisa da cromatina18. Este protocolo descreve um sistema eficiente para especificamente excisar e purificar domínios distintos da cromatina dos cromossomos de leveduras. Até onde sabemos, a pureza e o rendimento desta purificação em etapa única superam muitas das limitações dos métodos de purificação de cromatina locus-específicos, permitindo ...
Divulgações
Os autores não têm conflitos de interesse a declarar.
Agradecimentos
O trabalho no laboratório de S.H. foi apoiado pela DFG através do SFB1064 (projeto ID 213249687), do Conselho Europeu de Pesquisa (ERC Starting Grant 852798 ConflictResolution) e da Helmholtz Gesellschaft.
Materiais
| Name | Company | Catalog Number | Comments |
| Yeast strains | |||
| Control Strain: MATa; ura3Δ0; leu2Δ0; his3Δ1; met15Δ0; bar1::kanMX4; Chr I 212kb::LEU2 pTEF2-LEXA-TAP pGAL1-10 RecR | Section 1, see references 13 and 14 | ||
| Recombination Strain: MATa; ura3Δ0; leu2Δ0; his3Δ1; met15Δ0; bar1::kanMX4; RS_LEXA_NS-3_ARS316_NS+3_RS; Chr I 212kb::LEU2 pTEF2-LEXA-TAP pGAL1-10 RecR | Section 1, see reference 13 and 14 | ||
| Plasmid | |||
| K238 plasmid | Section 1, see reference 13 Storage: Store at -20 °C | ||
| K071 Spike-in plasmid DNA | Section 7.1, see reference 13 Storage: Store at -20 °C | ||
| Reagents | |||
| Acetone | Carl Roth | 5025.1 | Section 2 Storage: Store at room temperature |
| Ammonium acetate (NH4Ac) | Sigma Aldrich | A7262 | Section 6 and 7.1 Storage: Store at room temperature |
| Ammonium solution (NH4OH) 25% | Merck Millipore | 533003 | Section 6 Storage: Store at room temperature |
| Ammonium sulfate | Santa Cruz | Sc-29085 | Section 2 Storage: Store at room temperature |
| Bacto agar | BD (VWR) | 90000-760 | Section 3 Storage: Store at room temperature |
| Bacto peptone | BD (VWR) | 211820 | Section 3 Storage: Store at room temperature |
| β-Mercaptoethanol | Sigma Aldrich | 07604 | Section 7.2 Storage: Store at 4 °C |
| Chemiluminescent substrate kit | ThermoFisher | 34580 | Section 7.2 Storage: Store at 4 °C |
| Di-Sodium Hydrogen phosphate dodecahydrate | Merck | 1.06579.1000 | Section 2 and 7.1 Storage: Store at room temperature |
| Dithiothreitol (DTT) | ThermoFisher | 15508013 | Section 4 Storage: Store at 4 °C |
| Ethanol | Merck | 100983 | Section 7.1 Storage: Store at room temperature |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma Aldrich | ED | Section 7.1 Storage: Store at room temperature |
| Galactose (20% (w/v) stock) | Sigma Aldrich | G0625-1KG / 5KG | Section 3 Storage: Store at room temperature |
| Gel loading dye (6x) | BioLabs | B7024A | Section 7.1 Storage: Store at -20 °C |
| Glusose | Sigma-Aldrich | G8270 | Section Storage: Store at room temperature |
| Glycine | Carl Roth | .0079.4 | Section 2 Storage: Store at room temperature |
| Glycogen (5 mg/mL) | Invitrogen | AM9510 | Section 7.1 Storage: Store at -20 °C |
| Hydrochloric acid (HCl) | PanReac AppliChem | 182109.1211 | Section 2, 4 and 7.1 Storage: Store at room temperature |
| Magnesium Acetate (MgAc) | Bernd Kraft | 15274.2600/C035 | Section 4 Storage: Store at room temperature |
| Magnesium chloride (MgCl2) | Sigma Aldrich | M8266 | Section 6 Storage: Store at room temperature |
| Nu PAGE LDS sample buffer (4x) | Invitrogen | 2399549 | Section 7.2 Storage: Store at room temperature |
| Phenol/Chloroform/Isoamyl alcohol (25:24:1 v/v) | Invitrogen | 15593-031 | Section 7.1 Storage: Store at 4 °C |
| Potassium chloride (KCl) | Sigma | P9541 | Section 4 Storage: Store at room temperature |
| Radioactively labeled α-32P dATP (3,000 Ci/mmol, 10 mCi/mL) | Hartmann Analytic | SRP-203 | Section 7.1 Storage: Store at 4 °C |
| RadPrime labeling system | ThermoFisher | 18428-011 | Section 7.1 Storage: Store at -20 °C |
| Raffinose (20% (w/v) stock) | SERVA | 34140.03 | Section 3 Storage: Store at room temperature |
| Sodium chloride (NaCl) | Merck | K53710504142 | Section 7.1 Storage: Store at room temperature |
| Sodium citrate (Na3C6H5O7) | Sigma-Aldrich | 71402 | Section 7.1 Storage: Store at room temperature |
| Sodium hydroxide (NaOH) | Sigma Aldrich | S5881 | Section 7.1 Storage: Store at room temperature |
| Sodium n-dodecyl sulfate (SDS) (5% stock (w/v) ) | Alfa Aesar | A11183 | Section 7.1 Storage: Store at room temperature |
| Sodium phosphate monobasic | Sigma-Aldrich | 71496 | Section 2 and 7.1 Storage: Store at room temperature |
| Sodium azide | Santa Cruz Biotechnology | sc-208393 | Section 2 Storage: Store at -20 °C |
| Triethylamine | Sigma Aldrich | 90340 | Section 2 Storage: Store at room temperature |
| Tris base | Chem Cruz | SC-3715B | Section 2 and 4 Storage: Store at room temperature |
| Triton X-100 | Sigma Aldrich | X100 | Section 2 and 4 Storage: Store at room temperature |
| Tween-20 | Bernd Kraft | 18014332 | Section 4 Storage: Store at room temperature |
| Yeast extract | BD (VWR) | 212720 | Section 3 Storage: Store at room temperature |
| Yeast mating factor alpha (1 µg/mL stock ) | Biomol | Y2016.5 | Section 3 Storage: Store at -20 °C |
| Yeast Synthetic Drop-out medium Supplements without LEUCINE | Sigma Aldrich | Y1376 | Section 1, see reference 14 |
| Enzymes | |||
| HpaI restriction enzyme (5,000 U/mL) | NEB | R0105S | Section 7.1 Storage: Store at -20 °C |
| Protease and Phosphatase Inhibitor Cocktail (100x) | ThermoFisher Scientific | 78446 | Section 4 Storage: Store at4 °C |
| Proteinase K (10 mg/mL) | SERVA | 33756 | Section 7.1 Storage: Store at -20 °C |
| RNase A (10 mg/mL) | ThermoFisher | EN0531 | Section 7.1 Storage: Store at -20 °C |
| TEV protease (10000 U/µL) | NEB | P8112S | Section 5 Storage: Store at -20 °C |
| Materials | |||
| BcMag Epoxy-Activated Magnetic Beads | Bioclone Inc. | FC-102 | Section 2 Storage: Store at 4 °C |
| Dry ice | Section 4 | ||
| Low-binding centrifuge tubes 2.0 mL | Eppendorf | 22431102 | Section 4 |
| Microspin G-25 Columns | Cytiva | 27-5325-01 | Section 7.1 Storage: Store at room temperature |
| Parafilm | Merck | P7793 | Section 4 |
| Positive nylon membrane | Biozol | 11MEMP0001 | Section 7.1 Storage: Store at room temperature |
| PVDF transfer membrane | Immobilon-Merck Millipore | IPVH00010 | Section 7.2 Storage: Store at room temperature |
| SDS-PAGE gel 4-12% bis-tris (15 well, 1.5 mm) | Invitrogen | NP0336BOX | Section 7.2 Storage: Store at 4 °C |
| Syringe (25 mL) with luer fitting | Henke Sass Wolf | 4200-000V0 | Section 3 |
| Whatman paper (Grade 3MM CHR Cellulose Western Blotting Paper Sheet) | Cytiva | 3030-917 | Section 7.1 Storage: Store at room temperature |
| Antibodies | |||
| Anti-LexA, rabbit polyclonal IgG, DNA binding region antibody | Merck Millipore | 06-719 | Section 7.2 Storage: Store at -20 °C |
| Goat Anti-Rabbit IgG (H+L), Horseradish peroxidase conjugate | Invitrogen | G21234 | Section 7.2 Storage: Store at -20 °C |
| Peroxidase Anti-Peroxidase (PAP) antibody produced in rabbit for the detection of TAP-tagged proteins | Sigma Aldrich | P1291-500UL | Section 7.2 Storage: Store at -20 °C |
| Rabbit IgG antibodies | Sigma | I5006-100MG | Section 2 Storage: Store at 4 °C |
| Primers (10 µM) | |||
| ARS316: fwd 5'- CGGCATTATCGTACACAACCT, rev 5'- GTTCTTCGTTGCCTACATTTTCT | Section 7.1 | ||
| K071 Spike-in plasmid DNA: fwd: 5'-TTTTCGCTGCTTGTCCTTTT, rev 5'- CATTTTCGTCCTCCCAACAT | Section 7.1 | ||
| PCR fragment from yeast genomic DNA as a template for ARS316 amplification (for southern blot): fwd 5’- AAATTCTGCCCTTGATTCGT rev 5’- TTTGTTTATCTCATCACTAAT | Section 7.1 | ||
| PDC1: fwd 5'- CATGATCAGATGGGGCTTCA, rev 5'-ACCGGTGGTAGCGACTCTGT | Section 7.1 | ||
| Equipment | |||
| Coffee grinder | Gastroback | 42601 | Section 4 |
| Dewar flask | NAL GENE | 4150-2000 | Section 3 |
| DynaMag TM-2 magnetic rack | Invitrogen | 12321D | Section 4, 5 and 6 |
| Hybridization oven | Hybaid Mini10 | Ri418 | Section 2 |
| Microcentrifuge | Eppendorf | 5424R | Section 4 and 7.1 |
| UV-crosslinker | Analytikjena | 95-0174-02 | Section 7.1 |
Referências
- Kornberg, R. D., Lorch, Y. Twenty-five years of the nucleosome, fundamental particle of the eukaryote chromosome. Cell. 98 (3), 285-294 (1999).
- Gauchier, M., van Mierlo, G., Vermeulen, M., Déjardin, J. Purification and enrichment of specific chromatin loci. Nature Methods. 17 (4), 380-389 (2020).
- Korthout, T., et al. Decoding the chromatin proteome of a single genomic locus by DNA sequencing. PLoS Biology. 16 (7), e2005542 (2018).
- Antão, J. M., Mason, J. M., Déjardin, J., Kingston, R. E. Protein landscape at Drosophila melanogaster telomere-associated sequence repeats. Molecular and Cellular Biology. 32 (12), 2170-2182 (2012).
- Déjardin, J., Kingston, R. E. Purification of proteins associated with specific genomic loci. Cell. 136 (1), 175-186 (2009).
- Ide, S., Dejardin, J. End-targeting proteomics of isolated chromatin segments of a mammalian ribosomal RNA gene promoter. Nature Communications. 6, 6674 (2015).
- Unnikrishnan, A., Gafken, P. R., Tsukiyama, T. Dynamic changes in histone acetylation regulate origins of DNA replication. Nature Structural & Molecular Biology. 17 (4), 430 (2010).
- Buxton, K. E., et al. Elucidating protein-DNA interactions in human alphoid chromatin via hybridization capture and mass spectrometry. Journal of Proteome Research. 16 (9), 3433-3442 (2017).
- Kennedy-Darling, J., et al. Discovery of chromatin-associated proteins via sequence-specific capture and mass spectrometric protein identification in Saccharomyces cerevisiae. Journal of Proteome Research. 13 (8), 3810-3825 (2014).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355, 7111 (2017).
- Hamperl, S., et al. Purification of specific chromatin domains from single-copy gene loci in Saccharomyces cerevisiae. Functional Analysis of DNA and Chromatin. 1094, 329-341 (2014).
- Hamperl, S., et al. Compositional and structural analysis of selected chromosomal domains from Saccharomyces cerevisiae. Nucleic Acids Research. 42 (1), 2 (2014).
- Weiβ, M. Single-copy locus proteomics of early- and late-firing DNA replication origins identifies a role of Ask1/DASH complex in replication timing control. Cell Reports. 42 (2), 112045 (2023).
- Gietz, R. D., Schiestl, R. H. High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method. Nature Protocols. 2 (1), 31-34 (2007).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. Proceedings of the National Academy of Sciences of the United States of America. 76 (9), 4350-4354 (1979).
- Western blot membrane stripping for restaining protocol. Abcam Available from: https://www.abcam.com/protocols/western-blot-membrane-stripping-for-restaining-protocol (2023)
- Vermeulen, M., Déjardin, J. Locus-specific chromatin isolation. Nature Reviews. Molecular Cell Biology. 21 (5), 249-250 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados