Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Очистка хроматина локуса гена с одной копией у Saccharomyces cerevisiae
В этой статье
Резюме
В этом протоколе представлен локусоспецифический метод выделения хроматина, основанный на сайт-специфической рекомбинации, для очистки однокопийного гена, представляющего интерес в его нативном контексте хроматина, от почковавшихся дрожжей Saccharomyces cerevisiae.
Аннотация
Основной организационной единицей эукариотического хроматина является частица ядра нуклеосомы (NCP), которая состоит из ДНК, обернутой ~1,7 раза вокруг октамера гистона. Хроматин определяется как сущность NCP и многих других белковых комплексов, включая транскрипционные факторы, ремоделирование хроматина и модифицирующие ферменты. До сих пор неясно, как эти белок-ДНК-взаимодействия организуются на уровне специфических геномных локусов на разных стадиях клеточного цикла. В основном это связано с текущими техническими ограничениями, которые затрудняют получение точных измерений таких динамических взаимодействий. В этой статье мы опишем усовершенствованный метод, сочетающий сайт-специфичную рекомбинацию с эффективным одноступенчатым протоколом аффинной очистки для выделения интересующего нас однокопийного гена в его нативном хроматиновом состоянии. Этот метод позволяет надежно обогащать целевой локус с помощью геномного хроматина, что делает этот метод эффективной стратегией для идентификации и количественной оценки белковых взаимодействий непредвзятым и систематическим образом, например, с помощью масс-спектрометрии. В дополнение к такому анализу состава, нативный хроматин, очищенный этим методом, вероятно, отражает ситуацию in vivo в отношении позиционирования нуклеосом и модификаций гистонов и, следовательно, поддается дальнейшему структурному и биохимическому анализу хроматина, полученного практически из любого геномного локуса у дрожжей.
Введение
Динамическая организация эукариотических геномов в хроматин уплотняет ДНК, чтобы она помещалась в пределах ядра, обеспечивая при этом достаточную динамику для экспрессии генов и доступность регуляторных факторов. Отчасти эта универсальность опосредована нуклеосомой, основной единицей хроматина, которая состоит из основной частицы со 147.н. ДНК, обернутой ~1,7 раза вокруг октамера гистонов1. Нуклеосома представляет собой очень динамичную структуру по отношению к своему составу, с многочисленными вариантами гистонов и посттрансляционными модификациями (ПТМ) на N- и C-концевых хвостах гистонов. Кроме того, эукариотический хроматин взаимодействует с множеством других важных компонентов, таких как транскрипционные факторы, механизмы обработки ДНК и РНК, архитектурные белки, ферменты, участвующие в ремоделировании и модификации хроматина, и молекулы РНК, связанные с хроматином. Эти важнейшие механизмы, участвующие в транскрипции, репликации и репарации, требуют доступа к хроматину, который служит естественным субстратом для этих процессов. Следовательно, понимание молекулярных механизмов, лежащих в основе этих трансакций ДНК, требует точного определения коллективных изменений в структуре хроматина в конкретных областях генома, где эти механизмы сходятся и способствуют биологическим реакциям.
Несмотря на идентификацию многочисленных факторов хроматина с помощью генетики и исследований белок-белкового взаимодействия, проведение прямого, непредвзятого и всестороннего анализа взаимодействий хроматина на конкретных участках генома остается существенным препятствием 2,3. Первоначально для масс-спектрометрической идентификации ассоциированных белков 4,5,6,7 можно было выделить только очень распространенные участки генома (т.е. повторяющиеся локусы) или многокопийные плазмиды. Ряд новых подходов, основанных на прямой гибридизации зондов захвата с хроматинизированной ДНК, бесконтактном биотинилировании с использованием системы CRISPR-dCas9 или связывании последовательно-специфических белков-адаптеров с интересующим локусом, начал распутывать протеом однокопийных локусов из геномов дрожжей и млекопитающих 8,9,10. Однако все эти методы требуют сшивания формальдегида для стабилизации белок-ДНК взаимодействий и ультразвуковой обработки для солюбилизации хроматина для последующей очистки. В совокупности обе манипуляции исключают возможность последующих структурно-функциональных исследований очищенного хроматина.
Чтобы преодолеть эти ограничения, мы ранее разработали методологию, которая использует сайт-специфическую рекомбинацию для извлечения целевых хромосомных доменов из дрожжей11,12. По сути, интересующая область генома окружена сайт-специфической R-рекомбиназой из Zygosaccharomyces rouxi и одновременно включает группу из трех сайтов связывания ДНК для прокариотического транскрипционного репрессора белка LexA (LexA) в пределах одной и той же области. Дрожжевые клетки содержат экспрессионную кассету для одновременной экспрессии R-рекомбиназы и белка LexA, соединенного с меткой тандемной аффинной очистки (TAP). После индукции R-рекомбиназы фермент эффективно удаляет целевой участок из хромосомы в виде кольцевого домена хроматина. Этот домен может быть очищен с помощью белка-адаптера LexA-TAP, который связывается с сайтами связывания ДНК LexA, а также с аффинной опорой. Этот метод недавно был использован для выделения отдельных доменов хроматина, содержащих выбранные репликационные источники13-й хромосомы дрожжей.
Одним из основных преимуществ этого подхода ex vivo является то, что он позволяет проводить функциональный анализ изолированного материала. Например, репликационные исходные домены, очищенные с помощью этого метода, могут быть подвергнуты анализу репликации in vitro для оценки эффективности возбуждения исходного сырья в пробирке из нативных хроматиновых матриц in vivo . В конечном счете, биохимическая и функциональная характеристика выделенного материала может позволить восстановить ядерные процессы с использованием очищенных белков вместе с нативным хроматиновым шаблоном. Таким образом, эта методология открывает захватывающие возможности в исследовании хроматина, поскольку можно будет проследить коллективные изменения состава и структуры хроматина в определенной области генома, претерпевающей определенную хромосомную трансакцию.
протокол
Смотрите Таблицу материалов для получения подробной информации, связанной со всеми материалами и инструментами, используемыми в этом протоколе. В таблице 1 приведен список используемых решений, буферов и носителей.
1. Построение штамма рекомбинантных дрожжей
- Чтобы сконструировать рекомбинационно-компетентный штамм дрожжей, трансформируют SbfI-переваренную плазмиду K238 в штамм дрожжей с интегрированными LexA-связывающими сайтами и сайтами рекомбинации RS в интересующем локусе13,14.
ПРИМЕЧАНИЕ: Трансформированный фрагмент рестрикции SbfI содержит необходимую экспрессионную кассету для конститутивной экспрессии белка слияния LexA-TAP и R-рекомбиназы вместе с гомологичными последовательностями хромосомы I дрожжей для геномной интеграции экспрессионной кассеты путем гомологичной рекомбинации (рис. 1). - Для создания контрольного штамма берут изогенный штамм дрожжей, в котором отсутствуют интегрированные RS- и LexA-связывающие сайты, и трансформируют его с SbfI-расщепленной плазмидой K238 в тех же условиях, что и для рекомбинационно-компетентного штамма.
- Выберите компетентные дрожжевые клетки на основе маркера отбора LEU2 на агаровых пластинах SCD-LEU14.
2. Связывание антител IgG с эпоксидно-активированными магнитными шариками
ПРИМЕЧАНИЕ: Соедините антитела IgG с магнитными шариками, активированными эпоксидной смолой, в соответствии со следующим опубликованным протоколом11.
- Суспендировать 300 мг гранул, активированных эпоксидной смолой, в 10 мл 50% ацетона (300 мг соответствует ~5,1 ×10 10 шариков) в конической пробирке объемом 50 мл. Энергично встряхните на вихревом миксере.
- Центрифугируют пробирку с гранулами при 820 × г и 4 °C в течение 2 мин. Удалите надосадочную жидкость.
- Промойте шарики 3 раза 20 мл 0,1 М натрий-фосфатного буфера (pH 7,4). Удаляйте надосадочную жидкость после каждого этапа промывки центрифугированием, как описано в шаге 2.2.
- Суспендируйте шарики в 16 мл 0,1 М натрий-фосфатного буфера (pH 7,4) и осторожно вращайте в гибридизационной печи в течение 5 минут при комнатной температуре.
- Растворите IgG кролика (100 мг) в 7 мл dH2O (конечная концентрация: 14 мг/мл).
- Центрифугируют суспензию IgG при 13 000 × г и 4 °C в течение 10 мин для осветления суспензии.
- Переложите 3,5 мл надосадочной жидкости (что соответствует 50 мг кроличьих IgG) в новую коническую пробирку объемом 50 мл.
- Раствор IgG разбавляют 9,85 мл 0,1 М натрийфосфатного буфера (рН 7,4) с последующим добавлением по каплям 6,65 мл 3 М сульфата аммония в 0,1 М фосфата натрия (рН 7,4) при осторожном перемешивании.
ПРИМЕЧАНИЕ: Избегайте быстрого добавления сульфата аммония в раствор, так как высокая локальная концентрация соли вызовет осаждение IgG кролика. - Центрифугируют раствор IgG при 820 × g и 4 °C в течение 3 мин и добавляют полученную надосадочную жидкость к суспензии магнитного шарика.
- Инкубируйте пробирку в течение ночи или не менее 18 ч при 30 °C при осторожном вращении в гибридизационной печи.
- Удалите надосадочную жидкость, как описано в шаге 2.2.
- Промойте шарики 20 мл 100 мМ глицина-HCl (рН 2,5). Быстро удалите раствор, как описано в шаге 2.2, чтобы избежать денатурации полипептидов IgG.
- Промойте бусины один раз 20 мл 10 мМ Tris-HCl (pH 8,8). Аспирируйте надосадочную жидкость, как описано в шаге 2.2.
- Добавьте 20 мл 0,1М раствора триэтиламина в течение 5-10 мин при осторожном вращении для инактивации остаточных реакционноспособных эпоксидных групп. Удалите надосадочную жидкость, как описано в шаге 2.2.
- Промойте бусины 4 раза 20 мл PBS (pH 7,4) в течение 5 минут с легким вращением. Удаляйте надосадочную жидкость, как описано в шаге 2.2, после каждого этапа стирки.
- Промойте бусины 2 раза 20 мл PBS (pH 7,4) с 0,5% Triton X-100 (w/v) в течение 5 минут и 15 минут каждый при осторожном вращении в гибридизационной печи. Удалите надосадочную жидкость, как описано в шаге 2.2.
- Суспендировать шарики в конечном объеме 16 мл PBS (pH 7,4) с 0,02% азида натрия (w/v). Хранить в виде аликвот по 1 мл при температуре 4 °C до использования.
3. Культивирование и сбор дрожжевых клеток
- Высейте контрольные и рекомбинационные компетентные штаммы дрожжей с планшетов YPD в 5 мл среды YPR и инкубируйте в течение ночи при 30 °C и 200 об/мин.
- Высейте 2 мл этой культуры в 100 мл среды YPR и инкубируйте в течение ночи при 30 °C и 200 об/мин.
- Для каждого штамма дозируют 1 800 мл автоклавной среды YP и 200 мл автоклавного раствора рафинозы (20% по массе) в каждую из двух 5-литровых колб Эрленмейера (всего 4 л питательной среды для каждого штамма, разделенных на две колбы).
- Посев растущих дрожжевых клеток при наружном диаметре600 0,2 в соответствующую среду и инкубируют, как описано в шаге 3.1, в течение примерно 6 ч или до тех пор, пока клетки не достигнут желаемого наружного диаметра600 1,0. Чтобы обеспечить нормальный рост клеток, свободных от каких-либо загрязнений, проверяйте наружный диаметр с интервалом в 2 часа.
- Добавьте 200 мл галактозы (20% по массе), чтобы индуцировать сайт-специфическую рекомбинацию.
- Добавьте 110 мкл (50 нг/мл) YMCA (одновременно с галактозой, шаг 3,5), чтобы остановить клетки в фазе G1, и инкубируйте в течение 2 ч.
Примечание: Добавление YMFA к клеткам зависит от условий эксперимента и биологического вопроса, который необходимо решить. В частности, для этого эксперимента клетки задерживаются в фазе G1 для получения однородной клеточной популяции с лицензированным репликационным происхождением; в этом процессе пререпликативный комплекс связывается с источниками в фазе G1 до начала репликации ДНК в фазе S. - Переложите клеточную суспензию в центрифужные ведра объемом 1 л и центрифугируйте при температуре 6 000 × г и температуре 4 °C в течение 10 мин. Надосадочную жидкость выбросить, и повторно суспендировать клеточные гранулы в общем объеме 10-15 мл dH2O.
- Запечатайте шприц объемом 25 мл пробкой Луэра и поместите его в коническую пробирку объемом 50 мл, наполненную водой. Переложите клеточную суспензию в шприцевой узел и центрифугируйте при 2,397 × г в течение 10 мин при комнатной температуре. Выбросьте надосадочную жидкость.
- Выньте пробку Люэра из шприца и выдавите клетки в жидкий азот, чтобы сформировать клеточные «спагетти».
ПРИМЕЧАНИЕ: Использование 4 л питательной среды обеспечивает влажную массу конечных клеточных гранул 7-10 г. - Переложите спагетти с замороженными клетками в коническую пробирку объемом 50 мл и храните при температуре −80 °C до дальнейшего использования.
4. Очистка локуса хроматина
ПРИМЕЧАНИЕ: На рисунке 2 приведен схематический обзор этапов, выполняемых в этом протоколе локус-специфической очистки хроматина.
- Охладите коммерческую кофемолку, измельчив 30-50 г сухого льда, энергично встряхивая кофемолку 2 раза в течение 30 секунд каждый раз. После охлаждения выбросьте порошок сухого льда.
- Смешайте 3 г замороженных спагетти с дрожжами с ~30-50 г сухого льда в предварительно охлажденной кофемолке.
- Загерметизируйте стык между крышкой и измельчителем парапленкой, чтобы предотвратить потерю порошка дрожжевых клеток во время измельчения.
- Измельчите смесь дрожжевых клеток и сухого льда 10 раз в течение 30 секунд каждый раз, с интервалом 30 секунд между каждым раундом (общее время: ~10 минут). Чтобы сухой лед и клеточный порошок не прилипали к стенкам кофемолки, постоянно постукивайте по стенкам кофемолки во время измельчения.
- Переложите полученную дрожжевую клетку-сухой порошок льда в пластиковый стакан с помощью чистой и сухой лопатки.
- Держите стаканы при комнатной температуре, чтобы испарился сухой лед.
- Как только сухой лед испарится, добавьте 2,25 мл буфера MB с 1 ингибиторами протеазы и фосфатазы (750 мкл/г дрожжевых клеток) в порошок дрожжевых клеток.
- Энергично пипетируйте клеточно-буферную смесь, чтобы обеспечить полную суспензию клеток с буфером, и перелейте в коническую пробирку объемом 15 мл.
- Возьмите образцы для анализа ДНК (0,1%) и белка (0,05%) из полученной клеточной суспензии (экстракты сырых клеток [CCE]). Хранить при температуре −20 °C до дальнейшей обработки (описанной в разделе 7).
- Переложите клеточную суспензию в реакционные пробирки объемом 2 мл с низким связыванием и центрифугируйте при 21 130 × г в течение 30 мин при 4 °C, чтобы отделить клеточный мусор от сырого клеточного лизата.
- Смешайте надосадочную жидкость из всех пробирок в одну коническую пробирку объемом 15 мл.
- Возьмите образцы из надосадочной жидкости (вход [IN]) для анализа ДНК (0,1%) и белка (0,05%). Хранить при температуре −20 °C до дальнейшей обработки (описанной в разделе 7).
- Уравновешивают 500 мкл суспензии магнитных шариков, связанных с IgG (для каждого штамма дрожжей), промывая 2 раза 500 мкл холодного буфера MB с 1 ингибиторами протеазы и фосфатазы в течение 5 мин каждый при 4 °C при вращении при 20 об/мин. Удаляйте надосадочную жидкость из бусин с помощью магнитной решетки после каждого этапа стирки.
- Инкубируют магнитные шарики, связанные с IgG, в 500 мкл холодного буфера MB с 1x ингибиторами протеазы и фосфатазы в течение 1 ч при 4 °C при вращении при 20 об/мин. Выбросьте надосадочную жидкость из бусин с помощью магнитной стойки.
- Смешайте уравновешенную суспензию магнитных шариков с клеточным лизатом и инкубируйте при вращении при 20 об/мин в течение 2 ч при 4 °C.
- Перенесите полную суспензию лизата магнитных шариков в реакционные пробирки с низким связыванием.
- Отделите магнитные шарики, несущие интересующие кольца хроматина, от клеточного лизата с помощью магнитной стойки.
- Перелейте оставшуюся проточную часть (FT) из каждой пробирки в свежую коническую пробирку объемом 15 мл и возьмите образцы для анализа ДНК (0,1%) и белка (0,05%). Хранить при температуре −20 °C до дальнейшей обработки (описанной в разделе 7).
- Суспендируйте магнитные шарики из каждой пробирки в 300 мкл холодного буфера MB и объедините шарики в одну реакционную пробирку. Промыть 5 раз по 10 мин каждый с 750 мкл холодного буфера MB с 1 ингибитором протеазы и фосфатазы при 4 °C при вращении при 20 об/мин. Окончательную промывку проводят 750 мкл холодного буфера MB без ингибиторов протеазы и фосфатазы.
- Суспендировать шарики в 40 мкл холодного буфера МБ без ингибиторов протеазы и фосфатазы.
ПРИМЕЧАНИЕ: Окончательный объем элюата может быть скорректирован в соответствии с его предполагаемым последующим применением. Элюирование ТЭВ обычно эффективно работает в диапазоне 100-300 мкл.
5. Протеаза-опосредованное элюирование ТЭВ
- Чтобы высвободить кольцевые комплексы хроматина LexA-CBP из гранул, инкубируйте гранулы с 2 мкл 6-кратной рекомбинантной протеазы ТЭВ, помеченной His, в течение ночи при 4 °C и 450 об/мин в термомиксере.
- Отделите бисер от готового элюата с помощью магнитной стойки. Переложите элюат, содержащий расщепленные кольца хроматина, в новую реакционную пробирку объемом 1,5 мл.
- Снова поместите пробирки на магнитную решетку, чтобы отделить остатки гранул от конечного элюата.
- Ресуспендант гранул (TB) в 750 мкл холодного буфера переменного тока и возьмите образцы для анализа ДНК (0,1%) и белка (0,05%). Хранить при температуре −20 °C до дальнейшей обработки (описанной в разделе 7).
- Из окончательного элюата (TE) берут образцы для анализа ДНК (0,5%) и белка (0,25%). Хранить при температуре −20 °C до дальнейшей обработки (описанной в разделе 7).
ПРИМЕЧАНИЕ: Из-за малого объема конечного элюата процент образцов, собранных для анализа ДНК и белка, увеличивается, чтобы получить лучшее представление о содержании белка и ДНК в них во время анализа.
6. Элюирование денатурации
- Промыть шарики 2 раза в 750 мкл холодного буфера переменного тока в течение 20 минут каждый раз при 4 °C при вращении при 20 об/мин.
- Для извлечения оставшихся связанных комплексов LexA-хроматина проводят денатурационное элюирование, добавляя к гранулам 500 мкл 0,5М NH4OH, и инкубируют в течение 30 мин при комнатной температуре.
- Отделите гранулы от суспензии с помощью магнитной стойки и перенесите элюат в реакционную пробирку с низким связыванием.
- Снова инкубируйте шарики, как показано на шаге 6.2, чтобы восстановить максимальное количество локусов хроматина в элюате.
- Отделите шарики от суспензии с помощью магнитной стойки и соберите полученный элюат в ту же пробирку, содержащую элюат из шага 6.3.
- Ресуспендируйте шарики (DB) в 750 мкл dH2O и возьмите образцы для анализа ДНК (0,1%) и белка (0,05%).
- Из конечного элюата (ДЭ) берут образцы для анализа ДНК (0,5%) и белка (0,25%).
7. Анализ ДНК и белков
- Анализ ДНК
- Пробоподготовка
- К образцам ДНК добавляют 100 мкл буфера IRN и увеличивают объем dH2Oдо конечного объема 200 мкл.
- Добавьте 1 нг спайковой плазмидной ДНК K071.
- Добавьте 1 мкл РНКазы А (10 мг/мл) и инкубируйте при 37 °C в течение 1 ч.
- Добавьте 5 мкл протеиназы К (10 мг/мл) и 10 мкл SDS (20%) и инкубируйте в течение 1 ч при 56 °C.
- Добавьте 200 мкл фенола/хлороформа/изопропилового спирта (25:24:1) и тщательно перемешайте 2 раза в течение 10 секунд каждый раз.
- Центрифугируют образцы при 21 130 × г в течение 7 мин для разделения органической и водной фаз.
- Перелейте надосадочную жидкость в новые пробирки объемом 1,5 мл, содержащие 1,5 мкл гликогена (5 мг/мл) и 2,5 100% этанола.
- Инкубируйте пробирки при температуре −20 °C не менее 2 ч и не более ночи до осаждения ДНК.
- Центрифуга при 21 130 × г, 4 °C в течение 45 мин. Выбросьте надосадочную жидкость.
- Добавьте 150 мкл 70% этанола без ресуспендирования гранулы ДНК и снова центрифугируйте при 21 130 × г при 4 °C в течение 10 мин.
- Высушите гранулы ДНК при комнатной температуре в течение 10-15 мин и ресуспендируйте в 40 мкл dH2O.
- Выполните расщепление ферментом рестрикции для линеаризации ДНК, выделенной на этапе 7.1.1.11, эндонуклеазой рестрикции HpaI. Для реакционной смеси объемом 50 мкл инкубируют 40 мкл изолированной ДНК + 2 мкл фермента HpaI + 5 мкл буфера фермента рестрикции + 3 мкл dH2O в течение ночи при 37 °C.
- кПЦР-анализ
- Разбавляют рестрикционно-расщепленную ДНК в соотношении 1:5 (10 мкл рестрикционно-расщепленной ДНК, полученной на стадии 7.1.1.12, в 40 мкл dH2O).
- Используйте пары праймеров, предназначенные для области ARS316, PDC1 и спайковой плазмидной ДНК.
- Запустите qPCR с помощью программы, приведенной в таблице 2.
- Анализируйте результаты кПЦР путем относительного количественного определения, используя область ARS316 и спайковую плазмидную ДНК в качестве мишеней и PDC1 (или любой другой ген или область генома) в качестве референсного локуса.
- Оцените процент извлечения локуса ARS316 по сравнению с PDC1 путем нормализации относительных количественных значений праймеров ARS316 и PDC1 на процент объема фракции, взятой для каждого образца. Например, умножьте 0,1% для проточного значения на коэффициент 1 000 и 0,5% для элюирования ТЭВ на коэффициент 200.
- Нормализуйте проценты восстановления ARS316 и PDC1 с помощью относительных количественных значений спайковой плазмидной ДНК.
- Оцените обогащение локуса ARS316 по общему хроматину, оценив полученные относительные количественные целевые/референсные значения с помощью уравнения (1).
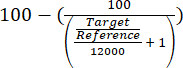 (1)
(1)
- Анализ Саузерн блоттинг
- К 20 мкл образца ДНК, расщепленного ферментом рестрикции, добавляют 5 мкл красителя с гелевой загрузкой.
- Прогонят образцы в 1% агарозном геле при 110 В в течение ~3 ч.
- Переложите гель на лоток и замочите его в растворе для депурирования при легком встряхивании на 20 минут.
- Смойте гель dH2O и замочите его в денатурационном растворе на 15 минут при встряхивании. Раствор для денатурации выбросьте.
- Замочите гель в денатурационном растворе на 15 мин при легком встряхивании.
- Смойте гель dH2O и промывайте его 2 раза в течение 15 минут каждый раз с помощью буфера для переноса с легким встряхиванием.
- Наполните лоток 1-1,5 л буфера для перекачки и поставьте его на устойчивую платформу.
- Отрежьте длинный кусок ватмана и положите его на платформу таким образом, чтобы два конца бумаги пропитались буфером для переноса.
- Отрежьте одну полоску положительной нейлоновой мембраны и четыре полоски ватмана, равные размеру геля.
- Положите на платформу два листа ватмана, смоченных в буфере для переноса.
- Положите гель лицевой стороной вниз на кусочки ватмана.
- Поверх геля положите положительную нейлоновую мембрану, а затем оставшиеся два кусочка ватмана, смоченные в трансферном буфере. Следите за тем, чтобы стек оставался влажным с буфером передачи.
- Удалите все застрявшие пузырьки воздуха, скрутив их стеклянной палочкой.
- Положите стопку бумажных полотенец поверх собранной стопки, а затем предмет весом 0,5 кг (например, стеклянную бутылку).
- Оставьте сборку для переноса на ночь при комнатной температуре.
- После переноса мембрану сшивают с помощью УФ-излучения с общей выходной энергией 1 200 Дж/см2.
- Для детектирования локуса ARS316 проводят южную блот-гибридизацию с использованием радиоактивных зондов, синтезированных с использованием референсной системы мечения ДНК.
ПРИМЕЧАНИЕ: Выполняйте все дальнейшие действия на льду, если не указано иное. - В реакционной пробирке с низким связыванием разводят 25-40 нг ПЦР-фрагмента (зонда) в 19 мкл dH2O и инкубируют при 95 °C в течение 5 мин. Остудить сразу на льду.
- Добавьте в пробирку по 1 мкл 500 мкМ dCTP, 500 мкМ dGTP и 500 мкМ dTTP.
- Добавьте 20 мкл буфера, содержащего случайные нуклеотидные гексамеры, поставляемые в комплекте.
- Добавьте в пробирку 5 мкл радиоактивно меченого нуклеотида [α-32P] dATP. Выполняйте все действия, связанные с радиоактивно меченым материалом, в защищенной среде, одобренной для радиоактивности.
- Добавьте 1 мкл фрагмента Кленова и инкубируйте при 37 °C на термомиксере в течение 15 мин для синтеза одноцепочечной ДНК.
- Добавьте 5 мкл стоп-буфера, чтобы остановить реакцию.
- Центрифугируйте колонку исключения размера в течение 2 минут при 1 500 × g , чтобы удалить буфер хранения. Отбросьте поток и поместите колонку в новую трубку.
- Пропустите зондовую смесь через колонку, чтобы удалить неинкорпорированные радиоактивные нуклеотиды. Центрифуга в течение 30 с при давлении 1 500 × г для сбора зонда.
- Добавьте 150 мкл ДНК сперматозоидов лосося (1:100), чтобы заблокировать неспецифическое связывание зонда с мембраной.
- Инкубируйте смесь зондов при 95 °C в течение 5 мин для денатурации двухцепочечной ДНК.
- Заморозьте на льду в течение нескольких секунд и центрифугируйте в течение нескольких секунд при 500 × г , чтобы капли воды конденсировались на крышке.
- Кратковременно промывают южную блоттинговую мембрану буфером для гибридизации в течение 5-10 мин с ротацией.
- Предварительно гибридизируют мембрану с буфером для гибридизации в течение 1 ч при 55 °C с вращением. Откажитесь от буфера гибридизации.
- Добавьте 15 мл свежего буфера для гибридизации (предварительно подогретого до 55 °C) в мембрану вместе с подготовленным зондом. Инкубируйте мембрану в течение ночи при температуре 55 °C с вращением.
- Выполните постгибридизационные промывки 2 раза по 15 мин каждая с 0,3x SSC, 0,1% SDS при 55 °C с вращением.
- Выполните постгибридизационные промывки 2 раза по 15 мин каждая с 0,1x SSC, 0,1% SDS при 55 °C с вращением.
- Выполните постгибридизационные промывки 2 раза по 15 мин каждая с 0,1x SSC, 1,5% SDS при 55 °C с вращением.
- Снимите мембрану с гибридизационной пробирки и подвергните мембрану воздействию люминографического экрана на срок до 3 дней, чтобы получить радиоактивные отпечатки на пленке.
- Получение изображений пленки с помощью системы лазерного сканирования с люминографической визуализацией.
- Пробоподготовка
- Анализ белков
- Пробоподготовка
- Отрегулируйте все образцы белка до их окончательных объемов (200 мкл, 100 мкл, 100 мкл, 20 мкл и 20 мкл для образцов экстракта сырых клеток, входных, проточных, гранул и элюата соответственно), добавив 1 мкл β-меркаптоэтанола, буфер для образцов PAGE LDS (1x) и dH2O.
- Денатурируют образцы при 95 °C в течение 5 мин.
- Вестерн-блоттинг-анализ
ПРИМЕЧАНИЕ: Выполняйте SDS-PAGE и вестерн-блоттинг по стандартным протоколам15,16.- Загрузите 15 мкл каждого образца в каждую лунку геля SDS-PAGE толщиной 1,5 мм.
- Проводят иммуноокрашивание блота с использованием антител PAP (1:2 000) и LexA (1:1 000) с ночной инкубацией при 4 °C. Развести в 5% растворе сухого молока/ПБСТ.
ПРИМЕЧАНИЕ: После ПАП-анализа блот может быть удален с помощью протокола мягкой стриппинга17 перед иммуноокрашиванием вторым антителом LexA.
- Пробоподготовка
Результаты
Очистка домена хроматина ARS316 ~1,4 kb была опосредована конститутивно экспрессируемым белком-адаптером LexA-TAP. В качестве отрицательного контроля мы проводили очистки с использованием изогенного штамма, экспрессирующего LexA-TAP, но не содержащего интегрированных RS и LexA-связывающих сайтов.
Обсуждение
Идентификация факторов и хроматинового ландшафта конкретной целевой области генома по-прежнему представляет собой серьезную проблему в исследованиях хроматина18. Этот протокол описывает эффективную систему специфического удаления и очистки различных доменов хроматина...
Раскрытие информации
У авторов нет конфликта интересов, который можно было бы раскрыть.
Благодарности
Работа в лаборатории S.H. поддерживалась DFG через SFB1064 (идентификатор проекта 213249687), Европейским исследовательским советом (ERC Starting Grant 852798 ConflictResolution) и Обществом Гельмгольца.
Материалы
| Name | Company | Catalog Number | Comments |
| Yeast strains | |||
| Control Strain: MATa; ura3Δ0; leu2Δ0; his3Δ1; met15Δ0; bar1::kanMX4; Chr I 212kb::LEU2 pTEF2-LEXA-TAP pGAL1-10 RecR | Section 1, see references 13 and 14 | ||
| Recombination Strain: MATa; ura3Δ0; leu2Δ0; his3Δ1; met15Δ0; bar1::kanMX4; RS_LEXA_NS-3_ARS316_NS+3_RS; Chr I 212kb::LEU2 pTEF2-LEXA-TAP pGAL1-10 RecR | Section 1, see reference 13 and 14 | ||
| Plasmid | |||
| K238 plasmid | Section 1, see reference 13 Storage: Store at -20 °C | ||
| K071 Spike-in plasmid DNA | Section 7.1, see reference 13 Storage: Store at -20 °C | ||
| Reagents | |||
| Acetone | Carl Roth | 5025.1 | Section 2 Storage: Store at room temperature |
| Ammonium acetate (NH4Ac) | Sigma Aldrich | A7262 | Section 6 and 7.1 Storage: Store at room temperature |
| Ammonium solution (NH4OH) 25% | Merck Millipore | 533003 | Section 6 Storage: Store at room temperature |
| Ammonium sulfate | Santa Cruz | Sc-29085 | Section 2 Storage: Store at room temperature |
| Bacto agar | BD (VWR) | 90000-760 | Section 3 Storage: Store at room temperature |
| Bacto peptone | BD (VWR) | 211820 | Section 3 Storage: Store at room temperature |
| β-Mercaptoethanol | Sigma Aldrich | 07604 | Section 7.2 Storage: Store at 4 °C |
| Chemiluminescent substrate kit | ThermoFisher | 34580 | Section 7.2 Storage: Store at 4 °C |
| Di-Sodium Hydrogen phosphate dodecahydrate | Merck | 1.06579.1000 | Section 2 and 7.1 Storage: Store at room temperature |
| Dithiothreitol (DTT) | ThermoFisher | 15508013 | Section 4 Storage: Store at 4 °C |
| Ethanol | Merck | 100983 | Section 7.1 Storage: Store at room temperature |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma Aldrich | ED | Section 7.1 Storage: Store at room temperature |
| Galactose (20% (w/v) stock) | Sigma Aldrich | G0625-1KG / 5KG | Section 3 Storage: Store at room temperature |
| Gel loading dye (6x) | BioLabs | B7024A | Section 7.1 Storage: Store at -20 °C |
| Glusose | Sigma-Aldrich | G8270 | Section Storage: Store at room temperature |
| Glycine | Carl Roth | .0079.4 | Section 2 Storage: Store at room temperature |
| Glycogen (5 mg/mL) | Invitrogen | AM9510 | Section 7.1 Storage: Store at -20 °C |
| Hydrochloric acid (HCl) | PanReac AppliChem | 182109.1211 | Section 2, 4 and 7.1 Storage: Store at room temperature |
| Magnesium Acetate (MgAc) | Bernd Kraft | 15274.2600/C035 | Section 4 Storage: Store at room temperature |
| Magnesium chloride (MgCl2) | Sigma Aldrich | M8266 | Section 6 Storage: Store at room temperature |
| Nu PAGE LDS sample buffer (4x) | Invitrogen | 2399549 | Section 7.2 Storage: Store at room temperature |
| Phenol/Chloroform/Isoamyl alcohol (25:24:1 v/v) | Invitrogen | 15593-031 | Section 7.1 Storage: Store at 4 °C |
| Potassium chloride (KCl) | Sigma | P9541 | Section 4 Storage: Store at room temperature |
| Radioactively labeled α-32P dATP (3,000 Ci/mmol, 10 mCi/mL) | Hartmann Analytic | SRP-203 | Section 7.1 Storage: Store at 4 °C |
| RadPrime labeling system | ThermoFisher | 18428-011 | Section 7.1 Storage: Store at -20 °C |
| Raffinose (20% (w/v) stock) | SERVA | 34140.03 | Section 3 Storage: Store at room temperature |
| Sodium chloride (NaCl) | Merck | K53710504142 | Section 7.1 Storage: Store at room temperature |
| Sodium citrate (Na3C6H5O7) | Sigma-Aldrich | 71402 | Section 7.1 Storage: Store at room temperature |
| Sodium hydroxide (NaOH) | Sigma Aldrich | S5881 | Section 7.1 Storage: Store at room temperature |
| Sodium n-dodecyl sulfate (SDS) (5% stock (w/v) ) | Alfa Aesar | A11183 | Section 7.1 Storage: Store at room temperature |
| Sodium phosphate monobasic | Sigma-Aldrich | 71496 | Section 2 and 7.1 Storage: Store at room temperature |
| Sodium azide | Santa Cruz Biotechnology | sc-208393 | Section 2 Storage: Store at -20 °C |
| Triethylamine | Sigma Aldrich | 90340 | Section 2 Storage: Store at room temperature |
| Tris base | Chem Cruz | SC-3715B | Section 2 and 4 Storage: Store at room temperature |
| Triton X-100 | Sigma Aldrich | X100 | Section 2 and 4 Storage: Store at room temperature |
| Tween-20 | Bernd Kraft | 18014332 | Section 4 Storage: Store at room temperature |
| Yeast extract | BD (VWR) | 212720 | Section 3 Storage: Store at room temperature |
| Yeast mating factor alpha (1 µg/mL stock ) | Biomol | Y2016.5 | Section 3 Storage: Store at -20 °C |
| Yeast Synthetic Drop-out medium Supplements without LEUCINE | Sigma Aldrich | Y1376 | Section 1, see reference 14 |
| Enzymes | |||
| HpaI restriction enzyme (5,000 U/mL) | NEB | R0105S | Section 7.1 Storage: Store at -20 °C |
| Protease and Phosphatase Inhibitor Cocktail (100x) | ThermoFisher Scientific | 78446 | Section 4 Storage: Store at4 °C |
| Proteinase K (10 mg/mL) | SERVA | 33756 | Section 7.1 Storage: Store at -20 °C |
| RNase A (10 mg/mL) | ThermoFisher | EN0531 | Section 7.1 Storage: Store at -20 °C |
| TEV protease (10000 U/µL) | NEB | P8112S | Section 5 Storage: Store at -20 °C |
| Materials | |||
| BcMag Epoxy-Activated Magnetic Beads | Bioclone Inc. | FC-102 | Section 2 Storage: Store at 4 °C |
| Dry ice | Section 4 | ||
| Low-binding centrifuge tubes 2.0 mL | Eppendorf | 22431102 | Section 4 |
| Microspin G-25 Columns | Cytiva | 27-5325-01 | Section 7.1 Storage: Store at room temperature |
| Parafilm | Merck | P7793 | Section 4 |
| Positive nylon membrane | Biozol | 11MEMP0001 | Section 7.1 Storage: Store at room temperature |
| PVDF transfer membrane | Immobilon-Merck Millipore | IPVH00010 | Section 7.2 Storage: Store at room temperature |
| SDS-PAGE gel 4-12% bis-tris (15 well, 1.5 mm) | Invitrogen | NP0336BOX | Section 7.2 Storage: Store at 4 °C |
| Syringe (25 mL) with luer fitting | Henke Sass Wolf | 4200-000V0 | Section 3 |
| Whatman paper (Grade 3MM CHR Cellulose Western Blotting Paper Sheet) | Cytiva | 3030-917 | Section 7.1 Storage: Store at room temperature |
| Antibodies | |||
| Anti-LexA, rabbit polyclonal IgG, DNA binding region antibody | Merck Millipore | 06-719 | Section 7.2 Storage: Store at -20 °C |
| Goat Anti-Rabbit IgG (H+L), Horseradish peroxidase conjugate | Invitrogen | G21234 | Section 7.2 Storage: Store at -20 °C |
| Peroxidase Anti-Peroxidase (PAP) antibody produced in rabbit for the detection of TAP-tagged proteins | Sigma Aldrich | P1291-500UL | Section 7.2 Storage: Store at -20 °C |
| Rabbit IgG antibodies | Sigma | I5006-100MG | Section 2 Storage: Store at 4 °C |
| Primers (10 µM) | |||
| ARS316: fwd 5'- CGGCATTATCGTACACAACCT, rev 5'- GTTCTTCGTTGCCTACATTTTCT | Section 7.1 | ||
| K071 Spike-in plasmid DNA: fwd: 5'-TTTTCGCTGCTTGTCCTTTT, rev 5'- CATTTTCGTCCTCCCAACAT | Section 7.1 | ||
| PCR fragment from yeast genomic DNA as a template for ARS316 amplification (for southern blot): fwd 5’- AAATTCTGCCCTTGATTCGT rev 5’- TTTGTTTATCTCATCACTAAT | Section 7.1 | ||
| PDC1: fwd 5'- CATGATCAGATGGGGCTTCA, rev 5'-ACCGGTGGTAGCGACTCTGT | Section 7.1 | ||
| Equipment | |||
| Coffee grinder | Gastroback | 42601 | Section 4 |
| Dewar flask | NAL GENE | 4150-2000 | Section 3 |
| DynaMag TM-2 magnetic rack | Invitrogen | 12321D | Section 4, 5 and 6 |
| Hybridization oven | Hybaid Mini10 | Ri418 | Section 2 |
| Microcentrifuge | Eppendorf | 5424R | Section 4 and 7.1 |
| UV-crosslinker | Analytikjena | 95-0174-02 | Section 7.1 |
Ссылки
- Kornberg, R. D., Lorch, Y. Twenty-five years of the nucleosome, fundamental particle of the eukaryote chromosome. Cell. 98 (3), 285-294 (1999).
- Gauchier, M., van Mierlo, G., Vermeulen, M., Déjardin, J. Purification and enrichment of specific chromatin loci. Nature Methods. 17 (4), 380-389 (2020).
- Korthout, T., et al. Decoding the chromatin proteome of a single genomic locus by DNA sequencing. PLoS Biology. 16 (7), e2005542 (2018).
- Antão, J. M., Mason, J. M., Déjardin, J., Kingston, R. E. Protein landscape at Drosophila melanogaster telomere-associated sequence repeats. Molecular and Cellular Biology. 32 (12), 2170-2182 (2012).
- Déjardin, J., Kingston, R. E. Purification of proteins associated with specific genomic loci. Cell. 136 (1), 175-186 (2009).
- Ide, S., Dejardin, J. End-targeting proteomics of isolated chromatin segments of a mammalian ribosomal RNA gene promoter. Nature Communications. 6, 6674 (2015).
- Unnikrishnan, A., Gafken, P. R., Tsukiyama, T. Dynamic changes in histone acetylation regulate origins of DNA replication. Nature Structural & Molecular Biology. 17 (4), 430 (2010).
- Buxton, K. E., et al. Elucidating protein-DNA interactions in human alphoid chromatin via hybridization capture and mass spectrometry. Journal of Proteome Research. 16 (9), 3433-3442 (2017).
- Kennedy-Darling, J., et al. Discovery of chromatin-associated proteins via sequence-specific capture and mass spectrometric protein identification in Saccharomyces cerevisiae. Journal of Proteome Research. 13 (8), 3810-3825 (2014).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355, 7111 (2017).
- Hamperl, S., et al. Purification of specific chromatin domains from single-copy gene loci in Saccharomyces cerevisiae. Functional Analysis of DNA and Chromatin. 1094, 329-341 (2014).
- Hamperl, S., et al. Compositional and structural analysis of selected chromosomal domains from Saccharomyces cerevisiae. Nucleic Acids Research. 42 (1), 2 (2014).
- Weiβ, M. Single-copy locus proteomics of early- and late-firing DNA replication origins identifies a role of Ask1/DASH complex in replication timing control. Cell Reports. 42 (2), 112045 (2023).
- Gietz, R. D., Schiestl, R. H. High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method. Nature Protocols. 2 (1), 31-34 (2007).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. Proceedings of the National Academy of Sciences of the United States of America. 76 (9), 4350-4354 (1979).
- Western blot membrane stripping for restaining protocol. Abcam Available from: https://www.abcam.com/protocols/western-blot-membrane-stripping-for-restaining-protocol (2023)
- Vermeulen, M., Déjardin, J. Locus-specific chromatin isolation. Nature Reviews. Molecular Cell Biology. 21 (5), 249-250 (2020).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены