A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
עותק יחיד של גן לוקוס כרומטין טיהור ב Saccharomyces cerevisiae
In This Article
Summary
פרוטוקול זה מציג שיטת בידוד כרומטין ספציפית ללוקוס המבוססת על רקומבינציה ספציפית לאתר כדי לטהר מוקד גן בעל עניין של עותק יחיד בהקשר הכרומטין הטבעי שלו משמרים ניצנים, Saccharomyces cerevisiae.
Abstract
היחידה הארגונית הבסיסית של הכרומטין האיקריוטי היא חלקיק הליבה הנוקלאוזומי (NCP), המורכב מדנ"א עטוף ~1.7 פעמים סביב אוקטמר היסטון. כרומטין מוגדר כישות של NCPs וקומפלקסים חלבוניים רבים אחרים, כולל גורמי שעתוק, עיצוב מחדש של כרומטין ושינוי אנזימים. עדיין לא ברור כיצד אינטראקציות חלבון-דנ"א אלה מתוזמרות ברמה של אתרים גנומיים ספציפיים במהלך שלבים שונים של מחזור התא. זה בעיקר בשל המגבלות הטכניות הנוכחיות, אשר מקשות על השגת מדידות מדויקות של אינטראקציות דינמיות כאלה. כאן, אנו מתארים שיטה משופרת המשלבת רקומבינציה ספציפית לאתר עם פרוטוקול טיהור זיקה יעיל בצעד אחד כדי לבודד מוקד גן בעל עניין בעותק יחיד במצב הכרומטין הטבעי שלו. השיטה מאפשרת העשרה איתנה של מוקד המטרה על פני כרומטין, מה שהופך טכניקה זו לאסטרטגיה יעילה לזיהוי וכימות אינטראקציות חלבונים באופן בלתי משוחד ושיטתי, למשל על ידי ספקטרומטריית מסות. בהמשך לניתוחי הרכב כאלה, כרומטין טבעי שטוהר בשיטה זו משקף ככל הנראה את המצב in vivo בנוגע למיקום נוקלאוזומים ושינויים בהיסטון, ולכן ניתן לבצע ניתוחים מבניים וביוכימיים נוספים של כרומטין הנגזר כמעט מכל מוקד גנומי בשמרים.
Introduction
הארגון הדינמי של גנומים אאוקריוטים לכרומטין דוחס את הדנ"א כך שיתאים לגבולות הגרעין תוך הבטחת דינמיקה מספקת לביטוי גנים ונגישות לגורמים רגולטוריים. בין השאר, רב-תכליתיות זו מתווכת על ידי הנוקלאוזום, היחידה הבסיסית של כרומטין, המורכבת מחלקיק ליבה עם 147 bp של DNA עטוף ~ 1.7 פעמים סביב אוקטמר היסטון1. הנוקלאוזום, הוא מבנה דינמי מאוד ביחס להרכבו, עם גרסאות היסטון רבות ושינויים פוסט-תרגומיים (PTM) על זנבות ההיסטון N ו-C. יתר על כן, כרומטין אאוקריוטי מקיים אינטראקציה עם מספר רב של מרכיבים חיוניים אחרים, כגון גורמי שעתוק, מכונות עיבוד DNA ו- RNA, חלבונים ארכיטקטוניים, אנזימים המעורבים בעיצוב מחדש ושינוי של כרומטין, ומולקולות RNA הקשורות לכרומטין. מכונות חיוניות אלה המעורבות בשעתוק, שכפול ותיקון דורשות גישה לכרומטין, המשמש כמצע הטבעי לתהליכים אלה. כתוצאה מכך, הבנת המנגנונים המולקולריים העומדים בבסיס עסקאות דנ"א אלה מחייבת הגדרה מדויקת של השינויים הקולקטיביים במבנה הכרומטין באזורים הגנומיים הספציפיים שבהם מכונות אלה מתכנסות ומאפשרות תגובות ביולוגיות.
למרות הזיהוי של גורמי כרומטין רבים באמצעות גנטיקה ומחקרי אינטראקציה חלבון-חלבון, ביצוע ניתוחים ישירים, בלתי משוחדים ומקיפים של אינטראקציות כרומטין באתרים גנומיים מסוימים נותר מכשול משמעותי 2,3. בתחילה, ניתן היה לבודד רק אזורים שופעים מאוד של הגנום (כלומר, לוקי חוזר) או פלסמידים מרובי עותקים בכמויות וטוהר מספיקים לזיהוי ספקטרומטרי מסה של החלבונים הקשורים 4,5,6,7. סדרה של גישות חדשות המבוססות על הכלאה ישירה של גשושיות לכידה לדנ"א כרומטיני, ביוטינילציה של קרבה באמצעות מערכת CRISPR-dCas9, או קשירה של חלבוני מתאם ספציפיים לרצף למוקד העניין החלו לפענח את הפרוטאום של אתרים בעלי עותק יחיד מגנומים של שמרים ויונקים 8,9,10. עם זאת, כל השיטות הללו דורשות הצלבת פורמלדהיד כדי לייצב את אינטראקציות החלבון-דנ"א וסוניקציה כדי להמיס את הכרומטין לטיהור לאחר מכן. יחד, שתי המניפולציות שוללות את האפשרות של מחקרים מבניים ופונקציונליים הבאים של הכרומטין המטוהר.
כדי להתגבר על מגבלות אלה, פיתחנו בעבר מתודולוגיה המשתמשת ברקומבינציה ספציפית לאתר כדי לחלץ תחומים כרומוזומליים ממוקדים משמרים11,12. בעיקרו של דבר, האזור הגנומי המעניין מוקף באתרי זיהוי (RS) עבור R-רקומבינאז ספציפי לאתר מ- Zygosaccharomyces rouxi ובו זמנית משלב קבוצה של שלושה אתרי קישור DNA עבור חלבון מדכא השעתוק הפרוקריוטי LexA (LexA) באותו אזור. תאי השמרים מכילים קסטת ביטוי לביטוי סימולטני של R-רקומבינאז וחלבון LexA המאוחה לתג טיהור זיקה טנדם (TAP). לאחר השראת R-רקומבינאז, האנזים מוציא ביעילות את אזור המטרה מהכרומוזום בצורה של תחום כרומטין מעגלי. ניתן לטהר תחום זה באמצעות חלבון מתאם LexA-TAP, הנקשר לאתרי קשירת הדנ"א של LexA, כמו גם לתמיכה באהדה. שיטה זו שימשה לאחרונה לבידוד תחומי כרומטין נפרדים המכילים מקורות שכפול נבחרים של כרומוזום שמרים III13.
אחד היתרונות העיקריים של גישת ex vivo זו הוא שהיא מאפשרת ניתוחים פונקציונליים של החומר המבודד. לדוגמה, תחומי מקור שכפול שטוהרו בשיטה זו יכולים להיות נתונים למבחני שכפול חוץ גופיים כדי להעריך את יעילות ירי המקור במבחנה מתבניות כרומטין מורכבות מקוריות in vivo . בסופו של דבר, האפיון הביוכימי והפונקציונלי של החומר המבודד עשוי לאפשר בנייה מחדש של תהליכים גרעיניים באמצעות חלבונים מטוהרים יחד עם תבנית הכרומטין המקורית. לסיכום, מתודולוגיה זו פותחת אפיק מרתק במחקר הכרומטין, שכן ניתן יהיה לעקוב אחר השינויים הקולקטיביים בהרכב ובמבנה הכרומטין של אזור גנומי מסוים העובר טרנזקציה כרומוזומלית מסוימת.
Protocol
עיין בטבלת החומרים לקבלת פרטים הקשורים לכל החומרים והמכשירים המשמשים בפרוטוקול זה. ראה טבלה 1 לקבלת רשימה של הפתרונות, המאגרים והמדיה שבהם נעשה שימוש.
1. בניית זן שמרים רקומביננטי
- כדי לבנות זן שמרים בעל יכולת רקומבינציה, הפוך פלסמיד K238 מעוכל SbfI לזן שמרים עם אתרי קישור משולבים של LexA ואתרי רקומבינציה RS במוקד העניין13,14.
הערה: מקטע הגבלת SbfI שעבר טרנספורמציה מכיל את קלטת הביטוי הנדרשת לביטוי מכונן של חלבון ההיתוך LexA-TAP ו-R-רקומבינאז יחד עם רצפים הומולוגיים של כרומוזום שמרים I לאינטגרציה גנומית של קלטת הביטוי באמצעות רקומבינציה הומולוגית (איור 1). - כדי לבנות זן בקרה, קחו זן שמרים איזוגני חסר אתרי קישור משולבים של RS ו-LexA, והפכו אותו לפלסמיד K238 מעוכל SbfI באותם תנאים שבהם נעשה שימוש בזן המוכשר לרקומבינציה.
- בחר את תאי השמרים המתאימים בהתבסס על סמן הבחירה LEU2 על לוחות אגר SCD-LEU14.
2. צימוד נוגדני IgG לחרוזים מגנטיים המופעלים על ידי אפוקסי
הערה: זוג נוגדני IgG לחרוזים מגנטיים המופעלים על ידי אפוקסי על פי פרוטוקול11 הבא שפורסם.
- להשהות 300 מ"ג של חרוזים המופעלים אפוקסי ב 10 מ"ל של 50% אצטון (300 מ"ג מתאים ~ 5.1 ×10 חרוזים) בצינור חרוטי 50 מ"ל. נערו במרץ על מערבל מערבולת.
- צנטריפוגה את הצינור המכיל את החרוזים ב 820 × גרם ו 4 ° C במשך 2 דקות. הסר את supernatant.
- לשטוף את החרוזים 3x עם 20 מ"ל של 0.1 M נתרן פוספט חיץ (pH 7.4). הסר את הסופרנאטנט לאחר כל שלב שטיפה באמצעות צנטריפוגה כמתואר בשלב 2.2.
- מרחפים את החרוזים ב-16 מ"ל של חיץ נתרן פוספט 0.1 מ' (pH 7.4), ומסובבים בעדינות בתנור הכלאה למשך 5 דקות בטמפרטורת החדר.
- להמיס את IgGs ארנב (100 מ"ג) ב 7 מ"ל של dH2O (ריכוז סופי: 14 מ"ג / מ"ל).
- צנטריפוגה את מתלה IgG ב 13,000 × גרם ו 4 ° C במשך 10 דקות כדי להבהיר את המתלים.
- מעבירים 3.5 מ"ל של supernatant (המקביל 50 מ"ג של ארנב IgGs) לצינור חרוטי חדש 50 מ"ל.
- לדלל את תמיסת IgG עם 9.85 מ"ל של 0.1 M נתרן פוספט חיץ (pH 7.4), ואחריו תוספת טיפה של 6.65 מ"ל של 3 M אמוניום סולפט ב 0.1 M פוספט נתרן (pH 7.4) תחת ערבוב עדין.
הערה: הימנע מהוספת אמוניום סולפט במהירות לתמיסה מכיוון שריכוז מקומי גבוה של המלח יגרום למשקעים של הארנב IgGs. - צנטריפוגו את תמיסת IgG בטמפרטורה של 820 × גרם ו-4°C למשך 3 דקות, והוסיפו את הסופרנאטנט המתקבל לתרחיף החרוזים המגנטי.
- לדגור על הצינור למשך הלילה או לפחות למשך 18 שעות בטמפרטורה של 30°C עם סיבוב עדין בתנור הכלאה.
- הסר את supernatant כמתואר בשלב 2.2.
- לשטוף את החרוזים עם 20 מ"ל של 100 mM גליצין-HCl (pH 2.5). הסר את התמיסה במהירות כמתואר בשלב 2.2 כדי למנוע דנטורציה של פוליפפטידים IgG.
- לשטוף את החרוזים פעם אחת עם 20 מ"ל של 10 mM Tris-HCl (pH 8.8). שאפו את הסופרנאטנט כמתואר בשלב 2.2.
- הוסף 20 מ"ל של תמיסת טריאתילאמין 0.1M למשך 5-10 דקות תחת סיבוב עדין כדי להשבית את קבוצות האפוקסי הריאקטיביות השיוריות. הסר את supernatant כמתואר בשלב 2.2.
- שטפו את החרוזים 4x עם 20 מ"ל PBS (pH 7.4) במשך 5 דקות עם סיבוב עדין. הסירו את הסופרנאטנט כמתואר בשלב 2.2 לאחר כל שלב כביסה.
- שטפו את החרוזים פעמיים עם 20 מ"ל PBS (pH 7.4) עם 0.5% Triton X-100 (w/v) למשך 5 דקות ו-15 דקות כל אחד תחת סיבוב עדין בתנור היברידי. הסר את supernatant כמתואר בשלב 2.2.
- להשהות את החרוזים בנפח סופי של 16 מ"ל של PBS (pH 7.4) עם 0.02% נתרן אזיד (w/v). יש לאחסן כ-1 מ"ל aliquots ב-4°C עד לשימוש.
3. תרבית תאי שמרים וקציר
- יש לחסן זני שמרים מוכשרים לבקרה ורקומבינציה מלוחות YPD ב-5 מ"ל של מדיום YPR, ולדגור למשך הלילה ב-30°C וב-200 סל"ד.
- יש לחסן 2 מ"ל של תרבית זו ב-100 מ"ל של מדיום YPR, ולדגור למשך הלילה ב-30 מעלות צלזיוס וב-200 סל"ד.
- עבור כל זן, יש לפזר 1,800 מ"ל של מדיום YP אוטוקלאבד ו-200 מ"ל של תמיסת רפינוז אוטוקלאבד (20% w/v) לכל אחת משתי צלוחיות ארלנמאייר 5 ליטר (4 ליטר מדיום תרבית בסך הכל לכל זן, מחולקות לשתי צלוחיות).
- חסן את תאי השמרים הגדלים ב- OD600 0.2 בתווך המתאים שלהם, ודגור כמתואר בשלב 3.1 במשך כ- 6 שעות או עד שהתאים מגיעים ל- OD600 הרצוי של 1.0. כדי להבטיח צמיחה תקינה של התאים, ללא כל זיהום, לבדוק את OD במרווחים של 2 שעות.
- הוסף 200 מ"ל גלקטוז (20% w/v) כדי לגרום לרקומבינציה ספציפית לאתר.
- הוסף 110 μL (50 ng/mL) של YMFA (במקביל לגלקטוז, שלב 3.5) כדי לעצור את התאים בשלב G1, ולדגור במשך 2 שעות.
הערה: הוספת YMFA לתאים תלויה בתנאי הניסוי ובשאלה הביולוגית שיש להתייחס אליה. בדיוק, עבור ניסוי זה, התאים נעצרים בשלב G1 כדי לקבל אוכלוסיית תאים הומוגנית עם מקורות שכפול מורשים; בתהליך זה, המכלול הקדם-שכפול נקשר למקורות בשלב G1 לפני תחילת שכפול הדנ"א בשלב S. - העבירו את מתלה התא לדלי צנטריפוגות בנפח 1 ליטר ולצנטריפוגות בטמפרטורה של 6,000 × גרם ו-4°C למשך 10 דקות. השליכו את הסופרנטנט, והשעו מחדש את כדורי התא בנפח כולל של 10-15 מ"ל של dH2O.
- אטמו מזרק 25 מ"ל עם תקע Luer, והניחו אותו בתוך צינור חרוטי 50 מ"ל מלא במים. מעבירים את מתלה התא למכלול המזרק והצנטריפוגה ב-2,397 × גרם למשך 10 דקות בטמפרטורת החדר. השליכו את הסופרנטנט.
- הסר את תקע Luer מן המזרק, ו extrude את התאים לתוך חנקן נוזלי כדי ליצור תא "ספגטי".
הערה: שימוש במדיום תרבית בנפח 4 ליטר מספק משקל רטוב של כדורי התא הסופי של 7-10 גרם. - מעבירים את ספגטי התא הקפוא לצינור חרוטי בנפח 50 מ"ל, ומאחסנים בטמפרטורה של 80°C עד לשימוש נוסף.
4. טיהור מוקד כרומטין
הערה: ראו איור 2 לקבלת סקירה סכמטית של השלבים המעורבים בפרוטוקול טיהור כרומטין ספציפי ללוקוס.
- קררו מטחנת קפה מסחרית על ידי טחינת 30-50 גרם קרח יבש על ידי ניעור נמרץ של מטחנת הקפה פעמיים למשך 30 שניות בכל פעם. לאחר הקירור, השליכו את אבקת הקרח היבש.
- מערבבים 3 גרם ספגטי תאי שמרים קפואים עם ~30-50 גרם קרח יבש במטחנת הקפה המקוררת מראש.
- אטמו את הצומת בין הפקק למטחנה עם פרפילם כדי למנוע אובדן של אבקת תאי שמרים במהלך הטחינה.
- טחנו את תערובת הקרח היבש בתאי שמרים 10x במשך 30 שניות בכל פעם, עם מרווחים של 30 שניות בין כל סיבוב (הערכת זמן כוללת: ~ 10 דקות). כדי למנוע מהקרח היבש ומאבקת התאים להידבק לדפנות מטחנת הקפה, יש להקיש על דפנות מטחנת הקפה ברציפות במהלך הטחינה.
- מעבירים את אבקת הקרח המייבשת בתאי השמרים המתקבלת לכוס פלסטיק בעזרת מרית נקייה ויבשה.
- שמור את הכוסות בטמפרטורת החדר כדי לאדות את הקרח היבש.
- לאחר שהקרח היבש מתאדה, הוסיפו 2.25 מ"ל של מאגר MB עם 1x פרוטאז ומעכבי פוספטאז (750 μL/g של תאי שמרים) לאבקת תאי השמרים.
- פיפטה תערובת חיץ התא במרץ כדי להבטיח את ההשעיה המלאה של התאים עם החיץ, ולהעביר צינור חרוטי 15 מ"ל.
- קח דגימות עבור ניתוח DNA (0.1%) וחלבון (0.05%) מתרחיף התא שנוצר (תמציות תאים גולמיים [CCE]). יש לאחסן בטמפרטורה של -20°C עד לעיבוד נוסף (המתואר בסעיף 7).
- העבירו את מתלה התא לצינורות תגובה של 2 מ"ל בעלי קשירה נמוכה, וצנטריפוגות בטמפרטורה של 21,130 × גרם למשך 30 דקות ב-4°C כדי להפריד את פסולת התא מליזט התא הגולמי.
- אגרו את הסופרנאטנטים מכל הצינורות לצינור חרוטי אחד של 15 מ"ל.
- קח דגימות מהסופרנאטנט (קלט [IN]) עבור DNA (0.1%) וניתוח חלבונים (0.05%). יש לאחסן בטמפרטורה של -20°C עד לעיבוד נוסף (המתואר בסעיף 7).
- יש לאזן 500 μL של תרחיף חרוזים מגנטי מצומד IgG (עבור כל זן שמרים) על ידי שטיפה 2x עם 500 μL של מאגר MB קר עם 1x protease ומעכבי phosphatase במשך 5 דקות כל אחד ב 4 ° C בסיבוב ב 20 סל"ד. השליכו את הסופרנאטנט מהחרוזים באמצעות מתלה מגנטי לאחר כל שלב כביסה.
- דגור על חרוזים מגנטיים מצומדים IgG ב 500 μL של מאגר MB קר עם 1x פרוטאז ומעכבי phosphatase במשך 1 שעה ב 4 ° C על סיבוב ב 20 סל"ד. השליכו את הסופרנאטנט מהחרוזים באמצעות מתלה מגנטי.
- מערבבים את תמיסת החרוזים המגנטיים המאוזנת עם התא ליזט, ודגרים עם סיבוב ב-20 סל"ד למשך שעתיים ב-4°C.
- העבירו את תרחיף הליזט המגנטי המלא של תאי החרוזים לצינורות תגובה בעלי קשירה נמוכה.
- הפרד את החרוזים המגנטיים, הנושאים כעת את טבעות הכרומטין המעניינות, מליזט התא באמצעות מדף מגנטי.
- העבר את יתרת הזרימה (FT) מכל צינור לצינור חרוטי טרי של 15 מ"ל, וקח דגימות לניתוח DNA (0.1%) וחלבון (0.05%). יש לאחסן בטמפרטורה של -20°C עד לעיבוד נוסף (המתואר בסעיף 7).
- השהה את החרוזים המגנטיים מכל צינור במאגר MB קר של 300 מיקרוליטר, ואגד את החרוזים לצינור תגובה אחד. יש לשטוף 5x במשך 10 דקות כל אחד עם 750 μL של מאגר MB קר עם 1x protease ומעכבי phosphatase ב 4 ° C בסיבוב ב 20 סל"ד. בצע את השטיפה הסופית עם 750 μL של חיץ MB קר ללא מעכבי פרוטאז ופוספטאז.
- להשהות את החרוזים ב 40 μL של מאגר MB קר ללא מעכבי פרוטאז ופוספטאז.
הערה: ניתן לכוונן את הנפח הסופי של eluate בהתאם ליישום הצפוי שלו במורד הזרם. ELUTION TEV בדרך כלל עובד ביעילות בטווח של 100-300 μL.
5. אלוציה בתיווך פרוטאז TEV
- כדי לשחרר קומפלקסים של טבעות כרומטין LexA-CBP מהחרוזים, יש לדגור על החרוזים עם 2 μL של פרוטאז TEV רקומביננטי 6x His-tagged למשך הלילה ב-4°C וב-450 סל"ד בתרמיקוסר.
- הפרד את החרוזים מהפולט הסופי באמצעות מדף מגנטי. מעבירים את האלואט, המכיל טבעות כרומטין שסועות, לצינור תגובה חדש של 1.5 מ"ל.
- שמור שוב את הצינורות על מדף מגנטי כדי להפריד את שאריות החרוזים מהפליטה הסופית.
- השהה מחדש את החרוזים (שחפת) ב-750 מיקרוליטר של חיץ AC קר, וקח דגימות לניתוח DNA (0.1%) וחלבון (0.05%). יש לאחסן בטמפרטורה של -20°C עד לעיבוד נוסף (המתואר בסעיף 7).
- מהאלואט הסופי (TE), לקחת דגימות לניתוח DNA (0.5%) וחלבון (0.25%). יש לאחסן בטמפרטורה של -20°C עד לעיבוד נוסף (המתואר בסעיף 7).
הערה: בשל הנפח הנמוך של ה- eluate הסופי, אחוז הדגימות שנאספו לניתוח DNA וחלבונים גדל כדי לקבל ייצוג טוב יותר של תכולת החלבון וה- DNA שלהם במהלך הניתוח.
6. Elution Denaturation
- שטפו את החרוזים 2x ב-750 מיקרוליטר של חיץ AC קר למשך 20 דקות בכל פעם ב-4°C בסיבוב ב-20 סל"ד.
- כדי לחלץ את שאר מתחמי LexA-כרומטין מאוגדים, בצע אלוציה דנטורציה על ידי הוספת 500 μL של 0.5M NH4OH לחרוזים, ודגרה במשך 30 דקות בטמפרטורת החדר.
- הפרידו את החרוזים מהמתלה באמצעות מתלה מגנטי, והעבירו את הפולט לצינור תגובה בעל קשירה נמוכה.
- לדגור על החרוזים שוב כמו בשלב 6.2 כדי לשחזר את מיקומי הכרומטין המרביים בפולט.
- הפרידו את החרוזים מהמתלה באמצעות מתלה מגנטי, ואגרו את הפולט שנוצר לאותו צינור המכיל את הפולט משלב 6.3.
- השהה מחדש את החרוזים (DB) ב- 750 μL של dH2O, וקח דגימות לניתוח DNA (0.1%) וחלבון (0.05%).
- מהאלואט הסופי (DE), קחו דגימות לניתוח DNA (0.5%) וחלבון (0.25%).
7. ניתוח DNA וחלבונים
- ניתוח DNA
- הכנת דוגמאות
- לדגימות הדנ"א, הוסף 100 μL של מאגר IRN, והגדל את הנפח עם dH2O לנפח סופי של 200 μL.
- הוסף 1 ng של K071 ספייק-אין פלסמיד DNA.
- הוסף 1 μL של RNAse A (10 מ"ג / מ"ל), ודגור ב 37 ° C במשך 1 שעות.
- הוסף 5 μL של proteinase K (10 מ"ג / מ"ל) ו 10 μL של SDS (20%), ודגור במשך 1 שעה ב 56 ° C.
- הוסף 200 μL של פנול/כלורופורם/איזופרופיל אלכוהול (25:24:1), ומערבל היטב 2x במשך 10 שניות בכל פעם.
- צנטריפוגה הדגימות ב 21,130 × גרם במשך 7 דקות כדי להפריד את הפאזה האורגנית והמימית.
- העבירו את הסופרנאטנט לצינורות חדשים של 1.5 מ"ל המכילים 1.5 מיקרוליטר גליקוגן (5 מ"ג/מ"ל) ו-2.5x 100% אתנול.
- לדגור על הצינורות ב -20 ° C למשך מינימום של 2 שעות ומקסימום של לילה כדי לזרז את ה- DNA.
- צנטריפוגה ב-21,130 × גרם, 4°C למשך 45 דקות. השליכו את הסופרנטנט.
- הוסף 150 μL של אתנול 70% מבלי להשעות מחדש את גלולת ה- DNA, וצנטריפוגה שוב ב 21,130 × גרם ב 4 ° C למשך 10 דקות.
- יבש את כדורי ה- DNA בטמפרטורת החדר למשך 10-15 דקות, והשהה מחדש ב 40 μL של dH2O.
- בצע עיכול אנזים הגבלה כדי ליניאריזציה של ה- DNA שבודד בשלב 7.1.1.11 עם אנדונוקלאז הגבלת HpaI. לקבלת תערובת תגובה של 50 μL, דגרו 40 μL של DNA מבודד + 2 μL של אנזים HpaI + 5 μL של מאגר אנזים הגבלה + 3 μL של dH2O לילה ב 37 °C.
- ניתוח qPCR
- לדלל את הדנ"א המעוכל על ידי הגבלה ביחס של 1:5 (10 μL של הדנ"א המעוכל על ידי הגבלה המתקבל משלב 7.1.1.12 ב 40 μL של dH2O).
- השתמש בזוגות פריימרים המיועדים לאזור ARS316, PDC1 ודנ"א פלסמיד ספייק-אין.
- הפעל qPCR באמצעות התוכנית המופיעה בטבלה 2.
- נתח את תוצאות ה- qPCR על ידי כימות יחסי באמצעות אזור ARS316 ו- DNA פלסמיד ספייק-אין כמטרות ו- PDC1 (או כל גן או אזור גנומי אחר) כמוקד הייחוס.
- הערך את אחוז שחזור הלוקוס של ARS316 על פני PDC1 על-ידי נרמול ערכי הכימות היחסי של הפריימר ARS316 ו- PDC1 לפי אחוז נפח השבר שנלקח עבור כל דגימה. לדוגמה, הכפל 0.1% עבור הזרימה בפקטור של 1,000 ו- 0.5% עבור פליטת TEV בפקטור של 200.
- נרמל את אחוזי ההתאוששות של ARS316 ו- PDC1 עם ערכי הכימות היחסיים של DNA פלסמיד ספייק.
- הערך את ההעשרה של מוקד ARS316 על פני הכרומטין הכולל על ידי הערכת יעד הכימות היחסי המתקבל / ערכי ייחוס באמצעות משוואה (1).
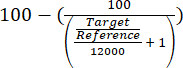 (1)
(1)
- ניתוח כתם דרומי
- ל-20 μL של דגימת DNA מעוכלת באנזים הגבלה, יש להוסיף 5 μL של צבע העמסת ג'ל.
- הפעל את הדגימות בג'ל אגרוז 1% ב 110 V במשך ~ 3 שעות.
- מעבירים את הג'ל למגש ומשרים אותו בתמיסת פירוק עם ניעור עדין למשך 20 דקות.
- שטפו את הג'ל עם dH2O, והשרו אותו בתמיסת דנטורציה למשך 15 דקות עם טלטול. השליכו את תמיסת הדנטורציה.
- השרו את הג'ל בתמיסת דנטורציה למשך 15 דקות בניעור עדין.
- שטפו את הג'ל עם dH2O, ושטפו אותו 2x במשך 15 דקות בכל פעם עם מאגר ההעברה עם ניעור עדין.
- מלא מגש עם 1-1.5 ליטר של מאגר העברה, והנח אותו על פלטפורמה יציבה.
- חותכים פיסת נייר Whatman ארוכה, ומניחים אותה על הפלטפורמה כך ששני קצוות הנייר יהיו ספוגים במאגר ההעברה.
- חותכים רצועה אחת של קרום ניילון חיובי וארבע רצועות נייר Whatman השוות לגודל הג'ל.
- הניחו שתי פיסות נייר Whatman ספוגות במאגר ההעברות על הרציף.
- מניחים את הג'ל עם הפנים כלפי מטה על גבי פיסות נייר Whatman.
- על גבי הג'ל, מניחים את קרום הניילון החיובי, ואחריו את שתי פיסות נייר Whatman הנותרות ספוגות במאגר העברה. הקפד לשמור על הערימה רטובה באמצעות מאגר ההעברה.
- הסר בועות אוויר לכודות על ידי גלגולן באמצעות מוט זכוכית.
- הניחו ערימה של מגבות נייר על גבי הערימה שהורכבה, ואחריה חפץ במשקל 0.5 ק"ג (למשל, בקבוק זכוכית).
- השאירו את ההרכבה להעברת לילה בטמפרטורת החדר.
- לאחר ההעברה, UV-crosslink הממברנה עם תפוקת אנרגיה כוללת של 1,200 J / cm2.
- לזיהוי מוקד ARS316, בצע הכלאה של כתם דרומי באמצעות בדיקות רדיואקטיביות המסונתזות באמצעות מערכת תיוג ה- DNA המוזכרת.
הערה: בצע את כל השלבים הבאים על קרח עד לציון אחר. - בצינור תגובה בעל קשירה נמוכה, יש לדלל 25-40 ננוגרם של מקטע PCR (בדיקה) ב-19 מיקרוליטר של dH2O, ולדגור ב-95°C למשך 5 דקות. מצננים מיד על קרח.
- הוסף 1 μL כל אחד של 500 μM dCTP, 500 μM dGTP ו- 500 μM dTTP לצינור.
- הוסף 20 μL של המאגר המכיל hexamers נוקלאוטידים אקראיים שסופקו עם הערכה.
- הוסף 5 μL של הנוקלאוטיד המסומן רדיואקטיבית [α-32P] dATP לצינור. בצע את כל השלבים הכוללים חומר המסומן רדיואקטיבית תחת סביבת הגנה מאושרת רדיואקטיביות.
- הוסף 1 μL של מקטע Klenow, ודגר ב 37 ° C על thermomixer במשך 15 דקות עבור סינתזת DNA חד גדילי.
- הוסף 5 μL של מאגר עצירה כדי לעצור את התגובה.
- צנטריפוגה עמודת אי הכללת גודל למשך 2 דקות ב- 1,500 × גרם כדי להסיר את מאגר האחסון. השליכו את הזרימה והניחו את העמוד בצינור טרי.
- העבירו את תערובת הגשושית דרך העמוד כדי להסיר נוקלאוטידים רדיואקטיביים לא משולבים. צנטריפוגה במשך 30 שניות ב 1,500 × גרם כדי לאסוף את הגשושית.
- הוסף 150 μL של DNA זרע סלמון (1:100) כדי לחסום את הקישור הלא ספציפי של הבדיקה לממברנה.
- דגרו על תערובת הגשושית בטמפרטורה של 95°C למשך 5 דקות כדי לנטרל את הדנ"א הדו-גדילי.
- הקפיאו על קרח למשך מספר שניות, וצנטריפוגה למשך מספר שניות בטמפרטורה של 500 × גרם כדי להוריד את טיפות המים המעובות על המכסה.
- לשטוף בקצרה את קרום הכתם הדרומי עם חיץ הכלאה במשך 5-10 דקות עם סיבוב.
- טרום הכלאה של הממברנה עם חיץ ההכלאה למשך שעה אחת ב 55 ° C עם סיבוב. השליכו את מאגר ההכלאה.
- הוסף 15 מ"ל של חיץ הכלאה טרי (מחומם מראש ב 55 ° C) לממברנה יחד עם הבדיקה המוכנה. לדגור את הממברנה לילה ב 55 °C עם סיבוב.
- בצעו שטיפות לאחר הכלאה פעמיים במשך 15 דקות כל אחת עם 0.3x SSC, 0.1% SDS ב-55°C עם סיבוב.
- בצעו שטיפות לאחר הכלאה פעמיים במשך 15 דקות כל אחת עם 0.1x SSC, 0.1% SDS ב-55°C עם סיבוב.
- בצעו שטיפות לאחר הכלאה פעמיים במשך 15 דקות כל אחת עם 0.1x SSC, 1.5% SDS ב-55°C עם סיבוב.
- הסר את הממברנה מצינור ההכלאה, וחשף את הממברנה למסך זרחן למשך עד 3 ימים כדי לקבל את הטביעות הרדיואקטיביות על הסרט.
- קבל תמונות של הסרט באמצעות מערכת סריקת לייזר זרחן.
- הכנת דוגמאות
- ניתוח חלבונים
- הכנת דוגמאות
- התאם את כל דגימות החלבון לנפחים הסופיים שלהן (200 μL, 100 μL, 100 μL, 20 μL ו- 20 μL עבור תמצית התאים הגולמיים, הקלט, הזרימה, החרוזים והדגימות הפולטות, בהתאמה) על ידי הוספת 1 μL של β-מרקפטואתנול, מאגר דגימות PAGE LDS (1x) ו- dH2O.
- נטרל את הדגימות ב 95 ° C למשך 5 דקות.
- ניתוח כתמים מערביים
הערה: בצע SDS-PAGE וניקוי מערבי באמצעות פרוטוקולים סטנדרטיים15,16.- טען 15 μL מכל דגימה לכל באר של ג'ל SDS-PAGE 1.5 מ"מ.
- ביצוע אימונוסטיין של הכתם באמצעות נוגדני PAP (1:2,000) ו-LexA (1:1,000) עם דגירה לילית ב-4°C. יש לדלל בתמיסת חלב יבש/PBST 5%.
הערה: לאחר ניתוח PAP, ניתן להסיר את הכתם באמצעות פרוטוקול הפשטה קלה17 לפני הכתמה חיסונית עם נוגדן LexA השני.
- הכנת דוגמאות
תוצאות
הטיהור של תחום הכרומטין ARS316 ~ 1.4 kb תווך על ידי חלבון מתאם LexA-TAP המתבטא באופן מכונן . כדי לשמש כבקרה שלילית, ביצענו טיהור באמצעות זן איזוגני המבטא LexA-TAP אך אינו מכיל אתרים משולבים של RS ו-LexA. איור 3 מדגים את תוצאות ניתוח הדנ"א מניסוי טיהור סטנדרטי שבוצע הן על קבוצת הבקרה והן על זן ב?...
Discussion
זיהוי הגורמים ונוף הכרומטין של אזור גנומי מטרה ספציפי ממשיך להוות אתגר מרכזי במחקר הכרומטין18. פרוטוקול זה מתאר מערכת יעילה לכריתה וטיהור ספציפית של תחומי כרומטין נפרדים מכרומוזומי שמרים. למיטב ידיעתנו, הטוהר והתשואה של טיהור חד-שלבי זה מתגברים על רבות מהמגבלות של שיטות טיהו?...
Disclosures
למחברים אין ניגודי עניינים לחשוף.
Acknowledgements
העבודה במעבדת S.H. נתמכה על ידי DFG באמצעות SFB1064 (מזהה פרויקט 213249687), מועצת המחקר האירופית (ERC Starting Grant 852798 ConflictResolution) והלמהולץ Gesellschaft.
Materials
| Name | Company | Catalog Number | Comments |
| Yeast strains | |||
| Control Strain: MATa; ura3Δ0; leu2Δ0; his3Δ1; met15Δ0; bar1::kanMX4; Chr I 212kb::LEU2 pTEF2-LEXA-TAP pGAL1-10 RecR | Section 1, see references 13 and 14 | ||
| Recombination Strain: MATa; ura3Δ0; leu2Δ0; his3Δ1; met15Δ0; bar1::kanMX4; RS_LEXA_NS-3_ARS316_NS+3_RS; Chr I 212kb::LEU2 pTEF2-LEXA-TAP pGAL1-10 RecR | Section 1, see reference 13 and 14 | ||
| Plasmid | |||
| K238 plasmid | Section 1, see reference 13 Storage: Store at -20 °C | ||
| K071 Spike-in plasmid DNA | Section 7.1, see reference 13 Storage: Store at -20 °C | ||
| Reagents | |||
| Acetone | Carl Roth | 5025.1 | Section 2 Storage: Store at room temperature |
| Ammonium acetate (NH4Ac) | Sigma Aldrich | A7262 | Section 6 and 7.1 Storage: Store at room temperature |
| Ammonium solution (NH4OH) 25% | Merck Millipore | 533003 | Section 6 Storage: Store at room temperature |
| Ammonium sulfate | Santa Cruz | Sc-29085 | Section 2 Storage: Store at room temperature |
| Bacto agar | BD (VWR) | 90000-760 | Section 3 Storage: Store at room temperature |
| Bacto peptone | BD (VWR) | 211820 | Section 3 Storage: Store at room temperature |
| β-Mercaptoethanol | Sigma Aldrich | 07604 | Section 7.2 Storage: Store at 4 °C |
| Chemiluminescent substrate kit | ThermoFisher | 34580 | Section 7.2 Storage: Store at 4 °C |
| Di-Sodium Hydrogen phosphate dodecahydrate | Merck | 1.06579.1000 | Section 2 and 7.1 Storage: Store at room temperature |
| Dithiothreitol (DTT) | ThermoFisher | 15508013 | Section 4 Storage: Store at 4 °C |
| Ethanol | Merck | 100983 | Section 7.1 Storage: Store at room temperature |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma Aldrich | ED | Section 7.1 Storage: Store at room temperature |
| Galactose (20% (w/v) stock) | Sigma Aldrich | G0625-1KG / 5KG | Section 3 Storage: Store at room temperature |
| Gel loading dye (6x) | BioLabs | B7024A | Section 7.1 Storage: Store at -20 °C |
| Glusose | Sigma-Aldrich | G8270 | Section Storage: Store at room temperature |
| Glycine | Carl Roth | .0079.4 | Section 2 Storage: Store at room temperature |
| Glycogen (5 mg/mL) | Invitrogen | AM9510 | Section 7.1 Storage: Store at -20 °C |
| Hydrochloric acid (HCl) | PanReac AppliChem | 182109.1211 | Section 2, 4 and 7.1 Storage: Store at room temperature |
| Magnesium Acetate (MgAc) | Bernd Kraft | 15274.2600/C035 | Section 4 Storage: Store at room temperature |
| Magnesium chloride (MgCl2) | Sigma Aldrich | M8266 | Section 6 Storage: Store at room temperature |
| Nu PAGE LDS sample buffer (4x) | Invitrogen | 2399549 | Section 7.2 Storage: Store at room temperature |
| Phenol/Chloroform/Isoamyl alcohol (25:24:1 v/v) | Invitrogen | 15593-031 | Section 7.1 Storage: Store at 4 °C |
| Potassium chloride (KCl) | Sigma | P9541 | Section 4 Storage: Store at room temperature |
| Radioactively labeled α-32P dATP (3,000 Ci/mmol, 10 mCi/mL) | Hartmann Analytic | SRP-203 | Section 7.1 Storage: Store at 4 °C |
| RadPrime labeling system | ThermoFisher | 18428-011 | Section 7.1 Storage: Store at -20 °C |
| Raffinose (20% (w/v) stock) | SERVA | 34140.03 | Section 3 Storage: Store at room temperature |
| Sodium chloride (NaCl) | Merck | K53710504142 | Section 7.1 Storage: Store at room temperature |
| Sodium citrate (Na3C6H5O7) | Sigma-Aldrich | 71402 | Section 7.1 Storage: Store at room temperature |
| Sodium hydroxide (NaOH) | Sigma Aldrich | S5881 | Section 7.1 Storage: Store at room temperature |
| Sodium n-dodecyl sulfate (SDS) (5% stock (w/v) ) | Alfa Aesar | A11183 | Section 7.1 Storage: Store at room temperature |
| Sodium phosphate monobasic | Sigma-Aldrich | 71496 | Section 2 and 7.1 Storage: Store at room temperature |
| Sodium azide | Santa Cruz Biotechnology | sc-208393 | Section 2 Storage: Store at -20 °C |
| Triethylamine | Sigma Aldrich | 90340 | Section 2 Storage: Store at room temperature |
| Tris base | Chem Cruz | SC-3715B | Section 2 and 4 Storage: Store at room temperature |
| Triton X-100 | Sigma Aldrich | X100 | Section 2 and 4 Storage: Store at room temperature |
| Tween-20 | Bernd Kraft | 18014332 | Section 4 Storage: Store at room temperature |
| Yeast extract | BD (VWR) | 212720 | Section 3 Storage: Store at room temperature |
| Yeast mating factor alpha (1 µg/mL stock ) | Biomol | Y2016.5 | Section 3 Storage: Store at -20 °C |
| Yeast Synthetic Drop-out medium Supplements without LEUCINE | Sigma Aldrich | Y1376 | Section 1, see reference 14 |
| Enzymes | |||
| HpaI restriction enzyme (5,000 U/mL) | NEB | R0105S | Section 7.1 Storage: Store at -20 °C |
| Protease and Phosphatase Inhibitor Cocktail (100x) | ThermoFisher Scientific | 78446 | Section 4 Storage: Store at4 °C |
| Proteinase K (10 mg/mL) | SERVA | 33756 | Section 7.1 Storage: Store at -20 °C |
| RNase A (10 mg/mL) | ThermoFisher | EN0531 | Section 7.1 Storage: Store at -20 °C |
| TEV protease (10000 U/µL) | NEB | P8112S | Section 5 Storage: Store at -20 °C |
| Materials | |||
| BcMag Epoxy-Activated Magnetic Beads | Bioclone Inc. | FC-102 | Section 2 Storage: Store at 4 °C |
| Dry ice | Section 4 | ||
| Low-binding centrifuge tubes 2.0 mL | Eppendorf | 22431102 | Section 4 |
| Microspin G-25 Columns | Cytiva | 27-5325-01 | Section 7.1 Storage: Store at room temperature |
| Parafilm | Merck | P7793 | Section 4 |
| Positive nylon membrane | Biozol | 11MEMP0001 | Section 7.1 Storage: Store at room temperature |
| PVDF transfer membrane | Immobilon-Merck Millipore | IPVH00010 | Section 7.2 Storage: Store at room temperature |
| SDS-PAGE gel 4-12% bis-tris (15 well, 1.5 mm) | Invitrogen | NP0336BOX | Section 7.2 Storage: Store at 4 °C |
| Syringe (25 mL) with luer fitting | Henke Sass Wolf | 4200-000V0 | Section 3 |
| Whatman paper (Grade 3MM CHR Cellulose Western Blotting Paper Sheet) | Cytiva | 3030-917 | Section 7.1 Storage: Store at room temperature |
| Antibodies | |||
| Anti-LexA, rabbit polyclonal IgG, DNA binding region antibody | Merck Millipore | 06-719 | Section 7.2 Storage: Store at -20 °C |
| Goat Anti-Rabbit IgG (H+L), Horseradish peroxidase conjugate | Invitrogen | G21234 | Section 7.2 Storage: Store at -20 °C |
| Peroxidase Anti-Peroxidase (PAP) antibody produced in rabbit for the detection of TAP-tagged proteins | Sigma Aldrich | P1291-500UL | Section 7.2 Storage: Store at -20 °C |
| Rabbit IgG antibodies | Sigma | I5006-100MG | Section 2 Storage: Store at 4 °C |
| Primers (10 µM) | |||
| ARS316: fwd 5'- CGGCATTATCGTACACAACCT, rev 5'- GTTCTTCGTTGCCTACATTTTCT | Section 7.1 | ||
| K071 Spike-in plasmid DNA: fwd: 5'-TTTTCGCTGCTTGTCCTTTT, rev 5'- CATTTTCGTCCTCCCAACAT | Section 7.1 | ||
| PCR fragment from yeast genomic DNA as a template for ARS316 amplification (for southern blot): fwd 5’- AAATTCTGCCCTTGATTCGT rev 5’- TTTGTTTATCTCATCACTAAT | Section 7.1 | ||
| PDC1: fwd 5'- CATGATCAGATGGGGCTTCA, rev 5'-ACCGGTGGTAGCGACTCTGT | Section 7.1 | ||
| Equipment | |||
| Coffee grinder | Gastroback | 42601 | Section 4 |
| Dewar flask | NAL GENE | 4150-2000 | Section 3 |
| DynaMag TM-2 magnetic rack | Invitrogen | 12321D | Section 4, 5 and 6 |
| Hybridization oven | Hybaid Mini10 | Ri418 | Section 2 |
| Microcentrifuge | Eppendorf | 5424R | Section 4 and 7.1 |
| UV-crosslinker | Analytikjena | 95-0174-02 | Section 7.1 |
References
- Kornberg, R. D., Lorch, Y. Twenty-five years of the nucleosome, fundamental particle of the eukaryote chromosome. Cell. 98 (3), 285-294 (1999).
- Gauchier, M., van Mierlo, G., Vermeulen, M., Déjardin, J. Purification and enrichment of specific chromatin loci. Nature Methods. 17 (4), 380-389 (2020).
- Korthout, T., et al. Decoding the chromatin proteome of a single genomic locus by DNA sequencing. PLoS Biology. 16 (7), e2005542 (2018).
- Antão, J. M., Mason, J. M., Déjardin, J., Kingston, R. E. Protein landscape at Drosophila melanogaster telomere-associated sequence repeats. Molecular and Cellular Biology. 32 (12), 2170-2182 (2012).
- Déjardin, J., Kingston, R. E. Purification of proteins associated with specific genomic loci. Cell. 136 (1), 175-186 (2009).
- Ide, S., Dejardin, J. End-targeting proteomics of isolated chromatin segments of a mammalian ribosomal RNA gene promoter. Nature Communications. 6, 6674 (2015).
- Unnikrishnan, A., Gafken, P. R., Tsukiyama, T. Dynamic changes in histone acetylation regulate origins of DNA replication. Nature Structural & Molecular Biology. 17 (4), 430 (2010).
- Buxton, K. E., et al. Elucidating protein-DNA interactions in human alphoid chromatin via hybridization capture and mass spectrometry. Journal of Proteome Research. 16 (9), 3433-3442 (2017).
- Kennedy-Darling, J., et al. Discovery of chromatin-associated proteins via sequence-specific capture and mass spectrometric protein identification in Saccharomyces cerevisiae. Journal of Proteome Research. 13 (8), 3810-3825 (2014).
- Liu, S. J., et al. CRISPRi-based genome-scale identification of functional long noncoding RNA loci in human cells. Science. 355, 7111 (2017).
- Hamperl, S., et al. Purification of specific chromatin domains from single-copy gene loci in Saccharomyces cerevisiae. Functional Analysis of DNA and Chromatin. 1094, 329-341 (2014).
- Hamperl, S., et al. Compositional and structural analysis of selected chromosomal domains from Saccharomyces cerevisiae. Nucleic Acids Research. 42 (1), 2 (2014).
- Weiβ, M. Single-copy locus proteomics of early- and late-firing DNA replication origins identifies a role of Ask1/DASH complex in replication timing control. Cell Reports. 42 (2), 112045 (2023).
- Gietz, R. D., Schiestl, R. H. High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method. Nature Protocols. 2 (1), 31-34 (2007).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. Proceedings of the National Academy of Sciences of the United States of America. 76 (9), 4350-4354 (1979).
- Western blot membrane stripping for restaining protocol. Abcam Available from: https://www.abcam.com/protocols/western-blot-membrane-stripping-for-restaining-protocol (2023)
- Vermeulen, M., Déjardin, J. Locus-specific chromatin isolation. Nature Reviews. Molecular Cell Biology. 21 (5), 249-250 (2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved