Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Microscopía de imagen de por vida (FLIM) de fluorescencia de células vivas multiparamétricas y producción de esferoides multicelulares
* Estos autores han contribuido por igual
En este artículo
Resumen
En este trabajo se describen diferentes métodos de formación de esferoides multicelulares para realizar un seguimiento de la microscopía multiparamétrica de células vivas. Mediante el uso de microscopía de imágenes de fluorescencia (FLIM), autofluorescencia celular, colorantes de tinción y nanopartículas, se demuestra el enfoque para el análisis del metabolismo celular, la hipoxia y la muerte celular en cáncer tridimensional vivo (3D) y esferoides derivados de células madre.
Resumen
Los esferoides tumorales multicelulares son un modelo popular de microagregados de tejidos en 3D para reproducir el microambiente tumoral, probar y optimizar terapias farmacológicas y utilizar biosensores y nanosensores en un contexto 3D. Su facilidad de producción, tamaño predecible, crecimiento y gradientes de nutrientes y metabolitos observados son importantes para recapitular el microambiente celular de nicho en 3D. Sin embargo, la heterogeneidad de los esferoides y la variabilidad de sus métodos de producción pueden influir en el metabolismo celular general, la viabilidad y la respuesta a los fármacos. Esto dificulta la elección de la metodología más adecuada, teniendo en cuenta los requisitos en tamaño, variabilidad, necesidades de biofabricación y uso como modelos de tejidos 3D in vitro en biología de células madre y cancerosas. En particular, la producción de esferoides puede influir en su compatibilidad con microscopías cuantitativas en vivo, como las imágenes metabólicas ópticas, la microscopía de imágenes de fluorescencia durante toda la vida útil (FLIM), el seguimiento de la hipoxia de esferoide con nanosensores o la viabilidad. Aquí, se presentan una serie de protocolos convencionales de formación de esferoides, destacando su compatibilidad con las microscopías vivas de campo amplio, confocales y de dos fotones. También se presenta la línea de imágenes de seguimiento para análisis con FLIM de autofluorescencia multiplexada y, utilizando varios tipos de esferoides de células madre y cáncer.
Introducción
Los esferoides multicelulares representan un grupo de modelos de tejidos en 3D obtenidos por la autoagregación de células y que presentan una forma esférica. Son ampliamente utilizados para imitar la interacción célula-célula y célula-matriz in vitro y para reproducir un contexto 3D dentro de una multitud de construcciones derivadas del cáncer y las células madre. Se emplean varias técnicas para reducir la adhesión celular y promover la agregación. Estos incluyen el método de caída colgante que se basa en la tensión superficial1; métodos de repelencia de la fijación de células, como placas de fijación ultrabajas, micromoldes y micropocillos 2,3; enfoque basado en ondas acústicas4; métodos de agregación inducida por flujo (matraces giratorios, biorreactores y dispositivos microfluídicos)5; formación asistida por partículas magnéticas6 y uso de matrices y andamios sintéticos y basados en ECM que promueven la agregación 7,8,9.
En la investigación, desarrollo y validación de nuevas terapias farmacológicas contra el cáncer, los esferoides son un modelo atractivo debido a su capacidad para recapitular los gradientes de difusión espacial limitados de nutrientes, productos de desecho yO2, lo que a menudo conduce a la formación de un núcleo necrótico, típico de los tumores sólidos10,11. Estos modelos in vitro más confiables y sofisticados desafían la necesidad de un uso extensivo de modelos animales (Ley de Modernización de la Administración de Alimentos y Medicamentos [FDA]2.0 12), de acuerdo con el principio de las 3R de la investigación con animales (reemplazo, reducción y refinamiento). Además del cáncer, los esferoides encuentran su aplicación en la investigación con células madre. Por ejemplo, las células madre pluripotentes tienen la capacidad de formar cuerpos embrioides (EB), que pueden utilizarse para la diferenciación de células madre pluripotentes inducidas (iPSC) hacia tipos de células especializadas que son difíciles de obtener directamente de los pacientes, como las células precursoras neurales13 o las células de la granulosa ovárica 13,14. Además, la formación de un EB suele ser el primer paso en el desarrollo de modelos de organoides más complejos, por ejemplo, el neuro15, el retinal16, el cardíaco17, el hígado18, el estómago19 y los organoides intestinales20. Factores como el tamaño, la reproducibilidad, el rendimiento y las aplicaciones posteriores deben tenerse en cuenta a la hora de elegir un método de formación de esferoides adecuado para los experimentos.
El aumento de la complejidad de la cultura 3D puede conducir a una mayor variabilidad en comparación con la cultura 2D. Factores como la composición de nutrientes21, la evaporación del medio22, la viscosidad23, el control del pH24, el método de formación de esferoides e incluso el tiempo en el cultivo25,26 pueden dar lugar a la obtención de esferoides de diferente morfología, tamaños, viabilidad y diferentes quimiorresistencias27,28. Investigaciones recientes demostraron que los gradientes de oxígeno de los esferoides no siempre son estáticos y se ven afectados por el método de formación, el tamaño del esferoide y la viscosidad extracelular, lo que afecta la heterogeneidad de los esferoides29. Para mejorar la reproducibilidad y la accesibilidad a los datos sobre esferoides, se ha desarrollado la base de conocimientos MISpheroID26, identificando la línea celular, el medio de cultivo, el método de formación y el tamaño de los esferoides como la información mínima para un resultado reproducible. Por lo tanto, se realizó una comparación detallada de múltiples métodos de alto rendimiento (SphericalPlate 5D, micromoldes hechos en laboratorio y moldes de microtejidos) y de baja adherencia (es decir, Biofloat y placas de 96 pocillos recubiertas de lipidure, tanto sin andamios como basados en andamios) (Figura 1 y Tabla 1), incluido el tamaño del pocillo (dada una estimación del tamaño máximo del esferoide), los consumibles utilizados, el tiempo de preparación y la posibilidad de monitorear los esferoides sin transportarlos a placas de microscopía. Este último permite estudios a largo plazo, mientras que los esferoides producidos con métodos de alto rendimiento a menudo dan lugar a experimentos de punto final. Todos los métodos, a excepción de las rejillas de la placa 5Dspheri, no aportan autofluorescencia no deseada, lo que permite su uso directo en microscopía.
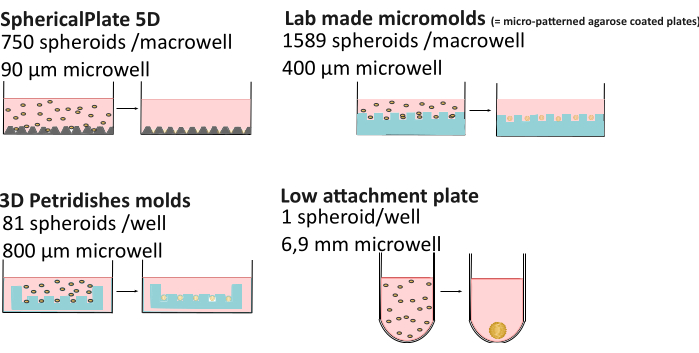
Figura 1: Explicación de los métodos de formación de esferoides. Métodos de alto rendimiento como el SphericalPlate 5D, que ha integrado micropocillos patentados en la placa, mientras que los micromoldes producidos en laboratorio y los moldes MicroTissue utilizan sellos para hacer múltiples micropocillos en agarosa (azul). Las placas de baja adherencia como Lipidure (Amsbio) y Biofloat (Sarstedt) utilizan un recubrimiento no adherente que inhibe la adhesión a la superficie celular y promueve la autoagregación celular. Haga clic aquí para ver una versión más grande de esta figura.
| 5D SpheriPlate | Micromoldes de producción propia | Microtejido | Métodos de apego bajo | |
| Número de esferoides/pocillo | 750 | 1589 | 81 | 1 |
| Diámetro del pozo | 90 μm | 400 μm | 800 μm | 1 milímetro |
| Volumen de cultivo | 1 mL | 5 mL | 1 mL | 200 μL |
| Otros consumibles | / | 7 mL de agarosa al 3% | 500 μL de agarosa al 2% | / * |
| Tiempo de preparación | 10 minutos | 2 h + 3 días de adaptación de medios | 0,5 h + 15 min adaptación de medios | 10–30 min + 1 h de secado |
| Monitorización | Sí | No** | Sí | Sí |
| Autofluorescente | Sí | No | No | No |
| Reutilizable | No | Sí | Sí | No** |
| Costar | €€ | € | €€€€ | €€€€: Recubrimiento y Matrigel |
| €€: Placa comercial de 96 pocillos | ||||
| *Algunas líneas celulares necesitan la adición de ECM (es decir, 2%-5% de Matrigel) para formar esferoides compactos. | ||||
| **El recubrimiento es reutilizable hasta que se agote. Sin embargo, cada placa consumirá una pequeña cantidad de medio y el polvo puede acumularse con el tiempo. La esterilización del filtro es necesaria regularmente. | ||||
Tabla 1: Comparación de múltiples métodos de formación de esferoides29. "Monitoreo": la capacidad de monitorear esferoide sin la necesidad de transferirlo a una placa de microscopía. €: 0-50€, €€: 50-150€ , €€€: 150-500€ , €€€€: >500€
La microscopía de fluorescencia permite el seguimiento directo de los aspectos biológicos clave dentro de los esferoides, incluida la muerte celular, la viabilidad, la proliferación, el metabolismo, la viscosidad e incluso las propiedades mecánicas30. La microscopía de imagen de fluorescencia (FLIM) proporciona una dimensión cuantitativa adicional para estudiar las interacciones de las sondas fluorescentes dentro de su (micro)entorno 31,32,33,34, lo que permite resolver los espectros de emisión superpuestos de acuerdo con diferentes tiempos de vida de emisión 35,36 y sondear el metabolismo celular a partir de la autofluorescencia celular intrínseca. Por lo tanto, compuestos autofluorescentes celulares tan extendidos como el fosfato de nicotinamida adenina dinucleótido (NAD(P)H), el mononucleótido de flavina (FMN), el dinucleótido de flavina y adenina (FAD), la protoporfirina IX y otros pueden medirse con FLIM de uno y dos fotones y sirven como "sensores" intrínsecos del catabolismo de la glucosa, la fosforilación oxidativa (OxPhos) y proporcionan una visión general del estado redox celular. El NAD(P)H existe en formas citoplasmáticas libres o en mitocondriales unidas a proteínas37,38. Del mismo modo, el estado oxidado de FAD es fluorescente con una vida útil más larga de la forma libre. Las microscopías de NAD(P)H y FAD generalmente involucran FLIM excitado por dos fotones, con el objetivo de prevenir el fotodaño de la muestra39. Con frecuencia, la "imagen metabólica óptica" FLIM puede combinarse con el uso de sondas basadas en colorantes, biosensores codificados genéticamente, microscopía de imágenes de fosforescencia (PLIM) y mediciones basadas en la intensidad radiométrica para proporcionar una imagen más completa del metabolismo, la oxigenación, la proliferación y la viabilidad celular de esferoides u organoides 29,30,31. Además, FLIM también puede combinarse con el método de transferencia de energía por resonancia de Förster (FRET) para medir la variación de la vida útil del fluoróforo donante cuando está en contacto cercano con el aceptor para investigar la unión de un fármaco con su dominio diana33,40,41.
Las imágenes FLIM adquiridas se analizan normalmente para calcular la vida útil píxel por píxel. En la actualidad, existen al menos 3 estrategias comunes utilizadas para obtener la vida útil de la fluorescencia: "FLIM rápido" semicuantitativo42 (a veces denominado "sentido tau"43,44), ajuste de la curva de decaimiento, utilizando un ajuste exponencial de uno, dos o tres elementos, y enfoque "sin ajuste" con transformación de fasores y análisis de diagramas de fasores. Dependiendo del proveedor, se puede utilizar software proporcionado (LAS X, Symphotime, SPCImage, etc.) o de código abierto (por ejemplo, FLIMfit45, FLIMJ46 u otros47) para manejar los datos FLIM medidos. Por lo general, el software proporcionado por el proveedor es útil para el análisis preliminar de datos, mientras que las soluciones de código abierto pueden proporcionar estudios más precisos utilizando, por ejemplo, diagramas de fasores y visualización en 3D.
A pesar de la utilidad y el atractivo de FLIM como método para estudiar esferoides, hay muy pocos protocolos experimentales disponibles, y existe una falta general de conocimiento en la elección del método de formación más apropiado para el éxito de experimentos de microscopía multiparamétrica en vivo con FLIM. Aquí, se presenta una comparación detallada de los protocolos de formación de esferoides comúnmente utilizados en función de su morfología, viabilidad y oxigenación con el nanosensor de detección de oxígeno (MMIR1) de rojo lejano e infrarrojo cercano (NIR) recientemente validado y caracterizado. La nanopartícula catiónica está impregnada con dos colorantes reporteros, el aza-BODIPY insensible al O2 de referencia (excitación 650 nm, emisión 675 nm) y la metaloporfirina sensible al NIR O2, PtTPTBPF (excitación 620 nm, emisión 760 nm). El MMIR1 permite el análisis en tiempo real de los gradientes de oxígeno en un microscopio de fluorescencia convencional (mediante análisis radiométrico) o en un microscopio de fosforescencia (PLIM) sin introducir toxicidad celular y permitiendo señales estables, monitorización a largo plazo y multiplexación25,29. Dependiendo de la necesidad de tinción con colorantes o nanosensores, el rendimiento de los esferoides o el tipo de célula, se puede elegir el protocolo de formación más adecuado. Dado que los estudios de viabilidad y oxigenación de los esferoides son relevantes para los estudios de esferoides derivados del cáncer y de las células madre, los protocolos presentados también incluyen ejemplos y resultados típicos esperados de NAD(P)H-FLIM y FAD-FLIM con estos modelos. Las tuberías de imágenes y análisis presentadas se dirigen a las plataformas de microscopía FLIM basadas en el recuento de fotones individuales correlacionadas en el tiempo más populares.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Generación de esferoides multicelulares
- Cultivo celular
NOTA: Los cultivos celulares se pueden recolectar de la American Type Culture Collection (ATCC), Lonza, Sigma-Aldrich u otros proveedores. ATCC proporciona toda la información de manejo requerida, incluidos los medios de crecimiento preferidos, los procedimientos de subcultivo, el nivel de bioseguridad, la tasa de crecimiento y los perfiles STR. En este caso, se utilizan 500 células/esferoide de la línea celular HCT116 de cáncer de colon humano en los medios 5A de McCoy (VWR, 392-0420) suplementados con un 10% de FBS y 1 mM de piruvato de sodio. Para experimentos a largo plazo que se monitorean diariamente, se pueden agregar 10 mM de HEPES, pH 7.2 a los medios.- Cultive el cultivo celular para alcanzar una confluencia del 70%-90%.
- Enjuague las células con PBS estéril precalentado (37 °C) (5 ml por matraz de 25cm2 o 10 ml por matraz de 75cm2 ).
- Añadir tripsina al 0,05% - 1 mM de solución de EDTA (0,5 mL por 25 cm² o 1 mL por matraz de 75cm2 ) e incubar durante 5-10 min a 37 °C con 5% de CO2, 95% de humedad para alcanzar el desprendimiento de células.
NOTA: Controle el desprendimiento de la célula bajo el microscopio de luz de transmisión (campo claro). La sobreexposición de las células a la solución enzimática disociante puede afectar su viabilidad. - Neutralice la tripsina añadiendo un exceso de medios de cultivo celular que contengan un 10% de FBS (al menos 5 mL de medio por 1 mL de solución de disociación).
NOTA: Para las células cultivadas en medios de cultivo con bajo contenido de FBS o sin FBS, la neutralización de tripsina se puede realizar con la adición de 0,5 mL de FBS al 100% de FBS al cultivo celular tratado con tripsina, seguido de centrifugación para transferir las células a sus medios de cultivo originales. - Disociar los agregados celulares mediante pipeteo para obtener una suspensión de una sola célula en el medio.
NOTA: El pipeteo con una pipeta serológica con una punta de pipeta de 1000 μL en la parte superior mejora enormemente la generación de suspensión unicelular en un gran volumen de cultivo de células en suspensión. - Utilice una cámara de recuento (hemocitómetro refrenado por Neubauer o alternativas) para contar el número de células por 1 mL de la suspensión celular.
- Diluir la suspensión celular para obtener el número deseado de células por mililitro.
- Añadir una solución concentrada de sonda deO2 (nanopartículas) a la suspensión celular a una concentración final de 10 μg/mL para el análisis radiométrico deO2.
NOTA: Para asegurar una suspensión celular homogénea (con la sonda), vuelva a suspender varias veces antes de la formación de esferoides. Si no se requiere la sonda deO2 , omita este paso y proceda con la formación de los esferoides. Se debe aplicar un protocolo modificado cuando se manejan iPSC. En resumen, las iPSC se cultivan en colonias en placas recubiertas de Geltrex y se pasan utilizando ReLeSR como se describe en el protocolo proporcionado por el proveedor48. El día de la formación de los esferoides, las colonias deben ser grandes, compactas y exhibir centros de múltiples capas con bordes distintos. Enjuague las células con PBS estéril precalentado. Añadir 1 mL de reactivo de disociación celular suave (GCDR) e incubar a 37 °C durante 8-10 min. Utilice una punta de 1000 μL para separar suavemente las células del pocillo y obtener una suspensión unicelular. Transfiera la suspensión de una sola célula a un tubo cónico estéril de 50 mL y agregue 4 mL de medio DMEM-F12 precalentado para neutralizar el GCDR. Lave el pocillo con 1 mL de DMEM-F12 y transfiéralo al resto de la suspensión celular. Centrifugar a 300 x g durante 5 min. Vuelva a suspender en 1 ml de medio apropiado para realizar más experimentos. Para los experimentos descritos en este manuscrito, se utilizó mTeSR + 10 μM Rock Inhibitor. Cuente y diluya la suspensión celular para obtener el número deseado de células por mililitro.
- Métodos de formación de esferoides
- Micromoldes de placas de Petri 3D
NOTA: Este método de alto rendimiento se utiliza para generar simultáneamente un gran número de esferoides (81 esferoides) en una matriz de micromoldes de 9 x 9 con 800 μm de diámetro y 800 μm de profundidad.- Enjuague los micromoldes para fundir placas de Petri 3D en dH2O y colóquelos en un recipiente esterilizable en autoclave.
- Mida 2 g de polvo de agarosa apto para electroforesis y colóquelo en un frasco de vidrio seco de 200 ml apto para autoclave.
NOTA: Asegúrese de que la botella y el polvo de agarosa estén secos, sin líquido ni humedad. - Micromoldes de autoclave para la fundición de placas de Petri 3D y botellas con polvo de agarosa durante 30 min en ciclo seco.
- Prepare una solución salina de 0,9 p/v % mediante la adición de 0,9 g de NaCl en 100 ml de agua ultrapura y esterilícela en autoclave.
NOTA: El NaCl es recomendado por el fabricante. Aumenta la estabilidad de la agarosa. - Prepare la solución de agarosa añadiendo la solución salina estéril al polvo de agarosa esterilizado. Atornille la tapa sin apretar para evitar la acumulación de presión. Agita la botella para mezclar el polvo de agarosa.
- Hervir y disolver el polvo de agarosa usando un horno de microondas. Detenga el microondas con frecuencia (~cada 10 s). Agite la botella y repita hasta que la agarosa se disuelva.
PRECAUCIÓN: La solución de agarosa está caliente y requiere un manejo cuidadoso. Agitar inmediatamente después del procedimiento de fusión puede hacer que la solución explote fuera del recipiente. Para evitar accidentes, utilice recipientes suficientemente grandes llenos hasta no más del 50% de su capacidad y utilice protección personal adecuada (guantes, guante de cocina, protección ocular y bata de laboratorio). - Deje que la solución de agarosa disuelta se enfríe a 60-70 °C. Utilizando técnicas y condiciones asépticas, pipetear la agarosa fundida en el micromolde (500 μL para la serie 12 o 330 μL para la serie 24).
NOTA: Evite la formación de burbujas al mezclar o pipetear la agarosa. Elimine las pequeñas burbujas atrapadas en las pequeñas características del micromolde pipeteando o raspando suavemente antes de que la agarosa se solidifique. - Deje que la agarosa se solidifique durante unos 2-3 minutos. A continuación, flexiona con cuidado el micromolde para retirar la placa de Petri 3D y transfierirla a una placa de cultivo de tejidos de 12 pocillos.
NOTA: La flexión excesiva del micromolde puede provocar la formación de grietas dentro del molde de agarosa. - Para equilibrar la placa de Petri 3D, agregue 2,5 mL/pocillo de medio de cultivo celular. Incubar durante 15 minutos o más. Retire el medio de cultivo y reemplácelo con medio fresco. Repita una vez más para equilibrar la placa de Petri 3D con un medio de cultivo.
NOTA: El protocolo puede interrumpirse aquí hasta la siembra de células. Para el almacenamiento a largo plazo (hasta 2 semanas a 4 °C), utilice una solución de PBS en lugar de un medio. - Retire completamente la placa de Petri 3D que rodea el medio de cultivo (o PBS) y retire con precisión el medio dentro de la placa de Petri 3D inclinando la placa de cultivo de tejidos.
- Siembre cuidadosamente 190 μL de suspensión celular que contiene 40.500 células gota a gota en la cámara de siembra celular (ver paso 1.1).
NOTA: El número de micropocillos en los sellos de agarosa determina el número de esferoides producidos por sello. En este caso, este molde de agarosa contiene 81 micropocillos (81 x 500 células/esferoide). La variación de la concentración celular en suspensión añadida a un macropocillo permite cambiar el número de células por esferoide, controlando así el tamaño del esferoide. - Espere ~ 10 minutos para que las células se asienten en las características del 3D Petri dsh. A continuación, añade 2 ml de medio al exterior de la placa de Petri 3D.
- Coloque la placa de cultivo de tejidos en la incubadora de cultivos celulares e intercambie el medio que rodea la placa de Petri 3D según sea necesario.
- Placa de fijación baja
NOTA: Este método se utiliza para generar un solo esferoide por pocillo. Las placas recubiertas (Lipidure o Biofloat) están disponibles comercialmente (omita los pasos 1.2.2.1-1.2.2.4). Alternativamente, el recubrimiento se puede comprar por separado y se puede usar para recubrir placas de pocillos múltiples sin tratar. Se recomienda llenar los pozos en los bordes de las placas de 96 pocillos con agua estéril o PBS debido a la evaporación más rápida en estos pocillos, limitando así el número de pozos para esferoides a 60. En caso de que se necesiten menos esferoides, llene los pozos vacíos circundantes con agua o PBS. Utilice puntas libres de polvo para el manejo de líquidos para evitar traer partículas pequeñas a los pozos, ya que interfieren con la formación de esferoides.- Prepare una solución de recubrimiento de 0,5 p/v % disolviendo 0,25 g del polvo polimérico en 50 ml de etanol en un recipiente de botella de vidrio. Filtrar-esterilizar el recubrimiento.
NOTA: La esterilización del filtro y todos los pasos siguientes deben llevarse a cabo en condiciones estériles bajo flujo laminar. - Agregue 200 μL de la solución de recubrimiento a cada pocillo de una placa de cultivo de fondo de 96 U.
- Incubar durante 1 min y retirar el exceso de recubrimiento.
NOTA: La solución de recubrimiento se puede usar varias veces. Almacenar en un recipiente de vidrio a temperatura ambiente (RT). No se recomiendan los recipientes de plástico, ya que el plástico puede disolverse parcialmente y convertirse en parte de la solución. Si hay polvo, esterilice el filtro con un filtro de polietersulfona (PES) o jeringa de nailon de 0,22 μm. - Deje que la placa de 96 pocillos se seque al aire durante aproximadamente 1 h.
NOTA: Si las células necesitan una matriz extracelular, continúe con el paso 1.2.3. Las placas recubiertas se pueden almacenar en RT cuando están envueltas en aluminio hasta por 1 mes. Al sembrar una cantidad menor de células/pocillo, centrifugar la placa durante 5 minutos a 300 g puede ayudar a reducir las células.
- Prepare una solución de recubrimiento de 0,5 p/v % disolviendo 0,25 g del polvo polimérico en 50 ml de etanol en un recipiente de botella de vidrio. Filtrar-esterilizar el recubrimiento.
- Protocolo de formación asistida por matriz extracelular
NOTA: Algunas líneas celulares no producen suficiente matriz extracelular (MEC) por sí mismas y necesitan la adición de MEC, como Matrigel, Cultrex o Geltrex, para formar esferoides compactos 49,50,51. Para tipos de células tales como el cáncer de mama MDA-MB-231, la papila dérmica humana, las células de cáncer de próstata y otras, es posible utilizar el paso 1.2.2 con las siguientes modificaciones52, que requieren la adición de ECM. Preferiblemente, use puntas libres de polvo para el manejo de líquidos para evitar que el polvo interfiera con la formación de esferoides. Los pasos 1 y 4-7 deben llevarse a cabo bajo una cabina de seguridad biológica (clase II).- Continúe con los pasos 1.2.2.1-1.2.2.4 (placas de fijación bajas) para el tratamiento de superficies.
- Enfríe previamente la placa de 96 pocillos a 4 °C en el refrigerador.
- Prepare la centrífuga con el adaptador correcto para la placa de 96 pocillos y preenfríela a 4 °C.
- Prepare una solución al 5% de matriz de membrana basal (BMM) en un medio de cultivo celular preenfriado (4 °C).
NOTA: BMM se entrecruza rápidamente en RT. Al manipular, mantenga las existencias y las soluciones en hielo. - Prepare la suspensión celular (consulte el paso 1.1).
- Agregue 50 μL de la solución de BMM en cada pocillo.
- Agregue suavemente 50 μL de suspensión celular en cada pocillo encima de la solución de BMM (25,000 celdas/pocillo).
NOTA: No arroje este volumen al pocillo, o de lo contrario, las células se extenderán por los lados de los pocillos y no se acumularán en el fondo. Se puede obtener un menor número de celdas por pocillo ajustando la densidad de celdas en consecuencia. No todas las células formarán esferoides en todas las densidades de siembra; La optimización debe realizarse en el tipo de celda y en cuanto a la dimensión deseada. Con los volúmenes proporcionados, la concentración final de BMM es del 2,5%. Si se necesita una concentración diferente, la solución madre debe prepararse a una concentración más baja o más alta. - Centrifugar la placa de 96 pocillos durante 5 minutos a 300 x g y 4 °C.
NOTA: Sin este paso, las células no se agregan correctamente en el fondo del pozo, lo que hace que se formen múltiples agregados más pequeños. La centrífuga debe enfriarse para evitar la reticulación en esta etapa. - Coloque la placa en la incubadora de cultivos celulares. Los agregados se consideran maduros el día 4 después de la siembra.
NOTA: Para obtener protocolos adicionales sobre la formación de esferoides, consulte el Archivo Suplementario 1.
- Micromoldes de placas de Petri 3D
2. Microscopía en vivo de esferoides
- Preparación de esferoides para el análisis de imágenes en vivo
NOTA: Dependiendo del diseño del experimento (p. ej., monitoreo a largo plazo o análisis de punto final, configuración del microscopio o propiedades espectrales de la fluorescencia medida) o debido a la incompatibilidad del método de producción de esferoides con la microscopía (p. ej., espesor de la muestra, autofluorescencia del material, flotación de esferoides durante la obtención de imágenes) monitoreo directo de esferoides en la placa, donde se produjeron, puede que no sea posible. El protocolo explica la preparación de esferoides para la obtención de imágenes, que son adecuados para la mayoría de los microscopios confocales y de campo amplio invertidos.- Preparar y precalentar (37 °C) los medios de imagen: DMEM suplementado con HEPES-Na, pH 7,2 (10 mM), piruvato de sodio (1 mM), L-glutamina (2 mM) y glucosa (5 mM), sin rojo de fenol.
NOTA: Se puede utilizar bicarbonato de sodio solo o en combinación con HEPES-Na, si se proporciona control deCO2 durante la obtención de imágenes24. Algunos tipos de cultivos celulares no pueden tolerar la presencia de HEPES. Dependiendo del diseño experimental, se puede modificar el contenido de piruvato, glutamina y glucosa. - Prepare placas de microscopía estériles (disponibles en el mercado o fabricadas en laboratorio) con superficies de vidrio de recubrimiento recubiertas (para una fuerte adhesión de esferoides) o no recubiertas (baja adherencia de esferoides) (espesor # 1.5).
NOTA: La necesidad y el tipo de recubrimiento dependen del tipo de célula, las propiedades de adhesión de los esferoides y la tasa de migración celular de la interfaz de cultivo 3D a 2D. Es importante tener esto en cuenta, ya que el recubrimiento puede facilitar la pérdida de organización 3D, cambiando la forma de los microgradientes en los esferoides y, como resultado, el comportamiento de las células. Para algunos experimentos (por ejemplo, las imágenes analizaron la respuesta a la estimulación del fármaco), se requiere una fuerte adherencia del esferoide a la superficie, y se prefiere un recubrimiento con gelatina, BMM, colágeno, colágeno / poli-D-lisina o poli-D-lisina. - Lave suavemente los esferoides teñidos con sonda de O2 de los micropocillos de la placa de agarosa microestampada o de 96 pocillos y transfiera los esferoides aún flotantes a un vial de 2 ml.
NOTA: Para asegurar la recolección de todos los esferoides del método de alto rendimiento, enjuague el molde de 1 a 3 veces con el volumen adicional de medios de cultivo, combinando todas las suspensiones de esferoides en un vial. En el caso de los esferoides en una placa de fijación baja, recoja los esferoides uno por uno de los pocillos individuales en un vial o directamente en una placa de microscopía si un pequeño número de esferoides es suficiente para el experimento. Al transferir esferoides grandes, corte el extremo de la punta de la pipeta para asegurarse de que no se dañe durante el pipeteo. - Deje el vial en posición vertical durante un máximo de 5 minutos para que los esferoides se asienten en el fondo del vial, formando una bolita visible.
- Retire el medio del tubo, dejando los esferoides intactos, y vuelva a suspenderlos suavemente en una cantidad suficiente de cultivo fresco.
NOTA: Para facilitar la manipulación de esferoides, la transferencia de esferoides a los medios de imagen también se puede realizar en esta etapa en pequeños lotes; Véanse los pasos 2.1.7 y 2.1.8. Los esferoides de los diferentes grupos experimentales deben ser tratados por igual, ya que la composición del medio y el tiempo de preacondicionamiento del medio pueden afectar su metabolismo. - Mientras los esferoides están flotando, transfiera un volumen igual de suspensión de esferoides por pocillo de la placa de microscopía.
- Incubar los esferoides durante 1-2 h a 37 °C en una incubadora de CO2 para asegurar su fijación a la superficie de la placa/pocillo de microscopía. Para la obtención de imágenes, continúe con el paso 2.1.9. Para la tinción de esferoides con sondas adicionales, proceda con el paso 2.1.8.
NOTA: La tasa de migración de células del esferoide 3D a la interfaz de superficie 2D es una función del tiempo. Para evitar la pérdida de organización 3D, el tiempo de incubación debe optimizarse con respecto al tipo de célula, el tipo de recubrimiento de la superficie y el diseño del experimento. Por ejemplo, HCT116, dependiendo del tamaño del esferoide, necesita al menos 2 h para una unión adecuada del esferoide a la superficie recubierta de colágeno IV/poli-D-lisina, mientras que la fijación y migración de hDPSC a la interfaz 2D es extremadamente rápida, lo que lleva a la pérdida de la organización 3D en 1-2 h. Para evitar la obtención de imágenes de "esferoides 2D" debido a una dispersión excesiva, se utilizan superficies de vidrio sin recubrimiento con un tiempo de incubación reducido. - Añada sondas de fluorescencia en las concentraciones recomendadas o empíricamente optimizadas a un volumen conocido de suspensión esferoide. Incubar durante 1 h a 37 °C CO2 incubadora antes de la toma de imágenes.
NOTA: Para el ensayo vivo/muerto, utilice yoduro de propidio y Calceína Green-AM en una concentración final estándar de 1 μg/mL. Para evitar el efecto tóxico de la tinción de yoduro de propidio en los cuerpos embrioides de iPSC, la concentración final de yoduro de propidio fue de 0,5 μg/mL. El tiempo de carga de la sonda puede prolongarse si la difusión de la sonda no es eficiente debido al gran tamaño del esferoide. El tiempo de carga siempre debe considerarse como parte del tiempo total de incubación necesario para la fijación del esferoide a la superficie. Si se necesita un tiempo más largo para la fijación de esferoides, el procedimiento de tinción debe organizarse al final de este período. Tenga en cuenta que un tiempo de incubación más prolongado puede conducir a la pérdida de la organización 3D. - Retire los medios de cultivo celular o los medios que contengan sondas fluorescentes y cámbielos por un volumen necesario de medios de imagen. Para asegurarse de que no haya ningún fondo de fluorescencia del medio en el camino de la imagen esferoide, repita el paso de intercambio de medios (lavado) hasta 5 veces.
NOTA: Para evitar la extracción de esferoides durante el intercambio de medios, se recomienda aspirar cuidadosamente los medios con una pipeta de 200 μL desde los bordes de las placas de microscopía y realizar la adición de medios por la pared o lateralmente en la placa de microscopía. - Proceda inmediatamente con el paso 2.2.1 del protocolo de imágenes.
NOTA: Un intervalo demasiado largo entre la preparación de la imagen y la adquisición real de la imagen puede influir en el metabolismo celular (por ejemplo, a través de cambios en la composición del medio), la viabilidad (algunas sondas fluorescentes utilizadas para el análisis de puntos finales tienen efectos tóxicos, que pueden estimular la muerte celular después de un largo período de incubación), así como provocar una pérdida de organización 3D. Si se deben comparar múltiples grupos de esferoides o condiciones experimentales, el diseño experimental debe desarrollarse en consecuencia para mantener el tiempo del tratamiento, el preacondicionamiento y los procedimientos de imagen lo más iguales posible entre los grupos analizados.
- Preparar y precalentar (37 °C) los medios de imagen: DMEM suplementado con HEPES-Na, pH 7,2 (10 mM), piruvato de sodio (1 mM), L-glutamina (2 mM) y glucosa (5 mM), sin rojo de fenol.
- Adquisición de imágenes
NOTA: El protocolo describe la obtención de imágenes multiparamétricas de esferoides vivos utilizando el microscopio confocal Stellaris 8 Falcon (Leica) y el software Leica Application Suite X (LAS X) versión 4.7. Sin embargo, solo se requerirán modificaciones menores para realizar dicho análisis en plataformas de microscopía alternativas.- Encienda la unidad de control de clima de temperatura 30-60 minutos antes de la toma de imágenes. Ajuste la velocidad de ventilación y la temperatura necesarias (35-37 °C).
NOTA: Si además del control de la temperatura, la concentración de gas (por ejemplo, CO2 uO2) debe controlarse durante la toma de imágenes, los dispositivos correspondientes también deben ponerse en marcha con anticipación para alcanzar las condiciones necesarias antes de la toma de imágenes. - Encienda el microscopio y los dispositivos conectados (es decir, el láser WLL, la computadora, la bomba de agua para el objetivo de inmersión en agua y otros bloques electrónicos operativos). Inicie el software de control del microscopio (por ejemplo, LAS X Machine Mode o Machine Mode with Environmental Control) suministrado con la configuración exacta del microscopio e inicialice la calibración de la platina.
- Elija el objetivo requerido en el software y aplique el fluido de inmersión si es necesario.
NOTA: Para la microscopía en vivo, se recomienda utilizar objetivos de inmersión en agua o glicerol que tengan una distancia de trabajo suficiente ('larga'), por ejemplo, HC Fluotar L 25x/0,95 W VIS IR (distancia de trabajo de 2,4 mm), HC PL Apo 40x/1,25 GLYC (distancia de trabajo de 0,35 mm) o al menos NA = 0,4 o superior para los objetivos de aire. La elección del aumento y la distancia de trabajo depende de la naturaleza y el tamaño de la muestra fotografiada y de las señales de fluorescencia medidas (brillo, rendimiento cuántico, eficiencia de tinción, véase, por ejemplo, la discusión sobre colorantes y nanopartículas53). Los objetos grandes (esferoides u organoides, de >500 μm de tamaño), los "biorreactores" o los chips microfluídicos requieren objetivos de larga distancia de trabajo y un aumento más bajo, mientras que el análisis de células individuales u orgánulos celulares requiere un gran aumento, a menudo logrado a través de imágenes de "mosaico". - Coloque la placa de microscopía con esferoides en el escenario. Ajuste el enfoque y encuentre un objeto/región de interés (ROI).
NOTA: Si se deben encontrar objetos pequeños, débilmente fluorescentes, de poco contraste o raros y es difícil encontrar el enfoque, se recomienda enfocar previamente las paredes de la placa de microscopía y "examinar" la superficie en busca del objeto de interés mediante serpentina, comenzando desde una de las esquinas del pocillo. - Elija la ventana Abrir proyecto y haga clic en el icono correspondiente, Crear un nuevo proyecto. Asigne un nombre estándar (por ejemplo, a partir de "YY-MM-DD+ descripción") al archivo del proyecto de investigación. Durante la toma de imágenes, todas las imágenes producidas se guardarán automáticamente en el archivo .lif del proyecto creado.
- Abra la ventana Adquisición . Establezca la longitud de onda de excitación del láser de luz blanca (WLL) y el rango requerido de detectores de escaneo híbridos o de resonancia (tipo HyD S, HyD X o HyD R) en función de las propiedades espectrales conocidas de la fluorescencia medida (espectros de excitación/absorbancia y emisión). Elija los tipos de escaneo Línea o Marco .
NOTA: Para la mayoría de los tintes fluorescentes disponibles comercialmente, las propiedades espectrales se pueden encontrar (o agregar) en el paquete LAS X Dye Assistant. Elija el detector con el rango de sensibilidad de los espectros adecuado compatible con las propiedades espectrales de la sonda y, en el caso de FLIM, compatible con el recuento de fotones (es decir, HyD X o HyD R). Para la adquisición de imágenes multiparamétricas, ajuste el WLL en múltiples posiciones de excitación (por ejemplo, para la obtención simultánea de imágenes de FAD/Flavins y dos canales de fluorescencia de la sonda radiométrica MMIR1 O2 - de referencia y sensible, los ajustes de excitación/emisión pueden ser 460 nm/510-590 nm HyD X1 y 614 nm/631-690 HyD X3 y 724-800 nm HyD R en consecuencia en una o dos secuencias de exploración secuenciales). Es importante asignar el detector adecuado para recoger la emisión, ya que los detectores pueden tener diferente sensibilidad espectral54. - (Opcional para FLIM) En la ventana Adquisición , elija el modo FLIM para realizar imágenes combinadas con el recuento de fotones (recopilación de desintegraciones). Inmediatamente, se abrirá un 'módulo FLIM adicional en el software LAS X' para navegar y analizar los datos de FLIM.
- (Opcional para FLIM) Elija la tasa de repetición de pulsos WLL en función de la vida útil media esperada del fluoróforo.
NOTA: La frecuencia del pulso láser debe ajustarse para recoger la desintegración de fluorescencia completa. Esto se puede hacer utilizando una función de selección de pulso instalada en el microscopio. La superposición de la decaimiento de la fluorescencia con el pulso láser conducirá al acortamiento de la vida útil estimada de la fluorescencia. Se recomienda tener intervalos de pulso de 4 a 5 veces más largos que el tiempo de vida promedio esperado de fluorescencia (por ejemplo, 25 ns/40 MHz para períodos de vida útil de hasta 5 ns). Muchos láseres pulsados tienen una tasa de repetición fija de 80 MHz (ideal solo para un rango de hasta 2-3 ns). Esto es importante para elegir los fluoróforos correctos para el experimento. - Inicie la vista previa de la imagen con el modo FAST LIVE y ajuste el enfoque preciso del objeto de imagen en una sección de interés.
PRECAUCIÓN: Siga estrictamente las reglas de seguridad del láser. Tenga siempre en cuenta las reglas de seguridad del láser y espere hasta que la imagen se haya detenido antes de encender la luz de transmisión, mirar dentro del ocular o la muestra.
NOTA: En el modo FAST LIVE , se aplica automáticamente a la imagen una resolución de alta velocidad de 600 Hz (que corresponde a una velocidad de fotogramas máxima de 4,43/s si se utiliza el modo de exploración X bidireccional), una resolución de 256 x 256 píxeles para mantener la fluorescencia a salvo del fotoblanqueo. Abra el orificio (por ejemplo, a 3-4 UA) y/o aumente la intensidad del láser si la señal de fluorescencia es demasiado débil para enfocar el objeto. Evite la recolección incompleta de descomposición. - (Opcional para FLIM) Al observar un histograma de intensidad de píxeles que aparece durante la obtención de imágenes en una ventana del módulo FLIM (modo Live ), ajuste la intensidad del láser / tamaño y la resolución adecuados para lograr el límite de velocidad de conteo de ~ 1 fotón / pulso láser (línea roja). Evite ir significativamente más alto que 1 para excluir el riesgo de efecto de acumulación. Si es necesario, ajuste la tasa de repetición de pulsos WLL para tener una colección completa de decaimiento en una ventana de decaimiento (para evitar una recolección de decaimiento incompleta, consulte el paso 2.2.8).
NOTA: Si el número de fotones (intensidad) no es suficiente para reconstruir un decaimiento confiable para el análisis de ajuste o la nube de diagrama de fasores, aplique varias repeticiones de escaneo (fotogramas o líneas, o configure el tiempo de escaneo), aumente la intensidad del láser y / o sacrifique la resolución (tamaño de ROI escaneado). Tenga en cuenta que la disminución de la tasa de repetición del láser requiere que se recojan más fotones para una reconstrucción confiable de la desintegración, y es posible que se requiera una corrección adicional de los parámetros de la imagen. Ser consciente del impacto potencial de la luz intensa y la iluminación continua durante mucho tiempo en la viabilidad y el metabolismocelular 55. El impacto negativo sobre la viabilidad y el metabolismo puede ser diferente en cada caso individual, dependiendo de la intensidad, la duración y la longitud de onda de la luz de excitación, así como de la modalidad de imagen (por ejemplo, imágenes confocales de un fotón frente a imágenes multifotónicas). Ajustar los parámetros de imagen en consecuencia y, si es necesario, controlar la viabilidad/muerte celular mediante la intensidad de Calcein Green-AM o yoduro de propidio en experimentos piloto56. Siempre que sea posible, se debe considerar la posibilidad de optimizar aún más el protocolo de tinción de la sonda de fluorescencia para alcanzar una señal de fluorescencia adecuada durante la microscopía en vivo. - (Opcional para 3D z-stack) Mientras está en Fast Live , establezca las coordenadas, la dirección de escaneo y atribuya su nombre a Comenzar y Terminar en la ventana de la pila Z (modo de escaneo XYZ). Elija el tamaño del paso Z o el número de pasos.
NOTA: Si bien el software calcula automáticamente el número "óptimo" de pasos, en función de la resolución utilizada y los parámetros de escaneo, la reconstrucción 3D en vivo normalmente puede requerir un número menor de pasos para lograr una adquisición rápida, por ejemplo, un tamaño de paso de 1-2 μm, un tamaño de pila de 50-100 μm, escaneo bidireccional, que requiere 2-3 minutos del tiempo total de escaneo. Tenga en cuenta que los orgánulos subcelulares, las células y el modelo celular 3D también pueden moverse durante las mediciones. Además, debido a la profundidad de penetración de la luz y los límites de dispersión, normalmente solo se puede lograr una profundidad de escaneo de 50-100 μm en FLIM confocal. - Cuando se hayan aplicado todos los ajustes necesarios, inicie la toma de imágenes.
- Asigne a la imagen un nombre apropiado.
- Busque el siguiente objeto de imagen en el modo de luz de transmisión y repita el procedimiento de imagen con los ajustes de imagen previamente optimizados (pasos 2.2.8-2.2.12).
NOTA: Para la comparación basada en la intensidad o el análisis de la relación de intensidad (por ejemplo, el análisis de oxigenación basado en la sonda MMIR O2), mantenga siempre la misma configuración de imagen para todos los objetos analizados (aumento y tipo de lente objetivo, intensidad del láser, potencia y frecuencia de pulso, longitud de onda de excitación, rango de los detectores, estenopeico, velocidad de escaneo, tiempo de permanencia de píxeles y resolución). Sin embargo, dado que la vida útil de la fluorescencia no depende de la intensidad de la fluorescencia y requiere que se recoja un número adecuado de fotones para un cálculo fiable, los parámetros de imagen FLIM se pueden reajustar a lo largo del experimento para mantener los números de fotones recogidos comparables entre diferentes tratamientos o condiciones experimentales. Por lo tanto, para el análisis multiparamétrico en el que se necesita un análisis basado en la intensidad y en el tiempo útil de la fluorescencia, se deben aplicar configuraciones de imagen universales optimizadas para todos los objetos de los grupos experimentales comparados. Para la comparación "solo FLIM", es posible comparar imágenes adquiridas con configuraciones de imagen ligeramente diferentes, ya que el software LAS X proporciona el cálculo de la IRF para la medición de imágenes individuales42. Sin embargo, para el análisis de ajuste de FLIM fuera de LAS X (por ejemplo, FLIMfit45), la función de respuesta del instrumento (IRF) debe medirse para cada condición de imagen diferente, ya que no se puede exportar desde el software de imagen. Por lo tanto, para simplificar el diseño experimental y la carga de trabajo, se recomienda aplicar la misma configuración de imágenes para todas las imágenes del conjunto de datos. A continuación, se pueden realizar las mediciones IRF correspondientes con el uso de fluoróforos de por vida apagados o de fluorescencia rápida (dentro del rango ps) con las propiedades de emisión del canal espectral medido 57,58,59, por luminiscencia de nanopartículas de oro60 o por señal de generación de segundo armónico para FLIM61 multifotónico. En el software LAS X, los parámetros de imagen previamente optimizados se pueden cargar para un nuevo proyecto haciendo clic con el botón derecho en el archivo de interés y eligiendo Aplicar configuración de imagen. - Una vez finalizada la sesión de imágenes, guarde el proyecto de imágenes. Para finalizar la sesión de imágenes, retire la muestra de la etapa de microscopía y limpie el objetivo del líquido de inmersión (si se usa) de acuerdo con el procedimiento estándar implementado en la instalación de imágenes. Cierre el proyecto y el software. Apague el microscopio, los láseres y todos los dispositivos conectados.
- Continúe con el análisis de datos de imágenes (paso 2.3).
- Encienda la unidad de control de clima de temperatura 30-60 minutos antes de la toma de imágenes. Ajuste la velocidad de ventilación y la temperatura necesarias (35-37 °C).
- Procesamiento de imágenes de fasores NAD(P)H/FAD-FLIM con módulo LAS X FLIM y FIJI
NOTA: El protocolo describe un análisis de la vida útil de la fluorescencia de los esferoides fotografiados para obtener datos en el dominio de la frecuencia en ejemplos de NAD(P)H y FAD/Flavins FLIM de autofluorescencia. La medición de la autofluorescencia de NAD(P)H se convirtió en un estándar de oro para el análisis metabólico, donde los componentes de la vida útil corta y más larga de la autofluorescencia de NAD(P)H se asocian con la glucólisis o la fosforilación oxidativa (OxPhos), respectivamente. Esto se puede analizar mediante el cambio en un diagrama de fasores a lo largo de la trayectoria metabólica hacia los estándares medidos de NAD(P)H libre o NAD(P)H unido a proteínas31,62. Para analizar la trayectoria del cambio metabólico, así como para comparar la posición de las nubes fasoriales (ver la NOTA a continuación paso 2.3.6) en un gráfico entre grupos experimentales, se implementó un análisis simplificado de coordenadas fasoriales, basado en el cálculo del centro geométrico (centroide) de la nube fasorial29. El protocolo descrito demuestra el cálculo de las coordenadas del centroide en FIJI y la medición de la distancia entre dos puntos en un diagrama de fasores utilizando coordenadas (por ejemplo, la distancia desde un centroide de la nube de fasores de autofluorescencia NAD(P)H esferoide hasta un punto teórico de "NAD(P)H libre"). Del mismo modo, se pueden analizar FAD y otras señales de autofluorescencia. Un conjunto de datos 1 con .lif (Se requiere el software LAS X) o .ptu Se proporcionan formatos de archivo para aprender este procedimiento (Legajo Complementario 2, Legajo Complementario 3, Legajo Complementario 4, Legajo Complementario 5, Legajo Complementario 6, Legajo Complementario 7, Legajo Complementario 8, Legajo Complementario 9, y Legajo Complementario 10).- Abra el módulo FLIM en LAS X, seleccione Abrir proyecto y cargue el archivo de imagen de esferoide (.lif) para el análisis NAD(P)H/FAD de autofluorescencia.
NOTA: Debido a posibles errores y pérdida de datos intermedios en el módulo FLIM, utilice una copia del archivo de imagen esferoide original (.lif) para el análisis NAD(P)H/FAD, manteniendo el archivo sin procesar. - Seleccione una sola imagen y navegue hasta la interfaz de análisis FLIM. Haga clic en Fasor para acceder al diagrama de fasores y activar el modo de análisis de fasores. Aplique un filtro (mediana o ondícula) y establezca el umbral para minimizar el ruido y mejorar la visibilidad de los datos para todos los análisis de fasores. Elige los armónicos. Para el análisis del cambio metabólico basado en los datos de NAD(P)H, proceda con los pasos 2.3.3-2.3.5. Para la comparación general de los diagramas de fasores, proceda desde el paso 2.3.6.
NOTA: Aplique la configuración de análisis (tipo de filtro, armónicos, umbral, agrupación y ROI de fasores) por igual a todas las imágenes de un conjunto de datos comparado. - (Opcional para el análisis de NAD(P)H) Elija cualquier imagen relacionada con el conjunto de datos y utilice la opción Cursor de relación de dibujo para dos componentes para ubicar con precisión la posición de 0,45 ns en un círculo universal de un espacio de trazado de fasores estándar. Esta posición se asignará a un tiempo de vida de fluorescencia promedio de una solución homogénea pura de NAD(P)H libre, que normalmente está cerca de la desintegración monoexponencial62.
NOTA: El NADH libre y el NAD(P)H libre tienen propiedades espectrales similares y valores similares de vida útil de fluorescencia en solución acuosa, con dos componentes de vida corta, 0,3 ns y 0,7 ns63. Por lo tanto, para la simplicidad del análisis basado en fasores y debido a una pequeña diferencia entre los componentes de la vida útil, se acepta que su decaimiento de fluorescencia es monoexponencial, lo que permite la asignación de la nube de fasores en un círculo universal. La vida media de referencia de una forma de NAD(P)H libre también puede medirse y trazarse en un espacio fasorial para un análisis similar. La elección del tiempo de vida de referencia se basó en la literatura62; nótese que en otras fuentes, se puede encontrar un valor ligeramente diferente de un NAD(P)H libre en solución (0,4 ns64). - (Opcional para el análisis de NAD(P)H) Exporte el diagrama de fasores con la vida útil de NAD(P)H libre asignada (consulte el paso 2.3.3) haciendo clic con el botón derecho en el gráfico y seleccionando Exportar datos. Exporte el diagrama fasorial como un archivo de formato .tiff a una carpeta de almacenamiento designada.
NOTA: El tamaño de píxel original de la imagen de trazado de fasores exportada desde el módulo LAS X FLIM es siempre de 1024 x 600 píxeles. Si se utiliza otro software para la exportación de datos y el análisis previo, asegúrese de que todas las imágenes de diagramas fasoriales se exporten con el mismo tamaño y resolución. - Para exportar la nube de fasores relacionada con esferoides, utilice la herramienta Dibujar cursor en el módulo LAS X FLIM para elegir el ROI del esferoide en la imagen. Exporte el diagrama de fasores generado como se describe en el paso 2.3.4.
NOTA: Las correspondientes coordenadas g y s (similares a x e y) del espacio fasorial se asignarán a cada píxel del ROI elegido, de acuerdo con su vida útil, transformada en un conjunto de datos en el dominio de la frecuencia64,65. El grupo de píxeles con valores similares de tf (tiempo de vida de fase) y tm (tiempo de vida de modulación) formará un patrón de nubes (nube de fasor) en un gráfico, donde el código de colores (con un rango de azul profundo a rojo) reflejará la abundancia de valores de tiempo de vida. Por la posición de la nube en un círculo universal o en el interior, se pueden distinguir los decaimientos mono o multiexponenciales. Algunos medios de medición exhiben una fuerte (auto)fluorescencia, lo que lleva a la aparición de una nube correspondiente en un diagrama de fasores, que no se puede eliminar simplemente con el umbral de medios de intensidad. Este patrón influirá en el cálculo de las coordenadas del centroide y debe excluirse del gráfico de fasores exportado. Trabajar con el ROI del esferoide permite la exclusión de los píxeles no relacionados del análisis de fasores posteriores. - Repita el procedimiento de exportación del diagrama de fasores para todos los ROI de esferoides en un conjunto de datos (consulte los pasos 2.3.4 y 2.3.6). Además, compruebe el conjunto de imágenes .tiff exportadas para garantizar el conjunto completo de datos para un análisis comparativo posterior y asegúrese de que todas las imágenes exportadas tengan el mismo tamaño de píxel (consulte la NOTA en el paso 2.3.4).
NOTA: En esta etapa del protocolo, el conjunto de imágenes debe incluir fasores con la ubicación de NAD(P)H libre (basada en la literatura o en datos obtenidos empíricamente) y todos los diagramas de fasores de ROI esferoidles (o patrones de ROI alternativos si es necesario para el análisis específico). A partir de este paso, se realizarán análisis adicionales en FIJI y luego en una hoja de cálculo. Usando la ventana de la herramienta Analizar , establezca la opción Escala en FIJI y asegúrese de que todas las imágenes de diagrama de fasores estén calibradas con el mismo tipo de unidad, por ejemplo, solo en píxeles. De lo contrario, establezca la longitud de la unidad en la ventana Establecer escala para la elegida (por ejemplo, para la escala basada en píxeles, coloque 1 en el campo Distancia en píxeles y establezca la unidad de longitud en Píxel). Para una mayor comparación, mida todos los datos exportados utilizando el mismo tipo de unidad. - (Opcional para el análisis de NAD(P)H) Determine la posición del punto de píxel de una vida útil de fluorescencia promedio de NAD(P)H libre en la imagen de diagrama de fasor exportada correspondiente (consulte el paso 2.3.3): Abra la imagen del fasor con FIJI, amplíe la imagen para visualizar claramente con una resolución de píxeles la intersección entre el semicírculo universal y la línea del cursor de relación para dos componentes ; utilice la herramienta ROI de rectángulo para seleccionar la intersección.
NOTA: Asegúrese de que la selección rectangular sea un área pequeña alrededor del punto de intersección para determinar con precisión sus coordenadas en el siguiente paso (paso 2.3.9). - (Opcional para el análisis de NAD(P)H) Abra la herramienta Analizar , elija la ventana Establecer mediciones y seleccione Centroide como parámetro de medición. Haga clic en Medir en la ventana de la herramienta Analizar para determinar las coordenadas del centroide del punto de referencia de NAD(P)H libre. Exporta estas coordenadas a una hoja de cálculo.
NOTA: Las coordenadas Free-NAD(P)H se utilizarán como punto de referencia para comparar las distancias desde este punto hasta la posición de la nube fasorial esferoide en un conjunto de datos (la forma de caracterizar el cambio metabólico entre la glucólisis y OxPhos en el análisis de autofluorescencia NAD(P)H FLIM) - Con FIJI, abra la imagen de la nube de fasores esferoides. Abra la herramienta Ventana de imagen , elija Ajustar y seleccione Umbral de color en la barra de herramientas. Seleccione el Método de umbral de elección (por ejemplo, Otsu) y establezca el Valor de tono y el Valor de brillo para reducir los parámetros para la selección de una parte de la nube fasorial con las coordenadas de píxeles más abundantes. Haga clic en Seleccionar para definir el área del clúster.
NOTA: Mantenga los mismos parámetros de umbral para todas las imágenes de diagrama fasorial, que deben analizarse. Para los datos NAD(P)H-FLIM presentados, se eligió el método de umbral Otsu con un valor de tono establecido de 9 y un valor de brillo de 160 y se aplicó a todas las imágenes de fasores. Alternativamente, el área seleccionada se puede copiar en el Administrador de ROI (siga la ruta Editar > Selección > Agregar al Administrador) para crear una biblioteca de ROI de fasor para un análisis más detallado. - Mientras mantiene la selección, calcule las coordenadas del centroide del área seleccionada siguiendo el procedimiento descrito en el paso 2.3.9. Exporte estas coordenadas al archivo de hoja de cálculo.
- Repita los pasos 2.3.9 y 2.3.10 para determinar las coordenadas del centroide de todas las imágenes de fasor de ROI para crear un conjunto de datos en la hoja de cálculo.
NOTA: El uso de la biblioteca ROI Manager ayuda a simplificar y organizar el análisis del ROI (consulte el paso 2.3.10) - (Opcional para el análisis de NAD(P)H) Abra la hoja de cálculo con las coordenadas de referencia exportadas de NAD(P)H libre y ROI esferoides de diferentes grupos de comparación. Calcule la distancia entre cada centroide de fasor esferoide individual para referenciar la posición libre de NAD(P)H utilizando coordenadas determinadas y la siguiente ecuación:
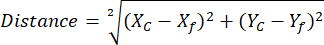
Donde, Xc e Yc son coordenadas de centroide, Xf e Yf son las coordenadas de referencia.
NOTA: La aplicación del parámetro centroide para determinar el cambio hacia el tiempo útil de referencia es apropiada solo en caso de que todos los centroides de un conjunto de datos se encuentren en la misma trayectoria lineal hacia el punto de referencia. Para comprobar esto, todos los puntos centroides del conjunto de datos deben trazarse junto con el punto de referencia en el mismo espacio de coordenadas, y se debe realizar la alineación de la tendencia lineal. Si el coeficiente R2 de la línea de tendencia lineal trazada a través de todos los puntos es cercano a 1 (por ejemplo, R2 es 0,8-0,99), se supone que el análisis de distancia es apropiado. - Organice todos los datos en consecuencia para la comparación y realice análisis estadísticos con el uso de cualquier software correspondiente (por ejemplo, Origin, MatLab). Elija la prueba estadística adecuada de acuerdo con las características del conjunto de datos (normalidad de distribución, número de unidades estadísticas, etc.).
NOTA: Para el análisis de NAD(P)H, compare los valores de distancia para caracterizar el cambio metabólico en función de las condiciones experimentales. Para la comparación de cualquier diagrama de fasor entre grupos experimentales, realice la comparación de las coordenadas de los centroides de la nube de fasores ROI.
- Abra el módulo FLIM en LAS X, seleccione Abrir proyecto y cargue el archivo de imagen de esferoide (.lif) para el análisis NAD(P)H/FAD de autofluorescencia.
Access restricted. Please log in or start a trial to view this content.
Resultados
Elección del método de formación de esferoides adecuado
El método de formación de esferoides seleccionado puede influir en gran medida en el tamaño, la forma, la densidad celular, la viabilidad y la sensibilidad al fármaco de los esferoides (Figura 2). Anteriormente, se compararon los efectos de múltiples métodos de alto rendimiento (SphericalPlate 5D, micromoldes hechos en laboratorio y moldes MicroTissue) y los métodos de baja adherencia de "rendimiento medio"...
Access restricted. Please log in or start a trial to view this content.
Discusión
Los esferoides multicelulares se están convirtiendo en un método de elección en los estudios de microentornos de nicho tumorales y de células madre, el descubrimiento de fármacos y el desarrollo de los "bloques de construcción de tejidos" para la biofabricación. La arquitectura interna heterogénea de los esferoides, los gradientes de nutrientes y la oxigenación pueden imitar los de los tejidos y tumores in vivo en un entorno relativamente simplificado y accesible. Con la necesidad de una mayor transpare...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Nada que revelar.
Agradecimientos
Este trabajo fue financiado por las becas del Fondo Especial de Investigación (BOF) de la Universidad de Gante (BOF/STA/202009/003; BOF/IOP/2022/058), Fundación de Investigación de Flandes (FWO, I001922N) y la Unión Europea, fliMAGIN3D-DN Horizon Europe-MSCA-DN Nº 101073507.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA | Gibco | 25300-054 | Also available from Sigma |
| 10 mL serological pipets | VWR | 612-3700 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 12 well cell-culture plates, sterile | Greiner bio-one | 665-180 | Similar products are also available from Sarstedt, Corning and other companies. |
| 12 Well Chamber slide, removable | Ibidi | 81201 | Also available from Grace Bio-Labs, ThermoFisher Scientific and others |
| 15 mL centrifuge tubes | Nerbe plus | 02-502-3001 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 3D Petri Dish micromolds | Microtissue | Z764000-6EA | |
| 6 well cell-culture plates, sterile | Greiner bio-one | 657160 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 70% ethanol | ChemLab | CL02.0537.5000 | |
| Biofloat | Sarstedt | 83.3925.400 | Commercial available coated 96-well plate for spheroid formation |
| Calcein Green-AM | Tebubio | AS-89201 | Apply in dilution 1:1000 |
| CellSens Dimension software | Olympus | version 3 | |
| Collagen from human placenta, type IV | Sigma | C5533 | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Confocal FLIM Microscope | Leica Microsystems | N/A | Stellaris 8 Falcon inverted microscope with white-light laser, HyD X detectors, climate / T control chamber (OkoLab), 25x/0.95 W objective |
| D(+)-Glucose | Merck | 8342 | Prepare 1 M stock solution, 1:100 for preparation of imaging medium (final concentration 10 mM) |
| Dulbecco's modified Eagle's medium (DMEM), phenol red-, glucose-, pyruvate- and glutamine-free | Sigma-Aldrich | D5030-10X1L | For preparation of imaging medium |
| Fetal Bovine Serum (FBS) | Gibco | 10270-098 | Also available from Sigma. Needs to be heat-inactivated before use. |
| HEPES (1M) | Gibco | 15630-080 | Dilution 1/100 for preparation of imaging medium (final concentration 10 mM) |
| Human colon cancer cells HCT116 | ATCC | ||
| ImageJ | NIH | version 1.54f | |
| Leica Application Suite X (LAS X) | Leica Microsystems | version 4.6.1.27508 | |
| L-glutamine | Gibco | 25030 | Also available from Sigma. Apply in dilution 1:100. |
| Lipidure-CM5206 | Amsbio | AMS.52000034GB1G | |
| McCoy's 5A, need addition of 1 mM Sodium Pyruvate and 10 mM HEPES | VWR | 392-0420 | Standard growth medium for HCT116 cells |
| micro-patterned 3D-printed PDMS stamps | N/A | N/A | Provided by the Centre for Microsystems Technology, Professor Dr. Jan Vanfleteren, Ghent University |
| NaCl | Chemlab | CL00.1429.100 | |
| Neubauer couting chamber | Fisher Scientific | 15980396 | |
| O2 probes: MMIR1 | N/A | N/A | Full characterization, validation and some applications can be found at: https://www.biorxiv.org/content/10.1101/2023.12.11.571110 v1 |
| PBS | Fisher scientific | Gibco18912014 | Dissolve PBS tablet in 500 mL of distilled water. |
| Pen Strep :Penicillin (10,000 U/mL) / streptomycin (10,000 μg/mL) 100x solution | Gibco | 15140-122 | Also available from Sigma. Apply in dilution 1:100. |
| Poly-D-lysine | Sigma | P6407-5mg | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Propidium Iodide | Sigma-Aldrich | 25535-16-4 | Cell death staining, use 1 µg/mL at 1h incubation |
| PVDF syringe filter 0.22 µm | Novolab | A35149 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Sodium pyruvate (100 mM) | Gibco | 11360-070 | Dilution 1/100 for preparation of imaging medium (final concentration 1mM) |
| SphericalPlate 5D 24-well | Kugelmeiers | SP5D-24W | |
| sterile petridish | Greiner bio-one | 633181 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (25 cm² ) | VWR | 734-2311 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (75 cm²) | VWR | 734-2313 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| U-bottom 96-well plate | VWR | 10062-900 | Similar products are also available from Sarstedt, Corning, Greiner Bio-one and other companies |
| Ultrapure Agarose | Invitrogen (Life Technologies) | 16500-500 | Other types of Agarose such as Agarose low melting point (A-9414, Sigma), Agarose for routine use (A-9539, Sigma) |
| Widefield fluorescence inverted microscope | Olympus | N/A | Inverted fluorescence microscope IX81, with motorised Z-axis control, CoolLED pE4000 (16 channels, 365-770 nm), ORCA-Flash4.0LT (Hamamatsu) cMOS camera, glass warming plate Okolab, CellSens Dimension v.3 software and air objectives 4x/0.13 UPlanFLN and 40x/0.6 LUCPlanFLN. (Optional, for high-resolution imaging) 60x/1.0 LUMPLFLN water |
Referencias
- Foty, R. A simple hanging drop cell culture protocol for generation of 3d spheroids. J Vis Exp. (51), e2720(2011).
- Moskovits, N., et al. Establishing 3-dimensional spheroids from patient-derived tumor samples and evaluating their sensitivity to drugs. J Vis Exp. (190), e64564(2022).
- Griner, L. M., et al. Generation of high-throughput three-dimensional tumor spheroids for drug screening. J Vis Exp. (139), e57476(2018).
- Qian, Y., Wei, X., Chen, K., Xu, M. Three-dimensional acoustic assembly device for mass manufacturing of cell spheroids. J Vis Exp. (200), e66078(2023).
- He, H., et al. Dynamic formation of cellular aggregates of chondrocytes and mesenchymal stem cells in spinner flask. Cell Prolif. 52 (4), e12587(2019).
- Perez, J. E., Nagle, I., Wilhelm, C. Magnetic molding of tumor spheroids: Emerging model for cancer screening. Biofabrication. 13 (1), 015018(2020).
- Kingsley, D. M., et al. Laser-based 3d bioprinting for spatial and size control of tumor spheroids and embryoid bodies. Acta Biomater. 95, 357-370 (2019).
- Guillaume, O., et al. Hybrid spheroid microscaffolds as modular tissue units to build macro-tissue assemblies for tissue engineering. Acta Biomater. 165, 72-85 (2023).
- Danilevicius, P., et al. laser-made 3D microscaffolds for tissue spheroid encagement. Biointerphases. 10 (2), 021011(2015).
- Jamieson, L. E., Harrison, D. J., Campbell, C. Chemical analysis of multicellular tumour spheroids. Analyst. 140 (12), 3910-3920 (2015).
- Dmitriev, R., Borisov, S., Jenkins, J., Papkovsky, D. Multiparametric imaging of tumor spheroids with ultra-bright and tunable nanoparticle O2 probes. SPIE BiOS. 9328, (2015).
- Zushin, P. -J. H., Mukherjee, S., Wu, J. C. FDA Modernization Act 2.0: transitioning beyond animal models with human cells, organoids, and AI/ML-based approaches. J Clin Invest. 133 (21), e175824(2023).
- Kim, D. -S., et al. Robust enhancement of neural differentiation from human es and ips cells regardless of their innate difference in differentiation propensity. Stem Cell Rev Rep. 6 (2), 270-281 (2010).
- Hart, D., Gutiérrez, D. R., Biason-Lauber, A. Generation of a human ovarian granulosa cell model from induced pluripotent stem cells. bioRxiv. , 2022(2022).
- Chiaradia, I., et al. Tissue morphology influences the temporal program of human brain organoid development. Cell Stem Cell. 30 (10), 1351-1367 (2023).
- Wagstaff, E. L., Ten Asbroek, A. L., Ten Brink, J. B., Jansonius, N. M., Bergen, A. A. An alternative approach to produce versatile retinal organoids with accelerated ganglion cell development. Sci Rep. 11 (1), 1101(2021).
- Thavandiran, N., et al. Design and formulation of functional pluripotent stem cell-derived cardiac microtissues. Proc Natl Acad Sci U S A. 110 (49), E4698-E4707 (2013).
- Harrison, S. P., et al. Scalable production of tissue-like vascularized liver organoids from human pscs. Exp Mol Med. 55 (9), 2005-2024 (2023).
- Noguchi, T. -A. K., Kurisaki, A. Formation of stomach tissue by organoid culture using mouse embryonic stem cells. Methods Mol Biol. 2017, 217-228 (2017).
- Takahashi, J., et al. Suspension culture in a rotating bioreactor for efficient generation of human intestinal organoids. Cell Reports Methods. 2 (11), 100337(2022).
- Lagziel, S., Gottlieb, E., Shlomi, T. Mind your media. Nat Metab. 2 (12), 1369-1372 (2020).
- Das, V., Fürst, T., Gurská, S., Džubák, P., Hajdúch, M. Reproducibility of uniform spheroid formation in 384-well plates: The effect of medium evaporation. J Biomol Screen. 21 (9), 923-930 (2016).
- Bera, K., et al. Extracellular fluid viscosity enhances cell migration and cancer dissemination. Nature. 611 (7935), 365-373 (2022).
- Michl, J., Park, K. C., Swietach, P. Evidence-based guidelines for controlling ph in mammalian live-cell culture systems. Commun Biol. 2, 144(2019).
- Okkelman, I. A., Vercruysse, C., Kondrashina, A. V., Borisov, S. M., Dmitriev, R. I. Affordable oxygen microscopy-assisted biofabrication of multicellular spheroids. J Vis Exp. (182), e63403(2022).
- Peirsman, A., et al. Mispheroid: A knowledgebase and transparency tool for minimum information in spheroid identity. Nat Methods. 18 (11), 1294-1303 (2021).
- Raghavan, S., et al. Comparative analysis of tumor spheroid generation techniques for differential in vitro drug toxicity. Oncotarget. 7 (13), 16948(2016).
- Froehlich, K., et al. Generation of multicellular breast cancer tumor spheroids: Comparison of different protocols. J Mammary Gland Biol Neoplasia. 21 (3-4), 89-98 (2016).
- Debruyne, A. C., et al. Live microscopy of multicellular spheroids with the multimodal near-infrared nanoparticles reveals differences in oxygenation gradients. ACS Nano. 18 (19), 12168-12186 (2024).
- Debruyne, A. C., Okkelman, I. A., Dmitriev, R. I. Balance between the cell viability and death in 3D. Semin Cell Dev Biol. 144, 55-66 (2023).
- Barroso, M., Monaghan, M. G., Niesner, R., Dmitriev, R. I. Probing organoid metabolism using fluorescence lifetime imaging microscopy (flim): The next frontier of drug discovery and disease understanding. Adv Drug Deliv Rev. 201, 115081(2023).
- Becker, W. Fluorescence lifetime imaging-techniques and applications. J Microsc. 247 (2), 119-136 (2012).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. J Cell Sci. 134 (9), 1-17 (2021).
- Sarder, P., Maji, D., Achilefu, S. Molecular probes for fluorescence lifetime imaging. Bioconjug Chem. 26 (6), 963-974 (2015).
- Alfonso-Garcia, A., et al. Mesoscopic fluorescence lifetime imaging: Fundamental principles, clinical applications and future directions. J Biophotonics. 14 (6), e202000472(2021).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: Fundamentals and advances in instrumentation, analysis, and applications. J Biomed Opt. 25 (7), 1-43 (2020).
- Yellen, G. Fueling thought: Management of glycolysis and oxidative phosphorylation in neuronal metabolism. J Cell Biol. 217 (7), 2235-2246 (2018).
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomark Med. 4 (2), 241-263 (2010).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of nad (p) h and fad. Antioxid Redox Signal. 30 (6), 875-889 (2019).
- Verma, A., et al. Fluorescence lifetime imaging for quantification of targeted drug delivery in varying tumor microenvironments. bioRxiv. , 2024(2024).
- Smith, J. T., et al. In vivo quantitative fret small animal imaging: Intensity versus lifetime-based fret. Biophys Rep. 3 (2), 100110(2023).
- Alvarez, L. A., et al. Application Note: SP8 Falcon: A novel concept in fluorescence lifetime imaging enabling video-rate confocal flim. Nat Methods. , (2019).
- Roberti, M. J., et al. TauSense: A fluorescence lifetime-based tool set for everyday imaging. Nat. Methods. , https://www.nature.com/articles/d42473-020-00364-w.pdf (2020).
- Auer, J. M. T., Murphy, L. C., Xiao, D., Li, D. U., Wheeler, A. P. Non-fitting flim-fret facilitates analysis of protein interactions in live zebrafish embryos. J Microsc. 291 (1), 43-56 (2023).
- Warren, S. C., et al. Rapid global fitting of large fluorescence lifetime imaging microscopy datasets. PLoS One. 8 (8), e70687(2013).
- Gao, D., et al. Flimj: An open-source imagej toolkit for fluorescence lifetime image data analysis. PloS One. 15 (12), e0238327(2020).
- Tullis, I. D. C., Ameer-Beg, S. M., Barber, P. R., Rankov, V., Vojnovic, B. Mapping femtosecond pulse front distortion and group velocity dispersion in multiphoton microscopy. Proc. SPIE 6089, Multiphoton Microscopy in the Biomedical Sciences VI. , 60890(2006).
- Zhou, Y., et al. One-step derivation of functional mesenchymal stem cells from human pluripotent stem cells. Bio Protoc. 8 (22), e3080(2018).
- Benton, G., Arnaoutova, I., George, J., Kleinman, H. K., Koblinski, J. Matrigel: From discovery and ECM mimicry to assays and models for cancer research. Adv Drug Deliv Rev. 79-80, 3-18 (2014).
- Badea, M. A., et al. Influence of matrigel on single-and multiple-spheroid cultures in breast cancer research. SLAS Discov. 24 (5), 563-578 (2019).
- Lang, S., Sharrard, R., Stark, M., Villette, J., Maitland, N. Prostate epithelial cell lines form spheroids with evidence of glandular differentiation in three-dimensional matrigel cultures. Br J Cancer. 85 (4), 590-599 (2001).
- Barra, J., et al. DMT1-dependent endosome-mitochondria interactions regulate mitochondrial iron translocation and metastatic outgrowth. Oncogene. 43 (9), 650-667 (2024).
- Dmitriev, R. I., Papkovsky, D. B. Intracellular probes for imaging oxygen concentration: How good are they. Methods Appl Fluoresc. 3 (3), 034001(2015).
- Schweikhard, V., et al. Application Note: The power HyD family of detectors for confocal microscopy. Nat Methods. , https://www.nature.com/articles/d42473-020-00398-0 (2020).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon flim microscopy. Sci Rep. 12 (1), 11938(2022).
- Bush, P. G., Wokosin, D. L., Hall, A. C. Two-versus one photon excitation laser scanning microscopy: Critical importance of excitation wavelength. Front Biosci. 12, 2646-2657 (2007).
- Liu, M., et al. Instrument response standard in time-resolved fluorescence spectroscopy at visible wavelength: Quenched fluorescein sodium. Appl Spectrosc. 68 (5), 577-583 (2014).
- Szabelski, M., et al. Collisional quenching of erythrosine b as a potential reference dye for impulse response function evaluation. Appl Spectrosc. 63 (3), 363-368 (2009).
- Chib, R., et al. Standard reference for instrument response function in fluorescence lifetime measurements in visible and near infrared. Meas Sci Technol. 27 (2), 027001(2015).
- Talbot, C. B., et al. Application of ultrafast gold luminescence to measuring the instrument response function for multispectral multiphoton fluorescence lifetime imaging. Opt Express. 19 (15), 13848-13861 (2011).
- Becker, W. Recording the instrument response function of a multiphoton flim system. Becker & Hickl. , Available from: https://www.becker-hickl.com/literature/application-notes/recording-the-instrument-response-function-of-a-multiphoton-flim-system/ (2007).
- Leben, R., Köhler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free nad (p) h fluorescence lifetime imaging. Int J Mol Sci. 20 (22), 5565(2019).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using nadh and nadph autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Gottlieb, D., Asadipour, B., Kostina, P., Ung, T. P. L., Stringari, C. FLUTE: A python gui for interactive phasor analysis of flim data. Biol Imaging. 3, e21(2023).
- Malacrida, L., Ranjit, S., Jameson, D. M., Gratton, E. The phasor plot: A universal circle to advance fluorescence lifetime analysis and interpretation. Annu Rev Biophys. 50, 575-593 (2021).
- Okkelman, I., Vandenberghe, W., Dmitriev, R. Role of preconditioning with oxygen and glucose deprivation in promoting differentiation of dental pulp stem cells in 3D culture. Mol Biol Cell. 34 (2), 1212(2022).
- Park, J. H., et al. The effect of bmp-mimetic peptide tethering bioinks on the differentiation of dental pulp stem cells (DPSCs) in 3D bioprinted dental constructs. Biofabrication. 12 (3), 035029(2020).
- Pașca, S. P. The rise of three-dimensional human brain cultures. Nature. 553 (7689), 437-445 (2018).
- Dmitriev, R. I., Zhdanov, A. V., Nolan, Y. M., Papkovsky, D. B. Imaging of neurosphere oxygenation with phosphorescent probes. Biomaterials. 34 (37), 9307-9317 (2013).
- Bryanskaya, E. O., et al. High levels of FAD autofluorescence indicate pathology preceding cell death. Biochim Biophys Acta Gen Subj. 1868 (1), 130520(2024).
- Han, S. J., Kwon, S., Kim, K. S. Challenges of applying multicellular tumor spheroids in preclinical phase. Cancer Cell Int. 21 (1), 152(2021).
- Okkelman, I. A., Neto, N., Papkovsky, D. B., Monaghan, M. G., Dmitriev, R. I. A deeper understanding of intestinal organoid metabolism revealed by combining fluorescence lifetime imaging microscopy (flim) and extracellular flux analyses. Redox Biol. 30, 101420(2020).
- Gstraunthaler, G., Seppi, T., Pfaller, W. Impact of culture conditions, culture media volumes, and glucose content on metabolic properties of renal epithelial cell cultures: Are renal cells in tissue culture hypoxic. Cell Physiol Biochem. 9 (3), 150-172 (1999).
- Glickman, R. D. Phototoxicity to the retina: Mechanisms of damage. Int J Toxicol. 21 (6), 473-490 (2002).
- Golub, A. S., Pittman, R. N. Monitoring Parameters of Oxygen Transport to Cells in the Microcirculation. Quenched-Phosphorescence Detection of Molecular Oxygen: Applications in Life Sciences. , The Royal Society of Chemistry. (2018).
- Nishigaki, T., Wood, C. D., Shiba, K., Baba, S. A., Darszon, A. Stroboscopic illumination using light-emitting diodes reduces phototoxicity in fluorescence cell imaging. Biotechniques. 41 (2), 191-197 (2006).
- Penjweini, R., Loew, H. G., Hamblin, M. R., Kratky, K. W. Long-term monitoring of live cell proliferation in presence of pvp-hypericin: A new strategy using ms pulses of led and the fluorescent dye cfse. J Microsc. 245 (1), 100-108 (2012).
- Carrasco Kind, M., et al. flimview: A software framework to handle, visualize and analyze flim data. F1000Research. 9, 574(2020).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. Pam: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophys J. 114 (7), 1518-1528 (2018).
- Chen, S. -J., Sinsuebphon, N., Barroso, M., Intes, X., Michalet, X. Optical Molecular Probes, Imaging and Drug Delivery. , Optica Publishing Group. (2021).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados