È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Produzione e microscopia multiparametrica per imaging a fluorescenza su cellule vive (FLIM) di sferoidi multicellulari
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, descriviamo diversi metodi di formazione di sferoidi multicellulari per eseguire la microscopia multiparametrica su cellule vive di follow-up. Utilizzando la microscopia a fluorescenza (FLIM), l'autofluorescenza cellulare, i coloranti di colorazione e le nanoparticelle, viene dimostrato l'approccio per l'analisi del metabolismo cellulare, dell'ipossia e della morte cellulare nel cancro tridimensionale (3D) vivo e negli sferoidi derivati da cellule staminali.
Abstract
Gli sferoidi tumorali multicellulari sono un popolare modello di microaggregato tissutale 3D per la riproduzione del microambiente tumorale, il test e l'ottimizzazione di terapie farmacologiche e l'utilizzo di biosensori e nanosensori in un contesto 3D. La loro facilità di produzione, le dimensioni prevedibili, la crescita e i gradienti di nutrienti e metaboliti osservati sono importanti per ricapitolare il microambiente cellulare di nicchia 3D. Tuttavia, l'eterogeneità degli sferoidi e la variabilità dei loro metodi di produzione possono influenzare il metabolismo cellulare complessivo, la vitalità e la risposta ai farmaci. Ciò rende difficile scegliere la metodologia più appropriata, considerando i requisiti di dimensioni, variabilità, esigenze di biofabbricazione e utilizzo come modelli di tessuto 3D in vitro nella biologia delle cellule staminali e tumorali. In particolare, la produzione di sferoidi può influenzare la loro compatibilità con microscopie quantitative dal vivo, come l'imaging metabolico ottico, la microscopia a fluorescenza (FLIM), il monitoraggio dell'ipossia sferoidale con nanosensori o la vitalità. Qui vengono presentati una serie di protocolli convenzionali di formazione di sferoidi, evidenziando la loro compatibilità con le microscopie a campo largo, confocale e a due fotoni. Viene inoltre presentato l'imaging di follow-up alla pipeline di analisi con FLIM multiplexato in autofluorescenza e, utilizzando vari tipi di cancro e sferoidi di cellule staminali.
Introduzione
Gli sferoidi multicellulari rappresentano un gruppo di modelli tissutali 3D ottenuti dall'autoaggregazione di cellule e che esibiscono una forma sferica. Sono ampiamente utilizzati per imitare l'interazione cellula-cellula e cellula-matrice in vitro e per riprodurre un contesto 3D all'interno di una moltitudine di costrutti derivati da cancro e cellule staminali. Diverse tecniche sono impiegate per ridurre l'attaccamento cellulare e promuovere l'aggregazione. Questi includono il metodo della goccia sospesa che si basa sulla tensione superficiale1; metodi di repulsione dell'attacco cellulare come piastre di attacco ultra-basse, microstampi e micropozzetti 2,3; approccio basato sulle onde acustiche4; metodi di aggregazione indotta dal flusso (palloni rotanti, bioreattori e dispositivi microfluidici)5; formazione assistita da particelle magnetiche6 e utilizzo di matrici e scaffold sintetici e basati su ECM che promuovono l'aggregazione 7,8,9.
Nella ricerca, nello sviluppo e nella validazione di nuove terapie farmacologiche sul cancro, gli sferoidi sono un modello attraente grazie alla loro capacità di ricapitolare i gradienti di nutrienti, prodotti di scarto e O2 limitati dalla diffusione spaziale, portando spesso alla formazione di un nucleo necrotico, tipico dei tumori solidi10,11. Questi modelli in vitro più affidabili e sofisticati sfidano la necessità di un uso estensivo di modelli animali (Food and Drug Administration [FDA] Modernization Act 2.012), secondo il principio delle 3R della ricerca sugli animali (sostituzione, riduzione e perfezionamento). Oltre al cancro, gli sferoidi trovano la loro applicazione nella ricerca sulle cellule staminali. Ad esempio, le cellule staminali pluripotenti hanno la capacità di formare corpi embrioidi (EB), che possono essere utilizzati per la differenziazione delle cellule staminali pluripotenti indotte (iPSC) verso tipi di cellule specializzate che sono difficili da ottenere direttamente dai pazienti, come le cellule precursori neurali13 o le cellule della granulosa ovarica 13,14. Inoltre, la formazione di un EB è spesso il primo passo nello sviluppo di modelli di organoidi più complessi, ad esempio neurale15, retinale16, cardiaco17, fegato18, stomaco19 e organoidi intestinali20. Fattori quali le dimensioni, la riproducibilità, la produttività e le applicazioni a valle dovrebbero essere considerati quando si sceglie un metodo di formazione di sferoidi appropriato per gli esperimenti.
L'aumento della complessità della cultura 3D può portare a una maggiore variabilità rispetto alla cultura 2D. Fattori come la composizione dei nutrienti21, l'evaporazione dei terreni22, la viscosità23, il controllo del pH24, il metodo di formazione degli sferoidi e persino il tempo trascorso nella coltura25,26 possono portare all'ottenimento di sferoidi di diversa morfologia, dimensioni, vitalità e diversa chemioresistenza27,28. Recenti ricerche hanno dimostrato che i gradienti di ossigeno sferoidale non sono sempre statici e sono influenzati dal metodo di formazione, dalle dimensioni degli sferoidi e dalla viscosità extracellulare, influenzando l'eterogeneità degli sferoidi29. Per migliorare la riproducibilità e l'accessibilità dei dati sugli sferoidi, è stata sviluppata la base di conoscenze MISpheroID26, identificando la linea cellulare, il terreno di coltura, il metodo di formazione e la dimensione degli sferoidi come informazioni minime per un risultato riproducibile. Pertanto, è stato effettuato un confronto dettagliato tra più metodi ad alta produttività (SphericalPlate 5D, microstampi realizzati in laboratorio e stampi per microtessuti) e a basso attaccamento (ad esempio, piastre a 96 pozzetti Biofloat e Lipidure-coated sia senza scaffold che basate su scaffold) (Figura 1 e Tabella 1), tra cui la dimensione del pozzetto (data una stima della dimensione massima dello sferoide), i materiali di consumo utilizzati, il tempo di preparazione e la possibilità di monitorare gli sferoidi senza trasportarli in piastre di microscopia. Quest'ultimo consente studi a lungo termine, mentre gli sferoidi prodotti con metodi ad alto rendimento spesso si traducono in esperimenti finali. Tutti i metodi, ad eccezione delle griglie della piastra 5DspheriLast, non apportano un'autofluorescenza indesiderata, consentendone così l'uso diretto in microscopia.
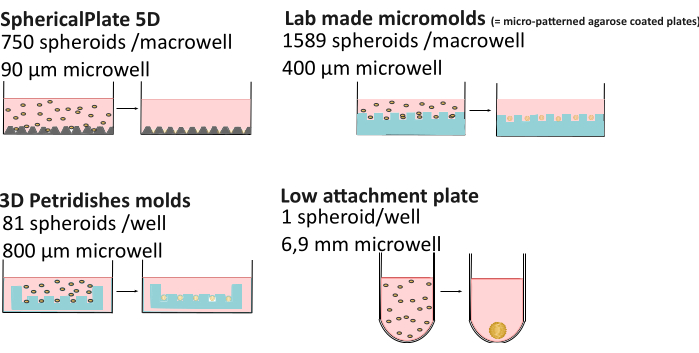
Figura 1: Spiegazione dei metodi di formazione degli sferoidi. Metodi ad alta produttività come la SphericalPlate 5D, che ha integrato micropozzetti brevettati nella piastra, mentre i microstampi prodotti in laboratorio e gli stampi MicroTissue utilizzano timbri per realizzare più micropozzetti in agarosio (blu). Le placche a basso attaccamento come Lipidure (Amsbio) e Biofloat (Sarstedt) utilizzano un rivestimento non aderente che inibisce l'adesione cellula-superficie e promuove l'autoaggregazione cellulare. Clicca qui per visualizzare una versione più grande di questa figura.
| Piastra SpheriPlate 5D | Microstampi autoprodotti | Microtessuto | Metodi di fissaggio bassi | |
| Numero di sferoidi/pozzetto | 750 | 1589 | 81 | 1 |
| Diametro pozzetto | 90 μm | 400 μm | 800 μm | 1 millimetro |
| Volume di coltura | 1 ml | 5 ml | 1 ml | 200 μl |
| Altri materiali di consumo | / | 7 mL di agarosio al 3% | 500 μl di agarosio al 2% | / * |
| Tempo di preparazione | 10 minuti | 2 ore + 3 giorni di adattamento dei media | 0,5 ore + 15 minuti di adattamento multimediale | 10-30 min + 1 ora di asciugatura |
| Monitoraggio | Sì | No** | Sì | Sì |
| Autofluorescente | Sì | No | No | No |
| Riutilizzabile | No | Sì | Sì | No** |
| Costo | €€ | € | €€€€ | €€€€: Rivestimento e Matrigel |
| €€: Piastra commerciale a 96 pozzetti | ||||
| *Alcune linee cellulari richiedono l'aggiunta di ECM (cioè 2%-5% di Matrigel) per formare sferoidi compatti. | ||||
| **Il rivestimento è riutilizzabile fino all'esaurimento. Tuttavia, ogni lastra consumerà una piccola quantità di materiale e la polvere può accumularsi nel tempo. La sterilizzazione del filtro è regolarmente necessaria. | ||||
Tabella 1: Confronto tra più metodi di formazione di sferoidi29. "Monitoraggio": la capacità di monitorare lo sferoide senza la necessità di trasferirlo su una capsula di microscopia. €: 0-50€, €€: 50-150€ , €€€: 150-500€ , €€€€: >500€
La microscopia a fluorescenza consente il monitoraggio diretto degli aspetti biologici chiave all'interno degli sferoidi, tra cui la morte cellulare, la vitalità, la proliferazione, il metabolismo, la viscosità e persino le proprietà meccaniche30. La microscopia a fluorescenza (FLIM) fornisce un'ulteriore dimensione quantitativa per lo studio delle interazioni delle sonde fluorescenti all'interno del loro (micro)ambiente 31,32,33,34, consentendo di risolvere gli spettri di emissione sovrapposti in base a diverse durate di emissione 35,36 e sondare il metabolismo cellulare basato sull'autofluorescenza cellulare intrinseca. Pertanto, composti autofluorescenti cellulari così diffusi come la nicotinammide adenina dinucleotide fosfato (NAD(P)H), la flavina mononucleotide (FMN), la flavina adenina dinucleotide (FAD), la protoporfirina IX e altri possono essere misurati con FLIM a uno e due fotoni e fungere da "sensori" intrinseci del catabolismo del glucosio, della fosforilazione ossidativa (OxPhos) e fornire una panoramica generale dello stato redox cellulare. Il NAD(P)H esiste in forme citoplasmatiche libere o in forme mitocondriali legate a proteine37,38. Allo stesso modo, lo stato ossidato del FAD è fluorescente con una durata più lunga della forma libera. Le microscopie NAD(P)H e FAD di solito utilizzano FLIM eccitate a due fotoni, con l'obiettivo di prevenire il fotodanneggiamento del campione39. Spesso, l'imaging metabolico ottico FLIM può essere combinato con l'uso di sonde basate su coloranti, biosensori geneticamente codificati, microscopia per imaging a tempo di fosforescenza (PLIM) e misurazioni basate sull'intensità raziometrica al fine di fornire un quadro più completo del metabolismo, dell'ossigenazione, della proliferazione e della vitalità cellularedi sferoidi o organoidi 29,30,31 . Inoltre, FLIM può anche essere combinato con il metodo di trasferimento di energia di risonanza di Förster (FRET) per misurare la variazione del tempo di vita del fluoroforo donatore quando è a stretto contatto con l'accettore per studiare il legame di un farmaco con il suo dominio target 33,40,41.
Le immagini FLIM acquisite vengono in genere analizzate per calcolare la durata pixel per pixel. Attualmente, ci sono almeno 3 strategie comuni utilizzate per ottenere il tempo di vita della fluorescenza: "FLIM veloce" semiquantitativo (a volte indicato come "senso tau"43,44), adattamento della curva di decadimento, utilizzando l'adattamento a uno, due o tre esponenziali e approccio "senza adattamento" con trasformazione dei fasori e analisi del diagramma dei fasori. A seconda del fornitore, per gestire i dati FLIM misurati è possibile utilizzare il software fornito (LAS X, Symphotime, SPCImage, ecc.) o open source (ad esempio, FLIMfit45, FLIMJ46 o altri47). In genere, il software fornito dal fornitore è utile per l'analisi preliminare dei dati, mentre le soluzioni open source possono fornire studi più accurati utilizzando, ad esempio, grafici dei fasori e visualizzazione 3D.
Nonostante l'utilità e l'attrattiva di FLIM come metodo per lo studio degli sferoidi, sono disponibili pochissimi protocolli sperimentali e c'è una generale mancanza di conoscenza nella scelta del metodo di formazione più appropriato per il successo degli esperimenti di microscopia multiparametrica dal vivo che coinvolgono FLIM. Qui, viene presentato un confronto dettagliato dei protocolli di formazione di sferoidi comunemente usati in base alla loro morfologia, vitalità e ossigenazione con il nanosensore di rilevamento dell'ossigeno (MMIR1) recentemente convalidato e caratterizzato nel lontano rosso e nel vicino infrarosso (NIR). La nanoparticella cationica è impregnata con due coloranti reporter, l'aza insensibile al riferimento O 2-BODIPY (eccitazione 650 nm, emissione 675 nm) e la metalloporfirina NIR O2-sensibile, PtTPTBPF (eccitazione 620 nm, emissione 760 nm). L'MMIR1 consente l'analisi in tempo reale dei gradienti di ossigeno su un microscopio a fluorescenza convenzionale (utilizzando l'analisi raziometrica) o un microscopio a fosforescenza (PLIM) senza introdurre tossicità cellulare e consentendo segnali stabili, monitoraggio a lungo termine e multiplexing25,29. A seconda della necessità di colorare con coloranti o nanosensori, della produttività degli sferoidi o del tipo di cellula, è possibile scegliere il protocollo di formazione più appropriato. Poiché gli studi sulla vitalità e l'ossigenazione degli sferoidi sono rilevanti per gli studi sul cancro e sugli sferoidi derivati da cellule staminali, i protocolli presentati includono anche esempi e risultati tipici attesi di NAD(P)H-FLIM e FAD-FLIM con questi modelli. Le pipeline di imaging e analisi presentate si rivolgono alle più diffuse piattaforme di microscopia FLIM basate sul conteggio di singoli fotoni correlati nel tempo.
Protocollo
1. Generazione di sferoidi multicellulari
- Coltura cellulare
NOTA: Le colture cellulari possono essere raccolte dall'American Type Culture Collection (ATCC), Lonza, Sigma-Aldrich o altri fornitori. L'ATCC fornisce tutte le informazioni necessarie per la manipolazione, compresi i terreni di crescita preferiti, le procedure di subcoltura, il livello di biosicurezza, il tasso di crescita e i profili STR. Qui, 500 cellule/sferoide della linea cellulare di cancro del colon umano HCT116 viene utilizzato nel terreno 5A di McCoy (VWR, 392-0420) integrato con il 10% di FBS e 1 mM di piruvato di sodio. Per esperimenti a lungo termine monitorati quotidianamente, è possibile aggiungere al terreno 10 mM HEPES, pH 7,2.- Coltiva la coltura cellulare per raggiungere una confluenza del 70%-90%.
- Sciacquare le celle con PBS sterile preriscaldato (37 °C) (5 mL per 25 cm2 o 10 mL per pallone da 75 cm2 ).
- Aggiungere la soluzione di tripsina allo 0,05% - 1 mM EDTA (0,5 mL per 25 cm² o 1 mLper 75 cm2 matraccia) e incubare per 5-10 minuti a 37 °C in 5% di CO2, 95% di umidità per raggiungere il distacco cellulare.
NOTA: Controllare il distacco delle cellule sotto il microscopio a luce di trasmissione (campo chiaro). La sovraesposizione delle cellule alla soluzione enzimatica dissociante può influire sulla loro vitalità. - Neutralizzare la tripsina aggiungendo un eccesso di terreno di coltura cellulare contenente il 10% di FBS (almeno 5 mL di terreno per 1 mL di soluzione di dissociazione).
NOTA: Per le cellule coltivate su terreni di coltura a basso FBS o privi di FBS, la neutralizzazione della tripsina può essere effettuata con l'aggiunta di 0,5 mL di FBS al 100% alla coltura cellulare trattata con tripsina, seguita dalla centrifugazione per trasferire le cellule al loro terreno di coltura originale. - Dissociare gli aggregati cellulari mediante pipettaggio per ottenere una sospensione di una singola cellula in un terreno.
NOTA: Il pipettaggio con una pipetta sierologica con un puntale da 1000 μl sulla parte superiore migliora notevolmente la generazione di sospensioni a singola cellula in un grande volume di colture cellulari in sospensione. - Utilizzare una camera di conteggio (emocitometro a righe di Neubauer o alternative) per contare il numero di cellule per 1 mL di sospensione cellulare.
- Diluire la sospensione cellulare per ottenere il numero desiderato di cellule per millilitro.
- Aggiungere una soluzione concentrata di sonda di O2 (nanoparticelle) alla sospensione cellulare a una concentrazione finale di 10 μg/mL per l'analisi raziometrica di O2.
NOTA: Per garantire una sospensione cellulare omogenea (con la sonda), risospendere più volte prima della formazione degli sferoidi. Se la sonda O2 non è necessaria, saltare questo passaggio e procedere con la formazione degli sferoidi. Quando si maneggiano le iPSC, è necessario applicare un protocollo modificato. In breve, le iPSC vengono coltivate in colonie su piastre rivestite di Geltrex e fatte passare utilizzando ReLeSR come descritto nel protocollo48 fornito dal fornitore. Il giorno della formazione degli sferoidi, le colonie dovrebbero essere grandi, compatte e mostrare centri multistrato con bordi distinti. Sciacquare le celle con PBS sterile preriscaldato. Aggiungere 1 mL di reagente di dissociazione cellulare delicata (GCDR) e incubare a 37 °C per 8-10 minuti. Utilizzare un puntale da 1000 μl per staccare delicatamente le cellule dal pozzetto e ottenere una sospensione a cella singola. Trasferire la sospensione di una singola cellula in una provetta conica sterile da 50 mL e aggiungere 4 mL di terreno DMEM-F12 preriscaldato per neutralizzare il GCDR. Lavare il pozzetto con 1 mL di DMEM-F12 e trasferirlo nel resto della sospensione cellulare. Centrifugare a 300 x g per 5 min. Risospendere in 1 mL di terreno appropriato per ulteriori esperimenti. Per gli esperimenti descritti in questo manoscritto, è stato utilizzato mTeSR + 10 μM Rock Inhibitor. Contare e diluire la sospensione cellulare per ottenere il numero desiderato di cellule per millilitro.
- Metodi di formazione degli sferoidi
- Micro-stampi per piastre di Petri 3D
NOTA: Questo metodo ad alta produttività viene utilizzato per generare simultaneamente un numero elevato di sferoidi (81 sferoidi) in un array di microstampi 9 x 9 con diametro di 800 μm e profondità di 800 μm.- Sciacquare i microstampi per la colata di piastre di Petri 3D in dH2O e metterli in un contenitore autoclavabile.
- Misurare 2 g di polvere di agarosio per elettroforesi e metterla in un flacone di vetro asciutto da 200 ml adatto all'autoclave.
NOTA: Assicurarsi che il flacone e la polvere di agarosio siano asciutti e privi di liquidi o umidità. - Microstampi in autoclave per la colata di piastre e bottiglie di Petri 3D con polvere di agarosio per 30 minuti su ciclo a secco.
- Preparare una soluzione salina allo 0,9 p/v% aggiungendo 0,9 g di NaCl in 100 mL di acqua ultrapura e sterilizzare in autoclave.
NOTA: NaCl è raccomandato dal produttore. Aumenta la stabilità dell'agarosio. - Preparare la soluzione di agarosio aggiungendo la soluzione salina sterile alla polvere di agarosio sterilizzata. Avvitare il coperchio senza stringere per evitare l'accumulo di pressione. Agitare il flacone per mescolare la polvere di agarosio.
- Far bollire e sciogliere la polvere di agarosio utilizzando un forno a microonde. Arrestare frequentemente il microonde (~ogni 10 s). Agitare la bottiglia e ripetere fino a quando l'agarosio non si sarà sciolto.
ATTENZIONE: La soluzione di agarosio è calda e richiede un'attenta manipolazione. L'agitazione immediatamente dopo la procedura di fusione può causare la fuoriuscita della soluzione dal recipiente. Per evitare incidenti, utilizzare recipienti sufficientemente grandi riempiti a non più del 50% della capacità e utilizzare un'adeguata protezione personale (guanti, guanto da forno, protezione per gli occhi e camice da laboratorio). - Lasciare raffreddare la soluzione di agarosio disciolta a 60-70 °C. Utilizzando tecniche e condizioni asettiche, pipettare l'agarosio fuso nel microstampo (500 μl per la serie 12 o 330 μl per la serie 24).
NOTA: Evitare la formazione di bolle durante la miscelazione o il pipettaggio dell'agarosio. Rimuovere eventuali piccole bolle intrappolate nelle piccole caratteristiche del microstampo mediante pipettaggio o raschiatura delicata prima che l'agarosio si solidifichi. - Lasciate solidificare l'agarosio per circa 2-3 minuti. Successivamente, flettere con cura il microstampo per rimuovere la piastra di Petri 3D e trasferirla su una piastra di coltura tissutale a 12 pozzetti.
NOTA: La flessione eccessiva del microstampo potrebbe portare alla formazione di crepe all'interno dello stampo di agarosio. - Per equilibrare la piastra di Petri 3D, aggiungere 2,5 mL/pozzetto di terreno di coltura cellulare. Incubare per 15 minuti o più. Rimuovere il terreno di coltura e sostituirlo con un terreno nuovo. Ripetere ancora una volta per equilibrare la piastra di Petri 3D con un terreno di coltura.
NOTA: Il protocollo può essere interrotto fino alla semina cellulare. Per la conservazione a lungo termine (fino a 2 settimane a 4 °C), utilizzare la soluzione PBS al posto del terreno. - Rimuovere completamente la piastra di Petri 3D che circonda il terreno di coltura (o PBS) e rimuovere accuratamente il terreno all'interno della piastra di Petri 3D inclinando la piastra di coltura tissutale.
- Seminare con cura 190 μl di sospensione cellulare contenente 40.500 cellule goccia per goccia nella camera di semina cellulare (vedere il passaggio 1.1).
NOTA: Il numero di micropozzetti nei timbri di agarosio determina il numero di sferoidi prodotti per timbro. In questo caso, questo stampo di agarosio contiene 81 micropozzetti (81 x 500 cellule/sferoide). La variazione della concentrazione di cellule in sospensione aggiunta a un macropozzetto consente di modificare il numero di cellule per sferoide, controllando così la dimensione dello sferoide. - Attendere ~10 minuti affinché le celle si stabilizzino nelle caratteristiche del Petri dsh 3D. Quindi, aggiungere 2 ml di terreno all'esterno della piastra di Petri 3D.
- Posizionare la piastra di coltura tissutale nell'incubatore per colture cellulari e sostituire il terreno che circonda la piastra di Petri 3D, se necessario.
- Piastra di fissaggio bassa
NOTA: Questo metodo viene utilizzato per generare un singolo sferoide per pozzetto. Le piastre rivestite (Lipidure o Biofloat) sono disponibili in commercio (saltare i passaggi 1.2.2.1-1.2.2.4). In alternativa, il rivestimento può essere acquistato separatamente e utilizzato per rivestire piastre multipozzetto non trattate. Si consiglia di riempire i pozzetti ai bordi delle piastre da 96 pozzetti con acqua sterile o PBS a causa dell'evaporazione più rapida in questi pozzetti, limitando così il numero di pozzetti per gli sferoidi a 60. Nel caso in cui siano necessari meno sferoidi, riempire i pozzi vuoti circostanti con acqua o PBS. Utilizzare punte prive di polvere per la manipolazione dei liquidi per evitare di portare piccole particelle nei pozzetti, poiché interferiscono con la formazione di sferoidi.- Preparare una soluzione di rivestimento 0,5 p/v% sciogliendo 0,25 g di polvere polimerica in 50 mL di etanolo in un contenitore di vetro. Filtrare-sterilizzare il rivestimento.
NOTA: La sterilizzazione del filtro e tutte le fasi successive devono essere eseguite in condizioni sterili a flusso laminare. - Aggiungere 200 μl della soluzione di rivestimento a ciascun pozzetto di una piastra di coltura con fondo a 96 U.
- Incubare per 1 minuto e rimuovere il rivestimento in eccesso.
NOTA: La soluzione di rivestimento può essere utilizzata più volte. Conservare in un contenitore di vetro a temperatura ambiente (RT). I contenitori di plastica non sono consigliati, poiché la plastica può dissolversi parzialmente e diventare parte della soluzione. Se è presente polvere, sterilizzare con filtri per siringa in polietersulfone (PES) da 0,22 μm o in nylon. - Lasciare asciugare la piastra a 96 pozzetti all'aria per circa 1 ora.
NOTA: Se le cellule necessitano di una matrice extracellulare, procedere al passaggio 1.2.3. Le lastre rivestite possono essere conservate presso RT se avvolte in alluminio per un massimo di 1 mese. Quando si semina un numero inferiore di cellule/pozzetto, centrifugare la piastra per 5 minuti a 300 g può aiutare a tirare giù le cellule.
- Preparare una soluzione di rivestimento 0,5 p/v% sciogliendo 0,25 g di polvere polimerica in 50 mL di etanolo in un contenitore di vetro. Filtrare-sterilizzare il rivestimento.
- Protocollo di formazione assistita da matrice extracellulare
NOTA: Alcune linee cellulari non producono abbastanza matrice extracellulare (ECM) e necessitano dell'aggiunta di ECM come Matrigel, Cultrex o Geltrex per formare sferoidi compatti 49,50,51. Per tipi di cellule come il cancro al seno MDA-MB-231, la papilla dermica umana, le cellule tumorali della prostata e altri, è possibile utilizzare il passaggio 1.2.2 con le seguenti modifiche52, che richiedono l'aggiunta di ECM. Utilizzare preferibilmente punte prive di polvere per la manipolazione dei liquidi per evitare che la polvere interferisca con la formazione di sferoidi. Le fasi 1 e 4-7 devono essere eseguite in una cabina di sicurezza biologica (classe II).- Procedere con i passaggi 1.2.2.1-1.2.2.4 (piastre di fissaggio basse) per il trattamento superficiale.
- Pre-raffreddare la piastra a 96 pozzetti a 4 °C in frigorifero.
- Preparare la centrifuga con l'adattatore corretto per la piastra a 96 pozzetti e preraffreddarla a 4 °C.
- Preparare una soluzione al 5% di matrice di membrana basale (BMM) in terreni di coltura cellulare pre-raffreddati (4 °C).
NOTA: BMM si reticolazione rapidamente a RT. Durante la manipolazione, tenere le scorte e le soluzioni in ghiaccio. - Preparare la sospensione cellulare (vedere il passaggio 1.1).
- Aggiungere 50 μl della soluzione di BMM in ciascun pozzetto.
- Aggiungere delicatamente 50 μl di sospensione cellulare in ciascun pozzetto sopra la soluzione BMM (25.000 cellule/pozzetto).
NOTA: Non far esplodere questo volume nel pozzo, altrimenti le celle si diffonderanno sui lati dei pozzetti e non si raccoglieranno sul fondo. Un numero inferiore di cellule per pozzetto può essere ottenuto regolando di conseguenza la densità cellulare. Non tutte le cellule formeranno sferoidi a tutte le densità di semina; L'ottimizzazione deve essere eseguita in base al tipo di cella e alla dimensione desiderata. Con i volumi forniti, la concentrazione finale di BMM è del 2,5%. Se è necessaria una concentrazione diversa, la soluzione madre deve essere preparata a una concentrazione inferiore/superiore. - Centrifugare la piastra a 96 pozzetti per 5 minuti a 300 x g e 4 °C.
NOTA: Senza questo passaggio, le cellule non si aggregano correttamente sul fondo del pozzo, causando la formazione di più aggregati più piccoli. La centrifuga deve essere raffreddata per evitare la reticolazione in questa fase. - Posizionare la piastra nell'incubatore per colture cellulari. Gli aggregati sono considerati maturi il giorno 4 dopo la semina.
NOTA: Per ulteriori protocolli sulla formazione degli sferoidi, fare riferimento al File supplementare 1.
- Micro-stampi per piastre di Petri 3D
2. Microscopia dal vivo di sferoidi
- Preparazione di sferoidi per l'analisi live imaging
NOTA: A seconda del disegno dell'esperimento (ad esempio, monitoraggio a lungo termine o analisi del punto finale, configurazione del microscopio o proprietà spettrali della fluorescenza misurata) o a causa dell'incompatibilità del metodo di produzione degli sferoidi con la microscopia (ad esempio, spessore del campione, autofluorescenza del materiale, fluttuazione degli sferoidi durante l'imaging) monitoraggio diretto degli sferoidi nella piastra, dove sono stati prodotti, potrebbe non essere possibile. Il protocollo spiega la preparazione degli sferoidi per l'imaging, che sono adatti per la maggior parte dei microscopi a campo largo invertito e confocali.- Preparare e preriscaldare i terreni di imaging (37 °C): DMEM integrato con HEPES-Na, pH 7,2 (10 mM), piruvato di sodio (1 mM), L-glutammina (2 mM) e glucosio (5 mM), senza rosso fenolo.
NOTA: Il bicarbonato di sodio da solo o in combinazione con HEPES-Na può essere utilizzato, se durante l'imaging24 viene fornito il controllo della CO2. Alcuni tipi di colture cellulari non tollerano la presenza di HEPES. A seconda del disegno sperimentale, il contenuto di piruvato, glutammina e glucosio può essere modificato. - Preparare piastre sterili per microscopia (disponibili in commercio o prodotte in laboratorio) con superfici in vetro di copertura rivestite (per una forte adesione sferoide) o non rivestite (bassa adesione sferoide) (spessore #1,5).
NOTA: La necessità e il tipo di rivestimento dipendono dal tipo di cellula, dalle proprietà di adesione degli sferoidi e dalla velocità della loro migrazione cellulare dall'interfaccia di coltura 3D a quella 2D. Questo è importante da considerare, in quanto il rivestimento può facilitare la perdita di organizzazione 3D, modificando la forma dei micro-gradienti negli sferoidi e, di conseguenza, il comportamento cellulare. Per alcuni esperimenti (ad esempio, l'imaging ha analizzato la risposta alla stimolazione farmacologica), è richiesta una forte aderenza sferoidale alla superficie ed è preferibile un rivestimento con gelatina, BMM, collagene, collagene/poli-D-lisina o poli-D-lisina. - Lavare delicatamente gli sferoidi colorati con sonda O2 dai micropozzetti dell'agarosio micromodellato o della piastra a 96 pozzetti e trasferire gli sferoidi ancora galleggianti in una fiala da 2 mL.
NOTA: Per garantire la raccolta di tutti gli sferoidi con il metodo ad alta produttività, sciacquare lo stampo 1-3 volte con il volume aggiuntivo di terreno di coltura, combinando tutte le sospensioni di sferoidi in un'unica fiala. Per gli sferoidi in una piastra di attacco bassa, raccogliere gli sferoidi uno per uno dai singoli pozzetti in una fiala o direttamente in una capsula per microscopia se un piccolo numero di sferoidi è sufficiente per l'esperimento. Quando si trasferiscono sferoidi di grandi dimensioni, tagliare l'estremità del puntale della pipetta per garantire che non si verifichino danni durante il pipettaggio. - Lasciare il flaconcino in posizione verticale per un massimo di 5 minuti per consentire agli sferoidi di depositarsi sul fondo del flaconcino, formando un pellet visibile.
- Rimuovete il terreno dal tubo, lasciando indisturbati gli sferoidi, e risospendeteli delicatamente in una quantità sufficiente di coltura fresca.
NOTA: Per comodità di manipolazione degli sferoidi, il trasferimento degli sferoidi al supporto di imaging può essere effettuato anche in questa fase in piccoli lotti; Vedere i passaggi 2.1.7 e 2.1.8. Gli sferoidi dei diversi gruppi sperimentali devono essere trattati allo stesso modo, poiché la composizione del terreno e il tempo di precondizionamento del terreno possono influenzare il loro metabolismo. - Mentre gli sferoidi galleggiano, trasferire un volume uguale di sospensione di sferoidi per pozzetto della capsula di microscopia.
- Incubare gli sferoidi per 1-2 ore a 37 °C in un incubatore di CO2 per garantire il loro fissaggio alla superficie della piastra/pozzetto per microscopia. Per l'imaging, procedere con il passaggio 2.1.9. Per la colorazione degli sferoidi con sonde aggiuntive, procedere con il passaggio 2.1.8.
NOTA: La velocità di migrazione delle celle dallo sferoide 3D all'interfaccia della superficie 2D è una funzione del tempo. Per evitare una perdita di organizzazione 3D, il tempo di incubazione deve essere ottimizzato rispetto al tipo di cellula, al tipo di rivestimento superficiale e al design dell'esperimento. Ad esempio, HCT116, a seconda delle dimensioni dello sferoide, richiede almeno 2 ore per un corretto attacco dello sferoide alla superficie rivestita di collagene IV/poli-D-lisina, mentre l'attacco e la migrazione dell'hDPSC all'interfaccia 2D sono estremamente rapidi, portando alla perdita dell'organizzazione 3D in 1-2 ore. Per evitare l'imaging di "sferoidi 2D" a causa di un'eccessiva diffusione, vengono utilizzate superfici di vetro non rivestite con un tempo di incubazione ridotto. - Aggiungere una o più sonde di fluorescenza nelle concentrazioni raccomandate o empiricamente ottimizzate a un volume noto di sospensione di sferoidi. Incubare per 1 ora a 37 °C con incubatore di CO2 prima dell'imaging.
NOTA: Per il saggio vivo/morto, utilizzare ioduro di propidio e Calceina Green-AM in una concentrazione finale standard di 1 μg/mL. Per evitare l'effetto tossico della colorazione con ioduro di propidio sui corpi embrioidi iPSC, la concentrazione finale di ioduro di propidio era di 0,5 μg/mL. Il tempo di caricamento della sonda può essere prolungato se la diffusione della sonda non è efficiente a causa delle grandi dimensioni degli sferoidi. Il tempo di caricamento deve sempre essere considerato come parte del tempo totale di incubazione necessario per l'adesione degli sferoidi alla superficie. Se è necessario un tempo più lungo per l'attacco degli sferoidi, la procedura di colorazione deve essere organizzata alla fine di questo periodo. Tieni presente che un tempo di incubazione più lungo potrebbe portare alla perdita dell'organizzazione 3D. - Rimuovere i terreni di coltura cellulare o i terreni contenenti sonde fluorescenti e sostituirli con un volume necessario di terreni di imaging. Per garantire che non vi siano fondi di fluorescenza del terreno durante l'imaging sferoidale, ripetere la fase di scambio del terreno (lavaggio) fino a 5 volte.
NOTA: Per evitare la rimozione degli sferoidi durante la sostituzione dei terreni, si consiglia di aspirare con cura i terreni con una pipetta da 200 μl dai bordi delle piastre per microscopia ed eseguire l'aggiunta di terreni dalla parete o lateralmente nella piastra per microscopia. - Procedere immediatamente con il passaggio 2.2.1 del protocollo di imaging.
NOTA: Una pausa troppo lunga tra la preparazione all'imaging e l'acquisizione effettiva dell'imaging può influenzare il metabolismo cellulare (ad esempio, attraverso la modifica della composizione del terreno), la vitalità (alcune sonde fluorescenti utilizzate per l'analisi degli endpoint hanno effetti tossici, che possono stimolare la morte cellulare dopo un lungo periodo di incubazione) e portare a una perdita di organizzazione 3D. Se è necessario confrontare più gruppi di sferoidi o condizioni sperimentali, il disegno sperimentale deve essere sviluppato di conseguenza per mantenere la tempistica del trattamento, del precondizionamento e delle procedure di imaging il più equa possibile tra i gruppi analizzati.
- Preparare e preriscaldare i terreni di imaging (37 °C): DMEM integrato con HEPES-Na, pH 7,2 (10 mM), piruvato di sodio (1 mM), L-glutammina (2 mM) e glucosio (5 mM), senza rosso fenolo.
- Acquisizione delle immagini
NOTA: Il protocollo descrive l'imaging multiparametrico di sferoidi vivi utilizzando il microscopio confocale Stellaris 8 Falcon (Leica) e il software Leica Application Suite X (LAS X) versione 4.7. Tuttavia, saranno necessarie solo piccole modifiche per eseguire tale analisi su piattaforme di microscopia alternative.- Accendere l'unità di controllo del climatizzatore della temperatura 30-60 minuti prima dell'imaging. Impostare la velocità di ventilazione e la temperatura necessarie (35-37 °C).
NOTA: Se oltre al controllo della temperatura, è necessario controllare la concentrazione di gas (ad es. CO2 o O2) durante l'imaging, anche i dispositivi corrispondenti devono essere avviati in anticipo per raggiungere le condizioni necessarie prima dell'imaging. - Accendere il microscopio e i dispositivi collegati (ad esempio, laser WLL, computer, pompa dell'acqua per l'obiettivo a immersione in acqua e altri blocchi elettronici operativi). Avviare il software di controllo del microscopio (ad esempio, LAS X Machine Mode o Machine Mode with Environmental Control) fornito con l'esatta configurazione del microscopio e inizializzare la calibrazione del tavolino.
- Scegliere l'obiettivo richiesto nel software e applicare il fluido di immersione, se necessario.
NOTA: Per la microscopia dal vivo, si consiglia di utilizzare obiettivi ad immersione in acqua o glicerolo con una distanza di lavoro sufficiente ("lunga"), ad esempio HC Fluotar L 25x/0,95 W VIS IR (distanza di lavoro 2,4 mm), HC PL Apo 40x/1,25 GLYC (distanza di lavoro 0,35 mm) o almeno NA = 0,4 o superiore per gli obiettivi in aria. La scelta dell'ingrandimento e della distanza di lavoro dipende dalla natura e dalle dimensioni del campione ripreso e dai segnali di fluorescenza misurati (luminosità, resa quantica, efficienza di colorazione, vedi ad esempio, discussione su coloranti e nanoparticelle53). Oggetti di grandi dimensioni (sferoidi o organoidi, dimensioni >500 μm), "bioreattori" o chip microfluidici richiedono obiettivi a lunga distanza di lavoro e un ingrandimento inferiore, mentre l'analisi di singole cellule o organelli cellulari richiede un elevato ingrandimento, spesso ottenuto tramite imaging "a mosaico". - Prepara il piatto per microscopia con gli sferoidi sul palco. Regola la messa a fuoco e trova un oggetto/regione di interesse (ROI).
NOTA: Se è necessario trovare oggetti piccoli, debolmente fluorescenti, con scarso contrasto o rari e trovare la messa a fuoco è difficile, si consiglia di mettere a fuoco in anticipo le pareti del piatto per microscopia e di "schermare" la superficie per l'oggetto di interesse mediante serpentina, partendo da uno degli angoli del pozzetto. - Scegliere la finestra Apri progetto e fare clic sull'icona corrispondente, Crea un nuovo progetto. Assegna un nome standard (ad esempio, a partire da "descrizione AA-MM-GG+") al file del progetto di ricerca. Durante l'imaging, tutte le immagini prodotte verranno salvate automaticamente nel file .lif del progetto creato.
- Aprire la finestra Acquisizione . Impostare la lunghezza d'onda di eccitazione del laser a luce bianca (WLL) e la gamma richiesta di rivelatori ibridi o a scansione di risonanza (tipo HyD S, HyD X o HyD R) in base alle proprietà spettrali note della fluorescenza misurata (spettri di eccitazione/assorbanza e di emissione). Scegliere i tipi di scansione Linea o Cornice .
NOTA: Per la maggior parte dei coloranti fluorescenti disponibili in commercio, le proprietà spettrali possono essere trovate (o aggiunte) nella confezione di LAS X Dye Assistant. Scegliere il rivelatore con l'intervallo di sensibilità spettrale appropriato compatibile con le proprietà spettrali della sonda e, nel caso di FLIM, compatibile con il conteggio dei fotoni (ad esempio, HyD X o HyD R). Per l'imaging multiparametrico, impostare la WLL in più posizioni di eccitazione (ad esempio, per l'imaging simultaneo di FAD/Flavine e due canali di fluorescenza della sonda raziometrica MMIR1 O2 - riferimento e sensibile, le impostazioni di eccitazione/emissione potrebbero essere 460 nm/510-590 nm HyD X1 e 614 nm/631-690 HyD X3 e 724-800 nm HyD R di conseguenza in una o due sequenze di scansione sequenziali). È importante assegnare il rivelatore appropriato per raccogliere l'emissione, poiché i rivelatori possono avere una diversa sensibilità spettrale54. - (Opzionale per FLIM) Nella finestra Acquisizione , scegliere la modalità FLIM per eseguire l'imaging combinato con il conteggio dei fotoni (raccolta del decadimento). Immediatamente, verrà aperto un ulteriore "modulo FLIM nel software LAS X" per navigare e analizzare i dati FLIM.
- (Opzionale per FLIM) Scegliere la frequenza di ripetizione dell'impulso WLL in base alla durata media prevista del fluoroforo.
NOTA: La frequenza dell'impulso laser deve essere regolata per raccogliere il decadimento completo della fluorescenza. Questa operazione può essere eseguita utilizzando una funzione di selezione degli impulsi installata sul microscopio. La sovrapposizione del decadimento della fluorescenza con l'impulso laser porterà alla riduzione della durata stimata della fluorescenza. Si raccomanda di avere intervalli di impulsi 4-5 volte più lunghi della durata media prevista della fluorescenza (ad esempio, 25 ns/40 MHz per durate fino a 5 ns). Molti laser pulsati hanno una frequenza di ripetizione fissa di 80 MHz (ideale solo per una portata fino a 2-3 ns). Questo è importante per la scelta dei fluorofori corretti per l'esperimento. - Avviare l'anteprima dell'immagine utilizzando la modalità FAST LIVE e regolare la messa a fuoco fine dell'oggetto di imaging su una sezione di interesse.
ATTENZIONE: Seguire rigorosamente le regole di sicurezza del laser. Considerare sempre le regole di sicurezza del laser e attendere che l'imaging si sia fermato prima di accendere la luce di trasmissione, guardare nell'oculare o nel campione.
NOTA: In modalità FAST LIVE , una scansione ad alta velocità di 600 Hz (corrisponde a una frequenza fotogrammi massima di 4,43/s se si utilizza la modalità di scansione X bidirezionale), una risoluzione di 256 x 256 pixel viene applicata automaticamente all'immagine per mantenere la fluorescenza al sicuro dal fotosbiancamento. Aprire il foro stenopeico (ad esempio, a 3-4 UA) e/o aumentare l'intensità del laser se il segnale di fluorescenza è troppo debole per mettere a fuoco l'oggetto. Evitare una raccolta di decadimento incompleta. - (Opzionale per FLIM) Osservando un istogramma dell'intensità dei pixel che appare durante l'imaging in una finestra del modulo FLIM (modalità Live ), regolare l'intensità del laser/le dimensioni del foro stenopeico e la risoluzione appropriate per raggiungere il limite di frequenza di conteggio ~1 fotone/impulso laser (linea rossa). Evitare di andare significativamente più in alto di 1 per escludere il rischio di effetto pile-up. Se necessario, regolare la frequenza di ripetizione dell'impulso WLL per avere una raccolta completa del decadimento in una finestra di decadimento (per evitare una raccolta del decadimento incompleta, vedere il passaggio 2.2.8).
NOTA: Se il numero di fotoni (intensità) non è sufficiente per ricostruire un decadimento affidabile per l'analisi di adattamento o la nuvola del diagramma dei fasori, applicare diverse ripetizioni di scansione (fotogrammi o linee, o impostare il tempo di scansione), aumentare l'intensità del laser e/o sacrificare la risoluzione (dimensione del ROI scansionato). Tenere presente che la riduzione della frequenza di ripetizione del laser richiede la raccolta di più fotoni per una ricostruzione affidabile del decadimento e potrebbe essere necessaria un'ulteriore correzione dei parametri di imaging. Essere consapevoli del potenziale impatto di una luce intensa e di un'illuminazione lunga e continua sulla vitalità cellulare e sul metabolismo55. L'impatto negativo sulla vitalità e sul metabolismo può essere diverso in ogni singolo caso, a seconda dell'intensità, della durata e della lunghezza d'onda della luce di eccitazione, nonché della modalità di imaging (ad esempio, imaging confocale a un fotone o multifotone). Regolare di conseguenza i parametri di imaging e, se necessario, controllare la vitalità/morte cellulare mediante Calcein Green-AM o intensità di ioduro di propidio negli esperimenti pilota56. Ove possibile, dovrebbero essere prese in considerazione ulteriori ottimizzazioni del protocollo di colorazione della sonda a fluorescenza per raggiungere un segnale di fluorescenza adeguato durante la microscopia dal vivo. - (Opzionale per lo z-stack 3D) In Fast Live impostare le coordinate, la direzione di scansione e attribuirle a Inizio e Fine nella finestra Z-stack (modalità di scansione XYZ). Scegli la dimensione del passo Z o il numero di passi.
NOTA: Mentre il software calcola automaticamente il numero "ottimale" di passaggi, in base alla risoluzione utilizzata e ai parametri di scansione, la ricostruzione 3D in tempo reale può normalmente richiedere un numero inferiore di passaggi per ottenere un'acquisizione rapida, ad esempio una dimensione del passo di 1-2 μm, una dimensione dello stack di 50-100 μm, una scansione bidirezionale, che richiede 2-3 minuti del tempo di scansione totale. Tieni presente che anche gli organelli subcellulari, le cellule e il modello cellulare 3D possono muoversi durante le misurazioni. Inoltre, a causa della profondità di penetrazione della luce e dei limiti di diffusione, in genere è possibile raggiungere solo una profondità di scansione di 50-100 μm su FLIM confocale. - Quando tutte le impostazioni necessarie sono state applicate, avviare l'imaging.
- Assegna all'immagine un nome appropriato.
- Cercare l'oggetto di imaging successivo in modalità luce di trasmissione e ripetere la procedura di imaging con le impostazioni di imaging precedentemente ottimizzate (passaggi 2.2.8-2.2.12).
NOTA: Per il confronto basato sull'intensità o l'analisi del rapporto di intensità (ad esempio, l'analisi dell'ossigenazione basata su sonda MMIR O2), mantenere sempre le stesse impostazioni di imaging per tutti gli oggetti analizzati (ingrandimento e tipo di lente dell'obiettivo, intensità laser, potenza e frequenza dell'impulso, lunghezza d'onda di eccitazione, portata dei rivelatori, foro stenopeico, velocità di scansione, tempo di permanenza dei pixel e risoluzione). Tuttavia, poiché la durata della fluorescenza non dipende dall'intensità della fluorescenza e richiede la raccolta di un numero appropriato di fotoni per un calcolo affidabile, i parametri di imaging FLIM possono essere riaggiustati nel corso dell'esperimento per mantenere il numero di fotoni raccolti confrontabile tra diversi trattamenti o condizioni sperimentali. Pertanto, per l'analisi multiparametrica in cui sono necessarie sia l'analisi basata sull'intensità che quella basata sulla durata della fluorescenza, è necessario applicare impostazioni di imaging universali ottimizzate per tutti gli oggetti nei gruppi sperimentali confrontati. Per il confronto "solo FLIM" è possibile confrontare le immagini acquisite con impostazioni di imaging leggermente diverse, poiché il software LAS X fornisce il calcolo dell'IRF per la misurazione delle singole immagini42. Tuttavia, per l'analisi FLIM-fitting al di fuori di LAS X (ad es. FLIMfit45) la funzione di risposta dello strumento (IRF) deve essere misurata per ogni diversa condizione di imaging, in quanto non può essere esportata dal software di imaging. Pertanto, per la semplicità del disegno sperimentale e del carico di lavoro, si consiglia di applicare le stesse impostazioni di imaging per tutte le immagini nel set di dati. Quindi, le corrispondenti misure IRF possono essere eseguite con l'uso di fluorofori temprati o a fluorescenza veloce (entro l'intervallo ps) con le proprietà di emissione del canale spettrale misurato 57,58,59, mediante luminescenza60 di nanoparticelle d'oro o mediante segnale di generazione di seconda armonica per FLIM 61 multifotone. Nel software LAS X, i parametri di imaging precedentemente ottimizzati possono essere caricati per un nuovo progetto facendo clic con il pulsante destro del mouse sul file di interesse e scegliendo Applica impostazioni immagine. - Al termine della sessione di imaging, salvare il progetto di imaging. Per finalizzare la sessione di imaging, rimuovere il campione dalla fase di microscopia e pulire l'obiettivo dal liquido di immersione (se utilizzato) secondo la procedura standard implementata nella struttura di imaging. Chiudi il progetto e il software. Spegnere il microscopio, i laser e tutti i dispositivi collegati.
- Procedere con l'analisi dei dati di imaging (passaggio 2.3).
- Accendere l'unità di controllo del climatizzatore della temperatura 30-60 minuti prima dell'imaging. Impostare la velocità di ventilazione e la temperatura necessarie (35-37 °C).
- Elaborazione delle immagini del fasore NAD(P)H/FAD-FLIM con modulo LAS X FLIM e FIJI
NOTA: Il protocollo descrive un'analisi del tempo di vita della fluorescenza di sferoidi imaged per dati nel dominio della frequenza su esempi di FLIM in autofluorescenza NAD(P)H e FAD/Flavine. La misurazione dell'autofluorescenza del NAD(P)H è diventata un gold standard per l'analisi metabolica, in cui i componenti del ciclo di vita dell'autofluorescenza del NAD(P)H brevi e più lunghi sono associati rispettivamente alla glicolisi o alla fosforilazione ossidativa (OxPhos). Questo può essere analizzato dallo spostamento su un grafico fasoriale lungo la traiettoria metabolica verso gli standard misurati di NAD(P)H libero o NAD(P)H legato alle proteine31,62. Per analizzare la traiettoria dello spostamento metabolico, nonché per confrontare la posizione delle nubi fasoriali (vedere la NOTA sotto il passaggio 2.3.6) su un grafico tra gruppi sperimentali, è stata implementata un'analisi semplificata delle coordinate fasoriali, basata sul calcolo del centro geometrico (centroide) della nube fasoriale29. Il protocollo descritto dimostra il calcolo delle coordinate del centroide nelle FIJI e la misurazione della distanza tra due punti su un grafico fasoriale utilizzando le coordinate (ad esempio, la distanza da un baricentro della nube di fasore di autofluorescenza dello sferoide NAD(P)H a un punto teorico "NAD(P)H libero"). Allo stesso modo, è possibile analizzare il FAD e altri segnali di autofluorescenza. Un set di dati 1 con .lif (è richiesto il software LAS X) o .ptu Formati di file per l'apprendimento di questa procedura è disponibile (Fascicolo supplementare 2, Fascicolo supplementare 3, Fascicolo supplementare 4, Fascicolo supplementare 5, Fascicolo supplementare 6, Fascicolo supplementare 7, Fascicolo supplementare 8, Fascicolo supplementare 9, e Fascicolo supplementare 10).- Aprire il modulo FLIM in LAS X, selezionare Apri progetto e caricare il file immagine sferoidale (.lif) per l'analisi NAD(P)H/FAD in autofluorescenza.
NOTA: A causa di potenziali bug e perdite di dati intermedi nel modulo FLIM, utilizzare una copia del file immagine sferoidale originale (.lif) per l'analisi NAD(P)H/FAD, mantenendo invariato il file raw. - Selezionare una singola immagine e accedere all'interfaccia di analisi FLIM. Fare clic su Phasor per accedere al grafico dei fasori e attivare la modalità di analisi dei fasori. Applica il filtro (Mediano o Wavelet) e imposta la soglia per ridurre al minimo il rumore e migliorare la visibilità dei dati per tutte le analisi dei fasori. Scegli le armoniche. Per l'analisi dello spostamento metabolico basata sui dati NAD(P)H, procedere con i passaggi 2.3.3-2.3.5. Per il confronto generale dei grafici dei fasori, procedere dal passaggio 2.3.6.
NOTA: Applica le impostazioni di analisi (tipo di filtro, armoniche, soglia, binning e ROI del fasore) allo stesso modo a tutte le immagini in un set di dati confrontato. - (Facoltativo per l'analisi NAD(P)H) Scegli un'immagine correlata al set di dati e utilizza l'opzione Cursore rapporto di disegno per due componenti per individuare con precisione la posizione di 0,45 ns su un cerchio universale di uno spazio di grafico a fasatori standard. Questa posizione sarà assegnata a un tempo medio di fluorescenza di una soluzione omogenea pura di NAD(P)H libero, che normalmente è vicino al decadimento mono-esponenziale62.
NOTA: Il NADH libero e il NAD(P)H libero hanno proprietà spettrali simili e valori simili di durata della fluorescenza in soluzione acquosa, con due componenti di breve durata, 0,3 ns e 0,7 ns63. Pertanto, per la semplicità dell'analisi basata sui fasori e a causa di una piccola differenza tra i componenti del tempo di vita, il loro decadimento della fluorescenza è accettato come mono-esponenziale, il che consente l'allocazione della nuvola di fasore su un cerchio universale. La durata media di riferimento di una forma di NAD(P)H libero può anche essere misurata e tracciata in uno spazio fasoriale per un'analisi simile. La durata di riferimento è stata scelta in base alla letteratura62; si noti che in altre fonti, si può trovare un valore leggermente diverso di un NAD(P)H libero in soluzione (0,4 ns64). - (Facoltativo per l'analisi NAD(P)H) Esportare il grafico dei fasori con la durata del NAD(P)H libero allocata (vedere il passaggio 2.3.3) facendo clic con il pulsante destro del mouse sul grafico e selezionando Esporta dati. Esportare il grafico dei fasori come file in formato .tiff in una cartella di archiviazione designata.
NOTA: La dimensione originale in pixel dell'immagine del grafico a fasi esportata dal modulo LAS X FLIM è sempre di 1024 x 600 pixel. Se viene utilizzato un altro software per l'esportazione dei dati e la pre-analisi, assicurarsi che tutte le immagini del grafico dei fasori vengano esportate con le stesse dimensioni e risoluzione. - Per esportare la nuvola di fasori relativa agli sferoidi, utilizzare lo strumento Disegna cursore nel modulo LAS X FLIM per scegliere la ROI sferoidale sull'immagine. Esportare il grafico dei fasori generato come descritto nel passaggio 2.3.4.
NOTA: Le corrispondenti coordinate g e s (simili a x e y) dello spazio dei fasori saranno assegnate ad ogni pixel della ROI scelta, in base alla loro durata, trasformate in un set di dati nel dominio della frequenza64,65. Il gruppo di pixel con valori simili di tf (durata di fase) e tm (durata di modulazione) formerà un modello di nuvole (nuvola di fasori) su un grafico, in cui la codifica dei colori (con un intervallo dal blu intenso al rosso) rifletterà l'abbondanza di valori di vita. Dalla posizione della nuvola su un cerchio universale o all'interno, si possono distinguere i decadimenti mono- o multi-esponenziali. Alcuni mezzi di misura mostrano una forte (auto)fluorescenza, che porta alla comparsa di una nuvola corrispondente su un grafico fasoriale, che non può essere semplicemente rimossa con la soglia di intensità. Questo modello influenzerà il calcolo delle coordinate del centroide e deve essere escluso dal grafico dei fasori esportato. Lavorare con il ROI sferoidale consente di escludere i pixel non correlati da ulteriori analisi dei fasori. - Ripetere la procedura di esportazione del grafico a fasi per tutte le ROI sferoidi in un set di dati (vedere i passaggi 2.3.4 e 2.3.6). Inoltre, controllare il set di immagini .tiff esportato per garantire l'insieme completo di dati per un'ulteriore analisi comparativa e assicurarsi che tutte le immagini esportate abbiano la stessa dimensione in pixel (vedere la NOTA nel passaggio 2.3.4).
NOTA: In questa fase del protocollo, il set di immagini deve includere i fasori con la posizione libera del NAD(P)H (basata sulla letteratura o sui dati ottenuti empiricamente) e tutti i grafici dei fasori ROI degli sferoidi (o modelli di ROI alternativi, se necessari per l'analisi specifica). Da questa fase, ulteriori analisi verranno effettuate nelle FIJI e successivamente in un foglio di calcolo. Utilizzando la finestra degli strumenti Analizza , l'opzione Imposta scala in FIJI e assicurarsi che tutte le immagini del grafico dei fasori siano calibrate con lo stesso tipo di unità, ad esempio solo in pixel. In caso contrario, impostare la lunghezza dell'unità nella finestra Imposta scala per quella scelta (ad esempio, per la scala basata sui pixel, inserire 1 nel campo Distanza in Pixel e impostare l'unità di lunghezza su Pixel). Per un ulteriore confronto, misurare tutti i dati esportati utilizzando lo stesso tipo di unità. - (Facoltativo per l'analisi NAD(P)H) Determinare la posizione del punto pixel di un tempo di vita medio della fluorescenza a NAD(P)H libero sull'immagine del grafico dei fasori esportata corrispondente (vedere il passaggio 2.3.3): aprire l'immagine del fasore con FIJI, ingrandire l'immagine per visualizzare chiaramente con una risoluzione in pixel l'intersezione tra il semicerchio universale e la linea del cursore del rapporto per due componenti ; usa lo strumento ROI rettangolo per selezionare l'intersezione.
NOTA: Assicurarsi che la selezione rettangolare sia una piccola area attorno al punto di intersezione per determinare con precisione le sue coordinate nel passaggio successivo (passaggio 2.3.9). - (Facoltativo per l'analisi NAD(P)H) Aprire lo strumento Analizza , scegliere la finestra Imposta misure e selezionare Centroide come parametro di misurazione. Fare clic su Misura nella finestra degli strumenti Analizza per determinare le coordinate del baricentro del punto di riferimento NAD(P)H libero. Esporta queste coordinate in un foglio di calcolo.
NOTA: Le coordinate Free-NAD(P)H saranno utilizzate come punto di riferimento per confrontare le distanze da questo punto alla posizione della nube di fasore sferoidale in un set di dati (il modo per caratterizzare lo spostamento metabolico tra glicolisi e OxPhos nell'analisi dell'autofluorescenza NAD(P)H FLIM) - Utilizzando FIJI, apri l'immagine della nube di fasori sferoidi. Apri lo strumento Finestra immagine , scegli Regola e seleziona Soglia colore nella barra degli strumenti. Selezionare il metodo di soglia scelto (ad esempio, Otsu) e impostare il valore di tonalità e il valore di luminosità per restringere i parametri per la selezione di una parte di nuvola di fasore con le coordinate pixel più abbondanti. Fare clic su Seleziona per definire l'area del cluster.
NOTA: Mantenere gli stessi parametri di soglia per tutte le immagini del grafico dei fasori, che devono essere analizzate. Per i dati NAD(P)H-FLIM presentati, il metodo di soglia Otsu con valore di tonalità impostato 9 e valore di luminosità 160 è stato scelto e applicato a tutte le immagini dei fasori. In alternativa, l'area selezionata può essere copiata in ROI Manager (seguire il percorso Modifica > Selezione > Aggiungi a Manager) per creare una libreria di ROI fasoriali per ulteriori analisi. - Mantenendo la selezione, calcolare le coordinate del baricentro dell'area selezionata seguendo la procedura descritta al punto 2.3.9. Esporta queste coordinate nel file del foglio di calcolo.
- Ripeti i passaggi 2.3.9 e 2.3.10 per determinare le coordinate del centroide per tutte le immagini del fasore ROI per creare un set di dati nel foglio di calcolo.
NOTA: L'utilizzo della libreria ROI Manager consente di semplificare e organizzare l'analisi del ROI (vedere il passaggio 2.3.10) - (Facoltativo per l'analisi NAD(P)H) Apri il foglio di calcolo con le coordinate del NAD(P)H di riferimento libero esportate e delle ROI sferoidi da diversi gruppi di confronto. Calcola la distanza tra ogni singolo centroide del fasore sferoidale per fare riferimento alla posizione di NAD(P)H libero utilizzando le coordinate determinate e la seguente equazione:
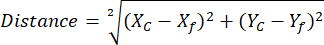
Dove, Xc e Yc sono le coordinate del baricentro, Xf e Yf sono le coordinate di riferimento.
NOTA: L'applicazione del parametro del baricentro per la determinazione dello spostamento verso la durata di riferimento è appropriata solo nel caso in cui tutti i centroidi di un set di dati giacciono sulla stessa traiettoria lineare verso il punto di riferimento. Per verificarlo, tutti i punti del baricentro del set di dati devono essere tracciati insieme al punto di riferimento nello stesso spazio di coordinate e deve essere eseguito l'allineamento lineare del trend. Se il coefficiente R2 della linea di tendenza lineare tracciata attraverso tutti i punti è vicino a 1 (ad esempio, R2 è 0,8-0,99), si presume che l'analisi della distanza sia appropriata. - Organizzare tutti i dati di conseguenza per il confronto ed eseguire analisi statistiche con l'uso di qualsiasi software corrispondente (ad esempio, Origin, MatLab). Scegliere il test statistico appropriato in base alle caratteristiche dell'insieme di dati (normalità della distribuzione, numero di unità statistiche, ecc.).
NOTA: Per l'analisi NAD(P)H, confrontare i valori di distanza per caratterizzare lo spostamento metabolico in base alle condizioni sperimentali. Per il confronto di qualsiasi grafico dei fasori tra gruppi sperimentali, eseguire il confronto delle coordinate dei centroidi delle nuvole dei fasori ROI.
- Aprire il modulo FLIM in LAS X, selezionare Apri progetto e caricare il file immagine sferoidale (.lif) per l'analisi NAD(P)H/FAD in autofluorescenza.
Risultati
Scegliere il metodo di formazione degli sferoidi appropriato
Il metodo di formazione degli sferoidi selezionato può influenzare notevolmente le dimensioni, la forma, la densità cellulare, la vitalità e la sensibilità ai farmaci degli sferoidi (Figura 2). In precedenza, gli effetti di più metodi ad alta produttività (SphericalPlate 5D, microstampi realizzati in laboratorio e stampi MicroTissue) e i metodi a basso attacco "a media produttività" (Biofloat e piastre a ...
Discussione
Gli sferoidi multicellulari stanno diventando un metodo di scelta negli studi di microambienti di nicchia di tumori e cellule staminali, nella scoperta di farmaci e nello sviluppo di "elementi costitutivi tissutali" per la biofabbricazione. L'architettura interna eterogenea degli sferoidi, i gradienti di nutrienti e l'ossigenazione possono imitare quelli dei tessuti e dei tumori in vivo in un ambiente relativamente semplificato e accessibile. Con la necessità di una maggiore trasparenza metodologica
Divulgazioni
Nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dalle sovvenzioni del Fondo speciale per la ricerca (BOF) dell'Università di Gand (BOF/STA/202009/003; BOF/IOP/2022/058), Fondazione per la ricerca delle Fiandre (FWO, I001922N) e l'Unione Europea, fliMAGIN3D-DN Horizon Europe-MSCA-DN n. 101073507.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA | Gibco | 25300-054 | Also available from Sigma |
| 10 mL serological pipets | VWR | 612-3700 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 12 well cell-culture plates, sterile | Greiner bio-one | 665-180 | Similar products are also available from Sarstedt, Corning and other companies. |
| 12 Well Chamber slide, removable | Ibidi | 81201 | Also available from Grace Bio-Labs, ThermoFisher Scientific and others |
| 15 mL centrifuge tubes | Nerbe plus | 02-502-3001 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 3D Petri Dish micromolds | Microtissue | Z764000-6EA | |
| 6 well cell-culture plates, sterile | Greiner bio-one | 657160 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 70% ethanol | ChemLab | CL02.0537.5000 | |
| Biofloat | Sarstedt | 83.3925.400 | Commercial available coated 96-well plate for spheroid formation |
| Calcein Green-AM | Tebubio | AS-89201 | Apply in dilution 1:1000 |
| CellSens Dimension software | Olympus | version 3 | |
| Collagen from human placenta, type IV | Sigma | C5533 | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Confocal FLIM Microscope | Leica Microsystems | N/A | Stellaris 8 Falcon inverted microscope with white-light laser, HyD X detectors, climate / T control chamber (OkoLab), 25x/0.95 W objective |
| D(+)-Glucose | Merck | 8342 | Prepare 1 M stock solution, 1:100 for preparation of imaging medium (final concentration 10 mM) |
| Dulbecco's modified Eagle's medium (DMEM), phenol red-, glucose-, pyruvate- and glutamine-free | Sigma-Aldrich | D5030-10X1L | For preparation of imaging medium |
| Fetal Bovine Serum (FBS) | Gibco | 10270-098 | Also available from Sigma. Needs to be heat-inactivated before use. |
| HEPES (1M) | Gibco | 15630-080 | Dilution 1/100 for preparation of imaging medium (final concentration 10 mM) |
| Human colon cancer cells HCT116 | ATCC | ||
| ImageJ | NIH | version 1.54f | |
| Leica Application Suite X (LAS X) | Leica Microsystems | version 4.6.1.27508 | |
| L-glutamine | Gibco | 25030 | Also available from Sigma. Apply in dilution 1:100. |
| Lipidure-CM5206 | Amsbio | AMS.52000034GB1G | |
| McCoy's 5A, need addition of 1 mM Sodium Pyruvate and 10 mM HEPES | VWR | 392-0420 | Standard growth medium for HCT116 cells |
| micro-patterned 3D-printed PDMS stamps | N/A | N/A | Provided by the Centre for Microsystems Technology, Professor Dr. Jan Vanfleteren, Ghent University |
| NaCl | Chemlab | CL00.1429.100 | |
| Neubauer couting chamber | Fisher Scientific | 15980396 | |
| O2 probes: MMIR1 | N/A | N/A | Full characterization, validation and some applications can be found at: https://www.biorxiv.org/content/10.1101/2023.12.11.571110 v1 |
| PBS | Fisher scientific | Gibco18912014 | Dissolve PBS tablet in 500 mL of distilled water. |
| Pen Strep :Penicillin (10,000 U/mL) / streptomycin (10,000 μg/mL) 100x solution | Gibco | 15140-122 | Also available from Sigma. Apply in dilution 1:100. |
| Poly-D-lysine | Sigma | P6407-5mg | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Propidium Iodide | Sigma-Aldrich | 25535-16-4 | Cell death staining, use 1 µg/mL at 1h incubation |
| PVDF syringe filter 0.22 µm | Novolab | A35149 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Sodium pyruvate (100 mM) | Gibco | 11360-070 | Dilution 1/100 for preparation of imaging medium (final concentration 1mM) |
| SphericalPlate 5D 24-well | Kugelmeiers | SP5D-24W | |
| sterile petridish | Greiner bio-one | 633181 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (25 cm² ) | VWR | 734-2311 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (75 cm²) | VWR | 734-2313 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| U-bottom 96-well plate | VWR | 10062-900 | Similar products are also available from Sarstedt, Corning, Greiner Bio-one and other companies |
| Ultrapure Agarose | Invitrogen (Life Technologies) | 16500-500 | Other types of Agarose such as Agarose low melting point (A-9414, Sigma), Agarose for routine use (A-9539, Sigma) |
| Widefield fluorescence inverted microscope | Olympus | N/A | Inverted fluorescence microscope IX81, with motorised Z-axis control, CoolLED pE4000 (16 channels, 365-770 nm), ORCA-Flash4.0LT (Hamamatsu) cMOS camera, glass warming plate Okolab, CellSens Dimension v.3 software and air objectives 4x/0.13 UPlanFLN and 40x/0.6 LUCPlanFLN. (Optional, for high-resolution imaging) 60x/1.0 LUMPLFLN water |
Riferimenti
- Foty, R. A simple hanging drop cell culture protocol for generation of 3d spheroids. J Vis Exp. (51), e2720 (2011).
- Moskovits, N., et al. Establishing 3-dimensional spheroids from patient-derived tumor samples and evaluating their sensitivity to drugs. J Vis Exp. (190), e64564 (2022).
- Griner, L. M., et al. Generation of high-throughput three-dimensional tumor spheroids for drug screening. J Vis Exp. (139), e57476 (2018).
- Qian, Y., Wei, X., Chen, K., Xu, M. Three-dimensional acoustic assembly device for mass manufacturing of cell spheroids. J Vis Exp. (200), e66078 (2023).
- He, H., et al. Dynamic formation of cellular aggregates of chondrocytes and mesenchymal stem cells in spinner flask. Cell Prolif. 52 (4), e12587 (2019).
- Perez, J. E., Nagle, I., Wilhelm, C. Magnetic molding of tumor spheroids: Emerging model for cancer screening. Biofabrication. 13 (1), 015018 (2020).
- Kingsley, D. M., et al. Laser-based 3d bioprinting for spatial and size control of tumor spheroids and embryoid bodies. Acta Biomater. 95, 357-370 (2019).
- Guillaume, O., et al. Hybrid spheroid microscaffolds as modular tissue units to build macro-tissue assemblies for tissue engineering. Acta Biomater. 165, 72-85 (2023).
- Danilevicius, P., et al. laser-made 3D microscaffolds for tissue spheroid encagement. Biointerphases. 10 (2), 021011 (2015).
- Jamieson, L. E., Harrison, D. J., Campbell, C. Chemical analysis of multicellular tumour spheroids. Analyst. 140 (12), 3910-3920 (2015).
- Dmitriev, R., Borisov, S., Jenkins, J., Papkovsky, D. Multiparametric imaging of tumor spheroids with ultra-bright and tunable nanoparticle O2 probes. SPIE BiOS. 9328, (2015).
- Zushin, P. -. J. H., Mukherjee, S., Wu, J. C. FDA Modernization Act 2.0: transitioning beyond animal models with human cells, organoids, and AI/ML-based approaches. J Clin Invest. 133 (21), e175824 (2023).
- Kim, D. -. S., et al. Robust enhancement of neural differentiation from human es and ips cells regardless of their innate difference in differentiation propensity. Stem Cell Rev Rep. 6 (2), 270-281 (2010).
- Hart, D., Gutiérrez, D. R., Biason-Lauber, A. Generation of a human ovarian granulosa cell model from induced pluripotent stem cells. bioRxiv. , 2022 (2022).
- Chiaradia, I., et al. Tissue morphology influences the temporal program of human brain organoid development. Cell Stem Cell. 30 (10), 1351-1367 (2023).
- Wagstaff, E. L., Ten Asbroek, A. L., Ten Brink, J. B., Jansonius, N. M., Bergen, A. A. An alternative approach to produce versatile retinal organoids with accelerated ganglion cell development. Sci Rep. 11 (1), 1101 (2021).
- Thavandiran, N., et al. Design and formulation of functional pluripotent stem cell-derived cardiac microtissues. Proc Natl Acad Sci U S A. 110 (49), E4698-E4707 (2013).
- Harrison, S. P., et al. Scalable production of tissue-like vascularized liver organoids from human pscs. Exp Mol Med. 55 (9), 2005-2024 (2023).
- Noguchi, T. -. A. K., Kurisaki, A. Formation of stomach tissue by organoid culture using mouse embryonic stem cells. Methods Mol Biol. 2017, 217-228 (2017).
- Takahashi, J., et al. Suspension culture in a rotating bioreactor for efficient generation of human intestinal organoids. Cell Reports Methods. 2 (11), 100337 (2022).
- Lagziel, S., Gottlieb, E., Shlomi, T. Mind your media. Nat Metab. 2 (12), 1369-1372 (2020).
- Das, V., Fürst, T., Gurská, S., Džubák, P., Hajdúch, M. Reproducibility of uniform spheroid formation in 384-well plates: The effect of medium evaporation. J Biomol Screen. 21 (9), 923-930 (2016).
- Bera, K., et al. Extracellular fluid viscosity enhances cell migration and cancer dissemination. Nature. 611 (7935), 365-373 (2022).
- Michl, J., Park, K. C., Swietach, P. Evidence-based guidelines for controlling ph in mammalian live-cell culture systems. Commun Biol. 2, 144 (2019).
- Okkelman, I. A., Vercruysse, C., Kondrashina, A. V., Borisov, S. M., Dmitriev, R. I. Affordable oxygen microscopy-assisted biofabrication of multicellular spheroids. J Vis Exp. (182), e63403 (2022).
- Peirsman, A., et al. Mispheroid: A knowledgebase and transparency tool for minimum information in spheroid identity. Nat Methods. 18 (11), 1294-1303 (2021).
- Raghavan, S., et al. Comparative analysis of tumor spheroid generation techniques for differential in vitro drug toxicity. Oncotarget. 7 (13), 16948 (2016).
- Froehlich, K., et al. Generation of multicellular breast cancer tumor spheroids: Comparison of different protocols. J Mammary Gland Biol Neoplasia. 21 (3-4), 89-98 (2016).
- Debruyne, A. C., et al. Live microscopy of multicellular spheroids with the multimodal near-infrared nanoparticles reveals differences in oxygenation gradients. ACS Nano. 18 (19), 12168-12186 (2024).
- Debruyne, A. C., Okkelman, I. A., Dmitriev, R. I. Balance between the cell viability and death in 3D. Semin Cell Dev Biol. 144, 55-66 (2023).
- Barroso, M., Monaghan, M. G., Niesner, R., Dmitriev, R. I. Probing organoid metabolism using fluorescence lifetime imaging microscopy (flim): The next frontier of drug discovery and disease understanding. Adv Drug Deliv Rev. 201, 115081 (2023).
- Becker, W. Fluorescence lifetime imaging-techniques and applications. J Microsc. 247 (2), 119-136 (2012).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. J Cell Sci. 134 (9), 1-17 (2021).
- Sarder, P., Maji, D., Achilefu, S. Molecular probes for fluorescence lifetime imaging. Bioconjug Chem. 26 (6), 963-974 (2015).
- Alfonso-Garcia, A., et al. Mesoscopic fluorescence lifetime imaging: Fundamental principles, clinical applications and future directions. J Biophotonics. 14 (6), e202000472 (2021).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: Fundamentals and advances in instrumentation, analysis, and applications. J Biomed Opt. 25 (7), 1-43 (2020).
- Yellen, G. Fueling thought: Management of glycolysis and oxidative phosphorylation in neuronal metabolism. J Cell Biol. 217 (7), 2235-2246 (2018).
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomark Med. 4 (2), 241-263 (2010).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of nad (p) h and fad. Antioxid Redox Signal. 30 (6), 875-889 (2019).
- Verma, A., et al. Fluorescence lifetime imaging for quantification of targeted drug delivery in varying tumor microenvironments. bioRxiv. , 2024 (2024).
- Smith, J. T., et al. In vivo quantitative fret small animal imaging: Intensity versus lifetime-based fret. Biophys Rep. 3 (2), 100110 (2023).
- Alvarez, L. A., et al. Application Note: SP8 Falcon: A novel concept in fluorescence lifetime imaging enabling video-rate confocal flim. Nat Methods. , (2019).
- Roberti, M. J., et al. TauSense: A fluorescence lifetime-based tool set for everyday imaging. Nat. Methods. , (2020).
- Auer, J. M. T., Murphy, L. C., Xiao, D., Li, D. U., Wheeler, A. P. Non-fitting flim-fret facilitates analysis of protein interactions in live zebrafish embryos. J Microsc. 291 (1), 43-56 (2023).
- Warren, S. C., et al. Rapid global fitting of large fluorescence lifetime imaging microscopy datasets. PLoS One. 8 (8), e70687 (2013).
- Gao, D., et al. Flimj: An open-source imagej toolkit for fluorescence lifetime image data analysis. PloS One. 15 (12), e0238327 (2020).
- Tullis, I. D. C., Ameer-Beg, S. M., Barber, P. R., Rankov, V., Vojnovic, B. Mapping femtosecond pulse front distortion and group velocity dispersion in multiphoton microscopy. Proc. SPIE 6089, Multiphoton Microscopy in the Biomedical Sciences VI. , 60890 (2006).
- Zhou, Y., et al. One-step derivation of functional mesenchymal stem cells from human pluripotent stem cells. Bio Protoc. 8 (22), e3080 (2018).
- Benton, G., Arnaoutova, I., George, J., Kleinman, H. K., Koblinski, J. Matrigel: From discovery and ECM mimicry to assays and models for cancer research. Adv Drug Deliv Rev. 79-80, 3-18 (2014).
- Badea, M. A., et al. Influence of matrigel on single-and multiple-spheroid cultures in breast cancer research. SLAS Discov. 24 (5), 563-578 (2019).
- Lang, S., Sharrard, R., Stark, M., Villette, J., Maitland, N. Prostate epithelial cell lines form spheroids with evidence of glandular differentiation in three-dimensional matrigel cultures. Br J Cancer. 85 (4), 590-599 (2001).
- Barra, J., et al. DMT1-dependent endosome-mitochondria interactions regulate mitochondrial iron translocation and metastatic outgrowth. Oncogene. 43 (9), 650-667 (2024).
- Dmitriev, R. I., Papkovsky, D. B. Intracellular probes for imaging oxygen concentration: How good are they. Methods Appl Fluoresc. 3 (3), 034001 (2015).
- Schweikhard, V., et al. Application Note: The power HyD family of detectors for confocal microscopy. Nat Methods. , (2020).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon flim microscopy. Sci Rep. 12 (1), 11938 (2022).
- Bush, P. G., Wokosin, D. L., Hall, A. C. Two-versus one photon excitation laser scanning microscopy: Critical importance of excitation wavelength. Front Biosci. 12, 2646-2657 (2007).
- Liu, M., et al. Instrument response standard in time-resolved fluorescence spectroscopy at visible wavelength: Quenched fluorescein sodium. Appl Spectrosc. 68 (5), 577-583 (2014).
- Szabelski, M., et al. Collisional quenching of erythrosine b as a potential reference dye for impulse response function evaluation. Appl Spectrosc. 63 (3), 363-368 (2009).
- Chib, R., et al. Standard reference for instrument response function in fluorescence lifetime measurements in visible and near infrared. Meas Sci Technol. 27 (2), 027001 (2015).
- Talbot, C. B., et al. Application of ultrafast gold luminescence to measuring the instrument response function for multispectral multiphoton fluorescence lifetime imaging. Opt Express. 19 (15), 13848-13861 (2011).
- Recording the instrument response function of a multiphoton flim system. Becker & Hickl Available from: https://www.becker-hickl.com/literature/application-notes/recording-the-instrument-response-function-of-a-multiphoton-flim-system/ (2007)
- Leben, R., Köhler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free nad (p) h fluorescence lifetime imaging. Int J Mol Sci. 20 (22), 5565 (2019).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using nadh and nadph autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Gottlieb, D., Asadipour, B., Kostina, P., Ung, T. P. L., Stringari, C. FLUTE: A python gui for interactive phasor analysis of flim data. Biol Imaging. 3, e21 (2023).
- Malacrida, L., Ranjit, S., Jameson, D. M., Gratton, E. The phasor plot: A universal circle to advance fluorescence lifetime analysis and interpretation. Annu Rev Biophys. 50, 575-593 (2021).
- Okkelman, I., Vandenberghe, W., Dmitriev, R. Role of preconditioning with oxygen and glucose deprivation in promoting differentiation of dental pulp stem cells in 3D culture. Mol Biol Cell. 34 (2), 1212 (2022).
- Park, J. H., et al. The effect of bmp-mimetic peptide tethering bioinks on the differentiation of dental pulp stem cells (DPSCs) in 3D bioprinted dental constructs. Biofabrication. 12 (3), 035029 (2020).
- Pașca, S. P. The rise of three-dimensional human brain cultures. Nature. 553 (7689), 437-445 (2018).
- Dmitriev, R. I., Zhdanov, A. V., Nolan, Y. M., Papkovsky, D. B. Imaging of neurosphere oxygenation with phosphorescent probes. Biomaterials. 34 (37), 9307-9317 (2013).
- Bryanskaya, E. O., et al. High levels of FAD autofluorescence indicate pathology preceding cell death. Biochim Biophys Acta Gen Subj. 1868 (1), 130520 (2024).
- Han, S. J., Kwon, S., Kim, K. S. Challenges of applying multicellular tumor spheroids in preclinical phase. Cancer Cell Int. 21 (1), 152 (2021).
- Okkelman, I. A., Neto, N., Papkovsky, D. B., Monaghan, M. G., Dmitriev, R. I. A deeper understanding of intestinal organoid metabolism revealed by combining fluorescence lifetime imaging microscopy (flim) and extracellular flux analyses. Redox Biol. 30, 101420 (2020).
- Gstraunthaler, G., Seppi, T., Pfaller, W. Impact of culture conditions, culture media volumes, and glucose content on metabolic properties of renal epithelial cell cultures: Are renal cells in tissue culture hypoxic. Cell Physiol Biochem. 9 (3), 150-172 (1999).
- Glickman, R. D. Phototoxicity to the retina: Mechanisms of damage. Int J Toxicol. 21 (6), 473-490 (2002).
- Golub, A. S., Pittman, R. N. Monitoring Parameters of Oxygen Transport to Cells in the Microcirculation. Quenched-Phosphorescence Detection of Molecular Oxygen: Applications in Life Sciences. , (2018).
- Nishigaki, T., Wood, C. D., Shiba, K., Baba, S. A., Darszon, A. Stroboscopic illumination using light-emitting diodes reduces phototoxicity in fluorescence cell imaging. Biotechniques. 41 (2), 191-197 (2006).
- Penjweini, R., Loew, H. G., Hamblin, M. R., Kratky, K. W. Long-term monitoring of live cell proliferation in presence of pvp-hypericin: A new strategy using ms pulses of led and the fluorescent dye cfse. J Microsc. 245 (1), 100-108 (2012).
- Carrasco Kind, M., et al. flimview: A software framework to handle, visualize and analyze flim data. F1000Research. 9, 574 (2020).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. Pam: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophys J. 114 (7), 1518-1528 (2018).
- Chen, S. -. J., Sinsuebphon, N., Barroso, M., Intes, X., Michalet, X. . Optical Molecular Probes, Imaging and Drug Delivery. , (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon