É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Produção e Microscopia de Imagem de Tempo de Vida de Fluorescência de Células Vivas Multiparâmetros (FLIM) de Esferoides Multicelulares
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, descrevemos diferentes métodos de formação de esferóides multicelulares para realizar microscopia de células vivas multiparâmetros de acompanhamento. Usando microscopia de imagem de fluorescência (FLIM), autofluorescência celular, corantes de coloração e nanopartículas, é demonstrada a abordagem para análise do metabolismo celular, hipóxia e morte celular em câncer tridimensional (3D) vivo e esferoides derivados de células-tronco.
Resumo
Os esferoides tumorais multicelulares são um modelo popular de microagregado de tecido 3D para reproduzir o microambiente tumoral, testar e otimizar terapias medicamentosas e usar bio e nanossensores em um contexto 3D. Sua facilidade de produção, tamanho previsível, crescimento e gradientes de nutrientes e metabólitos observados são importantes para recapitular o microambiente celular semelhante a um nicho 3D. No entanto, a heterogeneidade esferóide e a variabilidade de seus métodos de produção podem influenciar o metabolismo celular geral, a viabilidade e a resposta aos medicamentos. Isso dificulta a escolha da metodologia mais adequada, considerando os requisitos de tamanho, variabilidade, necessidades de biofabricação e uso como modelos de tecidos 3D in vitro em biologia de células-tronco e cânceres. Em particular, a produção de esferoides pode influenciar sua compatibilidade com microscopias quantitativas ao vivo, como imagens metabólicas ópticas, microscopia de imagem de fluorescência ao longo da vida (FLIM), monitoramento de hipóxia esferóide com nanossensores ou viabilidade. Aqui, uma série de protocolos convencionais de formação de esferóides são apresentados, destacando sua compatibilidade com as microscopias de campo amplo, confocais e de dois fótons ao vivo. A imagem de acompanhamento para análise pipeline com autofluorescência multiplexada FLIM e, usando vários tipos de câncer e esferóides de células-tronco, também é apresentada.
Introdução
Os esferoides multicelulares representam um grupo de modelos de tecidos 3D obtidos pela autoagregação de células e exibindo uma forma esférica. Eles são amplamente utilizados para imitar a interação célula-célula e célula-matriz in vitro e para reproduzir um contexto 3D dentro de uma infinidade de construções derivadas de câncer e células-tronco. Várias técnicas são empregadas para reduzir a ligação celular e promover a agregação. Isso inclui o método de queda suspensa com base na tensão superficial1; métodos de repelência de fixação celular, como placas de fixação ultrabaixa, micromoldes e micropoços 2,3; abordagem baseada em ondas acústicas4; métodos de agregação induzida por fluxo (frascos giratórios, biorreator e dispositivos microfluídicos)5; formação assistida por partículas magnéticas6 e uso de matrizes e andaimes sintéticos e baseados em ECM promotores de agregação 7,8,9.
Na pesquisa, desenvolvimento e validação de novas terapias medicamentosas contra o câncer, os esferoides são um modelo atraente devido à sua capacidade de recapitular os gradientes limitados por difusão espacial de nutrientes, resíduos eO2, muitas vezes levando à formação de um núcleo necrótico, típico dos tumores sólidos10,11. Esses modelos in vitro mais confiáveis e sofisticados desafiam a necessidade de uso extensivo de modelos animais (Food and Drug Administration [FDA] Modernization Act 2.012), de acordo com o princípio 3Rs da pesquisa animal (substituição, redução e refinamento). Além do câncer, os esferoides encontram sua aplicação na pesquisa com células-tronco. Por exemplo, as células-tronco pluripotentes têm a capacidade de formar corpos embrióides (EB), que podem ser usados para a diferenciação de células-tronco pluripotentes induzidas (iPSCs) em tipos de células especializadas que são difíceis de obter diretamente de pacientes, como células precursoras neurais13 ou células da granulosa ovariana 13,14. Além disso, a formação de um EB é frequentemente o primeiro passo no desenvolvimento de modelos organoides mais complexos, por exemplo, neural15, retinal16, cardíaco17, fígado18, estômago19 e organoides intestinais20. Fatores como tamanho, reprodutibilidade, rendimento e aplicações a jusante devem ser considerados ao escolher um método de formação de esferóides apropriado para os experimentos.
O aumento da complexidade da cultura 3D pode levar a uma maior variabilidade em comparação com a cultura 2D. Fatores como composição de nutrientes21, evaporação do meio22, viscosidade23, controle de pH24, método de formação de esferoides e até mesmo o tempo de cultura25,26 podem resultar na obtenção de esferoides de morfologia, tamanhos, viabilidade e quimiorresistência diferentes27,28. Pesquisas recentes demonstraram que os gradientes de oxigênio dos esferoides nem sempre são estáticos e são afetados pelo método de formação, tamanho do esferóide e viscosidade extracelular, afetando a heterogeneidade do esferóide29. Para melhorar a reprodutibilidade e a acessibilidade dos dados em esferoides, a base de conhecimento MISpheroID foi desenvolvida26, identificando linhagem celular, meio de cultura, método de formação e tamanho dos esferoides como as informações mínimas para um resultado reprodutível. Portanto, foi feita uma comparação detalhada de vários métodos de alto rendimento (SphericalPlate 5D, micromoldes feitos em laboratório e moldes de microtecido) e de baixa fixação (ou seja, placas de 96 poços revestidas com Biofloat e Lipidure, sem andaimes e baseadas em andaimes) (Figura 1 e Tabela 1), incluindo o tamanho do poço (dada uma estimativa do tamanho máximo do esferóide), consumíveis usados, tempo de preparação e a possibilidade de monitorar os esferoides sem transportá-los para placas de microscopia. Este último permite estudos de longo prazo, enquanto os esferoides produzidos com métodos de alto rendimento geralmente resultam em experimentos de endpoint. Todos os métodos, exceto as grades da Placa 5Dspheri, não trazem autofluorescência indesejada, possibilitando seu uso direto em microscopia.
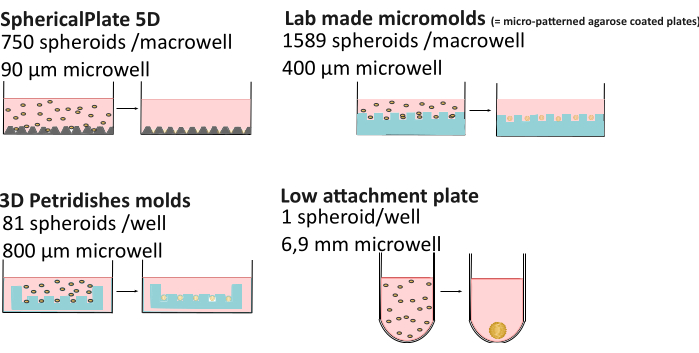
Figura 1: Métodos de formação de esferóides explicados. Métodos de alto rendimento, como o SphericalPlate 5D, que integrou micropoços patenteados na placa, enquanto os micromoldes produzidos em laboratório e os moldes MicroTissue usam carimbos para fazer vários micropoços em agarose (azul). Placas de baixa fixação, como Lipidure (Amsbio) e Biofloat (Sarstedt), usam um revestimento não aderente que inibe a adesão à superfície celular e promove a autoagregação celular. Clique aqui para ver uma versão maior desta figura.
| Placa 5D SpheriPlate | Micromoldes de produção própria | Microtecido | Métodos de fixação baixa | |
| Número de esferoides/poço | 750 | 1589 | 81 | 1 |
| Poço de diâmetro | 90 μm | 400 μm | 800 μm | 1 milímetro |
| Volume de cultura | 1 mL | 5 ml | 1 mL | 200 μL |
| Outros consumíveis | / | 7 mL de agarose a 3% | 500 μL de agarose a 2% | / * |
| Tempo de preparação | 10 minutos | 2 h + 3 dias de adaptação de mídia | 0,5 h + 15 min adaptação de mídia | 10–30 min + 1 h de secagem |
| Monitorização | Sim | Não** | Sim | Sim |
| Autofluorescente | Sim | Não | Não | Não |
| Reutilizável | Não | Sim | Sim | Não** |
| Custar | €€ | € | €€€€ | €€€€: Revestimento e Matrigel |
| €€: Placa comercial de 96 poços | ||||
| * Algumas linhagens celulares precisam de adição de ECM (ou seja, 2% a 5% de Matrigel) para formar esferóides compactos. | ||||
| **O revestimento é reutilizável até se esgotar. No entanto, cada placa consumirá uma pequena quantidade de mídia e a poeira pode se acumular com o tempo. A esterilização do filtro é necessária regularmente. | ||||
Tabela 1: Comparação de vários métodos de formação de esferóides29. "Monitorização": capacidade de monitorizar o esferóide sem necessidade de transferência para uma placa de microscopia. €: 0-50€, €€: 50-150€ , €€€: 150-500€ , €€€€: >500€
A microscopia de fluorescência permite o monitoramento direto dos principais aspectos biológicos dos esferoides, incluindo morte celular, viabilidade, proliferação, metabolismo, viscosidade e até propriedades mecânicas30. A microscopia de imagem de fluorescência ao longo da vida (FLIM) fornece uma dimensão quantitativa adicional para estudar as interações da sonda fluorescente dentro de seu (micro) ambiente31 , 32 , 33 , 34 , permitindo resolver os espectros de emissão sobrepostos de acordo com diferentes tempos de vida de emissão35 , 36 e sondar o metabolismo celular com base na autofluorescência celular intrínseca. Assim, compostos autofluorescentes celulares difundidos como nicotinamida adenina dinucleotídeo fosfato (NAD (P) H), mononucleotídeo de flavina (FMN), dinucleotídeo de flavina adenina (FAD), protoporfirina IX e outros podem ser medidos com FLIM de um e dois fótons e servem como 'sensores' intrínsecos do catabolismo da glicose, fosforilação oxidativa (OxPhos) e fornecem uma visão geral do estado redox celular. O NAD (P) H existe nas formas citoplasmáticas livres ou nas mitocôndrias ligadas a proteínas 37,38. Da mesma forma, o estado oxidado do FAD é fluorescente com uma vida útil mais longa da forma livre. As microscopias NAD(P)H e FAD geralmente envolvem FLIM excitado por dois fótons, com o objetivo de prevenir fotodanos à amostra39. Freqüentemente, o FLIM de 'imagem metabólica óptica' pode ser combinado com o uso de sondas à base de corantes, biossensores geneticamente codificados, microscopia de imagem de fosforescência ao longo da vida (PLIM) e medições baseadas em intensidade raciométrica, a fim de fornecer uma imagem mais completa do metabolismo esferóide ou organoide, oxigenação, proliferação e viabilidade celular29 , 30 , 31. Além disso, o FLIM também pode ser combinado com o método de transferência de energia de ressonância de Förster (FRET) para medir a variação ao longo da vida do fluoróforo do doador quando em contato próximo com o aceitador para investigar a ligação de um medicamento com seu domínio alvo 33,40,41.
As imagens FLIM adquiridas são normalmente analisadas para calcular o tempo de vida pixel a pixel. Atualmente, existem pelo menos 3 estratégias comuns usadas para obter o tempo de vida da fluorescência: 'FLIM rápido' semiquantitativo 42 (às vezes chamado de 'sentido tau' 43 , 44 ), ajuste da curva de decaimento, usando ajuste exponencial de um, dois ou três e abordagem 'sem ajuste' com transformação fasorial e análise de gráfico fasorial. Dependendo do fornecedor, o software fornecido (LAS X, Symphotime, SPCImage, etc.) ou de código aberto (por exemplo, FLIMfit45, FLIMJ46 ou outros47) pode ser usado para lidar com dados FLIM medidos. Normalmente, o software fornecido pelo fornecedor é útil para análise preliminar de dados, enquanto as soluções de código aberto podem fornecer estudos mais precisos usando, por exemplo, gráficos fasoriais e visualização 3D.
Apesar da utilidade e atratividade do FLIM como método para estudar esferoides, muito poucos protocolos experimentais estão disponíveis, e há uma falta geral de conhecimento na escolha do método de formação mais apropriado para experimentos bem-sucedidos de microscopia multiparamétrica ao vivo envolvendo FLIM. Aqui, uma comparação detalhada dos protocolos de formação de esferóides comumente usados é apresentada com base em sua morfologia, viabilidade e oxigenação com o nanossensor de detecção de oxigênio (MMIR1) recentemente validado e caracterizado em vermelho distante e infravermelho próximo (NIR). A nanopartícula catiônica é impregnada com dois corantes repórteres, o aza-BODIPY insensível ao Øde referência (excitação 650 nm, emissão 675 nm) e a metaloporfirina sensível ao NIR O2, PtTPTBPF (excitação 620 nm, emissão 760 nm). O MMIR1 permite a análise em tempo real de gradientes de oxigênio em um microscópio de fluorescência convencional (usando análise raciométrica) ou microscópio de fosforescência (PLIM) sem introduzir toxicidade celular e permitindo sinais estáveis, monitoramento de longo prazo e multiplexação25,29. Dependendo da necessidade de coloração com corantes ou nanossensores, rendimento de esferóides ou tipo de célula, o protocolo de formação mais adequado pode ser escolhido. Uma vez que os estudos de viabilidade e oxigenação de esferoides são relevantes para estudos de câncer e esferoides derivados de células-tronco, os protocolos apresentados também incluem exemplos e resultados típicos esperados de NAD(P)H-FLIM e FAD-FLIM com esses modelos. Os pipelines de imagem e análise apresentados têm como alvo as plataformas de microscopia FLIM baseadas em contagem de fótons únicos correlacionadas ao tempo mais populares.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Geração de esferóides multicelulares
- Cultura de células
NOTA: As culturas de células podem ser coletadas na American Type Culture Collection (ATCC), Lonza, Sigma-Aldrich ou outros fornecedores. O ATCC fornece todas as informações de manuseio necessárias, incluindo meios de crescimento preferidos, procedimentos de subcultura, nível de biossegurança, taxa de crescimento e perfis STR. Aqui, 500 células/esferóide da linha celular de câncer de cólon humano HCT116 são usadas no meio 5A de McCoy (VWR, 392-0420) suplementado com 10% de FBS e 1 mM de piruvato de sódio. Para experimentos de longo prazo que são monitorados diariamente, HEPES 10 mM, pH 7,2 pode ser adicionado ao meio.- Cultive a cultura de células para atingir 70% -90% de confluência.
- Enxaguar as células com PBS estéril pré-aquecido (37 °C) (5 ml para balão de 25 cm2 ou 10 ml por balão de 75 cm2 ).
- Adicione 0,05% de tripsina - 1 mM de solução de EDTA (0,5 mL por 25 cm² ou 1 mL por frasco de 75 cm2 ) e incube por 5-10 min a 37 ° C em 5% de CO2, 95% de umidade para atingir o descolamento celular.
NOTA: Controle o descolamento da célula sob o microscópio de luz de transmissão (campo claro). A superexposição das células à solução enzimática dissociante pode afetar sua viabilidade. - Neutralize a tripsina adicionando excesso de meio de cultura celular contendo 10% de FBS (pelo menos 5 mL de meio por 1 mL de solução de dissociação).
NOTA: Para células cultivadas em meios de cultura com baixo FBS ou sem FBS, a neutralização da tripsina pode ser feita com a adição de 0,5 mL de 100% de FBS à cultura de células tratadas com tripsina, seguida de centrifugação para transferir as células para seus meios de cultura originais. - Dissocie os agregados celulares por pipetagem para obter suspensão unicelular no meio.
NOTA: A pipetagem com uma pipeta sorológica com uma ponta de pipeta de 1000 μL na parte superior melhora muito a geração de suspensão de célula única em um grande volume de cultura de células em suspensão. - Use uma câmara de contagem (hemocitômetro com regras de Neubauer ou alternativas) para contar o número de células por 1 mL da suspensão celular.
- Diluir a suspensão celular para obter o número desejado de células por mililitro.
- Adicionar solução concentrada de sonda de O2 (nanopartículas) à suspensão celular a uma concentração final de 10 μg/ml para análise raciométrica de O2.
NOTA: Para garantir uma suspensão celular homogênea (com a sonda), ressuspenda várias vezes antes da formação do esferoide. Se a sonda O2 não for necessária, pule esta etapa e prossiga com a formação dos esferoides. Um protocolo modificado deve ser aplicado ao manusear iPSCs. Resumidamente, as iPSCs são cultivadas em colônias em placas revestidas com Geltrex e passadas usando ReLeSR conforme descrito no protocolo48 fornecido pelo fornecedor. No dia da formação dos esferóides, as colônias devem ser grandes, compactas e exibir centros de várias camadas com bordas distintas. Enxágue as células com PBS estéril pré-aquecido. Adicione 1 mL de reagente de dissociação celular suave (GCDR) e incube a 37 °C por 8-10 min. Use uma ponta de 1000 μL para separar suavemente as células do poço e obter uma suspensão unicelular. Transfira a suspensão de célula única para um tubo cônico estéril de 50 mL e adicione 4 mL de meio DMEM-F12 pré-aquecido para neutralizar o GCDR. Lave o poço com 1 mL de DMEM-F12 e transfira-o para o resto da suspensão celular. Centrifugue a 300 x g por 5 min. Ressuspenda em 1 mL de meio apropriado para experimentos adicionais. Para os experimentos descritos neste manuscrito, foi utilizado mTeSR + 10 μM Rock Inhibitor. Conte e dilua a suspensão celular para obter o número desejado de células por mililitro.
- Métodos de formação de esferóides
- Micro-moldes de placa de Petri 3D
NOTA: Este método de alto rendimento é usado para gerar simultaneamente um grande número de esferoides (81 esferoides) em uma matriz de micromoldes 9 x 9 com 800 μm de diâmetro e 800 μm de profundidade.- Enxágue os micro-moldes para fundição de placas de Petri 3D em dH2O e coloque em recipiente autoclavável.
- Meça 2 g de pó de agarose grau eletroforese e coloque-o em um frasco de vidro seco de 200 mL seguro para autoclave.
NOTA: Certifique-se de que a garrafa e o pó de agarose estejam secos, sem líquido ou umidade. - Micro-moldes de autoclave para fundição de placas e garrafas de Petri 3D com pó de agarose por 30 min em ciclo seco.
- Faça uma solução salina a 0,9 p/v% por adição de 0,9 g de NaCl em 100 mL de água ultrapura e esterilize em autoclavagem.
NOTA: O NaCl é recomendado pelo fabricante. Aumenta a estabilidade da agarose. - Prepare a solução de agarose adicionando a solução salina estéril ao pó de agarose esterilizado. Aparafuse a tampa frouxamente encaixada para evitar o acúmulo de pressão. Gire a garrafa para misturar o pó de agarose.
- Ferva e dissolva o pó de agarose usando um forno de micro-ondas. Pare o micro-ondas com frequência (~a cada 10 s). Agite o frasco e repita até que a agarose se dissolva.
CUIDADO: A solução de agarose está quente e requer manuseio cuidadoso. Agitar imediatamente após o processo de fusão pode fazer com que a solução estoure do recipiente. Para evitar acidentes, use recipientes suficientemente grandes cheios até 50% da capacidade e use proteção individual adequada (luvas, luva de forno, proteção para os olhos e jaleco). - Deixe a solução de agarose dissolvida esfriar até 60-70 °C. Usando técnicas e condições assépticas, pipete a agarose fundida no micromolde (500 μL para a série 12 ou 330 μL para a série 24).
NOTA: Evite a formação de bolhas ao misturar ou pipetar agarose. Remova quaisquer pequenas bolhas presas nas pequenas características do micromolde pipetando ou raspando suavemente antes que a agarose solidifique. - Deixe a agarose solidificar por cerca de 2-3 min. Em seguida, flexione cuidadosamente o micromolde para remover a placa de Petri 3D e transfira para uma placa de cultura de tecidos de 12 poços.
NOTA: A flexão excessiva do micromolde pode levar à formação de rachaduras dentro do molde de agarose. - Para equilibrar a placa de Petri 3D, adicione 2,5 mL/meio de cultura de células de poço. Incube por 15 min ou mais. Remova o meio de cultura e substitua-o por meio novo. Repita mais uma vez para equilibrar a placa de Petri 3D com um meio de cultura.
NOTA: O protocolo pode ser interrompido aqui até a semeadura celular. Para armazenamento de longo prazo (até 2 semanas a 4 °C), use a solução PBS em vez de média. - Remova completamente a placa de Petri 3D ao redor do meio de cultura (ou PBS) e remova com precisão o meio dentro da placa de Petri 3D inclinando a placa de cultura de tecidos.
- Semeie cuidadosamente 190 μL de suspensão celular contendo 40.500 células gota a gota na câmara de semeadura celular (consulte a etapa 1.1).
NOTA: O número de micro-poços em selos de agarose determina o número de esferóides produzidos por selo. Neste caso, este molde de agarose contém 81 micropoços (81 x 500 células/esferóide). A variação da concentração de células em suspensão adicionada a um macropoço permite alterar o número de células por esferoide, controlando assim o tamanho do esferoide. - Aguarde ~ 10 minutos para que as células se estabeleçam nos recursos do DSH de Petri 3D. Em seguida, adicione 2 mL de meio na parte externa da placa de Petri 3D.
- Coloque a placa de cultura de tecidos na incubadora de cultura de células e troque o meio ao redor da placa de Petri 3D conforme necessário.
- Placa de fixação baixa
NOTA: Este método é usado para gerar um único esferóide por poço. Placas revestidas (Lipidure ou Biofloat) estão disponíveis comercialmente (pule as etapas 1.2.2.1-1.2.2.4). Alternativamente, o revestimento pode ser adquirido separadamente e usado para revestir placas de vários poços não tratadas. Recomenda-se encher os poços nas bordas das placas de 96 poços com água estéril ou PBS devido à evaporação mais rápida nesses poços, limitando assim o número de poços para esferoides a 60. Caso sejam necessários menos esferoides, encha os poços vazios ao redor com água ou PBS. Use pontas sem poeira para manuseio de líquidos para evitar trazer pequenas partículas para os poços, pois elas interferem na formação de esferoides.- Preparar uma solução de revestimento a 0,5 p/v% dissolvendo 0,25 g do pó de polímero em 50 ml de etanol num recipiente de vidro em garrafa. Filtre o revestimento com filtro.
NOTA: A esterilização do filtro e todas as próximas etapas devem ser realizadas em condições estéreis sob fluxo laminar. - Adicione 200 μL da solução de revestimento a cada poço de uma placa de cultura de fundo em U 96.
- Incube por 1 min e retire o excesso de revestimento.
NOTA: A solução de revestimento pode ser usada várias vezes. Armazene em um recipiente de vidro em temperatura ambiente (RT). Recipientes de plástico não são recomendados, pois o plástico pode se dissolver parcialmente e se tornar parte da solução. Se houver poeira, esterilize o filtro com filtros de polietersulfona (PES) ou seringa de náilon de 0.22 μm. - Deixe a placa de 96 poços secar ao ar por cerca de 1 h.
NOTA: Se as células precisarem de uma matriz extracelular, prossiga para a etapa 1.2.3. As placas revestidas podem ser armazenadas no RT quando embrulhadas em alumínio por até 1 mês. Ao semear uma quantidade menor de células/poço, centrifugar a placa por 5 min a 300 g pode ajudar a puxar as células para baixo.
- Preparar uma solução de revestimento a 0,5 p/v% dissolvendo 0,25 g do pó de polímero em 50 ml de etanol num recipiente de vidro em garrafa. Filtre o revestimento com filtro.
- Protocolo de formação auxiliada por matriz extracelular
NOTA: Algumas linhagens celulares não produzem matriz extracelular (MEC) suficiente e precisam da adição de ECM, como Matrigel, Cultrex ou Geltrex, para formar esferóides compactos 49,50,51. Para tipos de células como câncer de mama MDA-MB-231, papila dérmica humana, células de câncer de próstata e outras, é possível usar a etapa 1.2.2 com as seguintes modificações52, exigindo a adição de MEC. De preferência, use pontas sem poeira para manuseio de líquidos para evitar que a poeira interfira na formação do esferoide. As etapas 1 e 4-7 devem ser executadas sob uma cabine de segurança biológica (classe II).- Prossiga com as etapas 1.2.2.1-1.2.2.4 (placas de fixação baixas) para tratamento de superfície.
- Pré-resfrie a placa de 96 poços a 4 °C na geladeira.
- Prepare a centrífuga com o adaptador correto para a placa de 96 poços e pré-ajuste para 4 °C.
- Preparar uma solução a 5% de matriz de membrana basal (BMM) em meios de cultura de células pré-arrefecidos (4 °C).
NOTA: O BMM reticula rapidamente em RT. Ao manusear, mantenha os estoques e as soluções no gelo. - Prepare a suspensão celular (consulte a etapa 1.1).
- Adicione 50 μL da solução BMM em cada poço.
- Adicione suavemente 50 μL de suspensão celular em cada poço em cima da solução BMM (25.000 células/poço).
NOTA: Não exploda este volume no poço, ou então as células se espalharão nas laterais dos poços e não se acumularão no fundo. Um número menor de células por poço pode ser obtido ajustando a densidade celular de acordo. Nem todas as células formarão esferóides em todas as densidades de semeadura; A otimização deve ser realizada por tipo de célula e dimensão desejada. Com os volumes fornecidos, a concentração final de BMM é de 2,5%. Se for necessária uma concentração diferente, a solução-mãe deve ser preparada a uma concentração mais baixa/mais elevada. - Centrifugue a placa de 96 poços por 5 min a 300 x g e 4 °C.
NOTA: Sem esta etapa, as células não se agregam adequadamente no fundo do poço, causando a formação de vários agregados menores. A centrífuga deve ser resfriada para evitar reticulação nesta fase. - Coloque a placa na incubadora de cultura de células. Os agregados são considerados maduros no dia 4 após a semeadura.
NOTA: Para protocolos adicionais sobre a formação de esferoides, consulte o Arquivo Suplementar 1.
- Micro-moldes de placa de Petri 3D
2. Microscopia ao vivo de esferóides
- Preparação de esferoides para análise de imagens ao vivo
NOTA: Dependendo do desenho do experimento (por exemplo, monitoramento de longo prazo ou análise de ponto final, configuração do microscópio ou propriedades espectrais da fluorescência medida) ou devido à incompatibilidade do método de produção de esferoides com a microscopia (por exemplo, espessura da amostra, autofluorescência do material, flutuação de esferoides durante a imagem) monitoramento direto de esferoides na placa, onde foram produzidos, pode não ser possível. O protocolo explica a preparação de esferoides para imagens, que são adequados para a maioria dos microscópios de campo amplo invertido e confocais.- Preparar e pré-aquecer (37 °C) os meios de imagem: DMEM suplementado com HEPES-Na, pH 7,2 (10 mM), piruvato de sódio (1 mM), L-glutamina (2 mM) e glicose (5 mM), sem vermelho de fenol.
NOTA: O bicarbonato de sódio sozinho ou em combinação com HEPES-Na pode ser usado, se o controle de CO2 for fornecido durante a imagem24. Alguns tipos de cultura de células não toleram a presença de HEPES. Dependendo do desenho experimental, o teor de piruvato, glutamina e glicose pode ser modificado. - Prepare placas de microscopia estéreis (disponíveis comercialmente ou feitas em laboratório) com superfícies de vidro de cobertura revestidas (para uma forte adesão esferoidal) ou não revestidas (baixa adesão esferoidal) (espessura # 1,5).
NOTA: A necessidade e o tipo de revestimento dependem do tipo de célula, das propriedades de adesão dos esferoides e da taxa de migração celular da interface de cultura 3D para 2D. É importante considerar isso, pois o revestimento pode facilitar a perda de organização 3D, alterando a forma dos microgradientes nos esferoides e, como resultado, o comportamento celular. Para alguns experimentos (por exemplo, imagens analisadas a resposta à estimulação medicamentosa), é necessária uma forte aderência esferoide à superfície e é preferível um revestimento com gelatina, BMM, colágeno, colágeno / poli-D-lisina ou poli-D-lisina. - Lave suavemente os esferóides corados com sonda de O2 dos micropoços da agarose micropadronizada ou da placa de 96 poços e transfira os esferóides ainda flutuantes para um frasco de 2 mL.
NOTA: Para garantir a coleta de todos os esferóides do método de alto rendimento, enxágue o molde 1-3 vezes com o volume adicional de meio de cultura, combinando todas as suspensões de esferóides em um frasco. Para esferóides em uma placa de fixação baixa, colete esferóides um por um de poços individuais para um frasco ou diretamente para uma placa de microscopia se um pequeno número de esferóides for suficiente para o experimento. Ao transferir esferoides grandes, corte a extremidade da ponta da pipeta para garantir que não haja danos durante a pipetagem. - Deixe o frasco na posição vertical por até 5 min para permitir que os esferóides se depositem no fundo do frasco, formando um pellet visível.
- Remova o meio do tubo, deixando os esferóides intactos, e ressuspenda-os suavemente em uma quantidade suficiente de cultura fresca.
NOTA: Para facilitar o manuseio de esferoides, a transferência de esferoides para o meio de imagem também pode ser feita nesta fase em pequenos lotes; Consulte as etapas 2.1.7 e 2.1.8. Os esferoides dos diferentes grupos experimentais devem ser tratados igualmente, pois a composição do meio e o tempo de pré-condicionamento do meio podem afetar seu metabolismo. - Enquanto os esferóides estiverem flutuando, transfira um volume igual de suspensão de esferóides por poço da placa de microscopia.
- Incubar os esferóides durante 1-2 h a 37 °C numa incubadora de CO2 para garantir a sua fixação à superfície da placa de microscopia/poço. Para obter imagens, prossiga com a etapa 2.1.9. Para coloração de esferóides com sondas adicionais, prossiga com a etapa 2.1.8.
NOTA: A taxa de migração celular do esferóide 3D para a interface de superfície 2D é uma função do tempo. Para evitar a perda de organização 3D, o tempo de incubação deve ser otimizado em relação ao tipo de célula, tipo de revestimento da superfície e ao design do experimento. Por exemplo, o HCT116, dependendo do tamanho do esferoide, precisa de pelo menos 2 h para uma fixação adequada do esferóide à superfície revestida de colágeno IV/poli-D-lisina, enquanto a fixação do hDPSC e a migração para a interface 2D são extremamente rápidas, levando à perda de organização 3D em 1-2 h. Para evitar a geração de imagens de 'esferóides 2D' devido ao espalhamento excessivo, superfícies de vidro não revestidas são usadas com o tempo de incubação reduzido. - Adicione sonda(s) de fluorescência em concentrações recomendadas ou empiricamente otimizadas a um volume conhecido de suspensão esferoide. Incubar durante 1 h a 37 °C CO2 incubadora antes da obtenção de imagens.
NOTA: Para ensaio vivo / morto, use iodeto de propídio e Calceína Green-AM em uma concentração final padrão de 1 μg / mL. Para evitar o efeito tóxico da coloração com iodeto de propídio nos corpos embrióides iPSC, a concentração final de iodeto de propídio foi de 0,5 μg/mL. O tempo de carregamento da sonda pode ser prolongado se a difusão da sonda não for eficiente devido ao grande tamanho do esferóide. O tempo de carregamento deve sempre ser considerado como parte do tempo total de incubação necessário para a fixação do esferóide à superfície. Se for necessário mais tempo para a fixação do esferóide, o procedimento de coloração deve ser organizado no final desse período. Esteja ciente de que um tempo de incubação mais longo pode levar à perda da organização 3D. - Remova os meios de cultura de células ou meios contendo sondas fluorescentes e troque-os por um volume necessário de meios de imagem. Para garantir que nenhum fundo de fluorescência do meio esteja no caminho da imagem esferoide, repita a etapa de troca de meio (lavagem) até 5 vezes.
NOTA: Para evitar a remoção do esferóide durante a troca de meios, recomenda-se aspirar cuidadosamente o meio com uma pipeta de 200 μL das bordas das placas de microscopia e realizar a adição de mídia pela parede ou lateralmente na placa de microscopia. - Prossiga imediatamente com a etapa 2.2.1 do protocolo de imagem.
NOTA: Um intervalo muito longo entre a preparação para a imagem e a aquisição real da imagem pode influenciar o metabolismo celular (por exemplo, por meio da composição alterada do meio), a viabilidade (algumas sondas fluorescentes usadas para análise de endpoint têm efeitos tóxicos, o que pode estimular a morte celular após um longo período de incubação), bem como levar a uma perda de organização 3D. Se vários grupos de esferoides ou condições experimentais devem ser comparados, o projeto experimental deve ser desenvolvido de acordo para manter o tempo do tratamento, pré-condicionamento e procedimentos de imagem o mais iguais possível entre os grupos analisados.
- Preparar e pré-aquecer (37 °C) os meios de imagem: DMEM suplementado com HEPES-Na, pH 7,2 (10 mM), piruvato de sódio (1 mM), L-glutamina (2 mM) e glicose (5 mM), sem vermelho de fenol.
- Aquisição de imagem
NOTA: O protocolo descreve a imagem multiparamétrica de esferoides vivos usando o microscópio confocal Stellaris 8 Falcon (Leica) e o software Leica Application Suite X (LAS X) versão 4.7. No entanto, apenas pequenas modificações serão necessárias para realizar tal análise em plataformas de microscopia alternativas.- Ligue a unidade de controle climático de temperatura 30-60 minutos antes da imagem. Defina a velocidade de ventilação e temperatura necessárias (35-37 °C).
NOTA: Se, além do controle de temperatura, a concentração de gás (por exemplo, CO2 ou O2) precisar ser controlada durante a imagem, os dispositivos correspondentes também devem ser iniciados com antecedência para atingir as condições necessárias antes da imagem. - Ligue o microscópio e os dispositivos conectados (ou seja, laser WLL, computador, bomba de água para a objetiva de imersão em água e outros blocos eletrônicos operacionais). Inicie o software de controle do microscópio (por exemplo, LAS X Machine Mode ou Machine Mode with Environmental Control) fornecido com a configuração exata do microscópio e inicialize a calibração da platina.
- Escolha a objetiva necessária no software e aplique o fluido de imersão, se necessário.
NOTA: Para microscopia ao vivo, recomenda-se o uso de objetivas de imersão em água ou glicerol com distância de trabalho suficiente ('longa'), por exemplo, HC Fluotar L 25x/0.95 W VIS IR (distância de trabalho de 2.4 mm), HC PL Apo 40x/1.25 GLYC (distância de trabalho de 0.35 mm) ou pelo menos NA = 0.4 ou superior para as objetivas de ar. A escolha da ampliação e da distância de trabalho depende da natureza e do tamanho da amostra fotografada e dos sinais de fluorescência medidos (brilho, rendimento quântico, eficiência de coloração, ver, por exemplo, discussão sobre corantes e nanopartículas53). Objetos grandes (esferoides ou organoides, tamanho de >500 μm), 'biorreator' ou chips microfluídicos requerem objetivas de longa distância de trabalho e menor ampliação, enquanto a análise de células individuais ou organelas celulares requer alta ampliação, muitas vezes obtida por meio de imagens em 'mosaico'. - Configure a placa de microscopia com esferóides no palco. Ajuste o foco e encontre um objeto/região de interesse (ROI).
NOTA: Se objetos pequenos, fracamente fluorescentes, de baixo contraste ou raros precisarem ser encontrados e for difícil encontrar o foco, recomenda-se pré-focar nas paredes da placa de microscopia e 'filtrar' a superfície para o objeto de interesse por serpentina, começando por um dos cantos do poço. - Escolha a janela Abrir projeto e clique no ícone correspondente, Criar um novo projeto. Dê um nome padrão (por exemplo, começando com "descrição YY-MM-DD+") para o arquivo do projeto de pesquisa. Durante a geração de imagens, todas as imagens produzidas serão salvas automaticamente no arquivo .lif do projeto criado.
- Abra a janela Aquisição . Defina o comprimento de onda de excitação do laser de luz branca (WLL) e a faixa necessária de detectores híbridos ou de varredura de ressonância (tipo HyD S, HyD X ou HyD R) com base nas propriedades espectrais conhecidas da fluorescência medida (espectros de excitação/absorbância e emissão). Escolha os tipos de digitalização Linha ou Quadro .
NOTA: Para a maioria dos corantes fluorescentes disponíveis comercialmente, as propriedades espectrais podem ser encontradas (ou adicionadas) no pacote LAS X Dye Assistant. Escolha o detector com a faixa de sensibilidade de espectro apropriada compatível com as propriedades espectrais da sonda e, no caso do FLIM, compatível com a contagem de fótons (ou seja, HyD X ou HyD R). Para imagens multiparamétricas, defina o WLL em várias posições de excitação (por exemplo, para imagens simultâneas de FAD/Flavinas e dois canais de fluorescência da sonda MMIR1 O2 raciométrica - referência e sensível, as configurações de excitação/emissão podem ser 460 nm/510-590 nm HyD X1 e 614 nm/631-690 HyD X3 e 724-800 nm HyD R de acordo com uma ou duas sequências de varredura sequencial). É importante atribuir o detector apropriado para coletar a emissão, pois os detectores podem ter sensibilidade espectral diferente54. - (Opcional para FLIM) Na janela Aquisição , escolha o modo FLIM para realizar imagens combinadas com a contagem de fótons (coleta de decaimento). Imediatamente, um 'módulo FLIM adicional no software LAS X' será aberto para navegar e analisar os dados FLIM.
- (Opcional para FLIM) Escolha a taxa de repetição de pulso WLL com base na vida útil média esperada do fluoróforo.
NOTA: A frequência do pulso do laser deve ser ajustada para coletar o decaimento total da fluorescência. Isso pode ser feito usando um recurso Pulse Picker instalado no microscópio. A sobreposição do decaimento da fluorescência com o pulso do laser levará ao encurtamento da vida útil estimada da fluorescência. Recomenda-se ter intervalos de pulso de 4 a 5 vezes mais longos do que a vida útil média esperada da fluorescência (por exemplo, 25 ns/40 MHz para vida útil de até 5 ns). Muitos lasers pulsados têm uma taxa de repetição fixa de 80 MHz (ideal apenas para uma faixa de até 2-3 ns). Isso é importante para escolher os fluoróforos corretos para o experimento. - Inicie a imagem de visualização usando o modo FAST LIVE e ajuste o foco fino do objeto de imagem em uma seção de interesse.
CUIDADO: Siga rigorosamente as regras de segurança do laser. Sempre considere as regras de segurança do laser e espere até que a imagem pare antes de ligar a luz de transmissão, olhar para a ocular ou para a amostra.
NOTA: No modo FAST LIVE , uma varredura de alta velocidade de 600 Hz (corresponde a uma taxa de quadros máxima de 4,43/s se o modo de varredura X bidirecional for usado), resolução de 256 x 256 pixels é aplicada automaticamente à imagem para manter a fluorescência protegida contra fotobranqueamento. Abra o orifício (por exemplo, para 3-4 UA) e/ou aumente a intensidade do laser se o sinal de fluorescência for muito fraco para focar no objeto. Evite a coleta de decomposição incompleta. - (Opcional para FLIM) Ao observar um histograma de intensidade de pixel que aparece durante a imagem em uma janela do módulo FLIM (modo Live ), ajuste a intensidade do laser/tamanho e a resolução do orifício apropriados para atingir a taxa de contagem ~ 1 limite de pulso de fóton/laser (linha vermelha). Evite ir significativamente acima de 1 para excluir o risco de efeito de empilhamento. Se necessário, ajuste a taxa de repetição de pulso WLL para ter uma coleção de decaimento completa em uma janela de decaimento (para evitar a coleta de decaimento incompleta, consulte a etapa 2.2.8).
NOTA: Se o número de fótons (intensidade) não for suficiente para reconstruir um decaimento confiável para análise de ajuste ou nuvem de gráfico fasorial, aplique várias repetições de varredura (quadros ou linhas, ou configure o tempo de varredura), aumente a intensidade do laser e/ou sacrifique a resolução (tamanho do ROI digitalizado). Esteja ciente de que diminuir a taxa de repetição do laser requer que mais fótons sejam coletados para uma reconstrução de decaimento confiável, e uma correção adicional dos parâmetros de imagem pode ser necessária. Esteja ciente do impacto potencial da luz intensa e da iluminação contínua longa na viabilidade e metabolismo celular55. O impacto negativo na viabilidade e no metabolismo pode ser diferente em cada caso individual, dependendo da intensidade, duração e comprimento de onda da luz de excitação, bem como da modalidade de imagem (por exemplo, imagem confocal de um fóton vs. multifóton). Ajustar os parâmetros de imagem de acordo e, se necessário, controlar a viabilidade/morte celular por Calceína Green-AM ou intensidade de iodeto de propídio em experimentos piloto56. Sempre que possível, otimizações adicionais do protocolo de coloração da sonda de fluorescência devem ser consideradas para atingir um sinal de fluorescência adequado durante a microscopia ao vivo. - (Opcional para pilha z 3D) Enquanto estiver no Fast Live , defina as coordenadas, a direção da varredura e atribua-as a Início e Fim na janela Z-stack (Modo de varredura XYZ). Escolha o tamanho do passo Z ou o número de passos.
NOTA: Embora o software calcule automaticamente o número "ideal" de etapas, com base na resolução usada e nos parâmetros de digitalização, a reconstrução 3D ao vivo normalmente pode exigir um número menor de etapas para obter uma aquisição rápida, por exemplo, tamanho de passo de 1-2 μm, tamanho de pilha de 50-100 μm, varredura bidirecional, exigindo 2-3 min do tempo total de varredura. Esteja ciente de que as organelas subcelulares, células e o modelo de célula 3D também podem se mover durante as medições. Além disso, devido à profundidade de penetração da luz e aos limites de dispersão, normalmente apenas 50-100 μm de profundidade de varredura no FLIM confocal podem ser alcançados. - Quando todas as configurações necessárias forem aplicadas, comece a criar imagens.
- Dê um nome apropriado à imagem.
- Procure o próximo objeto de imagem no modo de luz de transmissão e repita o procedimento de imagem com configurações de imagem previamente otimizadas (etapas 2.2.8 a 2.2.12).
NOTA: Para a comparação baseada em intensidade ou análise de razão de intensidade (por exemplo, análise de oxigenação baseada em sonda MMIR O2), sempre mantenha as mesmas configurações de imagem para todos os objetos analisados (ampliação e tipo de lente objetiva, intensidade do laser, potência e frequência de pulso, comprimento de onda de excitação, alcance dos detectores, orifício, velocidade de varredura, tempo de permanência e resolução de pixels). No entanto, como o tempo de vida da fluorescência não depende da intensidade da fluorescência e requer que um número apropriado de fótons seja coletado para um cálculo confiável, os parâmetros de imagem FLIM podem ser reajustados ao longo do experimento para manter os números de fótons coletados comparáveis entre diferentes tratamentos ou condições experimentais. Portanto, para análises multiparamétricas em que a análise baseada em intensidade e fluorescência é necessária, configurações de imagem universais otimizadas devem ser aplicadas para todos os objetos nos grupos experimentais comparados. Para comparação "somente FLIM", é possível comparar imagens adquiridas com configurações de imagem ligeiramente diferentes, pois o software LAS X fornece cálculo do IRF para medição de imagem individual42. No entanto, para análise de ajuste FLIM fora do LAS X (por exemplo, FLIMfit45), a função de resposta do instrumento (IRF) deve ser medida para cada condição de imagem diferente, pois não pode ser exportada do software de imagem. Assim, para a simplicidade do projeto experimental e da carga de trabalho, é recomendável aplicar as mesmas configurações de imagem para todas as imagens no conjunto de dados. Em seguida, as medições IRF correspondentes podem ser feitas com o uso de fluoróforos de tempo de vida de fluorescência extintos ou de fluorescência rápida (dentro da faixa de ps) com as propriedades de emissão do canal espectral medido 57,58,59, por luminescência de nanopartículas de ouro 60 ou por sinal de geração de segundo harmônico para FLIM multifóton61. No software LAS X, os parâmetros de imagem previamente otimizados podem ser carregados para um novo projeto clicando com o botão direito do mouse no arquivo de interesse e escolhendo Aplicar configurações de imagem. - Quando a sessão de imagem for finalizada, salve o projeto de imagem. Para finalizar a sessão de imagem, remova a amostra do estágio de microscopia e limpe a objetiva do líquido de imersão (se usado) de acordo com o procedimento padrão implementado na instalação de imagem. Feche o projeto e o software. Desligue o microscópio, os lasers e todos os dispositivos conectados.
- Prossiga com a análise dos dados de imagem (etapa 2.3).
- Ligue a unidade de controle climático de temperatura 30-60 minutos antes da imagem. Defina a velocidade de ventilação e temperatura necessárias (35-37 °C).
- Processamento de imagem fasorial NAD (P) H / FAD-FLIM com módulo LAS X FLIM e FIJI
NOTA: O protocolo descreve uma análise do tempo de vida de fluorescência de esferoides imageados para dados no domínio da frequência em exemplos de NAD(P)H e FAD/Flavins autofluorescência FLIM. A medição da autofluorescência NAD(P)H tornou-se um padrão-ouro para análise metabólica, onde componentes curtos e mais longos da vida útil da autofluorescência NAD(P)H estão associados à glicólise ou fosforilação oxidativa (OxPhos), respectivamente. Isso pode ser analisado pela mudança em um gráfico fasorial ao longo da trajetória metabólica em direção aos padrões medidos de NAD (P) H livre ou NAD (P) H ligado a proteínas31,62. Para analisar a trajetória do deslocamento metabólico, bem como para comparar a posição das nuvens fasoriais (ver a NOTA abaixo do passo 2.3.6) em um gráfico entre grupos experimentais, foi implementada uma análise simplificada das coordenadas fasoriais, com base no cálculo do centro geométrico (centróide) da nuvem fasorial29. O protocolo descrito demonstra o cálculo das coordenadas do centróide em FIJI e a medição da distância entre dois pontos em um gráfico fasorial usando coordenadas (por exemplo, a distância de um centróide da nuvem fasorial de autofluorescência NAD (P) H esferóide a um ponto teórico de "NAD (P) H livre"). Da mesma forma, FAD e outros sinais de autofluorescência podem ser analisados. Um conjunto de dados 1 com .lif (Software LAS X necessário) ou .ptu Formatos de arquivo para aprender este procedimento é fornecido (Processo suplementar 2, Processo suplementar 3, Processo suplementar 4, Processo suplementar 5, Processo suplementar 6, Processo suplementar 7, Processo suplementar 8, Processo suplementar 9, e Arquivo Suplementar 10).- Abra o módulo FLIM no LAS X, selecione Abrir projeto e carregue o arquivo de imagem esferóide (.lif) para análise de NAD(P)H/FAD de autofluorescência.
NOTA: Devido a possíveis bugs e perda de dados intermediária no módulo FLIM, use uma cópia do arquivo de imagem esferóide original (.lif) para análise NAD(P)H/FAD, mantendo o arquivo bruto inalterado. - Selecione uma única imagem e navegue até a interface de análise FLIM. Clique em Fasorial para acessar o gráfico fasorial e ativar o modo de análise fasorial. Aplique o filtro (Mediana ou Wavelet) e defina o Limite para minimizar o ruído e melhorar a visibilidade dos dados para todas as análises fasoriais. Escolha os harmônicos. Para análise da mudança metabólica com base nos dados de NAD(P)H, prossiga com as etapas 2.3.3-2.3.5. Para a comparação geral dos gráficos fasoriais, prossiga a partir da etapa 2.3.6.
NOTA: Aplique as configurações de análise (tipo de filtro, harmônicos, limite, binning e ROIs fasoriais) igualmente a todas as imagens em um conjunto de dados comparado. - (Opcional para análise NAD(P)H) Escolha qualquer imagem relacionada ao conjunto de dados e use a opção Cursor de taxa de desenho para dois componentes para localizar com precisão a posição de 0,45 ns em um círculo universal de um espaço de plotagem fasorial padrão. Esta posição será atribuída a um tempo de vida médio de fluorescência de uma solução homogênea pura de NAD (P) H, que normalmente está próxima do decaimento mono-exponencial62.
NOTA: O NADH livre e o NAD (P) H livre têm propriedades espectrais semelhantes e valores semelhantes de vida útil de fluorescência em solução aquosa, com dois componentes de vida útil curta, 0,3 ns e 0,7 ns63. Assim, pela simplicidade da análise baseada em fasores e devido a uma pequena diferença entre os componentes do tempo de vida, seu decaimento de fluorescência é aceito como monoexponencial, o que permite a alocação da nuvem fasorial em um círculo universal. O tempo médio de vida de referência de uma forma livre de NAD(P)H também pode ser medido e plotado em um espaço fasorial para uma análise semelhante. O tempo de vida de referência foi escolhido com base na literatura62; observe que em outras fontes, um valor ligeiramente diferente de um NAD (P) H livre em solução pode ser encontrado (0,4 ns64). - (Opcional para análise NAD(P)H) Exporte o gráfico fasorial com tempo de vida de NAD(P)H livre alocado (consulte a etapa 2.3.3) clicando com o botão direito do mouse no gráfico e selecionando Exportar dados. Exporte o gráfico fasorial como um arquivo de formato .tiff para uma pasta de armazenamento designada.
NOTA: O tamanho original do pixel da imagem de plotagem fasorial exportada do módulo LAS X FLIM é sempre 1024 x 600 pixels. Se outro software for usado para exportação e pré-análise de dados, certifique-se de que todas as imagens de plotagem fasorial sejam exportadas com o mesmo tamanho e resolução. - Para exportar nuvem fasorial relacionada ao esferoide, utilize a ferramenta Draw Cursor no módulo LAS X FLIM para escolher o ROI do esferóide na imagem. Exporte o gráfico fasorial gerado conforme descrito na etapa 2.3.4.
NOTA: As coordenadas g e s correspondentes (semelhantes a x e y) do espaço fasorial serão atribuídas a cada pixel da ROI escolhida, de acordo com seus tempos de vida, transformados em um conjunto de dados no domínio da frequência64,65. O cluster de pixels com valores semelhantes de tf (tempo de vida da fase) e tm (tempo de vida da modulação) formará um padrão de nuvem (nuvem fasor) em um gráfico, onde o código de cores (com um intervalo de azul profundo a vermelho) refletirá a abundância de valores de tempo de vida. Pela posição da nuvem em um círculo universal ou no interior, os decaimentos mono ou multi-exponenciais podem ser distinguidos. Alguns meios de medição exibem forte (auto)fluorescência, levando ao aparecimento de uma nuvem correspondente em um gráfico fasorial, que não pode ser simplesmente removida com a média do limiar de intensidade. Esse padrão influenciará o cálculo das coordenadas do centróide e deve ser excluído do gráfico fasorial exportado. Trabalhar com o ROI do esferóide permite a exclusão dos pixels não relacionados da análise fasorial posterior. - Repita o procedimento de exportação do gráfico fasorial para todas as ROIs esferóides em um conjunto de dados (consulte as etapas 2.3.4 e 2.3.6). Além disso, verifique o conjunto exportado de imagens .tiff para garantir o conjunto completo de dados para análise comparativa adicional e garantir que todas as imagens exportadas tenham o mesmo tamanho de pixel (consulte a NOTA na etapa 2.3.4).
NOTA: Nesta fase do protocolo, o conjunto de imagens deve incluir fasor com a localização de NAD(P)H livre (com base na literatura ou dados obtidos empiricamente) e todos os gráficos fasoriais de ROI esferóide (ou padrões alternativos de ROI, se necessário para a análise específica). A partir desta etapa, uma análise mais aprofundada será feita em FIJI e posteriormente em uma planilha. Usando a janela de ferramentas Analisar , defina a opção Escala no FIJI e certifique-se de que todas as imagens de plotagem fasorial sejam calibradas com o mesmo tipo de unidade, por exemplo, apenas em pixels. Caso contrário, defina o comprimento da unidade na janela Definir escala para a escolhida (por exemplo, para escala baseada em pixels, coloque 1 no campo Distância em pixels e defina a unidade de comprimento como Pixel). Para comparação adicional, meça todos os dados exportados usando o mesmo tipo de unidade. - (Opcional para análise NAD(P)H) Determine a posição do ponto de pixel de uma vida útil média de fluorescência de NAD(P)H livre na imagem de gráfico fasorial exportada correspondente (consulte a etapa 2.3.3): Abra a imagem fasorial com FIJI, amplie a imagem para visualizar claramente com uma resolução de pixel a interseção entre o semicírculo universal e a linha Cursor de proporção para dois componentes ; use a ferramenta ROI do retângulo para selecionar a interseção.
NOTA: Certifique-se de que a seleção retangular seja uma pequena área ao redor do ponto de interseção para determinação precisa de suas coordenadas na próxima etapa (etapa 2.3.9). - (Opcional para análise NAD(P)H) Abra a ferramenta Analisar , escolha a janela Definir medidas e selecione Centroide como um parâmetro de medição. Clique em Medir na janela de ferramentas Analisar para determinar as coordenadas do centróide do ponto de referência NAD(P)H livre. Exporte essas coordenadas para uma planilha.
NOTA: As coordenadas NAD (P) H livres serão usadas como ponto de referência para comparar as distâncias deste ponto até a posição da nuvem fasorial esferóide em um conjunto de dados (a maneira de caracterizar a mudança metabólica entre glicólise e OxPhos na análise de autofluorescência NAD (P) H FLIM) - Usando FIJI, abra a imagem da nuvem fasorial esferoide. Abra a ferramenta Janela de imagem , escolha Ajustar e selecione Limite de cor na barra de ferramentas. Selecione o Método de Limiar de escolha (por exemplo, Otsu) e defina o Valor de Matiz e o Valor de Brilho para restringir os parâmetros para a seleção de uma parte de nuvem fasorial com as coordenadas de pixel mais abundantes. Clique em Selecionar para definir a área do cluster.
NOTA: Mantenha os mesmos parâmetros de limite para todas as imagens de gráfico fasorial, que devem ser analisadas. Para os dados NAD(P)H-FLIM apresentados, o método de limiar Otsu com valor de matiz definido 9 e valor de brilho 160 foi escolhido e aplicado a todas as imagens fasoriais. Como alternativa, a área selecionada pode ser copiada para o ROI Manager (siga o caminho Editar > Seleção > Adicionar ao Manager) para criar uma biblioteca de ROIs fasoriais para análise posterior. - Mantendo a seleção, calcule as coordenadas do centróide da área selecionada seguindo o procedimento descrito na etapa 2.3.9. Exporte essas coordenadas para o arquivo de planilha.
- Repita as etapas 2.3.9 e 2.3.10 para determinar as coordenadas do centróide para todas as imagens fasoriais ROI para criar um conjunto de dados na planilha.
NOTA: O uso da biblioteca ROI Manager ajuda a simplificar e organizar a análise de ROI (consulte a etapa 2.3.10) - (Opcional para análise NAD(P)H) Abra a planilha com as coordenadas de referência de NAD(P)H livre e ROIs de esferoides exportadas de diferentes grupos de comparação. Calcule a distância entre cada centróide fasor esferóide individual para referenciar a posição de NAD(P)H livre usando coordenadas determinadas e a seguinte equação:
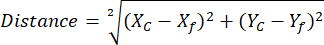
Onde, Xc e Yc são coordenadas centróides, Xf e Yf são as coordenadas de referência.
NOTA: A aplicação do parâmetro centróide para determinação do deslocamento em direção ao tempo de vida de referência é apropriada apenas no caso de todos os centróides de um conjunto de dados estarem na mesma trajetória linear em direção ao ponto de referência. Para verificar isso, todos os pontos centróides do conjunto de dados devem ser plotados junto com o ponto de referência no mesmo espaço de coordenadas, e o alinhamento da tendência linear deve ser realizado. Se o coeficiente R2 da linha de tendência linear traçada através de todos os pontos for próximo de 1 (por exemplo, R2 é 0,8-0,99), a análise de distância é considerada apropriada. - Organize todos os dados de acordo para comparação e realize análises estatísticas com o uso de qualquer software correspondente (por exemplo, Origin, MatLab). Escolha o teste estatístico apropriado de acordo com as características do conjunto de dados (normalidade da distribuição, número de unidades estatísticas, etc.).
NOTA: Para análise NAD(P)H, compare os valores de distância para caracterizar a mudança metabólica dependendo das condições experimentais. Para comparação de quaisquer gráficos fasoriais entre grupos experimentais, realize a comparação das coordenadas dos centróides da nuvem fasorial ROI.
- Abra o módulo FLIM no LAS X, selecione Abrir projeto e carregue o arquivo de imagem esferóide (.lif) para análise de NAD(P)H/FAD de autofluorescência.
Access restricted. Please log in or start a trial to view this content.
Resultados
Escolhendo o método apropriado de formação de esferoides
O método de formação de esferóides selecionado pode influenciar muito o tamanho, a forma, a densidade celular, a viabilidade e a sensibilidade aos medicamentos dos esferoides (Figura 2). Anteriormente, os efeitos de vários métodos de alto rendimento (SphericalPlate 5D, micromoldes feitos em laboratório e moldes MicroTissue) e os métodos de baixa fixação de 'rendimento médio' (placas de 96 poços revest...
Access restricted. Please log in or start a trial to view this content.
Discussão
Os esferoides multicelulares estão se tornando um método de escolha nos estudos de microambientes de nicho de tumores e células-tronco, descoberta de medicamentos e desenvolvimento dos 'blocos de construção de tecidos' para biofabricação. A arquitetura interna heterogênea dos esferoides, gradientes de nutrientes e oxigenação podem imitar os de tecidos e tumores in vivo em um ambiente relativamente simplificado e acessível. Com a necessidade de mais transparência metodológica26,28
Access restricted. Please log in or start a trial to view this content.
Divulgações
Nada a divulgar.
Agradecimentos
Este trabalho foi apoiado pelas bolsas do Fundo Especial de Pesquisa (BOF) da Universidade de Ghent (BOF/STA/202009/003; BOF/IOP/2022/058), Fundação de Investigação da Flandres (FWO, I001922N) e União Europeia, fliMAGIN3D-DN Horizon Europe-MSCA-DN n.º 101073507.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA | Gibco | 25300-054 | Also available from Sigma |
| 10 mL serological pipets | VWR | 612-3700 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 12 well cell-culture plates, sterile | Greiner bio-one | 665-180 | Similar products are also available from Sarstedt, Corning and other companies. |
| 12 Well Chamber slide, removable | Ibidi | 81201 | Also available from Grace Bio-Labs, ThermoFisher Scientific and others |
| 15 mL centrifuge tubes | Nerbe plus | 02-502-3001 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 3D Petri Dish micromolds | Microtissue | Z764000-6EA | |
| 6 well cell-culture plates, sterile | Greiner bio-one | 657160 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 70% ethanol | ChemLab | CL02.0537.5000 | |
| Biofloat | Sarstedt | 83.3925.400 | Commercial available coated 96-well plate for spheroid formation |
| Calcein Green-AM | Tebubio | AS-89201 | Apply in dilution 1:1000 |
| CellSens Dimension software | Olympus | version 3 | |
| Collagen from human placenta, type IV | Sigma | C5533 | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Confocal FLIM Microscope | Leica Microsystems | N/A | Stellaris 8 Falcon inverted microscope with white-light laser, HyD X detectors, climate / T control chamber (OkoLab), 25x/0.95 W objective |
| D(+)-Glucose | Merck | 8342 | Prepare 1 M stock solution, 1:100 for preparation of imaging medium (final concentration 10 mM) |
| Dulbecco's modified Eagle's medium (DMEM), phenol red-, glucose-, pyruvate- and glutamine-free | Sigma-Aldrich | D5030-10X1L | For preparation of imaging medium |
| Fetal Bovine Serum (FBS) | Gibco | 10270-098 | Also available from Sigma. Needs to be heat-inactivated before use. |
| HEPES (1M) | Gibco | 15630-080 | Dilution 1/100 for preparation of imaging medium (final concentration 10 mM) |
| Human colon cancer cells HCT116 | ATCC | ||
| ImageJ | NIH | version 1.54f | |
| Leica Application Suite X (LAS X) | Leica Microsystems | version 4.6.1.27508 | |
| L-glutamine | Gibco | 25030 | Also available from Sigma. Apply in dilution 1:100. |
| Lipidure-CM5206 | Amsbio | AMS.52000034GB1G | |
| McCoy's 5A, need addition of 1 mM Sodium Pyruvate and 10 mM HEPES | VWR | 392-0420 | Standard growth medium for HCT116 cells |
| micro-patterned 3D-printed PDMS stamps | N/A | N/A | Provided by the Centre for Microsystems Technology, Professor Dr. Jan Vanfleteren, Ghent University |
| NaCl | Chemlab | CL00.1429.100 | |
| Neubauer couting chamber | Fisher Scientific | 15980396 | |
| O2 probes: MMIR1 | N/A | N/A | Full characterization, validation and some applications can be found at: https://www.biorxiv.org/content/10.1101/2023.12.11.571110 v1 |
| PBS | Fisher scientific | Gibco18912014 | Dissolve PBS tablet in 500 mL of distilled water. |
| Pen Strep :Penicillin (10,000 U/mL) / streptomycin (10,000 μg/mL) 100x solution | Gibco | 15140-122 | Also available from Sigma. Apply in dilution 1:100. |
| Poly-D-lysine | Sigma | P6407-5mg | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Propidium Iodide | Sigma-Aldrich | 25535-16-4 | Cell death staining, use 1 µg/mL at 1h incubation |
| PVDF syringe filter 0.22 µm | Novolab | A35149 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Sodium pyruvate (100 mM) | Gibco | 11360-070 | Dilution 1/100 for preparation of imaging medium (final concentration 1mM) |
| SphericalPlate 5D 24-well | Kugelmeiers | SP5D-24W | |
| sterile petridish | Greiner bio-one | 633181 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (25 cm² ) | VWR | 734-2311 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (75 cm²) | VWR | 734-2313 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| U-bottom 96-well plate | VWR | 10062-900 | Similar products are also available from Sarstedt, Corning, Greiner Bio-one and other companies |
| Ultrapure Agarose | Invitrogen (Life Technologies) | 16500-500 | Other types of Agarose such as Agarose low melting point (A-9414, Sigma), Agarose for routine use (A-9539, Sigma) |
| Widefield fluorescence inverted microscope | Olympus | N/A | Inverted fluorescence microscope IX81, with motorised Z-axis control, CoolLED pE4000 (16 channels, 365-770 nm), ORCA-Flash4.0LT (Hamamatsu) cMOS camera, glass warming plate Okolab, CellSens Dimension v.3 software and air objectives 4x/0.13 UPlanFLN and 40x/0.6 LUCPlanFLN. (Optional, for high-resolution imaging) 60x/1.0 LUMPLFLN water |
Referências
- Foty, R. A simple hanging drop cell culture protocol for generation of 3d spheroids. J Vis Exp. (51), e2720(2011).
- Moskovits, N., et al. Establishing 3-dimensional spheroids from patient-derived tumor samples and evaluating their sensitivity to drugs. J Vis Exp. (190), e64564(2022).
- Griner, L. M., et al. Generation of high-throughput three-dimensional tumor spheroids for drug screening. J Vis Exp. (139), e57476(2018).
- Qian, Y., Wei, X., Chen, K., Xu, M. Three-dimensional acoustic assembly device for mass manufacturing of cell spheroids. J Vis Exp. (200), e66078(2023).
- He, H., et al. Dynamic formation of cellular aggregates of chondrocytes and mesenchymal stem cells in spinner flask. Cell Prolif. 52 (4), e12587(2019).
- Perez, J. E., Nagle, I., Wilhelm, C. Magnetic molding of tumor spheroids: Emerging model for cancer screening. Biofabrication. 13 (1), 015018(2020).
- Kingsley, D. M., et al. Laser-based 3d bioprinting for spatial and size control of tumor spheroids and embryoid bodies. Acta Biomater. 95, 357-370 (2019).
- Guillaume, O., et al. Hybrid spheroid microscaffolds as modular tissue units to build macro-tissue assemblies for tissue engineering. Acta Biomater. 165, 72-85 (2023).
- Danilevicius, P., et al. laser-made 3D microscaffolds for tissue spheroid encagement. Biointerphases. 10 (2), 021011(2015).
- Jamieson, L. E., Harrison, D. J., Campbell, C. Chemical analysis of multicellular tumour spheroids. Analyst. 140 (12), 3910-3920 (2015).
- Dmitriev, R., Borisov, S., Jenkins, J., Papkovsky, D. Multiparametric imaging of tumor spheroids with ultra-bright and tunable nanoparticle O2 probes. SPIE BiOS. 9328, (2015).
- Zushin, P. -J. H., Mukherjee, S., Wu, J. C. FDA Modernization Act 2.0: transitioning beyond animal models with human cells, organoids, and AI/ML-based approaches. J Clin Invest. 133 (21), e175824(2023).
- Kim, D. -S., et al. Robust enhancement of neural differentiation from human es and ips cells regardless of their innate difference in differentiation propensity. Stem Cell Rev Rep. 6 (2), 270-281 (2010).
- Hart, D., Gutiérrez, D. R., Biason-Lauber, A. Generation of a human ovarian granulosa cell model from induced pluripotent stem cells. bioRxiv. , 2022(2022).
- Chiaradia, I., et al. Tissue morphology influences the temporal program of human brain organoid development. Cell Stem Cell. 30 (10), 1351-1367 (2023).
- Wagstaff, E. L., Ten Asbroek, A. L., Ten Brink, J. B., Jansonius, N. M., Bergen, A. A. An alternative approach to produce versatile retinal organoids with accelerated ganglion cell development. Sci Rep. 11 (1), 1101(2021).
- Thavandiran, N., et al. Design and formulation of functional pluripotent stem cell-derived cardiac microtissues. Proc Natl Acad Sci U S A. 110 (49), E4698-E4707 (2013).
- Harrison, S. P., et al. Scalable production of tissue-like vascularized liver organoids from human pscs. Exp Mol Med. 55 (9), 2005-2024 (2023).
- Noguchi, T. -A. K., Kurisaki, A. Formation of stomach tissue by organoid culture using mouse embryonic stem cells. Methods Mol Biol. 2017, 217-228 (2017).
- Takahashi, J., et al. Suspension culture in a rotating bioreactor for efficient generation of human intestinal organoids. Cell Reports Methods. 2 (11), 100337(2022).
- Lagziel, S., Gottlieb, E., Shlomi, T. Mind your media. Nat Metab. 2 (12), 1369-1372 (2020).
- Das, V., Fürst, T., Gurská, S., Džubák, P., Hajdúch, M. Reproducibility of uniform spheroid formation in 384-well plates: The effect of medium evaporation. J Biomol Screen. 21 (9), 923-930 (2016).
- Bera, K., et al. Extracellular fluid viscosity enhances cell migration and cancer dissemination. Nature. 611 (7935), 365-373 (2022).
- Michl, J., Park, K. C., Swietach, P. Evidence-based guidelines for controlling ph in mammalian live-cell culture systems. Commun Biol. 2, 144(2019).
- Okkelman, I. A., Vercruysse, C., Kondrashina, A. V., Borisov, S. M., Dmitriev, R. I. Affordable oxygen microscopy-assisted biofabrication of multicellular spheroids. J Vis Exp. (182), e63403(2022).
- Peirsman, A., et al. Mispheroid: A knowledgebase and transparency tool for minimum information in spheroid identity. Nat Methods. 18 (11), 1294-1303 (2021).
- Raghavan, S., et al. Comparative analysis of tumor spheroid generation techniques for differential in vitro drug toxicity. Oncotarget. 7 (13), 16948(2016).
- Froehlich, K., et al. Generation of multicellular breast cancer tumor spheroids: Comparison of different protocols. J Mammary Gland Biol Neoplasia. 21 (3-4), 89-98 (2016).
- Debruyne, A. C., et al. Live microscopy of multicellular spheroids with the multimodal near-infrared nanoparticles reveals differences in oxygenation gradients. ACS Nano. 18 (19), 12168-12186 (2024).
- Debruyne, A. C., Okkelman, I. A., Dmitriev, R. I. Balance between the cell viability and death in 3D. Semin Cell Dev Biol. 144, 55-66 (2023).
- Barroso, M., Monaghan, M. G., Niesner, R., Dmitriev, R. I. Probing organoid metabolism using fluorescence lifetime imaging microscopy (flim): The next frontier of drug discovery and disease understanding. Adv Drug Deliv Rev. 201, 115081(2023).
- Becker, W. Fluorescence lifetime imaging-techniques and applications. J Microsc. 247 (2), 119-136 (2012).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. J Cell Sci. 134 (9), 1-17 (2021).
- Sarder, P., Maji, D., Achilefu, S. Molecular probes for fluorescence lifetime imaging. Bioconjug Chem. 26 (6), 963-974 (2015).
- Alfonso-Garcia, A., et al. Mesoscopic fluorescence lifetime imaging: Fundamental principles, clinical applications and future directions. J Biophotonics. 14 (6), e202000472(2021).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: Fundamentals and advances in instrumentation, analysis, and applications. J Biomed Opt. 25 (7), 1-43 (2020).
- Yellen, G. Fueling thought: Management of glycolysis and oxidative phosphorylation in neuronal metabolism. J Cell Biol. 217 (7), 2235-2246 (2018).
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomark Med. 4 (2), 241-263 (2010).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of nad (p) h and fad. Antioxid Redox Signal. 30 (6), 875-889 (2019).
- Verma, A., et al. Fluorescence lifetime imaging for quantification of targeted drug delivery in varying tumor microenvironments. bioRxiv. , 2024(2024).
- Smith, J. T., et al. In vivo quantitative fret small animal imaging: Intensity versus lifetime-based fret. Biophys Rep. 3 (2), 100110(2023).
- Alvarez, L. A., et al. Application Note: SP8 Falcon: A novel concept in fluorescence lifetime imaging enabling video-rate confocal flim. Nat Methods. , (2019).
- Roberti, M. J., et al. TauSense: A fluorescence lifetime-based tool set for everyday imaging. Nat. Methods. , https://www.nature.com/articles/d42473-020-00364-w.pdf (2020).
- Auer, J. M. T., Murphy, L. C., Xiao, D., Li, D. U., Wheeler, A. P. Non-fitting flim-fret facilitates analysis of protein interactions in live zebrafish embryos. J Microsc. 291 (1), 43-56 (2023).
- Warren, S. C., et al. Rapid global fitting of large fluorescence lifetime imaging microscopy datasets. PLoS One. 8 (8), e70687(2013).
- Gao, D., et al. Flimj: An open-source imagej toolkit for fluorescence lifetime image data analysis. PloS One. 15 (12), e0238327(2020).
- Tullis, I. D. C., Ameer-Beg, S. M., Barber, P. R., Rankov, V., Vojnovic, B. Mapping femtosecond pulse front distortion and group velocity dispersion in multiphoton microscopy. Proc. SPIE 6089, Multiphoton Microscopy in the Biomedical Sciences VI. , 60890(2006).
- Zhou, Y., et al. One-step derivation of functional mesenchymal stem cells from human pluripotent stem cells. Bio Protoc. 8 (22), e3080(2018).
- Benton, G., Arnaoutova, I., George, J., Kleinman, H. K., Koblinski, J. Matrigel: From discovery and ECM mimicry to assays and models for cancer research. Adv Drug Deliv Rev. 79-80, 3-18 (2014).
- Badea, M. A., et al. Influence of matrigel on single-and multiple-spheroid cultures in breast cancer research. SLAS Discov. 24 (5), 563-578 (2019).
- Lang, S., Sharrard, R., Stark, M., Villette, J., Maitland, N. Prostate epithelial cell lines form spheroids with evidence of glandular differentiation in three-dimensional matrigel cultures. Br J Cancer. 85 (4), 590-599 (2001).
- Barra, J., et al. DMT1-dependent endosome-mitochondria interactions regulate mitochondrial iron translocation and metastatic outgrowth. Oncogene. 43 (9), 650-667 (2024).
- Dmitriev, R. I., Papkovsky, D. B. Intracellular probes for imaging oxygen concentration: How good are they. Methods Appl Fluoresc. 3 (3), 034001(2015).
- Schweikhard, V., et al. Application Note: The power HyD family of detectors for confocal microscopy. Nat Methods. , https://www.nature.com/articles/d42473-020-00398-0 (2020).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon flim microscopy. Sci Rep. 12 (1), 11938(2022).
- Bush, P. G., Wokosin, D. L., Hall, A. C. Two-versus one photon excitation laser scanning microscopy: Critical importance of excitation wavelength. Front Biosci. 12, 2646-2657 (2007).
- Liu, M., et al. Instrument response standard in time-resolved fluorescence spectroscopy at visible wavelength: Quenched fluorescein sodium. Appl Spectrosc. 68 (5), 577-583 (2014).
- Szabelski, M., et al. Collisional quenching of erythrosine b as a potential reference dye for impulse response function evaluation. Appl Spectrosc. 63 (3), 363-368 (2009).
- Chib, R., et al. Standard reference for instrument response function in fluorescence lifetime measurements in visible and near infrared. Meas Sci Technol. 27 (2), 027001(2015).
- Talbot, C. B., et al. Application of ultrafast gold luminescence to measuring the instrument response function for multispectral multiphoton fluorescence lifetime imaging. Opt Express. 19 (15), 13848-13861 (2011).
- Becker, W. Recording the instrument response function of a multiphoton flim system. Becker & Hickl. , Available from: https://www.becker-hickl.com/literature/application-notes/recording-the-instrument-response-function-of-a-multiphoton-flim-system/ (2007).
- Leben, R., Köhler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free nad (p) h fluorescence lifetime imaging. Int J Mol Sci. 20 (22), 5565(2019).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using nadh and nadph autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Gottlieb, D., Asadipour, B., Kostina, P., Ung, T. P. L., Stringari, C. FLUTE: A python gui for interactive phasor analysis of flim data. Biol Imaging. 3, e21(2023).
- Malacrida, L., Ranjit, S., Jameson, D. M., Gratton, E. The phasor plot: A universal circle to advance fluorescence lifetime analysis and interpretation. Annu Rev Biophys. 50, 575-593 (2021).
- Okkelman, I., Vandenberghe, W., Dmitriev, R. Role of preconditioning with oxygen and glucose deprivation in promoting differentiation of dental pulp stem cells in 3D culture. Mol Biol Cell. 34 (2), 1212(2022).
- Park, J. H., et al. The effect of bmp-mimetic peptide tethering bioinks on the differentiation of dental pulp stem cells (DPSCs) in 3D bioprinted dental constructs. Biofabrication. 12 (3), 035029(2020).
- Pașca, S. P. The rise of three-dimensional human brain cultures. Nature. 553 (7689), 437-445 (2018).
- Dmitriev, R. I., Zhdanov, A. V., Nolan, Y. M., Papkovsky, D. B. Imaging of neurosphere oxygenation with phosphorescent probes. Biomaterials. 34 (37), 9307-9317 (2013).
- Bryanskaya, E. O., et al. High levels of FAD autofluorescence indicate pathology preceding cell death. Biochim Biophys Acta Gen Subj. 1868 (1), 130520(2024).
- Han, S. J., Kwon, S., Kim, K. S. Challenges of applying multicellular tumor spheroids in preclinical phase. Cancer Cell Int. 21 (1), 152(2021).
- Okkelman, I. A., Neto, N., Papkovsky, D. B., Monaghan, M. G., Dmitriev, R. I. A deeper understanding of intestinal organoid metabolism revealed by combining fluorescence lifetime imaging microscopy (flim) and extracellular flux analyses. Redox Biol. 30, 101420(2020).
- Gstraunthaler, G., Seppi, T., Pfaller, W. Impact of culture conditions, culture media volumes, and glucose content on metabolic properties of renal epithelial cell cultures: Are renal cells in tissue culture hypoxic. Cell Physiol Biochem. 9 (3), 150-172 (1999).
- Glickman, R. D. Phototoxicity to the retina: Mechanisms of damage. Int J Toxicol. 21 (6), 473-490 (2002).
- Golub, A. S., Pittman, R. N. Monitoring Parameters of Oxygen Transport to Cells in the Microcirculation. Quenched-Phosphorescence Detection of Molecular Oxygen: Applications in Life Sciences. , The Royal Society of Chemistry. (2018).
- Nishigaki, T., Wood, C. D., Shiba, K., Baba, S. A., Darszon, A. Stroboscopic illumination using light-emitting diodes reduces phototoxicity in fluorescence cell imaging. Biotechniques. 41 (2), 191-197 (2006).
- Penjweini, R., Loew, H. G., Hamblin, M. R., Kratky, K. W. Long-term monitoring of live cell proliferation in presence of pvp-hypericin: A new strategy using ms pulses of led and the fluorescent dye cfse. J Microsc. 245 (1), 100-108 (2012).
- Carrasco Kind, M., et al. flimview: A software framework to handle, visualize and analyze flim data. F1000Research. 9, 574(2020).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. Pam: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophys J. 114 (7), 1518-1528 (2018).
- Chen, S. -J., Sinsuebphon, N., Barroso, M., Intes, X., Michalet, X. Optical Molecular Probes, Imaging and Drug Delivery. , Optica Publishing Group. (2021).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados