Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Herstellung und Multiparameter-Lebendzell-Fluoreszenz-Lebensdauer-Bildgebungsmikroskopie (FLIM) von mehrzelligen Sphäroiden
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
In diesem Artikel beschreiben wir verschiedene Methoden zur Bildung von multizellulären Sphäroiden, um eine anschließende Multiparameter-Lebendzellmikroskopie durchzuführen. Unter Verwendung von Fluoreszenz-Lebensdauer-Bildgebungsmikroskopie (FLIM), zellulärer Autofluoreszenz, Färbefarbstoffen und Nanopartikeln wird der Ansatz zur Analyse des Zellstoffwechsels, der Hypoxie und des Zelltods bei lebenden dreidimensionalen (3D) Krebs- und Stammzell-abgeleiteten Sphäroiden demonstriert.
Zusammenfassung
Multizelluläre Tumor-Sphäroide sind ein beliebtes 3D-Gewebe-Mikroaggregatmodell für die Reproduktion von Tumormikroumgebungen, die Erprobung und Optimierung von Arzneimitteltherapien und den Einsatz von Bio- und Nanosensoren im 3D-Kontext. Ihre einfache Produktion, ihre vorhersehbare Größe, ihr Wachstum und ihre beobachteten Nährstoff- und Metabolitengradienten sind wichtig, um die 3D-nischenartige Zellmikroumgebung zu rekapitulieren. Die Heterogenität der Sphäroide und die Variabilität ihrer Produktionsmethoden können jedoch den gesamten Zellstoffwechsel, die Lebensfähigkeit und das Ansprechen auf das Arzneimittel beeinflussen. Dies macht es schwierig, die am besten geeignete Methodik zu wählen, wenn man die Anforderungen an Größe, Variabilität, Anforderungen an die Biofabrikation und die Verwendung als In-vitro-3D-Gewebemodelle in der Stamm- und Krebszellbiologie berücksichtigt. Insbesondere kann die Sphäroidproduktion ihre Kompatibilität mit quantitativen Lebendmikroskopien beeinflussen, wie z. B. optische metabolische Bildgebung, Fluoreszenzlebensdauer-Bildgebungsmikroskopie (FLIM), Überwachung der Sphäroidhypoxie mit Nanosensoren oder Viabilität. Hier wird eine Reihe konventioneller Protokolle zur Sphäroidbildung vorgestellt, die ihre Kompatibilität mit der Live-Weitfeld-, Konfokal- und Zwei-Photonen-Mikroskopie hervorheben. Die Follow-up-Bildgebung zur Analysepipeline mit Multiplex-Autofluoreszenz FLIM und unter Verwendung verschiedener Krebsarten und Stammzell-Sphäroide wird ebenfalls vorgestellt.
Einleitung
Mehrzellige Sphäroide stellen eine Gruppe von 3D-Gewebemodellen dar, die durch die Selbstaggregation von Zellen erhalten werden und eine kugelförmige Form aufweisen. Sie werden häufig verwendet, um Zell-Zell- und Zell-Matrix-Interaktionen in vitro nachzuahmen und einen 3D-Kontext in einer Vielzahl von Krebs- und Stammzell-abgeleiteten Konstrukten zu reproduzieren. Es werden verschiedene Techniken eingesetzt, um die Zelladhäsion zu reduzieren und die Aggregation zu fördern. Dazu gehören das Hanging-Drop-Verfahren, das sich auf die Oberflächenspannung1 stützt; Methoden zur Abwehr von Zellanhaftungen, wie z. B. Platten mit extrem geringer Anhaftung, Mikroformen und Mikrovertiefungen 2,3; akustischer wellenbasierter Ansatz4; strömungsinduzierte Aggregationsmethoden (Spinnerkolben, Bioreaktoren und mikrofluidische Geräte)5; magnetische partikelgestützte Bildung6 und Verwendung der aggregationsfördernden synthetischen und EZM-basierten Matrizen und Gerüste 7,8,9.
In der Krebsforschung, -entwicklung und -validierung neuer medikamentöser Therapien sind Sphäroide ein attraktives Modell, da sie in der Lage sind, die räumlich diffusionsbegrenzten Gradienten von Nährstoffen, Abfallprodukten und O2 zu rekapitulieren, was häufig zur Bildung eines nekrotischen Kerns führt, wie er für die soliden Tumoren typisch ist10,11. Diese zuverlässigeren und ausgefeilteren In-vitro-Modelle stellen die Notwendigkeit einer umfassenden Verwendung von Tiermodellen (Food and Drug Administration [FDA] Modernization Act 2.012) gemäß dem 3R-Prinzip der Tierversuche (Replacement, Reduction und Refinement) in Frage. Neben Krebs finden Sphäroide ihre Anwendung in der Stammzellforschung. Zum Beispiel haben pluripotente Stammzellen die Fähigkeit, Embryoidkörper (EB) zu bilden, die für die Differenzierung von induzierten pluripotenten Stammzellen (iPSCs) zu spezialisierten Zelltypen verwendet werden können, die nur schwer direkt von Patienten zu gewinnen sind, wie z. B. neurale Vorläuferzellen13 oder ovarielle Granulosazellen 13,14. Darüber hinaus ist die Bildung eines EB oft der erste Schritt bei der Entwicklung komplexerer Organoidmodelle, z. B. neuronale15, retinale16, kardiale17, Leber18, Magen19 und intestinale Organoide20. Faktoren wie Größe, Reproduzierbarkeit, Durchsatz und nachgelagerte Anwendungen sollten bei der Auswahl einer geeigneten Methode zur Sphäroidbildung für die Experimente berücksichtigt werden.
Die erhöhte Komplexität der 3D-Kultur kann zu einer höheren Variabilität im Vergleich zur 2D-Kultur führen. Faktoren wie die Nährstoffzusammensetzung21, die Verdunstung des Mediums22, die Viskosität23, die pH-Kontrolle24, das Verfahren zur Sphäroidbildung und sogar die Zeit in der Kultur25,26 können dazu führen, dass Sphäroide unterschiedlicher Morphologie, Größe, Lebensfähigkeit und unterschiedlicher Chemoresistenz erhaltenwerden 27,28. Neuere Forschungen haben gezeigt, dass sphäroide Sauerstoffgradienten nicht immer statisch sind und von der Bildungsmethode, der Sphäroidgröße und der extrazellulären Viskosität beeinflusst werden, was sich auf die Sphäroidheterogenität auswirkt29. Um die Reproduzierbarkeit und Zugänglichkeit von Daten zu Sphäroiden zu verbessern, wurde die MISpheroID-Wissensdatenbank26 entwickelt, in der die Zelllinie, das Kulturmedium, die Bildungsmethode und die Größe der Sphäroide als minimale Informationen für ein reproduzierbares Ergebnis identifiziert werden. Aus diesem Grund wurde ein detaillierter Vergleich mehrerer Methoden mit hohem Durchsatz (SphericalPlate 5D, im Labor hergestellte Mikroformen und Mikrogewebeformen) und Methoden mit geringer Befestigung (d. h. Biofloat- und Lipidure-beschichtete 96-Well-Platten, sowohl gerüstfrei als auch gerüstbasiert) durchgeführt (Abbildung 1 und Tabelle 1), einschließlich der Well-Größe (bei einer Schätzung der maximalen Sphäroidgröße), der verwendeten Verbrauchsmaterialien, der Vorbereitungszeit und der Möglichkeit, Sphäroide zu überwachen, ohne sie in Mikroskopieschalen zu transportieren. Letzteres ermöglicht Langzeitstudien, während Sphäroide, die mit Hochdurchsatzmethoden hergestellt werden, oft zu Endpunktexperimenten führen. Alle Methoden mit Ausnahme der Gitter der 5DspheriPlate bringen keine unerwünschte Autofluoreszenz mit sich, was ihren direkten Einsatz in der Mikroskopie ermöglicht.
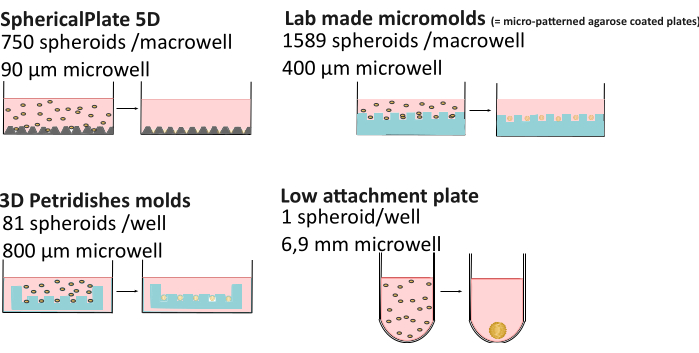
Abbildung 1: Erläuterung der Methoden zur Sphäroidbildung. Hochdurchsatzverfahren wie die SphericalPlate 5D, bei der patentierte Mikrowells in die Platte integriert sind, während die im Labor hergestellten Mikroformen und die MicroTissue-Formen Stempel verwenden, um mehrere Mikrowells in Agarose (blau) herzustellen. Platten mit geringer Bindung wie Lipidure (Amsbio) und Biofloat (Sarstedt) verwenden eine nicht haftende Beschichtung, die die Adhäsion der Zelloberfläche hemmt und die Selbstaggregation der Zellen fördert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| 5D SpheriPlate | Selbst hergestellte Mikroformen | Mikrogewebe | Methoden mit geringer Befestigung | |
| Anzahl der Sphäroide/Vertiefung | 750 | 1589 | 81 | 1 |
| Durchmesser gut | 90 μm | 400 μm | 800 μm | 1 mm |
| Kulturvolumen | 1 mL | 5 mL | 1 mL | 200 μL |
| Sonstige Verbrauchsmaterialien | / | 7 mL 3% Agarose | 500 μL 2 % Agarose | / * |
| Zubereitungszeit | ca. 10 Minuten | 2 h + 3 Tage Medienadaption | 0,5 h + 15 min Medienadaption | 10–30 min + 1 h Trocknung |
| Überwachung | Ja | Nein** | Ja | Ja |
| Autofluoreszierend | Ja | Nein | Nein | Nein |
| Mehrweg | Nein | Ja | Ja | Nein** |
| Kosten | €€ | € | €€€€ | €€€€: Beschichtung und Matrigel |
| €€: Kommerzielle 96-Well-Platte | ||||
| *Einige Zelllinien benötigen eine Zugabe von EZM (d. h. 2 % bis 5 % Matrigel), um kompakte Sphäroide zu bilden. | ||||
| **Die Beschichtung ist wiederverwendbar, bis sie aufgebraucht ist. Jede Platte verbraucht jedoch eine kleine Menge an Medien und Staub kann sich im Laufe der Zeit ansammeln. Eine Filtersterilisation ist regelmäßig erforderlich. | ||||
Tabelle 1: Vergleich mehrerer Methoden zur Sphäroidbildung29. "Monitoring": die Möglichkeit, das Sphäroid zu überwachen, ohne dass es zu einer Übertragung in eine Mikroskopieschüssel kommen muss. €: 0-50€, €€: 50-150€ , €€€: 150-500€ , €€€€: >500€
Die Fluoreszenzmikroskopie ermöglicht die direkte Überwachung der wichtigsten biologischen Aspekte in Sphäroiden, einschließlich Zelltod, Lebensfähigkeit, Proliferation, Stoffwechsel, Viskosität und sogar mechanischer Eigenschaften30. Die Fluoreszenzlebensdauer-Bildgebungsmikroskopie (FLIM) bietet eine zusätzliche quantitative Dimension für die Untersuchung der Wechselwirkungen von Fluoreszenzsonden in ihrer (Mikro-)Umgebung 31,32,33,34 und ermöglicht die Auflösung der überlappenden Emissionsspektren entsprechend unterschiedlicher Emissionslebensdauern35,36 und Untersuchung des Zellstoffwechsels auf der Grundlage der intrinsischen zellulären Autofluoreszenz. So können so weit verbreitete zelluläre autofluoreszierende Verbindungen wie Nicotinamid-Adenin-Dinukleotidphosphat (NAD(P)H), Flavin-Mononukleotid (FMN), Flavinadenin-Dinukleotid (FAD), Protoporphyrin IX und andere mit Ein- und Zwei-Photonen-FLIM gemessen werden und dienen als intrinsische "Sensoren" des Glukoseabbaus, der oxidativen Phosphorylierung (OxPhos) und bieten einen allgemeinen Überblick über den Redoxzustand der Zelle. NAD(P)H liegt in freier zytoplasmatischer oder in proteingebundener mitochondrialer Form vor37,38. In ähnlicher Weise ist der oxidierte Zustand von FAD fluoreszierend mit einer längeren Lebensdauer der freien Form. NAD(P)H- und FAD-Mikroskopie wird in der Regel mit Zwei-Photonen-angeregtem FLIM durchgeführt, um Photoschäden an der Probe zu verhindern39. Häufig kann die "optische metabolische Bildgebung" FLIM mit der Verwendung von farbstoffbasierten Sonden, genetisch kodierten Biosensoren, Phosphoreszenz-Lebensdauer-Bildgebungsmikroskopie (PLIM) und ratiometrischen intensitätsbasierten Messungen kombiniert werden, um ein vollständigeres Bild des Sphäroid- oder Organoidstoffwechsels, der Sauerstoffversorgung, der Proliferation und der Zellviabilität zu erhalten 29,30,31. Darüber hinaus kann FLIM auch mit der Förster-Resonanzenergietransfer-Methode (FRET) kombiniert werden, um die Lebensdauervariation des Donorfluorophors zu messen, wenn es in engem Kontakt mit dem Akzeptor steht, um die Bindung eines Wirkstoffs an seine Zieldomäne 33,40,41 zu untersuchen.
Die erfassten FLIM-Bilder werden in der Regel analysiert, um die Lebensdauer Pixel für Pixel zu berechnen. Derzeit gibt es mindestens 3 gängige Strategien, um die Fluoreszenzlebensdauer zu erhalten: semiquantitatives "schnelles FLIM"42 (manchmal auch als "Tau-Sinn" bezeichnet43,44), Zerfallskurvenanpassung unter Verwendung von ein-, zwei- oder dreiexponentieller Anpassung und "anpassungsfreier" Ansatz mit Phasortransformation und Phasorplot-Analyse. Je nach Anbieter kann entweder die bereitgestellte (LAS X, Symphotime, SPCImage usw.) oder Open-Source-Software (z. B. FLIMfit45, FLIMJ46 oder andere47) verwendet werden, um gemessene FLIM-Daten zu verarbeiten. In der Regel ist vom Anbieter bereitgestellte Software für die vorläufige Datenanalyse nützlich, während Open-Source-Lösungen genauere Studien ermöglichen können, z. B. mit Hilfe von Phasordiagrammen und 3D-Visualisierung.
Trotz der Nützlichkeit und Attraktivität von FLIM als Methode zur Untersuchung von Sphäroiden stehen nur sehr wenige experimentelle Protokolle zur Verfügung, und es besteht ein allgemeiner Mangel an Wissen über die Auswahl der am besten geeigneten Bildungsmethode für erfolgreiche multiparametrische Live-Mikroskopie-Experimente mit FLIM. In dieser Arbeit wird ein detaillierter Vergleich der häufig verwendeten Sphäroidbildungsprotokolle auf der Grundlage ihrer Morphologie, Viabilität und Oxygenierung mit dem kürzlich validierten und charakterisierten Sauerstoffsensor-Nanosensor (MMIR1) für Fernrot und Nahinfrarot (NIR) vorgestellt. Das kationische Nanopartikel ist mit zwei Reporterfarbstoffen imprägniert, dem Referenz-O 2-unempfindlichen aza-BODIPY (Anregung 650 nm, Emission 675 nm) und dem NIR O2-empfindlichen Metalloporphyrin, PtTPTBPF (Anregung 620 nm, Emission 760 nm). Das MMIR1 ermöglicht die Echtzeitanalyse von Sauerstoffgradienten mit einem herkömmlichen Fluoreszenzmikroskop (mittels ratiometrischer Analyse) oder einem Phosphoreszenz-Lebensdauermikroskop (PLIM) ohne zelluläre Toxizität und ermöglicht stabile Signale, Langzeitüberwachung und Multiplexing25,29. Abhängig von der Notwendigkeit, mit Farbstoffen oder Nanosensoren zu färben, dem Sphäroiddurchsatz oder dem Zelltyp kann das am besten geeignete Bildungsprotokoll ausgewählt werden. Da die Studien zur Lebensfähigkeit und Sauerstoffversorgung von Sphäroiden für Studien zu Krebs und Stammzell-abgeleiteten Sphäroiden relevant sind, enthalten die vorgestellten Protokolle auch Beispiele und erwartete typische Ergebnisse von NAD(P)H-FLIM und FAD-FLIM mit diesen Modellen. Die vorgestellten Bildgebungs- und Analysepipelines zielen auf die gängigsten zeitkorrelierten FLIM-Mikroskopieplattformen ab, die auf Einzelphotonenzählung basieren.
Protokoll
1. Erzeugung von mehrzelligen Sphäroiden
- Zellkultur
HINWEIS: Zellkulturen können von der American Type Culture Collection (ATCC), Lonza, Sigma-Aldrich oder anderen Anbietern entnommen werden. ATCC bietet alle erforderlichen Informationen zur Handhabung, einschließlich bevorzugter Wachstumsmedien, Subkulturverfahren, Biosicherheitsniveau, Wachstumsrate und STR-Profile. Hier werden 500 Zellen/Sphäroid der humanen Dickdarmkrebs-Zelllinie HCT116 in McCoy's 5A-Medien (VWR, 392-0420) verwendet, ergänzt mit 10% FBS und 1 mM Natriumpyruvat. Für Langzeitexperimente, die täglich überwacht werden, können dem Medium 10 mM HEPES, pH 7,2 zugesetzt werden.- Züchten Sie die Zellkultur, um eine Konfluenz von 70 % bis 90 % zu erreichen.
- Die Zellen werden mit vorgewärmtem (37 °C) sterilem PBS gespült (5 mL für 25 cm2 oder 10 mL pro 75 cm2 Kolben).
- 0,05 % Trypsin - 1 mM EDTA-Lösung (0,5 mL für 25 cm² oder 1 mL pro 75 cm2 Kolben) zugeben und 5-10 min bei 37 °C in 5 % CO2 und 95 % Feuchtigkeit inkubieren, um eine Zellablösung zu erreichen.
HINWEIS: Kontrollieren Sie die Zellablösung unter dem Transmissionslichtmikroskop (Hellfeldmikroskop). Die übermäßige Exposition der Zellen gegenüber der dissoziierenden Enzymlösung kann ihre Lebensfähigkeit beeinträchtigen. - Neutralisieren Sie Trypsin, indem Sie überschüssige Zellkulturmedien mit 10 % FBS (mindestens 5 ml Medien pro 1 ml Dissoziationslösung) hinzufügen.
HINWEIS: Bei Zellen, die auf niedrigen FBS- oder FBS-freien Kulturmedien kultiviert werden, kann die Trypsinneutralisation durch Zugabe von 0,5 ml 100 % FBS in die mit Trypsin behandelte Zellkultur erfolgen, gefolgt von einer Zentrifugation, um die Zellen auf ihre ursprünglichen Kulturmedien zu übertragen. - Dissoziieren Sie Zellaggregate durch Pipettieren, um eine Einzelzellsuspension in Medien zu erhalten.
HINWEIS: Das Pipettieren mit einer serologischen Pipette mit einer 1000-μl-Pipettenspitze auf der Oberseite verbessert die Erzeugung von Einzelzellsuspensionen in einem großen Volumen von Suspensionszellkulturen erheblich. - Verwenden Sie eine Zählkammer (Neubauer-Regel-Hämozytometer oder Alternativen), um die Zellzahl pro 1 ml der Zellsuspension zu zählen.
- Verdünnen Sie die Zellsuspension, um die gewünschte Anzahl von Zellen pro Milliliter zu erhalten.
- Geben Sie der Zellsuspension eine konzentrierte O2 -Sonde (Nanopartikel) Lösung in einer Endkonzentration von 10 μg/ml für die ratiometrische Analyse von O2.
HINWEIS: Um eine homogene Zellsuspension (mit der Sonde) zu gewährleisten, resuspendieren Sie die Suspension mehrmals, bevor Sie sich sphäroidbilden. Wenn die O2-Sonde nicht benötigt wird, überspringen Sie diesen Schritt und fahren Sie mit der Bildung der Sphäroide fort. Beim Umgang mit iPSCs sollte ein modifiziertes Protokoll angewendet werden. Kurz gesagt, iPS-Zellen werden in Kolonien auf Geltrex-beschichteten Platten gezüchtet und unter Verwendung von ReLeSR passageiert, wie in dem vom Hersteller bereitgestellten Protokollbeschrieben 48. Am Tag der Sphäroidbildung sollten die Kolonien groß und kompakt sein und mehrschichtige Zentren mit deutlichen Grenzen aufweisen. Spülen Sie die Zellen mit vorgewärmtem sterilem PBS. Fügen Sie 1 ml eines sanften Zelldissoziationsreagenzes (GCDR) hinzu und inkubieren Sie es 8-10 Minuten lang bei 37 °C. Verwenden Sie eine 1000-μl-Spitze, um Zellen vorsichtig aus der Vertiefung zu lösen und eine Einzelzellsuspension zu erhalten. Übertragen Sie die einzellige Suspension in ein steriles konisches 50-ml-Röhrchen und fügen Sie 4 ml vorgewärmtes DMEM-F12-Medium hinzu, um das GCDR zu neutralisieren. Waschen Sie die Vertiefung mit 1 mL DMEM-F12 und übertragen Sie es auf den Rest der Zellsuspension. Bei 300 x g 5 min zentrifugieren. Für weitere Versuche in 1 ml geeignetem Medium resuspendieren. Für die in diesem Manuskript beschriebenen Experimente wurde mTeSR + 10 μM Rock Inhibitor verwendet. Zählen und verdünnen Sie die Zellsuspension, um die gewünschte Anzahl von Zellen pro Milliliter zu erhalten.
- Methoden zur Sphäroidbildung
- 3D-Mikroformen für Petrischalen
HINWEIS: Dieses Hochdurchsatzverfahren wird verwendet, um gleichzeitig eine hohe Anzahl von Sphäroiden (81 Sphäroide) in einem 9 x 9 Mikromold-Array mit 800 μm Durchmesser und 800 μm Tiefe zu erzeugen.- Mikroformen zum Gießen von 3D-Petrischalen in dH2O spülen und in einen autoklavierbaren Behälter geben.
- Messen Sie 2 g Agarosepulver in Elektrophoresequalität ab und geben Sie es in eine trockene, autoklavensichere 200-ml-Glasflasche.
HINWEIS: Stellen Sie sicher, dass die Flasche und das Agarosepulver trocken und ohne Flüssigkeit oder Feuchtigkeit sind. - Autoklavieren Sie Mikroformen zum Gießen von 3D-Petrischalen und -flaschen mit Agarosepulver für 30 Minuten im Trockenzyklus.
- Eine 0,9 w/v% Kochsalzlösung durch Zugabe von 0,9 g NaCl in 100 mL Reinstwasser herstellen und durch Autoklavieren sterilisieren.
HINWEIS: NaCl wird vom Hersteller empfohlen. Es erhöht die Stabilität der Agarose. - Bereiten Sie die Agaroselösung vor, indem Sie die sterile Kochsalzlösung zum sterilisierten Agarosepulver hinzufügen. Schrauben Sie den Deckel locker auf, um einen Druckaufbau zu vermeiden. Schwenken Sie die Flasche, um das Agarosepulver zu mischen.
- Kochen Sie das Agarosepulver und lösen Sie es in der Mikrowelle auf. Stoppen Sie die Mikrowelle häufig (~alle 10 s). Schwenken Sie die Flasche und wiederholen Sie den Vorgang, bis sich die Agarose aufgelöst hat.
ACHTUNG: Die Agaroselösung ist heiß und erfordert eine sorgfältige Handhabung. Schütteln unmittelbar nach dem Schmelzvorgang kann dazu führen, dass die Lösung aus dem Gefäß platzt. Um Unfälle zu vermeiden, verwenden Sie ausreichend große Gefäße, die bis zu nicht mehr als 50 % des Fassungsvermögens gefüllt sind, und verwenden Sie einen geeigneten persönlichen Schutz (Handschuhe, Ofenhandschuh, Augenschutz und Laborkittel). - Die gelöste Agaroselösung auf 60-70 °C abkühlen lassen. Unter Verwendung aseptischer Techniken und Bedingungen pipettieren Sie geschmolzene Agarose in die Mikroform (500 μl für 12er Serien oder 330 μl für 24er Serien).
HINWEIS: Vermeiden Sie Blasenbildung beim Mischen oder Pipettieren von Agarose. Entfernen Sie alle eingeschlossenen kleinen Blasen in den kleinen Merkmalen der Mikroform durch Pipettieren oder vorsichtiges Verschrotten, bevor die Agarose erstarrt. - Die Agarose ca. 2-3 min fest werden lassen. Biegen Sie anschließend die Mikroform vorsichtig, um die 3D-Petrischale zu entfernen, und geben Sie sie auf eine 12-Well-Gewebekulturplatte.
HINWEIS: Eine Überbiegung der Mikroform kann zur Bildung von Rissen in der Agaroseform führen. - Um die 3D-Petrischale zu äquilibrieren, fügen Sie 2,5 ml/Well-Zellkulturmedium hinzu. 15 Minuten oder länger inkubieren. Entfernen Sie das Nährmedium und ersetzen Sie es durch frisches Medium. Wiederholen Sie den Vorgang erneut, um die 3D-Petrischale mit einem Nährmedium zu äquilibrieren.
HINWEIS: Das Protokoll kann hier bis zum Cell Seeding unterbrochen werden. Für die Langzeitlagerung (bis zu 2 Wochen bei 4 °C) ist PBS-Lösung anstelle von Medium zu verwenden. - Entfernen Sie die 3D-Petrischale, die das Kulturmedium (oder PBS) umgibt, vollständig und entfernen Sie das Medium in der 3D-Petrischale, indem Sie die Gewebekulturplatte kippen.
- 190 μl Zellsuspension mit 40.500 Zellen vorsichtig tropfenweise in die Zellaussaatkammer aussäen (siehe Schritt 1.1).
HINWEIS: Die Anzahl der Mikrovertiefungen in Agarose-Stempeln bestimmt die Anzahl der pro Stempel hergestellten Sphäroide. In diesem Fall enthält diese Agarose-Form 81 Mikrowells (81 x 500 Zellen/Sphäroide). Die Variation der Zellkonzentration in Suspension, die in einen Makrotopf gegeben wird, ermöglicht es, die Zellzahl pro Sphäroid zu ändern und so die Größe des Sphäroids zu steuern. - Warten Sie ~10 Minuten, bis sich die Zellen in den Eigenschaften des 3D-Petri-Dsh eingenisten. Geben Sie dann 2 ml Medium an die Außenseite der 3D-Petrischale.
- Legen Sie die Gewebekulturplatte in den Zellkultur-Inkubator und tauschen Sie das Medium, das die 3D-Petrischale umgibt, nach Bedarf aus.
- Niedrige Befestigungsplatte
HINWEIS: Diese Methode wird verwendet, um ein einzelnes Sphäroid pro Vertiefung zu erzeugen. Beschichtete Platten (Lipidure oder Biofloat) sind im Handel erhältlich (überspringen Sie die Schritte 1.2.2.1-1.2.2.4). Alternativ kann die Beschichtung separat erworben und zur Beschichtung von unbehandelten Multiwell-Platten verwendet werden. Es wird empfohlen, die Vertiefungen an den Rändern von 96-Well-Platten mit sterilem Wasser oder PBS zu füllen, da in diesen Wells schneller verdunstet, wodurch die Anzahl der Wells für Sphäroide auf 60 begrenzt wird. Falls weniger Sphäroide benötigt werden, füllen Sie die umliegenden leeren Brunnen mit Wasser oder PBS. Verwenden Sie staubfreie Spitzen für die Handhabung von Flüssigkeiten, um zu vermeiden, dass kleine Partikel in die Vertiefungen gelangen, da sie die Sphäroidbildung beeinträchtigen.- Bereiten Sie eine Beschichtungslösung mit 0,5 w/v% vor, indem Sie 0,25 g des Polymerpulvers in 50 mL Ethanol in einem Glasflaschenbehälter auflösen. Sterilisieren Sie die Beschichtung mit einem Filter.
HINWEIS: Die Filtersterilisation und alle weiteren Schritte müssen unter sterilen Bedingungen unter laminarer Strömung durchgeführt werden. - Geben Sie 200 μl der Beschichtungslösung in jede Vertiefung einer 96-U-Bodenkulturplatte.
- 1 min inkubieren und überschüssige Beschichtung entfernen.
HINWEIS: Die Beschichtungslösung kann mehrfach verwendet werden. In einem Glasbehälter bei Raumtemperatur (RT) lagern. Plastikbehälter werden nicht empfohlen, da sich der Kunststoff teilweise auflösen und Teil der Lösung werden kann. Wenn Staub vorhanden ist, sterilisieren Sie den Filter mit einem 0,22 μm Polyethersulfon (PES) oder Nylon-Spritzenfilter. - Lassen Sie die 96-Well-Platte ca. 1 h an der Luft trocknen.
HINWEIS: Wenn Zellen eine extrazelluläre Matrix benötigen, fahren Sie mit Schritt 1.2.3 fort. Beschichtete Platten können bei RT gelagert werden, wenn sie in Aluminium eingewickelt sind, bis zu 1 Monat. Wenn Sie eine geringere Menge an Zellen pro Vertiefung aussäen, kann das Zentrifugieren der Platte für 5 Minuten bei 300 g helfen, die Zellen nach unten zu ziehen.
- Bereiten Sie eine Beschichtungslösung mit 0,5 w/v% vor, indem Sie 0,25 g des Polymerpulvers in 50 mL Ethanol in einem Glasflaschenbehälter auflösen. Sterilisieren Sie die Beschichtung mit einem Filter.
- Extrazelluläres Matrix-gestütztes Bildungsprotokoll
HINWEIS: Einige Zelllinien produzieren selbst nicht genügend extrazelluläre Matrix (ECM) und benötigen die Zugabe von ECM, wie z. B. Matrigel, Cultrex oder Geltrex, um kompakte Sphäroide zu bilden 49,50,51. Für solche Zelltypen wie Brustkrebs MDA-MB-231, menschliche Hautpapillen, Prostatakrebszellen und andere ist es möglich, Schritt 1.2.2 mit den folgenden Modifikationen52 zu verwenden, die die Hinzufügung von EZM erfordern. Verwenden Sie vorzugsweise staubfreie Spitzen für die Handhabung von Flüssigkeiten, um zu vermeiden, dass Staub die Sphäroidbildung beeinträchtigt. Die Schritte 1 und 4-7 müssen unter einer biologischen Sicherheitswerkbank (Klasse II) durchgeführt werden.- Fahren Sie mit den Schritten 1.2.2.1 bis 1.2.2.4 (niedrige Befestigungsplatten) für die Oberflächenbehandlung fort.
- Die 96-Well-Platte bei 4 °C im Kühlschrank vorkühlen.
- Bereiten Sie die Zentrifuge mit dem richtigen Adapter für die 96-Well-Platte vor und kühlen Sie sie auf 4 °C vor.
- Bereiten Sie eine 5%ige Lösung der Basalmembranmatrix (BMM) in vorgekühlten (4 °C) Zellkulturmedien vor.
HINWEIS: BMM vernetzt schnell bei RT. Bewahren Sie bei der Handhabung Vorräte und Lösungen auf Eis auf. - Bereiten Sie die Zellsuspension vor (siehe Schritt 1.1).
- Geben Sie 50 μl der BMM-Lösung in jede Vertiefung.
- Geben Sie vorsichtig 50 μl Zellsuspension in jede Vertiefung auf der BMM-Lösung (25.000 Zellen/Vertiefung).
HINWEIS: Sprengen Sie dieses Volumen nicht in die Vertiefung, da sich die Zellen sonst an den Seiten der Vertiefungen ausbreiten und sich nicht am Boden sammeln. Eine geringere Anzahl von Zellen pro Vertiefung kann erreicht werden, indem die Zelldichte entsprechend angepasst wird. Nicht alle Zellen bilden Sphäroide bei allen Aussaatdichten; Die Optimierung muss je nach Zellentyp und gewünschter Dimension durchgeführt werden. Bei den bereitgestellten Volumina beträgt die Endkonzentration von BMM 2,5 %. Wenn eine andere Konzentration erforderlich ist, muss die Stammlösung in einer niedrigeren/höheren Konzentration hergestellt werden. - Die 96-Well-Platte wird 5 Minuten lang bei 300 x g und 4 °C zentrifugiert.
HINWEIS: Ohne diesen Schritt aggregieren die Zellen am Boden der Vertiefung nicht richtig, wodurch sich mehrere kleinere Aggregate bilden. Die Zentrifuge muss abgekühlt werden, um eine Vernetzung in dieser Phase zu vermeiden. - Stellen Sie die Platte in den Zellkultur-Inkubator. Die Zuschlagstoffe gelten am Tag 4 nach der Aussaat als reif.
HINWEIS: Weitere Protokolle zur Sphäroidbildung finden Sie in der Zusatzdatei 1.
- 3D-Mikroformen für Petrischalen
2. Live-Mikroskopie von Sphäroiden
- Aufbereitung von Sphäroiden für die Live-Bildgebung
HINWEIS: Abhängig von der Versuchsplanung (z. B. Langzeitüberwachung oder Endpunktanalyse, Mikroskopaufbau oder spektrale Eigenschaften der gemessenen Fluoreszenz) oder aufgrund der Inkompatibilität der Methode zur Herstellung von Sphäroiden mit der Mikroskopie (z. B. Probendicke, Autofluoreszenz des Materials, Schweben von Sphäroiden während der Bildgebung) direkte Überwachung der Sphäroide in der Platte, wo sie hergestellt wurden, möglicherweise nicht möglich. Das Protokoll erläutert die Vorbereitung von Sphäroiden für die Bildgebung, die für die meisten inversen Weitfeld- und Konfokalmikroskope geeignet sind.- Aufbereitung und Vorwärmen (37 °C) Bildgebungsmedien: DMEM ergänzt mit HEPES-Na, pH 7,2 (10 mM), Natriumpyruvat (1 mM), L-Glutamin (2 mM) und Glukose (5 mM), ohne Phenolrot.
HINWEIS: Natriumbicarbonat allein oder in Kombination mit HEPES-Na kann verwendet werden, wenn während der Bildgebung eine CO2 - Kontrolle vorliegt24. Einige Zellkulturtypen vertragen das Vorhandensein von HEPES nicht. Je nach Versuchsdesign können der Pyruvat-, Glutamin- und Glukosegehalt modifiziert werden. - Bereiten Sie sterile Mikroskopieschalen (kommerziell erhältlich oder im Labor hergestellt) mit beschichteten (für eine starke Sphäroidhaftung) oder unbeschichteten (geringe Sphäroidhaftung) Deckglasoberflächen (Dicke #1,5) vor.
HINWEIS: Der Bedarf und die Art der Beschichtung hängen vom Zelltyp, den Adhäsionseigenschaften der Sphäroide und der Geschwindigkeit ihrer Zellmigration von der 3D- zur 2D-Kulturschnittstelle ab. Dies ist wichtig zu berücksichtigen, da die Beschichtung den Verlust der 3D-Organisation erleichtern und die Form von Mikrogradienten in Sphäroiden und damit das Zellverhalten verändern kann. Für einige Experimente (z. B. bildgebende Verfahren zur Analyse der Reaktion auf Arzneimittelstimulation) ist eine starke Sphäroidhaftung an der Oberfläche erforderlich, und eine Beschichtung mit Gelatine, BMM, Kollagen, Kollagen/Poly-D-Lysin oder Poly-D-Lysin wird bevorzugt. - Waschen Sie die mit derO2-Sonde gefärbten Sphäroide vorsichtig aus den Mikrovertiefungen der mikrostrukturierten Agarose- oder 96-Well-Platte aus und überführen Sie die noch schwimmenden Sphäroide in ein 2-ml-Fläschchen.
HINWEIS: Um die Sammlung aller Sphäroide aus der Hochdurchsatzmethode zu gewährleisten, spülen Sie die Form 1-3 Mal mit dem zusätzlichen Volumen des Nährmediums und kombinieren Sie alle Sphäroidsuspensionen in einem Fläschchen. Bei Sphäroiden in einer niedrigen Anhaftungsplatte sind die Sphäroide einzeln aus einzelnen Vertiefungen in ein Fläschchen oder direkt in eine Mikroskopieschale zu sammeln, wenn eine kleine Anzahl von Sphäroiden für das Experiment ausreicht. Schneiden Sie beim Übertragen großer Sphäroide das Ende der Pipettenspitze ab, um sicherzustellen, dass beim Pipettieren keine Schäden entstehen. - Lassen Sie das Fläschchen bis zu 5 Minuten lang in vertikaler Position, damit sich die Sphäroide auf dem Boden des Fläschchens absetzen und ein sichtbares Pellet bilden.
- Nehmen Sie das Medium aus dem Röhrchen, lassen Sie die Sphäroide ungestört und resuspendieren Sie sie vorsichtig in einer ausreichenden Menge frischer Kultur.
HINWEIS: Zur Vereinfachung der Handhabung von Sphäroiden kann die Übertragung von Sphäroiden auf das Bildgebungsmedium in diesem Stadium auch in kleinen Chargen erfolgen. Siehe Schritte 2.1.7 und 2.1.8. Sphäroide aus den verschiedenen Versuchsgruppen sollten gleich behandelt werden, da die Medienzusammensetzung und die Vorkonditionierungszeit des Mediums ihren Stoffwechsel beeinflussen können. - Während die Sphäroide schweben, übertragen Sie ein gleiches Volumen der Sphäroidsuspension pro Vertiefung der Mikroskopieschale.
- Inkubieren Sie die Sphäroide für 1-2 h bei 37 °C in einem CO2 -Inkubator, um ihre Befestigung an der Oberfläche der Mikroskopieschale/-vertiefung sicherzustellen. Für das Imaging fahren Sie mit Schritt 2.1.9 fort. Für die Färbung von Sphäroiden mit zusätzlichen Sonden ist mit Schritt 2.1.8 fortzufahren.
HINWEIS: Die Rate der Zellmigration vom 3D-Sphäroid zur 2D-Oberflächenschnittstelle ist eine Funktion der Zeit. Um einen Verlust der 3D-Organisation zu vermeiden, muss die Inkubationszeit in Bezug auf den Zelltyp, die Art der Oberflächenbeschichtung und das Design des Experiments optimiert werden. Zum Beispiel benötigt HCT116 je nach Sphäroidgröße mindestens 2 h für eine korrekte Sphäroidbindung an die Kollagen IV/Poly-D-Lysin-beschichtete Oberfläche, während die hDPSC-Bindung und Migration zur 2D-Grenzfläche extrem schnell ist, was zu einem Verlust der 3D-Organisation in 1-2 Stunden führt. Um die Abbildung von "2D-Sphäroiden" aufgrund einer übermäßigen Ausbreitung zu vermeiden, werden unbeschichtete Glasoberflächen mit der verkürzten Inkubationszeit verwendet. - Fluoreszenzsonde(n) in empfohlenen oder empirisch optimierten Konzentrationen zu einem bekannten Volumen der Sphäroidsuspension hinzufügen. Vor der Bildgebung 1 h bei 37 °C CO2 -Inkubator inkubieren.
HINWEIS: Verwenden Sie für den Lebend-/Tot-Assay Propidiumiodid und Calcein Green-AM in einer Standard-Endkonzentration von 1 μg/ml. Um die toxische Wirkung der Propidiumiodid-Färbung auf iPSC-Embryoidkörper zu vermeiden, betrug die endgültige Propidiumiodidkonzentration 0,5 μg/ml. Die Ladezeit der Sonde kann verlängert werden, wenn die Diffusion der Sonde aufgrund der großen Sphäroidgröße nicht effizient ist. Die Ladezeit sollte immer als Teil der gesamten Inkubationszeit betrachtet werden, die für die Anheftung des Sphäroids an der Oberfläche benötigt wird. Wenn eine längere Zeit für die Anheftung der Sphäroide benötigt wird, sollte das Färbeverfahren am Ende dieses Zeitraums angeordnet werden. Beachten Sie, dass eine längere Inkubationszeit zum Verlust der 3D-Organisation führen kann. - Entfernen Sie Zellkulturmedien oder Medien, die Fluoreszenzsonden enthalten, und tauschen Sie sie gegen ein erforderliches Volumen an Bildgebungsmedien aus. Um sicherzustellen, dass sich kein Medienfluoreszenzhintergrund auf dem Weg der Sphäroidbildgebung befindet, wiederholen Sie den Schritt des Medienaustauschs (Waschen) bis zu 5 Mal.
HINWEIS: Um eine Sphäroidentfernung während des Medienwechsels zu vermeiden, wird empfohlen, das Medium vorsichtig mit einer 200-μl-Pipette von den Rändern der Mikroskopieschalen abzusaugen und die Zugabe des Mediums an der Wand oder seitlich in der Mikroskopieschale durchzuführen. - Fahren Sie sofort mit Schritt 2.2.1 des Bildgebungsprotokolls fort.
HINWEIS: Eine zu lange Pause zwischen der Vorbereitung auf die Bildgebung und der tatsächlichen Bildaufnahme kann den Zellstoffwechsel (z. B. durch eine veränderte Zusammensetzung des Mediums), die Lebensfähigkeit (einige Fluoreszenzsonden, die für die Endpunktanalyse verwendet werden, haben toxische Wirkungen, die den Zelltod nach einer langen Inkubationszeit stimulieren können) beeinflussen und zu einem Verlust der 3D-Organisation führen. Wenn mehrere Gruppen von Sphäroiden oder experimentelle Bedingungen verglichen werden müssen, muss das Versuchsdesign entsprechend entwickelt werden, um den Zeitpunkt der Behandlung, die Präkonditionierung und die bildgebenden Verfahren zwischen den analysierten Gruppen so gleich wie möglich zu halten.
- Aufbereitung und Vorwärmen (37 °C) Bildgebungsmedien: DMEM ergänzt mit HEPES-Na, pH 7,2 (10 mM), Natriumpyruvat (1 mM), L-Glutamin (2 mM) und Glukose (5 mM), ohne Phenolrot.
- Bilderfassung
HINWEIS: Das Protokoll beschreibt die multiparametrische Bildgebung von lebenden Sphäroiden mit dem konfokalen Mikroskop Stellaris 8 Falcon (Leica) und der Leica Application Suite X (LAS X) Software Version 4.7. Es sind jedoch nur geringfügige Modifikationen erforderlich, um eine solche Analyse auf alternativen Mikroskopieplattformen durchzuführen.- Schalten Sie das Temperatursteuergerät 30-60 Minuten vor der Aufnahme ein. Stellen Sie die erforderliche Belüftungsgeschwindigkeit und Temperatur (35-37 °C) ein.
HINWEIS: Wenn zusätzlich zur Temperaturregelung die Gaskonzentration (z. B. CO2 oderO 2) während der Bildgebung kontrolliert werden muss, sollten die entsprechenden Geräte ebenfalls im Voraus gestartet werden, um die erforderlichen Bedingungen vor der Bildgebung zu erreichen. - Schalten Sie das Mikroskop und die angeschlossenen Geräte ein (z. B. WLL-Laser, Computer, Wasserpumpe für das Wasserimmersionsobjektiv und andere elektronische Betriebsblöcke). Starten Sie die Mikroskopsteuerungssoftware (z. B. LAS X Machine Mode oder Machine Mode with Environmental Control), die mit der genauen Mikroskopeinrichtung geliefert wird, und initialisieren Sie die Tischkalibrierung.
- Wählen Sie das gewünschte Objektiv in der Software aus und tragen Sie bei Bedarf die Immersionsflüssigkeit auf.
HINWEIS: Für die Live-Mikroskopie wird empfohlen, entweder Wasser- oder Glycerin-Immersionsobjektive mit ausreichendem ("langem") Arbeitsabstand zu verwenden, z. B. HC Fluotar L 25x/0,95 W VIS IR (2,4 mm Arbeitsabstand), HC PL Apo 40x/1,25 GLYC (0,35 mm Arbeitsabstand) oder mindestens NA = 0,4 oder höher für die Luftobjektive. Die Wahl der Vergrößerung und des Arbeitsabstands hängt von der Art und Größe der abgebildeten Probe und den gemessenen Fluoreszenzsignalen ab (Helligkeit, Quantenausbeute, Färbeeffizienz, siehe z.B. Diskussion über Farbstoffe und Nanopartikel53). Große Objekte (Sphäroide oder Organoide, >500 μm Größe), "Bioreaktor"- oder Mikrofluidik-Chips erfordern Objektive mit großem Arbeitsabstand und eine geringere Vergrößerung, während die Analyse einzelner Zellen oder Zellorganellen eine hohe Vergrößerung erfordert, die häufig durch "Mosaik"-Bildgebung erreicht wird. - Stellen Sie die Mikroskopieschale mit Sphäroiden auf dem Tisch auf. Passen Sie den Fokus an und suchen Sie ein Objekt/einen Bereich of Interest (ROI).
HINWEIS: Wenn kleine, schwach fluoreszierende, kontrastarme oder seltene Objekte gefunden werden müssen und es schwierig ist, den Fokus zu finden, wird empfohlen, auf die Wände der Mikroskopieschüssel vorzufokussieren und die Oberfläche mit Serpentin von einer der Ecken der Vertiefung aus auf das Objekt des Interesses zu "screenen". - Wählen Sie das Fenster Projekt öffnen und klicken Sie auf das entsprechende Symbol Neues Projekt erstellen. Geben Sie der Forschungsprojektdatei einen Standardnamen (z. B. beginnend mit "JJ-MM-DD+ Beschreibung"). Während des Imagings werden alle produzierten Bilder automatisch in der erstellten .lif-Projektdatei gespeichert.
- Öffnen Sie das Fenster Erfassung . Stellen Sie die Anregungswellenlänge des Weißlichtlasers (WLL) und den erforderlichen Bereich von Hybrid- oder Resonanzdetektoren (Typ HyD S, HyD X oder HyD R) basierend auf den bekannten spektralen Eigenschaften der gemessenen Fluoreszenz (Anregungs-/Absorptions- und Emissionsspektren) ein. Wählen Sie die Scantypen Linie oder Rahmen aus.
HINWEIS: Bei den meisten kommerziell erhältlichen Fluoreszenzfarbstoffen sind die spektralen Eigenschaften im LAS X Dye Assistant-Paket zu finden (oder hinzugefügt). Wählen Sie den Detektor mit dem geeigneten Spektrenempfindlichkeitsbereich, der mit den spektralen Eigenschaften der Sonde und im Falle von FLIM mit der Photonenzählung kompatibel ist (d. h. HyD X oder HyD R). Für die multiparametrische Bildgebung stellen Sie die WLL in mehrere Anregungspositionen ein (z. B. für die gleichzeitige Abbildung von FAD/Flavinen und zwei Fluoreszenzkanälen der ratiometrischen MMIR1 O2-Sonde - Referenz und Empfindlichkeit können die Anregungs-/Emissionseinstellungen 460 nm/510-590 nm HyD X1 und 614 nm/631-690 HyD X3 und 724-800 nm HyD R entsprechend in einer oder zwei aufeinanderfolgenden Scansequenzen betragen). Es ist wichtig, den geeigneten Detektor zum Sammeln der Emission zuzuweisen, da Detektoren unterschiedliche spektrale Empfindlichkeitenaufweisen können 54. - (Optional für FLIM) Wählen Sie im Erfassungsfenster den FLIM-Modus , um die Bildgebung in Kombination mit der Photonenzählung (Zerfallssammlung) durchzuführen. Sofort wird ein zusätzliches "FLIM-Modul in der LAS X-Software" geöffnet, um FLIM-Daten zu navigieren und zu analysieren.
- (Optional für FLIM) Wählen Sie die WLL-Pulswiederholrate basierend auf der erwarteten durchschnittlichen Lebensdauer des Fluorophors.
HINWEIS: Die Frequenz des Laserpulses muss so eingestellt werden, dass der vollständige Fluoreszenzabfall erfasst wird. Dies kann mit einer Puls-Picker-Funktion erfolgen, die auf dem Mikroskop installiert ist. Die Überlappung des Fluoreszenzzerfalls mit dem Laserpuls führt zu einer Verkürzung der geschätzten Fluoreszenzlebensdauer. Es wird empfohlen, Pulsintervalle zu haben, die 4-5 Mal länger sind als die erwartete durchschnittliche Fluoreszenzlebensdauer (z. B. 25 ns/40 MHz für Lebensdauern bis zu 5 ns). Viele gepulste Laser haben eine feste Wiederholrate von 80 MHz (ideal nur für einen Bereich von bis zu 2-3 ns). Dies ist wichtig für die Auswahl der richtigen Fluorophore für das Experiment. - Starten Sie die Vorschaubildgebung im FAST LIVE-Modus und stellen Sie den Feinfokus des Bildobjekts auf einen interessanten Bereich ein.
ACHTUNG: Befolgen Sie strikt die Lasersicherheitsregeln. Beachten Sie immer die Sicherheitsregeln für Laser und warten Sie, bis die Bildgebung gestoppt ist, bevor Sie das Transmissionslicht einschalten, in das Okular oder auf die Probe schauen.
HINWEIS: Im FAST LIVE-Modus wird ein High-Speed-Scan von 600 Hz (entspricht einer maximalen Bildrate von 4,43/s im bidirektionalen X-Scan-Modus) mit einer Auflösung von 256 x 256 Pixeln automatisch auf das Bild angewendet, um die Fluoreszenz vor Photobleaching zu schützen. Öffnen Sie die Lochblende (z. B. auf 3-4 AE) und/oder erhöhen Sie die Laserintensität, wenn das Fluoreszenzsignal zu schwach ist, um auf das Objekt zu fokussieren. Vermeiden Sie eine unvollständige Fäulnissammlung. - (Optional für FLIM) Wenn Sie sich ein Pixelintensitätshistogramm ansehen, das während der Bildgebung in einem FLIM-Modulfenster (Live-Modus ) angezeigt wird, passen Sie die entsprechende Laserintensität/Lochblendengröße und -auflösung an, um die Zählrate von ~1 Photon/Laserpulsgrenze (rote Linie) zu erreichen. Vermeiden Sie es, deutlich höher als 1 zu gehen, um das Risiko eines Pile-up-Effekts auszuschließen. Passen Sie bei Bedarf die WLL-Impulswiederholrate an, um eine vollständige Decay-Sammlung in einem Decay-Fenster zu erhalten (um eine unvollständige Decay-Sammlung zu vermeiden, siehe Schritt 2.2.8).
HINWEIS: Wenn die Anzahl der Photonen (Intensität) nicht ausreicht, um einen zuverlässigen Zerfall für die Anpassungsanalyse oder die Phasorplot-Wolke zu rekonstruieren, wenden Sie mehrere Scan-Wiederholungen (Frames oder Linien oder Setup-Scanzeit) an, erhöhen Sie die Laserintensität und/oder opfern Sie die Auflösung (gescannte ROI-Größe). Beachten Sie, dass für eine zuverlässige Zerfallsrekonstruktion mehr Photonen gesammelt werden müssen, um den Zerfall zu rekonstruieren, und dass eine zusätzliche Korrektur der Bildgebungsparameter erforderlich sein kann. Seien Sie sich der möglichen Auswirkungen von intensivem Licht und langer kontinuierlicher Beleuchtung auf die Lebensfähigkeit und den Stoffwechsel der Zellenbewusst 55. Die negativen Auswirkungen auf die Lebensfähigkeit und den Stoffwechsel können in jedem Einzelfall unterschiedlich sein, abhängig von der Intensität, Dauer und Wellenlänge des Anregungslichts sowie der Bildgebungsmodalität (z. B. konfokale Ein-Photonen- vs. Multiphotonen-Bildgebung). Passen Sie die Bildgebungsparameter entsprechend an und kontrollieren Sie gegebenenfalls die Zellviabilität/den Zelltod durch Calcein Green-AM oder die Propidiumiodidintensität in Pilotversuchen56. Wo immer möglich, sollten weitere Optimierungen des Färbeprotokolls für Fluoreszenzsonden in Betracht gezogen werden, um ein adäquates Fluoreszenzsignal während der Live-Mikroskopie zu erreichen. - (Optional für 3D-Z-Stack) Legen Sie in Fast Live die Koordinaten und die Scanrichtung fest und ordnen Sie sie im Z-Stapel-Fenster (XYZ-Scan-Modus) Anfang und Ende zu. Wählen Sie die Größe des Z-Schritts oder die Anzahl der Schritte aus.
HINWEIS: Während die Software automatisch die "optimale" Anzahl von Schritten berechnet, basierend auf der verwendeten Auflösung und den Scanparametern, kann die Live-3D-Rekonstruktion normalerweise eine geringere Anzahl von Schritten erfordern, um eine schnelle Erfassung zu erreichen, z. B. Schrittweite von 1-2 μm, Stack-Größe von 50-100 μm, bidirektionales Scannen, das 2-3 Minuten der gesamten Scanzeit benötigt. Beachten Sie, dass sich die subzellulären Organellen, Zellen und das 3D-Zellmodell während der Messungen ebenfalls bewegen können. Darüber hinaus kann aufgrund der Lichteindringtiefe und der Streugrenzen typischerweise nur eine Scantiefe von 50-100 μm bei konfokalem FLIM erreicht werden. - Wenn alle erforderlichen Einstellungen übernommen wurden, starten Sie das Imaging.
- Geben Sie dem Bild einen passenden Namen.
- Suchen Sie im Transmissionslichtmodus nach dem nächsten Bildobjekt und wiederholen Sie den Bildvorgang mit zuvor optimierten Bildeinstellungen (Schritte 2.2.8-2.2.12).
HINWEIS: Behalten Sie für den intensitätsbasierten Vergleich oder die Analyse des Intensitätsverhältnisses (z. B. MMIR O 2-Sonden-basierte Oxygenierungsanalyse) immer die gleichen Bildgebungseinstellungen für alle analysierten Objekte bei (Vergrößerung und Objektivtyp, Laserintensität, Leistung und Pulsfrequenz, Anregungswellenlänge, Detektorbereich, Lochblende, Scangeschwindigkeit, Pixelverweilzeit und Auflösung). Da die Fluoreszenzlebensdauer jedoch nicht von der Fluoreszenzintensität abhängt und eine angemessene Anzahl von Photonen für eine zuverlässige Berechnung gesammelt werden muss, können die FLIM-Bildgebungsparameter im Laufe des Experiments neu angepasst werden, um die gesammelten Photonenzahlen zwischen verschiedenen Behandlungen oder experimentellen Bedingungen vergleichbar zu halten. Daher müssen für die multiparametrische Analyse, bei der sowohl eine intensitätsbasierte als auch eine auf der Fluoreszenzlebensdauer basierende Analyse erforderlich sind, optimierte universelle Bildgebungseinstellungen für alle Objekte in den verglichenen Versuchsgruppen angewendet werden. Für den "FLIM-only"-Vergleich ist es möglich, Bilder zu vergleichen, die mit leicht unterschiedlichen Bildeinstellungen aufgenommen wurden, da die LAS X-Software die Berechnung des IRF für die Einzelbildmessungermöglicht 42. Für FLIM-Anpassungsanalysen außerhalb von LAS X (z. B. FLIMfit45) sollte die Instrumentenreaktionsfunktion (IRF) jedoch für jede andere Bildgebungsbedingung gemessen werden, da sie nicht aus der Bildgebungssoftware exportiert werden kann. Aus Gründen der Vereinfachung des Versuchsdesigns und der Arbeitslast wird daher empfohlen, die gleichen Bildgebungseinstellungen für alle Bilder im Datensatz anzuwenden. Anschließend können die entsprechenden IRF-Messungen unter Verwendung von gequenchierten oder schnell fluoreszierenden Lebensdauerfluorophoren (innerhalb des ps-Bereichs) mit den Emissionseigenschaften des gemessenen Spektralkanals57,58,59, durch die Lumineszenz60 von Goldnanopartikeln oder durch ein Generationssignal der zweiten Harmonischen für das Multiphoton FLIM61 durchgeführt werden. In der LAS X-Software können die zuvor optimierten Imaging-Parameter für ein neues Projekt geladen werden, indem Sie mit der rechten Maustaste auf die gewünschte Datei klicken und Bildeinstellungen anwenden auswählen. - Wenn die Imaging-Sitzung abgeschlossen ist, speichern Sie das Imaging-Projekt. Um die Bildgebungssitzung abzuschließen, nehmen Sie die Probe aus dem Mikroskopietisch und reinigen Sie das Objektiv von der Tauchflüssigkeit (falls verwendet) gemäß dem in der Bildgebungseinrichtung implementierten Standardverfahren. Schließen Sie das Projekt und die Software. Schalten Sie das Mikroskop, die Laser und alle angeschlossenen Geräte aus.
- Fahren Sie mit der Analyse der Bilddaten fort (Schritt 2.3).
- Schalten Sie das Temperatursteuergerät 30-60 Minuten vor der Aufnahme ein. Stellen Sie die erforderliche Belüftungsgeschwindigkeit und Temperatur (35-37 °C) ein.
- NAD(P)H/FAD-FLIM Phasen-Bildverarbeitung mit LAS X FLIM Modul und FIJI
HINWEIS: Das Protokoll beschreibt eine Fluoreszenzlebensdaueranalyse von abgebildeten Sphäroiden für Frequenzbereichsdaten an Beispielen von NAD(P)H und FAD/Flavinen Autofluoreszenz FLIM. Die NAD(P)H-Autofluoreszenzmessung wurde zu einem Goldstandard für die Stoffwechselanalyse, bei der kurze und längere NAD(P)H-Autofluoreszenzlebensdauerkomponenten mit Glykolyse bzw. oxidativer Phosphorylierung (OxPhos) assoziiert sind. Dies kann durch die Verschiebung in einem Phasordiagramm entlang der metabolischen Trajektorie in Richtung der gemessenen Standards von freiem NAD(P)H oder proteingebundenem NAD(P)H analysiert werden31,62. Um den Verlauf der metabolischen Verschiebung zu analysieren und die Position der Phasorwolken (siehe Anmerkung unten in Schritt 2.3.6) auf einem Diagramm zwischen Versuchsgruppen zu vergleichen, wurde eine vereinfachte Phasorkoordinatenanalyse durchgeführt, die auf der Berechnung des geometrischen Zentrums (Schwerpunkt) der Phasorwolke basiert29. Das beschriebene Protokoll demonstriert die Berechnung von Schwerpunktkoordinaten in Fidschi und die Messung des Abstands zwischen zwei Punkten in einem Phasordiagramm unter Verwendung von Koordinaten (z.B. den Abstand von einem Schwerpunkt der sphäroiden NAD(P)H-Autofluoreszenz-Phasorwolke zu einem "freien NAD(P)H"-Punkt). In ähnlicher Weise können FAD und andere Autofluoreszenzsignale analysiert werden. Ein Datensatz 1 mit .lif (LAS X-Software erforderlich) oder .ptu Dateiformate zum Erlernen dieses Verfahrens zur Verfügung gestellt (Zusatzakte 2, Zusatzakte 3, Zusatzakte 4, Zusatzakte 5, Zusatzdatei 6, Zusatzakte 7, Zusatzakte 8, Zusatzakte 9, und Zusatzdatei 10).- Öffnen Sie das FLIM-Modul in LAS X, wählen Sie Projekt öffnen und laden Sie die Sphäroidbilddatei (.lif) für die Autofluoreszenz-NAD(P)H/FAD-Analyse.
HINWEIS: Verwenden Sie aufgrund möglicher Fehler und vorübergehender Datenverluste im FLIM-Modul eine Kopie der ursprünglichen Sphäroid-Bilddatei (.lif) für die NAD(P)H/FAD-Analyse, wobei Sie die Rohdatei unverändert lassen. - Wählen Sie ein einzelnes Bild aus und navigieren Sie zur FLIM-Analyseoberfläche. Klicken Sie auf Phasor, um auf das Phasordiagramm zuzugreifen und den Phasoranalysemodus zu aktivieren. Wenden Sie den Filter (Median oder Wavelet) an und legen Sie den Schwellenwert fest, um das Rauschen zu minimieren und die Datensichtbarkeit für alle Phasoranalysen zu verbessern. Wählen Sie die Oberschwingungen. Für die Analyse der metabolischen Verschiebung auf der Grundlage von NAD(P)H-Daten fahren Sie mit den Schritten 2.3.3-2.3.5 fort. Für den allgemeinen Vergleich von Phasordiagrammen gehen Sie von Schritt 2.3.6 aus.
HINWEIS: Wenden Sie die Analyseeinstellungen (Filtertyp, Oberschwingungen, Schwellenwerte, Binning und Phasor-ROIs) gleichermaßen auf alle Bilder in einem verglichenen Datensatz an. - (Optional für die NAD(P)H-Analyse) Wählen Sie ein beliebiges datasetbezogenes Bild aus, und verwenden Sie die Option Zeichner-Verhältnis-Cursor für zwei Komponenten , um die Position von 0,45 ns auf einem universellen Kreis eines Standard-Phasor-Plotraums genau zu lokalisieren. Diese Position wird einer durchschnittlichen Fluoreszenzlebensdauer einer reinen homogenen Lösung von freiem NAD(P)H zugeordnet, die normalerweise nahe dem monoexponentiellen Zerfall62 liegt.
HINWEIS: Freies NADH und freies NAD(P)H haben ähnliche spektrale Eigenschaften und ähnliche Werte der Fluoreszenzlebensdauer in wässriger Lösung, mit zwei Komponenten mit kurzer Lebensdauer, 0,3 ns und 0,7 ns63. Aus Gründen der Einfachheit der phasorbasierten Analyse und aufgrund eines geringen Unterschieds zwischen den Lebensdauerkomponenten wird daher angenommen, dass ihr Fluoreszenzzerfall mono-exponentiell ist, was eine Zuordnung der Phasorwolke zu einem universellen Kreis ermöglicht. Die durchschnittliche Referenzlebensdauer einer freien NAD(P)H-Form kann für eine ähnliche Analyse auch gemessen und in einem Phasorraum aufgetragen werden. Die Referenzlebensdauer wurde auf der Grundlage der Literaturgewählt 62; Es ist zu beachten, dass in anderen Quellen ein leicht abweichender Wert eines freien NAD(P)H in Lösung gefunden werden kann (0,4 ns64). - (Optional für die NAD(P)H-Analyse) Exportieren Sie das Phasordiagramm mit der zugewiesenen freien NAD(P)H-Lebensdauer (siehe Schritt 2.3.3), indem Sie mit der rechten Maustaste auf das Diagramm klicken und Daten exportieren auswählen. Exportieren Sie das Phasordiagramm als Datei im .tiff Format in einen bestimmten Speicherordner.
HINWEIS: Die ursprüngliche Pixelgröße des exportierten Phasorplot-Bildes aus dem LAS X FLIM-Modul beträgt immer 1024 x 600 Pixel. Wenn eine andere Software für den Datenexport und die Voranalyse verwendet wird, stellen Sie sicher, dass alle Phasor-Plot-Bilder mit der gleichen Größe und Auflösung exportiert werden. - Um Sphäroid-bezogene Phasorwolken zu exportieren, verwenden Sie das Werkzeug Cursor zeichnen im LAS X FLIM-Modul, um den Sphäroid-ROI auf dem Bild auszuwählen. Exportieren Sie das generierte Phasordiagramm, wie in Schritt 2.3.4 beschrieben.
ANMERKUNG: Die entsprechenden g- und s-Koordinaten (ähnlich wie x und y) des Phasorraums werden jedem Pixel des gewählten ROI entsprechend ihrer Lebensdauer zugewiesen und in einen Frequenzbereichsdatensatz64,65 transformiert. Der Cluster von Pixeln mit ähnlichen Werten von tf (Phasenlebensdauer) und tm (Modulationslebensdauer) bildet ein Wolkenmuster (Phasorwolke) in einem Diagramm, in dem die Farbcodierung (mit einem Bereich von tiefblau bis rot) die Häufigkeit der Lebensdauerwerte widerspiegelt. Durch die Position der Wolke auf einem universellen Kreis oder innerhalb des Himmels können die mono- oder multiexponentiellen Zerfälle unterschieden werden. Einige Messmedien weisen eine starke (Auto-)Fluoreszenz auf, was zum Auftreten einer entsprechenden Wolke auf einem Phasordiagramm führt, die nicht einfach mit Hilfe der Intensitätsschwelle entfernt werden kann. Dieses Muster beeinflusst die Berechnung der Schwerpunktkoordinaten und muss aus dem exportierten Phasordiagramm ausgeschlossen werden. Die Arbeit mit dem Sphäroid-ROI ermöglicht den Ausschluss der nicht verwandten Pixel von der weiteren Phasoranalyse. - Wiederholen Sie den Exportvorgang für das Phasordiagramm für alle Sphäroid-ROIs in einem Datensatz (siehe Schritte 2.3.4 und 2.3.6). Überprüfen Sie außerdem den exportierten Satz von .tiff Bildern, um sicherzustellen, dass der vollständige Datensatz für weitere vergleichende Analysen vorhanden ist, und stellen Sie sicher, dass alle exportierten Bilder die gleiche Pixelgröße haben (siehe HINWEIS in Schritt 2.3.4).
HINWEIS: In dieser Phase des Protokolls muss der Bildsatz einen Phasor mit der Position des freien NAD(P)H (basierend auf Literatur oder empirisch erhaltenen Daten) und alle Phasordiagramme des Sphäroid-ROI (oder alternativer ROI-Muster, falls für die spezifische Analyse erforderlich) enthalten. Von diesem Schritt aus erfolgt die weitere Analyse in Fidschi und anschließend in einer Tabelle. Stellen Sie im Werkzeugfenster "Analysieren " die Option "Maßstab festlegen " in FIJI sicher, dass alle Phasordiagrammbilder mit demselben Einheitentyp kalibriert sind, z. B. nur in Pixeln. Wenn nicht, legen Sie die Längeneinheit im Fenster "Maßstab festlegen " für die ausgewählte Einheit fest (geben Sie z. B. für den pixelbasierten Maßstab 1 in das Feld "Entfernung in Pixel" ein und setzen Sie die Längeneinheit auf Pixel). Messen Sie zum weiteren Vergleich alle exportierten Daten mit demselben Einheitentyp. - (Optional für die NAD(P)H-Analyse) Bestimmen Sie die Pixelpunktposition einer durchschnittlichen Fluoreszenzlebensdauer von freiem NAD(P)H auf dem entsprechenden exportierten Phasordiagramm (siehe Schritt 2.3.3): Öffnen Sie das Phasorbild mit FIJI, vergrößern Sie das Bild, um den Schnittpunkt zwischen dem universellen Halbkreis und der Linie Verhältniscursor für zwei Komponenten mit Pixelauflösung deutlich zu visualisieren; Verwenden Sie das Werkzeug Rechteck-ROI , um den Schnittpunkt auszuwählen.
HINWEIS: Stellen Sie sicher, dass die rechteckige Auswahl ein kleiner Bereich um den Schnittpunkt ist, um die Koordinaten im nächsten Schritt (Schritt 2.3.9) genau bestimmen zu können. - (Optional für die NAD(P)H-Analyse) Öffnen Sie das Analysewerkzeug, wählen Sie das Fenster Messwerte festlegen und wählen Sie Schwerpunkte als Messparameter aus. Klicken Sie im Werkzeugfenster Analysieren auf Messen, um die Schwerpunktkoordinaten des freien NAD(P)H-Referenzpunkts zu bestimmen. Exportieren Sie diese Koordinaten in eine Tabelle.
HINWEIS: Freie NAD(P)H-Koordinaten werden als Referenzpunkt verwendet, um die Entfernungen von diesem Punkt zur Position der Sphäroid-Phasorwolke in einem Datensatz zu vergleichen (die Art und Weise, wie die metabolische Verschiebung zwischen Glykolyse und OxPhos in der NAD(P)H FLIM-Autofluoreszenzanalyse charakterisiert wird) - Öffnen Sie mit FIJI das Bild der Sphäroid-Phasorwolke. Öffnen Sie das Bildfenster-Werkzeug, wählen Sie "Anpassen" und wählen Sie in der Symbolleiste "Farbschwellenwert" aus. Wählen Sie die Schwellenwertmethode Ihrer Wahl (z. B. Otsu) und legen Sie den Farbtonwert und den Helligkeitswert fest, um die Parameter für die Auswahl eines Phasorwolkenteils mit den häufigsten Pixelkoordinaten einzuschränken. Klicken Sie auf Auswählen , um den Clusterbereich zu definieren.
HINWEIS: Behalten Sie die gleichen Schwellenwertparameter für alle Phasor-Plot-Bilder bei, die analysiert werden müssen. Für die vorgestellten NAD(P)H-FLIM-Daten wurde die Schwellenwertmethode Otsu mit eingestelltem Farbtonwert 9 und Helligkeitswert 160 gewählt und auf alle Phasorbilder angewendet. Alternativ kann der ausgewählte Bereich in den ROI-Manager kopiert werden (folgen Sie dem Pfad Bearbeiten > Auswahl > Zum Manager hinzufügen), um eine Bibliothek mit Phasor-ROIs für die weitere Analyse zu erstellen. - Berechnen Sie unter Beibehaltung der Auswahl die Schwerpunktkoordinaten des ausgewählten Bereichs nach dem in Schritt 2.3.9 beschriebenen Verfahren. Exportieren Sie diese Koordinaten in die Tabellenkalkulationsdatei.
- Wiederholen Sie die Schritte 2.3.9 und 2.3.10, um die Schwerpunktkoordinaten für alle ROI-Phasorbilder zu ermitteln und einen Datensatz in der Tabelle zu erstellen.
HINWEIS: Die Verwendung der ROI-Bibliothek des ROI-Managers hilft bei der Vereinfachung und Organisation der ROI-Analyse (siehe Schritt 2.3.10) - (Optional für die NAD(P)H-Analyse) Öffnen Sie die Tabelle mit exportierten Referenz-ROI-Koordinaten für freie NAD(P)H und Sphäroide aus verschiedenen Vergleichsgruppen. Berechnen Sie den Abstand zwischen jedem einzelnen Sphäroid-Phasorschwerpunkt zur Referenz der freien NAD(P)H-Position unter Verwendung der ermittelten Koordinaten und der folgenden Gleichung:
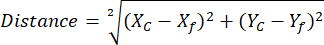
Dabei sind Xc und Yc Schwerpunktkoordinaten, Xf und Yf die Referenzkoordinaten.
HINWEIS: Die Anwendung des Schwerpunktparameters zur Bestimmung der Verschiebung in Richtung der Referenzlebensdauer ist nur dann geeignet, wenn alle Schwerpunkte aus einem Datensatz auf derselben linearen Trajektorie in Richtung des Referenzpunkts liegen. Um dies zu überprüfen, müssen alle Schwerpunktpunkte aus dem Datensatz zusammen mit dem Referenzpunkt im gleichen Koordinatenraum aufgetragen und die lineare Trendausrichtung durchgeführt werden. Wenn der R2-Koeffizient der linearen Trendlinie, die durch alle Punkte gezogen wird, nahe 1 liegt (z. B. R2 ist 0,8-0,99), wird davon ausgegangen, dass die Entfernungsanalyse angemessen ist. - Organisieren Sie alle Daten entsprechend für den Vergleich und führen Sie statistische Analysen mit Hilfe einer entsprechenden Software (z. B. Origin, MatLab) durch. Wählen Sie den geeigneten statistischen Test entsprechend den Merkmalen des Datensatzes (Verteilungsnormalverteilung, Anzahl der statistischen Einheiten usw.).
HINWEIS: Vergleichen Sie für die NAD(P)H-Analyse die Entfernungswerte, um die metabolische Verschiebung in Abhängigkeit von den experimentellen Bedingungen zu charakterisieren. Um Phasordiagramme zwischen experimentellen Gruppen zu vergleichen, führen Sie den Vergleich der ROI-Koordinaten der Phasorwolkenschwerpunkte durch.
- Öffnen Sie das FLIM-Modul in LAS X, wählen Sie Projekt öffnen und laden Sie die Sphäroidbilddatei (.lif) für die Autofluoreszenz-NAD(P)H/FAD-Analyse.
Ergebnisse
Auswahl der geeigneten Methode zur Bildung von Sphäroiden
Die gewählte Methode zur Bildung von Sphäroiden kann die Größe, Form, Zelldichte, Lebensfähigkeit und Empfindlichkeit von Sphäroiden stark beeinflussen (Abbildung 2). Zuvor wurden die Auswirkungen mehrerer Methoden mit hohem Durchsatz (SphericalPlate 5D, im Labor hergestellte Mikroformen und MicroTissue-Formen) und Methoden mit geringem Durchsatz (Biofloat- und Lipidure-beschichtete 96-Well-Platten) auf die ...
Diskussion
Multizelluläre Sphäroide werden zu einer Methode der Wahl bei der Untersuchung von Tumor- und Stammzell-Nischenmikroumgebungen, der Arzneimittelforschung und der Entwicklung der "Gewebebausteine" für die Biofabrikation. Die heterogene innere Architektur der Sphäroide, die Gradienten der Nährstoffe und die Sauerstoffversorgung können die von in vivo-Geweben und Tumoren in einer relativ vereinfachten und zugänglichen Umgebung nachahmen. Mit dem Bedarf an mehr methodischer Transparenz...
Offenlegungen
Nichts offenzulegen.
Danksagungen
Diese Arbeit wurde durch die Stipendien des Sonderforschungsfonds (BOF) der Universität Gent (BOF/STA/202009/003; BOF/IOP/2022/058), der Forschungsstiftung Flandern (FWO, I001922N) und der Europäischen Union, fliMAGIN3D-DN Horizon Europe-MSCA-DN Nr. 101073507.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA | Gibco | 25300-054 | Also available from Sigma |
| 10 mL serological pipets | VWR | 612-3700 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 12 well cell-culture plates, sterile | Greiner bio-one | 665-180 | Similar products are also available from Sarstedt, Corning and other companies. |
| 12 Well Chamber slide, removable | Ibidi | 81201 | Also available from Grace Bio-Labs, ThermoFisher Scientific and others |
| 15 mL centrifuge tubes | Nerbe plus | 02-502-3001 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 3D Petri Dish micromolds | Microtissue | Z764000-6EA | |
| 6 well cell-culture plates, sterile | Greiner bio-one | 657160 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 70% ethanol | ChemLab | CL02.0537.5000 | |
| Biofloat | Sarstedt | 83.3925.400 | Commercial available coated 96-well plate for spheroid formation |
| Calcein Green-AM | Tebubio | AS-89201 | Apply in dilution 1:1000 |
| CellSens Dimension software | Olympus | version 3 | |
| Collagen from human placenta, type IV | Sigma | C5533 | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Confocal FLIM Microscope | Leica Microsystems | N/A | Stellaris 8 Falcon inverted microscope with white-light laser, HyD X detectors, climate / T control chamber (OkoLab), 25x/0.95 W objective |
| D(+)-Glucose | Merck | 8342 | Prepare 1 M stock solution, 1:100 for preparation of imaging medium (final concentration 10 mM) |
| Dulbecco's modified Eagle's medium (DMEM), phenol red-, glucose-, pyruvate- and glutamine-free | Sigma-Aldrich | D5030-10X1L | For preparation of imaging medium |
| Fetal Bovine Serum (FBS) | Gibco | 10270-098 | Also available from Sigma. Needs to be heat-inactivated before use. |
| HEPES (1M) | Gibco | 15630-080 | Dilution 1/100 for preparation of imaging medium (final concentration 10 mM) |
| Human colon cancer cells HCT116 | ATCC | ||
| ImageJ | NIH | version 1.54f | |
| Leica Application Suite X (LAS X) | Leica Microsystems | version 4.6.1.27508 | |
| L-glutamine | Gibco | 25030 | Also available from Sigma. Apply in dilution 1:100. |
| Lipidure-CM5206 | Amsbio | AMS.52000034GB1G | |
| McCoy's 5A, need addition of 1 mM Sodium Pyruvate and 10 mM HEPES | VWR | 392-0420 | Standard growth medium for HCT116 cells |
| micro-patterned 3D-printed PDMS stamps | N/A | N/A | Provided by the Centre for Microsystems Technology, Professor Dr. Jan Vanfleteren, Ghent University |
| NaCl | Chemlab | CL00.1429.100 | |
| Neubauer couting chamber | Fisher Scientific | 15980396 | |
| O2 probes: MMIR1 | N/A | N/A | Full characterization, validation and some applications can be found at: https://www.biorxiv.org/content/10.1101/2023.12.11.571110 v1 |
| PBS | Fisher scientific | Gibco18912014 | Dissolve PBS tablet in 500 mL of distilled water. |
| Pen Strep :Penicillin (10,000 U/mL) / streptomycin (10,000 μg/mL) 100x solution | Gibco | 15140-122 | Also available from Sigma. Apply in dilution 1:100. |
| Poly-D-lysine | Sigma | P6407-5mg | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Propidium Iodide | Sigma-Aldrich | 25535-16-4 | Cell death staining, use 1 µg/mL at 1h incubation |
| PVDF syringe filter 0.22 µm | Novolab | A35149 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Sodium pyruvate (100 mM) | Gibco | 11360-070 | Dilution 1/100 for preparation of imaging medium (final concentration 1mM) |
| SphericalPlate 5D 24-well | Kugelmeiers | SP5D-24W | |
| sterile petridish | Greiner bio-one | 633181 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (25 cm² ) | VWR | 734-2311 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (75 cm²) | VWR | 734-2313 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| U-bottom 96-well plate | VWR | 10062-900 | Similar products are also available from Sarstedt, Corning, Greiner Bio-one and other companies |
| Ultrapure Agarose | Invitrogen (Life Technologies) | 16500-500 | Other types of Agarose such as Agarose low melting point (A-9414, Sigma), Agarose for routine use (A-9539, Sigma) |
| Widefield fluorescence inverted microscope | Olympus | N/A | Inverted fluorescence microscope IX81, with motorised Z-axis control, CoolLED pE4000 (16 channels, 365-770 nm), ORCA-Flash4.0LT (Hamamatsu) cMOS camera, glass warming plate Okolab, CellSens Dimension v.3 software and air objectives 4x/0.13 UPlanFLN and 40x/0.6 LUCPlanFLN. (Optional, for high-resolution imaging) 60x/1.0 LUMPLFLN water |
Referenzen
- Foty, R. A simple hanging drop cell culture protocol for generation of 3d spheroids. J Vis Exp. (51), e2720 (2011).
- Moskovits, N., et al. Establishing 3-dimensional spheroids from patient-derived tumor samples and evaluating their sensitivity to drugs. J Vis Exp. (190), e64564 (2022).
- Griner, L. M., et al. Generation of high-throughput three-dimensional tumor spheroids for drug screening. J Vis Exp. (139), e57476 (2018).
- Qian, Y., Wei, X., Chen, K., Xu, M. Three-dimensional acoustic assembly device for mass manufacturing of cell spheroids. J Vis Exp. (200), e66078 (2023).
- He, H., et al. Dynamic formation of cellular aggregates of chondrocytes and mesenchymal stem cells in spinner flask. Cell Prolif. 52 (4), e12587 (2019).
- Perez, J. E., Nagle, I., Wilhelm, C. Magnetic molding of tumor spheroids: Emerging model for cancer screening. Biofabrication. 13 (1), 015018 (2020).
- Kingsley, D. M., et al. Laser-based 3d bioprinting for spatial and size control of tumor spheroids and embryoid bodies. Acta Biomater. 95, 357-370 (2019).
- Guillaume, O., et al. Hybrid spheroid microscaffolds as modular tissue units to build macro-tissue assemblies for tissue engineering. Acta Biomater. 165, 72-85 (2023).
- Danilevicius, P., et al. laser-made 3D microscaffolds for tissue spheroid encagement. Biointerphases. 10 (2), 021011 (2015).
- Jamieson, L. E., Harrison, D. J., Campbell, C. Chemical analysis of multicellular tumour spheroids. Analyst. 140 (12), 3910-3920 (2015).
- Dmitriev, R., Borisov, S., Jenkins, J., Papkovsky, D. Multiparametric imaging of tumor spheroids with ultra-bright and tunable nanoparticle O2 probes. SPIE BiOS. 9328, (2015).
- Zushin, P. -. J. H., Mukherjee, S., Wu, J. C. FDA Modernization Act 2.0: transitioning beyond animal models with human cells, organoids, and AI/ML-based approaches. J Clin Invest. 133 (21), e175824 (2023).
- Kim, D. -. S., et al. Robust enhancement of neural differentiation from human es and ips cells regardless of their innate difference in differentiation propensity. Stem Cell Rev Rep. 6 (2), 270-281 (2010).
- Hart, D., Gutiérrez, D. R., Biason-Lauber, A. Generation of a human ovarian granulosa cell model from induced pluripotent stem cells. bioRxiv. , 2022 (2022).
- Chiaradia, I., et al. Tissue morphology influences the temporal program of human brain organoid development. Cell Stem Cell. 30 (10), 1351-1367 (2023).
- Wagstaff, E. L., Ten Asbroek, A. L., Ten Brink, J. B., Jansonius, N. M., Bergen, A. A. An alternative approach to produce versatile retinal organoids with accelerated ganglion cell development. Sci Rep. 11 (1), 1101 (2021).
- Thavandiran, N., et al. Design and formulation of functional pluripotent stem cell-derived cardiac microtissues. Proc Natl Acad Sci U S A. 110 (49), E4698-E4707 (2013).
- Harrison, S. P., et al. Scalable production of tissue-like vascularized liver organoids from human pscs. Exp Mol Med. 55 (9), 2005-2024 (2023).
- Noguchi, T. -. A. K., Kurisaki, A. Formation of stomach tissue by organoid culture using mouse embryonic stem cells. Methods Mol Biol. 2017, 217-228 (2017).
- Takahashi, J., et al. Suspension culture in a rotating bioreactor for efficient generation of human intestinal organoids. Cell Reports Methods. 2 (11), 100337 (2022).
- Lagziel, S., Gottlieb, E., Shlomi, T. Mind your media. Nat Metab. 2 (12), 1369-1372 (2020).
- Das, V., Fürst, T., Gurská, S., Džubák, P., Hajdúch, M. Reproducibility of uniform spheroid formation in 384-well plates: The effect of medium evaporation. J Biomol Screen. 21 (9), 923-930 (2016).
- Bera, K., et al. Extracellular fluid viscosity enhances cell migration and cancer dissemination. Nature. 611 (7935), 365-373 (2022).
- Michl, J., Park, K. C., Swietach, P. Evidence-based guidelines for controlling ph in mammalian live-cell culture systems. Commun Biol. 2, 144 (2019).
- Okkelman, I. A., Vercruysse, C., Kondrashina, A. V., Borisov, S. M., Dmitriev, R. I. Affordable oxygen microscopy-assisted biofabrication of multicellular spheroids. J Vis Exp. (182), e63403 (2022).
- Peirsman, A., et al. Mispheroid: A knowledgebase and transparency tool for minimum information in spheroid identity. Nat Methods. 18 (11), 1294-1303 (2021).
- Raghavan, S., et al. Comparative analysis of tumor spheroid generation techniques for differential in vitro drug toxicity. Oncotarget. 7 (13), 16948 (2016).
- Froehlich, K., et al. Generation of multicellular breast cancer tumor spheroids: Comparison of different protocols. J Mammary Gland Biol Neoplasia. 21 (3-4), 89-98 (2016).
- Debruyne, A. C., et al. Live microscopy of multicellular spheroids with the multimodal near-infrared nanoparticles reveals differences in oxygenation gradients. ACS Nano. 18 (19), 12168-12186 (2024).
- Debruyne, A. C., Okkelman, I. A., Dmitriev, R. I. Balance between the cell viability and death in 3D. Semin Cell Dev Biol. 144, 55-66 (2023).
- Barroso, M., Monaghan, M. G., Niesner, R., Dmitriev, R. I. Probing organoid metabolism using fluorescence lifetime imaging microscopy (flim): The next frontier of drug discovery and disease understanding. Adv Drug Deliv Rev. 201, 115081 (2023).
- Becker, W. Fluorescence lifetime imaging-techniques and applications. J Microsc. 247 (2), 119-136 (2012).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. J Cell Sci. 134 (9), 1-17 (2021).
- Sarder, P., Maji, D., Achilefu, S. Molecular probes for fluorescence lifetime imaging. Bioconjug Chem. 26 (6), 963-974 (2015).
- Alfonso-Garcia, A., et al. Mesoscopic fluorescence lifetime imaging: Fundamental principles, clinical applications and future directions. J Biophotonics. 14 (6), e202000472 (2021).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: Fundamentals and advances in instrumentation, analysis, and applications. J Biomed Opt. 25 (7), 1-43 (2020).
- Yellen, G. Fueling thought: Management of glycolysis and oxidative phosphorylation in neuronal metabolism. J Cell Biol. 217 (7), 2235-2246 (2018).
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomark Med. 4 (2), 241-263 (2010).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of nad (p) h and fad. Antioxid Redox Signal. 30 (6), 875-889 (2019).
- Verma, A., et al. Fluorescence lifetime imaging for quantification of targeted drug delivery in varying tumor microenvironments. bioRxiv. , 2024 (2024).
- Smith, J. T., et al. In vivo quantitative fret small animal imaging: Intensity versus lifetime-based fret. Biophys Rep. 3 (2), 100110 (2023).
- Alvarez, L. A., et al. Application Note: SP8 Falcon: A novel concept in fluorescence lifetime imaging enabling video-rate confocal flim. Nat Methods. , (2019).
- Roberti, M. J., et al. TauSense: A fluorescence lifetime-based tool set for everyday imaging. Nat. Methods. , (2020).
- Auer, J. M. T., Murphy, L. C., Xiao, D., Li, D. U., Wheeler, A. P. Non-fitting flim-fret facilitates analysis of protein interactions in live zebrafish embryos. J Microsc. 291 (1), 43-56 (2023).
- Warren, S. C., et al. Rapid global fitting of large fluorescence lifetime imaging microscopy datasets. PLoS One. 8 (8), e70687 (2013).
- Gao, D., et al. Flimj: An open-source imagej toolkit for fluorescence lifetime image data analysis. PloS One. 15 (12), e0238327 (2020).
- Tullis, I. D. C., Ameer-Beg, S. M., Barber, P. R., Rankov, V., Vojnovic, B. Mapping femtosecond pulse front distortion and group velocity dispersion in multiphoton microscopy. Proc. SPIE 6089, Multiphoton Microscopy in the Biomedical Sciences VI. , 60890 (2006).
- Zhou, Y., et al. One-step derivation of functional mesenchymal stem cells from human pluripotent stem cells. Bio Protoc. 8 (22), e3080 (2018).
- Benton, G., Arnaoutova, I., George, J., Kleinman, H. K., Koblinski, J. Matrigel: From discovery and ECM mimicry to assays and models for cancer research. Adv Drug Deliv Rev. 79-80, 3-18 (2014).
- Badea, M. A., et al. Influence of matrigel on single-and multiple-spheroid cultures in breast cancer research. SLAS Discov. 24 (5), 563-578 (2019).
- Lang, S., Sharrard, R., Stark, M., Villette, J., Maitland, N. Prostate epithelial cell lines form spheroids with evidence of glandular differentiation in three-dimensional matrigel cultures. Br J Cancer. 85 (4), 590-599 (2001).
- Barra, J., et al. DMT1-dependent endosome-mitochondria interactions regulate mitochondrial iron translocation and metastatic outgrowth. Oncogene. 43 (9), 650-667 (2024).
- Dmitriev, R. I., Papkovsky, D. B. Intracellular probes for imaging oxygen concentration: How good are they. Methods Appl Fluoresc. 3 (3), 034001 (2015).
- Schweikhard, V., et al. Application Note: The power HyD family of detectors for confocal microscopy. Nat Methods. , (2020).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon flim microscopy. Sci Rep. 12 (1), 11938 (2022).
- Bush, P. G., Wokosin, D. L., Hall, A. C. Two-versus one photon excitation laser scanning microscopy: Critical importance of excitation wavelength. Front Biosci. 12, 2646-2657 (2007).
- Liu, M., et al. Instrument response standard in time-resolved fluorescence spectroscopy at visible wavelength: Quenched fluorescein sodium. Appl Spectrosc. 68 (5), 577-583 (2014).
- Szabelski, M., et al. Collisional quenching of erythrosine b as a potential reference dye for impulse response function evaluation. Appl Spectrosc. 63 (3), 363-368 (2009).
- Chib, R., et al. Standard reference for instrument response function in fluorescence lifetime measurements in visible and near infrared. Meas Sci Technol. 27 (2), 027001 (2015).
- Talbot, C. B., et al. Application of ultrafast gold luminescence to measuring the instrument response function for multispectral multiphoton fluorescence lifetime imaging. Opt Express. 19 (15), 13848-13861 (2011).
- Recording the instrument response function of a multiphoton flim system. Becker & Hickl Available from: https://www.becker-hickl.com/literature/application-notes/recording-the-instrument-response-function-of-a-multiphoton-flim-system/ (2007)
- Leben, R., Köhler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free nad (p) h fluorescence lifetime imaging. Int J Mol Sci. 20 (22), 5565 (2019).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using nadh and nadph autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Gottlieb, D., Asadipour, B., Kostina, P., Ung, T. P. L., Stringari, C. FLUTE: A python gui for interactive phasor analysis of flim data. Biol Imaging. 3, e21 (2023).
- Malacrida, L., Ranjit, S., Jameson, D. M., Gratton, E. The phasor plot: A universal circle to advance fluorescence lifetime analysis and interpretation. Annu Rev Biophys. 50, 575-593 (2021).
- Okkelman, I., Vandenberghe, W., Dmitriev, R. Role of preconditioning with oxygen and glucose deprivation in promoting differentiation of dental pulp stem cells in 3D culture. Mol Biol Cell. 34 (2), 1212 (2022).
- Park, J. H., et al. The effect of bmp-mimetic peptide tethering bioinks on the differentiation of dental pulp stem cells (DPSCs) in 3D bioprinted dental constructs. Biofabrication. 12 (3), 035029 (2020).
- Pașca, S. P. The rise of three-dimensional human brain cultures. Nature. 553 (7689), 437-445 (2018).
- Dmitriev, R. I., Zhdanov, A. V., Nolan, Y. M., Papkovsky, D. B. Imaging of neurosphere oxygenation with phosphorescent probes. Biomaterials. 34 (37), 9307-9317 (2013).
- Bryanskaya, E. O., et al. High levels of FAD autofluorescence indicate pathology preceding cell death. Biochim Biophys Acta Gen Subj. 1868 (1), 130520 (2024).
- Han, S. J., Kwon, S., Kim, K. S. Challenges of applying multicellular tumor spheroids in preclinical phase. Cancer Cell Int. 21 (1), 152 (2021).
- Okkelman, I. A., Neto, N., Papkovsky, D. B., Monaghan, M. G., Dmitriev, R. I. A deeper understanding of intestinal organoid metabolism revealed by combining fluorescence lifetime imaging microscopy (flim) and extracellular flux analyses. Redox Biol. 30, 101420 (2020).
- Gstraunthaler, G., Seppi, T., Pfaller, W. Impact of culture conditions, culture media volumes, and glucose content on metabolic properties of renal epithelial cell cultures: Are renal cells in tissue culture hypoxic. Cell Physiol Biochem. 9 (3), 150-172 (1999).
- Glickman, R. D. Phototoxicity to the retina: Mechanisms of damage. Int J Toxicol. 21 (6), 473-490 (2002).
- Golub, A. S., Pittman, R. N. Monitoring Parameters of Oxygen Transport to Cells in the Microcirculation. Quenched-Phosphorescence Detection of Molecular Oxygen: Applications in Life Sciences. , (2018).
- Nishigaki, T., Wood, C. D., Shiba, K., Baba, S. A., Darszon, A. Stroboscopic illumination using light-emitting diodes reduces phototoxicity in fluorescence cell imaging. Biotechniques. 41 (2), 191-197 (2006).
- Penjweini, R., Loew, H. G., Hamblin, M. R., Kratky, K. W. Long-term monitoring of live cell proliferation in presence of pvp-hypericin: A new strategy using ms pulses of led and the fluorescent dye cfse. J Microsc. 245 (1), 100-108 (2012).
- Carrasco Kind, M., et al. flimview: A software framework to handle, visualize and analyze flim data. F1000Research. 9, 574 (2020).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. Pam: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophys J. 114 (7), 1518-1528 (2018).
- Chen, S. -. J., Sinsuebphon, N., Barroso, M., Intes, X., Michalet, X. . Optical Molecular Probes, Imaging and Drug Delivery. , (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten